Chlamydomonas reinhardtii
| Chlamydomonas reinhardtii | |
|---|---|
 | |
| Clasificación científica | |
| Clado : | Viridiplantae |
| División: | Clorófita |
| Clase: | Clorofíceas |
| Orden: | Clamidomonadales |
| Familia: | Clamidomonadáceas |
| Género: | Clamidomonas |
| Especies: | C. reinhardtii |
| Nombre binomial | |
| Chlamydomonas reinhardtii | |
Chlamydomonas reinhardtii es un alga verde unicelular de unos 10 micrómetros de diámetro que nada con dos flagelos . Tiene una pared celular formada por glicoproteínas ricas en hidroxiprolina , un gran cloroplasto en forma de copa, un gran pirenoide y un aparato ocular que detecta la luz.
Las especies de Chlamydomonas están ampliamente distribuidas en todo el mundo en el suelo y el agua dulce, de las cuales Chlamydomonas reinhardtii es una de las más comunes y extendidas. [1] C. reinhardtii es un organismo modelo biológico especialmente bien estudiado, en parte debido a su facilidad de cultivo y la capacidad de manipular su genética. Cuando se ilumina, C. reinhardtii puede crecer fotoautotróficamente, pero también puede crecer en la oscuridad si se le suministra carbono orgánico. Comercialmente, C. reinhardtii es de interés para producir biofármacos y biocombustibles, además de ser una valiosa herramienta de investigación para producir hidrógeno .
Historia
La cepa de laboratorio de tipo salvaje C. reinhardtii c137 (mt+) se origina a partir de un aislado recolectado cerca de Amherst, Massachusetts , en 1945 por Gilbert M. Smith. [2] [3]
El nombre de la especie se ha escrito de varias maneras diferentes debido a las diferentes transliteraciones del nombre del ruso: reinhardi , reinhardii y reinhardtii se refieren a la misma especie, C. reinhardtii Dangeard. [4]
Descripción
Las células de Chlamydomonas reinhardtii son mayoritariamente esféricas, pero pueden ser elipsoidales, ovoides, obovoides o asimétricas. Tienen una longitud de entre 10 y 22 μm y un ancho de entre 8 y 22 μm. La pared celular es delgada y carece de papila. Los flagelos tienen una longitud de entre 1,5 y 2 veces la del cuerpo celular. Las células contienen un único cloroplasto en forma de copa que recubre la parte inferior de la célula, con un único pirenoide basal. [1]
Mancha en el ojo
C. reinhardtii tiene un aparato ocular similar al de los dinoflagelados . [5] La mancha ocular está ubicada cerca del ecuador celular. Está compuesta por una capa de gránulos ricos en carotenoides en el cloroplasto que actúan como un reflector de luz. [6] La función principal de la mancha ocular es la fototaxis , que consiste en el movimiento (con los flagelos ) relacionado con un estímulo luminoso. [7] La fototaxis es crucial para el alga y permite la localización del entorno con condiciones de luz óptimas para la fotosíntesis. [8] La fototaxis puede ser positiva o negativa dependiendo de la intensidad de la luz. [5] La vía fototáctica consta de cuatro pasos que conducen a un cambio en el equilibrio de batido entre los dos flagelos (el flagelo cis que es el más cercano a la mancha ocular, y el flagelo trans que es el más alejado de la mancha ocular). [7]
Organismo modelo

Chlamydomonas se utiliza como organismo modelo para la investigación de cuestiones fundamentales en biología celular y molecular como:
- ¿Cómo se mueven las células?
- ¿Cómo responden las células a la luz?
- ¿Cómo se reconocen las células entre sí?
- ¿Cómo generan las células formas de onda flagelares regulares y repetibles ?
- ¿Cómo regulan las células su proteoma para controlar la longitud del flagelar ?
- ¿Cómo responden las células a los cambios en la nutrición mineral? (nitrógeno, azufre, etc.)
Existen muchos mutantes conocidos de C. reinhardtii . Estos mutantes son herramientas útiles para estudiar una variedad de procesos biológicos, incluyendo la motilidad flagelar, la fotosíntesis y la síntesis de proteínas . Dado que las especies de Chlamydomonas normalmente son haploides, los efectos de las mutaciones se observan inmediatamente sin necesidad de más cruces.
En 2007, se publicó la secuencia completa del genoma nuclear de C. reinhardtii . [9]
La canalrodopsina -1 y la canalrodopsina -2, proteínas que funcionan como canales de cationes activados por luz , se aislaron originalmente de C. reinhardtii . [10] [11] Estas proteínas y otras similares se utilizan cada vez más en el campo de la optogenética . [12]
Importancia mitocondrial
El genoma de C. reinhardtii es importante para el estudio mitocondrial, ya que es una especie en la que los genes de 6 de las 13 proteínas codificadas para las mitocondrias se encuentran en el núcleo de la célula, dejando 7 en las mitocondrias. [13] En especies fuera de Chlorophyceae, estos genes están presentes solo en las mitocondrias y no pueden expresarse de forma alotópica. Esto es importante para la prueba y el desarrollo de terapias para enfermedades mitocondriales genéticas.
Reproducción
Las células vegetativas de las especies reinhardtii son haploides con 17 cromosomas pequeños . Bajo la privación de nitrógeno , las células vegetativas se diferencian en gametos haploides. [14] Hay dos tipos de apareamiento , idénticos en apariencia, por lo tanto isógamos , y conocidos como mt(+) y mt(-) , que pueden fusionarse para formar un cigoto diploide . El cigoto no está flagelado y sirve como una forma latente de la especie en el suelo. A la luz, el cigoto experimenta meiosis y libera cuatro células haploides flageladas que reanudan el ciclo de vida vegetativo.
En condiciones ideales de crecimiento, las células pueden experimentar a veces dos o tres rondas de mitosis antes de que las células hijas sean liberadas de la pared celular vieja al medio. Por lo tanto, un solo paso de crecimiento puede dar como resultado 4 u 8 células hijas por célula madre.
El ciclo celular de esta alga verde unicelular puede sincronizarse alternando períodos de luz y oscuridad. La fase de crecimiento depende de la luz, mientras que, a partir de un punto designado como punto de transición o de compromiso, los procesos son independientes de la luz. [15]
Genética
El atractivo de las algas como organismo modelo ha aumentado recientemente con la liberación de varios recursos genómicos al dominio público. El borrador Chlre3 de la secuencia del genoma nuclear de Chlamydomonas preparado por el Joint Genome Institute del Departamento de Energía de los EE. UU. comprende 1557 estructuras que suman 120 Mb. Aproximadamente la mitad del genoma está contenido en 24 estructuras, todas de al menos 1,6 Mb de longitud. El ensamblaje actual del genoma nuclear está disponible en línea. [16]
El genoma mitocondrial de ~15,8 Kb (número de acceso a la base de datos: NC_001638) está disponible en línea en la base de datos NCBI. [17] El genoma completo del cloroplasto de ~203,8 Kb (número de acceso a la base de datos: NC_005353) está disponible en línea. [18] [19]
Además de los datos de secuencia genómica, existe una gran cantidad de datos de secuencias de expresión disponibles como bibliotecas de ADNc y etiquetas de secuencia expresada (EST). Hay siete bibliotecas de ADNc disponibles en línea. [20] Se puede comprar una biblioteca BAC en el Instituto de Genómica de la Universidad de Clemson. [21] También hay dos bases de datos de >50 000 [22] y >160 000 [23] EST disponibles en línea.
Una colección de mutantes de todo el genoma con sitios de inserción mapeados que cubren la mayoría de los genes nucleares [24] [25] está disponible: https://www.chlamylibrary.org/.
Se ha demostrado que el genoma de C. reinhardtii contiene N 6 -Metiladenosina (m 6 A), una marca común en procariotas pero mucho más rara en eucariotas. [26] Algunas investigaciones han indicado que 6mA en Chlamydomonas puede estar involucrado en el posicionamiento de los nucleosomas, ya que está presente en las regiones de enlace entre nucleosomas, así como cerca de los sitios de inicio de la transcripción de genes transcritos activamente. [27]
C. reinhardtii parece ser capaz de realizar varios procesos de reparación del ADN . [28] Estos incluyen la reparación recombinatoria , la reparación por rotura de cadena y la reparación por escisión. En particular, los cloroplastos de C. reinhardtii poseen un sistema eficiente para reparar roturas de doble cadena de ADN . [29] En los cloroplastos, la reparación recombinatoria homóloga del ADN se ve fuertemente estimulada por las roturas de doble cadena. [29]
Evolución experimental
This section is missing information about "multicellular", doi:10.1038/s41598-019-39558-8. (March 2023) |
Chlamydomonas se ha utilizado para estudiar distintos aspectos de la biología evolutiva y la ecología. Es un organismo de elección para muchos experimentos de selección porque:
- Tiene un tiempo de generación corto,
- Es a la vez autótrofo y heterótrofo facultativo .
- Puede reproducirse tanto sexual como asexualmente, y
- Ya existe una gran cantidad de información genética disponible.
Algunos ejemplos (no exhaustivos) de trabajo evolutivo realizado con Chlamydomonas incluyen la evolución de la reproducción sexual, [30] el efecto de la aptitud de las mutaciones, [31] y el efecto de la adaptación a diferentes niveles de CO 2 . [32]
Según una hipótesis teórica frecuentemente citada, [33] la reproducción sexual (en contraste con la reproducción asexual) se mantiene de manera adaptativa en ambientes benignos porque reduce la carga mutacional al combinar mutaciones deletéreas de diferentes líneas de descendencia y aumenta la aptitud media. Sin embargo, en un estudio experimental a largo plazo de C. reinhardtii , se obtuvo evidencia que contradecía esta hipótesis. En poblaciones sexuales, no se encontró que ocurriera la eliminación de mutaciones y no se encontró que la aptitud aumentara. [34]
Movimiento
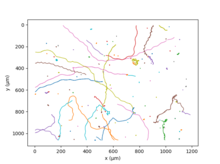
C. reinhardtii nada gracias a sus dos flagelos, [35] en un movimiento análogo al de la braza humana . Repitiendo este movimiento elemental 50 veces por segundo, el alga tiene una velocidad media de 70 μm/s; [36] la diversidad genética de las diferentes cepas da como resultado una enorme gama de valores para esta cantidad. Después de unos segundos de carrera, un batido asincrónico de los dos flagelos provoca un cambio aleatorio de dirección, un movimiento llamado " carrera y voltereta ". [35] A una escala temporal y espacial mayor, el movimiento aleatorio del alga puede describirse como un fenómeno de difusión activa. [37]
Técnicas de transformación del ADN
La transformación de genes se produce principalmente por recombinación homóloga en el cloroplasto y recombinación heteróloga en el núcleo. El genoma del cloroplasto de C. reinhardtii se puede transformar mediante bombardeo de partículas de microproyectiles o agitación con perlas de vidrio, sin embargo este último método es mucho menos eficiente. El genoma nuclear se ha transformado tanto con agitación con perlas de vidrio como con electroporación. El procedimiento biolístico parece ser la forma más eficiente de introducir ADN en el genoma del cloroplasto. Esto se debe probablemente a que el cloroplasto ocupa más de la mitad del volumen de la célula, lo que proporciona al microproyectil un objetivo grande. Se ha demostrado que la electroporación es la forma más eficiente de introducir ADN en el genoma nuclear con frecuencias máximas de transformación dos órdenes de magnitud más altas que las obtenidas mediante el método de perlas de vidrio. [ cita requerida ]
Usos prácticos
Producción de productos biofarmacéuticos
La C. reinhardtii modificada genéticamente se ha utilizado para producir una proteína amiloide sérica de mamíferos (requiere cita), una proteína de anticuerpo humano (requiere cita), un factor de crecimiento endotelial vascular humano , una posible vacuna terapéutica contra el virus del papiloma humano 16 , [38] una posible vacuna contra la malaria (una vacuna de algas comestibles ), [39] y un fármaco de diseño complejo que podría utilizarse para tratar el cáncer. [40]
Fuente de proteína alternativa
Se ha sugerido que C. reinhardtii es una nueva fuente nutricional a base de algas. En comparación con la Chlorella y la Spirulina , se descubrió que C. reinhardtii tiene más ácido alfa-linolénico y una menor cantidad de metales pesados, a la vez que contiene todos los aminoácidos esenciales y un contenido proteico similar. [41] Triton Algae Innovations estaba desarrollando un producto proteico alternativo comercial elaborado a partir de C. reinhardtii .
Fuente limpia de producción de hidrógeno
En 1939, el investigador alemán Hans Gaffron (1902-1979), que en ese momento trabajaba en la Universidad de Chicago, descubrió el metabolismo del hidrógeno de las algas verdes unicelulares. C. reinhardtii y otras algas verdes pueden, en determinadas circunstancias, dejar de producir oxígeno y pasar a producir hidrógeno. Esta reacción, llevada a cabo por la hidrogenasa , una enzima activa únicamente en ausencia de oxígeno, es de corta duración. Durante los treinta años siguientes, Gaffron y su equipo elaboraron la mecánica básica de esta producción fotosintética de hidrógeno por parte de las algas. [42]
Para aumentar la producción de hidrógeno, los investigadores están siguiendo varias vías.
- La primera vía consiste en desacoplar la hidrogenasa de la fotosíntesis. De esta manera, la acumulación de oxígeno ya no puede inhibir la producción de hidrógeno. Y, si se va un paso más allá modificando la estructura de la enzima hidrogenasa, se consigue que la hidrogenasa sea insensible al oxígeno. Esto hace posible una producción continua de hidrógeno. En este caso, el flujo de electrones necesario para esta producción ya no procede de la producción de azúcares, sino de la descomposición de su propia reserva de almidón . [43]
- Una segunda vía es interrumpir temporalmente, mediante manipulación genética de la hidrogenasa, el proceso de fotosíntesis, lo que impide que el oxígeno alcance un nivel en el que pueda detener la producción de hidrógeno. [44]
- La tercera vía, investigada principalmente por los investigadores en la década de 1950, son los métodos químicos o mecánicos de eliminación del O2 producido por la actividad fotosintética de las células de las algas. Estos han incluido la adición de eliminadores de O2, el uso de reductores añadidos y la purga de los cultivos con gases inertes. [45] Sin embargo, estos métodos no son inherentemente escalables y pueden no ser aplicables a los sistemas aplicados. Han aparecido nuevas investigaciones sobre el tema de la eliminación de oxígeno de los cultivos de algas y pueden eliminar los problemas de incrustaciones.
- Se ha investigado la cuarta vía, es decir, el uso de sales de cobre para disociar la acción de la hidrogenasa de la producción de oxígeno. [46]
- Se ha sugerido que la quinta vía redirige el flujo de electrones fotosintéticos desde la fijación de CO 2 en el ciclo de Calvin a la hidrogenasa mediante la aplicación de pulsos de luz cortos a las algas anaeróbicas [47] o agotando el cultivo de CO 2 . [48]
Véase también
Referencias
- ^ ab Ettl, H. (1983). Ettl, H.; Gerloff, J.; Heynig, H.; Mollenhauer, D. (eds.). Clorofita. 1. Parte / Parte 1: Phytomonadina . Süßwasserflora von Mitteleuropa. vol. 9. VEB Gustav Fischer Verlag. págs. XIV + 808. ISBN 978-3-8274-2659-8.
- ^ "CC-125 tipo salvaje mt+ 137c". Lista de la colección principal del Centro de Chlamydomonas . Archivado desde el original el 27 de julio de 2009. Consultado el 9 de marzo de 2009 .
- ^ Harris, Stern y Witman 2009
- ^ "Taxonomía de Chlamydomonas". Universidad de Montreal.
- ^ ab Ueki, Noriko; Ide, Takahiro; Mochiji, Shota; Kobayashi, Yuki; Tokutsu, Ryutaro; Ohnishi, Norikazu; Yamaguchi, Katsushi; Shigenobu, Shuji; Tanaka, Kan; Minagawa, junio; Hisabori, Toru; Hirono, Masafumi; Wakabayashi, Ken-Ichi (2016). "Determinación del signo fototáctico dependiente de las manchas oculares en Chlamydomonas reinhardtii". Actas de la Academia Nacional de Ciencias . 113 (19): 5299–5304. Código Bib : 2016PNAS..113.5299U. doi : 10.1073/pnas.1525538113 . PMC 4868408 . PMID 27122315.
- ^ Foster KW, Smyth RD (diciembre de 1980). "Antenas de luz en algas fototácticas". Microbiol Rev . 44 (4): 572–630. doi :10.1128/mr.44.4.572-630.1980. PMC 373196 . PMID 7010112.
- ^ ab Hegemann, P.; Berthold, P. "13. Fotorreceptores sensoriales y control de la actividad flagelar por la luz, págs. 395-430". En Harris, Stern y Witman (2009).
- ^ Demmig-Adams, B.; Adams, WW (1992). "Fotoprotección y otras respuestas de las plantas al estrés lumínico intenso". Revisión anual de fisiología vegetal y biología molecular de plantas . 43 : 599–626. doi :10.1146/annurev.pp.43.060192.003123.
- ^ Merchant; Prochnik, SE; Vallon, O; Harris, EH; Karpowicz, SJ; Witman, GB; Terry, A; Salamov, A; et al. (2007). "El genoma de Chlamydomonas revela la evolución de funciones clave en animales y plantas". Science . 318 (5848): 245–250. Bibcode :2007Sci...318..245M. doi :10.1126/science.1143609. PMC 2875087 . PMID 17932292.
- ^ Nagel G, Ollig D, Fuhrmann M, et al. (28 de junio de 2002). "Canal rodopsina-1: un canal de protones activado por luz en algas verdes". Ciencia . 296 (5577): 2395–8. Código bibliográfico : 2002 Ciencia... 296.2395N. doi : 10.1126/ciencia.1072068. PMID 12089443. S2CID 206506942.
- ^ Lagali PS, Balya D, Awatramani GB, Münch TA, Kim DS, Busskamp V, Cepko CL, Roska B (junio de 2008). "Los canales activados por luz dirigidos a las células bipolares ON restauran la función visual en la degeneración de la retina". Nature Neuroscience . 11 (6): 667–75. doi :10.1038/nn.2117. PMID 18432197. S2CID 6798764.
- ^ Boyden ES, et al. (3 de mayo de 2011). "Una historia de la optogenética: el desarrollo de herramientas para controlar circuitos cerebrales con luz". F1000 Biology Reports . 3 (11): 11. doi : 10.3410/B3-11 . PMC 3155186 . PMID 21876722.
- ^ Figueroa-Martínez F, Funes S, Franzén LG, González-Halphen D (mayo de 2008). "Reconstrucción de la maquinaria de importación de proteínas mitocondriales de Chlamydomonas reinhardtii". Genética . 179 (1): 149–155. doi :10.1534/genetics.108.087965. PMC 2390594 . PMID 18493047.
- ^ Sager R, Granick S (julio de 1954). "Control nutricional de la sexualidad en Chlamydomonas reinhardi". J. Gen. Physiol . 37 (6): 729–42. doi :10.1085/jgp.37.6.729. PMC 2147466 . PMID 13174779.
- ^ Oldenhof H.; Zachleder V.; den Ende H. (2006). "Regulación del ciclo celular mediante luz azul y roja en Chlamydomonas reinhardtii (Chlorophyta)". Eur. J. Phycol . 41 (3): 313–320. Bibcode :2006EJPhy..41..313O. doi : 10.1080/09670260600699920 .
- ^ "Chlamydomonas reinhardtii v3.0".
- ^ "Mitocondria de Chlamydomonas reinhardtii, genoma completo". Nucleótido . Biblioteca Nacional de Medicina. 3 de abril de 2023. Secuencia de referencia del NCBI: NC_001638.1.
- ^ "Cloroplasto de Chlamydomonas reinhardtii, genoma completo". Nucleótido . Biblioteca Nacional de Medicina. 3 de abril de 2023. Secuencia de referencia del NCBI: NC_005353.1.
- ^ "Portal del genoma del cloroplasto de Chlamydomonas".
- ^ "Chlamydomonas Center — Libraries". Archivado desde el original el 19 de octubre de 2004. Consultado el 28 de septiembre de 2006 .
- ^ "CUGI". Archivado desde el original el 26 de diciembre de 2014. Consultado el 3 de abril de 2006 .
- ^ "[KDRI]Índice EST de Chlamydomonas reinhardtii".
- ^ "Buscar en ChlamyEST". Chlamy Center. Archivado desde el original el 4 de febrero de 2005. Consultado el 28 de septiembre de 2006 .
- ^ Li, Xiaobo; Zhang, Ru; Patena, Weronika; Gang, Spencer S.; Blum, Sean R.; Ivanova, Nina; Yue, Rebecca; Robertson, Jacob M.; Lefebvre, Paul A.; Fitz-Gibbon, Sorel T.; Grossman, Arthur R.; Jonikas, Martin C. (1 de febrero de 2016). "Una biblioteca de mutantes indexada y mapeada permite estudios de genética inversa de procesos biológicos en Chlamydomonas reinhardtii". La célula vegetal . 28 (2): 367–387. doi :10.1105/tpc.15.00465. ISSN 1040-4651. PMC 4790863 . PMID 26764374.
- ^ Li, Xiaobo; Patena, Weronika; Fauser, Friedrich; Jinkerson, Robert E.; Saroussi, Shai; Meyer, Moritz T.; Ivanova, Nina; Robertson, Jacob M.; Yue, Rebeca; Zhang, Ru; Vilarrasa-Blasi, Josep; Wittkopp, Tyler M.; Ramundo, Silvia; Blum, Sean R.; Vaya, Audrey; Laudon, Mateo; Srikumar, Tharan; Lefebvre, Paul A.; Grossman, Arthur R.; Jonikas, Martin C. (abril de 2019). "Una biblioteca de mutantes de algas de todo el genoma y un análisis funcional identifican los genes necesarios para la fotosíntesis eucariota". Genética de la Naturaleza . 51 (4): 627–635. doi :10.1038/s41588-019-0370-6. ISSN 1546-1718. Número de modelo : PMID 30886426 .
- ^ Hattman, S; Kenny, C; Berger, L; Pratt, K (septiembre de 1978). "Estudio comparativo de la metilación del ADN en tres eucariotas unicelulares". Journal of Bacteriology . 135 (3): 1156–7. doi :10.1128/JB.135.3.1156-1157.1978. PMC 222496 . PMID 99431.
- ^ Fu, sí; Luo, Guan-Zheng; Chen, Kai; Deng, Xin; Yu, Miao; Han, Dalí; Hao, Ziyang; Liu, Jianzhao; Lu, Xingyu; Doré, Luis C.; Weng, Xiaocheng; Ji, Quanjiang; Mets, Laurens; Él, Chuan (mayo de 2015). "N6-metildesoxiadenosina marca los sitios de inicio de la transcripción activa en Chlamydomonas". Celúla . 161 (4): 879–892. doi :10.1016/j.cell.2015.04.010. PMC 4427561 . PMID 25936837.
- ^ Vlcek D, Sevcovicová A, Sviezená B, Gálová E, Miadoková E (enero de 2008). "Chlamydomonas reinhardtii: un sistema modelo conveniente para el estudio de la reparación del ADN en eucariotas fotoautotróficos". Curr Genet . 53 (1): 1–22. doi :10.1007/s00294-007-0163-9. PMID 17992532.
- ^ ab Dürrenberger F, Thompson AJ, Herrin DL, Rochaix JD (septiembre de 1996). "Recombinación inducida por rotura de doble cadena en cloroplastos de Chlamydomonas reinhardtii". Nucleic Acids Res . 24 (17): 3323–31. doi :10.1093/nar/24.17.3323. PMC 146090 . PMID 8811085.
- ^ Colegrave N (2002). "El sexo libera el límite de velocidad de la evolución". Nature . 420 (6916): 664–6. Bibcode :2002Natur.420..664C. doi :10.1038/nature01191. hdl : 1842/692 . PMID 12478292. S2CID 4382757.
- ^ De Visser JA, Hoekstra RF, Van den Ende H (1996). "El efecto del sexo y las mutaciones deletéreas sobre la aptitud en Chlamydomonas ". Proc. R. Soc. Lond. B. 263 ( 1367): 193–200. doi :10.1098/rspb.1996.0031. JSTOR 50473.
- ^ Collins, Bell (2004). "Consecuencias fenotípicas de 1000 generaciones de selección en niveles elevados de CO2 en un alga verde". Nature . 431 (7008): 566–9. Bibcode :2004Natur.431..566C. doi :10.1038/nature02945. PMID 15457260. S2CID 4354542.
- ^ Kondrashov AS (octubre de 1984). "Mutaciones deletéreas como factor evolutivo. 1. La ventaja de la recombinación". Genet. Res . 44 (2): 199–217. doi : 10.1017/s0016672300026392 . PMID 6510714.
- ^ Renaut S, Replansky T, Heppleston A, Bell G (noviembre de 2006). "La ecología y la genética de la aptitud física en Chlamydomonas. XIII. Aptitud física de poblaciones sexuales y asexuales a largo plazo en entornos benignos". Evolution . 60 (11): 2272–9. doi :10.1554/06-084.1 (inactivo 2024-07-30). PMID 17236420. S2CID 18977144.
{{cite journal}}: CS1 maint: DOI inactive as of July 2024 (link) - ^ ab Polin, Marco; Tuval, Idan; Drescher, Knut; Gollub, JP; Goldstein, Raymond E. (24 de julio de 2009). "Chlamydomonas nada con dos "marchas" en una versión eucariota de la locomoción de correr y dar volteretas". Science . 325 (5939): 487–490. Bibcode :2009Sci...325..487P. doi :10.1126/science.1172667. ISSN 0036-8075. PMID 19628868. S2CID 10530835.
- ^ García, Michaël (9 de julio de 2013). Hydrodynamique de micro-nageurs (PhD) (en francés). Universidad de Grenoble. tel-00952771.
- ^ Goldstein, Raymond E (23 de julio de 2018). "¿Son los resultados teóricos 'resultados'?". eLife . 7 : e40018. doi : 10.7554/eLife.40018 . ISSN 2050-084X. PMC 6056240 . PMID 30033910.
- ^ Demurtas OC; Massa S; Ferrante P; Venuti A; Franconi R; et al. (2013). "Una vacuna contra el virus del papiloma humano 16 E7 derivada de Chlamydomonas induce protección tumoral específica". PLOS ONE . 8 (4): e61473. Bibcode :2013PLoSO...861473D. doi : 10.1371/journal.pone.0061473 . PMC 3634004 . PMID 23626690.
- ^ "Biólogos producen una posible vacuna contra la malaria a partir de algas". PhysOrg. 16 de mayo de 2012.
- ^ "Ingeniería de algas para crear un fármaco 'de diseño' anticancerígeno complejo". PhysOrg. 10 de diciembre de 2012.
- ^ Darwish, Randa; Gedi, Mohamed; Akepach, Patchaniya; Assaye, Hirut; Zaky, Abderlahman; Gray, David (26 de septiembre de 2020). "Chlamydomonas reinhardtii es un posible complemento alimenticio con capacidad para superar a la clorella y la espirulina". Applied Sciences . 10 (19): 6736. doi : 10.3390/app10196736 . hdl : 20.500.11820/20a558d8-9745-4613-8203-d86234aa4762 . Consultado el 26 de agosto de 2021 .
- ^ Anastasios Melis; Thomas Happe (2004). "Senderos de investigación sobre hidrógeno en algas verdes: desde Hans Gaffron hasta nuevas fronteras" (PDF) . Photosynthesis Research . 80 (1–3): 401–9. Bibcode :2004PhoRe..80..401M. doi :10.1023/B:PRES.0000030421.31730.cb. PMID 16328836. S2CID 7188276.
- ^ Laurent Cournac; Florence Musa; Laetitia Bernarda; Geneviève Guedeneya; Paulette Vignaisb; Gilles Peltie (2002). "Pasos limitantes de la producción de hidrógeno en Chlamydomonas reinhardtii y Synechocystis PCC 6803 analizados mediante transitorios de intercambio de gases inducidos por la luz". Revista Internacional de Energía del Hidrógeno . 27 (11/12): 1229–37. Código Bibliográfico :2002IJHE...27.1229C. doi :10.1016/S0360-3199(02)00105-2.
- ^ Anastasios Melis. "Producción de biocombustibles de hidrógeno e hidrocarburos mediante fotosíntesis de microalgas". Archivado desde el original el 2008-04-03 . Consultado el 2008-04-07 .
- ^ Kosourov, S.; Tsyganov, A.; Seibert, M.; Ghirardi, M. (junio de 2002). "Fotoproducción sostenida de hidrógeno por Chlamydomonas reinhardtii: efectos de los parámetros de cultivo". Biotecnología. Bioing . 78 (7): 731–40. doi :10.1002/bit.10254. PMID 12001165.
- ^ Fernandez VM, Rua ML, Reyes P, Cammack R, Hatchikian EC (noviembre de 1989). "Inhibición de la hidrogenasa de Desulfovibrio gigas con sales de cobre y otros iones metálicos". Eur. J. Biochem . 185 (2): 449–54. doi : 10.1111/j.1432-1033.1989.tb15135.x . PMID 2555191.
- ^ Kosourov, S.; Jokel, M.; Aro, E.-M.; Allahverdiyeva, Y. (marzo de 2018). "Un nuevo enfoque para la fotoproducción sostenida y eficiente de H2 por Chlamydomonas reinhardtii". Energy & Environmental Science . 11 (6): 1431–6. doi : 10.1039/C8EE00054A .
- ^ Nagy, V.; Podmaniczki, A.; Vidal-Meireles, A.; Tengölics, R.; Kovács, L.; Rákhely, G.; Scoma, A.; Tóth SZ. (marzo de 2018). "Producción de H2 sostenible y eficiente basada en la división del agua en algas verdes, lograda mediante la limitación del sustrato del ciclo de Calvin–Benson–Bassham". Biotecnología para biocombustibles . 11 : 69. doi : 10.1186/s13068-018-1069-0 . PMC 5858145 . PMID 29560024.
Lectura adicional
- Aoyama, H.; Kuroiwa, T.; Nakamura, S. (2009). "El comportamiento dinámico de las mitocondrias en cigotos vivos durante la maduración y la meiosis en Chlamydomonas reinhardtii ". Eur. J. Phycol . 44 (4): 497–507. Bibcode :2009EJPhy..44..497A. doi :10.1080/09670260903272599.
- Jamers, A.; Lenjou, M.; Deraedt, P.; van Bockstaele, D.; Blust, R.; de Coen, W. (2009). "Análisis citométrico de flujo de las algas verdes Chlamydomonas reinhadtii (Chlorophyceae) expuestas al cadmio". Eur. J. Phycol.'. 44 (4): 541–550. Código Bibliográfico :2009EJPhy..44..541J. doi :10.1080/09670260903118214.
- Harris, EH; Stern, DB; Witman, GB, eds. (2009). The Chlamydomonas Sourcebook. Vol. 1–3 (2.ª ed.). Académico. ISBN 978-0-12-370873-1.
Enlaces externos
- Centro de recursos de Chlamydomonas: "Un repositorio central para recibir, catalogar, preservar y distribuir cultivos silvestres y mutantes confiables y de alta calidad del alga verde Chlamydomonas reinhardtii , así como reactivos moleculares y kits útiles para la educación y la investigación".
- Portal de Genómica Comparativa de Plantas: recursos sobre Chlamydomonas reinhardtii del Instituto Conjunto del Genoma del Departamento de Energía
- Guiry, MD; Guiry, GM "Chlamydomonas reinhardtii". AlgaeBase . Publicación electrónica mundial, Universidad Nacional de Irlanda, Galway.
- Célula de Chlamydomonas reinhardtii, ciclo de vida, cepas, tipos de apareamiento: base de datos archivada.
