Aminoácido

Los aminoácidos son compuestos orgánicos que contienen grupos funcionales tanto de aminoácidos como de ácidos carboxílicos . [1] Aunque existen más de 500 aminoácidos en la naturaleza, los más importantes son los 22 α-aminoácidos incorporados a las proteínas . [2] Solo estos 22 aparecen en el código genético de la vida. [3] [4]
Los aminoácidos se pueden clasificar según la ubicación de los grupos funcionales estructurales centrales ( aminoácidos alfa (α) , beta (β) , gamma (γ) , etc.); otras categorías se relacionan con la polaridad , la ionización y el tipo de grupo de cadena lateral ( alifático , acíclico , aromático , polar , etc.). En forma de proteínas, los residuos de aminoácidos forman el segundo componente más grande ( siendo el agua el más grande) de los músculos humanos y otros tejidos . [5] Más allá de su papel como residuos en las proteínas, los aminoácidos participan en varios procesos como el transporte de neurotransmisores y la biosíntesis . Se cree que desempeñaron un papel clave para permitir la vida en la Tierra y su surgimiento . [6]
La Comisión Conjunta de Nomenclatura Bioquímica de la IUPAC y la IUBMB nombra formalmente a los aminoácidos en términos de la estructura "neutra" ficticia que se muestra en la ilustración. Por ejemplo, el nombre sistemático de la alanina es ácido 2-aminopropanoico, basado en la fórmula CH 3 −CH(NH 2 )−COOH . La Comisión justificó este enfoque de la siguiente manera: [7]
Los nombres y fórmulas sistemáticos que se dan se refieren a formas hipotéticas en las que los grupos amino no están protonados y los grupos carboxilo no están disociados. Esta convención es útil para evitar diversos problemas de nomenclatura, pero no debe interpretarse como que estas estructuras representan una fracción apreciable de las moléculas de aminoácidos.
Historia
Los primeros aminoácidos fueron descubiertos a principios del siglo XIX. [8] [9] En 1806, los químicos franceses Louis-Nicolas Vauquelin y Pierre Jean Robiquet aislaron un compuesto de los espárragos que posteriormente se denominó asparagina , el primer aminoácido en ser descubierto. [10] [11] La cistina fue descubierta en 1810, [12] aunque su monómero, la cisteína , permaneció sin descubrir hasta 1884. [13] [11] [a] La glicina y la leucina fueron descubiertas en 1820. [14] El último de los 20 aminoácidos comunes en ser descubierto fue la treonina en 1935 por William Cumming Rose , quien también determinó los aminoácidos esenciales y estableció los requerimientos diarios mínimos de todos los aminoácidos para un crecimiento óptimo. [15] [16]
La unidad de la categoría química fue reconocida por Wurtz en 1865, pero no le dio un nombre particular. [17] El primer uso del término "aminoácido" en el idioma inglés data de 1898, [18] mientras que el término alemán, Aminosäure , se utilizó antes. [19] Se descubrió que las proteínas producen aminoácidos después de la digestión enzimática o hidrólisis ácida . En 1902, Emil Fischer y Franz Hofmeister propusieron independientemente que las proteínas se forman a partir de muchos aminoácidos, por lo que se forman enlaces entre el grupo amino de un aminoácido con el grupo carboxilo de otro, lo que resulta en una estructura lineal que Fischer denominó " péptido ". [20]
Estructura general

Los 2- , alfa- o α-aminoácidos [21] tienen la fórmula genérica H 2 NCHRCOOH en la mayoría de los casos, [b] donde R es un sustituyente orgánico conocido como " cadena lateral ". [22]
De los cientos de aminoácidos descritos, 22 son proteinogénicos ("constructores de proteínas"). [23] [24] [25] Son estos 22 compuestos los que se combinan para dar una amplia gama de péptidos y proteínas ensamblados por los ribosomas . [26] Los aminoácidos no proteinogénicos o modificados pueden surgir de la modificación postraduccional o durante la síntesis de péptidos no ribosómicos .
Quiralidad
El átomo de carbono que se encuentra junto al grupo carboxilo se denomina carbono α . En los aminoácidos proteinogénicos, lleva la amina y el grupo R o cadena lateral específicos de cada aminoácido. Con cuatro sustituyentes distintos, el carbono α es estereogénico en todos los α-aminoácidos excepto en la glicina. Todos los aminoácidos proteogénicos quirales tienen la configuración L. Son enantiómeros "levógiros" , que se refieren a los estereoisómeros del carbono alfa.
Se han encontrado algunos D -aminoácidos ("diestros") en la naturaleza, por ejemplo, en envolturas bacterianas , como neuromodulador ( D - serina ) y en algunos antibióticos . [27] [28] En raras ocasiones, los residuos de D -aminoácidos se encuentran en proteínas y se convierten a partir del L -aminoácido como una modificación postraduccional . [29] [c]
Cadenas laterales
Cadenas laterales con carga polar
Cinco aminoácidos poseen una carga a pH neutro. A menudo, estas cadenas laterales aparecen en las superficies de las proteínas para permitir su solubilidad en agua, y las cadenas laterales con cargas opuestas forman importantes contactos electrostáticos llamados puentes salinos que mantienen las estructuras dentro de una sola proteína o entre proteínas interconectadas. [32] Muchas proteínas unen metales en sus estructuras de manera específica, y estas interacciones suelen estar mediadas por cadenas laterales cargadas, como aspartato , glutamato e histidina . En determinadas condiciones, cada grupo formador de iones puede estar cargado, formando sales dobles. [33]
Los dos aminoácidos con carga negativa a pH neutro son el aspartato (Asp, D) y el glutamato (Glu, E). Los grupos carboxilato aniónicos se comportan como bases de Brønsted en la mayoría de las circunstancias. [32] Las enzimas en entornos de pH muy bajo, como la proteasa aspártica pepsina en los estómagos de los mamíferos, pueden tener residuos catalíticos de aspartato o glutamato que actúan como ácidos de Brønsted.

Existen tres aminoácidos con cadenas laterales que son cationes a pH neutro: arginina (Arg, R), lisina (Lys, K) e histidina (His, H). La arginina tiene un grupo guanidino cargado y la lisina un grupo alquil amino cargado, y están completamente protonados a pH 7. El grupo imidazol de la histidina tiene un pKa de 6,0, y solo está protonado en un 10 % a pH neutro. Debido a que la histidina se encuentra fácilmente en sus formas básica y ácida conjugada, a menudo participa en transferencias catalíticas de protones en reacciones enzimáticas. [32]
Cadenas laterales polares sin carga
Los aminoácidos polares sin carga serina (Ser, S), treonina (Thr, T), asparagina (Asn, N) y glutamina (Gln, Q) forman fácilmente enlaces de hidrógeno con agua y otros aminoácidos. [32] No se ionizan en condiciones normales, siendo una excepción destacada la serina catalítica en las serina proteasas . Este es un ejemplo de perturbación grave y no es característico de los residuos de serina en general. La treonina tiene dos centros quirales, no solo el centro quiral L (2 S ) en el carbono α compartido por todos los aminoácidos aparte de la glicina aquiral, sino también (3 R ) en el carbono β. La especificación estereoquímica completa es (2 S ,3 R )- L - treonina .
Cadenas laterales hidrofóbicas
Las interacciones de aminoácidos no polares son la principal fuerza impulsora detrás de los procesos que pliegan las proteínas en sus estructuras tridimensionales funcionales. [32] Ninguna de las cadenas laterales de estos aminoácidos se ioniza fácilmente y, por lo tanto, no tiene pKa s , con la excepción de la tirosina (Tyr, Y). El hidroxilo de la tirosina puede desprotonarse a un pH alto formando el fenolato con carga negativa. Debido a esto, se podría colocar a la tirosina en la categoría de aminoácidos polares sin carga, pero su muy baja solubilidad en agua coincide bien con las características de los aminoácidos hidrófobos.
Cadenas laterales para casos especiales
Varias cadenas laterales no están bien descritas por las categorías cargadas, polares e hidrofóbicas. La glicina (Gly, G) podría considerarse un aminoácido polar ya que su pequeño tamaño significa que su solubilidad está determinada en gran medida por los grupos amino y carboxilato. Sin embargo, la falta de cualquier cadena lateral proporciona a la glicina una flexibilidad única entre los aminoácidos con grandes ramificaciones para el plegamiento de proteínas. [32] La cisteína (Cys, C) también puede formar enlaces de hidrógeno fácilmente, lo que la colocaría en la categoría de aminoácidos polares, aunque a menudo se puede encontrar en estructuras de proteínas que forman enlaces covalentes, llamados enlaces disulfuro , con otras cisteínas. Estos enlaces influyen en el plegamiento y la estabilidad de las proteínas, y son esenciales en la formación de anticuerpos . La prolina (Pro, P) tiene una cadena lateral de alquilo y podría considerarse hidrofóbica, pero debido a que la cadena lateral se une nuevamente al grupo amino alfa, se vuelve particularmente inflexible cuando se incorpora a las proteínas. Similar a la glicina, esto influye en la estructura de la proteína de una manera única entre los aminoácidos. La selenocisteína (Sec, U) es un aminoácido raro que no está codificado directamente por el ADN, pero que se incorpora a las proteínas a través del ribosoma. La selenocisteína tiene un potencial redox menor en comparación con la cisteína similar y participa en varias reacciones enzimáticas únicas. [34] La pirrolisina (Pyl, O) es otro aminoácido que no está codificado en el ADN, pero que los ribosomas sintetizan en proteínas. [35] Se encuentra en especies arqueales donde participa en la actividad catalítica de varias metiltransferasas.
β- y γ-aminoácidos
Aminoácidos con la estructura NH+3-CXY-CXY-CO−2, como la β-alanina , un componente de la carnosina y algunos otros péptidos, son β-aminoácidos. Los que tienen la estructura NH+3−CXY−CXY−CXY−CO−2son γ-aminoácidos, y así sucesivamente, donde X e Y son dos sustituyentes (uno de los cuales normalmente es H). [7]
Zwitteriones

Las formas naturales comunes de los aminoácidos tienen una estructura zwitteriónica , con −NH+3( −NH+2− en el caso de la prolina) y −CO−2grupos funcionales unidos al mismo átomo de C, y por lo tanto son α-aminoácidos, y son los únicos que se encuentran en las proteínas durante la traducción en el ribosoma. En solución acuosa a pH cercano a la neutralidad, los aminoácidos existen como zwitteriones , es decir, como iones dipolares con NH+3y CO−2en estados cargados, por lo que la estructura general es NH+3-CrH-CO−2A pH fisiológico, las llamadas "formas neutras" −NH 2 −CHR−CO 2 H no están presentes en ningún grado medible. [36] Aunque las dos cargas en la estructura zwitterion suman cero, es engañoso llamar "sin carga" a una especie con una carga neta de cero.
En condiciones fuertemente ácidas (pH por debajo de 3), el grupo carboxilato se protona y la estructura se convierte en un ácido carboxílico de amonio, NH+3−CHR−CO 2 H . Esto es relevante para enzimas como la pepsina, que son activas en ambientes ácidos como el estómago y los lisosomas de los mamíferos , pero no se aplica significativamente a las enzimas intracelulares. En condiciones altamente básicas (pH mayor de 10, que normalmente no se observa en condiciones fisiológicas), el grupo amonio se desprotona para dar NH 2 −CHR−CO−2.
Aunque en química se utilizan diversas definiciones de ácidos y bases, la única que resulta útil para la química en disolución acuosa es la de Brønsted : [37] [38] un ácido es una especie que puede donar un protón a otra especie, y una base es una que puede aceptar un protón. Este criterio se utiliza para etiquetar los grupos en la ilustración anterior. Las cadenas laterales de carboxilato de los residuos de aspartato y glutamato son las principales bases de Brønsted en las proteínas. Del mismo modo, la lisina, la tirosina y la cisteína actuarán normalmente como un ácido de Brønsted. La histidina en estas condiciones puede actuar tanto como un ácido de Brønsted como una base.
Punto isoeléctrico

En el caso de los aminoácidos con cadenas laterales sin carga, el zwitterión predomina en valores de pH entre los dos valores de p K a , pero coexiste en equilibrio con pequeñas cantidades de iones netos negativos y positivos. En el punto medio entre los dos valores de p K a , la cantidad traza de iones netos negativos y la cantidad traza de iones netos positivos se equilibran, de modo que la carga neta promedio de todas las formas presentes es cero. [39] Este pH se conoce como el punto isoeléctrico p I , por lo que p I = 1/2 ( pKa1 + pKa2 ) .
En el caso de los aminoácidos con cadenas laterales cargadas, interviene el p K a de la cadena lateral. Por tanto, en el caso del aspartato o el glutamato con cadenas laterales negativas, el grupo amino terminal se encuentra esencialmente en su totalidad en la forma cargada −NH+3, pero esta carga positiva debe equilibrarse con el estado con solo un grupo carboxilato C-terminal cargado negativamente. Esto ocurre a medio camino entre los dos valores de p K a del carboxilato : p I = 1/2 (p K a1 + p K a(R) ), donde p K a(R) es la cadena lateral p K a . [38]
Consideraciones similares se aplican a otros aminoácidos con cadenas laterales ionizables, incluidos no sólo el glutamato (similar al aspartato), sino también la cisteína, la histidina, la lisina, la tirosina y la arginina con cadenas laterales positivas.
Los aminoácidos tienen movilidad cero en la electroforesis en su punto isoeléctrico, aunque este comportamiento suele aprovecharse más en el caso de péptidos y proteínas que en el de aminoácidos individuales. Los zwitteriones tienen una solubilidad mínima en su punto isoeléctrico, y algunos aminoácidos (en particular, aquellos con cadenas laterales no polares) pueden aislarse por precipitación a partir del agua ajustando el pH al punto isoeléctrico requerido.
Propiedades fisicoquímicas
Los 20 aminoácidos canónicos se pueden clasificar según sus propiedades. Los factores importantes son la carga, la hidrofilicidad o hidrofobicidad , el tamaño y los grupos funcionales. [28] Estas propiedades influyen en la estructura de las proteínas y las interacciones proteína-proteína . Las proteínas solubles en agua tienden a tener sus residuos hidrófobos ( Leu , Ile , Val , Phe y Trp ) enterrados en el medio de la proteína, mientras que las cadenas laterales hidrófilas están expuestas al disolvente acuoso. (En bioquímica , un residuo se refiere a un monómero específico dentro de la cadena polimérica de un polisacárido , proteína o ácido nucleico ). Las proteínas integrales de membrana tienden a tener anillos externos de aminoácidos hidrófobos expuestos que los anclan en la bicapa lipídica . Algunas proteínas de membrana periféricas tienen un parche de aminoácidos hidrófobos en su superficie que se adhiere a la membrana. De manera similar, las proteínas que tienen que unirse a moléculas con carga positiva tienen superficies ricas en aminoácidos con carga negativa, como el glutamato y el aspartato , mientras que las proteínas que se unen a moléculas con carga negativa tienen superficies ricas en aminoácidos con carga positiva, como la lisina y la arginina . Por ejemplo, la lisina y la arginina están presentes en grandes cantidades en las regiones de baja complejidad de las proteínas de unión a ácidos nucleicos. [40] Existen varias escalas de hidrofobicidad de los residuos de aminoácidos. [41]
Algunos aminoácidos tienen propiedades especiales. La cisteína puede formar enlaces disulfuro covalentes con otros residuos de cisteína. La prolina forma un ciclo con la estructura principal del polipéptido y la glicina es más flexible que otros aminoácidos.
La glicina y la prolina están fuertemente presentes en regiones de baja complejidad de proteínas tanto eucariotas como procariotas, mientras que lo opuesto es el caso con la cisteína, la fenilalanina, el triptófano, la metionina, la valina, la leucina y la isoleucina, que son altamente reactivas, complejas o hidrófobas. [40] [42] [43]
Muchas proteínas sufren una serie de modificaciones postraduccionales , mediante las cuales se unen grupos químicos adicionales a las cadenas laterales de los residuos de aminoácidos, produciendo a veces lipoproteínas (que son hidrófobas) [44] o glicoproteínas (que son hidrófilas) [45], lo que permite que la proteína se adhiera temporalmente a una membrana. Por ejemplo, una proteína de señalización puede unirse y luego desprenderse de una membrana celular, porque contiene residuos de cisteína a los que se les puede agregar el ácido graso palmítico y luego eliminarlo. [46]
Tabla de abreviaturas y propiedades de aminoácidos estándar
Aunque los símbolos de una letra están incluidos en la tabla, la IUPAC-IUBMB recomienda [7] que "el uso de los símbolos de una letra se restrinja a la comparación de secuencias largas".
La notación de una letra fue elegida por la IUPAC-IUB basándose en las siguientes reglas: [47]
- Se utilizan letras iniciales cuando no hay ambigüedad: C cisteína, H histidina, I isoleucina, M metionina, S serina, V valina, [47]
- Cuando se necesita una asignación arbitraria, se da prioridad a los aminoácidos estructuralmente más simples: A Alanina, G glicina, L leucina, P prolina, T treonina, [47]
- La F PH enilalanina y la R a R ginina se asignan por ser fonéticamente sugerentes, [47]
- El triptófano W se asigna en función del anillo doble que sugiere visualmente la voluminosa letra W, [47]
- La lisina K y la tirosina Y se asignan alfabéticamente como las más cercanas a sus iniciales L y T (nótese que se evitó la U por su similitud con V, mientras que la X se reservó para aminoácidos indeterminados o atípicos); para la tirosina también se propuso el mnemónico t Y rosina, [48]
- El aspartato D se asignó arbitrariamente, con el mnemónico propuesto ácido aspártico D ic; [49] El glutamato E se asignó en secuencia alfabética siendo más grande por solo un grupo metileno –CH2–, [48]
- La N asparagina se asignó arbitrariamente, con el mnemónico propuesto asparagi N e; [49] La Q glutamina se asignó en secuencia alfabética de las que aún estaban disponibles (nótese nuevamente que se evitó O debido a la similitud con D), con el mnemónico propuesto Q lutamina. [49]
| Aminoácido | Símbolos de 3 y 1 letra | Cadena lateral | Índice de hidropatía [50] | Absortividad molar [51] | Masa molecular | Abundancia de proteínas (%) [52] | Codificación genética estándar, notación IUPAC | ||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 3 | 1 | Clase | Polaridad química [53] | Carga neta a pH 7,4 [53] | Longitud de onda, λmáx . (nm) | Coeficiente ε (mM −1 ·cm −1 ) | |||||
| Alanina | Ala | A | Alifático | No polar | Neutral | 1.8 | 89.094 | 8,76 | GCN | ||
| Arginina | Argento | R | Catión fijo | Polar básico | Positivo | -4,5 | 174.203 | 5,78 | MONSEÑOR, CGY [54] | ||
| Asparagina | ASN | norte | Amida | Polar | Neutral | -3,5 | 132.119 | 3.93 | AAY | ||
| Aspartato | Áspid | D | Anión | Base de Brønsted | Negativo | -3,5 | 133.104 | 5.49 | Gay | ||
| Cisteína | Cis | do | Tiol | Ácido de Brønsted | Neutral | 2.5 | 250 | 0.3 | 121.154 | 1.38 | UGY |
| Glutamina | Gln | Q | Amida | Polar | Neutral | -3,5 | 146.146 | 3.9 | AUTO | ||
| Glutamato | pegamento | mi | Anión | Base de Brønsted | Negativo | -3,5 | 147.131 | 6.32 | GAR | ||
| Glicina | Gly | GRAMO | Alifático | No polar | Neutral | -0,4 | 75.067 | 7.03 | GGN | ||
| Histidina | Su | yo | Catiónico | Ácido y base de Brønsted | Positivo, 10% Neutral, 90% | -3,2 | 211 | 5.9 | 155.156 | 2.26 | ISLA PEQUEÑA |
| Isoleucina | Isla | I | Alifático | No polar | Neutral | 4.5 | 131.175 | 5.49 | AHU | ||
| Leucina | Leu | yo | Alifático | No polar | Neutral | 3.8 | 131.175 | 9.68 | YUR, CUY [55] | ||
| Lisina | Lis | K | Catión | Ácido de Brønsted | Positivo | -3,9 | 146.189 | 5.19 | AAR | ||
| Metionina | Conocí | METRO | Tioéter | No polar | Neutral | 1.9 | 149.208 | 2.32 | AGO | ||
| Fenilalanina | fen | F | Aromático | No polar | Neutral | 2.8 | 257, 206, 188 | 0,2, 9,3, 60,0 | 165.192 | 3.87 | UUY |
| Prolina | Pro | PAG | Cíclico | No polar | Neutral | -1,6 | 115.132 | 5.02 | CCN | ||
| Serina | Ser | S | Hidroxílico | Polar | Neutral | -0,8 | 105.093 | 7.14 | UCN, AGY | ||
| Treonina | El | yo | Hidroxílico | Polar | Neutral | -0,7 | 119.119 | 5.53 | ACN | ||
| Triptófano | Trp | Yo | Aromático | No polar | Neutral | -0,9 | 280, 219 | 5.6, 47.0 | 204.228 | 1.25 | UGG |
| Tirosina | Tiro | Y | Aromático | Ácido de Brønsted | Neutral | -1,3 | 274, 222, 193 | 1.4, 8.0, 48.0 | 181.191 | 2.91 | UAY |
| Valina | Val | V | Alifático | No polar | Neutral | 4.2 | 117.148 | 6.73 | PISTOLA | ||
En algunas especies, dos aminoácidos adicionales están codificados por codones que normalmente se interpretan como codones de terminación :
| Aminoácidos 21 y 22 | 3 letras | 1 letra | Masa molecular |
|---|---|---|---|
| Selenocisteína | Segundo | tú | 168.064 |
| Pirrolisina | Pyl | Oh | 255.313 |
Además de los códigos de aminoácidos específicos, los marcadores de posición se utilizan en casos en los que el análisis químico o cristalográfico de un péptido o proteína no puede determinar de manera concluyente la identidad de un residuo. También se utilizan para resumir motivos conservados de secuencias proteicas . El uso de letras individuales para indicar conjuntos de residuos similares es similar al uso de códigos de abreviatura para bases degeneradas . [56] [57]
| Aminoácidos ambiguos | 3 letras | 1 letra | Aminoácidos incluidos | Codones incluidos |
|---|---|---|---|---|
| Cualquiera / desconocido | Xaa | incógnita | Todo | NNN |
| Asparagina o aspartato | Asc | B | Re, N | RAYO |
| Glutamina o glutamato | Gracias | O | mi, q | RAE |
| Leucina o isoleucina | Xle | Yo | Yo, yo | YTR, ATH, CTY [58] |
| Hidrofóbico | Φ | V, yo, l, f, w, y, m | NTN, TAY, TGG | |
| Aromático | Ohmio | F, W, Y, H | YWY, TTY, TGG [59] | |
| Alifático (no aromático) | O | V, yo, l, m | VTN, TTR [60] | |
| Pequeño | π | P, G, A, S | BCN, RGY, GGR | |
| Hidrofílico | o | S, T, H, N, Q, E, D, K, R | VAN, WCN, CGN, AGY [61] | |
| Cargado positivamente | + | K, R, H | ARR, LLORAR, CGR | |
| Cargado negativamente | − | Re, mi | Ganar |
A veces se utiliza Unk en lugar de Xaa , pero es menos estándar.
Ter o * (de terminación) se utiliza en la notación para las mutaciones en proteínas cuando aparece un codón de terminación. No corresponde a ningún aminoácido. [62]
Además, muchos aminoácidos no estándar tienen un código específico. Por ejemplo, varios fármacos peptídicos, como Bortezomib y MG132 , se sintetizan artificialmente y conservan sus grupos protectores , que tienen códigos específicos. Bortezomib es Pyz -Phe-boroLeu, y MG132 es Z -Leu-Leu-Leu-al. Para ayudar en el análisis de la estructura de las proteínas, están disponibles análogos de aminoácidos fotorreactivos . Estos incluyen fotoleucina ( pLeu ) y fotometionina ( pMet ). [63]
Aparición y funciones en bioquímica
Aminoácidos proteinogénicos
Los aminoácidos son los precursores de las proteínas. [26] Se unen mediante reacciones de condensación para formar cadenas de polímeros cortas llamadas péptidos o cadenas más largas llamadas polipéptidos o proteínas. Estas cadenas son lineales y no ramificadas, y cada residuo de aminoácido dentro de la cadena está unido a dos aminoácidos vecinos. En la naturaleza, el proceso de elaboración de proteínas codificadas por material genético de ARN se llama traducción e implica la adición paso a paso de aminoácidos a una cadena de proteína en crecimiento por una ribozima que se llama ribosoma . [64] El orden en el que se agregan los aminoácidos se lee a través del código genético a partir de una plantilla de ARNm , que es un ARN derivado de uno de los genes del organismo .
Veintidós aminoácidos se incorporan de forma natural a los polipéptidos y se denominan aminoácidos proteinogénicos o naturales. [28] De estos, 20 están codificados por el código genético universal. Los 2 restantes, selenocisteína y pirrolisina , se incorporan a las proteínas mediante mecanismos sintéticos únicos. La selenocisteína se incorpora cuando el ARNm que se está traduciendo incluye un elemento SECIS , que hace que el codón UGA codifique selenocisteína en lugar de un codón de terminación. [65] La pirrolisina es utilizada por algunas arqueas metanogénicas en enzimas que utilizan para producir metano . Está codificada con el codón UAG, que normalmente es un codón de terminación en otros organismos. [66]
Varios estudios evolutivos independientes han sugerido que Gly, Ala, Asp, Val, Ser, Pro, Glu, Leu, Thr pueden pertenecer a un grupo de aminoácidos que constituyeron el código genético temprano, mientras que Cys, Met, Tyr, Trp, His, Phe pueden pertenecer a un grupo de aminoácidos que constituyeron adiciones posteriores al código genético. [67] [68] [69]
Aminoácidos estándar y no estándar
Los 20 aminoácidos que están codificados directamente por los codones del código genético universal se denominan aminoácidos estándar o canónicos . Una forma modificada de metionina ( N -formilmetionina ) se incorpora a menudo en lugar de la metionina como aminoácido inicial de las proteínas en bacterias, mitocondrias y plástidos (incluidos los cloroplastos). Otros aminoácidos se denominan no estándar o no canónicos . La mayoría de los aminoácidos no estándar también son no proteinogénicos (es decir, no pueden incorporarse a las proteínas durante la traducción), pero dos de ellos son proteinogénicos, ya que pueden incorporarse traduccionalmente a las proteínas explotando información no codificada en el código genético universal.
Los dos aminoácidos proteinogénicos no estándar son la selenocisteína (presente en muchos no eucariotas así como en la mayoría de los eucariotas, pero no codificada directamente por el ADN) y la pirrolisina (que se encuentra solo en algunas arqueas y al menos en una bacteria ). La incorporación de estos aminoácidos no estándar es rara. Por ejemplo, 25 proteínas humanas incluyen selenocisteína en su estructura primaria, [70] y las enzimas caracterizadas estructuralmente (selenoenzimas) emplean selenocisteína como la fracción catalítica en sus sitios activos. [71] La pirrolisina y la selenocisteína se codifican a través de codones variantes. Por ejemplo, la selenocisteína está codificada por el codón de terminación y el elemento SECIS . [72] [73] [74]
La N -formilmetionina (que suele ser el aminoácido inicial de las proteínas en bacterias, mitocondrias y cloroplastos ) se considera generalmente una forma de metionina en lugar de un aminoácido proteinogénico separado. Las combinaciones de codón- ARNt que no se encuentran en la naturaleza también se pueden utilizar para "expandir" el código genético y formar nuevas proteínas conocidas como aloproteínas que incorporan aminoácidos no proteinogénicos . [75] [76] [77]
Aminoácidos no proteinogénicos
Además de los 22 aminoácidos proteinogénicos , se conocen muchos otros aminoácidos no proteinogénicos , que no se encuentran en las proteínas (por ejemplo, carnitina , GABA , levotiroxina ) o que no son producidos directamente y de forma aislada por la maquinaria celular estándar. Por ejemplo, la hidroxiprolina se sintetiza a partir de la prolina . Otro ejemplo es la selenometionina .
Los aminoácidos no proteinogénicos que se encuentran en las proteínas se forman mediante modificación postraduccional . Dichas modificaciones también pueden determinar la localización de la proteína, por ejemplo, la adición de grupos hidrófobos largos puede hacer que una proteína se una a una membrana fosfolipídica . [78] Ejemplos:
- La carboxilación del glutamato permite una mejor unión de los cationes calcio , [79]
- La hidroxiprolina , generada por hidroxilación de la prolina , es un componente principal del colágeno del tejido conectivo . [80]
- La hipusina, en el factor de iniciación de la traducción EIF5A , contiene una modificación de la lisina. [81]
Algunos aminoácidos no proteinogénicos no se encuentran en las proteínas. Algunos ejemplos incluyen el ácido 2-aminoisobutírico y el neurotransmisor ácido gamma-aminobutírico . Los aminoácidos no proteinogénicos a menudo aparecen como intermediarios en las vías metabólicas de los aminoácidos estándar; por ejemplo, la ornitina y la citrulina aparecen en el ciclo de la urea , parte del catabolismo de los aminoácidos (véase más adelante). [82] Una rara excepción al predominio de los α-aminoácidos en biología es el β-aminoácido beta alanina (ácido 3-aminopropanoico), que se utiliza en plantas y microorganismos en la síntesis de ácido pantoténico (vitamina B 5 ), un componente de la coenzima A . [83]
En la nutrición de los mamíferos
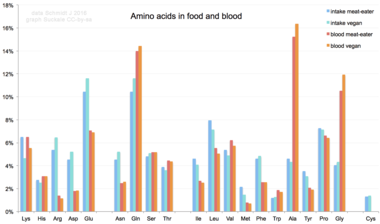
Los aminoácidos no son un componente típico de los alimentos: los animales comen proteínas. La proteína se descompone en aminoácidos en el proceso de digestión. Luego se utilizan para sintetizar nuevas proteínas, otras biomoléculas o se oxidan a urea y dióxido de carbono como fuente de energía. [84] La vía de oxidación comienza con la eliminación del grupo amino por una transaminasa ; luego, el grupo amino se introduce en el ciclo de la urea . El otro producto de la transamidación es un cetoácido que ingresa al ciclo del ácido cítrico . [85] Los aminoácidos glucogénicos también se pueden convertir en glucosa, a través de la gluconeogénesis . [86]
De los 20 aminoácidos estándar, nueve ( His , Ile , Leu , Lys , Met , Phe , Thr , Trp y Val ) se denominan aminoácidos esenciales porque el cuerpo humano no puede sintetizarlos a partir de otros compuestos al nivel necesario para el crecimiento normal, por lo que deben obtenerse de los alimentos. [87] [88] [89]
Aminoácidos semiesenciales y condicionalmente esenciales y requerimientos juveniles
Además, la cisteína, la tirosina y la arginina se consideran aminoácidos semiesenciales, y la taurina, un ácido aminosulfónico semiesencial en los niños. Algunos aminoácidos son condicionalmente esenciales para ciertas edades o condiciones médicas. Los aminoácidos esenciales también pueden variar de una especie a otra. [d] Las vías metabólicas que sintetizan estos monómeros no están completamente desarrolladas. [90] [91]
Funciones no proteicas
Las catecolaminas y las aminas traza se sintetizan a partir de la fenilalanina y la tirosina en los seres humanos. |
Muchos aminoácidos proteinogénicos y no proteinogénicos tienen funciones biológicas más allá de ser precursores de proteínas y péptidos. En los seres humanos, los aminoácidos también tienen papeles importantes en diversas vías biosintéticas. Las defensas contra los herbívoros en las plantas a veces emplean aminoácidos. [95] Ejemplos:
Aminoácidos estándar
- El triptófano es un precursor del neurotransmisor serotonina . [96]
- La tirosina (y su precursor, la fenilalanina) son precursores de los neurotransmisores catecolaminas dopamina , epinefrina y norepinefrina y de varias aminas traza .
- La fenilalanina es un precursor de la fenetilamina y la tirosina en los seres humanos. En las plantas, es un precursor de varios fenilpropanoides , que son importantes en el metabolismo de las plantas.
- La glicina es un precursor de las porfirinas como el hemo . [97]
- La arginina es un precursor del óxido nítrico . [98]
- La ornitina y la S -adenosilmetionina son precursoras de poliaminas . [99]
- El aspartato , la glicina y la glutamina son precursores de nucleótidos . [100] Sin embargo, no se conocen todas las funciones de otros aminoácidos no estándar abundantes.
Funciones de los aminoácidos no estándar
- La carnitina se utiliza en el transporte de lípidos .
- El ácido gamma-aminobutírico es un neurotransmisor. [101]
- El 5-HTP (5-hidroxitriptófano) se utiliza para el tratamiento experimental de la depresión. [102]
- L -DOPA ( L -dihidroxifenilalanina) para el tratamiento del Parkinson , [103]
- La eflornitina inhibe la ornitina descarboxilasa y se utiliza en el tratamiento de la enfermedad del sueño . [104]
- La canavanina , un análogo de la arginina que se encuentra en muchas legumbres , es un antialimentario que protege a la planta de los depredadores. [105]
- La mimosina, presente en algunas legumbres, es otro posible antialimentario . [106] Este compuesto es un análogo de la tirosina y puede envenenar a los animales que pastan estas plantas.
Usos en la industria
Alimento para animales
A veces se añaden aminoácidos a los alimentos para animales porque algunos de los componentes de estos alimentos, como la soja , tienen niveles bajos de algunos de los aminoácidos esenciales , especialmente de lisina, metionina, treonina y triptófano. [107] Asimismo, los aminoácidos se utilizan para quelar cationes metálicos con el fin de mejorar la absorción de minerales de los suplementos alimenticios. [108]
Alimento
La industria alimentaria es un importante consumidor de aminoácidos, especialmente ácido glutámico , que se utiliza como potenciador del sabor , [109] y aspartamo (éster 1-metílico de aspartilfenilalanina), que se utiliza como edulcorante artificial . [110] Los fabricantes a veces añaden aminoácidos a los alimentos para aliviar los síntomas de deficiencias minerales, como la anemia, mejorando la absorción de minerales y reduciendo los efectos secundarios negativos de la suplementación con minerales inorgánicos. [111]
Bloques de construcción químicos
Los aminoácidos son materias primas de bajo costo que se utilizan en la síntesis de grupos quirales como bloques de construcción enantioméricamente puros . [112] [113]
Los aminoácidos se utilizan en la síntesis de algunos cosméticos . [107]
Usos aspiracionales
Fertilizante
La capacidad quelante de los aminoácidos se utiliza a veces en fertilizantes para facilitar la llegada de minerales a las plantas con el fin de corregir deficiencias minerales, como la clorosis férrica. Estos fertilizantes también se utilizan para prevenir la aparición de deficiencias y mejorar la salud general de las plantas. [114]
Plásticos biodegradables
Los aminoácidos han sido considerados como componentes de polímeros biodegradables, que tienen aplicaciones como embalajes ecológicos y en medicina en la administración de fármacos y la construcción de implantes protésicos . [115] Un ejemplo interesante de tales materiales es el poliaspartato , un polímero biodegradable soluble en agua que puede tener aplicaciones en pañales desechables y en la agricultura. [116] Debido a su solubilidad y capacidad para quelar iones metálicos, el poliaspartato también se utiliza como un agente antical biodegradable y un inhibidor de la corrosión . [117] [118]
Síntesis

Síntesis química
La producción comercial de aminoácidos suele depender de bacterias mutantes que producen en exceso aminoácidos individuales utilizando la glucosa como fuente de carbono. Algunos aminoácidos se producen mediante conversiones enzimáticas de intermediarios sintéticos. El ácido 2-aminotiazolin-4-carboxílico es un intermediario en una síntesis industrial de L -cisteína , por ejemplo. El ácido aspártico se produce mediante la adición de amoníaco al fumarato utilizando una liasa. [111]
Biosíntesis
En las plantas, el nitrógeno se asimila primero en compuestos orgánicos en forma de glutamato , formado a partir de alfa-cetoglutarato y amoníaco en la mitocondria. Para otros aminoácidos, las plantas utilizan transaminasas para mover el grupo amino del glutamato a otro alfa-cetoácido. Por ejemplo, la aspartato aminotransferasa convierte el glutamato y el oxaloacetato en alfa-cetoglutarato y aspartato. [119] Otros organismos también utilizan transaminasas para la síntesis de aminoácidos.
Los aminoácidos no estándar se forman generalmente a través de modificaciones de los aminoácidos estándar. Por ejemplo, la homocisteína se forma a través de la vía de transulfuración o por la desmetilación de la metionina a través del metabolito intermedio S -adenosilmetionina [120] , mientras que la hidroxiprolina se forma mediante una modificación postraduccional de la prolina [121] .
Los microorganismos y las plantas sintetizan muchos aminoácidos poco comunes. Por ejemplo, algunos microbios producen ácido 2-aminoisobutírico y lantionina , que es un derivado de la alanina con puentes de sulfuro. Ambos aminoácidos se encuentran en lantibióticos peptídicos como la alameticina . [122] Sin embargo, en las plantas, el ácido 1-aminociclopropano-1-carboxílico es un pequeño aminoácido cíclico disustituido que es un intermediario en la producción de la hormona vegetal etileno . [123]
Síntesis primordial
Se supone que la formación de aminoácidos y péptidos precede y quizás induce el surgimiento de la vida en la Tierra . Los aminoácidos pueden formarse a partir de precursores simples bajo diversas condiciones. [124] El metabolismo químico superficial de aminoácidos y compuestos muy pequeños puede haber llevado a la acumulación de aminoácidos, coenzimas y pequeñas moléculas de carbono basadas en fosfato. [125] [ cita(s) adicional(es) necesaria(s) ] Los aminoácidos y bloques de construcción similares podrían haber sido elaborados en protopéptidos , y los péptidos se consideran actores clave en el origen de la vida. [126]
En el famoso experimento de Urey-Miller , el paso de un arco eléctrico a través de una mezcla de metano, hidrógeno y amoníaco produce una gran cantidad de aminoácidos. Desde entonces, los científicos han descubierto una variedad de formas y componentes por los cuales puede haber ocurrido la formación potencialmente prebiótica y la evolución química de los péptidos, como los agentes de condensación, el diseño de péptidos autorreplicantes y una serie de mecanismos no enzimáticos por los cuales los aminoácidos podrían haber surgido y elaborado hasta convertirse en péptidos. [126] Varias hipótesis invocan la síntesis de Strecker por la cual el cianuro de hidrógeno, aldehídos simples, amoníaco y agua producen aminoácidos. [124]
Según una revisión, los aminoácidos, e incluso los péptidos, "aparecen con bastante regularidad en los diversos caldos experimentales que se han dejado cocinar a partir de sustancias químicas simples. Esto se debe a que los nucleótidos son mucho más difíciles de sintetizar químicamente que los aminoácidos". Para un orden cronológico, sugiere que debe haber habido un "mundo proteínico" o al menos un "mundo polipeptídico", posiblemente seguido más tarde por el " mundo del ARN " y el " mundo del ADN ". [127] Los mapeos de codones -aminoácidos pueden ser el sistema de información biológica en el origen primordial de la vida en la Tierra. [128] Si bien los aminoácidos y, en consecuencia, los péptidos simples deben haberse formado en diferentes escenarios geoquímicos probados experimentalmente, la transición de un mundo abiótico a las primeras formas de vida aún está en gran medida sin resolver. [129]
Reacciones
Los aminoácidos experimentan las reacciones esperadas de los grupos funcionales constituyentes. [130] [131]
Formación de enlaces peptídicos

Como tanto los grupos amina como los ácidos carboxílicos de los aminoácidos pueden reaccionar para formar enlaces amida, una molécula de aminoácido puede reaccionar con otra y unirse a través de un enlace amida. Esta polimerización de aminoácidos es lo que crea las proteínas. Esta reacción de condensación produce el enlace peptídico recién formado y una molécula de agua. En las células, esta reacción no ocurre directamente; en cambio, el aminoácido se activa primero mediante la unión a una molécula de ARN de transferencia a través de un enlace éster . Este aminoacil-ARNt se produce en una reacción dependiente de ATP llevada a cabo por una aminoacil-ARNt sintetasa . [132] Este aminoacil-ARNt es entonces un sustrato para el ribosoma, que cataliza el ataque del grupo amino de la cadena proteica en elongación sobre el enlace éster. [133] Como resultado de este mecanismo, todas las proteínas producidas por los ribosomas se sintetizan comenzando en su extremo N y avanzando hacia su extremo C.
Sin embargo, no todos los enlaces peptídicos se forman de esta manera. En algunos casos, los péptidos son sintetizados por enzimas específicas. Por ejemplo, el tripéptido glutatión es una parte esencial de las defensas de las células contra el estrés oxidativo. Este péptido se sintetiza en dos pasos a partir de aminoácidos libres. [134] En el primer paso, la gamma-glutamilcisteína sintetasa condensa la cisteína y el glutamato a través de un enlace peptídico formado entre el carboxilo de la cadena lateral del glutamato (el carbono gamma de esta cadena lateral) y el grupo amino de la cisteína. Luego, este dipéptido es condensado con glicina por la glutatión sintetasa para formar glutatión. [135]
En química, los péptidos se sintetizan mediante una variedad de reacciones. Una de las más utilizadas en la síntesis de péptidos en fase sólida utiliza los derivados de oxima aromática de aminoácidos como unidades activadas. Estos se agregan en secuencia a la cadena peptídica en crecimiento, que está unida a un soporte de resina sólida. [136] Las bibliotecas de péptidos se utilizan en el descubrimiento de fármacos a través de un cribado de alto rendimiento . [137]
La combinación de grupos funcionales permite que los aminoácidos sean ligandos polidentados eficaces para quelatos de metal-aminoácido. [138] Las múltiples cadenas laterales de los aminoácidos también pueden sufrir reacciones químicas.
Catabolismo

* Glucogénicos , con los productos que tienen la capacidad de formar glucosa por gluconeogénesis
* Cetogénicos , con los productos que no tienen la capacidad de formar glucosa. Estos productos aún pueden usarse para la cetogénesis o la síntesis de lípidos .
* Aminoácidos catabolizados en productos glucogénicos y cetogénicos.
La degradación de un aminoácido a menudo implica la desaminación al mover su grupo amino a α-cetoglutarato, formando glutamato . Este proceso involucra transaminasas, a menudo las mismas que se usan en la aminación durante la síntesis. En muchos vertebrados, el grupo amino se elimina a través del ciclo de la urea y se excreta en forma de urea . Sin embargo, la degradación de aminoácidos puede producir ácido úrico o amoníaco en su lugar. Por ejemplo, la serina deshidratasa convierte la serina en piruvato y amoníaco. [100] Después de la eliminación de uno o más grupos amino, el resto de la molécula a veces se puede usar para sintetizar nuevos aminoácidos, o se puede usar para obtener energía ingresando a la glucólisis o al ciclo del ácido cítrico , como se detalla en la imagen de la derecha.
Complejización
Los aminoácidos son ligandos bidentados que forman complejos de aminoácidos con metales de transición . [140]
Análisis químico
El contenido total de nitrógeno de la materia orgánica está formado principalmente por los grupos amino de las proteínas. El nitrógeno total Kjeldahl ( TKN ) es una medida de nitrógeno ampliamente utilizada en el análisis de aguas (residuales), suelo, alimentos, piensos y materia orgánica en general. Como sugiere el nombre, se aplica el método Kjeldahl . Hay métodos más sensibles disponibles. [141] [142]
Véase también
Notas
- ^ El descubrimiento tardío se explica por el hecho de que la cisteína se oxida a cistina en el aire.
- ^ La prolina y otros aminoácidos cíclicos son una excepción a esta fórmula general. La ciclización del α-aminoácido crea la amina secundaria correspondiente. A veces se las denomina iminoácidos .
- ^ La convención L y D para la configuración de aminoácidos no se refiere a la actividad óptica del aminoácido en sí, sino a la actividad óptica del isómero de gliceraldehído a partir del cual ese aminoácido puede, en teoría, sintetizarse ( el D -gliceraldehído es dextrógiro; el L -gliceraldehído es levógiro). Una convención alternativa es utilizar los designadores ( S ) y ( R ) para especificar la configuración absoluta . [30] Casi todos los aminoácidos en las proteínas son ( S ) en el carbono α, siendo la cisteína ( R ) y la glicina no quirales . [31] La cisteína tiene su cadena lateral en la misma ubicación geométrica que los otros aminoácidos, pero la terminología R / S se invierte porque el azufre tiene un número atómico más alto en comparación con el oxígeno carboxílico, lo que le da a la cadena lateral una mayor prioridad según las reglas de secuencia de Cahn-Ingold-Prelog .
- ^ Por ejemplo, los rumiantes como las vacas obtienen una serie de aminoácidos a través de microbios en las dos primeras cámaras del estómago .
Referencias
- ^ Nelson DL, Cox MM (2005). Principios de bioquímica (4.ª ed.). Nueva York: W. H. Freeman. ISBN 0-7167-4339-6.
- ^ Flissi, Areski; Ricart, Emma; Campart, Clementine; Caballero, Mickael; Dufresne, Yoann; Michalik, Juraj; Jacques, Philippe; Flahaut, Christophe; Lisacek, Frédérique; Leclère, Valérie; Pupin, Maude (2020). "Norine: actualización del recurso de péptidos no ribosomales". Investigación de ácidos nucleicos . 48 (D1): D465-D469. doi :10.1093/nar/gkz1000. PMC 7145658 . PMID 31691799.
- ^ Richard Cammack, ed. (2009). «Newsletter 2009». Comité de Nomenclatura Bioquímica de la IUPAC y NC-IUBMB. Pirrolisina. Archivado desde el original el 12 de septiembre de 2017. Consultado el 16 de abril de 2012 .
- ^ Rother, Michael; Krzycki, Joseph A. (1 de enero de 2010). "Selenocisteína, pirrolisina y el metabolismo energético único de las arqueas metanogénicas". Archaea . 2010 : 1–14. doi : 10.1155/2010/453642 . ISSN 1472-3646. PMC 2933860 . PMID 20847933.
- ^ Latham MC (1997). «Capítulo 8. Composición corporal, funciones de los alimentos, metabolismo y energía». Nutrición humana en el mundo en desarrollo . Serie Alimentación y Nutrición – N.º 29. Roma: Organización de las Naciones Unidas para la Alimentación y la Agricultura. Archivado desde el original el 8 de octubre de 2012. Consultado el 9 de septiembre de 2012 .
- ^ Luisi, Pier Luigi (13 de julio de 2006). El surgimiento de la vida: de los orígenes químicos a la biología sintética. Cambridge University Press. p. 13. ISBN 9781139455640. Recuperado el 5 de agosto de 2024 .
Por supuesto, si en la Tierra solo hubiera habido dicetopiperazinas y no aminoácidos; o si los azúcares no tuvieran el tamaño que tienen; o si los lípidos fueran tres veces más cortos, entonces no tendríamos vida.
- ^ abc "Nomenclatura y simbolismo de aminoácidos y péptidos". Comisión Conjunta IUPAC-IUB sobre Nomenclatura Bioquímica. 1983. Archivado desde el original el 9 de octubre de 2008. Consultado el 17 de noviembre de 2008 .
- ^ Vickery HB, Schmidt CL (1931). "La historia del descubrimiento de los aminoácidos". Chem . Rev. 9 (2): 169–318. doi :10.1021/cr60033a001.
- ^ Hansen S (mayo de 2015). "Die Entdeckung der proteinogenen Aminosäuren von 1805 en París hasta 1935 en Illinois" (PDF) (en alemán). Berlina. Archivado desde el original (PDF) el 1 de diciembre de 2017.
- ^ Vauquelin LN, Robiquet PJ (1806). "El descubrimiento de un nuevo principio vegetal en Asparagus sativus". Anales de Chimié . 57 : 88–93.
- ^ ab Anfinsen CB, Edsall JT, Richards FM (1972). Avances en química de proteínas. Nueva York: Academic Press. pp. 99, 103. ISBN 978-0-12-034226-6.
- ^ Wollaston WH (1810). "Sobre el óxido quístico, una nueva especie de cálculo urinario". Philosophical Transactions of the Royal Society . 100 : 223–230. doi :10.1098/rstl.1810.0015. S2CID 110151163.
- ^ Baumann E (1884). «Über cytostin und cystein». Z Physiol Chem . 8 (4): 299–305. Archivado desde el original el 14 de marzo de 2011. Consultado el 28 de marzo de 2011 .
- ^ Braconnot HM (1820). "Sobre la conversión de matières animales en nuevas sustancias par le moyen de l'acide sulfurique". Annales de Chimie et de Physique . 2da Serie. 13 : 113-125.
- ^ Simoni RD, Hill RL, Vaughan M (septiembre de 2002). "El descubrimiento del aminoácido treonina: el trabajo de William C. Rose [artículo clásico]". The Journal of Biological Chemistry . 277 (37): E25. doi : 10.1016/S0021-9258(20)74369-3 . PMID 12218068. Archivado desde el original el 10 de junio de 2019 . Consultado el 4 de julio de 2015 .
- ^ McCoy RH, Meyer CE, Rose WC (1935). "Experimentos de alimentación con mezclas de aminoácidos altamente purificados. VIII. Aislamiento e identificación de un nuevo aminoácido esencial". Journal of Biological Chemistry . 112 : 283–302. doi : 10.1016/S0021-9258(18)74986-7 .
- ^ Menten, P. Dictionnaire de chimie: Une approche étymologique et historique . De Boeck, Bruselas. enlace Archivado el 28 de diciembre de 2019 en Wayback Machine .
- ^ Harper D. "amino-". Diccionario Etimológico Online . Archivado desde el original el 2 de diciembre de 2017. Consultado el 19 de julio de 2010 .
- ^ Paal C (1894). "Ueber die Einwirkung von Phenyl-i-cyanat auf organische Aminosäuren". Berichte der Deutschen Chemischen Gesellschaft . 27 : 974–979. doi :10.1002/cber.189402701205. Archivado desde el original el 25 de julio de 2020.
- ^ Fruton JS (1990). "Capítulo 5: Emil Fischer y Franz Hofmeister". Contrastes en el estilo científico: grupos de investigación en ciencias químicas y bioquímicas . Vol. 191. Sociedad Filosófica Americana. págs. 163-165. ISBN 978-0-87169-191-0.
- ^ "Alpha amino acid". Merriam-Webster Medical . Archivado desde el original el 3 de enero de 2015. Consultado el 3 de enero de 2015 ..
- ^ Clark, Jim (agosto de 2007). "Introducción a los aminoácidos". chemguide . Archivado desde el original el 30 de abril de 2015 . Consultado el 4 de julio de 2015 .
- ^ Jakubke HD, Sewald N (2008). "Aminoácidos". Péptidos de la A a la Z: una enciclopedia concisa . Alemania: Wiley-VCH. p. 20. ISBN 9783527621170. Archivado desde el original el 17 de mayo de 2016 . Consultado el 5 de enero de 2016 – a través de Google Books.
- ^ Pollegioni L, Servi S, eds. (2012). Aminoácidos no naturales: métodos y protocolos . Métodos en biología molecular. Vol. 794. Humana Press. p. v. doi :10.1007/978-1-61779-331-8. ISBN . 978-1-61779-331-8. OCLC 756512314. S2CID 3705304.
- ^ Hertweck C (octubre de 2011). "Biosíntesis y carga de pirrolisina, el 22.º aminoácido codificado genéticamente". Angewandte Chemie International Edition . 50 (41): 9540–9541. doi :10.1002/anie.201103769. PMID 21796749. S2CID 5359077.

- ^ ab "Capítulo 1: Las proteínas son las moléculas trabajadoras del cuerpo". Las estructuras de la vida . Instituto Nacional de Ciencias Médicas Generales. 27 de octubre de 2011. Archivado desde el original el 7 de junio de 2014 . Consultado el 20 de mayo de 2008 .
- ^ Michal G, Schomburg D, eds. (2012). Vías bioquímicas: un atlas de bioquímica y biología molecular (2.ª ed.). Oxford: Wiley-Blackwell. pág. 5. ISBN 978-0-470-14684-2.
- ^ abc Creighton TH (1993). "Capítulo 1" . Proteínas: estructuras y propiedades moleculares . San Francisco: WH Freeman. ISBN 978-0-7167-7030-5.
- ^ Genchi, Giuseppe (1 de septiembre de 2017). "Una visión general de los d-aminoácidos". Aminoácidos . 49 (9): 1521–1533. doi :10.1007/s00726-017-2459-5. ISSN 1438-2199. PMID 28681245. S2CID 254088816.
- ^ Cahn, RS ; Ingold, CK ; Prelog, V. (1966). "Especificación de la quiralidad molecular". Angewandte Chemie International Edition . 5 (4): 385–415. doi :10.1002/anie.196603851.
- ^ Hatem SM (2006). "Determinación por cromatografía de gases de enantiómeros de aminoácidos en tabaco y vinos embotellados". Universidad de Giessen. Archivado desde el original el 22 de enero de 2009. Consultado el 17 de noviembre de 2008 .
- ^ abcdef Garrett, Reginald H.; Grisham, Charles M. (2010). Bioquímica (4.ª ed.). Belmont, CA: Brooks/Cole, Cengage Learning. págs. 74, 134–176, 430–442. ISBN 978-0-495-10935-8.OCLC 297392560 .
- ^ Novikov, Anton P.; Safonov, Alexey V.; German, Konstantin E.; Grigoriev, Mikhail S. (1 de diciembre de 2023). "Qué tipo de interacciones podemos obtener al pasar de iones zwitter a iones "dritter": las interacciones anión-anión C–O⋯Re(O4) y Re–O⋯Re(O4) marcan una diferencia estructural entre el perrenato de L-histidinio y el pertecnetato". CrystEngComm . 26 : 61–69. doi :10.1039/D3CE01164J. ISSN 1466-8033. S2CID 265572280.
- ^ Papp, Laura Vanda; Lu, Jun; Holmgren, Arne; Khanna, Kum Kum (1 de julio de 2007). "Del selenio a las selenoproteínas: síntesis, identidad y su papel en la salud humana". Antioxidantes y señalización redox . 9 (7): 775–806. doi :10.1089/ars.2007.1528. ISSN 1523-0864. PMID 17508906.
- ^ Hao, Bing; Gong, Weimin; Ferguson, Tsuneo K.; James, Carey M.; Krzycki, José A.; Chan, Michael K. (24 de mayo de 2002). "Un nuevo residuo codificado por UAG en la estructura de una metanógeno metiltransferasa". Ciencia . 296 (5572): 1462-1466. Código bibliográfico : 2002 Ciencia... 296.1462H. doi : 10.1126/ciencia.1069556. ISSN 0036-8075. PMID 12029132. S2CID 35519996.
- ^ Steinhardt, J.; Reynolds, JA (1969). Equilibrios múltiples en proteínas . Nueva York: Academic Press. pp. 176–21. ISBN 978-0126654509.
- ^ Brønsted, JN (1923). "Einige Bemerkungen über den Begriff der Säuren und Basen" [Observaciones sobre el concepto de ácidos y bases]. Recueil des Travaux Chimiques des Pays-Bas . 42 (8): 718–728. doi :10.1002/recl.19230420815.
- ^ ab Vollhardt, K. Peter C. (2007). Química orgánica: estructura y función. Neil Eric Schore (5.ª ed.). Nueva York: WH Freeman. págs. 58–66. ISBN 978-0-7167-9949-8.OCLC 61448218 .
- ^ Fennema OR (19 de junio de 1996). Química de los alimentos, 3.ª edición . CRC Press. págs. 327-328. ISBN 978-0-8247-9691-4.
- ^ ab Ntountoumi C, Vlastaridis P, Mossialos D, Stathopoulos C, Iliopoulos I, Promponas V, et al. (noviembre de 2019). "Las regiones de baja complejidad en las proteínas de los procariotas desempeñan papeles funcionales importantes y están altamente conservadas". Nucleic Acids Research . 47 (19): 9998–10009. doi :10.1093/nar/gkz730. PMC 6821194 . PMID 31504783.
- ^ Urry DW (2004). "El cambio en la energía libre de Gibbs para la asociación hidrofóbica: Derivación y evaluación por medio de transiciones inversas de temperatura". Chemical Physics Letters . 399 (1–3): 177–183. Bibcode :2004CPL...399..177U. doi :10.1016/S0009-2614(04)01565-9.
- ^ Marcotte EM, Pellegrini M, Yeates TO, Eisenberg D (octubre de 1999). "Un censo de repeticiones de proteínas". Revista de biología molecular . 293 (1): 151–60. doi :10.1006/jmbi.1999.3136. PMID 10512723.
- ^ Haerty W, Golding GB (octubre de 2010). Bonen L (ed.). "Secuencias de baja complejidad y repeticiones de un solo aminoácido: no solo secuencias de péptidos "basura"". Genome . 53 (10): 753–62. doi :10.1139/G10-063. PMID 20962881.
- ^ Magee T, Seabra MC (abril de 2005). "Acilación y prenilación de proteínas grasas: lo que está de moda en las grasas". Current Opinion in Cell Biology . 17 (2): 190–196. doi :10.1016/j.ceb.2005.02.003. PMID 15780596.
- ^ Pilobello KT, Mahal LK (junio de 2007). "Descifrando el glicocódigo: la complejidad y el desafío analítico de la glicomics". Current Opinion in Chemical Biology . 11 (3): 300–305. doi :10.1016/j.cbpa.2007.05.002. PMID 17500024.
- ^ Smotrys JE, Linder ME (2004). "Palmitoilación de proteínas de señalización intracelular: regulación y función". Revista Anual de Bioquímica . 73 (1): 559–587. doi :10.1146/annurev.biochem.73.011303.073954. PMID 15189153.
- ^ abcde "Comisión IUPAC-IUB sobre nomenclatura bioquímica: una notación de una letra para secuencias de aminoácidos". Journal of Biological Chemistry . 243 (13): 3557–3559. 10 de julio de 1968. doi : 10.1016/S0021-9258(19)34176-6 .
- ^ ab Saffran, M. (abril de 1998). "Nombres de aminoácidos y juegos de salón: desde nombres triviales hasta códigos de una sola letra, los nombres de aminoácidos han puesto a prueba la memoria de los estudiantes. ¿Es posible una nomenclatura más racional?". Educación bioquímica . 26 (2): 116–118. doi :10.1016/S0307-4412(97)00167-2.
- ^ abc Adoga, Godwin I; Nicholson, Bh (enero de 1988). "Cartas al editor". Educación bioquímica . 16 (1): 49. doi :10.1016/0307-4412(88)90026-X.
- ^ Kyte J, Doolittle RF (mayo de 1982). "Un método simple para mostrar el carácter hidropático de una proteína". Journal of Molecular Biology . 157 (1): 105–132. CiteSeerX 10.1.1.458.454 . doi :10.1016/0022-2836(82)90515-0. PMID 7108955.
- ^ Freifelder D (1983). Bioquímica física (2.ª ed.). WH Freeman and Company. ISBN 978-0-7167-1315-9.[ página necesaria ]
- ^ Kozlowski LP (enero de 2017). "Proteome-pI: base de datos de puntos isoeléctricos del proteoma". Nucleic Acids Research . 45 (D1): D1112–D1116. doi :10.1093/nar/gkw978. PMC 5210655 . PMID 27789699.
- ^ ab Hausman RE, Cooper GM (2004). La célula: un enfoque molecular . Washington, DC: ASM Press. p. 51. ISBN 978-0-87893-214-6.
- ^ Los codones también pueden expresarse mediante: CGN, AGR
- ^ Los codones también pueden expresarse mediante: CUN, UUR
- ^ Aasland R, Abrams C, Ampe C, Ball LJ, Bedford MT, Cesareni G, Gimona M, Hurley JH, Jarchau T, Lehto VP, Lemmon MA, Linding R, Mayer BJ, Nagai M, Sudol M, Walter U, Winder SJ (febrero de 2002). "Normalización de la nomenclatura de motivos peptídicos como ligandos de dominios proteicos modulares". FEBS Letters . 513 (1): 141–144. doi :10.1111/j.1432-1033.1968.tb00350.x. PMID 11911894.
- ^ Comisión de Nomenclatura Bioquímica de la IUPAC-IUB (1972). "Una notación de una letra para secuencias de aminoácidos". Química Pura y Aplicada . 31 (4): 641–645. doi : 10.1351/pac197231040639 . PMID 5080161.
- ^ Los codones también se pueden expresar mediante: CTN, ATH, TTR; MTY, YTR, ATA; MTY, HTA, YTG
- ^ Los codones también pueden expresarse mediante: TWY, CAY, TGG
- ^ Los codones también pueden expresarse mediante: NTR, VTY
- ^ Los codones también pueden expresarse mediante: VAN, WCN, MGY, CGP
- ^ "HGVS: nomenclatura de variantes de secuencia, recomendaciones de proteínas". Archivado desde el original el 24 de septiembre de 2021 . Consultado el 23 de septiembre de 2021 .
- ^ Suchanek M, Radzikowska A, Thiele C (abril de 2005). "La fotoleucina y la fotometionina permiten la identificación de interacciones proteína-proteína en células vivas". Nature Methods . 2 (4): 261–267. doi : 10.1038/nmeth752 . PMID 15782218.
- ^ Rodnina MV, Beringer M, Wintermeyer W (enero de 2007). "Cómo los ribosomas forman enlaces peptídicos". Tendencias en ciencias bioquímicas . 32 (1): 20–26. doi :10.1016/j.tibs.2006.11.007. PMID 17157507.
- ^ Driscoll DM, Copeland PR (2003). "Mecanismo y regulación de la síntesis de selenoproteína". Revista Anual de Nutrición . 23 (1): 17–40. doi :10.1146/annurev.nutr.23.011702.073318. PMID 12524431.
- ^ Krzycki JA (diciembre de 2005). "La codificación genética directa de la pirrolisina". Current Opinion in Microbiology . 8 (6): 706–712. doi :10.1016/j.mib.2005.10.009. PMID 16256420.
- ^ Wong, JT-F. (1975). "Una teoría de coevolución del código genético". Actas de la Academia Nacional de Ciencias . 72 (5): 1909–1912. Bibcode :1975PNAS...72.1909T. doi : 10.1073/pnas.72.5.1909 . PMC 432657 . PMID 1057181.
- ^ Trifonov EN (diciembre de 2000). "Orden temporal de consenso de aminoácidos y evolución del código de tripletes". Gene . 261 (1): 139–151. doi :10.1016/S0378-1119(00)00476-5. PMID 11164045.
- ^ Higgs PG, Pudritz RE (junio de 2009). "Una base termodinámica para la síntesis de aminoácidos prebióticos y la naturaleza del primer código genético". Astrobiología . 9 (5): 483–90. arXiv : 0904.0402 . Bibcode :2009AsBio...9..483H. doi :10.1089/ast.2008.0280. PMID 19566427. S2CID 9039622.
- ^ Kryukov GV, Castellano S, Novoselov SV, Lobanov AV, Zehtab O, Guigó R, Gladyshev VN (mayo de 2003). «Caracterización de los selenoproteomas de los mamíferos». Science . 300 (5624): 1439–1443. Bibcode :2003Sci...300.1439K. doi :10.1126/science.1083516. PMID 12775843. S2CID 10363908. Archivado desde el original el 23 de julio de 2018 . Consultado el 12 de junio de 2019 .
- ^ Gromer S, Urig S, Becker K (enero de 2004). "El sistema de tiorredoxina: de la ciencia a la clínica". Medicinal Research Reviews . 24 (1): 40–89. doi :10.1002/med.10051. PMID 14595672. S2CID 1944741.
- ^ Tjong H (2008). Modelado de las contribuciones electrostáticas al plegamiento y unión de proteínas (tesis doctoral). Florida State University. p. 1 nota al pie. Archivado desde el original el 28 de enero de 2020 . Consultado el 28 de enero de 2020 .
- ^ Stewart L, Burgin AB (2005). "Síntesis de genes completos: un futuro Gene-O-Matic". Fronteras en el diseño y descubrimiento de fármacos . 1 . Bentham Science Publishers : 299. doi :10.2174/1574088054583318. ISBN 978-1-60805-199-1. ISSN 1574-0889. Archivado desde el original el 14 de abril de 2021 . Consultado el 5 de enero de 2016 .
- ^ Elzanowski A, Ostell J (7 de abril de 2008). «The Genetic Codes». Centro Nacional de Información Biotecnológica (NCBI). Archivado desde el original el 20 de agosto de 2016. Consultado el 10 de marzo de 2010 .
- ^ Xie J, Schultz PG (diciembre de 2005). "Adición de aminoácidos al repertorio genético". Current Opinion in Chemical Biology . 9 (6): 548–554. doi :10.1016/j.cbpa.2005.10.011. PMID 16260173.
- ^ Wang Q, Parrish AR, Wang L (marzo de 2009). "Expansión del código genético para estudios biológicos". Química y biología . 16 (3): 323–336. doi :10.1016/j.chembiol.2009.03.001. PMC 2696486 . PMID 19318213.
- ^ Simon M (2005). Computación emergente: énfasis en la bioinformática . Nueva York: AIP Press/Springer Science+Business Media. pp. 105–106. ISBN 978-0-387-22046-8.
- ^ Blenis J, Resh MD (diciembre de 1993). "Localización subcelular especificada por la acilación y fosforilación de proteínas". Current Opinion in Cell Biology . 5 (6): 984–989. doi :10.1016/0955-0674(93)90081-Z. PMID 8129952.
- ^ Vermeer C (marzo de 1990). "Proteínas que contienen gamma-carboxiglutamato y la carboxilasa dependiente de la vitamina K". The Biochemical Journal . 266 (3): 625–636. doi :10.1042/bj2660625. PMC 1131186 . PMID 2183788.
- ^ Bhattacharjee A, Bansal M (marzo de 2005). "Estructura del colágeno: la triple hélice de Madrás y el escenario actual". IUBMB Life . 57 (3): 161–172. doi :10.1080/15216540500090710. PMID 16036578. S2CID 7211864.
- ^ Park MH (febrero de 2006). "La síntesis postraduccional de un aminoácido derivado de poliamina, la hipusina, en el factor de iniciación de la traducción eucariota 5A (eIF5A)". Journal of Biochemistry . 139 (2): 161–169. doi :10.1093/jb/mvj034. PMC 2494880 . PMID 16452303.
- ^ Curis E, Nicolis I, Moinard C, Osowska S, Zerrouk N, Bénazeth S, Cynober L (noviembre de 2005). "Casi todo sobre citrulina en mamíferos". Aminoácidos . 29 (3): 177–205. doi :10.1007/s00726-005-0235-4. PMID 16082501. S2CID 23877884.
- ^ Coxon KM, Chakauya E, Ottenhof HH, Whitney HM, Blundell TL, Abell C, Smith AG (agosto de 2005). "Biosíntesis de pantotenato en plantas superiores". Biochemical Society Transactions . 33 (Pt 4): 743–746. doi :10.1042/BST0330743. PMID 16042590.
- ^ Sakami W, Harrington H (1963). "Metabolismo de aminoácidos". Revista Anual de Bioquímica . 32 (1): 355–398. doi :10.1146/annurev.bi.32.070163.002035. PMID 14144484.
- ^ Brosnan JT (abril de 2000). "Glutamato, en la interfaz entre el metabolismo de los aminoácidos y los carbohidratos". The Journal of Nutrition . 130 (4S Suppl): 988S–990S. doi : 10.1093/jn/130.4.988S . PMID 10736367.
- ^ Young VR, Ajami AM (septiembre de 2001). "Glutamina: ¿el emperador o su ropa?". The Journal of Nutrition . 131 (9 Suppl): 2449S–2459S, 2486S–2487S. doi : 10.1093/jn/131.9.2449S . PMID 11533293.
- ^ Young VR (agosto de 1994). "Requerimientos de aminoácidos en adultos: argumentos a favor de una revisión importante de las recomendaciones actuales". The Journal of Nutrition . 124 (8 Suppl): 1517S–1523S. doi : 10.1093/jn/124.suppl_8.1517S . PMID 8064412.
- ^ Fürst P, Stehle P (junio de 2004). "¿Cuáles son los elementos esenciales necesarios para la determinación de los requerimientos de aminoácidos en humanos?". The Journal of Nutrition . 134 (6 Suppl): 1558S–1565S. doi : 10.1093/jn/134.6.1558S . PMID 15173430.
- ^ Reeds PJ (julio de 2000). "Aminoácidos prescindibles e indispensables para los seres humanos". The Journal of Nutrition . 130 (7): 1835S–1840S. doi : 10.1093/jn/130.7.1835S . PMID 10867060.
- ^ Imura K, Okada A (enero de 1998). "Metabolismo de aminoácidos en pacientes pediátricos". Nutrition . 14 (1): 143–148. doi :10.1016/S0899-9007(97)00230-X. PMID 9437700.
- ^ Lourenço R, Camilo ME (2002). "Taurina: ¿un aminoácido condicionalmente esencial en humanos? Una visión general en salud y enfermedad". Nutricion Hospitalaria . 17 (6): 262–270. PMID 12514918.
- ^ Broadley KJ (marzo de 2010). "Los efectos vasculares de las trazas de aminas y anfetaminas". Farmacología y terapéutica . 125 (3): 363–375. doi :10.1016/j.pharmthera.2009.11.005. PMID 19948186.
- ^ Lindemann L, Hoener MC (mayo de 2005). "Un renacimiento de las aminas traza inspirado en una nueva familia de GPCR". Tendencias en ciencias farmacológicas . 26 (5): 274–281. doi :10.1016/j.tips.2005.03.007. PMID 15860375.
- ^ Wang X, Li J, Dong G, Yue J (febrero de 2014). "Los sustratos endógenos del CYP2D cerebral". Revista Europea de Farmacología . 724 : 211–218. doi :10.1016/j.ejphar.2013.12.025. PMID 24374199.
- ^ Hylin JW (1969). "Péptidos y aminoácidos tóxicos en alimentos y piensos". Revista de química agrícola y alimentaria . 17 (3): 492–496. doi :10.1021/jf60163a003.
- ^ Savelieva KV, Zhao S, Pogorelov VM, Rajan I, Yang Q, Cullinan E, Lanthorn TH (2008). Bartolomucci A (ed.). "La alteración genética de ambos genes de la triptófano hidroxilasa reduce drásticamente la serotonina y afecta el comportamiento en modelos sensibles a los antidepresivos". PLOS ONE . 3 (10): e3301. Bibcode :2008PLoSO...3.3301S. doi : 10.1371/journal.pone.0003301 . PMC 2565062 . PMID 18923670.
- ^ Shemin D, Rittenberg D (diciembre de 1946). "La utilización biológica de la glicina para la síntesis de la protoporfirina de la hemoglobina". The Journal of Biological Chemistry . 166 (2): 621–625. doi : 10.1016/S0021-9258(17)35200-6 . PMID 20276176. Archivado desde el original el 7 de mayo de 2022 . Consultado el 3 de noviembre de 2008 .
- ^ Tejero J, Biswas A, Wang ZQ, Page RC, Haque MM, Hemann C, Zweier JL, Misra S, Stuehr DJ (noviembre de 2008). "Estabilización y caracterización de un intermediario de reacción de hemo-oxi en la óxido nítrico sintasa inducible". The Journal of Biological Chemistry . 283 (48): 33498–33507. doi : 10.1074/jbc.M806122200 . PMC 2586280 . PMID 18815130.
- ^ Rodríguez-Caso C, Montañez R, Cascante M, Sánchez-Jiménez F, Medina MA (agosto de 2006). "Modelado matemático del metabolismo de poliaminas en mamíferos". The Journal of Biological Chemistry . 281 (31): 21799–21812. doi : 10.1074/jbc.M602756200 . hdl : 10630/32289 . PMID 16709566.
- ^ ab Stryer L, Berg JM, Tymoczko JL (2002). Bioquímica (5ª ed.). Nueva York: WH Freeman. págs. 693–698. ISBN 978-0-7167-4684-3.
- ^ Petroff OA (diciembre de 2002). "GABA y glutamato en el cerebro humano". The Neuroscientist . 8 (6): 562–573. doi :10.1177/1073858402238515. PMID 12467378. S2CID 84891972.
- ^ Turner EH, Loftis JM, Blackwell AD (marzo de 2006). "Serotonina a la carta: suplementación con el precursor de serotonina 5-hidroxitriptófano". Farmacología y terapéutica . 109 (3): 325–338. doi :10.1016/j.pharmthera.2005.06.004. PMID 16023217. S2CID 2563606. Archivado desde el original el 13 de abril de 2020. Consultado el 12 de junio de 2019 .
- ^ Kostrzewa RM, Nowak P, Kostrzewa JP, Kostrzewa RA, Brus R (marzo de 2005). "Peculiaridades del tratamiento con L-DOPA de la enfermedad de Parkinson". Aminoácidos . 28 (2): 157–164. doi :10.1007/s00726-005-0162-4. PMID 15750845. S2CID 33603501.
- ^ Heby O, Persson L, Rentala M (agosto de 2007). "Enzimas biosintéticas de poliaminas dirigidas a la enfermedad del sueño africana, la enfermedad de Chagas y la leishmaniasis". Amino Acids . 33 (2): 359–366. doi :10.1007/s00726-007-0537-9. PMID 17610127. S2CID 26273053.
- ^ Rosenthal GA (2001). "L-canavanina: un aleloquímico insecticida de plantas superiores". Aminoácidos . 21 (3): 319–330. doi :10.1007/s007260170017. PMID 11764412. S2CID 3144019.
- ^ Hammond, Andrew C. (1 de mayo de 1995). "Toxicosis por Leucaena y su control en rumiantes". Journal of Animal Science . 73 (5): 1487–1492. doi :10.2527/1995.7351487x. PMID 7665380. Archivado desde el original el 7 de mayo de 2022 . Consultado el 7 de mayo de 2022 .
- ^ ab Leuchtenberger W, Huthmacher K, Drauz K (noviembre de 2005). "Producción biotecnológica de aminoácidos y derivados: estado actual y perspectivas". Applied Microbiology and Biotechnology . 69 (1): 1–8. doi :10.1007/s00253-005-0155-y. PMID 16195792. S2CID 24161808.
- ^ Ashmead HD (1993). El papel de los quelatos de aminoácidos en la nutrición animal . Westwood: Noyes Publications.
- ^ Garattini S (abril de 2000). "Ácido glutámico, veinte años después". The Journal of Nutrition . 130 (4S Suppl): 901S–909S. doi : 10.1093/jn/130.4.901S . PMID 10736350.
- ^ Stegink LD (julio de 1987). "La historia del aspartamo: un modelo para la prueba clínica de un aditivo alimentario". The American Journal of Clinical Nutrition . 46 (1 Suppl): 204–215. doi :10.1093/ajcn/46.1.204. PMID 3300262.
- ^ ab Drauz K, Grayson I, Kleemann A, Krimmer HP, Leuchtenberger W, Weckbecker C (2007). "Aminoácidos". Enciclopedia de química industrial de Ullmann . Weinheim: Wiley-VCH. doi :10.1002/14356007.a02_057.pub2. ISBN 978-3527306732.
- ^ Hanessian S (1993). "Reflexiones sobre la síntesis total de productos naturales: arte, artesanía, lógica y el enfoque de Chiron". Química pura y aplicada . 65 (6): 1189–1204. doi : 10.1351/pac199365061189 . S2CID 43992655.
- ^ Blaser HU (1992). "El pool quiral como fuente de catalizadores enantioselectivos y auxiliares". Chemical Reviews . 92 (5): 935–952. doi :10.1021/cr00013a009.
- ^ Ashmead HD (1986). Alimentación foliar de plantas con quelatos de aminoácidos . Park Ridge: Noyes Publications.
- ^ Sanda F, Endo T (1999). "Síntesis y funciones de polímeros basados en aminoácidos". Química y física macromolecular . 200 (12): 2651–2661. doi : 10.1002/(SICI)1521-3935(19991201)200:12<2651::AID-MACP2651>3.0.CO;2-P .
- ^ Gross RA, Kalra B (agosto de 2002). «Polímeros biodegradables para el medio ambiente». Science . 297 (5582): 803–807. Bibcode :2002Sci...297..803G. doi :10.1126/science.297.5582.803. PMID 12161646. Archivado desde el original el 25 de julio de 2020 . Consultado el 12 de junio de 2019 .
- ^ Low KC, Wheeler AP, Koskan LP (1996). Ácido poliaspártico comercial y sus usos . Serie Avances en química. Vol. 248. Washington, DC: American Chemical Society .
- ^ Thombre SM, Sarwade BD (2005). "Síntesis y biodegradabilidad del ácido poliaspártico: una revisión crítica". Journal of Macromolecular Science, Parte A. 42 ( 9): 1299–1315. doi :10.1080/10601320500189604. S2CID 94818855.
- ^ Jones RC, Buchanan BB, Gruissem W (2000). Bioquímica y biología molecular de las plantas. Rockville, Maryland: Sociedad Estadounidense de Fisiólogos Vegetales. pp. 371–372. ISBN 978-0-943088-39-6.
- ^ Brosnan JT, Brosnan ME (junio de 2006). "Los aminoácidos que contienen azufre: una descripción general". The Journal of Nutrition . 136 (6 Suppl): 1636S–1640S. doi : 10.1093/jn/136.6.1636S . PMID 16702333.
- ^ Kivirikko KI, Pihlajaniemi T (1998). "Colágeno hidroxilasas y la subunidad de la proteína disulfuro isomerasa de las prolil 4-hidroxilasas". Avances en enzimología y áreas relacionadas de la biología molecular . Avances en enzimología y áreas relacionadas de la biología molecular. Vol. 72. págs. 325–398. doi :10.1002/9780470123188.ch9. ISBN 9780470123188. Número de identificación personal 9559057.
- ^ Whitmore L, Wallace BA (mayo de 2004). "Análisis de la composición de la secuencia de peptaibol: implicaciones para la síntesis in vivo y la formación de canales". Revista Europea de Biofísica . 33 (3): 233–237. doi :10.1007/s00249-003-0348-1. PMID 14534753. S2CID 24638475.
- ^ Alexander L, Grierson D (octubre de 2002). "Biosíntesis y acción del etileno en el tomate: un modelo para la maduración de frutos climatéricos". Journal of Experimental Botany . 53 (377): 2039–2055. doi : 10.1093/jxb/erf072 . PMID 12324528.
- ^ ab Kitadai, Norio; Maruyama, Shigenori (2018). "Orígenes de los componentes básicos de la vida: una revisión". Fronteras de la geociencia . 9 (4): 1117-1153. Código Bib : 2018GeoFr...9.1117K. doi : 10.1016/j.gsf.2017.07.007 . S2CID 102659869.
- ^ Danchin, Antoine (12 de junio de 2017). "Del metabolismo químico a la vida: el origen del proceso de codificación genética". Beilstein Journal of Organic Chemistry . 13 (1): 1119–1135. doi :10.3762/bjoc.13.111. ISSN 1860-5397. PMC 5480338 . PMID 28684991.
- ^ ab Frenkel-Pinter, Moran; Samanta, Mousumi; Ashkenasy, Gonen; Leman, Luke J. (10 de junio de 2020). "Péptidos prebióticos: centros moleculares en el origen de la vida" . Chemical Reviews . 120 (11): 4707–4765. doi :10.1021/acs.chemrev.9b00664. ISSN 0009-2665. PMID 32101414. S2CID 211536416.
- ^ Milner-White, E. James (6 de diciembre de 2019). "Estructuras tridimensionales de proteínas en el origen de la vida". Interface Focus . 9 (6): 20190057. doi :10.1098/rsfs.2019.0057. PMC 6802138 . PMID 31641431.
- ^ Chatterjee, Sankar; Yadav, Surya (junio de 2022). "La coevolución de biomoléculas y sistemas de información prebióticos en el origen de la vida: un modelo de visualización para ensamblar el primer gen". Vida . 12 (6): 834. Bibcode :2022Life...12..834C. doi : 10.3390/life12060834 . ISSN 2075-1729. PMC 9225589 . PMID 35743865.
- ^ Kirschning, Andreas (26 de mayo de 2021). "El par coenzima/proteína y la evolución molecular de la vida". Natural Product Reports . 38 (5): 993–1010. doi : 10.1039/D0NP00037J . ISSN 1460-4752. PMID 33206101. S2CID 227037164.
- ^ Elmore DT, Barrett GC (1998). Aminoácidos y péptidos . Cambridge, Reino Unido: Cambridge University Press. pp. 48–60. ISBN 978-0-521-46827-5.
- ^ Gutteridge A, Thornton JM (noviembre de 2005). "Entender el conjunto de herramientas catalíticas de la naturaleza". Tendencias en ciencias bioquímicas . 30 (11): 622–629. doi :10.1016/j.tibs.2005.09.006. PMID 16214343.
- ^ Ibba M, Söll D (mayo de 2001). "El renacimiento de la síntesis de aminoacil-ARNt". EMBO Reports . 2 (5): 382–387. doi :10.1093/embo-reports/kve095. PMC 1083889 . PMID 11375928.
- ^ Lengyel P, Söll D (junio de 1969). "Mecanismo de la biosíntesis de proteínas". Bacteriological Reviews . 33 (2): 264–301. doi :10.1128/MMBR.33.2.264-301.1969. PMC 378322 . PMID 4896351.
- ^ Wu G, Fang YZ, Yang S, Lupton JR, Turner ND (marzo de 2004). "Metabolismo del glutatión y sus implicaciones para la salud". The Journal of Nutrition . 134 (3): 489–492. doi : 10.1093/jn/134.3.489 . PMID 14988435.
- ^ Meister A (noviembre de 1988). "Metabolismo del glutatión y su modificación selectiva". The Journal of Biological Chemistry . 263 (33): 17205–17208. doi : 10.1016/S0021-9258(19)77815-6 . PMID 3053703.
- ^ Carpino LA (1992). "1-Hidroxi-7-azabenzotriazol. Un aditivo de acoplamiento de péptidos eficiente". Revista de la Sociedad Química Americana . 115 (10): 4397–4398. doi :10.1021/ja00063a082.
- ^ Marasco D, Perretta G, Sabatella M, Ruvo M (octubre de 2008). "Perspectivas pasadas y futuras de las bibliotecas de péptidos sintéticos". Current Protein & Peptide Science . 9 (5): 447–467. doi :10.2174/138920308785915209. PMID 18855697.
- ^ Konara S, Gagnona K, Clearfield A, Thompson C, Hartle J, Ericson C, Nelson C (2010). "Determinación estructural y caracterización de bis-glicinatos de cobre y zinc con cristalografía de rayos X y espectrometría de masas". Journal of Coordination Chemistry . 63 (19): 3335–3347. doi :10.1080/00958972.2010.514336. S2CID 94822047.
- ^ Stipanuk MH (2006). Aspectos bioquímicos, fisiológicos y moleculares de la nutrición humana (2.ª ed.). Saunders Elsevier.
- ^ Dghaym RD, Dhawan R, Arndtsen BA (septiembre de 2001). "El uso de monóxido de carbono e iminas como sintones derivados de péptidos: una síntesis fácil catalizada por paladio de imidazolinas derivadas de α-aminoácidos". Angewandte Chemie . 40 (17): 3228–3230. doi :10.1002/(SICI)1521-3773(19980703)37:12<1634::AID-ANIE1634>3.0.CO;2-C. PMID 29712039.
- ^ Muñoz-Huerta RF, Guevara-Gonzalez RG, Contreras-Medina LM, Torres-Pacheco I, Prado-Olivarez J, Ocampo-Velazquez RV (agosto de 2013). "Una revisión de los métodos para detectar el estado del nitrógeno en las plantas: ventajas, desventajas y avances recientes". Sensores . 13 (8). Basilea, Suiza: 10823–43. Bibcode :2013Senso..1310823M. doi : 10.3390/s130810823 . PMC 3812630 . PMID 23959242.
- ^ Martin PD, Malley DF, Manning G, Fuller L (2002). "Determinación del carbono orgánico y nitrógeno del suelo a nivel de campo utilizando espectroscopia de infrarrojo cercano". Revista Canadiense de Ciencias del Suelo . 82 (4): 413–422. doi :10.4141/S01-054.
Lectura adicional
- Tymoczko JL (2012). "Composición y estructura de las proteínas". Bioquímica . Nueva York: WH Freeman and company. págs. 28-31. ISBN. 9781429229364.
- Doolittle RF (1989). "Redundancias en secuencias de proteínas". En Fasman GD (ed.). Predicciones de la estructura de las proteínas y los principios de la conformación de las proteínas . Nueva York: Plenum Press . pp. 599–623. ISBN 978-0-306-43131-9. Código LCCN 89008555.
- Nelson DL, Cox MM (2000). Principios de bioquímica de Lehninger (3.ª ed.). Worth Publishers . ISBN 978-1-57259-153-0. Número de serie LCCN 99049137.
- Meierhenrich U (2008). Aminoácidos y la asimetría de la vida (PDF) . Berlín: Springer Verlag . ISBN 978-3-540-76885-2. LCCN 2008930865. Archivado desde el original (PDF) el 12 de enero de 2012.



