Bacteria
| Bacteria | |
|---|---|
.jpg/440px-E._coli_Bacteria_(7316101966).jpg) | |
| Micrografía electrónica de barrido de bacilos de Escherichia coli | |
| Clasificación científica | |
| Dominio: | Bacterias Woese et al. 1990 |
| Filos | |
Véase § Phyla | |
| Sinónimos | |
| |
Bacterias ( / b æ k ˈ t ɪər i ə / ;sg.: bacteria) son organismos ubicuos, en su mayoría de vida libre, que a menudo consisten en unacélula biológica. Constituyen un grandominiodemicroorganismosprocariotas . Por lo general, de unos pocosmicrómetrosde longitud, las bacterias estuvieron entre las primeras formas de vida que aparecieron enla Tierray están presentes en la mayoría de sushábitats. Las bacterias habitan el suelo, el agua,las fuentes termales ácidas,los desechos radiactivosy labiosfera profundadecorteza terrestre. Las bacterias desempeñan un papel vital en muchas etapas delciclo de los nutrientesal reciclar nutrientes y lafijación de nitrógenode laatmósfera. El ciclo de los nutrientes incluye ladescomposicióndecadáveres; las bacterias son responsables de lade putrefacciónen este proceso. En las comunidades biológicas que rodeanlos respiraderos hidrotermalesylas filtraciones frías,extremófilasproporcionan los nutrientes necesarios para mantener la vida al convertir compuestos disueltos, comoel sulfuro de hidrógenoyel metano, en energía. Las bacterias también viven enmutualistas,comensalesyparasitariascon plantas y animales. La mayoría de las bacterias no han sido caracterizadas y hay muchas especies que no se puedencultivaren el laboratorio. El estudio de las bacterias se conoce comobacteriología, una rama dela microbiología.
Al igual que todos los animales, los seres humanos son portadores de una gran cantidad de bacterias (aproximadamente entre 10 13 y 10 14 ). [2] La mayoría se encuentran en el intestino , aunque hay muchas en la piel. La mayoría de las bacterias que se encuentran en el interior y exterior del cuerpo son inofensivas o se vuelven inofensivas gracias a los efectos protectores del sistema inmunitario , y muchas son beneficiosas , [3] en particular las que se encuentran en el intestino. Sin embargo, varias especies de bacterias son patógenas y causan enfermedades infecciosas , como el cólera , la sífilis , el ántrax , la lepra , la tuberculosis , el tétanos y la peste bubónica . Las enfermedades bacterianas mortales más comunes son las infecciones respiratorias . Los antibióticos se utilizan para tratar las infecciones bacterianas y también se utilizan en la agricultura, lo que hace que la resistencia a los antibióticos sea un problema creciente. Las bacterias son importantes en el tratamiento de aguas residuales y la descomposición de derrames de petróleo , la producción de queso y yogur mediante fermentación , la recuperación de oro, paladio , cobre y otros metales en el sector minero ( biominería , biolixiviación ), así como en la biotecnología y la fabricación de antibióticos y otros productos químicos.
Las bacterias , que en el pasado eran plantas que formaban parte de la clase Schizomycetes ("hongos de fisión"), ahora se clasifican como procariotas . A diferencia de las células de los animales y otros eucariotas , las células bacterianas no contienen un núcleo y rara vez albergan orgánulos rodeados de membranas . Aunque el término bacteria incluía tradicionalmente a todos los procariotas, la clasificación científica cambió después del descubrimiento en la década de 1990 de que los procariotas consisten en dos grupos muy diferentes de organismos que evolucionaron a partir de un ancestro común antiguo . Estos dominios evolutivos se denominan Bacteria y Archaea . [4]
Etimología

La palabra bacteria es el plural del neolatino bacterium , que es la latinización del griego antiguo βακτήριον ( baktḗrion ), [5] el diminutivo de βακτηρία ( baktēría ), que significa "bastón, bastón", [6] porque los primeros que se descubrieron tenían forma de bastón . [7] [8]
Origen y evolución temprana

Los ancestros de las bacterias eran microorganismos unicelulares que fueron las primeras formas de vida que aparecieron en la Tierra, hace unos 4 mil millones de años. [10] Durante unos 3 mil millones de años, la mayoría de los organismos eran microscópicos, y las bacterias y las arqueas eran las formas de vida dominantes. [11] [12] [13] Aunque existen fósiles bacterianos , como los estromatolitos , su falta de morfología distintiva impide que se utilicen para examinar la historia de la evolución bacteriana o para fechar el momento del origen de una especie bacteriana en particular. Sin embargo, las secuencias genéticas se pueden utilizar para reconstruir la filogenia bacteriana , y estos estudios indican que las bacterias divergieron primero del linaje arqueal/eucariota. [14] El ancestro común más reciente (MRCA) de bacterias y arqueas fue probablemente un hipertermófilo que vivió hace unos 2500 millones a 3200 millones de años. [15] [16] [17] La vida más temprana en la tierra puede haber sido la de las bacterias hace unos 3220 millones de años. [18]
Las bacterias también estuvieron involucradas en la segunda gran divergencia evolutiva, la de las arqueas y los eucariotas. [19] [20] Aquí, los eucariotas resultaron de la entrada de bacterias antiguas en asociaciones endosimbióticas con los ancestros de las células eucariotas, que posiblemente estaban relacionadas con las arqueas. [21] [22] Esto implicó la fagocitosis por parte de células protoeucariotas de simbiontes alfaproteobacterianos para formar mitocondrias o hidrogenosomas , que todavía se encuentran en todos los Eukarya conocidos (a veces en forma altamente reducida , por ejemplo, en los antiguos protozoos "amitocondriales"). Más tarde, algunos eucariotas que ya contenían mitocondrias también fagocitaron organismos similares a las cianobacterias , lo que llevó a la formación de cloroplastos en algas y plantas. Esto se conoce como endosimbiosis primaria . [23]
Hábitat
Las bacterias son omnipresentes y viven en todos los hábitats posibles del planeta, incluido el suelo, el agua, las profundidades de la corteza terrestre e incluso en entornos tan extremos como las fuentes termales ácidas y los desechos radiactivos. [24] [25] Se cree que hay aproximadamente 2×10 30 bacterias en la Tierra, [26] formando una biomasa que solo es superada por las plantas. [27] Son abundantes en lagos y océanos, en el hielo ártico y en los manantiales geotermales [28] donde proporcionan los nutrientes necesarios para sustentar la vida al convertir compuestos disueltos, como el sulfuro de hidrógeno y el metano , en energía. [29] Viven sobre y en plantas y animales. La mayoría no causan enfermedades, son beneficiosas para sus entornos y son esenciales para la vida. [3] [30] El suelo es una rica fuente de bacterias y unos pocos gramos contienen alrededor de mil millones de ellas. Todas son esenciales para la ecología del suelo, descomponiendo los desechos tóxicos y reciclando los nutrientes. Incluso se encuentran en la atmósfera y un metro cúbico de aire contiene alrededor de cien millones de células bacterianas. Los océanos y los mares albergan alrededor de 3 x 10 26 bacterias que proporcionan hasta el 50% del oxígeno que respiramos los humanos. [31] Solo alrededor del 2% de las especies bacterianas han sido completamente estudiadas. [32]
| Hábitat | Especies | Referencia |
|---|---|---|
| Frío (menos 15 °C en la Antártida) | Criptoendolitos | [33] |
| Caliente ( géiseres de 70–100 °C ) | Termo acuático | [32] |
| Radiación, 5M Rad | Deinococo radiodurans | [33] |
| Salina, 47% de sal ( Mar Muerto , Gran Lago Salado ) | varias especies | [32] [33] |
| pH ácido 3 | varias especies | [24] |
| pH alcalino 12,8 | betaproteobacteria | [33] |
| Espacio (6 años en un satélite de la NASA ) | Bacillus subtilis | [33] |
| 3,2 km bajo tierra | varias especies | [33] |
| Alta presión ( Fosa de las Marianas – 1200 atm ) | Moritella, Shewanella y otras | [33] |
Morfología
Tamaño . Las bacterias presentan una amplia diversidad de formas y tamaños. Las células bacterianas tienen aproximadamente una décima parte del tamaño de las células eucariotas y suelen tener una longitud de entre 0,5 y 5,0 micrómetros . Sin embargo, algunas especies son visibles a simple vista; por ejemplo, Thiomargarita namibiensis mide hasta medio milímetro de largo, [34] Epulopiscium fishelsoni alcanza los 0,7 mm, [35] y Thiomargarita magnifica puede alcanzar incluso los 2 cm de longitud, que es 50 veces más grande que otras bacterias conocidas. [36] [37] Entre las bacterias más pequeñas se encuentran los miembros del género Mycoplasma , que miden solo 0,3 micrómetros, tan pequeños como los virus más grandes . [38] Algunas bacterias pueden ser incluso más pequeñas, pero estas ultramicrobacterias no están bien estudiadas. [39]
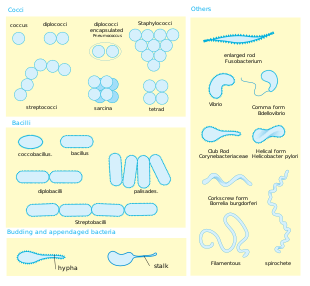
Forma . La mayoría de las especies bacterianas son esféricas, llamadas cocos ( singular coccus , del griego kókkos , grano, semilla), o con forma de bastón, llamadas bacilos ( singular bacillus, del latín baculus , palo). [40] Algunas bacterias, llamadas vibrio , tienen forma de bastón ligeramente curvado o de coma; otras pueden tener forma de espiral, llamadas espirilas , o estar muy enrolladas, llamadas espiroquetas . Se han descrito una pequeña cantidad de otras formas inusuales, como las bacterias con forma de estrella. [41] Esta amplia variedad de formas está determinada por la pared celular bacteriana y el citoesqueleto y es importante porque puede influir en la capacidad de las bacterias para adquirir nutrientes, adherirse a superficies, nadar a través de líquidos y escapar de los depredadores . [42] [43]

Multicelularidad . La mayoría de las especies bacterianas existen como células individuales; otras se asocian en patrones característicos: Neisseria forma diploides (pares), los estreptococos forman cadenas y los estafilococos se agrupan en grupos de "racimos de uvas". Las bacterias también pueden agruparse para formar estructuras multicelulares más grandes, como los filamentos alargados de las especies de Actinomycetota , los agregados de las especies de Myxobacteria y las hifas complejas de las especies de Streptomyces . [45] Estas estructuras multicelulares a menudo solo se ven en ciertas condiciones. Por ejemplo, cuando carecen de aminoácidos, las mixobacterias detectan las células circundantes en un proceso conocido como detección de quórum , migran unas hacia otras y se agregan para formar cuerpos fructíferos de hasta 500 micrómetros de largo y que contienen aproximadamente 100.000 células bacterianas. [46] En estos cuerpos fructíferos, las bacterias realizan tareas separadas; Por ejemplo, aproximadamente una de cada diez células migran a la parte superior de un cuerpo fructífero y se diferencian en un estado latente especializado llamado mixospora, que es más resistente a la desecación y otras condiciones ambientales adversas. [47]
Biopelículas . Las bacterias a menudo se adhieren a las superficies y forman agregaciones densas llamadas biopelículas [48] y formaciones más grandes conocidas como tapetes microbianos . [49] Estas biopelículas y tapetes pueden variar desde unos pocos micrómetros de espesor hasta medio metro de profundidad, y pueden contener múltiples especies de bacterias, protistas y arqueas. Las bacterias que viven en biopelículas muestran una disposición compleja de células y componentes extracelulares, formando estructuras secundarias, como microcolonias , a través de las cuales hay redes de canales para permitir una mejor difusión de nutrientes. [50] [51] En entornos naturales, como el suelo o las superficies de las plantas, la mayoría de las bacterias están unidas a las superficies en biopelículas. [52] Las biopelículas también son importantes en medicina, ya que estas estructuras a menudo están presentes durante infecciones bacterianas crónicas o en infecciones de dispositivos médicos implantados , y las bacterias protegidas dentro de las biopelículas son mucho más difíciles de matar que las bacterias individuales aisladas. [53]
Estructura celular

Estructuras intracelulares
La célula bacteriana está rodeada por una membrana celular , que está hecha principalmente de fosfolípidos . Esta membrana encierra el contenido de la célula y actúa como una barrera para retener nutrientes, proteínas y otros componentes esenciales del citoplasma dentro de la célula. [54] A diferencia de las células eucariotas , las bacterias generalmente carecen de grandes estructuras unidas a la membrana en su citoplasma, como un núcleo , mitocondrias , cloroplastos y otros orgánulos presentes en las células eucariotas. [55] Sin embargo, algunas bacterias tienen orgánulos unidos a proteínas en el citoplasma que compartimentan aspectos del metabolismo bacteriano, [56] [57] como el carboxisoma . [58] Además, las bacterias tienen un citoesqueleto de múltiples componentes para controlar la localización de proteínas y ácidos nucleicos dentro de la célula, y para gestionar el proceso de división celular . [59] [60] [61]
Muchas reacciones bioquímicas importantes , como la generación de energía, ocurren debido a gradientes de concentración a través de las membranas, lo que crea una diferencia de potencial análoga a una batería. La falta general de membranas internas en las bacterias significa que estas reacciones, como el transporte de electrones , ocurren a través de la membrana celular entre el citoplasma y el exterior de la célula o periplasma . [62] Sin embargo, en muchas bacterias fotosintéticas, la membrana plasmática está muy plegada y llena la mayor parte de la célula con capas de membrana que recogen luz. [63] Estos complejos que recogen luz pueden incluso formar estructuras encerradas en lípidos llamadas clorosomas en las bacterias verdes del azufre . [64]

Las bacterias no tienen un núcleo rodeado por una membrana y su material genético es típicamente un único cromosoma bacteriano circular de ADN ubicado en el citoplasma en un cuerpo de forma irregular llamado nucleoide . [65] El nucleoide contiene el cromosoma con sus proteínas asociadas y ARN . Al igual que todos los demás organismos , las bacterias contienen ribosomas para la producción de proteínas, pero la estructura del ribosoma bacteriano es diferente a la de los eucariotas y las arqueas. [66]
Algunas bacterias producen gránulos de almacenamiento de nutrientes intracelulares, como glucógeno , [67] polifosfato , [68] azufre [69] o polihidroxialcanoatos . [70] Bacterias como las cianobacterias fotosintéticas , producen vacuolas de gas internas , que utilizan para regular su flotabilidad, lo que les permite moverse hacia arriba o hacia abajo en capas de agua con diferentes intensidades de luz y niveles de nutrientes. [71]
Estructuras extracelulares
Alrededor del exterior de la membrana celular se encuentra la pared celular . Las paredes celulares bacterianas están hechas de peptidoglicano (también llamado mureína), que está hecho de cadenas de polisacáridos reticuladas por péptidos que contienen D- aminoácidos . [72] Las paredes celulares bacterianas son diferentes de las paredes celulares de las plantas y los hongos , que están hechas de celulosa y quitina , respectivamente. [73] La pared celular de las bacterias también es distinta de la de las aqueas, que no contienen peptidoglicano. La pared celular es esencial para la supervivencia de muchas bacterias, y el antibiótico penicilina (producido por un hongo llamado Penicillium ) es capaz de matar bacterias al inhibir un paso en la síntesis de peptidoglicano. [73]
En líneas generales, existen dos tipos diferentes de pared celular en las bacterias, que las clasifican en bacterias Gram-positivas y bacterias Gram-negativas . Los nombres se originan a partir de la reacción de las células a la tinción de Gram , una prueba de larga data para la clasificación de especies bacterianas. [74]
Las bacterias grampositivas poseen una pared celular gruesa que contiene muchas capas de peptidoglicano y ácidos teicoicos . Por el contrario, las bacterias gramnegativas tienen una pared celular relativamente delgada que consiste en unas pocas capas de peptidoglicano rodeadas por una segunda membrana lipídica que contiene lipopolisacáridos y lipoproteínas . La mayoría de las bacterias tienen la pared celular gramnegativa, y solo los miembros del grupo Bacillota y actinomycetota (anteriormente conocidas como bacterias grampositivas de bajo G+C y alto G+C, respectivamente) tienen la disposición grampositiva alternativa. [75] Estas diferencias en la estructura pueden producir diferencias en la susceptibilidad a los antibióticos; por ejemplo, la vancomicina puede matar solo bacterias grampositivas y es ineficaz contra patógenos gramnegativos , como Haemophilus influenzae o Pseudomonas aeruginosa . [76] Algunas bacterias tienen estructuras de pared celular que no son ni clásicamente grampositivas ni gramnegativas. Esto incluye bacterias clínicamente importantes como las micobacterias, que tienen una pared celular gruesa de peptidoglicano como una bacteria Gram-positiva, pero también una segunda capa externa de lípidos. [77]
En muchas bacterias, una capa S de moléculas de proteína dispuestas de manera rígida cubre el exterior de la célula. [78] Esta capa proporciona protección química y física para la superficie celular y puede actuar como una barrera de difusión macromolecular . Las capas S tienen diversas funciones y se sabe que actúan como factores de virulencia en las especies de Campylobacter y contienen enzimas de superficie en Bacillus stearothermophilus . [79] [80]
Los flagelos son estructuras proteicas rígidas, de unos 20 nanómetros de diámetro y hasta 20 micrómetros de longitud, que se utilizan para la movilidad . Los flagelos son impulsados por la energía liberada por la transferencia de iones a través de un gradiente electroquímico a través de la membrana celular. [81]
Las fimbrias (a veces llamadas " pili de unión ") son filamentos finos de proteína, generalmente de 2 a 10 nanómetros de diámetro y hasta varios micrómetros de longitud. Se distribuyen sobre la superficie de la célula y se parecen a pelos finos cuando se observan bajo el microscopio electrónico . [82] Se cree que las fimbrias están involucradas en la unión a superficies sólidas o a otras células, y son esenciales para la virulencia de algunos patógenos bacterianos. [83] Los pili ( sing . pilus) son apéndices celulares, ligeramente más grandes que las fimbrias, que pueden transferir material genético entre células bacterianas en un proceso llamado conjugación , donde se denominan pili de conjugación o pili sexuales (ver genética bacteriana, a continuación). [84] También pueden generar movimiento, donde se denominan pili tipo IV . [85]
Muchas bacterias producen glicocálix para rodear sus células [86] y su complejidad estructural varía: desde una capa viscosa desorganizada de sustancias poliméricas extracelulares hasta una cápsula altamente estructurada . Estas estructuras pueden proteger a las células de ser absorbidas por células eucariotas como los macrófagos (parte del sistema inmunológico humano ). [87] También pueden actuar como antígenos y participar en el reconocimiento celular, además de ayudar a la adhesión a superficies y la formación de biopelículas. [88]
El ensamblaje de estas estructuras extracelulares depende de los sistemas de secreción bacterianos , que transfieren proteínas desde el citoplasma al periplasma o al entorno que rodea a la célula. Se conocen muchos tipos de sistemas de secreción y estas estructuras suelen ser esenciales para la virulencia de los patógenos, por lo que se estudian intensamente. [88]
Endosporas
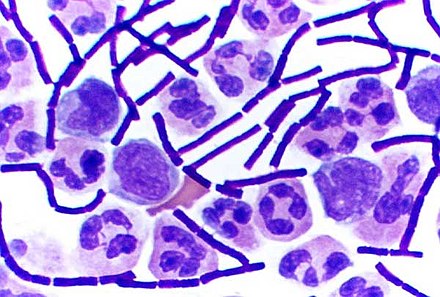
Algunos géneros de bacterias Gram-positivas, como Bacillus , Clostridium , Sporohalobacter , Anaerobacter y Heliobacterium , pueden formar estructuras latentes y altamente resistentes llamadas endosporas . [90] Las endosporas se desarrollan dentro del citoplasma de la célula; generalmente, se desarrolla una sola endospora en cada célula. [91] Cada endospora contiene un núcleo de ADN y ribosomas rodeado por una capa de corteza y protegido por una capa rígida multicapa compuesta de peptidoglicano y una variedad de proteínas. [91]
Las endosporas no muestran un metabolismo detectable y pueden sobrevivir a tensiones físicas y químicas extremas, como altos niveles de luz ultravioleta , radiación gamma , detergentes , desinfectantes , calor, congelación, presión y desecación . [92] En este estado latente, estos organismos pueden permanecer viables durante millones de años. [93] [94] [95] Las endosporas incluso permiten que las bacterias sobrevivan a la exposición al vacío y la radiación del espacio exterior , lo que lleva a la posibilidad de que las bacterias puedan distribuirse por todo el Universo mediante polvo espacial , meteoroides , asteroides , cometas , planetoides o panspermia dirigida . [96] [97]
Las bacterias formadoras de endosporas pueden causar enfermedades; por ejemplo, el ántrax se puede contraer por la inhalación de endosporas de Bacillus anthracis , y la contaminación de heridas punzantes profundas con endosporas de Clostridium tetani causa tétanos , que, como el botulismo , es causado por una toxina liberada por las bacterias que crecen a partir de las esporas. [98] La infección por Clostridioides difficile , un problema común en los entornos de atención médica, es causada por bacterias formadoras de esporas. [99]
Metabolismo
Las bacterias exhiben una variedad extremadamente amplia de tipos metabólicos . [100] La distribución de rasgos metabólicos dentro de un grupo de bacterias se ha utilizado tradicionalmente para definir su taxonomía , pero estos rasgos a menudo no corresponden con las clasificaciones genéticas modernas. [101] El metabolismo bacteriano se clasifica en grupos nutricionales sobre la base de tres criterios principales: la fuente de energía , los donadores de electrones utilizados y la fuente de carbono utilizada para el crecimiento. [102]
Las bacterias fototróficas obtienen energía de la luz mediante la fotosíntesis , mientras que las bacterias quimiotróficas descomponen compuestos químicos mediante oxidación , [103] impulsando el metabolismo mediante la transferencia de electrones de un donante de electrones determinado a un aceptor terminal de electrones en una reacción redox . Los quimiotrofos se dividen además por los tipos de compuestos que utilizan para transferir electrones. Las bacterias que obtienen electrones de compuestos inorgánicos como el hidrógeno, el monóxido de carbono o el amoníaco se denominan litotrofos , mientras que las que utilizan compuestos orgánicos se denominan organotrofos . [103] Aún así, más específicamente, los organismos aeróbicos utilizan el oxígeno como aceptor terminal de electrones, mientras que los organismos anaeróbicos utilizan otros compuestos como el nitrato , el sulfato o el dióxido de carbono. [103]
Muchas bacterias, llamadas heterótrofas , obtienen su carbono de otro carbono orgánico . Otras, como las cianobacterias y algunas bacterias púrpuras , son autótrofas , lo que significa que obtienen carbono celular fijando dióxido de carbono . [104] En circunstancias inusuales, el gas metano puede ser utilizado por bacterias metanotróficas como fuente de electrones y como sustrato para el anabolismo del carbono . [105]
| Tipo nutricional | Fuente de energía | Fuente de carbono | Ejemplos |
|---|---|---|---|
| Fotótrofos | Luz del sol | Compuestos orgánicos (fotoheterótrofos) o fijación de carbono (fotoautótrofos) | Cianobacterias , bacterias verdes del azufre , cloroflexotas o bacterias púrpuras |
| Litótrofos | Compuestos inorgánicos | Compuestos orgánicos (litoheterótrofos) o fijación de carbono (litoautótrofos) | Thermodesulfobacteriota , Hydrogenophilaceae o Nitrospirota |
| Organótrofos | Compuestos orgánicos | Compuestos orgánicos (quimioheterótrofos) o fijación de carbono (quimioautótrofos) | Bacillus , Clostridium o Enterobacteriaceae |
En muchos sentidos, el metabolismo bacteriano proporciona rasgos que son útiles para la estabilidad ecológica y para la sociedad humana. Por ejemplo, los diazótrofos tienen la capacidad de fijar el gas nitrógeno utilizando la enzima nitrogenasa . [106] Este rasgo, que se puede encontrar en bacterias de la mayoría de los tipos metabólicos enumerados anteriormente, [107] conduce a los procesos ecológicamente importantes de desnitrificación , reducción de sulfato y acetogénesis , respectivamente. [108] Los procesos metabólicos bacterianos son impulsores importantes en las respuestas biológicas a la contaminación ; por ejemplo, las bacterias reductoras de sulfato son en gran medida responsables de la producción de las formas altamente tóxicas de mercurio ( metil- y dimetilmercurio ) en el medio ambiente. [109] Los anaerobios no respiratorios utilizan la fermentación para generar energía y poder reductor, secretando subproductos metabólicos (como el etanol en la elaboración de cerveza) como desechos. Los anaerobios facultativos pueden cambiar entre la fermentación y diferentes aceptores terminales de electrones dependiendo de las condiciones ambientales en las que se encuentren. [110]
Crecimiento y reproducción



A diferencia de los organismos multicelulares, los aumentos en el tamaño celular ( crecimiento celular ) y la reproducción por división celular están estrechamente vinculados en los organismos unicelulares. Las bacterias crecen hasta un tamaño fijo y luego se reproducen a través de fisión binaria , una forma de reproducción asexual . [112] En condiciones óptimas, las bacterias pueden crecer y dividirse extremadamente rápido, y algunas poblaciones bacterianas pueden duplicarse tan rápido como cada 17 minutos. [113] En la división celular, se producen dos células hijas de clones idénticos . Algunas bacterias, mientras todavía se reproducen asexualmente, forman estructuras reproductivas más complejas que ayudan a dispersar las células hijas recién formadas. Los ejemplos incluyen la formación del cuerpo fructífero por mixobacterias y la formación de hifas aéreas por especies de Streptomyces , o gemación. La gemación implica que una célula forme una protuberancia que se desprende y produce una célula hija. [114]
En el laboratorio, las bacterias se cultivan generalmente utilizando medios sólidos o líquidos. [115] Los medios de crecimiento sólidos , como las placas de agar , se utilizan para aislar cultivos puros de una cepa bacteriana. Sin embargo, los medios de crecimiento líquidos se utilizan cuando se requiere la medición del crecimiento o de grandes volúmenes de células. El crecimiento en medios líquidos agitados se produce como una suspensión celular uniforme, lo que hace que los cultivos sean fáciles de dividir y transferir, aunque es difícil aislar bacterias individuales de los medios líquidos. El uso de medios selectivos (medios con nutrientes específicos añadidos o deficientes, o con antibióticos añadidos) puede ayudar a identificar organismos específicos. [116]
La mayoría de las técnicas de laboratorio para el cultivo de bacterias utilizan altos niveles de nutrientes para producir grandes cantidades de células de forma barata y rápida. [115] Sin embargo, en entornos naturales, los nutrientes son limitados, lo que significa que las bacterias no pueden seguir reproduciéndose indefinidamente. Esta limitación de nutrientes ha llevado a la evolución de diferentes estrategias de crecimiento (véase la teoría de selección r/K ). Algunos organismos pueden crecer extremadamente rápido cuando los nutrientes están disponibles, como la formación de floraciones de algas y cianobacterias que a menudo ocurren en lagos durante el verano. [117] Otros organismos tienen adaptaciones a entornos hostiles, como la producción de múltiples antibióticos por Streptomyces que inhiben el crecimiento de microorganismos competidores. [118] En la naturaleza, muchos organismos viven en comunidades (por ejemplo, biopelículas ) que pueden permitir un mayor suministro de nutrientes y protección contra el estrés ambiental. [52] Estas relaciones pueden ser esenciales para el crecimiento de un organismo o grupo de organismos en particular ( sintrofia ). [119]
El crecimiento bacteriano sigue cuatro fases. Cuando una población de bacterias ingresa por primera vez a un entorno con alto contenido de nutrientes que permite el crecimiento, las células necesitan adaptarse a su nuevo entorno. La primera fase de crecimiento es la fase de retraso , un período de crecimiento lento cuando las células se adaptan al entorno con alto contenido de nutrientes y se preparan para un crecimiento rápido. La fase de retraso tiene altas tasas de biosíntesis, ya que se producen las proteínas necesarias para un crecimiento rápido. [120] [121] La segunda fase de crecimiento es la fase logarítmica , también conocida como fase exponencial. La fase logarítmica está marcada por un crecimiento exponencial rápido . La velocidad a la que crecen las células durante esta fase se conoce como tasa de crecimiento ( k ), y el tiempo que tardan las células en duplicarse se conoce como tiempo de generación ( g ). Durante la fase logarítmica, los nutrientes se metabolizan a máxima velocidad hasta que uno de los nutrientes se agota y comienza a limitar el crecimiento. La tercera fase de crecimiento es la fase estacionaria y es causada por nutrientes agotados. Las células reducen su actividad metabólica y consumen proteínas celulares no esenciales. La fase estacionaria es una transición de un crecimiento rápido a un estado de respuesta al estrés y hay una mayor expresión de genes involucrados en la reparación del ADN , el metabolismo antioxidante y el transporte de nutrientes . [122] La fase final es la fase de muerte donde las bacterias se quedan sin nutrientes y mueren. [123]
Genética

La mayoría de las bacterias tienen un solo cromosoma circular que puede variar en tamaño desde solo 160.000 pares de bases en la bacteria endosimbiótica Carsonella ruddii , [125] a 12.200.000 pares de bases (12,2 Mbp) en la bacteria que habita en el suelo Sorangium cellulosum . [126] Hay muchas excepciones a esto; por ejemplo, algunas especies de Streptomyces y Borrelia contienen un solo cromosoma lineal, [127] [128] mientras que algunas especies de Vibrio contienen más de un cromosoma. [129] Algunas bacterias contienen plásmidos , pequeñas moléculas extracromosómicas de ADN que pueden contener genes para varias funciones útiles como resistencia a antibióticos , capacidades metabólicas o varios factores de virulencia . [130]
Los genomas bacterianos suelen codificar entre unos cientos y unos miles de genes. Los genes de los genomas bacterianos suelen ser una única extensión continua de ADN. Aunque existen varios tipos diferentes de intrones en las bacterias, estos son mucho más raros que en los eucariotas. [131]
Las bacterias, como organismos asexuales, heredan una copia idéntica del genoma de sus progenitores y son clonales . Sin embargo, todas las bacterias pueden evolucionar por selección a partir de cambios en su material genético (ADN) causados por recombinación genética o mutaciones . Las mutaciones surgen de errores cometidos durante la replicación del ADN o de la exposición a mutágenos . Las tasas de mutación varían ampliamente entre diferentes especies de bacterias e incluso entre diferentes clones de una misma especie de bacteria. [132] Los cambios genéticos en los genomas bacterianos surgen de mutaciones aleatorias durante la replicación o de "mutaciones dirigidas por estrés", donde los genes involucrados en un proceso particular que limita el crecimiento tienen una mayor tasa de mutación. [133]
Algunas bacterias transfieren material genético entre células. Esto puede ocurrir de tres formas principales. Primero, las bacterias pueden absorber ADN exógeno de su entorno en un proceso llamado transformación . [134] Muchas bacterias pueden absorber naturalmente ADN del entorno, mientras que otras deben ser alteradas químicamente para inducirlas a absorber ADN. [135] El desarrollo de la competencia en la naturaleza generalmente se asocia con condiciones ambientales estresantes y parece ser una adaptación para facilitar la reparación del daño del ADN en las células receptoras. [136] En segundo lugar, los bacteriófagos pueden integrarse en el cromosoma bacteriano, introduciendo ADN extraño en un proceso conocido como transducción . Existen muchos tipos de bacteriófagos; algunos infectan y lisan a sus bacterias hospedadoras , mientras que otros se insertan en el cromosoma bacteriano. [ 137] Las bacterias resisten la infección por fagos a través de sistemas de modificación de restricción que degradan el ADN extraño [138] y un sistema que usa secuencias CRISPR para retener fragmentos de los genomas de fagos con los que las bacterias han estado en contacto en el pasado, lo que les permite bloquear la replicación del virus a través de una forma de interferencia de ARN . [139] [140] En tercer lugar, las bacterias pueden transferir material genético a través del contacto celular directo mediante conjugación . [141]
En circunstancias normales, la transducción, la conjugación y la transformación implican la transferencia de ADN entre bacterias individuales de la misma especie, pero ocasionalmente la transferencia puede ocurrir entre individuos de diferentes especies bacterianas, y esto puede tener consecuencias significativas, como la transferencia de resistencia a los antibióticos. [142] [143] En tales casos, la adquisición de genes de otras bacterias o del medio ambiente se denomina transferencia horizontal de genes y puede ser común en condiciones naturales. [144]
Comportamiento
Movimiento

Muchas bacterias son móviles (capaces de moverse por sí mismas) y lo hacen utilizando una variedad de mecanismos. Los más estudiados de estos son los flagelos , filamentos largos que son girados por un motor en la base para generar un movimiento similar al de una hélice. [145] El flagelo bacteriano está formado por unas 20 proteínas, y se requieren aproximadamente otras 30 proteínas para su regulación y ensamblaje. [145] El flagelo es una estructura giratoria impulsada por un motor reversible en la base que utiliza el gradiente electroquímico a través de la membrana para obtener energía. [146]

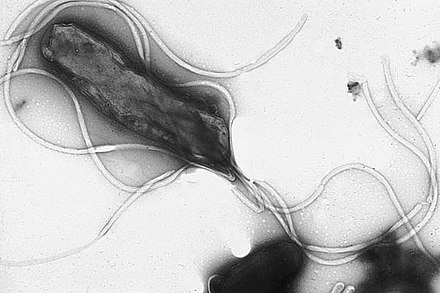
Las bacterias pueden usar los flagelos de diferentes maneras para generar diferentes tipos de movimiento. Muchas bacterias (como E. coli ) tienen dos modos distintos de movimiento: movimiento hacia adelante (nadar) y volteretas. Las volteretas les permiten reorientarse y hacen de su movimiento un paseo aleatorio tridimensional . [147] Las especies bacterianas difieren en el número y disposición de los flagelos en su superficie; algunas tienen un solo flagelo ( monótricas ), un flagelo en cada extremo ( anfítricas ), grupos de flagelos en los polos de la célula ( lofótricas ), mientras que otras tienen flagelos distribuidos por toda la superficie de la célula ( perítricas ). Los flagelos de un grupo de bacterias, las espiroquetas , se encuentran entre dos membranas en el espacio periplásmico. Tienen un cuerpo helicoidal distintivo que se retuerce mientras se mueve. [145]
Otros dos tipos de movimiento bacteriano se denominan motilidad de espasmo , que se basa en una estructura llamada pilus tipo IV [148] , y motilidad de deslizamiento , que utiliza otros mecanismos. En la motilidad de espasmo, el pilus en forma de bastón se extiende desde la célula, se une a un sustrato y luego se retrae, tirando de la célula hacia adelante. [149]
Las bacterias móviles son atraídas o repelidas por ciertos estímulos en comportamientos llamados taxs : estos incluyen quimiotaxis , fototaxis , taxis energéticos y magnetotaxis . [150] [151] [152] En un grupo peculiar, las mixobacterias, las bacterias individuales se mueven juntas para formar ondas de células que luego se diferencian para formar cuerpos fructíferos que contienen esporas. [47] Las mixobacterias se mueven solo cuando están en superficies sólidas, a diferencia de E. coli , que es móvil en medios líquidos o sólidos. [153]
Varias especies de Listeria y Shigella se desplazan dentro de las células huésped usurpando el citoesqueleto , que normalmente se utiliza para mover orgánulos dentro de la célula. Al promover la polimerización de actina en un polo de sus células, pueden formar una especie de cola que las empuja a través del citoplasma de la célula huésped. [154]
Comunicación
Algunas bacterias tienen sistemas químicos que generan luz. Esta bioluminiscencia se da a menudo en bacterias que viven en asociación con peces, y la luz probablemente sirve para atraer a los peces u otros animales grandes. [155]
Las bacterias a menudo funcionan como agregados multicelulares conocidos como biopelículas , intercambiando una variedad de señales moleculares para la comunicación intercelular y participando en un comportamiento multicelular coordinado. [156] [157]
Los beneficios comunitarios de la cooperación multicelular incluyen una división celular del trabajo , el acceso a recursos que no pueden ser utilizados eficazmente por células individuales, la defensa colectiva contra antagonistas y la optimización de la supervivencia de la población mediante la diferenciación en distintos tipos de células. [156] Por ejemplo, las bacterias en biopelículas pueden tener una resistencia más de quinientas veces mayor a los agentes antibacterianos que las bacterias "planctónicas" individuales de la misma especie. [157]
Un tipo de comunicación intercelular mediante una señal molecular se denomina detección de quórum , que sirve para determinar si la densidad de población local es suficiente para sustentar la inversión en procesos que solo tienen éxito si un gran número de organismos similares se comportan de manera similar, como excretar enzimas digestivas o emitir luz. [158] [159] La detección de quórum permite a las bacterias coordinar la expresión genética y producir, liberar y detectar autoinductores o feromonas que se acumulan con el crecimiento de la población celular. [160]
Clasificación e identificación


La clasificación busca describir la diversidad de especies bacterianas nombrando y agrupando organismos en función de similitudes. Las bacterias se pueden clasificar en función de la estructura celular, el metabolismo celular o las diferencias en los componentes celulares, como el ADN , los ácidos grasos , los pigmentos, los antígenos y las quinonas . [116] Si bien estos esquemas permitieron la identificación y clasificación de cepas bacterianas, no estaba claro si estas diferencias representaban variación entre especies distintas o entre cepas de la misma especie. Esta incertidumbre se debió a la falta de estructuras distintivas en la mayoría de las bacterias, así como a la transferencia lateral de genes entre especies no relacionadas. [162] Debido a la transferencia lateral de genes, algunas bacterias estrechamente relacionadas pueden tener morfologías y metabolismos muy diferentes. Para superar esta incertidumbre, la clasificación bacteriana moderna enfatiza la sistemática molecular , utilizando técnicas genéticas como la determinación de la relación citosina- guanina , la hibridación genoma-genoma, así como la secuenciación de genes que no han sufrido una transferencia lateral extensa de genes, como el gen ARNr . [163] La clasificación de las bacterias se determina mediante la publicación en el International Journal of Systematic Bacteriology, [164] y el Bergey's Manual of Systematic Bacteriology. [165] El Comité Internacional de Bacteriología Sistemática (ICSB) mantiene reglas internacionales para la denominación de bacterias y categorías taxonómicas y para la clasificación de las mismas en el Código Internacional de Nomenclatura de Bacterias . [166]
Históricamente, las bacterias se consideraban parte de Plantae , el reino vegetal, y se las llamaba "Schizomycetes" (hongos de fisión). [167] Por esta razón, las bacterias colectivas y otros microorganismos en un huésped a menudo se denominan "flora". [168] El término "bacteria" se aplicaba tradicionalmente a todos los procariotas unicelulares microscópicos. Sin embargo, la sistemática molecular mostró que la vida procariota constaba de dos dominios separados , originalmente llamados Eubacteria y Archaebacteria, pero ahora llamados Bacteria y Archaea que evolucionaron independientemente de un ancestro común antiguo. [4] Las arqueas y los eucariotas están más estrechamente relacionados entre sí que cualquiera de ellos con las bacterias. Estos dos dominios, junto con Eukarya, son la base del sistema de tres dominios , que actualmente es el sistema de clasificación más utilizado en microbiología. [169] Sin embargo, debido a la introducción relativamente reciente de la sistemática molecular y un rápido aumento en el número de secuencias de genoma disponibles, la clasificación bacteriana sigue siendo un campo cambiante y en expansión. [170] [171] Por ejemplo, Cavalier-Smith argumentó que las arqueas y los eucariotas evolucionaron a partir de bacterias grampositivas. [172]
La identificación de bacterias en el laboratorio es particularmente relevante en medicina , donde el tratamiento correcto está determinado por la especie bacteriana que causa una infección. En consecuencia, la necesidad de identificar patógenos humanos fue un impulso importante para el desarrollo de técnicas para identificar bacterias. [173]
La tinción de Gram , desarrollada en 1884 por Hans Christian Gram , caracteriza a las bacterias basándose en las características estructurales de sus paredes celulares. [174] [74] Las capas gruesas de peptidoglicano en la pared celular "Gram-positiva" se tiñen de púrpura, mientras que la pared celular delgada "Gram-negativa" aparece rosa. [174] Al combinar la morfología y la tinción de Gram, la mayoría de las bacterias pueden clasificarse como pertenecientes a uno de los cuatro grupos (cocos Gram-positivos, bacilos Gram-positivos, cocos Gram-negativos y bacilos Gram-negativos). Algunos organismos se identifican mejor mediante tinciones distintas a la tinción de Gram, en particular las micobacterias o Nocardia , que muestran resistencia a los ácidos en tinciones de Ziehl–Neelsen o similares. [175] Otros organismos pueden necesitar ser identificados por su crecimiento en medios especiales, o por otras técnicas, como la serología . [176]
Las técnicas de cultivo están diseñadas para promover el crecimiento e identificar bacterias particulares mientras se restringe el crecimiento de las otras bacterias en la muestra. [177] A menudo, estas técnicas están diseñadas para muestras específicas; por ejemplo, una muestra de esputo se tratará para identificar organismos que causan neumonía , mientras que las muestras de heces se cultivan en medios selectivos para identificar organismos que causan diarrea mientras se previene el crecimiento de bacterias no patógenas. Las muestras que normalmente son estériles, como sangre , orina o líquido cefalorraquídeo , se cultivan en condiciones diseñadas para cultivar todos los organismos posibles. [116] [178] Una vez que se ha aislado un organismo patógeno, se puede caracterizar aún más por su morfología, patrones de crecimiento (como crecimiento aeróbico o anaeróbico ), patrones de hemólisis y tinción. [179]
Al igual que con la clasificación bacteriana, la identificación de bacterias utiliza cada vez más métodos moleculares, [180] y espectroscopia de masas . [181] La mayoría de las bacterias no han sido caracterizadas y hay muchas especies que no se pueden cultivar en el laboratorio. [182] Los diagnósticos que utilizan herramientas basadas en ADN, como la reacción en cadena de la polimerasa , son cada vez más populares debido a su especificidad y velocidad, en comparación con los métodos basados en cultivos. [183] Estos métodos también permiten la detección e identificación de células " viables pero no cultivables " que son metabólicamente activas pero que no se dividen. [184] Sin embargo, incluso utilizando estos métodos mejorados, el número total de especies bacterianas no se conoce y ni siquiera se puede estimar con certeza. Siguiendo la clasificación actual, hay un poco menos de 9.300 especies conocidas de procariotas, que incluyen bacterias y arqueas; [185] pero los intentos de estimar el número real de diversidad bacteriana han variado de 10 7 a 10 9 especies totales, e incluso estas estimaciones diversas pueden estar erradas en muchos órdenes de magnitud. [186] [187]
Filos
Los siguientes filos han sido publicados válidamente según el Código Bacteriológico : [188]
- Acidobacterias
- Actinomicetos
- Aquificota
- Armatimonadota
- Atribacterota
- Bacillota
- Bacteroidota
- Balneario
- Bdellovibrionota
- Caldisericota
- Calditrichota
- Campylobacterota
- Clamidiota
- Clorobiota
- Cloroflexota
- Crisiogenota
- Coprotermobacterota
- Cianobacterias
- Deferribacterota
- Deinococota
- Dictoglomota
- Microbiota elusiva
- Fibrobacterias
- Fusobacterium
- Gemmatimonadota
- Ignavibacterium
- Lentisphaerota
- Micoplasma matota
- Mixococota
- Nitrospinota
- Nitrospirota
- Planctomicetos
- Pseudomonas aeruginosa
- Rodotermota
- Espiroquetas
- Sinergistota
- Termodesulfobacteriotas
- Termomicrobiota
- Termogota
- Verrucomicrobiota
Interacciones con otros organismos
A pesar de su aparente simplicidad, las bacterias pueden formar asociaciones complejas con otros organismos. Estas asociaciones simbióticas pueden dividirse en parasitismo , mutualismo y comensalismo . [190]
Comensales
La palabra " comensalismo " se deriva de la palabra "comensal", que significa "comer en la misma mesa" [191] y todas las plantas y animales están colonizados por bacterias comensales. En los seres humanos y otros animales, millones de ellas viven en la piel, las vías respiratorias, el intestino y otros orificios. [192] [193] Conocidas como "flora normal", [194] o "comensales", [195] estas bacterias normalmente no causan daño, pero ocasionalmente pueden invadir otros sitios del cuerpo y causar infecciones. Escherichia coli es un comensal en el intestino humano, pero puede causar infecciones del tracto urinario. [196] De manera similar, los estreptococos, que son parte de la flora normal de la boca humana, pueden causar enfermedades cardíacas . [197]
Depredadores
Algunas especies de bacterias matan y luego consumen otros microorganismos; estas especies se denominan bacterias depredadoras . [198] Entre ellas se incluyen organismos como Myxococcus xanthus , que forma enjambres de células que matan y digieren cualquier bacteria que encuentran. [199] Otros depredadores bacterianos se adhieren a sus presas para digerirlas y absorber nutrientes o invaden otra célula y se multiplican dentro del citosol. [200] Se cree que estas bacterias depredadoras evolucionaron a partir de saprófagos que consumían microorganismos muertos, a través de adaptaciones que les permitieron atrapar y matar a otros organismos. [201]
Mutualistas
Ciertas bacterias forman asociaciones espaciales estrechas que son esenciales para su supervivencia. Una de estas asociaciones mutualistas, llamada transferencia de hidrógeno entre especies, ocurre entre grupos de bacterias anaeróbicas que consumen ácidos orgánicos , como ácido butírico o ácido propiónico , y producen hidrógeno , y arqueas metanogénicas que consumen hidrógeno. [202] Las bacterias en esta asociación son incapaces de consumir los ácidos orgánicos ya que esta reacción produce hidrógeno que se acumula en su entorno. Solo la asociación íntima con las arqueas que consumen hidrógeno mantiene la concentración de hidrógeno lo suficientemente baja como para permitir que las bacterias crezcan. [203]
En el suelo, los microorganismos que residen en la rizosfera (una zona que incluye la superficie de la raíz y el suelo que se adhiere a la raíz después de una suave agitación) llevan a cabo la fijación de nitrógeno , convirtiendo el gas nitrógeno en compuestos nitrogenados. [204] Esto sirve para proporcionar una forma de nitrógeno fácilmente absorbible para muchas plantas, que no pueden fijar el nitrógeno por sí mismas. Muchas otras bacterias se encuentran como simbiontes en humanos y otros organismos. Por ejemplo, la presencia de más de 1.000 especies bacterianas en la flora intestinal humana normal de los intestinos puede contribuir a la inmunidad intestinal, sintetizar vitaminas , como el ácido fólico , la vitamina K y la biotina , convertir azúcares en ácido láctico (ver Lactobacillus ), así como fermentar carbohidratos complejos no digeribles . [205] [206] [207] La presencia de esta flora intestinal también inhibe el crecimiento de bacterias potencialmente patógenas (generalmente a través de la exclusión competitiva ) y estas bacterias beneficiosas se venden en consecuencia como suplementos dietéticos probióticos . [208]
Casi toda la vida animal depende de las bacterias para sobrevivir, ya que solo las bacterias y algunas arqueas poseen los genes y las enzimas necesarias para sintetizar vitamina B 12 , también conocida como cobalamina , y proporcionarla a través de la cadena alimentaria. La vitamina B 12 es una vitamina soluble en agua que participa en el metabolismo de cada célula del cuerpo humano. Es un cofactor en la síntesis de ADN y en el metabolismo de los ácidos grasos y aminoácidos . Es particularmente importante en el funcionamiento normal del sistema nervioso a través de su papel en la síntesis de mielina . [209]
Patógenos


El cuerpo está continuamente expuesto a muchas especies de bacterias, incluyendo comensales benéficos, que crecen en la piel y las mucosas , y saprofitos , que crecen principalmente en el suelo y en materia en descomposición . La sangre y los fluidos tisulares contienen nutrientes suficientes para sostener el crecimiento de muchas bacterias. El cuerpo tiene mecanismos de defensa que le permiten resistir la invasión microbiana de sus tejidos y le dan una inmunidad natural o resistencia innata contra muchos microorganismos . [210] A diferencia de algunos virus , las bacterias evolucionan relativamente lentamente por lo que muchas enfermedades bacterianas también ocurren en otros animales. [211]
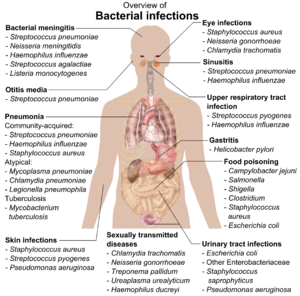
Si las bacterias forman una asociación parasitaria con otros organismos, se clasifican como patógenos. [212] Las bacterias patógenas son una de las principales causas de muerte y enfermedad humana y causan infecciones como el tétanos (causado por Clostridium tetani ), fiebre tifoidea , difteria , sífilis , cólera , enfermedades transmitidas por alimentos , lepra (causada por Mycobacterium leprae ) y tuberculosis (causada por Mycobacterium tuberculosis ). [213] Una causa patógena para una enfermedad médica conocida solo puede descubrirse muchos años después, como fue el caso de Helicobacter pylori y la enfermedad de úlcera péptica . [214] Las enfermedades bacterianas también son importantes en la agricultura , y las bacterias causan manchas en las hojas , fuego bacteriano y marchitez en las plantas, así como enfermedad de Johne , mastitis , salmonela y ántrax en animales de granja. [215]

Cada especie de patógeno tiene un espectro característico de interacciones con sus huéspedes humanos . Algunos organismos, como Staphylococcus o Streptococcus , pueden causar infecciones de la piel, neumonía , meningitis y sepsis , una respuesta inflamatoria sistémica que produce shock , vasodilatación masiva y muerte. [216] Sin embargo, estos organismos también son parte de la flora humana normal y generalmente existen en la piel o en la nariz sin causar ninguna enfermedad. Otros organismos invariablemente causan enfermedades en humanos, como Rickettsia , que son parásitos intracelulares obligados capaces de crecer y reproducirse solo dentro de las células de otros organismos. Una especie de Rickettsia causa tifus , mientras que otra causa fiebre maculosa de las Montañas Rocosas . Chlamydia , otro filo de parásitos intracelulares obligados, contiene especies que pueden causar neumonía o infección del tracto urinario y pueden estar involucradas en la enfermedad cardíaca coronaria . [217] Algunas especies, como Pseudomonas aeruginosa , Burkholderia cenocepacia y Mycobacterium avium , son patógenos oportunistas y causan enfermedades principalmente en personas inmunodeprimidas o con fibrosis quística . [218] [219] Algunas bacterias producen toxinas , que causan enfermedades. [220] Estas son endotoxinas , que provienen de células bacterianas rotas, y exotoxinas , que son producidas por bacterias y liberadas al medio ambiente. [221] La bacteria Clostridium botulinum , por ejemplo, produce una potente exotoxina que causa parálisis respiratoria, y Salmonellae produce una endotoxina que causa gastroenteritis. [221] Algunas exotoxinas se pueden convertir en toxoides , que se utilizan como vacunas para prevenir la enfermedad. [222]
Las infecciones bacterianas pueden tratarse con antibióticos , que se clasifican como bactericidas si matan las bacterias o bacteriostáticos si solo previenen el crecimiento bacteriano. Hay muchos tipos de antibióticos, y cada clase inhibe un proceso que es diferente en el patógeno del que se encuentra en el huésped. Un ejemplo de cómo los antibióticos producen toxicidad selectiva son el cloranfenicol y la puromicina , que inhiben el ribosoma bacteriano , pero no el ribosoma eucariota estructuralmente diferente. [223] Los antibióticos se utilizan tanto en el tratamiento de enfermedades humanas como en la agricultura intensiva para promover el crecimiento animal, donde pueden estar contribuyendo al rápido desarrollo de resistencia a los antibióticos en las poblaciones bacterianas. [224] Las infecciones se pueden prevenir con medidas antisépticas , como esterilizar la piel antes de perforarla con la aguja de una jeringa, y con el cuidado adecuado de los catéteres permanentes. Los instrumentos quirúrgicos y dentales también se esterilizan para prevenir la contaminación por bacterias. Los desinfectantes como la lejía se utilizan para matar bacterias u otros patógenos en las superficies para prevenir la contaminación y reducir aún más el riesgo de infección. [225]
Importancia en la tecnología y la industria
Las bacterias, a menudo bacterias de ácido láctico , como las especies de Lactobacillus y Lactococcus , en combinación con levaduras y mohos , se han utilizado durante miles de años en la preparación de alimentos fermentados , como queso , encurtidos , salsa de soja , chucrut , vinagre , vino y yogur . [226] [227]
La capacidad de las bacterias para degradar una variedad de compuestos orgánicos es notable y se ha utilizado en el procesamiento de desechos y la biorremediación . Las bacterias capaces de digerir los hidrocarburos del petróleo se utilizan a menudo para limpiar derrames de petróleo . [228] Se agregó fertilizante a algunas de las playas de Prince William Sound en un intento de promover el crecimiento de estas bacterias naturales después del derrame de petróleo del Exxon Valdez en 1989. Estos esfuerzos fueron efectivos en playas que no estaban demasiado cubiertas de petróleo. Las bacterias también se utilizan para la biorremediación de desechos tóxicos industriales . [229] En la industria química , las bacterias son más importantes en la producción de productos químicos enantioméricamente puros para su uso como productos farmacéuticos o agroquímicos . [230]
Las bacterias también se pueden utilizar en lugar de pesticidas en el control biológico de plagas . Esto implica comúnmente Bacillus thuringiensis (también llamado BT), una bacteria Gram-positiva que habita en el suelo. Las subespecies de esta bacteria se utilizan como insecticidas específicos para lepidópteros bajo nombres comerciales como Dipel y Thuricide. [231] Debido a su especificidad, estos pesticidas se consideran respetuosos con el medio ambiente , con poco o ningún efecto sobre los humanos, la vida silvestre , los polinizadores y la mayoría de los demás insectos beneficiosos . [232] [233]
Debido a su capacidad de crecer rápidamente y la relativa facilidad con la que pueden ser manipuladas, las bacterias son los caballos de batalla para los campos de la biología molecular , la genética y la bioquímica . Al realizar mutaciones en el ADN bacteriano y examinar los fenotipos resultantes, los científicos pueden determinar la función de los genes, las enzimas y las vías metabólicas en las bacterias, y luego aplicar este conocimiento a organismos más complejos. [234] Este objetivo de comprender la bioquímica de una célula alcanza su expresión más compleja en la síntesis de enormes cantidades de datos cinéticos de enzimas y de expresión genética en modelos matemáticos de organismos completos. Esto se puede lograr en algunas bacterias bien estudiadas, y ahora se están produciendo y probando modelos del metabolismo de Escherichia coli . [235] [236] Esta comprensión del metabolismo y la genética bacteriana permite el uso de la biotecnología para bioingeniería de bacterias para la producción de proteínas terapéuticas, como insulina , factores de crecimiento o anticuerpos . [237] [238]
Debido a su importancia para la investigación en general, las muestras de cepas bacterianas se aíslan y se conservan en Centros de Recursos Biológicos . Esto garantiza la disponibilidad de la cepa para los científicos de todo el mundo. [239]
Historia de la bacteriología

Las bacterias fueron observadas por primera vez por el microscopista holandés Antonie van Leeuwenhoek en 1676, utilizando un microscopio de lente única de su propio diseño. Luego publicó sus observaciones en una serie de cartas a la Royal Society de Londres . [240] Las bacterias fueron el descubrimiento microscópico más notable de Leeuwenhoek. Su tamaño estaba justo en el límite de lo que sus lentes simples podían resolver y, en uno de los paréntesis más sorprendentes en la historia de la ciencia, nadie más las volvería a ver durante más de un siglo. [241] Sus observaciones también incluyeron protozoos a los que llamó animálculos , y sus hallazgos fueron analizados nuevamente a la luz de los hallazgos más recientes de la teoría celular . [242]
Christian Gottfried Ehrenberg introdujo la palabra "bacteria" en 1828. [243] De hecho, su Bacterium era un género que contenía bacterias con forma de bastón que no formaban esporas, [244] a diferencia de Bacillus , un género de bacterias con forma de bastón que formaban esporas definido por Ehrenberg en 1835. [245]
En 1859, Louis Pasteur demostró que el crecimiento de microorganismos causa el proceso de fermentación y que este crecimiento no se debe a la generación espontánea ( las levaduras y los mohos , comúnmente asociados con la fermentación, no son bacterias, sino hongos ). Junto con su contemporáneo Robert Koch , Pasteur fue uno de los primeros defensores de la teoría de los gérmenes de la enfermedad . [246] Antes de ellos, Ignaz Semmelweis y Joseph Lister se habían dado cuenta de la importancia de las manos desinfectadas en el trabajo médico. Semmelweis, quien en la década de 1840 formuló sus reglas para el lavado de manos en el hospital, antes del advenimiento de la teoría de los gérmenes, atribuyó la enfermedad a la "materia orgánica animal en descomposición". Sus ideas fueron rechazadas y su libro sobre el tema condenado por la comunidad médica. Sin embargo, después de Lister, los médicos comenzaron a desinfectarse las manos en la década de 1870. [247]
Robert Koch, pionero de la microbiología médica, trabajó en el cólera , el ántrax y la tuberculosis . En su investigación sobre la tuberculosis, Koch finalmente demostró la teoría de los gérmenes, por la que recibió un Premio Nobel en 1905. [248] En los postulados de Koch , estableció criterios para comprobar si un organismo es la causa de una enfermedad , y estos postulados todavía se utilizan en la actualidad. [249]
Se dice que Ferdinand Cohn es el fundador de la bacteriología y estudió las bacterias desde 1870. Cohn fue el primero en clasificar las bacterias según su morfología. [250] [251]
Aunque en el siglo XIX se sabía que las bacterias son la causa de muchas enfermedades, no había tratamientos antibacterianos eficaces disponibles. [252] En 1910, Paul Ehrlich desarrolló el primer antibiótico, al cambiar los colorantes que teñían selectivamente a Treponema pallidum (la espiroqueta que causa la sífilis ) por compuestos que mataban selectivamente al patógeno. [253] Ehrlich, que había recibido el Premio Nobel en 1908 por su trabajo en inmunología , fue pionero en el uso de colorantes para detectar e identificar bacterias, y su trabajo fue la base de la tinción de Gram y la tinción de Ziehl-Neelsen . [254]
Un gran paso adelante en el estudio de las bacterias se produjo en 1977 cuando Carl Woese reconoció que las arqueas tienen una línea de descendencia evolutiva separada de las bacterias. [255] Esta nueva taxonomía filogenética dependía de la secuenciación del ARN ribosómico 16S y dividía a los procariotas en dos dominios evolutivos, como parte del sistema de tres dominios . [4]
Véase también
Referencias
- ^ "31. Vida antigua: microfósiles de sílex de ápice". www.lpi.usra.edu . Consultado el 12 de marzo de 2022 .
- ^ Sender R, Fuchs S, Milo R (19 de agosto de 2016). "Estimaciones revisadas del número de células humanas y bacterianas en el cuerpo". PLOS Biology . 14 (8): e1002533. doi : 10.1371/journal.pbio.1002533 . ISSN 1545-7885. PMC 4991899 . PMID 27541692.
- ^ ab McCutcheon JP (octubre de 2021). "La genómica y la biología celular de las infecciones intracelulares beneficiosas para el huésped". Revisión anual de biología celular y del desarrollo . 37 (1): 115–142. doi : 10.1146/annurev-cellbio-120219-024122 . PMID: 34242059. S2CID : 235786110.
- ^ abc Hall 2008, pág. 145.
- ^ βακτήριον. Liddell, Henry George ; Scott, Robert ; Un léxico griego-inglés en el Proyecto Perseo .
- ^ βακτηρία en Liddell y Scott .
- ^ Harper D. "bacteria". Diccionario Etimológico en Línea .
- ^ desde Krasner 2014, pág. 74.
- ^ Woese CR, Kandler O, Wheelis ML (junio de 1990). "Hacia un sistema natural de organismos: propuesta para los dominios Archaea, Bacteria y Eucarya". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 87 (12): 4576–79. Bibcode :1990PNAS...87.4576W. doi : 10.1073/pnas.87.12.4576 . PMC 54159 . PMID 2112744.
- ^ Hall 2008, pág. 84.
- ^ Godoy-Vitorino F (julio de 2019). "Ecología microbiana humana y la nueva medicina en ascenso". Annals of Translational Medicine . 7 (14): 342. doi : 10.21037/atm.2019.06.56 . PMC 6694241 . PMID 31475212.
- ^ Schopf JW (julio de 1994). «Tasas dispares, destinos diferentes: el ritmo y el modo de evolución cambiaron desde el Precámbrico hasta el Fanerozoico». Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (15): 6735–42. Bibcode :1994PNAS...91.6735S. doi : 10.1073/pnas.91.15.6735 . PMC 44277 . PMID 8041691.
- ^ DeLong EF, Pace NR (agosto de 2001). "Diversidad ambiental de bacterias y arqueas". Biología sistemática . 50 (4): 470–78. CiteSeerX 10.1.1.321.8828 . doi :10.1080/106351501750435040. PMID 12116647.
- ^ Brown JR, Doolittle WF (diciembre de 1997). "Archaea y la transición de procariota a eucariota". Microbiology and Molecular Biology Reviews . 61 (4): 456–502. doi :10.1128/mmbr.61.4.456-502.1997. PMC 232621 . PMID 9409149.
- ^ Daum B, Gold V (junio de 2018). "Twitch or swim: toward the understanding of procariota motion based on the type IV pilus blueprint" (Contracción o natación: hacia la comprensión del movimiento procariota basado en el modelo del pilus tipo IV). Química biológica . 399 (7): 799–808. doi :10.1515/hsz-2018-0157. hdl : 10871/33366 . PMID: 29894297. S2CID : 48352675.
- ^ Di Giulio M (diciembre de 2003). "El ancestro universal y el ancestro de las bacterias eran hipertermófilos". Journal of Molecular Evolution . 57 (6): 721–30. Bibcode :2003JMolE..57..721D. doi :10.1007/s00239-003-2522-6. PMID 14745541. S2CID 7041325.
- ^ Battistuzzi FU, Feijao A, Hedges SB (noviembre de 2004). "Una escala de tiempo genómica de la evolución procariota: perspectivas sobre el origen de la metanogénesis, la fototrofia y la colonización de la tierra". BMC Evolutionary Biology . 4 : 44. doi : 10.1186/1471-2148-4-44 . PMC 533871 . PMID 15535883.
- ^ Homann M, Sansjofre P, Van Zuilen M, Heubeck C, Gong J, Killingsworth B, et al. (23 de julio de 2018). "Vida microbiana y ciclo biogeoquímico en la tierra hace 3220 millones de años" (PDF) . Nature Geoscience . 11 (9): 665–671. Bibcode :2018NatGe..11..665H. doi :10.1038/s41561-018-0190-9. S2CID 134935568.
- ^ Gabaldón T (octubre de 2021). «Origen y evolución temprana de la célula eucariota». Revista Anual de Microbiología . 75 (1): 631–647. doi :10.1146/annurev-micro-090817-062213. PMID 34343017. S2CID 236916203. Archivado desde el original el 19 de agosto de 2022 . Consultado el 19 de agosto de 2022 .
- ^ Callier V (8 de junio de 2022). «Mitocondrias y el origen de los eucariotas». Revista Knowable . doi : 10.1146/knowable-060822-2 . Consultado el 19 de agosto de 2022 .
- ^ Poole AM, Penny D (enero de 2007). "Evaluación de hipótesis sobre el origen de los eucariotas". BioEssays . 29 (1): 74–84. doi :10.1002/bies.20516. PMID 17187354.
- ^ Dyall SD, Brown MT, Johnson PJ (abril de 2004). "Invasiones antiguas: de endosimbiontes a organelos". Science . 304 (5668): 253–257. Bibcode :2004Sci...304..253D. doi :10.1126/science.1094884. PMID 15073369. S2CID 19424594.
- ^ Stephens TG, Gabr A, Calatrava V, Grossman AR, Bhattacharya D (septiembre de 2021). "¿Por qué la endosimbiosis primaria es tan rara?". The New Phytologist . 231 (5): 1693–1699. doi : 10.1111/nph.17478 . PMC 8711089 . PMID 34018613.
- ^ ab Baker-Austin C, Dopson M (abril de 2007). "Vida en ácido: homeostasis del pH en acidófilos". Tendencias en microbiología . 15 (4): 165–171. doi :10.1016/j.tim.2007.02.005. PMID 17331729.
- ^ Jeong SW, Choi YJ (octubre de 2020). "Microorganismos extremófilos para el tratamiento de contaminantes tóxicos en el medio ambiente". Moléculas . 25 (21): 4916. doi : 10.3390/molecules25214916 . PMC 7660605 . PMID 33114255.
- ^ Flemming HC, Wuertz S (abril de 2019). "Bacterias y arqueas en la Tierra y su abundancia en biopelículas". Nature Reviews. Microbiología . 17 (4): 247–260. doi :10.1038/s41579-019-0158-9. PMID 30760902. S2CID 61155774.
- ^ Bar-On YM, Phillips R, Milo R (junio de 2018). "La distribución de la biomasa en la Tierra". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 115 (25): 6506–6511. Bibcode :2018PNAS..115.6506B. doi : 10.1073/pnas.1711842115 . PMC 6016768 . PMID 29784790.
- ^ Wheelis 2008, pág. 362.
- ^ Kushkevych I, Procházka J, Gajdács M, Rittmann SK, Vítězová M (junio de 2021). "Fisiología molecular de bacterias anaeróbicas fototróficas de azufre púrpura y verde". Revista Internacional de Ciencias Moleculares . 22 (12): 6398. doi : 10.3390/ijms22126398 . PMC 8232776 . PMID 34203823.
- ^ Wheelis 2008, pág. 6.
- ^ Pommerville 2014, págs. 3–6.
- ^ abc Krasner 2014, pág. 38.
- ^ abcdefg Pommerville 2014, pág. 134.
- ^ Schulz HN, Jorgensen BB (2001). "Big bacteria". Revisión anual de microbiología . 55 : 105–137. doi :10.1146/annurev.micro.55.1.105. PMID 11544351. S2CID 18168018.
- ^ Williams C (2011). "¿A quién llamas simple?". New Scientist . 211 (2821): 38–41. doi :10.1016/S0262-4079(11)61709-0.
- ^ Volland JM, Gonzalez-Rizzo S, Gros O, Tyml T, Ivanova N, Schulz F, Goudeau D, Elisabeth NH, Nath N, Udwary D, Malmstrom RR (18 de febrero de 2022). "Una bacteria de un centímetro de largo con ADN compartimentado en orgánulos unidos a la membrana". bioRxiv (preimpresión). doi :10.1101/2022.02.16.480423. S2CID 246975579.
- ^ Sanderson K (junio de 2022). "La bacteria más grande jamás encontrada es sorprendentemente compleja". Nature . doi :10.1038/d41586-022-01757-1. PMID 35750919. S2CID 250022076.
- ^ Robertson J, Gomersall M, Gill P (noviembre de 1975). "Mycoplasma hominis: crecimiento, reproducción y aislamiento de células pequeñas viables". Journal of Bacteriology . 124 (2): 1007–1018. doi :10.1128/JB.124.2.1007-1018.1975. PMC 235991 . PMID 1102522.
- ^ Velimirov B (2001). "Nanobacterias, ultramicrobacterias y formas de inanición: una búsqueda de la bacteria metabolizadora más pequeña". Microbios y entornos . 16 (2): 67–77. doi : 10.1264/jsme2.2001.67 .
- ^ Dusenbery DB (2009). Vivir a escala micro . Cambridge, Massachusetts: Harvard University Press . Págs. 20-25. ISBN. 978-0-674-03116-6.
- ^ Yang DC, Blair KM, Salama NR (marzo de 2016). "Mantenerse en forma: el impacto de la forma celular en la supervivencia bacteriana en diversos entornos". Microbiology and Molecular Biology Reviews . 80 (1): 187–203. doi :10.1128/MMBR.00031-15. PMC 4771367 . PMID 26864431.
- ^ Cabeen MT, Jacobs-Wagner C (agosto de 2005). "Forma de la célula bacteriana". Nature Reviews. Microbiology . 3 (8): 601–10. doi :10.1038/nrmicro1205. PMID 16012516. S2CID 23938989.
- ^ Young KD (septiembre de 2006). "El valor selectivo de la forma bacteriana". Microbiology and Molecular Biology Reviews . 70 (3): 660–703. doi :10.1128/MMBR.00001-06. PMC 1594593 . PMID 16959965.
- ^ Crawford 2007, pág. xi.
- ^ Claessen D, Rozen DE, Kuipers OP, Søgaard-Andersen L, van Wezel GP (febrero de 2014). "Soluciones bacterianas para la multicelularidad: una historia de biopelículas, filamentos y cuerpos fructíferos". Reseñas de la naturaleza. Microbiología . 12 (2): 115–24. doi :10.1038/nrmicro3178. hdl : 11370/0db66a9c-72ef-4e11-a75d-9d1e5827573d . PMID 24384602. S2CID 20154495.
- ^ Shimkets LJ (1999). "Señalización intercelular durante el desarrollo del cuerpo fructífero de Myxococcus xanthus". Revisión anual de microbiología . 53 : 525–49. doi :10.1146/annurev.micro.53.1.525. PMID 10547700.
- ^ ab Kaiser D (2004). "Señalización en mixobacterias". Revisión anual de microbiología . 58 : 75–98. doi :10.1146/annurev.micro.58.030603.123620. PMID 15487930.
- ^ Wheelis 2008, pág. 75.
- ^ Mandal A, Dutta A, Das R, Mukherjee J (junio de 2021). "El papel de las comunidades microbianas intermareales en el secuestro de dióxido de carbono y la eliminación de contaminantes: una revisión". Boletín de contaminación marina . 170 : 112626. Bibcode :2021MarPB.17012626M. doi :10.1016/j.marpolbul.2021.112626. PMID 34153859.
- ^ Donlan RM (septiembre de 2002). "Biofilms: vida microbiana en superficies". Enfermedades infecciosas emergentes . 8 (9): 881–90. doi :10.3201/eid0809.020063. PMC 2732559 . PMID 12194761.
- ^ Branda SS, Vik S, Friedman L, Kolter R (enero de 2005). "Biofilms: the matrix revisited". Tendencias en microbiología . 13 (1): 20–26. doi :10.1016/j.tim.2004.11.006. PMID 15639628.
- ^ ab Davey ME, O'toole GA (diciembre de 2000). "Biopelículas microbianas: de la ecología a la genética molecular". Microbiology and Molecular Biology Reviews . 64 (4): 847–67. doi :10.1128/MMBR.64.4.847-867.2000. PMC 99016 . PMID 11104821.
- ^ Donlan RM, Costerton JW (abril de 2002). "Biofilms: mecanismos de supervivencia de microorganismos clínicamente relevantes". Clinical Microbiology Reviews . 15 (2): 167–93. doi :10.1128/CMR.15.2.167-193.2002. PMC 118068 . PMID 11932229.
- ^ Slonczewski JL, Foster JW (2013). Microbiología: una ciencia en evolución (tercera edición). Nueva York: WW Norton. pág. 82. ISBN 978-0-393-12367-8.
- ^ Feijoo-Siota L, Rama JL, Sánchez-Pérez A, Villa TG (julio de 2017). "Consideraciones sobre los nucleoides bacterianos". Microbiología y Biotecnología Aplicadas . 101 (14): 5591–602. doi :10.1007/s00253-017-8381-7. PMID 28664324. S2CID 10173266.
- ^ Bobik TA (mayo de 2006). "Organelos poliédricos que compartimentan los procesos metabólicos bacterianos". Applied Microbiology and Biotechnology . 70 (5): 517–25. doi :10.1007/s00253-005-0295-0. PMID 16525780. S2CID 8202321.
- ^ Yeates TO, Kerfeld CA, Heinhorst S, Cannon GC, Shively JM (septiembre de 2008). "Organelos basados en proteínas en bacterias: carboxisomas y microcompartimentos relacionados". Nature Reviews. Microbiology . 6 (9): 681–91. doi :10.1038/nrmicro1913. PMID 18679172. S2CID 22666203.
- ^ Kerfeld CA, Sawaya MR, Tanaka S, Nguyen CV, Phillips M, Beeby M, Yeates TO (agosto de 2005). "Estructuras proteicas que forman la cubierta de los orgánulos bacterianos primitivos". Science . 309 (5736): 936–38. Bibcode :2005Sci...309..936K. CiteSeerX 10.1.1.1026.896 . doi :10.1126/science.1113397. PMID 16081736. S2CID 24561197.
- ^ Gitai Z (marzo de 2005). "La nueva biología celular bacteriana: partes móviles y arquitectura subcelular". Cell . 120 (5): 577–86. doi : 10.1016/j.cell.2005.02.026 . PMID 15766522. S2CID 8894304.
- ^ Shih YL, Rothfield L (septiembre de 2006). "El citoesqueleto bacteriano". Microbiology and Molecular Biology Reviews . 70 (3): 729–54. doi :10.1128/MMBR.00017-06. PMC 1594594 . PMID 16959967.
- ^ Norris V, den Blaauwen T, Cabin-Flaman A, Doi RH, Harshey R, Janniere L, Jimenez-Sanchez A, Jin DJ, Levin PA, Mileykovskaya E, Minsky A, Saier M, Skarstad K (marzo de 2007). "Taxonomía funcional de hiperestructuras bacterianas". Microbiology and Molecular Biology Reviews . 71 (1): 230–53. doi :10.1128/MMBR.00035-06. PMC 1847379 . PMID 17347523.
- ^ Pommerville 2014, págs. 120-121.
- ^ Bryant DA, Frigaard NU (noviembre de 2006). "Fotosíntesis y fototrofia procariotas iluminadas". Tendencias en microbiología . 14 (11): 488–96. doi :10.1016/j.tim.2006.09.001. PMID 16997562.
- ^ Psencík J, Ikonen TP, Laurinmäki P, Merckel MC, Butcher SJ, Serimaa RE, Tuma R (agosto de 2004). "Organización lamelar de pigmentos en clorosomas, los complejos de recolección de luz de las bacterias fotosintéticas verdes". Revista biofísica . 87 (2): 1165–72. Código Bibliográfico :2004BpJ....87.1165P. doi :10.1529/biophysj.104.040956. PMC 1304455 . PMID 15298919.
- ^ Thanbichler M, Wang SC, Shapiro L (octubre de 2005). "El nucleoide bacteriano: una estructura altamente organizada y dinámica". Journal of Cellular Biochemistry . 96 (3): 506–21. doi : 10.1002/jcb.20519 . PMID 15988757. S2CID 25355087.
- ^ Poehlsgaard J, Douthwaite S (noviembre de 2005). "El ribosoma bacteriano como objetivo de los antibióticos". Nature Reviews. Microbiology . 3 (11): 870–81. doi :10.1038/nrmicro1265. PMID 16261170. S2CID 7521924.
- ^ Yeo M, Chater K (marzo de 2005). "La interacción del metabolismo del glucógeno y la diferenciación proporciona una perspectiva sobre la biología del desarrollo de Streptomyces coelicolor". Microbiología . 151 (Pt 3): 855–61. doi : 10.1099/mic.0.27428-0 . PMID 15758231. Archivado desde el original el 29 de septiembre de 2007.
- ^ Shiba T, Tsutsumi K, Ishige K, Noguchi T (marzo de 2000). "Polifosfato inorgánico y polifosfato quinasa: sus nuevas funciones biológicas y aplicaciones". Bioquímica. Biokhimiia . 65 (3): 315–23. PMID 10739474. Archivado desde el original el 25 de septiembre de 2006.
- ^ Brune DC (junio de 1995). "Aislamiento y caracterización de proteínas de glóbulos de azufre de Chromatium vinosum y Thiocapsa roseopersicina". Archivos de Microbiología . 163 (6): 391–99. Bibcode :1995ArMic.163..391B. doi :10.1007/BF00272127. PMID 7575095. S2CID 22279133.
- ^ Kadouri D, Jurkevitch E, Okon Y, Castro-Sowinski S (2005). "Importancia ecológica y agrícola de los polihidroxialcanoatos bacterianos". Critical Reviews in Microbiology . 31 (2): 55–67. doi :10.1080/10408410590899228. PMID 15986831. S2CID 4098268.
- ^ Walsby AE (marzo de 1994). "Vesículas de gas". Microbiological Reviews . 58 (1): 94–144. doi :10.1128/MMBR.58.1.94-144.1994. PMC 372955 . PMID 8177173.
- ^ van Heijenoort J (marzo de 2001). "Formación de las cadenas de glicano en la síntesis de peptidoglicano bacteriano". Glycobiology . 11 (3): 25R–36R. doi : 10.1093/glycob/11.3.25R . PMID 11320055. S2CID 46066256.
- ^ ab Koch AL (octubre de 2003). "La pared bacteriana como objetivo de ataque: investigación pasada, presente y futura". Clinical Microbiology Reviews . 16 (4): 673–87. doi :10.1128/CMR.16.4.673-687.2003. PMC 207114 . PMID 14557293.
- ^ ab Gram HC (1884). "Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten". Fortschr. Med . 2 : 185–89.
- ^ Hugenholtz P (2002). "Explorando la diversidad procariota en la era genómica". Genome Biology . 3 (2): REVIEWS0003. doi : 10.1186/gb-2002-3-2-reviews0003 . PMC 139013 . PMID 11864374.
- ^ Walsh FM, Amyes SG (octubre de 2004). "Microbiología y mecanismos de resistencia a fármacos de patógenos totalmente resistentes" (PDF) . Current Opinion in Microbiology . 7 (5): 439–44. doi :10.1016/j.mib.2004.08.007. PMID 15451497.
- ^ Alderwick LJ, Harrison J, Lloyd GS, Birch HL (marzo de 2015). "La pared celular micobacteriana: peptidoglicano y arabinogalactano". Cold Spring Harbor Perspectives in Medicine . 5 (8): a021113. doi :10.1101/cshperspect.a021113. PMC 4526729 . PMID 25818664.
- ^ Fagan RP, Fairweather NF (marzo de 2014). "Biogénesis y funciones de las capas S bacterianas" (PDF) . Nature Reviews. Microbiology . 12 (3): 211–22. doi :10.1038/nrmicro3213. PMID 24509785. S2CID 24112697.
- ^ Thompson SA (diciembre de 2002). "Capas superficiales de Campylobacter (capas S) y evasión inmunitaria". Anales de Periodontología . 7 (1): 43–53. doi :10.1902/annals.2002.7.1.43. PMC 2763180 . PMID 16013216.
- ^ Beveridge TJ, Pouwels PH, Sára M, Kotiranta A, Lounatmaa K, Kari K, Kerosuo E, Haapasalo M, Egelseer EM, Schocher I, Sleytr UB, Morelli L, Callegari ML, Nomellini JF, Bingle WH, Smit J, Leibovitz E, Lemaire M, Miras I, Salamitou S, Béguin P, Ohayon H, Gounon P, Matuschek M, Koval SF (junio de 1997). "Funciones de las capas S". Reseñas de microbiología FEMS . 20 (1–2): 99–149. doi :10.1111/j.1574-6976.1997.tb00305.x. PMID 9276929.
- ^ Kojima S, Blair DF (2004). El motor flagelar bacteriano: estructura y función de una máquina molecular compleja . Revista internacional de citología. Vol. 233. págs. 93-134. doi :10.1016/S0074-7696(04)33003-2. ISBN . 978-0-12-364637-8. Número de identificación personal 15037363.
- ^ Wheelis 2008, pág. 76.
- ^ Cheng RA, Wiedmann M (2020). "Avances recientes en nuestra comprensión de la diversidad y los roles de las fimbrias chaperona-usher en la facilitación del tropismo tisular y del hospedador de Salmonella". Frontiers in Cellular and Infection Microbiology . 10 : 628043. doi : 10.3389/fcimb.2020.628043 . PMC 7886704 . PMID 33614531.
- ^ Silverman PM (febrero de 1997). "Hacia una biología estructural de la conjugación bacteriana". Microbiología molecular . 23 (3): 423–29. doi : 10.1046/j.1365-2958.1997.2411604.x . PMID 9044277. S2CID 24126399.
- ^ Costa TR, Felisberto-Rodrigues C, Meir A, Prevost MS, Redzej A, Trokter M, Waksman G (junio de 2015). "Sistemas de secreción en bacterias gramnegativas: perspectivas estructurales y mecanicistas". Nature Reviews. Microbiología . 13 (6): 343–59. doi :10.1038/nrmicro3456. PMID 25978706. S2CID 8664247.
- ^ Luong P, Dube DH (julio de 2021). "Desmantelamiento del glicocáliz bacteriano: herramientas químicas para sondear, perturbar y obtener imágenes de los glicanos bacterianos". Química bioorgánica y medicinal . 42 : 116268. doi :10.1016/j.bmc.2021.116268. ISSN 0968-0896. PMC 8276522. PMID 34130219 .
- ^ Stokes RW, Norris-Jones R, Brooks DE, Beveridge TJ, Doxsee D, Thorson LM (octubre de 2004). "La capa externa rica en glicanos de la pared celular de Mycobacterium tuberculosis actúa como una cápsula antifagocítica que limita la asociación de la bacteria con los macrófagos". Infección e inmunidad . 72 (10): 5676–86. doi :10.1128/IAI.72.10.5676-5686.2004. PMC 517526 . PMID 15385466.
- ^ ab Kalscheuer R, Palacios A, Anso I, Cifuente J, Anguita J, Jacobs WR, Guerin ME, Prados-Rosales R (julio de 2019). "La cápsula de Mycobacterium tuberculosis: una estructura celular con implicaciones clave en la patogénesis". The Biochemical Journal . 476 (14): 1995–2016. doi :10.1042/BCJ20190324. PMC 6698057 . PMID 31320388.
- ^ Jernigan JA, Stephens DS, Ashford DA, Omenaca C, Topiel MS, Galbraith M, Tapper M, Fisk TL, Zaki S, Popovic T, Meyer RF, Quinn CP, Harper SA, Fridkin SK, Sejvar JJ, Shepard CW, McConnell M, Guarner J, Shieh WJ, Malecki JM, Gerberding JL, Hughes JM, Perkins BA (2001). "Ántrax por inhalación relacionado con el bioterrorismo: los primeros 10 casos notificados en los Estados Unidos". Enfermedades infecciosas emergentes . 7 (6): 933–44. doi :10.3201/eid0706.010604. PMC 2631903 . PMID 11747719.
- ^ Nicholson WL, Munakata N, Horneck G, Melosh HJ, Setlow P (septiembre de 2000). "Resistencia de las endosporas de Bacillus a ambientes terrestres y extraterrestres extremos". Microbiology and Molecular Biology Reviews . 64 (3): 548–72. doi :10.1128/MMBR.64.3.548-572.2000. PMC 99004 . PMID 10974126.
- ^ ab McKenney PT, Driks A, Eichenberger P (enero de 2013). "La endospora de Bacillus subtilis: ensamblaje y funciones de la capa multicapa". Nature Reviews. Microbiology . 11 (1): 33–44. doi : 10.1038/nrmicro2921 . PMC 9910062 . PMID 23202530. S2CID 205498395.
- ^ Nicholson WL, Fajardo-Cavazos P, Rebeil R, Slieman TA, Riesenman PJ, Law JF, Xue Y (agosto de 2002). "Endosporas bacterianas y su importancia en la resistencia al estrés". Antonie van Leeuwenhoek . 81 (1–4): 27–32. doi :10.1023/A:1020561122764. PMID 12448702. S2CID 30639022.
- ^ Vreeland RH, Rosenzweig WD, Powers DW (octubre de 2000). "Aislamiento de una bacteria halotolerante de 250 millones de años de antigüedad a partir de un cristal de sal primario". Nature . 407 (6806): 897–900. Bibcode :2000Natur.407..897V. doi :10.1038/35038060. PMID 11057666. S2CID 9879073.
- ^ Cano RJ, Borucki MK (mayo de 1995). "Reactivación e identificación de esporas bacterianas en ámbar dominicano de entre 25 y 40 millones de años". Science . 268 (5213): 1060–64. Bibcode :1995Sci...268.1060C. doi :10.1126/science.7538699. PMID 7538699.
- ^ "Disputa por bacterias antiguas". BBC News . 7 de junio de 2001 . Consultado el 26 de abril de 2020 .
- ^ Nicholson WL, Schuerger AC, Setlow P (abril de 2005). "El entorno solar UV y la resistencia a la radiación UV de las esporas bacterianas: consideraciones para el transporte de la Tierra a Marte mediante procesos naturales y vuelos espaciales tripulados". Mutation Research . 571 (1–2): 249–64. Bibcode :2005MRFMM.571..249N. doi :10.1016/j.mrfmmm.2004.10.012. PMID 15748651.
- ^ "Colonizar la galaxia es difícil. ¿Por qué no enviar bacterias en su lugar?". The Economist . 12 de abril de 2018. ISSN 0013-0613 . Consultado el 26 de abril de 2020 .
- ^ Revitt-Mills SA, Vidor CJ, Watts TD, Lyras D, Rood JI, Adams V (mayo de 2019). "Plásmidos de virulencia de los clostridios patógenos". Microbiology Spectrum . 7 (3). doi :10.1128/microbiolspec.GPP3-0034-2018. PMID 31111816. S2CID 160013108.
- ^ Reigadas E, van Prehn J, Falcone M, Fitzpatrick F, Vehreschild MJ, Kuijper EJ, Bouza E (julio de 2021). "Cómo: intervenciones profilácticas para la prevención de la infección por Clostridioides difficile". Microbiología clínica e infecciones . 27 (12): 1777–1783. doi : 10.1016/j.cmi.2021.06.037 . hdl : 1887/3249077 . PMID 34245901.
- ^ Nealson KH (enero de 1999). "Microbiología post-vikinga: nuevos enfoques, nuevos datos, nuevos conocimientos". Orígenes de la vida y evolución de la biosfera . 29 (1): 73–93. Bibcode :1999OLEB...29...73N. doi :10.1023/A:1006515817767. PMID 11536899. S2CID 12289639.
- ^ Xu J (junio de 2006). "Ecología microbiana en la era de la genómica y la metagenómica: conceptos, herramientas y avances recientes". Ecología molecular . 15 (7): 1713–31. doi : 10.1111/j.1365-294X.2006.02882.x . PMID 16689892. S2CID 16374800.
- ^ Zillig W (diciembre de 1991). "Bioquímica comparativa de arqueas y bacterias". Current Opinion in Genetics & Development . 1 (4): 544–51. doi :10.1016/S0959-437X(05)80206-0. PMID 1822288.
- ^ abc Slonczewski JL, Foster JW. Microbiología: una ciencia en evolución (3.ª ed.). WW Norton & Company. págs. 491–44.
- ^ Hellingwerf KJ, Crielaard W, Hoff WD, Matthijs HC, Mur LR, van Rotterdam BJ (1994). "Fotobiología de bacterias". Antonie van Leeuwenhoek (manuscrito enviado). 65 (4): 331–47. doi :10.1007/BF00872217. PMID 7832590. S2CID 23438926.
- ^ Dalton H (junio de 2005). "La conferencia Leeuwenhoek de 2000: la historia natural y no natural de las bacterias oxidantes de metano". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias biológicas . 360 (1458): 1207–22. doi :10.1098/rstb.2005.1657. PMC 1569495. PMID 16147517 .
- ^ Imran A, Hakim S, Tariq M, Nawaz MS, Laraib I, Gulzar U, Hanif MK, Siddique MJ, Hayat M, Fraz A, Ahmad M (2021). "Diazotrofos para reducir las crisis de contaminación por nitrógeno: una mirada profunda a las raíces". Frontiers in Microbiology . 12 : 637815. doi : 10.3389/fmicb.2021.637815 . PMC 8180554 . PMID 34108945.
- ^ Zehr JP, Jenkins BD, Short SM, Steward GF (julio de 2003). "Diversidad de genes de la nitrogenasa y estructura de la comunidad microbiana: una comparación entre sistemas". Microbiología ambiental . 5 (7): 539–54. Bibcode :2003EnvMi...5..539Z. doi : 10.1046/j.1462-2920.2003.00451.x . PMID 12823187.
- ^ Kosugi Y, Matsuura N, Liang Q, Yamamoto-Ikemoto R (octubre de 2020). "Tratamiento de aguas residuales mediante el proceso de "Reducción de sulfato, desnitrificación Anammox y nitrificación parcial (SRDAPN)"". Chemosphere . 256 : 127092. Bibcode :2020Chmsp.25627092K. doi :10.1016/j.chemosphere.2020.127092. PMID 32559887. S2CID 219476361.
- ^ Morel FM, Kraepiel AM, Amyot M (1998). "El ciclo químico y la bioacumulación del mercurio". Revista anual de ecología y sistemática . 29 : 543–66. doi :10.1146/annurev.ecolsys.29.1.543. S2CID 86336987.
- ^ Ślesak I, Kula M, Ślesak H, Miszalski Z, Strzałka K (agosto de 2019). "¿Cómo definir la anaerobiosis obligatoria? Una visión evolutiva del sistema de respuesta antioxidante y las primeras etapas de la evolución de la vida en la Tierra". Biología y medicina de radicales libres . 140 : 61–73. doi : 10.1016/j.freeradbiomed.2019.03.004 . PMID 30862543.
- ^ Stewart EJ, Madden R, Paul G, Taddei F (febrero de 2005). "Envejecimiento y muerte en un organismo que se reproduce por división morfológicamente simétrica". PLOS Biology . 3 (2): e45. doi : 10.1371/journal.pbio.0030045 . PMC 546039 . PMID 15685293.
- ^ Koch AL (2002). "Control del ciclo celular bacteriano mediante crecimiento citoplasmático". Critical Reviews in Microbiology . 28 (1): 61–77. doi :10.1080/1040-840291046696. PMID 12003041. S2CID 11624182.
- ^ Pommerville 2014, pág. 138.
- ^ Pommerville 2014, pág. 557.
- ^ desde Wheelis 2008, pág. 42.
- ^ abc Thomson RB, Bertram H (diciembre de 2001). "Diagnóstico de laboratorio de infecciones del sistema nervioso central". Clínicas de enfermedades infecciosas de Norteamérica . 15 (4): 1047–71. doi :10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ Paerl HW, Fulton RS, Moisander PH, Dyble J (abril de 2001). "Floraciones de algas nocivas en agua dulce, con énfasis en las cianobacterias". TheScientificWorldJournal . 1 : 76–113. doi : 10.1100/tsw.2001.16 . PMC 6083932 . PMID 12805693.
- ^ Challis GL, Hopwood DA (noviembre de 2003). "Sinergia y contingencia como fuerzas impulsoras de la evolución de la producción de metabolitos secundarios múltiples por especies de Streptomyces". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (Supl. 2): 14555–61. Bibcode :2003PNAS..10014555C. doi : 10.1073/pnas.1934677100 . PMC 304118 . PMID 12970466.
- ^ Kooijman SA, Auger P, Poggiale JC, Kooi BW (agosto de 2003). "Pasos cuantitativos en la simbiogénesis y la evolución de la homeostasis". Biological Reviews of the Cambridge Philosophical Society . 78 (3): 435–63. doi :10.1017/S1464793102006127. PMID 14558592. S2CID 41072709.
- ^ Bertrand RL (2019). "La fase de latencia es un período dinámico, organizado, adaptativo y evolutivo que prepara a las bacterias para la división celular". Journal of Bacteriology . 201 (7): e00697-18. doi :10.1128/JB.00697-18. PMC 6416914 . PMID 30642990.
- ^ Prats C, López D, Giró A, Ferrer J, Valls J (agosto de 2006). "Modelado individual de cultivos bacterianos para estudiar las causas microscópicas de la fase de latencia". Journal of Theoretical Biology . 241 (4): 939–53. Bibcode :2006JThBi.241..939P. doi :10.1016/j.jtbi.2006.01.029. PMID 16524598.
- ^ Hecker M, Völker U (2001). Respuesta general al estrés de Bacillus subtilis y otras bacterias . Avances en fisiología microbiana. Vol. 44. págs. 35–91. doi :10.1016/S0065-2911(01)44011-2. ISBN 978-0-12-027744-5. Número de identificación personal 11407115.
- ^ Slonczewski JL, Foster JW. Microbiología: una ciencia en evolución (3.ª ed.). WW Norton & Company. pág. 143.
- ^ Leppänen M, Sundberg LR, Laanto E, de Freitas Almeida GM, Papponen P, Maasilta IJ (agosto de 2017). "Obtención de imágenes de colonias bacterianas y de la interacción fago-bacteria con una resolución subnanométrica mediante microscopía de iones de helio". Advanced Biosystems . 1 (8): e1700070. doi :10.1002/adbi.201700070. PMID 32646179. S2CID 90960276.
- ^ Nakabachi A, Yamashita A, Toh H, Ishikawa H, Dunbar HE, Moran NA, Hattori M (octubre de 2006). "El genoma de 160 kilobases del endosimbionte bacteriano Carsonella". Ciencia . 314 (5797): 267. doi :10.1126/science.1134196. PMID 17038615. S2CID 44570539.
- ^ Pradella S, Hans A, Spröer C, Reichenbach H, Gerth K, Beyer S (diciembre de 2002). "Caracterización, tamaño del genoma y manipulación genética de la mixobacteria Sorangium cellulosum So ce56". Archivos de Microbiología . 178 (6): 484–92. Código Bibliográfico :2002ArMic.178..484P. doi :10.1007/s00203-002-0479-2. PMID 12420170. S2CID 21023021.
- ^ Hinnebusch J, Tilly K (diciembre de 1993). "Plásmidos lineales y cromosomas en bacterias". Microbiología molecular . 10 (5): 917–22. doi :10.1111/j.1365-2958.1993.tb00963.x. PMID 7934868. S2CID 23852021.
- ^ Lin YS, Kieser HM, Hopwood DA, Chen CW (diciembre de 1993). "El ADN cromosómico de Streptomyces lividans 66 es lineal". Microbiología molecular . 10 (5): 923–33. doi :10.1111/j.1365-2958.1993.tb00964.x. PMID 7934869. S2CID 8536066.
- ^ Val ME, Soler-Bistué A, Bland MJ, Mazel D (diciembre de 2014). "Gestión de genomas multipartitos: el modelo de Vibrio cholerae". Current Opinion in Microbiology . 22 : 120–26. doi :10.1016/j.mib.2014.10.003. PMID 25460805. S2CID 215743285.
- ^ Kado CI (octubre de 2014). "Acontecimientos históricos que dieron origen al campo de la biología de plásmidos". Microbiology Spectrum . 2 (5): 3. doi : 10.1128/microbiolspec.PLAS-0019-2013 . PMID 26104369.
- ^ Belfort M , Reaban ME, Coetzee T, Dalgaard JZ (julio de 1995). "Intrones e inteínas procariotas: una panoplia de forma y función". Journal of Bacteriology . 177 (14): 3897–903. doi :10.1128/jb.177.14.3897-3903.1995. PMC 177115 . PMID 7608058.
- ^ Denamur E, Matic I (mayo de 2006). "Evolución de las tasas de mutación en bacterias". Microbiología molecular . 60 (4): 820–27. doi : 10.1111/j.1365-2958.2006.05150.x . PMID 16677295. S2CID 20713095.
- ^ Wright BE (mayo de 2004). "Mutaciones adaptativas dirigidas por estrés y evolución". Microbiología molecular . 52 (3): 643–50. doi : 10.1111/j.1365-2958.2004.04012.x . PMID 15101972. S2CID 1071308.
- ^ Chen I, Dubnau D (marzo de 2004). "Absorción de ADN durante la transformación bacteriana". Nature Reviews. Microbiology . 2 (3): 241–49. doi :10.1038/nrmicro844. PMID 15083159. S2CID 205499369.
- ^ Johnsborg O, Eldholm V, Håvarstein LS (diciembre de 2007). "Transformación genética natural: prevalencia, mecanismos y función". Investigación en microbiología . 158 (10): 767–78. doi : 10.1016/j.resmic.2007.09.004 . PMID 17997281.
- ^ Bernstein H, Bernstein C, Michod RE (2012). "La reparación del ADN como función adaptativa primaria del sexo en bacterias y eucariotas". Capítulo 1: págs. 1–49 en: DNA Repair: New Research , Sakura Kimura y Sora Shimizu (eds.). Nova Sci. Publ., Hauppauge, NY ISBN 978-1-62100-808-8 .
- ^ Brüssow H, Canchaya C, Hardt WD (septiembre de 2004). "Fagos y la evolución de patógenos bacterianos: desde reordenamientos genómicos hasta conversión lisogénica". Microbiology and Molecular Biology Reviews . 68 (3): 560–602, tabla de contenidos. doi :10.1128/MMBR.68.3.560-602.2004. PMC 515249 . PMID 15353570.
- ^ Bickle TA, Krüger DH (junio de 1993). "Biología de la restricción del ADN". Microbiological Reviews . 57 (2): 434–50. doi :10.1128/MMBR.57.2.434-450.1993. PMC 372918 . PMID 8336674.
- ^ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P (marzo de 2007). "CRISPR proporciona resistencia adquirida contra virus en procariotas". Science . 315 (5819): 1709–12. Bibcode :2007Sci...315.1709B. doi :10.1126/science.1138140. hdl : 20.500.11794/38902 . PMID 17379808. S2CID 3888761.
- ^ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, Dickman MJ, Makarova KS, Koonin EV, van der Oost J (agosto de 2008). "Los pequeños ARN CRISPR guían la defensa antiviral en procariotas". Ciencia . 321 (5891): 960–64. Código Bib : 2008 Ciencia... 321.. 960B. doi : 10.1126/ciencia.1159689. PMC 5898235 . PMID 18703739.
- ^ Cabezón E, Ripoll-Rozada J, Peña A, de la Cruz F, Arechaga I (enero de 2015). "Hacia un modelo integrado de conjugación bacteriana". Reseñas de microbiología FEMS . 39 (1): 81–95. doi : 10.1111/1574-6976.12085 . PMID 25154632. S2CID 34245150.
- ^ Michod RE, Bernstein H, Nedelcu AM (mayo de 2008). "Valor adaptativo del sexo en patógenos microbianos" (PDF) . Infección, genética y evolución . 8 (3): 267–85. Bibcode :2008InfGE...8..267M. doi :10.1016/j.meegid.2008.01.002. PMID 18295550. Archivado (PDF) desde el original el 30 de diciembre de 2016.
- ^ Hastings PJ, Rosenberg SM, Slack A (septiembre de 2004). "Transferencia lateral de resistencia a los antibióticos inducida por antibióticos". Tendencias en microbiología . 12 (9): 401–14. doi :10.1016/j.tim.2004.07.003. PMID 15337159.
- ^ Davison J (septiembre de 1999). "Intercambio genético entre bacterias en el medio ambiente". Plasmid . 42 (2): 73–91. doi :10.1006/plas.1999.1421. PMID 10489325.
- ^ abc Kim KW (diciembre de 2017). "Observaciones con microscopio electrónico de apéndices superficiales procariotas". Journal of Microbiology (Seúl, Corea) . 55 (12): 919–26. doi :10.1007/s12275-017-7369-4. PMID 29214488. S2CID 89872403.
- ^ Macnab RM (diciembre de 1999). "El flagelo bacteriano: hélice rotatoria reversible y aparato de exportación tipo III". Journal of Bacteriology . 181 (23): 7149–53. doi :10.1128/JB.181.23.7149-7153.1999. PMC 103673 . PMID 10572114.
- ^ Wu M, Roberts JW, Kim S, Koch DL, DeLisa MP (julio de 2006). "Dinámica bacteriana colectiva revelada utilizando una técnica de seguimiento de partículas desenfocadas a escala poblacional tridimensional". Applied and Environmental Microbiology . 72 (7): 4987–94. Bibcode :2006ApEnM..72.4987W. doi :10.1128/AEM.00158-06. PMC 1489374 . PMID 16820497.
- ^ Mattick JS (2002). "Pili tipo IV y motilidad espasmódica". Revisión anual de microbiología . 56 : 289–314. doi :10.1146/annurev.micro.56.012302.160938. PMID 12142488.
- ^ Merz AJ, So M, Sheetz MP (septiembre de 2000). "La retracción del pilus potencia la motilidad bacteriana por espasmos". Nature . 407 (6800): 98–102. Bibcode :2000Natur.407...98M. doi :10.1038/35024105. PMID 10993081. S2CID 4425775.
- ^ Lux R, Shi W (julio de 2004). "Movimientos en bacterias guiados por quimiotaxis". Critical Reviews in Oral Biology and Medicine . 15 (4): 207–20. doi : 10.1177/154411130401500404 . PMID 15284186.
- ^ Schweinitzer T, Josenhans C (julio de 2010). "Taxis energéticos bacterianos: ¿una estrategia global?". Archivos de Microbiología . 192 (7): 507–20. Bibcode :2010ArMic.192..507S. doi :10.1007/s00203-010-0575-7. PMC 2886117. PMID 20411245 .
- ^ Frankel RB, Bazylinski DA, Johnson MS, Taylor BL (agosto de 1997). "Magnetoaerotaxis en bacterias cocoideas marinas". Revista biofísica . 73 (2): 994–1000. Código Bibliográfico :1997BpJ....73..994F. doi :10.1016/S0006-3495(97)78132-3. PMC 1180996 . PMID 9251816.
- ^ Nan B, Zusman DR (2011). "Descubriendo el misterio de la motilidad de deslizamiento en las mixobacterias". Revisión anual de genética . 45 : 21–39. doi :10.1146/annurev-genet-110410-132547. PMC 3397683. PMID 21910630 .
- ^ Goldberg MB (diciembre de 2001). "Motilidad basada en actina de patógenos microbianos intracelulares". Microbiology and Molecular Biology Reviews . 65 (4): 595–626, tabla de contenidos. doi :10.1128/MMBR.65.4.595-626.2001. PMC 99042 . PMID 11729265.
- ^ Calcagnile M, Tredici SM, Talà A, Alifano P (diciembre de 2019). "Semiquímicos bacterianos e interacciones transreino con insectos y plantas". Insectos . 10 (12): 441. doi : 10.3390/insects10120441 . PMC 6955855 . PMID 31817999.
- ^ ab Shapiro JA (1998). "Pensando en las poblaciones bacterianas como organismos multicelulares" (PDF) . Revista anual de microbiología . 52 : 81–104. doi :10.1146/annurev.micro.52.1.81. PMID 9891794. Archivado desde el original (PDF) el 17 de julio de 2011.
- ^ ab Costerton JW, Lewandowski Z, Caldwell DE, Korber DR, Lappin-Scott HM (1995). "Biopelículas microbianas". Revisión anual de microbiología . 49 : 711–45. doi :10.1146/annurev.mi.49.100195.003431. PMID 8561477.
- ^ Miller DP, Lamont RJ (2019). "Sistemas de señalización en bacterias orales". Inmunidad y microbioma de la mucosa oral . Avances en medicina y biología experimental. Vol. 1197. págs. 27–43. doi :10.1007/978-3-030-28524-1_3. ISBN 978-3-030-28523-4. Número de identificación personal 31732932. Número de identificación personal 208063186.
- ^ Abisado RG, Benomar S, Klaus JR, Dandekar AA, Chandler JR (mayo de 2018). "Detección de quórum bacteriano e interacciones con la comunidad microbiana". mBio . 9 (3). doi :10.1128/mBio.02331-17. PMC 5964356 . PMID 29789364.
- ^ Miller MB, Bassler BL (2001). "Detección de quórum en bacterias". Revisión anual de microbiología . 55 : 165–99. doi :10.1146/annurev.micro.55.1.165. PMID 11544353. S2CID 1099089.
- ^ Zhu Q, Mai U, Pfeiffer W, Janssen S, Asnicar F, Sanders JG, et al. (diciembre de 2019). "La filogenómica de 10.575 genomas revela proximidad evolutiva entre los dominios de bacterias y arqueas". Nature Communications . 10 (1): 5477. Bibcode :2019NatCo..10.5477Z. doi :10.1038/s41467-019-13443-4. PMC 6889312 . PMID 31792218.
- ^ Boucher Y, Douady CJ, Papke RT, Walsh DA, Boudreau ME, Nesbø CL, Case RJ, Doolittle WF (2003). "Transferencia lateral de genes y los orígenes de los grupos procariotas". Revisión anual de genética . 37 : 283–328. doi :10.1146/annurev.genet.37.050503.084247. PMID 14616063.
- ^ Olsen GJ, Woese CR, Overbeek R (enero de 1994). "Los vientos del cambio (evolutivo): insuflando nueva vida a la microbiología". Journal of Bacteriology . 176 (1): 1–6. doi :10.2172/205047. PMC 205007 . PMID 8282683.
- ^ "IJSEM Home". Ijs.sgmjournals.org. 28 de octubre de 2011. Archivado desde el original el 19 de octubre de 2011. Consultado el 4 de noviembre de 2011 .
- ^ "Bergey's Manual Trust". Bergeys.org. Archivado desde el original el 7 de noviembre de 2011. Consultado el 4 de noviembre de 2011 .
- ^ Hedlund BP, Dodsworth JA, Staley JT (junio de 2015). "El panorama cambiante de la exploración de la biodiversidad microbiana y sus implicaciones para la sistemática". Microbiología sistemática y aplicada . 38 (4): 231–36. Bibcode :2015SyApM..38..231H. doi :10.1016/j.syapm.2015.03.003. PMID 25921438.
- ^ "Schizomycetes". Diccionario médico Merriam-Webster. Consultado el 3 de agosto de 2021.
- ^ Brown MM, Horswill AR (noviembre de 2020). "Staphylococcus epidermidis: ¿amigo o enemigo de la piel?". PLOS Pathogens . 16 (11): e1009026. doi : 10.1371/journal.ppat.1009026 . PMC 7660545 . PMID 33180890.
- ^ Gupta RS (2000). "Las relaciones evolutivas naturales entre procariotas". Critical Reviews in Microbiology . 26 (2): 111–31. CiteSeerX 10.1.1.496.1356 . doi :10.1080/10408410091154219. PMID 10890353. S2CID 30541897.
- ^ Rappé MS, Giovannoni SJ (2003). "La mayoría microbiana no cultivada". Revisión anual de microbiología . 57 : 369–94. doi :10.1146/annurev.micro.57.030502.090759. PMID 14527284. S2CID 10781051.
- ^ Doolittle RF (junio de 2005). "Aspectos evolutivos de la biología del genoma completo". Current Opinion in Structural Biology . 15 (3): 248–53. doi :10.1016/j.sbi.2005.04.001. PMID 15963888.
- ^ Cavalier-Smith T (enero de 2002). "El origen neomurano de las arqueobacterias, la raíz negibacteriana del árbol universal y la megaclasificación bacteriana". Revista internacional de microbiología sistemática y evolutiva . 52 (Pt 1): 7–76. doi : 10.1099/00207713-52-1-7 . PMID 11837318.
- ^ Pommerville 2014, pág. 15−31.
- ^ desde Krasner 2014, pág. 77.
- ^ Woods GL, Walker DH (julio de 1996). "Detección de infecciones o agentes infecciosos mediante el uso de tinciones citológicas e histológicas". Clinical Microbiology Reviews . 9 (3): 382–404. doi :10.1128/CMR.9.3.382. PMC 172900 . PMID 8809467.
- ^ Tang S, Orsi RH, Luo H, Ge C, Zhang G, Baker RC, Stevenson A, Wiedmann M (2019). "Evaluación y comparación de métodos de caracterización y subtipificación molecular para Salmonella". Frontiers in Microbiology . 10 : 1591. doi : 10.3389/fmicb.2019.01591 . PMC 6639432 . PMID 31354679.
- ^ Krasner 2014, págs. 87–89.
- ^ Weinstein MP (marzo de 1994). "Importancia clínica de los hemocultivos". Clinics in Laboratory Medicine . 14 (1): 9–16. doi :10.1016/S0272-2712(18)30390-1. PMID 8181237.
- ^ Riley LW (noviembre de 2018). "Métodos de laboratorio en epidemiología molecular: infecciones bacterianas". Microbiology Spectrum . 6 (6). doi :10.1128/microbiolspec.AME-0004-2018. PMID 30387415. S2CID 54234977.
- ^ Lenkowski M, Nijakowski K, Kaczmarek M, Surdacka A (marzo de 2021). "La técnica de amplificación isotérmica mediada por bucle en el diagnóstico periodontal: una revisión sistemática". Revista de medicina clínica . 10 (6): 1189. doi : 10.3390/jcm10061189 . PMC 8000232 . PMID 33809163.
- ^ Alizadeh M, Yousefi L, Pakdel F, Ghotaslou R, Rezaee MA, Khodadadi E, Oskouei MA, Soroush Barhaghi MH, Kafil HS (2021). "Aplicaciones de la espectroscopia de masas MALDI-TOF en microbiología clínica". Avances en ciencias farmacológicas y farmacéuticas . 2021 : 9928238. doi : 10.1155/2021/9928238 . PMC 8121603. PMID 34041492 .
- ^ Dudek NK, Sun CL, Burstein D (2017). "Nueva diversidad microbiana y potencial funcional en el microbioma oral de los mamíferos marinos" (PDF) . Current Biology . 27 (24): 3752–62. Bibcode :2017CBio...27E3752D. doi : 10.1016/j.cub.2017.10.040 . PMID 29153320. S2CID 43864355.
- ^ Louie M, Louie L, Simor AE (agosto de 2000). "El papel de la tecnología de amplificación de ADN en el diagnóstico de enfermedades infecciosas". CMAJ . 163 (3): 301–09. doi :10.1016/s1381-1169(00)00220-x. PMC 80298 . PMID 10951731. Archivado desde el original el 14 de junio de 2006.
- ^ Oliver JD (febrero de 2005). «El estado viable pero no cultivable en bacterias». Journal of Microbiology . 43 Spec No: 93–100. PMID 15765062. Archivado desde el original el 28 de septiembre de 2007.
- ^ Euzéby JP (8 de diciembre de 2011). «Número de nombres publicados». Lista de nombres procariotas con posición en la nomenclatura . Archivado desde el original el 19 de enero de 2012. Consultado el 10 de diciembre de 2011 .
- ^ Curtis TP, Sloan WT, Scannell JW (agosto de 2002). "Estimación de la diversidad procariota y sus límites". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (16): 10494–99. Bibcode :2002PNAS...9910494C. doi : 10.1073/pnas.142680199 . PMC 124953 . PMID 12097644.
- ^ Schloss PD, Handelsman J (diciembre de 2004). "Estado del censo microbiano". Microbiology and Molecular Biology Reviews . 68 (4): 686–91. doi :10.1128/MMBR.68.4.686-691.2004. PMC 539005 . PMID 15590780.
- ^ Oren A, Garrity GM (2021). "Publicación válida de los nombres de cuarenta y dos filos de procariotas". Int J Syst Evol Microbiol . 71 (10): 5056. doi : 10.1099/ijsem.0.005056 . PMID 34694987. S2CID 239887308.
- ^ Fisher B, Harvey RP, Champe PC (2007). "Capítulo 33". Reseñas ilustradas de Lippincott: microbiología (Serie Reseñas ilustradas de Lippincott) . Hagerstwon, MD: Lippincott Williams & Wilkins. págs. 367–92. ISBN 978-0-7817-8215-9.
- ^ Mushegian AA, Ebert D (enero de 2016). "Repensar el" mutualismo "en diversas comunidades huéspedes-simbiontes". Bioensayos . 38 (1): 100–8. doi :10.1002/bies.201500074. PMID 26568407. S2CID 31661712.
- ^ Harper D. "comensalismo". Diccionario Etimológico Online .
- ^ Sears CL (octubre de 2005). "Una asociación dinámica: celebrando nuestra flora intestinal". Anaerobe . 11 (5): 247–51. doi :10.1016/j.anaerobe.2005.05.001. PMID 16701579.
- ^ Khan R, Petersen FC, Shekhar S (2019). "Bacterias comensales: un actor emergente en la defensa contra patógenos respiratorios". Frontiers in Immunology . 10 : 1203. doi : 10.3389/fimmu.2019.01203 . PMC 6554327 . PMID 31214175.
- ^ Roscoe DL, Chow AW (marzo de 1988). "Flora normal e inmunidad de las mucosas de la cabeza y el cuello". Clínicas de enfermedades infecciosas de Norteamérica . 2 (1): 1–19. doi :10.1016/S0891-5520(20)30163-X. PMID 3074102.
- ^ Shiao SL, Kershaw KM, Limon JJ, You S, Yoon J, Ko EY, Guarnerio J, Potdar AA, McGovern DP, Bose S, Dar TB, Noe P, Lee J, Kubota Y, Maymi VI, Davis MJ, Henson RM, Choi RY, Yang W, Tang J, Gargus M, Prince AD, Zumsteg ZS, Underhill DM (julio de 2021). "Las bacterias y hongos comensales regulan diferencialmente las respuestas tumorales a la radioterapia". Célula cancerosa . 39 (9): 1202–1213.e6. doi :10.1016/j.ccell.2021.07.002. PMC 8830498 . PMID 34329585.
- ^ Lyapichev KA, Ivashkevich Y, Chernov Y, Chinenov D, Shpot E, Bessonov AA, Dabaja BS, Konoplev S (2021). "El linfoma MALT de la vejiga urinaria muestra un marcado predominio femenino, una distribución geográfica desigual y una posible etiología infecciosa". Investigación e informes en urología . 13 : 49–62. doi : 10.2147/RRU.S283366 . PMC 7873029 . PMID 33575225.
- ^ Eleyan L, Khan AA, Musollari G, Chandiramani AS, Shaikh S, Salha A, Tarmahomed A, Harky A (abril de 2021). "Endocarditis infecciosa en la población pediátrica". Revista Europea de Pediatría . 180 (10): 3089–3100. doi :10.1007/s00431-021-04062-7. PMID 33852085. S2CID 233225250.
- ^ Martin MO (septiembre de 2002). "Procariotas depredadores: una oportunidad de investigación emergente". Revista de microbiología molecular y biotecnología . 4 (5): 467–77. PMID 12432957.
- ^ Velicer GJ, Stredwick KL (agosto de 2002). "Evolución social experimental con Myxococcus xanthus". Antonie van Leeuwenhoek . 81 (1–4): 155–64. doi :10.1023/A:1020546130033. PMID 12448714. S2CID 20018104.
- ^ Bauer A, Forchhammer K (mayo de 2021). "Depredación bacteriana de cianobacterias". Fisiología microbiana . 31 (2): 99–108. doi : 10.1159/000516427 . ISSN 2673-1665. PMID 34010833.
- ^ Velicer GJ, Mendes-Soares H (enero de 2009). "Depredadores bacterianos". Current Biology . 19 (2): R55–56. Bibcode :2009CBio...19..R55V. doi : 10.1016/j.cub.2008.10.043 . PMID 19174136. S2CID 5432036.
- ^ Stams AJ, de Bok FA, Plugge CM, van Eekert MH, Dolfing J, Schraa G (marzo de 2006). "Transferencia de electrones exocelular en comunidades microbianas anaeróbicas". Microbiología ambiental . 8 (3): 371–82. Bibcode :2006EnvMi...8..371S. doi :10.1111/j.1462-2920.2006.00989.x. PMID 16478444.
- ^ Zinser ER (agosto de 2018). "Protección cruzada contra el peróxido de hidrógeno por microbios auxiliares: los impactos en la cianobacteria Prochlorococcus y otros beneficiarios en las comunidades marinas". Informes de microbiología ambiental . 10 (4): 399–411. Bibcode :2018EnvMR..10..399Z. doi : 10.1111/1758-2229.12625 . PMID 29411546.
- ^ Barea JM, Pozo MJ, Azcón R, Azcón-Aguilar C (julio 2005). "Cooperación microbiana en la rizosfera". Journal of Experimental Botany . 56 (417): 1761–78. doi : 10.1093/jxb/eri197 . PMID 15911555.
- ^ O'Hara AM, Shanahan F (julio de 2006). "La flora intestinal como un órgano olvidado". EMBO Reports . 7 (7): 688–93. doi :10.1038/sj.embor.7400731. PMC 1500832 . PMID 16819463.
- ^ Zoetendal EG, Vaughan EE, de Vos WM (marzo de 2006). "Un mundo microbiano dentro de nosotros". Microbiología molecular . 59 (6): 1639–50. doi : 10.1111/j.1365-2958.2006.05056.x . PMID 16553872. S2CID 37602619.
- ^ Gorbach SL (febrero de 1990). "Bacterias del ácido láctico y salud humana". Anales de Medicina . 22 (1): 37–41. doi :10.3109/07853899009147239. PMID 2109988.
- ^ Salminen SJ, Gueimonde M, Isolauri E (mayo de 2005). "Probióticos que modifican el riesgo de enfermedad". The Journal of Nutrition . 135 (5): 1294–98. doi : 10.1093/jn/135.5.1294 . PMID 15867327.
- ^ Watanabe F, Bito T (enero de 2018). "Fuentes de vitamina B12 e interacción microbiana". Experimental Biology and Medicine . 243 (2): 148–58. doi :10.1177/1535370217746612. PMC 5788147 . PMID 29216732.
- ^ Pommerville 2014, págs. 16-21.
- ^ Clark 2010, pág. 215.
- ^ Wheelis 2008, pág. 44.
- ^ Clark 2010, págs. 30, 195, 233, 236.
- ^ Miller AK, Williams SM (julio de 2021). "La infección por Helicobacter pylori provoca efectos tanto protectores como nocivos en la salud y la enfermedad humanas". Genes and Immunity . 22 (4): 218–226. doi :10.1038/s41435-021-00146-4. PMC 8390445 . PMID 34244666.
- ^ Schwarz S, Enne VI, van Duijkeren E (octubre de 2016). "40 años de artículos veterinarios en JAC: ¿qué hemos aprendido?". The Journal of Antimicrobial Chemotherapy . 71 (10): 2681–90. doi : 10.1093/jac/dkw363 . PMID 27660260.
- ^ Fish DN (febrero de 2002). "Terapia antimicrobiana óptima para la sepsis". American Journal of Health-System Pharmacy . 59 (Supl. 1): S13–19. doi : 10.1093/ajhp/59.suppl_1.S13 . PMID 11885408.
- ^ Belland RJ, Ouellette SP, Gieffers J, Byrne GI (febrero de 2004). "Chlamydia pneumoniae y aterosclerosis". Microbiología celular . 6 (2): 117–27. doi : 10.1046/j.1462-5822.2003.00352.x . PMID 14706098. S2CID 45218449.
- ^ Heise ER (febrero de 1982). "Enfermedades asociadas con la inmunosupresión". Environmental Health Perspectives . 43 : 9–19. doi :10.2307/3429162. JSTOR 3429162. PMC 1568899 . PMID 7037390.
- ^ Saiman L (2004). "Microbiología de la enfermedad pulmonar por fibrosis quística temprana". Paediatric Respiratory Reviews . 5 (Supl A): S367–69. doi :10.1016/S1526-0542(04)90065-6. PMID 14980298.
- ^ Pommerville 2014, pág. 118.
- ^ desde Pommerville 2014, págs. 646–47.
- ^ Krasner 2014, págs. 165, 369.
- ^ Yonath A, Bashan A (2004). "Cristalografía ribosomal: la iniciación, la formación de enlaces peptídicos y la polimerización de aminoácidos se ven obstaculizadas por los antibióticos". Revisión anual de microbiología . 58 : 233–51. doi : 10.1146/annurev.micro.58.030603.123822 . PMID 15487937.
- ^ Khachatourians GG (noviembre de 1998). "Uso agrícola de antibióticos y evolución y transferencia de bacterias resistentes a los antibióticos". CMAJ . 159 (9): 1129–36. PMC 1229782 . PMID 9835883.
- ^ Kuo J (octubre de 2017). "Procesos de desinfección". Water Environment Research . 89 (10): 1206–44. Bibcode :2017WaEnR..89.1206K. doi : 10.2175/106143017X15023776270278 . PMID 28954657.
- ^ Johnson ME, Lucey JA (abril de 2006). "Principales avances tecnológicos y tendencias en el queso". Journal of Dairy Science . 89 (4): 1174–78. doi : 10.3168/jds.S0022-0302(06)72186-5 . PMID 16537950.
- ^ Krasner 2014, págs. 25-26.
- ^ Cohen Y (diciembre de 2002). "Biorremediación de petróleo mediante tapetes microbianos marinos". Microbiología internacional . 5 (4): 189–93. doi :10.1007/s10123-002-0089-5. PMID 12497184. S2CID 26039323.
- ^ Neves LC, Miyamura TT, Moraes DA, Penna TC, Converti A (2006). "Métodos de biofiltración para la eliminación de residuos fenólicos". Applied Biochemistry and Biotechnology . 129–132 (1–3): 130–52. doi :10.1385/ABAB:129:1:130. PMID 16915636. S2CID 189905816.
- ^ Liese A, Filho MV (diciembre de 1999). "Producción de productos químicos finos mediante biocatálisis". Current Opinion in Biotechnology . 10 (6): 595–603. doi :10.1016/S0958-1669(99)00040-3. PMID 10600695.
- ^ Aronson AI, Shai Y (febrero de 2001). "Por qué las toxinas insecticidas de Bacillus thuringiensis son tan eficaces: características únicas de su modo de acción". FEMS Microbiology Letters . 195 (1): 1–8. doi : 10.1111/j.1574-6968.2001.tb10489.x . PMID 11166987.
- ^ Bozsik A (julio de 2006). "Susceptibilidad de Coccinella septempunctata (Coleoptera: Coccinellidae) adulta a insecticidas con diferentes modos de acción". Pest Management Science . 62 (7): 651–54. doi :10.1002/ps.1221. PMID 16649191.
- ^ Chattopadhyay A, Bhatnagar NB, Bhatnagar R (2004). "Toxinas insecticidas bacterianas". Critical Reviews in Microbiology . 30 (1): 33–54. doi :10.1080/10408410490270712. PMID 15116762. S2CID 1580984.
- ^ Serres MH, Gopal S, Nahum LA, Liang P, Gaasterland T, Riley M (2001). "Una actualización funcional del genoma de Escherichia coli K-12". Genome Biology . 2 (9): RESEARCH0035. doi : 10.1186/gb-2001-2-9-research0035 . PMC 56896 . PMID 11574054.
- ^ Almaas E, Kovács B, Vicsek T, Oltvai ZN, Barabási AL (febrero de 2004). "Organización global de los flujos metabólicos en la bacteria Escherichia coli". Naturaleza . 427 (6977): 839–43. arXiv : q-bio/0403001 . Código Bib : 2004Natur.427..839A. doi : 10.1038/naturaleza02289. PMID 14985762. S2CID 715721.
- ^ Reed JL, Vo TD, Schilling CH, Palsson BO (2003). "Un modelo a escala del genoma expandido de Escherichia coli K-12 (iJR904 GSM/GPR)". Genome Biology . 4 (9): R54. doi : 10.1186/gb-2003-4-9-r54 . PMC 193654 . PMID 12952533.
- ^ Walsh G (abril de 2005). "Insulinas terapéuticas y su fabricación a gran escala". Applied Microbiology and Biotechnology . 67 (2): 151–59. doi :10.1007/s00253-004-1809-x. PMID 15580495. S2CID 5986035.
- ^ Graumann K, Premstaller A (febrero de 2006). "Fabricación de proteínas terapéuticas recombinantes en sistemas microbianos". Biotechnology Journal . 1 (2): 164–86. doi :10.1002/biot.200500051. PMID 16892246. S2CID 24702839.
- ^ Rabsch W, Helm RA, Eisenstark A (febrero de 2004). "Diversidad de tipos de fagos entre cultivos archivados de la colección Demerec de cepas de Salmonella enterica serovar Typhimurium". Microbiología aplicada y ambiental . 70 (2): 664–69. Bibcode :2004ApEnM..70..664R. doi :10.1128/aem.70.2.664-669.2004. PMC 348941 . PMID 14766539.
- ^ Ruedas 2008.
- ^ Asimov I (1982). Enciclopedia biográfica de ciencia y tecnología de Asimov (2.ª ed.). Garden City, Nueva York: Doubleday and Company. pág. 143.
- ^ Pommerville 2014, pág. 7.
- ^ Ehrenberg CG (1828). Symbolae Physioe. Animalia evertebrata . Berlín: Decas prima.
- ^ Breed RS, Conn HJ (mayo de 1936). "El estatus del término genérico Bacterium Ehrenberg 1828". Revista de bacteriología . 31 (5): 517–18. doi :10.1128/jb.31.5.517-518.1936. PMC 543738 . PMID 16559906.
- ^ Ehrenberg CG (1835). Dritter Beitrag zur Erkenntniss grosser Organization in der Richtung des kleinsten Raumes [ Tercera contribución al conocimiento de la gran organización en dirección al espacio más pequeño ] (en alemán). Berlín: Physikalische Abhandlungen der Koeniglichen Akademie der Wissenschaften. págs. 143–336.
- ^ "Los documentos de Pasteur sobre la teoría de los gérmenes". Sitio web sobre derecho médico y de salud pública del Centro de Derecho de la LSU, artículos históricos sobre salud pública. Archivado desde el original el 18 de diciembre de 2006. Consultado el 23 de noviembre de 2006 .
- ^ "'Lávate las manos' fue un consejo médico controvertido en su momento". Historia . 6 de marzo de 2020. Archivado desde el original el 11 de agosto de 2023.
- ^ "El Premio Nobel de Fisiología o Medicina 1905". Nobelprize.org. Archivado desde el original el 10 de diciembre de 2006. Consultado el 22 de noviembre de 2006 .
- ^ O'Brien SJ, Goedert JJ (octubre de 1996). "El VIH causa el SIDA: los postulados de Koch se cumplen". Current Opinion in Immunology . 8 (5): 613–18. doi :10.1016/S0952-7915(96)80075-6. PMID 8902385.
- ^ Chung KT. "Ferdinand Julius Cohn (1828–1898): pionero de la bacteriología" (PDF) . Departamento de Microbiología y Ciencias Celulares Moleculares, Universidad de Memphis. Archivado (PDF) desde el original el 27 de julio de 2011.
- ^ Drews, Gerhart (1999). «Ferdinand Cohn, fundador de la microbiología moderna» (PDF) . ASM News . 65 (8): 547–52. Archivado desde el original (PDF) el 13 de julio de 2017.
- ^ Thurston AJ (diciembre de 2000). "De sangre, inflamación y heridas de bala: la historia del control de la sepsis". The Australian and New Zealand Journal of Surgery . 70 (12): 855–61. doi : 10.1046/j.1440-1622.2000.01983.x . PMID 11167573.
- ^ Schwartz RS (marzo de 2004). "Las balas mágicas de Paul Ehrlich". The New England Journal of Medicine . 350 (11): 1079–80. doi :10.1056/NEJMp048021. PMID 15014180.
- ^ "Biografía de Paul Ehrlich". Nobelprize.org. Archivado desde el original el 28 de noviembre de 2006. Consultado el 26 de noviembre de 2006 .
- ^ Woese CR, Fox GE (noviembre de 1977). "Estructura filogenética del dominio procariota: los reinos primarios". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 74 (11): 5088–90. Bibcode :1977PNAS...74.5088W. doi : 10.1073/pnas.74.11.5088 . PMC 432104 . PMID 270744.
Bibliografía
- Clark D (2010). Gérmenes, genes y civilización: cómo las epidemias moldearon quiénes somos hoy . Upper Saddle River, NJ: FT Press. ISBN 978-0-13-701996-0.OCLC 473120711 .
- Crawford D (2007). Compañeros mortales: cómo los microbios moldearon nuestra historia . Oxford Nueva York: Oxford University Press. ISBN 978-0-19-956144-5.OCLC 183198723 .
- Hall B (2008). La evolución de Strickberger: la integración de genes, organismos y poblaciones . Sudbury, Mass.: Jones y Bartlett. ISBN 978-0-7637-0066-9.OCLC 85814089 .
- Krasner R (2014). El desafío microbiano: una perspectiva de salud pública . Burlington, Mass.: Jones & Bartlett Learning. ISBN 978-1-4496-7375-8.OCLC 794228026 .
- Pommerville JC (2014). Fundamentos de microbiología (10.ª ed.). Boston: Jones y Bartlett. ISBN 978-1-284-03968-9.
- Wheelis M (2008). Principios de la microbiología moderna . Sudbury, Mass.: Jones and Bartlett Publishers. ISBN 978-0-7637-1075-0.OCLC 67392796 .
Enlaces externos
- Libro de texto en línea sobre bacteriología (2015)