Interferencia de ARN
La interferencia de ARN ( RNAi ) es un proceso biológico en el que las moléculas de ARN participan en la supresión específica de secuencia de la expresión génica por ARN bicatenario , a través de la represión traduccional o transcripcional. Históricamente, la RNAi se conocía con otros nombres, entre ellos cosupresión , silenciamiento génico postranscripcional (PTGS) y quelling . El estudio detallado de cada uno de estos procesos aparentemente diferentes dilucidó que la identidad de estos fenómenos era en realidad RNAi. Andrew Fire y Craig C. Mello compartieron el Premio Nobel de Fisiología o Medicina de 2006 por su trabajo sobre RNAi en el gusano nematodo Caenorhabditis elegans , que publicaron en 1998. Desde el descubrimiento de RNAi y sus potenciales reguladores, se ha hecho evidente que RNAi tiene un inmenso potencial en la supresión de genes deseados. RNAi ahora se conoce como preciso, eficiente, estable y mejor que la terapia antisentido para la supresión génica. [1] Se puede desarrollar ARN antisentido producido intracelularmente por un vector de expresión y encontrarlo útil como nuevo agente terapéutico. [2]
Dos tipos de pequeñas moléculas de ácido ribonucleico (ARN), el microARN (miARN) y el pequeño ARN interferente ( siARN ), son fundamentales para los componentes de la vía del ARNi. Una vez que se degrada el ARNm, se produce un silenciamiento postranscripcional a medida que se impide la traducción de proteínas. La transcripción se puede inhibir a través del mecanismo de silenciamiento pretranscripcional del ARNi, a través del cual un complejo enzimático cataliza la metilación del ADN en posiciones genómicas complementarias al ARNi o miARN complejado. El ARNi tiene un papel importante en la defensa de las células contra secuencias de nucleótidos parásitas (p. ej., virus o transposones ) y también influye en el desarrollo de los organismos.
La vía de RNAi es un proceso natural que se encuentra en muchos eucariotas y células animales. Es iniciada por la enzima Dicer , que escinde moléculas largas de ARN bicatenario (dsRNA) en fragmentos cortos de doble cadena de aproximadamente 21 a 23 nucleótidos de siRNA. Cada siRNA se desenrolla en dos ARN monocatenarios (ssRNA), la cadena pasajera (sentido) y la cadena guía (antisentido). La cadena pasajera es luego escindida por la proteína Argonaute 2 (Ago2). La cadena pasajera se degrada y la cadena guía se incorpora al complejo de silenciamiento inducido por ARN (RISC). El ensamblaje RISC luego se une y degrada el ARNm objetivo. Específicamente, esto se logra cuando la cadena guía se empareja con una secuencia complementaria en una molécula de ARNm e induce la escisión por Ago2, un componente catalítico del RISC. En algunos organismos, este proceso se propaga sistémicamente, a pesar de las concentraciones molares inicialmente limitadas de ARNi. [3]
El ARNi es una herramienta de investigación valiosa, tanto en cultivos celulares como en organismos vivos , porque el ARN bicatenario sintético introducido en las células puede inducir de forma selectiva y robusta la supresión de genes específicos de interés. El ARNi puede utilizarse para análisis a gran escala que inhiben sistemáticamente cada gen (y las proteínas posteriores que codifica) en la célula, lo que puede ayudar a identificar los componentes necesarios para un proceso celular particular o un evento como la división celular . La vía también se utiliza como una herramienta práctica para alimentos, medicamentos e insecticidas . [4]
Mecanismo celular

El RNAi es un proceso de silenciamiento génico dependiente del ARN que está controlado por RISC y es iniciado por moléculas cortas de ARN bicatenario en el citoplasma de una célula, donde interactúan con el componente catalítico RISC Argonaute . [6] Cuando el dsRNA es exógeno (procedente de una infección por un virus con un genoma de ARN o manipulaciones de laboratorio), el ARN se importa directamente al citoplasma y Dicer lo escinde en fragmentos cortos. El dsRNA iniciador también puede ser endógeno (originario de la célula), como en los pre-microRNA expresados a partir de genes codificadores de ARN en el genoma. Las transcripciones primarias de dichos genes se procesan primero para formar la estructura característica de tallo-bucle del pre-miRNA en el núcleo , luego se exportan al citoplasma. Por lo tanto, las dos vías del dsRNA, exógena y endógena, convergen en el RISC. [7]
El dsRNA exógeno inicia el RNAi activando la proteína ribonucleasa Dicer, [8] que se une y escinde los dsRNA en plantas, o los ARN de horquilla corta (shRNA) en humanos, para producir fragmentos bicatenarios de 20-25 pares de bases con un saliente de 2 nucleótidos en el extremo 3'. [9] Los estudios bioinformáticos sobre los genomas de múltiples organismos sugieren que esta longitud maximiza la especificidad del gen diana y minimiza los efectos no específicos. [10] Estos fragmentos bicatenarios cortos se denominan siRNA . Estos siRNA luego se separan en hebras simples y se integran en un RISC activo, por RISC-Loading Complex (RLC). RLC incluye Dicer-2 y R2D2, y es crucial para unir Ago2 y RISC. [11] El factor 11 asociado a la proteína de unión a TATA (TAF11) ensambla el RLC al facilitar la tetramerización de Dcr-2-R2D2, lo que aumenta la afinidad de unión al ARNi en 10 veces. La asociación con TAF11 convertiría el complejo R2-D2-Iniciador (RDI) en el RLC. [12] R2D2 lleva dominios de unión al ARN bicatenario en tándem para reconocer el extremo termodinámicamente estable de los dúplex de ARNi , mientras que Dicer-2 lleva el otro extremo menos estable. La carga es asimétrica: el dominio MID de Ago2 reconoce el extremo termodinámicamente estable del ARNi. Por lo tanto, la cadena "pasajera" (sentido) cuyo extremo 5' es descartado por MID es expulsada, mientras que la cadena "guía" (antisentido) guardada coopera con AGO para formar el RISC. [11]
Después de la integración en el RISC, los siRNA se aparean con su ARNm objetivo y lo escinden, evitando así que se lo use como plantilla de traducción . [13] A diferencia del siRNA , un complejo RISC cargado con miRNA escanea los ARNm citoplasmáticos en busca de una posible complementariedad. En lugar de una escisión destructiva (por Ago2), los miRNA se dirigen a las regiones 3' no traducidas (UTR) de los ARNm donde normalmente se unen con una complementariedad imperfecta, bloqueando así el acceso de los ribosomas para la traducción. [14]
El dsRNA exógeno es detectado y unido por una proteína efectora, conocida como RDE-4 en C. elegans y R2D2 en Drosophila , que estimula la actividad de Dicer. [15] Se desconoce el mecanismo que produce esta especificidad de longitud y esta proteína solo se une a dsRNA largos. [15]
En C. elegans, esta respuesta de iniciación se amplifica a través de la síntesis de una población de ARNi "secundarios" durante la cual los ARNi iniciadores o "primarios" producidos por Dicer se utilizan como plantillas. [16] Estos ARNi "secundarios" son estructuralmente distintos de los ARNi producidos por Dicer y parecen ser producidos por una ARN polimerasa dependiente de ARN (RdRP). [17]
MicroARN

Los microARN (miARN) son ARN no codificantes codificados genómicamente que ayudan a regular la expresión génica , particularmente durante el desarrollo . [18] El fenómeno de RNAi, ampliamente definido, incluye los efectos de silenciamiento génico inducidos endógenamente de los miARN, así como el silenciamiento desencadenado por dsARN extraño. Los miARN maduros son estructuralmente similares a los siARN producidos a partir de dsARN exógeno, pero antes de alcanzar la madurez, los miARN primero deben sufrir una extensa modificación postranscripcional . Un miARN se expresa a partir de un gen codificador de ARN mucho más largo como una transcripción primaria conocida como pri-miARN que se procesa, en el núcleo celular , a una estructura de tallo-bucle de 70 nucleótidos llamada pre-miARN por el complejo microprocesador . Este complejo consta de una enzima ARNasa III llamada Drosha y una proteína de unión a dsARN DGCR8 . La porción dsRNA de este pre-miRNA se une y escinde por Dicer para producir la molécula de miRNA madura que se puede integrar en el complejo RISC; por lo tanto, el miRNA y el siRNA comparten la misma maquinaria celular descendente. [19] Primero, el miRNA codificado viral se describió en el virus de Epstein-Barr (VEB). [20] A partir de entonces, se ha descrito un número cada vez mayor de microRNA en virus. VIRmiRNA es un catálogo completo que cubre el microRNA viral, sus objetivos y los miRNA antivirales [21] (ver también el recurso VIRmiRNA: http://crdd.osdd.net/servers/virmirna/).
Los siRNA derivados de precursores de dsRNA largos difieren de los miRNA en que los miRNA, especialmente los de animales, típicamente tienen un apareamiento de bases incompleto con un objetivo e inhiben la traducción de muchos ARNm diferentes con secuencias similares. Por el contrario, los siRNA típicamente tienen un apareamiento de bases perfecto e inducen la escisión del ARNm solo en un único objetivo específico. [22] En Drosophila y C. elegans , los miRNA y siRNA son procesados por proteínas Argonaute y enzimas Dicer distintas. [23] [24]
Tres regiones principales no traducidas y microARN
Las tres regiones no traducidas principales (3'-UTR) de los ARNm suelen contener secuencias reguladoras que provocan la interferencia del ARN postranscripcional. Estas 3'-UTR suelen contener tanto sitios de unión para los miARN como para las proteínas reguladoras. Al unirse a sitios específicos dentro de la 3'-UTR, los miARN pueden disminuir la expresión génica de varios ARNm, ya sea inhibiendo la traducción o causando directamente la degradación de la transcripción. La 3'-UTR también puede tener regiones silenciadoras que se unen a proteínas represoras que inhiben la expresión de un ARNm.
El 3′-UTR a menudo contiene elementos de respuesta a microARN (MRE) . Los MRE son secuencias a las que se unen los microARN. Estos son motivos predominantes dentro de los 3′-UTR. Entre todos los motivos reguladores dentro de los 3′-UTR (por ejemplo, incluidas las regiones silenciadoras), los MRE constituyen aproximadamente la mitad de los motivos.
En 2023, el sitio web miRBase , [25] un archivo de secuencias y anotaciones de miRNA , enumeraba 28.645 entradas en 271 especies biológicas. De estas, 1.917 miRNA se encontraban en loci de miRNA humanos anotados. Se predijo que los miRNA tendrían un promedio de alrededor de cuatrocientos ARNm diana (que afectan la expresión de varios cientos de genes). [26] Friedman et al. [26] estiman que >45.000 sitios diana de miRNA dentro de los 3′UTR de ARNm humano se conservan por encima de los niveles de fondo, y >60% de los genes codificadores de proteínas humanas han estado bajo presión selectiva para mantener el emparejamiento con miRNA.
Experimentos directos muestran que un único miRNA puede reducir la estabilidad de cientos de ARNm únicos. [27] Otros experimentos muestran que un único miRNA puede reprimir la producción de cientos de proteínas, pero que esta represión a menudo es relativamente leve (menos del doble). [28] [29]
Los efectos de la desregulación de la expresión genética por parte de los microARN parecen ser importantes en el cáncer. [30] Por ejemplo, en los cánceres gastrointestinales, se han identificado nueve microARN alterados epigenéticamente y eficaces para regular negativamente las enzimas de reparación del ADN. [31]
Los efectos de la desregulación de la expresión genética por parte de los miRNA también parecen ser importantes en trastornos neuropsiquiátricos, como la esquizofrenia, el trastorno bipolar, la depresión mayor, la enfermedad de Parkinson, la enfermedad de Alzheimer y los trastornos del espectro autista. [32] [33] [34]
Activación y catálisis de RISC
El dsRNA exógeno es detectado y unido por una proteína efectora, conocida como RDE-4 en C. elegans y R2D2 en Drosophila , que estimula la actividad de Dicer. [15] Esta proteína solo se une a dsRNA largos, pero se desconoce el mecanismo que produce esta especificidad de longitud. [15] Esta proteína de unión al ARN facilita luego la transferencia de siRNA escindidos al complejo RISC. [35]
En C. elegans, esta respuesta de iniciación se amplifica a través de la síntesis de una población de ARNi "secundarios" durante la cual los ARNi iniciadores o "primarios" producidos por Dicer se utilizan como plantillas. [16] Estos ARNi "secundarios" son estructuralmente distintos de los ARNi producidos por Dicer y parecen ser producidos por una ARN polimerasa dependiente de ARN (RdRP). [17]


Los componentes activos de un complejo de silenciamiento inducido por ARN (RISC) son endonucleasas llamadas proteínas Argonaute, que escinden la cadena de ARNm diana complementaria a su ARNi unido . [6] Como los fragmentos producidos por Dicer son de doble cadena, cada uno podría, en teoría, producir un ARNi funcional . Sin embargo, solo una de las dos cadenas, conocida como cadena guía , se une a Argonaute y dirige el silenciamiento del gen. La otra cadena anti-guía o cadena pasajera se degrada durante la activación de RISC. [36] Aunque primero se creyó que una helicasa dependiente de ATP separaba estas dos cadenas, [37] el proceso demostró ser independiente de ATP y realizado directamente por los componentes proteicos de RISC. [3] [38] Sin embargo, un análisis cinético in vitro de RNAi en presencia y ausencia de ATP mostró que puede requerirse ATP para desenrollar y eliminar la cadena de ARNm escindida del complejo RISC después de la catálisis. [39] La cadena guía tiende a ser aquella cuyo extremo 5' está emparejado de manera menos estable con su complemento, [40] pero la selección de la cadena no se ve afectada por la dirección en la que Dicer corta el dsRNA antes de la incorporación de RISC. [41] En cambio, la proteína R2D2 puede servir como factor diferenciador al unirse al extremo 5' más estable de la cadena pasajera. [42]
La base estructural de la unión del ARN a la proteína Argonauta se examinó mediante cristalografía de rayos X del dominio de unión de un Argonauta unido al ARN. Aquí, el extremo 5′ fosforilado de la cadena de ARN entra en un bolsillo superficial básico conservado y hace contactos a través de un catión divalente (un átomo con dos cargas positivas) como el magnesio y por apilamiento aromático (un proceso que permite que más de un átomo comparta un electrón pasándolo de un lado a otro) entre el nucleótido 5′ en el ARNi y un residuo de tirosina conservado . Se cree que este sitio forma un sitio de nucleación para la unión del ARNi a su ARNm diana. [43] El análisis del efecto inhibidor de los desajustes en el extremo 5' o 3' de la cadena guía ha demostrado que el extremo 5' de la cadena guía es probablemente responsable de la coincidencia y la unión con el ARNm objetivo, mientras que el extremo 3' es responsable de organizar físicamente el ARNm objetivo en una región RISC favorable a la escisión. [39]
No se entiende cómo el complejo RISC activado localiza los ARNm complementarios dentro de la célula. Aunque se ha propuesto que el proceso de escisión está vinculado a la traducción , la traducción del ARNm diana no es esencial para la degradación mediada por RNAi. [44] De hecho, el RNAi puede ser más eficaz contra los ARNm dianas que no se traducen. [45] Las proteínas Argonaute se localizan en regiones específicas en el citoplasma llamadas P-bodies (también cuerpos citoplasmáticos o cuerpos GW), que son regiones con altas tasas de descomposición del ARNm; [46] La actividad de miRNA también se agrupa en P-bodies. [47] La interrupción de P-bodies disminuye la eficiencia de RNAi, lo que sugiere que son un sitio crítico en el proceso de RNAi. [48]
Silenciamiento transcripcional

Los componentes de la vía RNAi se utilizan en muchos eucariotas en el mantenimiento de la organización y estructura de sus genomas . La modificación de histonas y la inducción asociada de la formación de heterocromatina sirven para regular negativamente los genes pretranscripcionalmente ; [ 50] este proceso se conoce como silenciamiento transcripcional inducido por ARN (RITS), y se lleva a cabo por un complejo de proteínas llamado complejo RITS. En la levadura de fisión , este complejo contiene Argonaute, una proteína cromodominio Chp1 y una proteína llamada Tas3 de función desconocida. [51] Como consecuencia, la inducción y propagación de regiones heterocromáticas requiere las proteínas Argonaute y RdRP. [52] De hecho, la eliminación de estos genes en la levadura de fisión S. pombe interrumpe la metilación de histonas y la formación del centrómero , [53] causando una anafase lenta o estancada durante la división celular . [54] En algunos casos, se ha observado que procesos similares asociados con la modificación de histonas regulan positivamente la transcripción de genes. [55]
El mecanismo por el cual el complejo RITS induce la formación y organización de la heterocromatina no se entiende bien. La mayoría de los estudios se han centrado en la región de tipo de apareamiento en la levadura de fisión, que puede no ser representativa de las actividades en otras regiones/organismos genómicos. En el mantenimiento de las regiones de heterocromatina existentes, RITS forma un complejo con ARNi complementarios a los genes locales y se une de manera estable a las histonas metiladas locales, actuando co-transcripcionalmente para degradar cualquier transcripción de pre-ARNm naciente que sea iniciada por la ARN polimerasa . La formación de dicha región de heterocromatina, aunque no su mantenimiento, depende de Dicer, presumiblemente porque Dicer es necesario para generar el complemento inicial de ARNi que se dirigen a las transcripciones posteriores. [56] Se ha sugerido que el mantenimiento de la heterocromatina funciona como un bucle de retroalimentación de autorreforzamiento, ya que los nuevos ARNi se forman a partir de las transcripciones nacientes ocasionales por RdRP para su incorporación a los complejos RITS locales. [57] La relevancia de las observaciones de las regiones de apareamiento y los centrómeros de la levadura de fisión para los mamíferos no está clara, ya que el mantenimiento de la heterocromatina en las células de mamíferos puede ser independiente de los componentes de la vía del ARNip. [58]
Interacción con la edición de ARN
El tipo de edición de ARN que es más frecuente en eucariotas superiores convierte nucleótidos de adenosina en inosina en dsRNAs a través de la enzima adenosina desaminasa (ADAR). [59] Se propuso originalmente en 2000 que las vías de edición de ARN RNAi y A→I podrían competir por un sustrato de dsRNA común. [60] Algunos pre-miRNAs experimentan edición de ARN A→I [61] [62] y este mecanismo puede regular el procesamiento y la expresión de miRNA maduros. [62] Además, al menos un ADAR mamífero puede secuestrar siRNAs de componentes de la vía RNAi. [63] Otro respaldo para este modelo proviene de estudios sobre cepas de C. elegans ADAR-nulas que indican que la edición de ARN A→I puede contrarrestar el silenciamiento de genes endógenos y transgenes por RNAi. [64]

Variación entre organismos
Los organismos varían en su capacidad para captar dsRNA extraño y usarlo en la vía RNAi. Los efectos de RNAi pueden ser tanto sistémicos como hereditarios en plantas y C. elegans , aunque no en Drosophila o mamíferos. En plantas, se cree que RNAi se propaga por la transferencia de siRNA entre células a través de plasmodesmos (canales en las paredes celulares que permiten la comunicación y el transporte). [37] La heredabilidad proviene de la metilación de promotores diana por RNAi; el nuevo patrón de metilación se copia en cada nueva generación de la célula. [66] Una amplia distinción general entre plantas y animales radica en la orientación de los miRNA producidos endógenamente; en plantas, los miRNA suelen ser perfectamente o casi perfectamente complementarios a sus genes diana e inducen la escisión directa del mRNA por RISC, mientras que los miRNA de los animales tienden a ser más divergentes en secuencia e inducen la represión de la traducción. [65] Este efecto traduccional puede producirse inhibiendo las interacciones de los factores de iniciación de la traducción con la cola de poliadenina del ARNm . [67]
Algunos protozoos eucariotas como Leishmania major y Trypanosoma cruzi carecen por completo de la vía RNAi. [68] [69] La mayoría o todos los componentes también faltan en algunos hongos , más notablemente el organismo modelo Saccharomyces cerevisiae . [70] La presencia de RNAi en otras especies de levadura en ciernes como Saccharomyces castellii y Candida albicans , demuestra además que la inducción de dos proteínas relacionadas con RNAi de S. castellii facilita RNAi en S. cerevisiae . [71] El hecho de que ciertos ascomicetos y basidiomicetos carezcan de vías RNAi indica que las proteínas necesarias para el silenciamiento de ARN se han perdido independientemente de muchos linajes de hongos , posiblemente debido a la evolución de una nueva vía con función similar, o a la falta de ventaja selectiva en ciertos nichos . [72]
Sistemas procariotas relacionados
La expresión génica en procariotas está influenciada por un sistema basado en ARN similar en algunos aspectos al RNAi. En este caso, los genes que codifican ARN controlan la abundancia o la traducción del ARNm al producir un ARN complementario que se une a un ARNm. Sin embargo, estos ARN reguladores no se consideran generalmente análogos a los microARN porque la enzima Dicer no está involucrada. [73] Se ha sugerido que los sistemas de interferencia CRISPR en procariotas son análogos a los sistemas RNAi eucariotas, aunque ninguno de los componentes proteicos es ortólogo . [74]
Funciones biológicas
Esta sección necesita ser actualizada . ( Mayo de 2020 ) |
Inmunidad
El ARNi es una parte vital de la respuesta inmune a los virus y otro material genético extraño , especialmente en plantas donde también puede prevenir la autopropagación de transposones. [75] Plantas como Arabidopsis thaliana expresan múltiples homólogos de Dicer que están especializados para reaccionar de manera diferente cuando la planta está expuesta a diferentes virus. [76] Incluso antes de que la vía del ARNi se entendiera completamente, se sabía que el silenciamiento genético inducido en las plantas podría propagarse por toda la planta en un efecto sistémico y podría transferirse de las plantas de stock a las plantas de vástago a través del injerto . [77] Desde entonces, este fenómeno ha sido reconocido como una característica del sistema inmunológico de la planta que permite que toda la planta responda a un virus después de un encuentro localizado inicial. [78] En respuesta, muchos virus de plantas han desarrollado mecanismos elaborados para suprimir la respuesta del ARNi. [79] Estos incluyen proteínas virales que se unen a fragmentos cortos de ARN bicatenario con extremos salientes de cadena sencilla, como los producidos por Dicer. [80] Algunos genomas de plantas también expresan ARNi endógenos en respuesta a la infección por tipos específicos de bacterias . [81] Estos efectos pueden ser parte de una respuesta generalizada a los patógenos que regula negativamente cualquier proceso metabólico en el huésped que ayude al proceso de infección. [82]
Aunque los animales generalmente expresan menos variantes de la enzima Dicer que las plantas, el RNAi en algunos animales produce una respuesta antiviral. Tanto en Drosophila juvenil como adulta , el RNAi es importante en la inmunidad innata antiviral y es activo contra patógenos como el virus X de Drosophila . [83] [84] Un papel similar en la inmunidad puede operar en C. elegans , ya que las proteínas Argonaute se regulan positivamente en respuesta a virus y gusanos que sobreexpresan componentes de la vía RNAi son resistentes a la infección viral. [85] [86]
El papel del ARNi en la inmunidad innata de los mamíferos es poco conocido y hay relativamente pocos datos disponibles. Sin embargo, la existencia de virus que codifican genes capaces de suprimir la respuesta del ARNi en células de mamíferos puede ser una evidencia a favor de una respuesta inmune de los mamíferos dependiente del ARNi, [87] [88] aunque esta hipótesis ha sido cuestionada por estar poco fundamentada. [89] Se han presentado evidencias de la existencia de una vía antiviral funcional del ARNi en células de mamíferos. [90] [91]
También existen otras funciones del ARNi en virus de mamíferos, como los miRNA expresados por el virus del herpes que pueden actuar como desencadenantes de la organización de la heterocromatina para mediar la latencia viral. [92]
Regulación negativa de los genes
Los miRNA expresados endógenamente, incluyendo tanto los miRNA intrónicos como los intergénicos , son más importantes en la represión traduccional [65] y en la regulación del desarrollo, especialmente en el momento de la morfogénesis y el mantenimiento de tipos de células indiferenciadas o incompletamente diferenciadas como las células madre . [93] El papel de los miRNA expresados endógenamente en la regulación negativa de la expresión génica se describió por primera vez en C. elegans en 1993. [94] En las plantas, esta función se descubrió cuando se demostró que el "microRNA JAW" de Arabidopsis estaba involucrado en la regulación de varios genes que controlan la forma de la planta. [95] En las plantas, la mayoría de los genes regulados por los miRNA son factores de transcripción ; [96] por lo tanto, la actividad de los miRNA es particularmente amplia y regula redes genéticas completas durante el desarrollo modulando la expresión de genes reguladores clave, incluyendo factores de transcripción así como proteínas F-box . [97] En muchos organismos, incluyendo los humanos, los miRNA están vinculados a la formación de tumores y la desregulación del ciclo celular . Aquí, los miRNA pueden funcionar como oncogenes y supresores de tumores . [98]
Evolución
Con base en un análisis filogenético basado en la parsimonia , el ancestro común más reciente de todos los eucariotas probablemente ya poseía una vía temprana de RNAi; se piensa que la ausencia de la vía en ciertos eucariotas es una característica derivada. [99] Este sistema ancestral de RNAi probablemente contenía al menos una proteína similar a Dicer, una Argonauta, una proteína PIWI y una ARN polimerasa dependiente de ARN que también puede haber desempeñado otras funciones celulares. Un estudio de genómica comparativa a gran escala indica asimismo que el grupo corona eucariota ya poseía estos componentes, que pueden haber tenido entonces asociaciones funcionales más estrechas con sistemas generalizados de degradación de ARN como el exosoma . [100] Este estudio también sugiere que la familia de proteínas Argonauta de unión al ARN, que comparten los eucariotas, la mayoría de las arqueas y al menos algunas bacterias (como Aquifex aeolicus ), es homóloga y evolucionó originalmente a partir de componentes del sistema de iniciación de la traducción .
Aplicaciones
Vía de ARNi para la supresión de genes
La desactivación de genes es un método utilizado para reducir la expresión de genes específicos de un organismo. Esto se logra mediante el proceso natural de RNAi. [6] Esta técnica de desactivación de genes utiliza una molécula de ARNi de doble cadena que se sintetiza con una secuencia complementaria al gen de interés. La cascada de RNAi comienza una vez que la enzima Dicer comienza a procesar el ARNi. El resultado final del proceso conduce a la degradación del ARNm y destruye cualquier instrucción necesaria para construir ciertas proteínas. Con este método, los investigadores pueden disminuir (pero no eliminar por completo) la expresión de un gen objetivo. El estudio de los efectos de esta disminución en la expresión puede mostrar el papel fisiológico o el impacto de los productos del gen objetivo. [101] [102]
Efectos fuera del objetivo de la eliminación de genes
Se han hecho grandes esfuerzos en biología computacional para diseñar reactivos de dsRNA exitosos que maximicen la eliminación de genes pero minimicen los efectos "fuera de objetivo". Los efectos fuera de objetivo surgen cuando un ARN introducido tiene una secuencia de bases que puede emparejarse con múltiples genes y, por lo tanto, reducir la expresión de estos. Dichos problemas ocurren con mayor frecuencia cuando el dsRNA contiene secuencias repetitivas. Se ha estimado a partir del estudio de los genomas de humanos, C. elegans y S. pombe que aproximadamente el 10% de los posibles siRNA tienen importantes efectos fuera de objetivo. [10] Se han desarrollado una multitud de herramientas de software que implementan algoritmos para el diseño de siRNA generales [103] [104] específicos de mamíferos, [105] y específicos de virus [106] que se verifican automáticamente para detectar una posible reactividad cruzada.
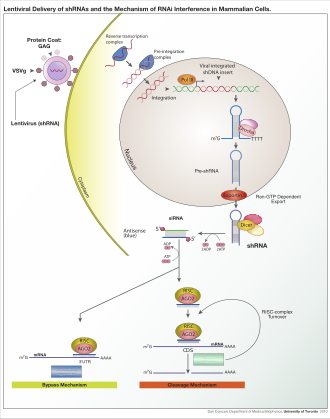
Dependiendo del organismo y del sistema experimental, el ARN exógeno puede ser una cadena larga diseñada para ser escindida por Dicer, o ARN cortos diseñados para servir como sustratos de ARNi . En la mayoría de las células de mamíferos, se utilizan ARN más cortos porque las moléculas de ARN bicatenario largo inducen la respuesta de interferón de mamíferos , una forma de inmunidad innata que reacciona de manera no específica al material genético extraño. [107] Los ovocitos de ratón y las células de embriones de ratón tempranos carecen de esta reacción al ARNbc exógeno y, por lo tanto, son un sistema modelo común para estudiar los efectos de la eliminación de genes de mamíferos. [108] También se han desarrollado técnicas de laboratorio especializadas para mejorar la utilidad del ARNi en sistemas de mamíferos al evitar la introducción directa de ARNi , por ejemplo, mediante la transfección estable con un plásmido que codifica la secuencia apropiada a partir de la cual se pueden transcribir los ARNi , [109] o mediante sistemas de vectores lentivirales más elaborados que permiten la activación o desactivación inducible de la transcripción, conocida como ARNi condicional . [110] [111]
Medicamentos
.jpg/440px-Drosophila_melanogaster_-_side_(aka).jpg)

La técnica de inactivación de genes mediante terapias de ARNi ha demostrado ser exitosa en estudios clínicos controlados aleatorizados. Estos medicamentos son una clase creciente de fármacos basados en ARNi que disminuyen la expresión de proteínas codificadas por ciertos genes. Hasta la fecha, las autoridades regulatorias de los EE. UU. y Europa han aprobado cinco medicamentos de ARNi: patisiran (2018), givosiran (2019), lumasiran (2020), inclisiran (2020 en Europa con aprobación prevista en los EE. UU. en 2021) y vutrisiran (2022). [112] [113] [114] [115]
Si bien todas las terapias de ARNi aprobadas por los organismos reguladores actuales se centran en enfermedades que se originan en el hígado, otros medicamentos bajo investigación apuntan a una serie de áreas de enfermedades que incluyen enfermedades cardiovasculares, trastornos hemorrágicos, trastornos por consumo de alcohol, fibrosis quística, gota, carcinoma y trastornos oculares.
Patisiran es el primer medicamento basado en ARNi de doble cadena aprobado en 2018 y desarrollado por Alnylam Pharmaceuticals . Patisiran utiliza la cascada de ARNi para suprimir el gen que codifica para TTR (transtiretina). Las mutaciones en este gen pueden causar el plegamiento incorrecto de una proteína responsable de la amiloidosis ATTR hereditaria . Para lograr una respuesta terapéutica, patisiran está envuelto por una membrana de nanopartículas lipídicas que facilita el cruce hacia el citoplasma. Una vez dentro de la célula, el ARNi comienza a procesarse por la enzima Dicer. Patisiran es administrado por un profesional de la salud a través de una infusión intravenosa con una dosis basada en el peso corporal. Las advertencias y precauciones incluyen el riesgo de reacciones relacionadas con la infusión y niveles reducidos de vitamina A (suero). [116]
En 2019, la FDA y la EMA aprobaron givosiran para el tratamiento de adultos con porfiria hepática aguda (AHP). [117] La FDA también otorgó a givosiran una designación de terapia innovadora , designación de revisión prioritaria y designación de medicamento huérfano para el tratamiento de la porfiria hepática aguda (AHP) en noviembre de 2019. [118] Para 2020, givosiran recibió la aprobación de la EMA. [119] Givosiran es un ARNi que descompone el ARNm de la sintasa del ácido aminolevulínico 1 (ALAS1) en el hígado. La descomposición del ARNm de ALAS1 evita que se acumulen toxinas (responsables de los ataques neuroviscerales y la enfermedad de AHP) como el ácido aminolevulínico (ALA) y el porfobilinógeno (PBG). [120] [121] [122] [123] Para facilitar la entrada al citoplasma, givosiran utiliza ligandos GalNAc y entra en las células del hígado. El medicamento se administra por vía subcutánea por un profesional sanitario y la dosis se establece en función del peso corporal. Las advertencias y precauciones incluyen el riesgo de reacciones anafilácticas, toxicidad hepática, toxicidad renal y reacciones en el lugar de la inyección. [124]
Lumasiran fue aprobado como un medicamento basado en ARNi en 2020 para su uso tanto en la Unión Europea como en los Estados Unidos. [125] [126] Este medicamento se utiliza para el tratamiento de la hiperoxaluria primaria tipo 1 (PH1) en poblaciones pediátricas y adultas. El fármaco está diseñado para reducir la producción de oxalato hepático y los niveles de oxalato urinario a través de RNAi al dirigirse al ARNm de la hidroxiácido oxidasa 1 (HAO1) para su descomposición. La reducción de los niveles de la enzima HAO1 reduce la oxidación del glicolato a glioxilato (que es un sustrato para el oxalato). Lumasiran es administrado por vía subcutánea por un profesional de la salud con una dosis basada en el peso corporal. [127] Los datos de ensayos clínicos controlados aleatorizados indican que la reacción adversa más común que se notificó fueron reacciones en el lugar de la inyección. Estas reacciones fueron leves y estuvieron presentes en el 38 por ciento de los pacientes tratados con lumasiran. [128]
En 2022, la FDA y la EMA aprobaron el vutrisiran para el tratamiento de adultos con amiloidosis hereditaria mediada por transtiretina con polineuropatía en estadio 1 o 2. [129] [130] El vutrisiran está diseñado para descomponer el ARNm que codifica la transtiretina .
Otros fármacos en investigación que utilizan el ARNip están siendo desarrollados por compañías farmacéuticas como Arrowhead Pharmaceuticals , Dicerna, Alnylam Pharmaceuticals , Amgen y Sylentis. Estos medicamentos cubren una variedad de objetivos a través del ARNip y enfermedades.
Terapias de ARNi en investigación en desarrollo:
| Droga | Objetivo | Sistema de entrega | Enfermedad | Fase | Estado | Compañía | Identificador |
|---|---|---|---|---|---|---|---|
| ALN–VSP02 | KSP y VEGF | PNL | Tumores sólidos | I | Terminado | Productos farmacéuticos Alnylam | NCT01158079 |
| ARNi–EphA2–DOPC | EphA2 | PNL | Cánceres avanzados | I | Reclutamiento | Centro Oncológico MD Anderson | NCT01591356 |
| Atu027 | PKN3 | PNL | Tumores sólidos | I | Terminado | Terapéutica del Silencio | NCT00938574 |
| TKM–080301 | PLK1 | PNL | Cáncer | I | Reclutamiento | Tekmira Farmacéutica | NCT01262235 |
| TKM–100201 | VP24, VP35, L-polimerasa del ébola de Zaire | PNL | Infección por el virus del Ébola | I | Reclutamiento | Tekmira Farmacéutica | NCT01518881 |
| ALN–RSV01 | Nucleocápside del virus respiratorio sincitial | ARNi desnudo | Infecciones por virus respiratorio sincitial | II | Terminado | Productos farmacéuticos Alnylam | NCT00658086 |
| PRO-040201 | ApoB | PNL | Hipercolesterolemia | I | Finalizado | Tekmira Farmacéutica | NCT00927459 |
| ALN–PCS02 | PCSK9 | PNL | Hipercolesterolemia | I | Terminado | Productos farmacéuticos Alnylam | NCT01437059 |
| ALN-TTR02 | TTR | PNL | Amiloidosis mediada por transtiretina | II | Reclutamiento | Productos farmacéuticos Alnylam | NCT01617967 |
| CALAA-01 | RRM2 | Ciclodextrina NP | Tumores sólidos | I | Activo | Farmacéutica Calando | NCT00689065 |
| TD101 | K6a (mutación N171K) | ARNi desnudo | Paquioniquia congénita | I | Terminado | Proyecto Paquioniquia Congénita | NCT00716014 |
| AGN211745 | VEGFR1 | ARNi desnudo | Degeneración macular relacionada con la edad, neovascularización coroidea | II | Finalizado | Alergan | NCT00395057 |
| Certificado de aptitud para el empleo (QPI-1007) | CASP2 | ARNi desnudo | Atrofia óptica, neuropatía óptica isquémica anterior no arterítica | I | Terminado | Productos farmacéuticos Quark | NCT01064505 |
| I5NP | pág. 53 | ARNi desnudo | Lesión renal, insuficiencia renal aguda | I | Terminado | Productos farmacéuticos Quark | NCT00554359 |
| Retraso en la función del injerto, complicaciones del trasplante renal | Yo, yo | Reclutamiento | Productos farmacéuticos Quark | NCT00802347 | |||
| PF-655 (PF-04523655) | RTP801 (Objetivo propietario) | ARNi desnudo | Neovascularización coroidea, retinopatía diabética, edema macular diabético | II | Activo | Productos farmacéuticos Quark | NCT01445899 |
| Cargador siG12D | KRAS | Polímero LODER | Cáncer de páncreas | II | Reclutamiento | Semilla silenciada | NCT01676259 |
| Bevasiranib | Factor de crecimiento endotelial vascular (VEGF) | ARNi desnudo | Edema macular diabético, degeneración macular | II | Terminado | Salud Opko | NCT00306904 |
| SYL1001 | Virus de la papila traumática 1 | ARNi desnudo | Dolor ocular, síndrome del ojo seco | Yo, yo | Reclutamiento | Sylentis | NCT01776658 |
| SYL040012 | ADRB2 | ARNi desnudo | Hipertensión ocular, glaucoma de ángulo abierto | II | Reclutamiento | Sylentis | NCT01739244 |
| CEQ508 | CTNNB1 | ARNm portador de Escherichia coli | Poliposis adenomatosa familiar | Yo, yo | Reclutamiento | Marina Biotecnología | Desconocido |
| RXi-109 | GFC | Compuesto de ARNip autoadministrable | Prevención de cicatrices cicatriciales | I | Reclutamiento | Productos farmacéuticos RXi | NCT01780077 |
| ALN-TTRsc | TTR | Conjugado de ARNip y GalNAc | Amiloidosis mediada por transtiretina | I | Reclutamiento | Productos farmacéuticos Alnylam | NCT01814839 |
| Arco-520 | Regiones conservadas del VHB | DPC | VHB | I | Reclutamiento | Investigación de Arrowhead | NCT01872065 |
Categorización jurídica y cuestiones jurídicas en un futuro próximo
En la actualidad, tanto los miRNA como los siRNA se sintetizan químicamente y, por lo tanto, están legalmente clasificados dentro de la UE y en los EE. UU. como medicamentos "simples". Pero como los siRNA de bioingeniería (BERA) están en desarrollo, se clasificarían como medicamentos biológicos, al menos en la UE. El desarrollo de la tecnología de los BERA plantea la cuestión de la categorización de los medicamentos que tienen el mismo mecanismo de acción pero se producen química o biológicamente. Esta falta de coherencia debería abordarse. [131]
Mecanismos de entrega
Para lograr el potencial clínico del ARNi, el ARNi debe transportarse de manera eficiente a las células de los tejidos objetivo. Sin embargo, hay varias barreras que deben solucionarse antes de que pueda usarse clínicamente. Por ejemplo, el ARNi "desnudo" es susceptible a varios obstáculos que reducen su eficacia terapéutica. [132] Además, una vez que el ARNi ha ingresado al torrente sanguíneo, el ARN desnudo puede degradarse por nucleasas séricas y puede estimular el sistema inmunológico innato. [132] Debido a su tamaño y naturaleza altamente polianiónica (que contiene cargas negativas en varios sitios), las moléculas de ARNi no modificadas no pueden ingresar fácilmente a las células a través de la membrana celular. Por lo tanto, se debe utilizar ARNi artificial o encapsulado en nanopartículas . Si el ARNi se transfiere a través de la membrana celular, pueden ocurrir toxicidades no deseadas si las dosis terapéuticas no se optimizan, y los ARNi pueden exhibir efectos fuera del objetivo (por ejemplo, regulación negativa no deseada de genes con complementariedad de secuencia parcial ). [133] Incluso después de ingresar a las células, se requiere una dosificación repetida ya que sus efectos se diluyen en cada división celular. En respuesta a estos posibles problemas y barreras, dos enfoques ayudan a facilitar la entrega de ARNi a las células objetivo: nanopartículas lipídicas y conjugados. [134]
Nanopartículas lipídicas
Las nanopartículas lipídicas (LNP) se basan en estructuras similares a liposomas que generalmente están hechas de un centro acuoso rodeado por una capa lipídica. [135] Un subconjunto de estructuras liposomales utilizadas para administrar medicamentos a los tejidos descansan en vesículas unilamelares grandes (LUV) que pueden tener un tamaño de 100 nm. Los mecanismos de administración de LNP se han convertido en una fuente cada vez mayor de ácidos nucleicos envolventes y pueden incluir plásmidos , CRISPR y ARNm . [136]
El primer uso aprobado de nanopartículas lipídicas como mecanismo de administración de fármacos comenzó en 2018 con el fármaco de ARNi patisiran, desarrollado por Alnylam Pharmaceuticals. Dicerna Pharmaceuticals, Persomics , Sanofi y Sirna Therapeutics también trabajaron para llevar terapias de ARNi al mercado. [137] [138]
Otras aplicaciones recientes incluyen dos vacunas contra la COVID-19 aprobadas por la FDA: mRNA-1273, desarrollada por Moderna y BNT162b , desarrollada por una colaboración entre Pfizer y BioNtech . [139] Estas dos vacunas utilizan nanopartículas lipídicas para administrar el ARNm del antígeno. Encapsular la molécula de ARNm en nanopartículas lipídicas fue un avance fundamental para producir vacunas de ARNm viables, resolviendo una serie de barreras técnicas clave en la administración de la molécula de ARNm a la célula huésped, distribuida a través de la apolipoproteína E (apoE) en el receptor de lipoproteína de baja densidad (LDLR). En diciembre de 2020, Novartis anunció que los resultados positivos de los estudios de eficacia de fase III consideraron que el inclisirán era un tratamiento para la hipercolesterolemia familiar heterocigótica (HeFH) y la enfermedad cardiovascular aterosclerótica (ASCVD). [140]
Conjugados
Además de los LNP, las terapias de ARNi se han dirigido a la administración a través de conjugados de ARNi (por ejemplo, GalNAc, carbohidratos, péptidos, aptámeros, anticuerpos). [141] Se han desarrollado terapias que utilizan conjugados de ARNi para enfermedades raras o genéticas como la porfiria hepática aguda (AHP), la hemofilia , la hiperoxaluria primaria (PH) y la amiloidosis ATTR hereditaria , así como otras enfermedades cardiometabólicas como la hipertensión y la esteatohepatitis no alcohólica (NASH). [142]
Biotecnología
El ARNi se ha utilizado para una variedad de otras aplicaciones, incluidos alimentos, cultivos e insecticidas. El uso de la vía del ARNi ha desarrollado numerosos productos, como alimentos como manzanas árticas , tabaco sin nicotina, café descafeinado, vegetación fortificada con nutrientes y cultivos hipoalergénicos. [143] [144] [145] El uso emergente del ARNi tiene el potencial de desarrollar muchos otros productos para uso futuro.
Infección viral
El tratamiento antiviral es una de las primeras aplicaciones médicas basadas en RNAi propuestas, y se han desarrollado dos tipos diferentes. El primer tipo es para dirigirse a los ARN virales. Muchos estudios han demostrado que dirigirse a los ARN virales puede suprimir la replicación de numerosos virus, incluidos el VIH , [146] VPH , [147] hepatitis A , [148] hepatitis B , [149] virus de la influenza , [150] [151] [152] [153] virus respiratorio sincitial (VSR), [153] coronavirus del SARS (SARS-CoV), [153] adenovirus [153] y virus del sarampión . [154] La otra estrategia es bloquear las entradas virales iniciales dirigiéndose a los genes de la célula huésped. [155] Por ejemplo, la supresión de los receptores de quimiocinas ( CXCR4 y CCR5 ) en las células huésped puede prevenir la entrada viral del VIH. [156]
Cáncer
Si bien la quimioterapia tradicional puede matar eficazmente las células cancerosas, la falta de especificidad para discriminar entre células normales y cancerosas en estos tratamientos suele causar efectos secundarios graves. Numerosos estudios han demostrado que el ARNi puede proporcionar un enfoque más específico para inhibir el crecimiento tumoral al dirigirse a los genes relacionados con el cáncer (es decir, el oncogén ). [157] También se ha propuesto que el ARNi puede mejorar la sensibilidad de las células cancerosas a los agentes quimioterapéuticos , lo que proporciona un enfoque terapéutico combinatorio con la quimioterapia. [158] Otro posible tratamiento basado en el ARNi es inhibir la invasión y la migración celular . [159]
En comparación con la quimioterapia u otros fármacos contra el cáncer, los fármacos de ARNi tienen muchas ventajas. [160] El ARNi actúa en la etapa postranscripcional de la expresión génica, por lo que no modifica ni cambia el ADN de forma perjudicial. [160] El ARNi también se puede utilizar para producir una respuesta específica de una determinada manera, como por ejemplo reduciendo la supresión de la expresión génica. [160] En una sola célula cancerosa, el ARNi puede provocar una supresión drástica de la expresión génica con solo unas pocas copias. [160] Esto sucede silenciando los genes que promueven el cáncer con ARNi, así como dirigiéndose a una secuencia de ARNm. [160]
Los fármacos de ARNi tratan el cáncer silenciando ciertos genes que lo promueven. [160] Esto se hace complementando los genes del cáncer con el ARNi, por ejemplo, manteniendo las secuencias de ARNm de acuerdo con el fármaco de ARNi. [160] Lo ideal sería inyectar o modificar químicamente el ARNi para que pueda llegar a las células cancerosas de manera más eficiente. [160] La captación y regulación del ARNi está controlada por los riñones. [160]
Enfermedades neurológicas
Las estrategias de RNAi también muestran potencial para tratar enfermedades neurodegenerativas . Estudios en células y en ratones han demostrado que la focalización específica en genes productores de beta amiloide (por ejemplo, BACE1 y APP) mediante RNAi puede reducir significativamente la cantidad de péptido Aβ que se correlaciona con la causa de la enfermedad de Alzheimer . [161] [162] [163] Además, estos enfoques basados en el silenciamiento también proporcionan resultados prometedores en el tratamiento de la enfermedad de Parkinson y la enfermedad de Poliglutamina . [164] [165] [166]
Estimulación de la respuesta inmune
El sistema inmunológico humano se divide en dos ramas separadas: el sistema inmunológico innato y el sistema inmunológico adaptativo. [167] El sistema inmunológico innato es la primera defensa contra las infecciones y responde a los patógenos de manera genérica. [167] Por otro lado, el sistema inmunológico adaptativo, un sistema que se desarrolló más tarde que el innato, está compuesto principalmente por células B y T altamente especializadas que están entrenadas para reaccionar a porciones específicas de moléculas patógenas. [167]
El desafío entre los patógenos antiguos y los nuevos ha ayudado a crear un sistema de células y partículas protegidas que se denominan marco seguro. [167] Este marco ha proporcionado a los humanos un ejército de sistemas que buscan y destruyen partículas invasoras, como patógenos, organismos microscópicos, parásitos e infecciones. [167] El marco seguro de los mamíferos se ha desarrollado para incorporar ARNi como una herramienta para indicar la contaminación viral, lo que ha permitido que el ARNi cree una intensa respuesta inmune innata. [167]
El ARNi está controlado por el sistema inmunológico innato, que puede dividirse en respuestas inflamatorias agudas y respuestas antivirales. [167] La respuesta inflamatoria se crea con señales de pequeñas moléculas de señalización o citocinas. [167] Estas incluyen interleucina-1 (IL-1), interleucina-6 (IL-6), interleucina-12 (IL-12) y factor de necrosis tumoral α (TNF-α). [167] El sistema inmunológico innato genera inflamación y respuestas antivirales, que causan la liberación de receptores de reconocimiento de patrones (PRR). [167] Estos receptores ayudan a etiquetar qué patógenos son virus, hongos o bacterias. [167] Además, la importancia del ARNi y el sistema inmunológico innato es incluir más PRR para ayudar a reconocer diferentes estructuras de ARN. [167] Esto hace que sea más probable que el ARNi cause una respuesta inmunoestimulante en caso de que aparezca el patógeno. [167]
Alimento
La RNAi se ha utilizado para modificar genéticamente plantas con el fin de que produzcan niveles más bajos de toxinas vegetales naturales. Estas técnicas aprovechan el fenotipo estable y hereditario de la RNAi en las plantas. Las semillas de algodón son ricas en proteínas dietéticas , pero contienen de forma natural el producto terpenoide tóxico gosipol , lo que las hace inadecuadas para el consumo humano. La RNAi se ha utilizado para producir plantas de algodón cuyas semillas contienen niveles reducidos de delta-cadineno sintasa , una enzima clave en la producción de gosipol, sin afectar la producción de la enzima en otras partes de la planta, donde el gosipol es en sí mismo importante para prevenir daños causados por plagas de plantas. [168]
Los esfuerzos de desarrollo han reducido con éxito los niveles de alérgenos en las plantas de tomate [169] y la fortificación de plantas como los tomates con antioxidantes dietéticos . [170] El silenciamiento de la alfa-amilasa mediante ARNi también se ha utilizado para disminuir el crecimiento del hongo Aspergillus flavus en el maíz, que de otro modo habría contaminado los granos con aflatoxinas peligrosas . [171] El silenciamiento de la sintasa del factor lacrimógeno en las cebollas ha producido cebollas sin lágrimas y se ha utilizado ARNi en los genes BP1 de las semillas de colza para mejorar la fotosíntesis. [172] Los genes SBEIIa y SBEIIb del trigo se han utilizado en el trigo para producir niveles más altos de amilosa con el fin de mejorar la función intestinal, [173] y Travella et al. En 2006 se empleó RNAi para la genómica funcional en una investigación de razas de trigo hexaploide , mientras que Scofield et al. utilizaron el silenciamiento génico inducido por virus (VIGS, un subtipo de RNAi) en 2005 para investigar el mecanismo de resistencia proporcionado por Lr21 contra la roya de la hoja del trigo en el trigo hexaploide . [174]
Insecticidas
El RNAi se está desarrollando como insecticida y se emplean múltiples enfoques, entre ellos la ingeniería genética y la aplicación tópica. [4] Las células del intestino medio de algunos insectos absorben las moléculas de dsRNA en un proceso conocido como RNAi ambiental. [175] En algunos insectos, el efecto es sistémico, ya que la señal se propaga por todo el cuerpo del insecto (lo que se conoce como RNAi sistémico). [176]
Los animales expuestos al ARNi en dosis millones de veces superiores a los niveles previstos de exposición humana no muestran efectos adversos. [177] El ARNi tiene efectos variables en diferentes especies de lepidópteros (mariposas y polillas). [178]
Drosophila spp., Bombyx mori , Locusta spp., Spodoptera spp., Tribolium castaneum , Nilaparvata lugens , Helicoverpa armigera y Apis mellifera son modelos que se han utilizado ampliamente para aprender cómo funciona el RNAi dentro de taxones particulares de insectos. Musca domestica tiene dosgenes Ago2 y Glossina morsitans tres, como encontraron Lewis et al. 2016 y Hain et al. 2010. [179] [180] En el caso de lavía miRNA, Diuraphis noxia tiene dos Ago1 , M. domestica dos Dcr1 , Acyrthosiphon pisum dos de Ago1 y Loqs y Dcr1 y cuatro de Pasha . Mientras que en el piRNA , G. morsitans y A. pisum tienen dos o tres Ago3 cada uno. [180] Esto ha llevado a la identificación de futurosobjetivos de desarrollo de insecticidas , y los modos de acción y las razones de la resistencia de otros insecticidas a los insecticidas. [180]
Plantas transgénicas
Se han creado cultivos transgénicos para expresar ARN de doble cadena, cuidadosamente seleccionados para silenciar genes cruciales en las plagas objetivo. Estos ARN de doble cadena están diseñados para afectar solo a los insectos que expresan secuencias genéticas específicas. Como prueba de principio , en 2009 un estudio mostró ARN que podrían matar a cualquiera de las cuatro especies de moscas de la fruta sin dañar a las otras tres. [4]
Actual
Otra posibilidad es suministrar dsRNA sin ingeniería genética. Una de ellas consiste en añadirlos al agua de riego . Las moléculas son absorbidas por el sistema vascular de las plantas y envenenan a los insectos que se alimentan de ellas. Otra posibilidad consiste en rociar dsRNA como un pesticida convencional. Esto permitiría una adaptación más rápida a la resistencia. Tales métodos requerirían fuentes de dsRNA de bajo coste que actualmente no existen. [4]
Genómica funcional
Los enfoques para el diseño de bibliotecas de ARNi de todo el genoma pueden requerir más sofisticación que el diseño de un solo ARNi para un conjunto definido de condiciones experimentales. Las redes neuronales artificiales se utilizan con frecuencia para diseñar bibliotecas de ARNi [181] y para predecir su probable eficiencia en la eliminación de genes. [182] El cribado genómico masivo se considera ampliamente como un método prometedor para la anotación del genoma y ha desencadenado el desarrollo de métodos de cribado de alto rendimiento basados en microarreglos . [183] [184]
Cribado a escala genómica
La investigación de la interferencia del ARN a escala del genoma se basa en la tecnología de cribado de alto rendimiento (HTS, por sus siglas en inglés). La tecnología HTS de la interferencia del ARN permite el cribado de pérdida de función en todo el genoma y se utiliza ampliamente en la identificación de genes asociados con fenotipos específicos. Esta tecnología ha sido aclamada como una posible segunda ola genómica, después de la primera ola genómica de plataformas de descubrimiento de polimorfismos de nucleótidos únicos y microarrays de expresión génica . [185] Una de las principales ventajas del cribado de la interferencia del ARN a escala del genoma es su capacidad de interrogar simultáneamente miles de genes. Con la capacidad de generar una gran cantidad de datos por experimento, el cribado de la interferencia del ARN a escala del genoma ha llevado a una explosión de las tasas de generación de datos. Explotar conjuntos de datos tan grandes es un desafío fundamental, que requiere métodos estadísticos/bioinformáticos adecuados. El proceso básico del cribado de la interferencia del ARN basado en células incluye la elección de una biblioteca de interferencia del ARN, tipos de células robustos y estables, transfección con agentes de interferencia del ARN, tratamiento/incubación, detección de señales, análisis e identificación de genes importantes o dianas terapéuticas. [186]
Historia
Descubrimiento del ARNi

El proceso de RNAi se denominó "co-supresión" y "supresión" cuando se observó antes del conocimiento de un mecanismo relacionado con el ARN. El descubrimiento de RNAi fue precedido primero por observaciones de inhibición transcripcional por ARN antisentido expresado en plantas transgénicas , [188] y más directamente por informes de resultados inesperados en experimentos realizados por científicos de plantas en los Estados Unidos y los Países Bajos a principios de la década de 1990. [189] En un intento de alterar los colores de las flores en petunias , los investigadores introdujeron copias adicionales de un gen que codifica la chalcona sintasa , una enzima clave para la pigmentación de las flores en plantas de petunias de color de flor normalmente rosa o violeta. Se esperaba que el gen sobreexpresado diera como resultado flores más oscuras, pero en cambio causó que algunas flores tuvieran pigmento púrpura menos visible, a veces en patrones abigarrados, lo que indica que la actividad de la chalcona sintasa había disminuido sustancialmente o se había suprimido de una manera específica del contexto. Esto se explicaría más tarde como el resultado de la inserción del transgén adyacente a los promotores en la dirección opuesta en varias posiciones a lo largo de los genomas de algunos transformantes, lo que conduce a la expresión de transcripciones antisentido y al silenciamiento de genes cuando estos promotores están activos. [ cita requerida ] Otra observación temprana de RNAi provino de un estudio del hongo Neurospora crassa , [190] aunque no se reconoció inmediatamente como relacionado. Una investigación adicional del fenómeno en plantas indicó que la regulación negativa se debía a la inhibición postranscripcional de la expresión génica a través de una mayor tasa de degradación del ARNm. [191] Este fenómeno se denominó cosupresión de la expresión génica , pero el mecanismo molecular siguió siendo desconocido. [192]
Poco tiempo después, los virólogos de plantas que trabajaban en mejorar la resistencia de las plantas a las enfermedades virales observaron un fenómeno inesperado similar. Si bien se sabía que las plantas que expresaban proteínas específicas de virus mostraban una mayor tolerancia o resistencia a la infección viral, no se esperaba que las plantas que solo portaban regiones cortas y no codificantes de secuencias de ARN viral mostraran niveles similares de protección. Los investigadores creían que el ARN viral producido por transgenes también podría inhibir la replicación viral. [193] El experimento inverso, en el que se introdujeron secuencias cortas de genes de plantas en virus, mostró que el gen objetivo se suprimía en una planta infectada. [194] Este fenómeno se denominó "silenciamiento génico inducido por virus" (VIGS), [174] y el conjunto de tales fenómenos se denominó colectivamente silenciamiento génico postranscripcional. [195]
Después de estas observaciones iniciales en plantas, los laboratorios buscaron este fenómeno en otros organismos. [196] [197] El primer caso de silenciamiento de ARN en animales se documentó en 1996, cuando Guo y Kemphues observaron que, al introducir ARN sentido y antisentido en el ARNm par-1 en Caenorhabditis elegans , se producía la degradación del mensaje par-1. [198] Se pensaba que esta degradación era provocada por el ARN monocatenario (ssRNA), pero dos años más tarde, en 1998, Fire y Mello descubrieron que esta capacidad de silenciar la expresión del gen par-1 era en realidad provocada por el ARN bicatenario (dsRNA). [198] El artículo de 1998 de Craig C. Mello y Andrew Fire en Nature informó de un potente efecto de silenciamiento genético tras inyectar ARN bicatenario en C. elegans . [199] Al investigar la regulación de la producción de proteínas musculares, observaron que ni las inyecciones de ARNm ni de ARN antisentido tenían efecto sobre la producción de proteínas, pero el ARN bicatenario silenciaba con éxito el gen objetivo. Como resultado de este trabajo, acuñaron el término RNAi . Este descubrimiento representó la primera identificación del agente causal del fenómeno. Fire y Mello recibieron el Premio Nobel de Fisiología o Medicina de 2006. [6] [200]
Terapéutica de ARNi
Justo después del descubrimiento revolucionario de Fire y Mello, Elbashir et al. descubrieron que, al utilizar ARN interferente pequeño (siRNA) fabricado sintéticamente, era posible silenciar secuencias específicas de un gen, en lugar de silenciar el gen entero. [201] Solo un año después, McCaffrey y sus colegas demostraron que este silenciamiento específico de secuencia tenía aplicaciones terapéuticas al apuntar a una secuencia del virus de la hepatitis C en ratones transgénicos . [202] Desde entonces, varios investigadores han estado intentando expandir las aplicaciones terapéuticas del RNAi, buscando específicamente apuntar a genes que causan varios tipos de cáncer . [203] [204] En 2006, las primeras aplicaciones que llegaron a los ensayos clínicos fueron en el tratamiento de la degeneración macular y el virus respiratorio sincitial . [205] Cuatro años más tarde se inició el primer ensayo clínico de fase I en humanos, utilizando un sistema de administración de nanopartículas para apuntar a tumores sólidos . [206]
La FDA aprobó el primer fármaco basado en ARNi ( patisiran ) en 2018. Givosiran y lumasiran obtuvieron posteriormente la aprobación de la FDA para el tratamiento de AHP y PH1 en 2019 y 2020, respectivamente. [112] Inclisiran recibió la aprobación de la EMA en 2020 para el tratamiento del colesterol alto y actualmente está bajo revisión por parte de la FDA. [207]
Véase también
Referencias
- ^ Saurabh S, Vidyarthi AS, Prasad D (marzo de 2014). "Interferencia de ARN: del concepto a la realidad en la mejora de los cultivos". Planta . 239 (3): 543–64. Bibcode :2014Plant.239..543S. doi : 10.1007/s00425-013-2019-5 . PMID 24402564.
- ^ Weiss B, Davidkova G, Zhou LW (marzo de 1999). "Terapia génica con ARN antisentido para estudiar y modular procesos biológicos". Ciencias de la vida celular y molecular . 55 (3): 334–58. doi :10.1007/s000180050296. PMC 11146801. PMID 10228554. S2CID 9448271 .
- ^ ab Matranga C, Tomari Y, Shin C, Bartel DP, Zamore PD (noviembre de 2005). "La escisión de la cadena pasajera facilita el ensamblaje de siRNA en complejos enzimáticos de ARNi que contienen Ago2". Cell . 123 (4): 607–20. doi : 10.1016/j.cell.2005.08.044 . PMID 16271386.
- ^ abcd Kupferschmidt K (agosto de 2013). "Una dosis letal de ARN". Science . 341 (6147): 732–3. Bibcode :2013Sci...341..732K. doi : 10.1126/science.341.6147.732 . PMID 23950525.
- ^ Macrae IJ, Zhou K, Li F, Repic A, Brooks AN, Cande WZ, et al. (enero de 2006). "Base estructural para el procesamiento de ARN bicatenario por Dicer". Science . 311 (5758): 195–8. Bibcode :2006Sci...311..195M. doi :10.1126/science.1121638. PMID 16410517. S2CID 23785494.
- ^ abcd Daneholt B. «Información avanzada: interferencia del ARN». Premio Nobel de Fisiología o Medicina 2006. Archivado desde el original el 20 de enero de 2007. Consultado el 25 de enero de 2007 .
- ^ Bagasra O, Prilliman KR (agosto de 2004). "Interferencia de ARN: el sistema inmunológico molecular". Revista de histología molecular . 35 (6): 545–53. CiteSeerX 10.1.1.456.1701 . doi :10.1007/s10735-004-2192-8. PMID 15614608. S2CID 2966105.
- ^ Bernstein E, Caudy AA, Hammond SM, Hannon GJ (enero de 2001). "Función de una ribonucleasa bidentada en el paso de iniciación de la interferencia del ARN". Nature . 409 (6818): 363–6. Bibcode :2001Natur.409..363B. doi :10.1038/35053110. PMID 11201747. S2CID 4371481.

- ^ Siomi H, Siomi MC (enero de 2009). "En camino hacia la lectura del código de interferencia del ARN". Nature . 457 (7228): 396–404. Bibcode :2009Natur.457..396S. doi :10.1038/nature07754. PMID 19158785. S2CID 205215974.
Zamore PD, Tuschl T, Sharp PA, Bartel DP (marzo de 2000). "RNAi: el ARN bicatenario dirige la escisión dependiente de ATP del ARNm en intervalos de 21 a 23 nucleótidos". Cell . 101 (1): 25–33. doi : 10.1016/S0092-8674(00)80620-0 . PMID 10778853.
Vermeulen A, Behlen L, Reynolds A, Wolfson A, Marshall WS, Karpilow J, et al. (mayo de 2005). "Las contribuciones de la estructura del dsRNA a la especificidad y eficiencia de Dicer". ARN . 11 (5): 674–82. doi :10.1261/rna.7272305. PMC 1370754 . PMID 15811921.
Castanotto D, Rossi JJ (enero de 2009). "Las promesas y los escollos de las terapias basadas en la interferencia del ARN". Nature . 457 (7228): 426–33. Bibcode :2009Natur.457..426C. doi :10.1038/nature07758. PMC 2702667 . PMID 19158789. - ^ ab Qiu S, Adema CM, Lane T (2005). "Un estudio computacional de los efectos fuera del objetivo de la interferencia del ARN". Investigación de ácidos nucleicos . 33 (6): 1834–47. doi :10.1093/nar/gki324. PMC 1072799 . PMID 15800213.
- ^ ab Nakanishi K (septiembre de 2016). "Anatomía de RISC: ¿cómo activan los ARN pequeños y las chaperonas las proteínas Argonaute?". Wiley Interdisciplinary Reviews: RNA . 7 (5): 637–60. doi :10.1002/wrna.1356. PMC 5084781 . PMID 27184117.
- ^ Liang C, Wang Y, Murota Y, Liu X, Smith D, Siomi MC, et al. (septiembre de 2015). "TAF11 ensambla el complejo de carga RISC para mejorar la eficiencia de RNAi". Molecular Cell . 59 (5): 807–18. doi :10.1016/j.molcel.2015.07.006. PMC 4560963 . PMID 26257286.
- ^ Ahlquist P (mayo de 2002). "ARN polimerasas dependientes de ARN, virus y silenciamiento de ARN". Science . 296 (5571): 1270–3. Bibcode :2002Sci...296.1270A. doi :10.1126/science.1069132. PMID 12016304. S2CID 42526536.
- ^ Roberts TC (2015). "La maquinaria de microARN". MicroARN: ciencia básica . Avances en medicina experimental y biología. Vol. 887. págs. 15-30. doi :10.1007/978-3-319-22380-3_2. ISBN 978-3-319-22379-7. Número de identificación personal 26662984.
- ^ abcd Parker G, Eckert D, Bass B (2006). "RDE-4 se une preferentemente a dsRNA largo y su dimerización es necesaria para la escisión de dsRNA a siRNA". ARN . 12 (5): 807–18. doi :10.1261/rna.2338706. PMC 1440910 . PMID 16603715.
- ^ ab Baulcombe DC (enero de 2007). "Biología molecular. Silenciamiento amplificado". Science . 315 (5809): 199–200. doi :10.1126/science.1138030. PMID 17218517. S2CID 46285020.
- ^ ab Pak J, Fire A (enero de 2007). "Poblaciones distintas de efectores primarios y secundarios durante la intercepción del ARN en C. elegans". Science . 315 (5809): 241–4. Bibcode :2007Sci...315..241P. doi :10.1126/science.1132839. PMID 17124291. S2CID 46620298.
- ^ Wang QL, Li ZH (mayo de 2007). "Las funciones de los microARN en plantas". Frontiers in Bioscience . 12 : 3975–82. doi :10.2741/2364. PMC 2851543 . PMID 17485351. S2CID 23014413.
Zhao Y, Srivastava D (abril de 2007). "Una visión evolutiva de la función de los microARN". Tendencias en ciencias bioquímicas . 32 (4): 189–97. doi :10.1016/j.tibs.2007.02.006. PMID 17350266. - ^ Gregory RI, Chendrimada TP, Shiekhattar R (2006). "Biogénesis de microARN: aislamiento y caracterización del complejo microprocesador". Protocolos de microARN . Métodos en biología molecular. Vol. 342. págs. 33–47. doi :10.1385/1-59745-123-1:33. ISBN. 978-1-59745-123-9. Número de identificación personal 16957365.
- ^ Pfeffer S, Zavolan M, Grässer FA, Chien M, Russo JJ, Ju J, et al. (abril de 2004). "Identificación de microARN codificados por virus". Science . 304 (5671): 734–6. Bibcode :2004Sci...304..734P. doi :10.1126/science.1096781. PMID 15118162. S2CID 25287167.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 de enero de 2014). "VIRmiRNA: un recurso completo para los miRNA virales validados experimentalmente y sus objetivos". Base de datos . 2014 : bau103. doi :10.1093/database/bau103. PMC 4224276 . PMID 25380780.
- ^ Pillai RS, Bhattacharyya SN, Filipowicz W (2007). "Represión de la síntesis de proteínas por miRNAs: ¿cuántos mecanismos?". Trends Cell Biol . 17 (3): 118–26. doi :10.1016/j.tcb.2006.12.007. PMID 17197185.
- ^ Okamura K, Ishizuka A, Siomi H, Siomi M (2004). "Funciones distintas de las proteínas Argonautas en pequeñas vías de escisión de ARN dirigidas por ARN". Desarrollo de genes . 18 (14): 1655–66. doi :10.1101/gad.1210204. PMC 478188 . PMID 15231716.
- ^ Lee Y, Nakahara K, Pham J, Kim K, He Z, Sontheimer E, et al. (2004). "Roles distintos de Dicer-1 y Dicer-2 de Drosophila en las vías de silenciamiento de siRNA/miRNA". Cell . 117 (1): 69–81. doi : 10.1016/S0092-8674(04)00261-2 . PMID 15066283.
- ^ miRBase.org
- ^ ab Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "La mayoría de los ARNm de mamíferos son dianas conservadas de los microARN". Genome Res . 19 (1): 92–105. doi :10.1101/gr.082701.108. PMC 2612969 . PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, et al. (febrero de 2005). "El análisis de microarrays muestra que algunos microARN regulan negativamente un gran número de ARNm objetivo". Nature . 433 (7027): 769–73. Bibcode :2005Natur.433..769L. doi :10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (septiembre de 2008). "Cambios generalizados en la síntesis de proteínas inducida por microARN". Nature . 455 (7209): 58–63. Bibcode :2008Natur.455...58S. doi :10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (septiembre de 2008). "El impacto de los microARN en la producción de proteínas". Nature . 455 (7209): 64–71. Bibcode :2008Natur.455...64B. doi :10.1038/nature07242. PMC 2745094 . PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, et al. (Julio de 2011). "Mecanismos y papel de la desregulación de microARN en la aparición y progresión del cáncer". Genética y Biología Molecular . 34 (3): 363–70. doi :10.1590/S1415-47572011000300001. PMC 3168173 . PMID 21931505.
- ^ Bernstein C, Bernstein H (mayo de 2015). "Reducción epigenética de la reparación del ADN en la progresión del cáncer gastrointestinal". Revista mundial de oncología gastrointestinal . 7 (5): 30–46. doi : 10.4251/wjgo.v7.i5.30 . PMC 4434036 . PMID 25987950.
- ^ Maffioletti E, Tardito D, Gennarelli M, Bocchio-Chiavetto L (2014). "Microespías del cerebro a la periferia: nuevas pistas a partir de estudios sobre microARN en trastornos neuropsiquiátricos". Frontiers in Cellular Neuroscience . 8 : 75. doi : 10.3389/fncel.2014.00075 . PMC 3949217 . PMID 24653674.
- ^ Mellios N, Sur M (2012). "El papel emergente de los microARN en la esquizofrenia y los trastornos del espectro autista". Frontiers in Psychiatry . 3 : 39. doi : 10.3389/fpsyt.2012.00039 . PMC 3336189 . PMID 22539927.
- ^ Geaghan M, Cairns MJ (agosto de 2015). "MicroARN y desregulación postranscripcional en psiquiatría". Psiquiatría biológica . 78 (4): 231–9. doi : 10.1016/j.biopsych.2014.12.009 . hdl : 1959.13/1335073 . PMID 25636176.
- ^ Liu Q, Rand TA, Kalidas S, Du F, Kim HE, Smith DP, et al. (septiembre de 2003). "R2D2, un puente entre los pasos de iniciación y efector de la vía de RNAi de Drosophila". Science . 301 (5641): 1921–5. Bibcode :2003Sci...301.1921L. doi :10.1126/science.1088710. PMID 14512631. S2CID 41436233.
- ^ Gregory RI, Chendrimada TP, Cooch N, Shiekhattar R (noviembre de 2005). "El RISC humano acopla la biogénesis de microARN y el silenciamiento génico postranscripcional". Cell . 123 (4): 631–40. doi : 10.1016/j.cell.2005.10.022 . PMID 16271387.
- ^ ab Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, et al. (2004). Biología celular molecular (5.ª ed.). WH Freeman: Nueva York, NY. ISBN 978-0-7167-4366-8.
- ^ Leuschner PJ, Ameres SL, Kueng S, Martinez J (marzo de 2006). "Escisión de la cadena pasajera de ARNi durante el ensamblaje de RISC en células humanas". EMBO Reports . 7 (3): 314–20. doi :10.1038/sj.embor.7400637. PMC 1456892 . PMID 16439995.
- ^ ab Haley B, Zamore PD (julio de 2004). "Análisis cinético del complejo enzimático de ARNi". Nature Structural & Molecular Biology . 11 (7): 599–606. doi :10.1038/nsmb780. PMID 15170178. S2CID 12400060.
- ^ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (octubre de 2003). "Asimetría en el ensamblaje del complejo enzimático de ARNi". Cell . 115 (2): 199–208. doi : 10.1016/S0092-8674(03)00759-1 . PMID 14567917.
- ^ Preall JB, He Z, Gorra JM, Sontheimer EJ (marzo de 2006). "La selección de cadenas cortas de ARN interferente es independiente de la polaridad de procesamiento de dsRNA durante la intercepción de ARN en Drosophila". Current Biology . 16 (5): 530–5. Bibcode :2006CBio...16..530P. doi : 10.1016/j.cub.2006.01.061 . PMID 16527750.
- ^ Tomari Y, Matranga C, Haley B, Martinez N, Zamore PD (noviembre de 2004). "Un sensor de proteínas para la asimetría de ARNip". Ciencia . 306 (5700): 1377–80. Código bibliográfico : 2004 Ciencia... 306.1377T. doi : 10.1126/ciencia.1102755. PMID 15550672. S2CID 31558409.
- ^ Ma JB, Yuan YR, Meister G, Pei Y, Tuschl T, Patel DJ (marzo de 2005). "Base estructural para el reconocimiento específico del extremo 5' del ARN guía por la proteína Piwi de A. fulgidus". Nature . 434 (7033): 666–70. Bibcode :2005Natur.434..666M. doi :10.1038/nature03514. PMC 4694588 . PMID 15800629.
- ^ Sen G, Wehrman T, Blau H (2005). "La traducción del ARNm no es un requisito previo para la escisión del ARNm mediada por ARN interferentes pequeños". Diferenciación . 73 (6): 287–93. doi :10.1111/j.1432-0436.2005.00029.x. PMID 16138829. S2CID 41117614.
- ^ Gu S, Rossi J (2005). "Desacoplamiento de RNAi de la traducción activa en células de mamíferos". ARN . 11 (1): 38–44. doi :10.1261/rna.7158605. PMC 1370689 . PMID 15574516.
- ^ Sen G, Blau H (2005). "Argonaute 2/RISC reside en sitios de desintegración del ARNm de mamíferos conocidos como cuerpos citoplasmáticos". Nat Cell Biol . 7 (6): 633–6. doi :10.1038/ncb1265. PMID 15908945. S2CID 6085169.
- ^ Lian S, Jakymiw A, Eystathioy T, Hamel J, Fritzler M, Chan E (2006). "Cuerpos GW, microARN y el ciclo celular". Ciclo celular . 5 (3): 242–5. doi : 10.4161/cc.5.3.2410 . PMID 16418578.
- ^ Jakymiw A, Lian S, Eystathioy T, Li S, Satoh M, Hamel J, et al. (2005). "La disrupción de los cuerpos P afecta la interferencia del ARN en mamíferos". Nat Cell Biol . 7 (12): 1267–74. doi :10.1038/ncb1334. PMID 16284622. S2CID 36630239.
- ^ Hammond S, Bernstein E, Beach D, Hannon G (2000). "Una nucleasa dirigida por ARN media el silenciamiento génico postranscripcional en células de Drosophila". Nature . 404 (6775): 293–6. Bibcode :2000Natur.404..293H. doi :10.1038/35005107. PMID 10749213. S2CID 9091863.

- ^ Holmquist GP, Ashley T (2006). "Organización cromosómica y modificación de la cromatina: influencia en la función y evolución del genoma". Cytogenetic and Genome Research . 114 (2): 96–125. doi :10.1159/000093326. PMID 16825762. S2CID 29910065.
- ^ Verdel A, Jia S, Gerber S, Sugiyama T, Gygi S, Grewal SI, et al. (enero de 2004). "Orientación de la heterocromatina mediada por ARNi por el complejo RITS". Science . 303 (5658): 672–6. Bibcode :2004Sci...303..672V. doi :10.1126/science.1093686. PMC 3244756 . PMID 14704433.
- ^ Irvine DV, Zaratiegui M, Tolia NH, Goto DB, Chitwood DH, Vaughn MW, et al. (agosto de 2006). "El corte de Argonaute es necesario para el silenciamiento y la propagación heterocromática". Science . 313 (5790): 1134–7. Bibcode :2006Sci...313.1134I. doi :10.1126/science.1128813. PMID 16931764. S2CID 42997104.
- ^ Volpe TA, Kidner C, Hall IM, Teng G, Grewal SI, Martienssen RA (septiembre de 2002). "Regulación del silenciamiento heterocromático y la metilación de la lisina-9 de la histona H3 mediante RNAi". Science . 297 (5588): 1833–7. Bibcode :2002Sci...297.1833V. doi : 10.1126/science.1074973 . PMID 12193640. S2CID 2613813.
- ^ Volpe T, Schramke V, Hamilton GL, White SA, Teng G, Martienssen RA, et al. (2003). "La interferencia del ARN es necesaria para la función normal del centrómero en la levadura de fisión". Investigación cromosómica . 11 (2): 137–46. doi :10.1023/A:1022815931524. PMID 12733640. S2CID 23813417.
- ^ Li LC, Okino ST, Zhao H, Pookot D, Place RF, Urakami S, et al. (noviembre de 2006). "Small dsRNAs induce transcriptional activation in human cells" (Los ARNdc pequeños inducen la activación transcripcional en células humanas). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (46): 17337–42. Bibcode :2006PNAS..10317337L. doi : 10.1073/pnas.0607015103 . PMC 1859931 . PMID 17085592.
- ^ Noma K, Sugiyama T, Cam H, Verdel A, Zofall M, Jia S, et al. (noviembre de 2004). "RITS actúa en cis para promover el silenciamiento transcripcional y postranscripcional mediado por interferencia de ARN". Nature Genetics . 36 (11): 1174–80. doi : 10.1038/ng1452 . PMID 15475954.
- ^ Sugiyama T, Cam H, Verdel A, Moazed D, Grewal SI (enero de 2005). "La ARN polimerasa dependiente de ARN es un componente esencial de un bucle autoimpuesto que acopla el ensamblaje de la heterocromatina a la producción de ARNi". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (1): 152–7. Bibcode :2005PNAS..102..152S. doi : 10.1073/pnas.0407641102 . PMC 544066 . PMID 15615848.
- ^ Wang F, Koyama N, Nishida H, Haraguchi T, Reith W, Tsukamoto T (junio de 2006). "El ensamblaje y mantenimiento de la heterocromatina iniciado por repeticiones transgénicas son independientes de la vía de interferencia del ARN en células de mamíferos". Biología molecular y celular . 26 (11): 4028–40. doi :10.1128/MCB.02189-05. PMC 1489094 . PMID 16705157.
- ^ Bass BL (2002). "Edición de ARN por adenosina deaminasas que actúan sobre el ARN". Revisión anual de bioquímica . 71 : 817–46. doi :10.1146/annurev.biochem.71.110601.135501. PMC 1823043 . PMID 12045112.
- ^ Bass BL (abril de 2000). "ARN bicatenario como plantilla para el silenciamiento génico". Cell . 101 (3): 235–8. doi : 10.1016/S0092-8674(02)71133-1 . PMID 10847677.
- ^ Luciano DJ, Mirsky H, Vendetti NJ, Maas S (agosto de 2004). "Edición de ARN de un precursor de miARN". ARN . 10 (8): 1174–7. doi :10.1261/rna.7350304. PMC 1370607 . PMID 15272117.
- ^ ab Yang W, Chendrimada TP, Wang Q, Higuchi M, Seeburg PH, Shiekhattar R, et al. (enero de 2006). "Modulación del procesamiento y la expresión de microARN a través de la edición de ARN por desaminasas ADAR". Nature Structural & Molecular Biology . 13 (1): 13–21. doi :10.1038/nsmb1041. PMC 2950615 . PMID 16369484.
- ^ Yang W, Wang Q, Howell KL, Lee JT, Cho DS, Murray JM, et al. (febrero de 2005). "La ARN desaminasa ADAR1 limita la eficacia del ARN interferente corto en células de mamíferos". The Journal of Biological Chemistry . 280 (5): 3946–53. doi : 10.1074/jbc.M407876200 . PMC 2947832 . PMID 15556947.
- ^ Nishikura K (diciembre de 2006). "El editor se encuentra con el silenciador: diálogo cruzado entre la edición de ARN y la interferencia de ARN". Nature Reviews Molecular Cell Biology . 7 (12): 919–31. doi :10.1038/nrm2061. PMC 2953463 . PMID 17139332.
- ^ abc Saumet A, Lecellier CH (2006). "Silenciamiento del ARN antiviral: ¿nos parecemos a las plantas?". Retrovirology . 3 (1): 3. doi : 10.1186/1742-4690-3-3 . PMC 1363733 . PMID 16409629.
- ^ Jones L, Ratcliff F, Baulcombe DC (mayo de 2001). "El silenciamiento génico transcripcional dirigido por ARN en plantas puede heredarse independientemente del ARN desencadenante y requiere Met1 para su mantenimiento". Current Biology . 11 (10): 747–57. Bibcode :2001CBio...11..747J. doi : 10.1016/S0960-9822(01)00226-3 . PMID 11378384. S2CID 16789197.
- ^ Humphreys DT, Westman BJ, Martin DI, Preiss T (noviembre de 2005). "Los microARN controlan la iniciación de la traducción inhibiendo el factor de iniciación eucariota 4E/cap y la función de cola de poli(A)". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (47): 16961–6. Bibcode :2005PNAS..10216961H. doi : 10.1073/pnas.0506482102 . PMC 1287990 . PMID 16287976.
- ^ DaRocha WD, Otsu K, Teixeira SM, Donelson JE (febrero de 2004). "Pruebas de interferencia de ARN citoplasmático (RNAi) y construcción de un sistema promotor T7 inducible por tetraciclina en Trypanosoma cruzi". Parasitología molecular y bioquímica . 133 (2): 175–86. doi :10.1016/j.molbiopara.2003.10.005. PMID 14698430.
- ^ Robinson KA, Beverley SM (mayo de 2003). "Mejoras en la eficiencia de la transfección y pruebas de métodos de interferencia de ARN (ARNi) en el parásito protozoario Leishmania". Parasitología molecular y bioquímica . 128 (2): 217–28. doi :10.1016/S0166-6851(03)00079-3. PMID 12742588.
- ^ Aravind L, Watanabe H, Lipman DJ, Koonin EV (octubre de 2000). "Pérdida específica de linaje y divergencia de genes funcionalmente vinculados en eucariotas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (21): 11319–24. Bibcode :2000PNAS...9711319A. doi : 10.1073/pnas.200346997 . PMC 17198 . PMID 11016957.
- ^ Drinnenberg IA, Weinberg DE, Xie KT, Mower JP, Wolfe KH, Fink GR, et al. (octubre de 2009). "RNAi en levadura en ciernes". Science . 326 (5952): 544–550. Bibcode :2009Sci...326..544D. doi :10.1126/science.1176945. PMC 3786161 . PMID 19745116.
- ^ Nakayashiki H, Kadotani N, Mayama S (julio de 2006). «Evolución y diversificación de proteínas silenciadoras de ARN en hongos» (PDF) . Journal of Molecular Evolution . 63 (1): 127–35. Bibcode :2006JMolE..63..127N. doi :10.1007/s00239-005-0257-2. PMID 16786437. S2CID 22639035. Archivado desde el original (PDF) el 6 de agosto de 2020 . Consultado el 4 de diciembre de 2019 .
- ^ Morita T, Mochizuki Y, Aiba H (marzo de 2006). "La represión translacional es suficiente para el silenciamiento génico por ARN pequeños no codificantes bacterianos en ausencia de destrucción del ARNm". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (13): 4858–63. Bibcode :2006PNAS..103.4858M. doi : 10.1073/pnas.0509638103 . PMC 1458760 . PMID 16549791.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (marzo de 2006). "Un supuesto sistema inmunológico basado en la interferencia de ARN en procariotas: análisis computacional de la maquinaria enzimática prevista, analogías funcionales con la interferencia de ARN eucariota y mecanismos de acción hipotéticos". Biology Direct . 1 : 7. doi : 10.1186/1745-6150-1-7 . PMC 1462988 . PMID 16545108.
- ^ Stram Y, Kuzntzova L (junio de 2006). "Inhibición de virus por interferencia de ARN". Virus Genes . 32 (3): 299–306. doi :10.1007/s11262-005-6914-0. PMC 7088519 . PMID 16732482.
- ^ Blevins T, Rajeswaran R, Shivaprasad PV, Beknazariants D, Si-Ammour A, Park HS, et al. (2006). "Cuatro Dicers de plantas median la biogénesis de ARN pequeño viral y el silenciamiento inducido por virus de ADN". Nucleic Acids Research . 34 (21): 6233–46. doi :10.1093/nar/gkl886. PMC 1669714 . PMID 17090584.
- ^ Palauqui JC, Elmayan T, Pollien JM, Vaucheret H (agosto de 1997). "Silenciamiento sistémico adquirido: el silenciamiento postranscripcional específico del transgén se transmite por injerto de cepas silenciadas a vástagos no silenciados". The EMBO Journal . 16 (15): 4738–45. doi :10.1093/emboj/16.15.4738. PMC 1170100 . PMID 9303318.
- ^ Voinnet O (agosto de 2001). "Silenciamiento de ARN como sistema inmunológico de las plantas contra los virus". Tendencias en genética . 17 (8): 449–59. doi :10.1016/S0168-9525(01)02367-8. PMID 11485817.
- ^ Lucy AP, Guo HS, Li WX, Ding SW (abril de 2000). "Supresión del silenciamiento génico postranscripcional por una proteína viral vegetal localizada en el núcleo". The EMBO Journal . 19 (7): 1672–80. doi :10.1093/emboj/19.7.1672. PMC 310235 . PMID 10747034.
- ^ Mérai Z, Kerényi Z, Kertész S, Magna M, Lakatos L, Silhavy D (junio de 2006). "La unión del ARN bicatenario puede ser una estrategia general del ARN viral de las plantas para suprimir el silenciamiento del ARN". Revista de Virología . 80 (12): 5747–56. doi :10.1128/JVI.01963-05. PMC 1472586 . PMID 16731914.
- ^ Katiyar-Agarwal S, Morgan R, Dahlbeck D, Borsani O, Villegas A, Zhu JK, et al. (noviembre de 2006). "Un ARNi endógeno inducible por patógenos en la inmunidad de las plantas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (47): 18002–7. Bibcode :2006PNAS..10318002K. doi : 10.1073/pnas.0608258103 . PMC 1693862 . PMID 17071740.
- ^ Fritz JH, Girardin SE, Philpott DJ (junio de 2006). "Defensa inmunitaria innata a través de la interferencia del ARN". Science's STKE . 2006 (339): pe27. doi :10.1126/stke.3392006pe27. PMID 16772641. S2CID 33972766.
- ^ Zambon RA, Vakharia VN, Wu LP (mayo de 2006). "La RNAi es una respuesta inmunitaria antiviral contra un virus dsRNA en Drosophila melanogaster". Microbiología celular . 8 (5): 880–9. doi : 10.1111/j.1462-5822.2006.00688.x . PMID 16611236. S2CID 32439482.
- ^ Wang XH, Aliyari R, Li WX, Li HW, Kim K, Carthew R, et al. (abril de 2006). "La interferencia del ARN dirige la inmunidad innata contra los virus en la Drosophila adulta". Science . 312 (5772): 452–4. Bibcode :2006Sci...312..452W. doi :10.1126/science.1125694. PMC 1509097 . PMID 16556799.
- ^ Lu R, Maduro M, Li F, Li HW, Broitman-Maduro G, Li WX, et al. (agosto de 2005). "Replicación de virus animales y silenciamiento antiviral mediado por ARNi en Caenorhabditis elegans". Nature . 436 (7053): 1040–1043. Bibcode :2005Natur.436.1040L. doi :10.1038/nature03870. PMC 1388260 . PMID 16107851.
- ^ Wilkins C, Dishongh R, Moore SC, Whitt MA, Chow M, Machaca K (agosto de 2005). "La interferencia del ARN es un mecanismo de defensa antiviral en Caenorhabditis elegans". Nature . 436 (7053): 1044–7. Bibcode :2005Natur.436.1044W. doi :10.1038/nature03957. PMID 16107852. S2CID 4431035.
- ^ Berkhout B, Haasnoot J (mayo de 2006). "La interacción entre la infección viral y la maquinaria de interferencia del ARN celular". FEBS Letters . 580 (12): 2896–902. Bibcode :2006FEBSL.580.2896B. doi :10.1016/j.febslet.2006.02.070. PMC 7094296 . PMID 16563388.
- ^ Schütz S, Sarnow P (enero de 2006). "Interacción de los virus con la vía de interferencia del ARN en mamíferos". Virología . 344 (1): 151–7. doi : 10.1016/j.virol.2005.09.034 . PMID 16364746.
- ^ Cullen BR (junio de 2006). "¿Está implicada la interferencia del ARN en la inmunidad antiviral intrínseca en los mamíferos?". Nature Immunology . 7 (6): 563–7. doi :10.1038/ni1352. PMID 16715068. S2CID 23467688.
- ^ Maillard PV, Ciaudo C, Marchais A, Li Y, Jay F, Ding SW, et al. (octubre de 2013). "Interferencia de ARN antiviral en células de mamíferos". Science . 342 (6155): 235–8. Bibcode :2013Sci...342..235M. doi :10.1126/science.1241930. PMC 3853215 . PMID 24115438.
- ^ Li Y, Lu J, Han Y, Fan X, Ding SW (octubre de 2013). "La interferencia del ARN funciona como un mecanismo de inmunidad antiviral en mamíferos". Science . 342 (6155): 231–4. Bibcode :2013Sci...342..231L. doi :10.1126/science.1241911. PMC 3875315 . PMID 24115437.
- ^ Li HW, Ding SW (octubre de 2005). "Silenciamiento antiviral en animales". FEBS Letters . 579 (26): 5965–73. Bibcode :2005FEBSL.579.5965L. doi :10.1016/j.febslet.2005.08.034. PMC 1350842 . PMID 16154568.
- ^ Carrington JC, Ambros V (julio de 2003). "El papel de los microARN en el desarrollo de plantas y animales". Science . 301 (5631): 336–8. Bibcode :2003Sci...301..336C. doi :10.1126/science.1085242. PMID 12869753. S2CID 43395657.
- ^ Lee RC, Feinbaum RL, Ambros V (diciembre de 1993). "El gen heterocrónico lin-4 de C. elegans codifica ARN pequeños con complementariedad antisentido con lin-14". Cell . 75 (5): 843–54. doi : 10.1016/0092-8674(93)90529-Y . PMID 8252621.
- ^ Palatnik JF, Allen E, Wu X, Schommer C, Schwab R, Carrington JC y col. (Septiembre de 2003). "Control de la morfogénesis foliar por microARN". Naturaleza . 425 (6955): 257–63. Código Bib :2003Natur.425..257P. doi : 10.1038/naturaleza01958. PMID 12931144. S2CID 992057.
- ^ Zhang B, Pan X, Cobb GP, Anderson TA (enero de 2006). "MicroARN vegetal: una pequeña molécula reguladora con gran impacto". Biología del desarrollo . 289 (1): 3–16. doi : 10.1016/j.ydbio.2005.10.036 . PMID 16325172.
- ^ Jones-Rhoades MW, Bartel DP, Bartel B (2006). "MicroRNAS y sus funciones reguladoras en plantas". Revisión anual de biología vegetal . 57 : 19–53. doi :10.1146/annurev.arplant.57.032905.105218. PMID 16669754. S2CID 13010154.
- ^ Zhang B, Pan X, Cobb GP, Anderson TA (febrero de 2007). "microARN como oncogenes y supresores tumorales". Biología del desarrollo . 302 (1): 1–12. doi : 10.1016/j.ydbio.2006.08.028 . PMID 16989803.
- ^ Cerutti H, Casas-Mollano JA (agosto de 2006). "Sobre el origen y las funciones del silenciamiento mediado por ARN: desde los protistas hasta el hombre". Current Genetics . 50 (2): 81–99. doi :10.1007/s00294-006-0078-x. PMC 2583075 . PMID 16691418.
- ^ Anantharaman V, Koonin EV, Aravind L (abril de 2002). "Genómica comparativa y evolución de las proteínas implicadas en el metabolismo del ARN". Nucleic Acids Research . 30 (7): 1427–64. doi :10.1093/nar/30.7.1427. PMC 101826 . PMID 11917006.
- ^ Voorhoeve PM, Agami R (enero de 2003). "Knockdown se levanta". Tendencias en biotecnología . 21 (1): 2–4. doi :10.1016/S0167-7799(02)00002-1. PMID 12480342.
- ^ Munkácsy G, Sztupinszki Z, Herman P, Bán B, Pénzváltó Z, Szarvas N, et al. (septiembre de 2016). "La validación de la eficiencia del silenciamiento de ARNi utilizando datos de matrices genéticas muestra una tasa de falla del 18,5% en 429 experimentos independientes". Terapia Molecular: Ácidos Nucleicos . 5 (9): e366. doi :10.1038/mtna.2016.66. PMC 5056990 . PMID 27673562.
- ^ Naito Y, Yamada T, Matsumiya T, Ui-Tei K, Saigo K, Morishita S (julio de 2005). "dsCheck: software de búsqueda de ARN fuera del objetivo altamente sensible para la interferencia de ARN mediada por ARN de doble cadena". Nucleic Acids Research . 33 (edición del servidor web): W589–91. doi :10.1093/nar/gki419. PMC 1160180 . PMID 15980542.
- ^ Henschel A, Buchholz F, Habermann B (julio de 2004). "DEQOR: una herramienta basada en la web para el diseño y control de calidad de ARNi". Nucleic Acids Research . 32 (número del servidor web): W113–20. doi :10.1093/nar/gkh408. PMC 441546 . PMID 15215362.
- ^ Naito Y, Yamada T, Ui-Tei K, Morishita S, Saigo K (julio de 2004). "siDirect: software de diseño de ARNip altamente eficaz y específico para la interferencia de ARN de mamíferos". Investigación de ácidos nucleicos . 32 (problema del servidor web): W124–9. doi :10.1093/nar/gkh442. PMC 441580 . PMID 15215364.
- ^ Naito Y, Ui-Tei K, Nishikawa T, Takebe Y, Saigo K (julio de 2006). "siVirus: software de diseño de ARNsi antiviral basado en la web para secuencias virales altamente divergentes". Nucleic Acids Research . 34 (edición del servidor web): W448–50. doi :10.1093/nar/gkl214. PMC 1538817 . PMID 16845046.
- ^ Reynolds A, Anderson EM, Vermeulen A, Fedorov Y, Robinson K, Leake D, et al. (junio de 2006). "La inducción de la respuesta del interferón por ARNi depende del tipo de célula y de la longitud del dúplex". ARN . 12 (6): 988–93. doi :10.1261/rna.2340906. PMC 1464853 . PMID 16611941.
- ^ Stein P, Zeng F, Pan H, Schultz RM (octubre de 2005). "Ausencia de efectos no específicos de la interferencia del ARN desencadenada por el ARN bicatenario largo en ovocitos de ratón". Biología del desarrollo . 286 (2): 464–71. doi : 10.1016/j.ydbio.2005.08.015 . PMID 16154556.
- ^ Brummelkamp TR, Bernards R , Agami R (abril de 2002). "Un sistema para la expresión estable de ARN interferentes cortos en células de mamíferos". Science . 296 (5567): 550–3. Bibcode :2002Sci...296..550B. doi :10.1126/science.1068999. hdl : 1874/15573 . PMID 11910072. S2CID 18460980.
- ^ Tiscornia G, Tergaonkar V, Galimi F, Verma IM (mayo de 2004). "Interferencia de ARN inducible por recombinasa CRE mediada por vectores lentivirales". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 (19): 7347–51. Bibcode :2004PNAS..101.7347T. doi : 10.1073/pnas.0402107101 . PMC 409921 . PMID 15123829.
- ^ Ventura A, Meissner A, Dillon CP, McManus M, Sharp PA, Van Parijs L, et al. (julio de 2004). "Interferencia condicional de ARN regulada por Cre-lox a partir de transgenes". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 (28): 10380–5. Bibcode :2004PNAS..10110380V. doi : 10.1073/pnas.0403954101 . PMC 478580 . PMID 15240889.
- ^ ab Adams D, González-Duarte A, O'Riordan WD, Yang CC, Ueda M, Kristen AV, et al. (5 de julio de 2018). "Patisiran, un terapéutico de ARNi, para la amiloidosis hereditaria por transtiretina". Revista de Medicina de Nueva Inglaterra . 379 (1): 11-21. doi : 10.1056/NEJMoa1716153 . hdl : 2445/138257 . PMID 29972753. S2CID 205102839.
- ^ Balwani M, Sardh E, Ventura P, Peiró PA, Rees DC, Stölzel U, et al. (11 de junio de 2020). "Ensayo de fase 3 de givosiran terapéutico con ARNi para la porfiria intermitente aguda". New England Journal of Medicine . 382 (24): 2289–2301. doi :10.1056/NEJMoa1913147. ISSN 0028-4793. PMID 32521132.
- ^ Garrelfs SF, Frishberg Y, Hulton SA, Koren MJ, O'Riordan WD, Cochat P, et al. (1 de abril de 2021). "Lumasiran, un terapéutico de ARNi para la hiperoxaluria primaria tipo 1". La Revista de Medicina de Nueva Inglaterra . 384 (13): 1216-1226. doi :10.1056/NEJMoa2021712. ISSN 1533-4406. PMID 33789010.
- ^ Adams D, Tournev IL, Taylor MS, Coelho T, Planté-Bordeneuve V, Berk JL, et al. (marzo de 2023). "Eficacia y seguridad de vutrisiran para pacientes con amiloidosis hereditaria mediada por transtiretina con polineuropatía: un ensayo clínico aleatorizado". Amiloide . 30 (1): 18–26. doi :10.1080/13506129.2022.2091985. hdl : 1959.4/unsworks_80922 . ISSN 1744-2818. PMID 35875890.
- ^ "ETIQUETA: ONPATTRO- inyección de patisiran, complejo lipídico". Daily Med . 10 de mayo de 2021.
- ^ "Paquete de aprobación de medicamentos: inyección de GIVLAARI (givosiran)". FDA .
- ^ Gonzalez-Aseguinolaza G (11 de junio de 2020). "Givosiran: interferencia de ARN en ejecución para combatir los ataques de porfiria". New England Journal of Medicine . 382 (28–4793): 2366–2367. doi :10.1056/NEJMe2010986. PMID 32521139. S2CID 219592948.
- ^ "Givlaari". Agencia Europea de Medicamentos .
- ^ Puy H, Gouya L, Deybach JC (2010). "Porfirias". Lancet . 375 (9718): 924–937. doi :10.1016/S0140-6736(09)61925-5. PMID 20226990. S2CID 208791867.
- ^ Simon A, Pompilus F, Querbes W, Wei A, Strzok S, Penz C, et al. (2018). "Perspectiva del paciente sobre la porfiria intermitente aguda con ataques frecuentes: una enfermedad con manifestaciones intermitentes y crónicas". Paciente . 11 (5): 527–537. doi :10.1007/s40271-018-0319-3. PMC 6132435 . PMID 29915990.
- ^ Pischik E, Kauppinen R (2015). "Una actualización del tratamiento clínico de la porfiria intermitente aguda". Aplicación de la genética clínica . 8 : 201–214. doi : 10.2147/TACG.S48605 . PMC 4562648 . PMID 26366103.
- ^ Bissell DM, Lai JC, Meister RK, Blanc PD (2015). "El papel del ácido delta-aminolevulínico en los síntomas de la porfiria aguda". The American Journal of Medicine . 128 (3): 313–317. doi :10.1016/j.amjmed.2014.10.026. PMC 4339446 . PMID 25446301.
- ^ "ETIQUETA: GIVLAARI- solución inyectable de givosiran sódico". Daily Med . 5 de abril de 2022.
- ^ "Paquete de aprobación de medicamentos: OXLUMO". FDA .
- ^ "Oxlumo". Agencia Europea de Medicamentos .
- ^ "ETIQUETA: OXLUMO- inyección de lumasiran, solución". Medicina diaria . 2 de diciembre de 2021.
- ^ Garrelfs SF, Frishberg Y, Hulton SA, Koren MJ, O'Riordan WD, Cochat P, et al. (1 de abril de 2021). "Lumasiran, un terapéutico de ARNi para la hiperoxaluria primaria tipo 1". Revista de Medicina de Nueva Inglaterra . 384 (13): 1216-1226. doi : 10.1056/NEJMoa2021712 . PMID 33789010. S2CID 232482623.
- ^ Investigación Cf (27 de noviembre de 2023). "Instantáneas de ensayos de fármacos: AMVUTTRA". FDA .
- ^ "Amvuttra | Agencia Europea de Medicamentos". www.ema.europa.eu . Consultado el 11 de enero de 2024 .
- ^ Guerriaud M, Kohli E (2022). "Medicamentos basados en ARN y regulación: hacia una evolución necesaria de las definiciones emitidas desde la legislación de la Unión Europea". Frontiers in Medicine . 9 . doi : 10.3389/fmed.2022.1012497 . ISSN 2296-858X. PMC 9618588 . PMID 36325384.
- ^ ab Kanasty R, Dorkin JR, Vegas A, Anderson D (noviembre de 2013). "Materiales de administración para terapias con ARNip". Nature Materials . 12 (11): 967–77. Bibcode :2013NatMa..12..967K. doi :10.1038/nmat3765. PMID 24150415.
- ^ Wittrup A, Lieberman J (septiembre de 2015). "Derribando la enfermedad: un informe de progreso sobre terapias con ARNi". Nature Reviews Genetics . 16 (9): 543–52. doi :10.1038/nrg3978. PMC 4756474 . PMID 26281785.
- ^ De-Souza EA, Camara H, Salgueiro WG, Moro RP, Knittel TL, Tonon G, et al. (mayo de 2019). "La interferencia de ARN puede dar lugar a fenotipos inesperados en Caenorhabditis elegans". Investigación de ácidos nucleicos . 47 (8): 3957–3969. doi : 10.1093/nar/gkz154. PMC 6486631 . PMID 30838421.
- ^ Li W, Szoka FC (24 de marzo de 2007). "Nanopartículas basadas en lípidos para la administración de ácidos nucleicos". Pharm Res . 24 (3): 438–449. doi :10.1007/s11095-006-9180-5. PMID 17252188. S2CID 9995555.
- ^ Leung AK, Tam YY, Cullis PR (2014). "Nanopartículas lipídicas para la administración de ARN interferente de corta distancia". Vectores no virales para terapia génica: transferencia génica basada en lípidos y polímeros . Avances en genética. Vol. 88. págs. 71–110. doi :10.1016/B978-0-12-800148-6.00004-3. ISBN 978-0-12-800148-6. PMC 7149983 . PMID 25409604.
- ^ John LaMattina (15 de abril de 2014). "La apuesta de las grandes farmacéuticas por el ARNi demuestra que las nuevas tecnologías no garantizan el éxito de la I+D". Forbes .
- ^ Eric Bender (1 de septiembre de 2014). "La segunda venida del ARNi". The Scientist .
- ^ Baden LR (2021). "Eficacia y seguridad de la vacuna ARNm-1273 contra el SARS-CoV-2". New England Journal of Medicine . 384 (5): 403–416. doi :10.1056/NEJMoa2035389. PMC 7787219 . PMID 33378609.
- ^ Fitzgerald K, White S, Borodovsky A, Bettencourt BR, Strahs A, Clausen V (enero de 2017). "Un inhibidor terapéutico de ARNi altamente duradero de PCSK9". New England Journal of Medicine . 376 (1): 41–51. doi :10.1056/NEJMoa1609243. PMC 5778873 . PMID 27959715.
- ^ Kruspe S, Giangrande P (2017). "Quimeras de aptámeros y ARNi: descubrimiento, progreso y perspectivas futuras". Biomedicines . 5 (4): 45. doi : 10.3390/biomedicines5030045 . PMC 5618303 . PMID 28792479.
- ^ Balwani M, Sardh E, Ventura P, Peiró PA, Rees DC, Stölzel U, et al. (11 de junio de 2020). "Ensayo de fase 3 de givosiran terapéutico con ARNi para la porfiria intermitente aguda". New England Journal of Medicine . 382 (24): 2289–2301. doi : 10.1056/NEJMoa1913147 . PMID 32521132. S2CID 219586624.
- ^ Andersson M, Melander M, Pojmark P, Larsson H, Bülow L, Hofvander P (2006). "Supresión génica dirigida mediante interferencia de ARN: un método eficiente para la producción de líneas de patata con alto contenido de amilosa". Journal of Biotechnology . 123 (2): 137–148. doi :10.1016/j.jbiotec.2005.11.001. PMID 16466822.
- ^ Frizzi A, Huang S (2010). "Aprovechamiento de las vías de silenciamiento del ARN para la biotecnología vegetal". Revista de biotecnología vegetal . 8 (6): 655–677. doi : 10.1111/j.1467-7652.2010.00505.x . PMID 20331529.
- ^ Parrott W, Chassy B, Ligon J, Meyer L, Patrick J, Zhou J, et al. (2010). "Aplicación de los principios de evaluación de la seguridad de alimentos y piensos para evaluar los enfoques transgénicos de modulación genética en cultivos". Asociación Británica de Investigación Biológica Industrial . 48 (7): 1773–1790. doi :10.1016/j.fct.2010.04.017. PMID 20399824.
- ^ Berkhout B (abril de 2004). "Interferencia de ARN como método antiviral: dirigiéndose al VIH-1". Current Opinion in Molecular Therapeutics . 6 (2): 141–5. PMID 15195925.
- ^ Jiang M, Milner J (septiembre de 2002). "Silenciamiento selectivo de la expresión génica viral en células de carcinoma cervical humano positivas para VPH tratadas con ARNi, un cebador de interferencia de ARN". Oncogene . 21 (39): 6041–8. doi : 10.1038/sj.onc.1205878 . PMID 12203116.
- ^ Kusov Y, Kanda T, Palmenberg A, Sgro JY, Gauss-Müller V (junio de 2006). "Silenciamiento de la infección por el virus de la hepatitis A mediante pequeñas ARN interferentes". Journal of Virology . 80 (11): 5599–610. doi :10.1128/jvi.01773-05. PMC 1472172 . PMID 16699041.
- ^ Jia F, Zhang YZ, Liu CM (octubre de 2006). "Un sistema basado en retrovirus para silenciar de forma estable los genes del virus de la hepatitis B mediante interferencia de ARN". Biotechnology Letters . 28 (20): 1679–85. doi : 10.1007/s10529-006-9138-z . PMID 16900331. S2CID 34511611.
- ^ Li YC, Kong LH, Cheng BZ, Li KS (diciembre de 2005). "Construcción de vectores de expresión de ARNi del virus de la influenza y sus efectos inhibidores en la multiplicación del virus de la influenza". Enfermedades aviares . 49 (4): 562–73. doi :10.1637/7365-041205R2.1. PMID 16405000. S2CID 86214047.
- ^ Khanna M, Saxena L, Rajput R, Kumar B, Prasad R (2015). "Silenciamiento genético: un enfoque terapéutico para combatir las infecciones por el virus de la gripe". Future Microbiology . 10 (1): 131–40. doi :10.2217/fmb.14.94. PMID 25598342.
- ^ Rajput R, Khanna M, Kumar P, Kumar B, Sharma S, Gupta N, et al. (diciembre de 2012). "El ARN interferente pequeño dirigido a la transcripción del gen no estructural 1 inhibe la replicación del virus de la influenza A en ratones experimentales". Nucleic Acid Therapeutics . 22 (6): 414–22. doi :10.1089/nat.2012.0359. PMID 23062009.
- ^ abcd Asha K, Kumar P, Sanicas M, Meseko CA, Khanna M, Kumar B (diciembre de 2018). "Avances en terapias basadas en ácidos nucleicos contra infecciones virales respiratorias". Revista de medicina clínica . 8 (1): 6. doi : 10.3390/jcm8010006 . PMC 6351902 . PMID 30577479.
- ^ Hu L, Wang Z, Hu C, Liu X, Yao L, Li W, et al. (2005). "Inhibición de la multiplicación del virus del sarampión en cultivos celulares mediante interferencia de ARN". Acta Virologica . 49 (4): 227–34. PMID 16402679.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (2014). "VIRmiRNA: un recurso completo para microARN virales validados experimentalmente y sus objetivos". Base de datos . 2014 . doi :10.1093/database/bau103. PMC 4224276 . PMID 25380780.
- ^ Crowe S (2003). "La supresión de la expresión del receptor de quimiocinas mediante la interferencia del ARN permite la inhibición de la replicación del VIH-1, por Martínez et al". AIDS . 17 (Suppl 4): S103–5. doi : 10.1097/00002030-200317004-00014 . PMID 15080188.
- ^ Fuchs U, Damm-Welk C, Borkhardt A (agosto de 2004). "Silenciamiento de genes relacionados con enfermedades mediante pequeñas ARN interferentes". Medicina molecular actual . 4 (5): 507–17. doi :10.2174/1566524043360492. PMID 15267222.
- ^ Cioca DP, Aoki Y, Kiyosawa K (febrero de 2003). "La interferencia del ARN es una vía funcional con potencial terapéutico en líneas celulares de leucemia mieloide humana". Cancer Gene Therapy . 10 (2): 125–33. doi : 10.1038/sj.cgt.7700544 . PMID 12536201.
- ^ Lapteva N, Yang AG, Sanders DE, Strube RW, Chen SY (enero de 2005). "La supresión de CXCR4 por un ARN interferente pequeño anula el crecimiento del tumor de mama in vivo". Terapia génica del cáncer . 12 (1): 84–9. doi : 10.1038/sj.cgt.7700770 . PMID 15472715.
- ^ abcdefghi Xu CF, Wang J (1 de febrero de 2015). "Sistemas de administración para el desarrollo de fármacos de ARNip en la terapia del cáncer". Revista asiática de ciencias farmacéuticas . 10 (1): 1–12. doi : 10.1016/j.ajps.2014.08.011 .
- ^ Singer O, Marr RA, Rockenstein E, Crews L, Coufal NG, Gage FH, et al. (octubre de 2005). "La focalización de BACE1 con ARNi mejora la neuropatología de la enfermedad de Alzheimer en un modelo transgénico". Nature Neuroscience . 8 (10): 1343–9. doi :10.1038/nn1531. PMID 16136043. S2CID 6978101.
- ^ Rodríguez-Lebrón E, Gouvion CM, Moore SA, Davidson BL, Paulson HL (septiembre de 2009). "El ARNi específico de alelos mitiga la progresión fenotípica en un modelo transgénico de la enfermedad de Alzheimer". Molecular Therapy . 17 (9): 1563–73. doi :10.1038/mt.2009.123. PMC 2835271 . PMID 19532137.
- ^ Piedrahita D, Hernández I, López-Tobón A, Fedorov D, Obara B, Manjunath BS, et al. (octubre de 2010). "El silenciamiento de CDK5 reduce los ovillos neurofibrilares en ratones transgénicos con Alzheimer". La Revista de Neurociencia . 30 (42): 13966–76. doi :10.1523/jneurosci.3637-10.2010. PMC 3003593 . PMID 20962218.
- ^ Raoul C, Barker SD, Aebischer P (marzo de 2006). "Modelado y corrección de enfermedades neurodegenerativas por interferencia de ARN basado en virus". Terapia génica . 13 (6): 487–95. doi : 10.1038/sj.gt.3302690 . PMID 16319945.
- ^ Harper SQ, Staber PD, He X, Eliason SL, Martins IH, Mao Q, et al. (abril de 2005). "La interferencia de ARN mejora las anormalidades motoras y neuropatológicas en un modelo de ratón con enfermedad de Huntington". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (16): 5820–5. Bibcode :2005PNAS..102.5820H. doi : 10.1073/pnas.0501507102 . PMC 556303 . PMID 15811941.
- ^ Boudreau RL, Rodríguez-Lebrón E, Davidson BL (abril de 2011). "Medicina de ARNi para el cerebro: avances y desafíos". Human Molecular Genetics . 20 (R1): R21–7. doi :10.1093/hmg/ddr137. PMC 3095054 . PMID 21459775.
- ^ abcdefghijklm Whitehead KA, Dahlman JE, Langer RS, Anderson DG (2011). "¿Silenciamiento o estimulación? Administración de ARNi y el sistema inmunológico". Revisión anual de ingeniería química y biomolecular . 2 : 77–96. doi :10.1146/annurev-chembioeng-061010-114133. PMID 22432611. S2CID 28803811.
- ^ Sunilkumar G, Campbell LM, Puckhaber L, Stipanovic RD, Rathore KS (noviembre de 2006). "Ingeniería de semillas de algodón para su uso en nutrición humana mediante la reducción específica de tejidos del gosipol tóxico". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (48): 18054–9. doi : 10.1073/pnas.0605389103 . PMC 1838705 . PMID 17110445.
- ^ Le LQ, Lorenz Y, Scheurer S, Fötisch K, Enrique E, Bartra J, et al. (marzo de 2006). "Diseño de frutos de tomate con alergenicidad reducida mediante inhibición de la expresión de ns-LTP (Lyc e 3) mediada por dsRNAi". Revista de biotecnología vegetal . 4 (2): 231–42. doi : 10.1111/j.1467-7652.2005.00175.x . PMID 17177799.
- ^ Niggeweg R, Michael AJ, Martin C (junio de 2004). "Ingeniería de plantas con mayores niveles del antioxidante ácido clorogénico". Nature Biotechnology . 22 (6): 746–54. doi :10.1038/nbt966. PMID 15107863. S2CID 21588259.
- ^ Gilbert MK, Majumdar R, Rajasekaran K, Chen ZY, Wei Q, Sickler CM, et al. (junio de 2018). "El silenciamiento basado en la interferencia de ARN del gen de la alfa-amilasa (amy1) en Aspergillus flavus disminuye el crecimiento fúngico y la producción de aflatoxina en granos de maíz". Planta . 247 (6): 1465–1473. Bibcode :2018Plant.247.1465G. doi :10.1007/s00425-018-2875-0. PMID 29541880. S2CID 3918937.
- ^ Katoch R, Thakur N (marzo de 2013). "Interferencia de ARN: una técnica prometedora para la mejora de cultivos tradicionales". Revista Internacional de Ciencias de la Alimentación y Nutrición . 64 (2): 248–59. doi :10.3109/09637486.2012.713918. PMID 22861122. S2CID 45212581.
- ^ Katoch R, Thakur N (marzo de 2013). "Avances en la tecnología de interferencia de ARN y su impacto en la mejora nutricional, el control de enfermedades e insectos en plantas". Applied Biochemistry and Biotechnology . 169 (5): 1579–605. doi :10.1007/s12010-012-0046-5. PMID 23322250. S2CID 23733295.
- ^ ab Downie RC, Lin M, Corsi B, Ficke A, Lillemo M, Oliver RP, et al. (27 de julio de 2021). "Septoria Nodorum Blotch of Wheat: Disease Management and Resistance Breeding in the Face of Changing Disease Dynamics and a Changing Environment" (Mancha de trigo causada por Septoria nodorum: manejo de enfermedades y mejoramiento de la resistencia frente a la dinámica cambiante de las enfermedades y un entorno cambiante). Fitopatología . 111 (6). Sociedad Fitopatológica Estadounidense : PHYTO–07–20–028. doi :10.1094/phyto-07-20-0280-rvw. hdl : 20.500.11937/83208 . ISSN 0031-949X. PMID 33245254. S2CID 227181536.
- ^ Ivashuta S, Zhang Y, Wiggins BE, Ramaseshadri P, Segers GC, Johnson S, et al. (mayo de 2015). "ARNi ambiental en insectos herbívoros". ARN . 21 (5): 840–50. doi :10.1261/rna.048116.114. PMC 4408792 . PMID 25802407.
- ^ Miller SC, Miyata K, Brown SJ, Tomoyasu Y (2012). "Disección de la interferencia sistémica del ARN en el escarabajo rojo de la harina Tribolium castaneum: parámetros que afectan la eficiencia del ARNi". PLOS ONE . 7 (10): e47431. Bibcode :2012PLoSO...747431M. doi : 10.1371/journal.pone.0047431 . PMC 3484993 . PMID 23133513.
- ^ Petrick JS, Friedrich GE, Carleton SM, Kessenich CR, Silvanovich A, Zhang Y, et al. (noviembre de 2016). "ARN DvSnf7 activo contra el gusano de la raíz del maíz: evaluación toxicológica de dosis repetidas por vía oral en apoyo de la seguridad en humanos y mamíferos". Toxicología y farmacología regulatorias . 81 : 57–68. doi : 10.1016/j.yrtph.2016.07.009 . PMID 27436086.
- ^ Terenius O, Papanicolaou A, Garbutt JS, Eleftherianos I, Huvenne H, Kanginakudru S, et al. (febrero de 2011). "Interferencia de ARN en lepidópteros: una descripción general de estudios exitosos y no exitosos e implicaciones para el diseño experimental". Journal of Insect Physiology . 57 (2): 231–45. Bibcode :2011JInsP..57..231T. doi :10.1016/j.jinsphys.2010.11.006. hdl : 1854/LU-1101411 . PMID 21078327.
- ^ Mongelli V, Saleh MC (29 de septiembre de 2016). "No hay que silenciar a los insectos: vías de ARN pequeño y respuestas antivirales en insectos" (PDF) . Revisión anual de virología . 3 (1). Revisiones anuales : 573–589. doi :10.1146/annurev-virology-110615-042447. ISSN 2327-056X. PMID 27741406. S2CID 38499958.
- ^ abc Zhu KY, Palli SR (7 de enero de 2020). "Mecanismos, aplicaciones y desafíos de la interferencia del ARN de insectos". Revisión anual de entomología . 65 (1). Revisiones anuales : 293–311. doi :10.1146/annurev-ento-011019-025224. ISSN 0066-4170. PMC 9939233 . PMID 31610134. S2CID 204702574.
- ^ Huesken D, Lange J, Mickanin C, Weiler J, Asselbergs F, Warner J, et al. (agosto de 2005). "Diseño de una biblioteca de ARNi de todo el genoma utilizando una red neuronal artificial". Nature Biotechnology . 23 (8): 995–1001. doi :10.1038/nbt1118. PMID 16025102. S2CID 11030533.
- ^ Ge G, Wong GW, Luo B (octubre de 2005). "Predicción de la eficiencia de eliminación de ARNi mediante modelos de redes neuronales artificiales". Comunicaciones de investigación bioquímica y biofísica . 336 (2): 723–8. doi :10.1016/j.bbrc.2005.08.147. PMID 16153609.
- ^ Janitz M, Vanhecke D, Lehrach H (2006). "Interferencia de ARN de alto rendimiento en genómica funcional". ARN hacia la medicina . Manual de farmacología experimental. Vol. 173. págs. 97-104. doi :10.1007/3-540-27262-3_5. ISBN 978-3-540-27261-8. Número de identificación personal 16594612.
- ^ Vanhecke D, Janitz M (febrero de 2005). "Genómica funcional mediante interferencia de ARN de alto rendimiento". Drug Discovery Today . 10 (3): 205–12. doi :10.1016/S1359-6446(04)03352-5. hdl : 11858/00-001M-0000-0010-86E7-8 . PMID 15708535. S2CID 9934291.
- ^ Matson RS (2005). Aplicación de la tecnología de microarrays genómicos y proteómicos en el descubrimiento de fármacos . CRC Press. p. 6. ISBN 978-0-8493-1469-8.
- ^ Zhang XHD (2011). Cribado óptimo de alto rendimiento: diseño experimental práctico y análisis de datos para la investigación de ARNi a escala genómica. Cambridge University Press. págs. ix–xiii. ISBN 978-0-521-73444-8.
- ^ Matzke MA, Matzke AJ (2004). "Plantando las semillas de un nuevo paradigma". PLOS Biol . 2 (5): e133. doi : 10.1371/journal.pbio.0020133 . PMC 406394 . PMID 15138502.
- ^ Ecker JR, Davis RW (agosto de 1986). "Inhibición de la expresión génica en células vegetales mediante la expresión de ARN antisentido". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 83 (15): 5372–6. Bibcode :1986PNAS...83.5372E. doi : 10.1073/pnas.83.15.5372 . PMC 386288 . PMID 16593734.
- ^ Napoli C, Lemieux C, Jorgensen R (abril de 1990). "La introducción de un gen de chalcona sintasa quimérico en petunia da como resultado una cosupresión reversible de genes homólogos en trans". The Plant Cell . 2 (4): 279–289. doi :10.1105/tpc.2.4.279. PMC 159885 . PMID 12354959.
- ^ Romano N, Macino G (noviembre de 1992). "Quelling: inactivación transitoria de la expresión génica en Neurospora crassa mediante transformación con secuencias homólogas". Microbiología molecular . 6 (22): 3343–53. doi :10.1111/j.1365-2958.1992.tb02202.x. PMID 1484489. S2CID 31234985.
- ^ Van Blokland R, Van der Geest N, Mol JN, Kooter JM (1994). "La supresión mediada por transgenes de la expresión de la chalcona sintasa en Petunia hybrida resulta de un aumento en la renovación del ARN". Plant J . 6 (6): 861–77. doi : 10.1046/j.1365-313X.1994.6060861.x .
- ^ Mol JN, van der Krol AR (1991). Ácidos nucleicos y proteínas antisentido: fundamentos y aplicaciones . M. Dekker. pp. 4, 136. ISBN 978-0-8247-8516-1.
- ^ Covey S, Al-Kaff N, Lángara A, Turner D (1997). "Las plantas combaten la infección mediante el silenciamiento de genes". Nature . 385 (6619): 781–2. Código Bibliográfico :1997Natur.385..781C. doi :10.1038/385781a0. S2CID 43229760.
- ^ Kumagai MH, Donson J, della-Cioppa G, Harvey D, Hanley K, Grill LK (febrero de 1995). "Inhibición citoplasmática de la biosíntesis de carotenoides con ARN derivado de virus". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 92 (5): 1679–83. Bibcode :1995PNAS...92.1679K. doi : 10.1073/pnas.92.5.1679 . PMC 42583 . PMID 7878039.
- ^ Ratcliff F, Harrison BD, Baulcombe DC (junio de 1997). "Una similitud entre la defensa viral y el silenciamiento de genes en plantas". Science . 276 (5318): 1558–60. doi :10.1126/science.276.5318.1558. PMID 18610513.
- ^ Guo S, Kemphues KJ (mayo de 1995). "par-1, un gen necesario para establecer la polaridad en los embriones de C. elegans, codifica una supuesta quinasa Ser/Thr que se distribuye asimétricamente". Cell . 81 (4): 611–20. doi : 10.1016/0092-8674(95)90082-9 . PMID 7758115.
- ^ Pal-Bhadra M, Bhadra U, Birchler JA (agosto de 1997). "Cosupresión en Drosophila: el silenciamiento génico de la alcohol deshidrogenasa por transgenes white-Adh es dependiente de Polycomb". Cell . 90 (3): 479–90. doi : 10.1016/S0092-8674(00)80508-5 . PMID 9267028.
- ^ ab Sen GL, Blau HM (julio de 2006). "Una breve historia del ARNi: el silencio de los genes". FASEB Journal . 20 (9): 1293–9. doi : 10.1096/fj.06-6014rev . PMID 16816104. S2CID 12917676.
- ^ Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC (febrero de 1998). "Interferencia genética potente y específica por ARN bicatenario en Caenorhabditis elegans". Nature . 391 (6669): 806–11. Bibcode :1998Natur.391..806F. doi :10.1038/35888. PMID 9486653. S2CID 4355692.
- ^ Daneholt B (2 de octubre de 2006). «El Premio Nobel de Fisiología o Medicina 2006». nobelprize.org . Consultado el 30 de octubre de 2017 .
- ^ Elbashir S, Harborth J, Lendeckel W, et al. (2001). "Los dúplex de ARN de 21 nucleótidos median la interferencia del ARN en células de mamíferos cultivadas". Nature . 411 (6836): 494–498. Bibcode :2001Natur.411..494E. doi :10.1038/35078107. PMID 11373684. S2CID 710341.
- ^ McCaffrey AP, Meuse L, Pham TT, Conklin DS, Hannon GJ , Kay MA (julio de 2002). "Interferencia de ARN en ratones adultos". Nature . 418 (6893): 38–9. Bibcode :2002Natur.418...38M. doi :10.1038/418038a. PMID 12097900. S2CID 4361399.

- ^ Devi GR (septiembre de 2006). "Enfoques basados en ARNi en la terapia del cáncer". Terapia génica del cáncer . 13 (9): 819–29. doi : 10.1038/sj.cgt.7700931 . PMID: 16424918.
- ^ Wall NR, Shi Y (octubre de 2003). "ARN pequeño: ¿se puede aprovechar la interferencia del ARN con fines terapéuticos?". Lancet . 362 (9393): 1401–3. doi :10.1016/s0140-6736(03)14637-5. PMID 14585643. S2CID 25034627.
- ^ Sah D (2006). "Potencial terapéutico de la interferencia de ARN para trastornos neurológicos". Life Sci . 79 (19): 1773–80. doi :10.1016/j.lfs.2006.06.011. PMID 16815477.
- ^ Davis ME , Zuckerman JE, Choi CH, Seligson D, Tolcher A, Alabi CA , et al. (abril de 2010). "Evidencia de RNAi en humanos a partir de siRNA administrado sistémicamente a través de nanopartículas dirigidas". Nature . 464 (7291): 1067–70. Bibcode :2010Natur.464.1067D. doi :10.1038/nature08956. PMC 2855406. PMID 20305636 .
- ^ "Novartis recibe la aprobación de la UE para Leqvio(inclisiran), un siRNA de primera clase para reducir el colesterol con dos dosis al año". Novartis . 11 de diciembre de 2020.
Enlaces externos
- Descripción general del proceso de ARNi, de The Naked Scientists de la Universidad de Cambridge
- Animación del proceso de RNAi, de Nature
- NOVA scienceNOW explica el ARNi: un video de 15 minutos de la transmisión de Nova que se emitió en PBS el 26 de julio de 2005
- Silenciamiento de genomas Archivado el 10 de agosto de 2019 en Wayback Machine Experimentos de interferencia de ARN (ARNi) y bioinformática en C. elegans para la educación. Del Centro de aprendizaje de ADN Dolan del Laboratorio Cold Spring Harbor.
- Cribado de ARNi en C. elegans en formato líquido de 96 pocillos y su aplicación a la identificación sistemática de interacciones genéticas (un protocolo)
- Dos "personas gusano" estadounidenses ganan el Nobel por su trabajo sobre el ARN, según el New York Times
- Enfoque web en Terapia Molecular: "El desarrollo del ARNi como estrategia terapéutica", una colección de artículos gratuitos sobre el ARNi como estrategia terapéutica.
- GenomeRNAi: una base de datos de fenotipos de experimentos de detección de interferencia de ARN en Drosophila melanogaster y Homo sapiens
- Herramientas de interferencia de ARN Archivado el 19 de junio de 2018 en Wayback Machine Herramientas de interferencia de ARN prediseñadas y personalizadas





