Expresión genética
| Parte de una serie sobre |
| Bioquímica |
|---|
 |
La expresión génica es el proceso por el cual la información de un gen se utiliza en la síntesis de un producto génico funcional que le permite producir productos finales, proteínas o ARN no codificante , y en última instancia afectar un fenotipo . Estos productos son a menudo proteínas , pero en los genes no codificantes de proteínas, como el ARN de transferencia (ARNt) y el ARN nuclear pequeño (ARNpn) , el producto es un ARN no codificante funcional . El proceso de expresión génica es utilizado por todos los seres vivos conocidos: eucariotas (incluidos los organismos multicelulares ), procariotas ( bacterias y arqueas ) y utilizado por los virus , para generar la maquinaria macromolecular para la vida.
En genética , la expresión génica es el nivel más fundamental en el que el genotipo da lugar al fenotipo , es decir , al rasgo observable. La información genética almacenada en el ADN representa el genotipo, mientras que el fenotipo resulta de la "interpretación" de esa información. Dichos fenotipos suelen manifestarse mediante la síntesis de proteínas que controlan la estructura y el desarrollo del organismo, o que actúan como enzimas que catalizan vías metabólicas específicas.
Todos los pasos del proceso de expresión génica pueden ser modulados (regulados), incluyendo la transcripción , el empalme del ARN , la traducción y la modificación postraduccional de una proteína. La regulación de la expresión génica permite controlar el momento, la ubicación y la cantidad de un producto génico determinado (proteína o ARNnc) presente en una célula y puede tener un efecto profundo en la estructura y función celular. La regulación de la expresión génica es la base de la diferenciación celular , el desarrollo , la morfogénesis y la versatilidad y adaptabilidad de cualquier organismo . Por lo tanto, la regulación génica puede servir como sustrato para el cambio evolutivo.
Mecanismo
Transcripción

La producción de una copia de ARN a partir de una cadena de ADN se denomina transcripción y la realizan las ARN polimerasas , que añaden un ribonucleótido a la vez a una cadena de ARN en crecimiento según la ley de complementariedad de las bases de los nucleótidos. Este ARN es complementario a la cadena de ADN molde 3′ → 5′, [1] con la excepción de que las timinas (T) se reemplazan por uracilos (U) en el ARN y hay posibles errores.
En las bacterias, la transcripción la lleva a cabo un único tipo de ARN polimerasa, que necesita unirse a una secuencia de ADN llamada caja Pribnow con la ayuda de la proteína del factor sigma (factor σ) para iniciar la transcripción. En los eucariotas, la transcripción se realiza en el núcleo por tres tipos de ARN polimerasas, cada una de las cuales necesita una secuencia de ADN especial llamada promotor y un conjunto de proteínas de unión al ADN ( factores de transcripción ) para iniciar el proceso (véase la regulación de la transcripción a continuación). La ARN polimerasa I es responsable de la transcripción de los genes del ARN ribosómico (ARNr). La ARN polimerasa II (Pol II) transcribe todos los genes codificantes de proteínas, pero también algunos ARN no codificantes ( p. ej ., ARNsn, ARNsno o ARN largos no codificantes ). La ARN polimerasa III transcribe ARNr 5S , genes de ARN de transferencia (ARNt) y algunos ARN pequeños no codificantes ( p. ej. , 7SK ). La transcripción termina cuando la polimerasa encuentra una secuencia llamada terminador .
Procesamiento de ARNm
Mientras que la transcripción de genes codificadores de proteínas procariotas crea ARN mensajero (ARNm) que está listo para ser traducido a proteína, la transcripción de genes eucariotas deja una transcripción primaria de ARN (pre-ARN), que primero tiene que sufrir una serie de modificaciones para convertirse en un ARN maduro. Los tipos y pasos involucrados en los procesos de maduración varían entre preARN codificantes y no codificantes; es decir, aunque las moléculas de preARN tanto para ARNm como para ARNt sufren empalme, los pasos y la maquinaria involucrados son diferentes. [2] El procesamiento del ARN no codificante se describe a continuación (maduración del ARN no codificante).
El procesamiento del pre-ARNm incluye el recubrimiento 5' , que es un conjunto de reacciones enzimáticas que agregan 7-metilguanosina (m 7 G) al extremo 5' del pre-ARNm y, por lo tanto, protegen al ARN de la degradación por exonucleasas . [3] Luego, el recubrimiento m 7 G se une mediante el complejo de unión al recubrimiento heterodímero (CBC20/CBC80), que ayuda en la exportación del ARNm al citoplasma y también protege al ARN del desprotegido . [4]
Otra modificación es la escisión 3' y la poliadenilación . [5] Se producen si la secuencia señal de poliadenilación (5'- AAUAAA-3') está presente en el pre-ARNm, que generalmente está entre la secuencia codificante de la proteína y el terminador. [6] El pre-ARNm se escinde primero y luego se agregan una serie de ~200 adeninas (A) para formar la cola de poli(A), que protege al ARN de la degradación. [7] La cola de poli(A) está unida por múltiples proteínas de unión a poli(A) (PABP) necesarias para la exportación del ARNm y la reiniciación de la traducción. [8] En el proceso inverso de la deadenilación, las colas de poli(A) son acortadas por la exonucleasa CCR4-Not 3'-5', lo que a menudo conduce a la descomposición completa de la transcripción. [9]
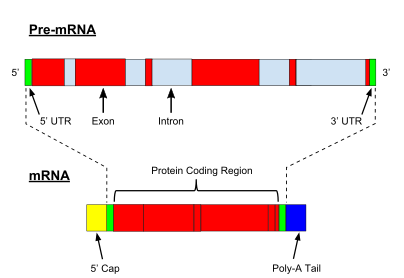
Una modificación muy importante del pre-ARNm eucariota es el empalme del ARN . La mayoría de los pre-ARNm eucariotas consisten en segmentos alternados llamados exones e intrones . [10] Durante el proceso de empalme, un complejo catalítico ARN-proteína conocido como espliceosoma cataliza dos reacciones de transesterificación , que eliminan un intrón y lo liberan en forma de estructura de lazo, y luego empalman los exones vecinos. [11] En ciertos casos, algunos intrones o exones pueden eliminarse o retenerse en el ARNm maduro. [12] Este llamado empalme alternativo crea una serie de transcripciones diferentes que se originan a partir de un solo gen. Debido a que estas transcripciones pueden traducirse potencialmente en diferentes proteínas, el empalme extiende la complejidad de la expresión génica eucariota y el tamaño del proteoma de una especie . [13]
El procesamiento extensivo del ARN puede ser una ventaja evolutiva que ha sido posible gracias al núcleo de los eucariotas. En los procariotas, la transcripción y la traducción se producen juntas, mientras que en los eucariotas, la membrana nuclear separa los dos procesos, lo que da tiempo para que se produzca el procesamiento del ARN. [14]
Maduración del ARN no codificante
En la mayoría de los organismos, los genes no codificantes (ARNnc) se transcriben como precursores que sufren un procesamiento posterior. En el caso de los ARN ribosómicos (ARNr), a menudo se transcriben como un pre-ARNr que contiene uno o más ARNr. El pre-ARNr se escinde y modifica ( 2'- O -metilación y formación de pseudouridina ) en sitios específicos por aproximadamente 150 especies diferentes de ARN pequeño restringido al nucléolo, llamadas ARNsno. Los ARNsno se asocian con proteínas, formando snoRNP. Mientras que la parte del ARNsno se aparea con el ARN objetivo y, por lo tanto, coloca la modificación en un sitio preciso, la parte proteica realiza la reacción catalítica. En eucariotas, en particular una snoRNP llamada ARNasa, MRP escinde el pre-ARNr 45S en los ARNr 28S , 5.8S y 18S . Los factores de procesamiento del ARNr y del ARN forman grandes agregados llamados nucléolo . [15]
En el caso del ARN de transferencia (ARNt), por ejemplo, la secuencia 5′ es eliminada por la ARNasa P [ 16], mientras que el extremo 3′ es eliminado por la enzima ARNt Z [17] y la cola CCA 3′ no moldeada es añadida por una nucleotidil transferasa [18] . En el caso del micro ARN (miARN) , los miARN se transcriben primero como transcripciones primarias o pri-miARN con una tapa y una cola de poli-A y se procesan en estructuras cortas de tallo-bucle de 70 nucleótidos conocidas como pre-miARN en el núcleo celular por las enzimas Drosha y Pasha . Después de ser exportado, se procesa para madurar miARN en el citoplasma mediante la interacción con la endonucleasa Dicer , que también inicia la formación del complejo de silenciamiento inducido por ARN (RISC) , compuesto por la proteína Argonaute .
Incluso los snRNA y snoRNA sufren una serie de modificaciones antes de convertirse en parte del complejo RNP funcional. [19] Esto se hace ya sea en el nucleoplasma o en los compartimentos especializados llamados cuerpos de Cajal . [20] Sus bases son metiladas o pseudouridiniladas por un grupo de pequeños ARN específicos del cuerpo de Cajal (scaRNA) , que son estructuralmente similares a los snoRNA. [21]
Exportación de ARN
En los eucariotas, la mayor parte del ARN maduro debe exportarse al citoplasma desde el núcleo . Si bien algunos ARN funcionan en el núcleo, muchos ARN se transportan a través de los poros nucleares y hacia el citosol . [22] La exportación de ARN requiere la asociación con proteínas específicas conocidas como exportinas. Las moléculas de exportina específicas son responsables de la exportación de un tipo de ARN determinado. El transporte de ARNm también requiere la asociación correcta con el complejo de unión de exones (EJC), que garantiza que se complete el procesamiento correcto del ARNm antes de la exportación. En algunos casos, los ARN se transportan adicionalmente a una parte específica del citoplasma, como una sinapsis ; luego son remolcados por proteínas motoras que se unen a través de proteínas de enlace a secuencias específicas (llamadas "códigos postales") en el ARN. [23]
Traducción
Para algunos ARN no codificantes, el ARN maduro es el producto génico final. [24] En el caso del ARN mensajero (ARNm), el ARN es un portador de información que codifica la síntesis de una o más proteínas. El ARNm que lleva una única secuencia de proteína (común en eucariotas) es monocistrónico , mientras que el ARNm que lleva múltiples secuencias de proteínas (común en procariotas) se conoce como policistrónico .
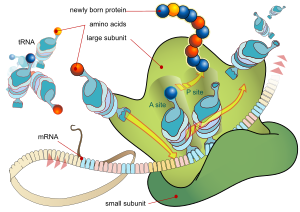
Cada ARNm consta de tres partes: una región no traducida 5' (5'UTR), una región codificante de proteínas o marco de lectura abierto (ORF) y una región no traducida 3' (3'UTR). La región codificante lleva información para la síntesis de proteínas codificada por el código genético para formar tripletes. Cada triplete de nucleótidos de la región codificante se llama codón y corresponde a un sitio de unión complementario a un triplete anticodón en el ARN de transferencia. Los ARN de transferencia con la misma secuencia anticodón siempre llevan un tipo idéntico de aminoácido . Luego, los aminoácidos se encadenan entre sí por el ribosoma de acuerdo con el orden de tripletes en la región codificante. El ribosoma ayuda al ARN de transferencia a unirse al ARN mensajero y toma el aminoácido de cada ARN de transferencia y crea una proteína sin estructura a partir de él. [25] [26] Cada molécula de ARNm se traduce en muchas moléculas de proteína, en promedio ~2800 en mamíferos. [27] [28]
En los procariotas, la traducción generalmente ocurre en el punto de transcripción (cotranscripcionalmente), a menudo utilizando un ARN mensajero que todavía está en proceso de creación. En los eucariotas, la traducción puede ocurrir en una variedad de regiones de la célula dependiendo de dónde se supone que está la proteína que se está escribiendo. Las ubicaciones principales son el citoplasma para las proteínas citoplasmáticas solubles y la membrana del retículo endoplasmático para las proteínas que se exportan desde la célula o se insertan en una membrana celular . Las proteínas que se supone que se producen en el retículo endoplasmático se reconocen a mitad del proceso de traducción. Esto está gobernado por la partícula de reconocimiento de señales , una proteína que se une al ribosoma y lo dirige al retículo endoplasmático cuando encuentra un péptido señal en la cadena de aminoácidos en crecimiento (nasciente). [29]
Plegable

Cada proteína existe como un polipéptido desplegado o una espiral aleatoria cuando se traduce de una secuencia de ARNm a una cadena lineal de aminoácidos . Este polipéptido carece de cualquier estructura tridimensional desarrollada (el lado izquierdo de la figura vecina). El polipéptido luego se pliega en su estructura tridimensional característica y funcional a partir de una espiral aleatoria . [30] Los aminoácidos interactúan entre sí para producir una estructura tridimensional bien definida, la proteína plegada (el lado derecho de la figura) conocida como el estado nativo . La estructura tridimensional resultante está determinada por la secuencia de aminoácidos ( dogma de Anfinsen ). [31]
La estructura tridimensional correcta es esencial para el funcionamiento, aunque algunas partes de las proteínas funcionales pueden permanecer sin plegar . [32] La falta de plegamiento en la forma deseada generalmente produce proteínas inactivas con diferentes propiedades, incluidos priones tóxicos . Se cree que varias enfermedades neurodegenerativas y de otro tipo son resultado de la acumulación de proteínas mal plegadas . [33] Muchas alergias son causadas por el plegamiento de las proteínas, ya que el sistema inmunológico no produce anticuerpos para ciertas estructuras proteínicas. [34]
Las enzimas llamadas chaperonas ayudan a la proteína recién formada a alcanzar ( plegarse ) la estructura tridimensional que necesita para funcionar. [35] De manera similar, las chaperonas de ARN ayudan a los ARN a alcanzar sus formas funcionales. [36] Ayudar al plegamiento de proteínas es una de las principales funciones del retículo endoplásmico en los eucariotas.
Translocación
Las proteínas secretoras de eucariotas o procariotas deben ser translocadas para entrar en la vía secretora. Las proteínas recién sintetizadas son dirigidas al canal de translocación Sec61 eucariota o SecYEG procariota por péptidos señal . La eficiencia de la secreción de proteínas en eucariotas depende en gran medida del péptido señal que se haya utilizado. [37]
Transporte de proteínas
Muchas proteínas están destinadas a otras partes de la célula que no sean el citosol y se utiliza una amplia gama de secuencias de señalización (o péptidos señal) para dirigir las proteínas a donde se supone que deben estar. [38] [39] En los procariotas, este es normalmente un proceso simple debido a la compartimentación limitada de la célula. [40] Sin embargo, en los eucariotas hay una gran variedad de procesos de orientación diferentes para garantizar que la proteína llegue al orgánulo correcto. [41]
No todas las proteínas permanecen dentro de la célula y muchas se exportan, por ejemplo, las enzimas digestivas , las hormonas y las proteínas de la matriz extracelular . En los eucariotas la vía de exportación está bien desarrollada y el principal mecanismo para la exportación de estas proteínas es la translocación al retículo endoplasmático, seguida del transporte a través del aparato de Golgi . [42] [43]
Regulación de la expresión genética

La regulación de la expresión génica es el control de la cantidad y el momento de aparición del producto funcional de un gen. El control de la expresión es vital para permitir que una célula produzca los productos génicos que necesita cuando los necesita; a su vez, esto le da a las células la flexibilidad para adaptarse a un entorno variable, señales externas, daños a la célula y otros estímulos. En términos más generales, la regulación génica le da a la célula el control sobre toda la estructura y función, y es la base de la diferenciación celular , la morfogénesis y la versatilidad y adaptabilidad de cualquier organismo.
Se utilizan numerosos términos para describir los tipos de genes dependiendo de cómo se regulan; estos incluyen:
- Un gen constitutivo es un gen que se transcribe continuamente a diferencia de un gen facultativo, que sólo se transcribe cuando es necesario.
- Un gen de mantenimiento es un gen que se requiere para mantener la función celular básica y, por lo tanto, se expresa típicamente en todos los tipos de células de un organismo. Algunos ejemplos incluyen actina , GAPDH y ubiquitina . Algunos genes de mantenimiento se transcriben a una tasa relativamente constante y estos genes se pueden usar como punto de referencia en experimentos para medir las tasas de expresión de otros genes.
- Un gen facultativo es un gen que sólo se transcribe cuando es necesario, a diferencia de un gen constitutivo.
- Un gen inducible es un gen cuya expresión responde a un cambio ambiental o depende de la posición en el ciclo celular.
Cualquier paso de la expresión génica puede ser modulado, desde el paso de transcripción ADN-ARN hasta la modificación postraduccional de una proteína. La estabilidad del producto génico final, ya sea ARN o proteína, también contribuye al nivel de expresión del gen: un producto inestable da como resultado un nivel de expresión bajo. En general, la expresión génica se regula a través de cambios [44] en el número y tipo de interacciones entre moléculas [45] que influyen colectivamente en la transcripción del ADN [46] y la traducción del ARN. [47]
Algunos ejemplos sencillos de dónde es importante la expresión genética son:
- Control de la expresión de insulina para que dé una señal para la regulación de la glucosa en sangre .
- Inactivación del cromosoma X en mamíferos hembras para evitar una "sobredosis" de los genes que contiene.
- Los niveles de expresión de ciclina controlan la progresión a través del ciclo celular eucariota .
Regulación transcripcional

La regulación de la transcripción se puede dividir en tres vías principales de influencia: genética (interacción directa de un factor de control con el gen), interacción de modulación de un factor de control con la maquinaria de transcripción y epigenética (cambios no secuenciales en la estructura del ADN que influyen en la transcripción). [48] [49]

La interacción directa con el ADN es el método más simple y directo por el cual una proteína cambia los niveles de transcripción. [50] Los genes a menudo tienen varios sitios de unión de proteínas alrededor de la región codificante con la función específica de regular la transcripción. [51] Hay muchas clases de sitios de unión de ADN reguladores conocidos como potenciadores , aislantes y silenciadores . [52] Los mecanismos para regular la transcripción son variados, desde bloquear sitios de unión clave en el ADN para la ARN polimerasa hasta actuar como activador y promover la transcripción al ayudar a la unión de la ARN polimerasa. [53]
La actividad de los factores de transcripción se modula aún más mediante señales intracelulares que provocan modificaciones postraduccionales de las proteínas, como la fosforilación , la acetilación o la glicosilación . [54] Estos cambios influyen en la capacidad de un factor de transcripción para unirse, directa o indirectamente, al ADN promotor, para reclutar a la ARN polimerasa o para favorecer la elongación de una molécula de ARN recién sintetizada. [55]
La membrana nuclear en eucariotas permite una mayor regulación de los factores de transcripción por la duración de su presencia en el núcleo, que está regulada por cambios reversibles en su estructura y por la unión de otras proteínas. [56] Los estímulos ambientales o señales endocrinas [57] pueden causar la modificación de las proteínas reguladoras [58] provocando cascadas de señales intracelulares [59] , que resultan en la regulación de la expresión genética.
Se ha hecho evidente que existe una influencia significativa de los efectos no específicos de la secuencia de ADN sobre la transcripción. [60] Estos efectos se denominan epigenéticos e involucran la estructura de orden superior del ADN, las proteínas de unión al ADN no específicas de la secuencia y la modificación química del ADN. [61] En general, los efectos epigenéticos alteran la accesibilidad del ADN a las proteínas y, por lo tanto, modulan la transcripción. [62]

En los eucariotas, la estructura de la cromatina , controlada por el código de las histonas , regula el acceso al ADN con impactos significativos en la expresión de genes en las áreas de eucromatina y heterocromatina . [63]
Potenciadores, factores de transcripción, complejos mediadores y bucles de ADN en la transcripción de mamíferos

La expresión génica en los mamíferos está regulada por muchos elementos cis-reguladores , incluidos los promotores centrales y los elementos proximales al promotor que se encuentran cerca de los sitios de inicio de la transcripción de los genes, aguas arriba en el ADN (hacia la región 5' de la cadena sentido ). Otros módulos cis-reguladores importantes se localizan en regiones del ADN que están distantes de los sitios de inicio de la transcripción. Estos incluyen potenciadores , silenciadores , aisladores y elementos de anclaje. [64] Los potenciadores y sus factores de transcripción asociados tienen un papel principal en la regulación de la expresión génica. [65]
Los potenciadores son regiones del genoma que regulan los genes. Los potenciadores controlan los programas de expresión génica específicos de cada tipo de célula, generalmente recorriendo largas distancias para acercarse físicamente a los promotores de sus genes objetivo. [66] Múltiples potenciadores, cada uno a menudo a decenas o cientos de miles de nucleótidos de distancia de sus genes objetivo, se unen a los promotores de sus genes objetivo y se coordinan entre sí para controlar la expresión génica. [66]
La ilustración muestra un potenciador que da una vuelta para acercarse al promotor de un gen diana. El bucle está estabilizado por un dímero de una proteína conectora (por ejemplo, el dímero de CTCF o YY1 ). Un miembro del dímero está anclado a su motivo de unión en el potenciador y el otro miembro está anclado a su motivo de unión en el promotor (representado por los zigzags rojos en la ilustración). [67] Varios factores de transcripción específicos de la función celular (entre los aproximadamente 1.600 factores de transcripción en una célula humana) [68] generalmente se unen a motivos específicos en un potenciador. [69] Una pequeña combinación de estos factores de transcripción unidos al potenciador, cuando se acerca a un promotor por un bucle de ADN, gobierna el nivel de transcripción del gen diana. El mediador (un complejo que generalmente consta de aproximadamente 26 proteínas en una estructura interactuante) comunica señales reguladoras de los factores de transcripción unidos al ADN del potenciador directamente a la enzima ARN polimerasa II (pol II) unida al promotor. [70]
Los potenciadores, cuando están activos, generalmente se transcriben a partir de ambas cadenas de ADN con ARN polimerasas que actúan en dos direcciones diferentes, produciendo dos eARN como se ilustra en la figura. [71] Un potenciador inactivo puede estar unido a un factor de transcripción inactivo. La fosforilación del factor de transcripción puede activarlo y ese factor de transcripción activado puede entonces activar el potenciador al que está unido (ver la pequeña estrella roja que representa la fosforilación del factor de transcripción unido al potenciador en la ilustración). [72] Un potenciador activado comienza la transcripción de su ARN antes de activar la transcripción del ARN mensajero de su gen objetivo. [73]
Metilación y desmetilación del ADN en la regulación transcripcional

La metilación del ADN es un mecanismo generalizado de influencia epigenética en la expresión génica, que se observa en bacterias y eucariotas y tiene funciones en el silenciamiento hereditario de la transcripción y la regulación de la transcripción. La metilación ocurre con mayor frecuencia en una citosina (ver Figura). La metilación de la citosina ocurre principalmente en secuencias de dinucleótidos donde una citosina es seguida por una guanina, un sitio CpG . El número de sitios CpG en el genoma humano es de aproximadamente 28 millones. [74] Dependiendo del tipo de célula, aproximadamente el 70% de los sitios CpG tienen una citosina metilada. [75]
La metilación de la citosina en el ADN tiene un papel importante en la regulación de la expresión génica. La metilación de los CpG en una región promotora de un gen suele reprimir la transcripción génica [76], mientras que la metilación de los CpG en el cuerpo de un gen aumenta la expresión. [77] Las enzimas TET desempeñan un papel central en la desmetilación de las citosinas metiladas. La desmetilación de los CpG en un promotor génico por la actividad de la enzima TET aumenta la transcripción del gen. [78]
Regulación transcripcional en el aprendizaje y la memoria

En una rata, el condicionamiento contextual del miedo (CFC) es una experiencia de aprendizaje dolorosa. Un solo episodio de CFC puede dar lugar a un recuerdo de miedo que dure toda la vida. [79] Después de un episodio de CFC, la metilación de la citosina se altera en las regiones promotoras de aproximadamente el 9,17 % de todos los genes en el ADN de las neuronas del hipocampo de una rata. [80] El hipocampo es donde se almacenan inicialmente los nuevos recuerdos. Después del CFC, alrededor de 500 genes han aumentado la transcripción (a menudo debido a la desmetilación de los sitios CpG en una región promotora) y alrededor de 1000 genes han disminuido la transcripción (a menudo debido a la 5-metilcitosina recién formada en los sitios CpG en una región promotora). El patrón de genes inducidos y reprimidos dentro de las neuronas parece proporcionar una base molecular para la formación de la primera memoria transitoria de este evento de entrenamiento en el hipocampo del cerebro de la rata. [80]
Se han establecido algunos mecanismos específicos que guían nuevas metilaciones de ADN y nuevas desmetilaciones de ADN en el hipocampo durante el establecimiento de la memoria (ver [81] para el resumen). Un mecanismo incluye guiar la isoforma corta de la enzima de desmetilación de ADN TET1 , TET1s, a aproximadamente 600 ubicaciones en el genoma. La guía se realiza mediante la asociación de TET1s con la proteína EGR1 , un factor de transcripción importante en la formación de la memoria. Llevar TET1s a estas ubicaciones inicia la desmetilación de ADN en esos sitios, regulando al alza los genes asociados. Un segundo mecanismo involucra a DNMT3A2, una isoforma de empalme de la ADN metiltransferasa DNMT3A, que agrega grupos metilo a las citosinas en el ADN. Esta isoforma es inducida por la actividad sináptica, y su ubicación de acción parece estar determinada por modificaciones postraduccionales de histonas (un código de histonas ). Los nuevos ARN mensajeros resultantes son luego transportados por partículas RNP mensajeras (gránulos neuronales) a las sinapsis de las neuronas, donde pueden traducirse en proteínas que afectan las actividades de las sinapsis. [81]
En particular, el gen del factor neurotrófico derivado del cerebro ( BDNF ) se conoce como un "gen del aprendizaje". [82] Después del CFC hubo una regulación positiva de la expresión del gen BDNF , relacionada con la disminución de la metilación de CpG de ciertos promotores internos del gen, y esto se correlacionó con el aprendizaje. [82]
Regulación transcripcional en el cáncer
La mayoría de los promotores de genes contienen una isla CpG con numerosos sitios CpG . [83] Cuando muchos de los sitios CpG del promotor de un gen están metilados, el gen se silencia. [84] Los cánceres colorrectales suelen tener de 3 a 6 mutaciones impulsoras y de 33 a 66 mutaciones autoestopistas o pasajeras. [85] Sin embargo, el silenciamiento transcripcional puede ser de mayor importancia que la mutación a la hora de provocar la progresión al cáncer. Por ejemplo, en los cánceres colorrectales, entre 600 y 800 genes se silencian transcripcionalmente por la metilación de la isla CpG (véase regulación de la transcripción en el cáncer ). La represión transcripcional en el cáncer también puede producirse por otros mecanismos epigenéticos , como la expresión alterada de microARN . [86] En el cáncer de mama, la represión transcripcional de BRCA1 puede producirse con mayor frecuencia por la sobretranscripción del microARN-182 que por la hipermetilación del promotor BRCA1 (véase Baja expresión de BRCA1 en cánceres de mama y de ovario ).
Regulación postranscripcional
En los eucariotas, donde se requiere la exportación de ARN antes de que sea posible la traducción, se cree que la exportación nuclear proporciona un control adicional sobre la expresión génica. Todo el transporte hacia dentro y hacia fuera del núcleo se realiza a través del poro nuclear y el transporte está controlado por una amplia gama de proteínas importinas y exportinas . [87]
La expresión de un gen que codifica una proteína solo es posible si el ARN mensajero que lleva el código sobrevive lo suficiente para ser traducido. [41] En una célula típica, una molécula de ARN solo es estable si se la protege específicamente de la degradación. [88] La degradación del ARN tiene particular importancia en la regulación de la expresión en células eucariotas donde el ARNm tiene que viajar distancias significativas antes de ser traducido. [89] En eucariotas, el ARN se estabiliza mediante ciertas modificaciones postranscripcionales, en particular la tapa 5' y la cola poliadenilada . [90]
La degradación intencional del ARNm se utiliza no solo como un mecanismo de defensa contra el ARN extraño (normalmente de virus), sino también como una vía de desestabilización del ARNm . [91] Si una molécula de ARNm tiene una secuencia complementaria a un ARN interferente pequeño , entonces se la selecciona para su destrucción a través de la vía de interferencia del ARN . [92]
Tres regiones principales no traducidas y microARN
Las tres regiones no traducidas principales (3′UTR) de los ARN mensajeros (ARNm) suelen contener secuencias reguladoras que influyen postranscripcionalmente en la expresión génica. Estas 3′-UTR suelen contener tanto sitios de unión para microARN (miARN) como para proteínas reguladoras. [93] Al unirse a sitios específicos dentro de la 3′-UTR, los miARN pueden disminuir la expresión génica de varios ARNm ya sea inhibiendo la traducción o causando directamente la degradación de la transcripción. [94] La 3′-UTR también puede tener regiones silenciadoras que se unen a proteínas represoras que inhiben la expresión de un ARNm. [95]
El 3′-UTR a menudo contiene elementos de respuesta a microARN (MRE) . Los MRE son secuencias a las que se unen los microARN. Estos son motivos predominantes dentro de los 3′-UTR. Entre todos los motivos reguladores dentro de los 3′-UTR (por ejemplo, incluidas las regiones silenciadoras), los MRE constituyen aproximadamente la mitad de los motivos. [96]
En 2014, el sitio web miRBase , [97] un archivo de secuencias y anotaciones de miRNA , enumeraba 28.645 entradas en 233 especies biológicas. De estas, 1.881 miRNA se encontraban en loci de miRNA humanos anotados. Se predijo que los miRNA tendrían un promedio de alrededor de cuatrocientos ARNm diana (que afectan la expresión de varios cientos de genes). [98] Friedman et al. [98] estiman que >45.000 sitios diana de miRNA dentro de los 3′UTR de ARNm humano se conservan por encima de los niveles de fondo, y >60% de los genes codificadores de proteínas humanas han estado bajo presión selectiva para mantener el emparejamiento con miRNA.
Experimentos directos muestran que un único miRNA puede reducir la estabilidad de cientos de ARNm únicos. [99] Otros experimentos muestran que un único miRNA puede reprimir la producción de cientos de proteínas, pero que esta represión a menudo es relativamente leve (menos del doble). [100] [101]
Los efectos de la desregulación de la expresión genética por parte de los microARN parecen ser importantes en el cáncer. [102] Por ejemplo, en los cánceres gastrointestinales, se han identificado nueve microARN alterados epigenéticamente y eficaces para regular negativamente las enzimas de reparación del ADN. [103]
Los efectos de la desregulación de la expresión genética por parte de los miRNA también parecen ser importantes en trastornos neuropsiquiátricos, como la esquizofrenia, el trastorno bipolar, la depresión mayor, la enfermedad de Parkinson, la enfermedad de Alzheimer y los trastornos del espectro autista. [104] [105]
Regulación traslacional
La regulación directa de la traducción es menos frecuente que el control de la transcripción o la estabilidad del ARNm, pero se utiliza ocasionalmente. [106] La inhibición de la traducción de proteínas es un objetivo principal de las toxinas y los antibióticos , por lo que pueden matar una célula anulando su control normal de la expresión genética. [107] Los inhibidores de la síntesis de proteínas incluyen el antibiótico neomicina y la toxina ricina . [108]
Modificaciones postraduccionales
Las modificaciones postraduccionales (PTM) son modificaciones covalentes de las proteínas. Al igual que el empalme del ARN, ayudan a diversificar significativamente el proteoma. Estas modificaciones suelen estar catalizadas por enzimas. Además, procesos como las adiciones covalentes a los residuos de la cadena lateral de aminoácidos a menudo pueden revertirse mediante otras enzimas. Sin embargo, algunos, como la escisión proteolítica de la estructura principal de la proteína, son irreversibles. [109]
Las PTM desempeñan muchas funciones importantes en la célula. [110] Por ejemplo, la fosforilación está principalmente implicada en la activación y desactivación de proteínas y en las vías de señalización. [111] Las PTM están implicadas en la regulación transcripcional: una función importante de la acetilación y la metilación es la modificación de la cola de las histonas, que altera la accesibilidad del ADN para la transcripción. [109] También se pueden ver en el sistema inmunológico, donde la glicosilación juega un papel clave. [112] Un tipo de PTM puede iniciar otro tipo de PTM, como se puede ver en cómo la ubiquitinación marca las proteínas para su degradación a través de la proteólisis. [109] La proteólisis, además de estar implicada en la descomposición de las proteínas, también es importante para activarlas y desactivarlas, y para regular procesos biológicos como la transcripción del ADN y la muerte celular. [113]
Medición

La medición de la expresión genética es una parte importante de muchas ciencias biológicas , ya que la capacidad de cuantificar el nivel en el que se expresa un gen en particular dentro de una célula, tejido u organismo puede proporcionar mucha información valiosa. Por ejemplo, la medición de la expresión genética puede:
- Identificar la infección viral de una célula ( expresión de proteína viral ).
- Determinar la susceptibilidad de un individuo al cáncer ( expresión de oncogenes ).
- Descubra si una bacteria es resistente a la penicilina ( expresión de betalactamasa ).
- El perfil de expresión genética evalúa un panel de genes para ayudar a comprender el mecanismo fundamental de una célula. Esto se utiliza cada vez más en la terapia contra el cáncer para dirigir la quimioterapia específica . ( Consulte RNA-Seq y DNA_microarray para obtener más detalles).
De manera similar, el análisis de la localización de la expresión de proteínas es una herramienta poderosa, y esto puede hacerse a escala de organismo o celular. La investigación de la localización es particularmente importante para el estudio del desarrollo en organismos multicelulares y como indicador de la función de las proteínas en células individuales. Lo ideal es que la medición de la expresión se realice detectando el producto génico final (para muchos genes, este es la proteína); sin embargo, a menudo es más fácil detectar uno de los precursores, típicamente el ARNm , e inferir los niveles de expresión génica a partir de estas mediciones.
cuantificación de ARNm
Los niveles de ARNm se pueden medir cuantitativamente mediante transferencia Northern , que proporciona información sobre el tamaño y la secuencia de las moléculas de ARNm. [114] Una muestra de ARN se separa en un gel de agarosa y se hibrida con una sonda de ARN marcada radiactivamente que es complementaria a la secuencia diana. [115] Luego, el ARN radiomarcado se detecta mediante una autorradiografía . [116] Debido a que el uso de reactivos radiactivos hace que el procedimiento requiera mucho tiempo y sea potencialmente peligroso, se han desarrollado métodos alternativos de etiquetado y detección, como las químicas de digoxigenina y biotina. [117] Las desventajas percibidas de la transferencia Northern son que se requieren grandes cantidades de ARN y que la cuantificación puede no ser completamente precisa, ya que implica medir la fuerza de la banda en una imagen de un gel. [118] Por otro lado, la información adicional del tamaño del ARNm de la transferencia Northern permite la discriminación de transcripciones empalmadas alternativamente. [119] [120]
Otro método para medir la abundancia de ARNm es la RT-qPCR. En esta técnica, la transcripción inversa es seguida por la PCR cuantitativa . La transcripción inversa genera primero una plantilla de ADN a partir del ARNm; esta plantilla monocatenaria se llama ADNc . La plantilla de ADNc se amplifica luego en el paso cuantitativo, durante el cual la fluorescencia emitida por las sondas de hibridación marcadas o los colorantes intercalantes cambia a medida que avanza el proceso de amplificación del ADN . [121] Con una curva estándar cuidadosamente construida, la qPCR puede producir una medición absoluta del número de copias del ARNm original, típicamente en unidades de copias por nanolitro de tejido homogeneizado o copias por célula. [122] La qPCR es muy sensible (la detección de una sola molécula de ARNm es teóricamente posible), pero puede ser costosa dependiendo del tipo de reportero utilizado; las sondas de oligonucleótidos marcadas con fluorescencia son más caras que los colorantes fluorescentes intercalantes no específicos. [123]
Para el perfil de expresión , o análisis de alto rendimiento de muchos genes dentro de una muestra, se puede realizar PCR cuantitativa para cientos de genes simultáneamente en el caso de matrices de baja densidad. [124] Un segundo enfoque es la micromatriz de hibridación . Una única matriz o "chip" puede contener sondas para determinar los niveles de transcripción para cada gen conocido en el genoma de uno o más organismos. [125] Alternativamente, se pueden utilizar tecnologías "basadas en etiquetas" como el análisis en serie de la expresión génica (SAGE) y RNA-Seq , que pueden proporcionar una medida relativa de la concentración celular de diferentes ARNm. [126] Una ventaja de los métodos basados en etiquetas es la "arquitectura abierta", que permite la medición exacta de cualquier transcripción, con una secuencia conocida o desconocida. [127] La secuenciación de próxima generación (NGS), como RNA-Seq , es otro enfoque, que produce grandes cantidades de datos de secuencia que se pueden hacer coincidir con un genoma de referencia. Aunque la NGS es comparativamente costosa, consume mucho tiempo y recursos, puede identificar polimorfismos de un solo nucleótido , variantes de empalme y genes nuevos, y también se puede utilizar para perfilar la expresión en organismos para los que hay poca o ninguna información de secuencia disponible. [128]
Perfiles de ARN en Wikipedia
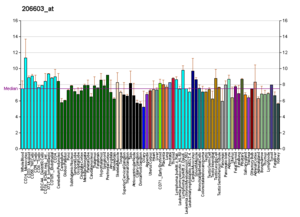
Perfiles como estos se encuentran para casi todas las proteínas que aparecen en Wikipedia. Son generados por organizaciones como el Instituto de Genómica de la Fundación de Investigación Novartis y el Instituto Europeo de Bioinformática . Se puede encontrar información adicional buscando en sus bases de datos (para ver un ejemplo del transportador GLUT4 que se muestra aquí, consulte la cita). [129] Estos perfiles indican el nivel de expresión de ADN (y, por lo tanto, de ARN producido) de una determinada proteína en un determinado tejido, y están codificados por colores en las imágenes ubicadas en el Recuadro de Proteínas en el lado derecho de cada página de Wikipedia.
Cuantificación de proteínas
En el caso de los genes que codifican proteínas, el nivel de expresión se puede evaluar directamente mediante varios métodos con algunas analogías claras con las técnicas de cuantificación de ARNm.
Uno de los métodos más utilizados es realizar un Western blot contra la proteína de interés. [130] Esto proporciona información sobre el tamaño de la proteína además de su identidad. Una muestra (a menudo lisado celular ) se separa en un gel de poliacrilamida , se transfiere a una membrana y luego se prueba con un anticuerpo contra la proteína de interés. El anticuerpo puede conjugarse con un fluoróforo o con peroxidasa de rábano picante para la obtención de imágenes y/o cuantificación. La naturaleza basada en gel de este ensayo hace que la cuantificación sea menos precisa, pero tiene la ventaja de poder identificar modificaciones posteriores de la proteína, por ejemplo, proteólisis o ubiquitinación, a partir de cambios en el tamaño.
Correlación ARNm-proteína
Si bien la transcripción refleja directamente la expresión génica, el número de copias de moléculas de ARNm no se correlaciona directamente con el número de moléculas de proteína traducidas a partir del ARNm. La cuantificación tanto de la proteína como del ARNm permite una correlación de los dos niveles. La regulación de cada paso de la expresión génica puede afectar la correlación, como se muestra para la regulación de la traducción [28] o la estabilidad de la proteína. [131] Los factores postraduccionales, como el transporte de proteínas en células altamente polares, [132] también pueden influir en la correlación ARNm-proteína medida.
Localización

El análisis de la expresión no se limita a la cuantificación; también se puede determinar la localización. El ARNm se puede detectar con una cadena de ARNm complementaria adecuadamente marcada y la proteína se puede detectar mediante anticuerpos marcados. A continuación, la muestra analizada se observa mediante microscopía para identificar dónde se encuentra el ARNm o la proteína.

Al reemplazar el gen con una nueva versión fusionada a un marcador de proteína fluorescente verde o similar, la expresión puede cuantificarse directamente en células vivas. Esto se hace mediante imágenes utilizando un microscopio de fluorescencia . Es muy difícil clonar una proteína fusionada a GFP en su ubicación nativa en el genoma sin afectar los niveles de expresión, por lo que este método a menudo no se puede utilizar para medir la expresión génica endógena. Sin embargo, se usa ampliamente para medir la expresión de un gen introducido artificialmente en la célula, por ejemplo a través de un vector de expresión . Al fusionar una proteína diana con un reportero fluorescente, el comportamiento de la proteína, incluida su localización celular y nivel de expresión, se puede cambiar significativamente.
El ensayo inmunoabsorbente ligado a enzimas funciona mediante el uso de anticuerpos inmovilizados en una placa de microtitulación para capturar las proteínas de interés de las muestras añadidas al pocillo. Mediante el uso de un anticuerpo de detección conjugado con una enzima o un fluoróforo, la cantidad de proteína unida se puede medir con precisión mediante detección fluorométrica o colorimétrica . El proceso de detección es muy similar al de un Western blot, pero al evitar los pasos de gel se puede lograr una cuantificación más precisa.
Sistema de expresión
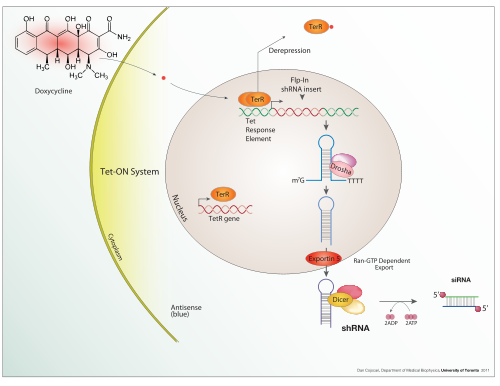
Un sistema de expresión es un sistema diseñado específicamente para la producción de un producto génico de elección. Normalmente se trata de una proteína, aunque también puede ser ARN, como el ARNt o una ribozima . Un sistema de expresión consta de un gen, normalmente codificado por ADN , y la maquinaria molecular necesaria para transcribir el ADN en ARNm y traducir el ARNm en proteína utilizando los reactivos proporcionados. En el sentido más amplio, esto incluye todas las células vivas, pero el término se utiliza más normalmente para referirse a la expresión como herramienta de laboratorio. Por tanto, un sistema de expresión suele ser artificial de alguna manera. Sin embargo, los sistemas de expresión son un proceso fundamentalmente natural. Los virus son un excelente ejemplo, ya que se replican utilizando la célula huésped como sistema de expresión para las proteínas y el genoma virales.
Expresión inducible
La doxiciclina también se utiliza en la activación transcripcional controlada por tetraciclina "Tet-on" y "Tet-off" para regular la expresión transgénica en organismos y cultivos celulares .
En la naturaleza
Además de estas herramientas biológicas, ciertas configuraciones de ADN observadas de forma natural (genes, promotores, potenciadores, represores) y la maquinaria asociada en sí se denominan sistemas de expresión. Este término se utiliza normalmente en el caso en que un gen o un conjunto de genes se activa en condiciones bien definidas, por ejemplo, el sistema de expresión de interruptor represor simple en el fago Lambda y el sistema de operador lac en las bacterias. Varios sistemas de expresión naturales se utilizan directamente o se modifican y se utilizan para sistemas de expresión artificiales, como el sistema de expresión Tet-on y Tet-off .
Redes de genes
En ocasiones, se ha considerado a los genes como nodos de una red, en la que las entradas son proteínas como los factores de transcripción y las salidas son el nivel de expresión génica. El nodo en sí mismo realiza una función, y el funcionamiento de estas funciones se ha interpretado como la realización de una especie de procesamiento de información dentro de las células y determina el comportamiento celular.
Las redes de genes también se pueden construir sin formular un modelo causal explícito. Este suele ser el caso cuando se ensamblan redes a partir de grandes conjuntos de datos de expresión. [133] La covariación y correlación de la expresión se calcula a través de una gran muestra de casos y mediciones (a menudo datos del transcriptoma o del proteoma ). La fuente de variación puede ser experimental o natural (observacional). Hay varias formas de construir redes de expresión genética, pero un enfoque común es calcular una matriz de todas las correlaciones de expresión por pares en diferentes condiciones, puntos temporales o individuos y convertir la matriz (después de establecer un umbral en algún valor de corte) en una representación gráfica en la que los nodos representan genes, transcripciones o proteínas y los bordes que conectan estos nodos representan la fuerza de la asociación (véase GeneNetwork GeneNetwork 2). [134]
Técnicas y herramientas
Las siguientes técnicas experimentales se utilizan para medir la expresión genética y se enumeran en orden cronológico aproximado, comenzando por las tecnologías más antiguas y consolidadas. Se dividen en dos grupos según su grado de multiplexidad .
- Técnicas de complejidad baja a media:
- Técnicas de plexación superior:
- SABIO [136]
- Microarray de ADN [137]
- Matriz de mosaicos [138]
- Secuenciación de ARN [139]
Bases de datos de expresión genética
- Ómnibus de expresión genética (GEO) en NCBI [140]
- Atlas de expresiones en el EBI
- Bgee Bgee en el Instituto Suizo de Bioinformática SIB
- Base de datos de expresión genética de ratones en el Laboratorio Jackson
- CollecTF : una base de datos de sitios de unión de factores de transcripción validados experimentalmente en bacterias. [141]
- COLOMBOS: colección de compendios de expresión bacteriana. [142]
- Base de datos de microarrays de muchos microbios: datos microbianos de Affymetrix [143]
Véase también
- Prueba de expresión molecular AlloMap
- Marcadores
- Secuencia EPIC
- Etiqueta de secuencia expresada
- Atlas de expresión
- Perfiles de expresión
- Estructura genética
- Ingeniería genética
- Organismo genéticamente modificado
- Lista de bases de datos biológicas
- Lista de genes humanos
- Gen oscilante
- Paramutación
- Producción de proteínas
- Purificación de proteínas
- Ribonomica
- Cresta
- Herramienta de creación de perfiles de secuencias
- Ráfaga transcripcional
- Ruido transcripcional
- Transcripción de función desconocida
Referencias
- ^ Brueckner F, Armache KJ, Cheung A, Damsma GE, Kettenberger H, Lehmann E, et al. (febrero de 2009). "Estudios de estructura y función del complejo de elongación de la ARN polimerasa II". Acta Crystallographica. Sección D, Cristalografía biológica . 65 (Pt 2): 112–120. Bibcode :2009AcCrD..65..112B. doi :10.1107/S0907444908039875. PMC 2631633 . PMID 19171965.
- ^ Krebs JE, Goldstein ES, Kilpatrick ST (2 de marzo de 2017). Genes de Lewin XII . Burlington, MA: Jones & Bartlett Learning. ISBN 978-1-284-10449-3.OCLC 965781334 .
- ^ Ramanathan A, Robb GB, Chan SH (septiembre de 2016). "Recubrimiento del ARNm: funciones biológicas y aplicaciones". Nucleic Acids Research . 44 (16): 7511–7526. doi :10.1093/nar/gkw551. PMC 5027499 . PMID 27317694.
- ^ Gonatopoulos-Pournatzis T, Cowling VH (enero de 2014). "Complejo de unión a capuchón (CBC)". The Biochemical Journal . 457 (2): 231–242. doi :10.1042/BJ20131214. PMC 3901397 . PMID 24354960.
- ^ Neve J, Patel R, Wang Z, Louey A, Furger AM (julio de 2017). "Escisión y poliadenilación: la terminación del mensaje expande la regulación génica". RNA Biology . 14 (7): 865–890. doi :10.1080/15476286.2017.1306171. PMC 5546720 . PMID 28453393.
- ^ Borodulina OR, Kramerov DA (septiembre de 2008). "Las transcripciones sintetizadas por la ARN polimerasa III pueden poliadenilarse de una manera dependiente de AAUAAA". ARN . 14 (9): 1865–1873. doi :10.1261/rna.1006608. PMC 2525947 . PMID 18658125.
- ^ Munoz-Tello P, Rajappa L, Coquille S, Thore S (2015). "Poliuridilación en eucariotas: una modificación del extremo 3' que regula la vida del ARN". BioMed Research International . 2015 : 968127. doi : 10.1155/2015/968127 . PMC 4442281 . PMID 26078976.
- ^ Passmore LA, Coller J (febrero de 2022). "Funciones de las colas de poli(A) del ARNm en la regulación de la expresión génica eucariota". Nature Reviews. Molecular Cell Biology . 23 (2): 93–106. doi :10.1038/s41580-021-00417-y. PMC 7614307 . PMID 34594027.
- ^ Morozov IY, Jones MG, Razak AA, Rigden DJ, Caddick MX (enero de 2010). "La modificación de CUCU del ARNm promueve la descapuchación y la degradación de la transcripción en Aspergillus nidulans". Biología molecular y celular . 30 (2): 460–469. doi :10.1128/MCB.00997-09. PMC 2798463 . PMID 19901075.
- ^ Darnell JE (abril de 2013). "Reflexiones sobre la historia del procesamiento del pre-ARNm y aspectos destacados del conocimiento actual: una imagen unificada". ARN . 19 (4): 443–460. doi :10.1261/rna.038596.113. PMC 3677254 . PMID 23440351.
- ^ Zhang L, Vielle A, Espinosa S, Zhao R (mayo de 2019). "ARN en el espliceosoma: perspectiva a partir de las estructuras crioEM". Wiley Interdisciplinary Reviews. ARN . 10 (3): e1523. doi :10.1002/wrna.1523. PMC 6450755 . PMID 30729694.
- ^ Hossain MA, Rodriguez CM, Johnson TL (octubre de 2011). "Características clave del gen SUS1 de Saccharomyces cerevisiae de dos intrones contribuyen a su empalme alternativo". Nucleic Acids Research . 39 (19): 8612–8627. doi :10.1093/nar/gkr497. PMC 3201863 . PMID 21749978.
- ^ Baralle FE, Giudice J (julio de 2017). "Empalme alternativo como regulador del desarrollo y la identidad tisular". Nature Reviews. Molecular Cell Biology . 18 (7): 437–451. doi :10.1038/nrm.2017.27. PMC 6839889 . PMID 28488700.
- ^ Baum B, Spang A (diciembre de 2023). "Sobre el origen del núcleo: una hipótesis". Microbiology and Molecular Biology Reviews . 87 (4): e0018621. doi :10.1128/mmbr.00186-21. PMC 10732040 . PMID 38018971.
- ^ Sirri V, Urcuqui-Inchima S, Roussel P, Hernandez-Verdun D (enero de 2008). "Nucleolo: el fascinante cuerpo nuclear". Histoquímica y biología celular . 129 (1): 13–31. doi :10.1007/s00418-007-0359-6. PMC 2137947 . PMID 18046571.
- ^ Frank DN, Pace NR (1998). "Ribonucleasa P: unidad y diversidad en una ribozima procesadora de ARNt". Revisión anual de bioquímica . 67 : 153–180. doi : 10.1146/annurev.biochem.67.1.153 . PMID 9759486.
- ^ Ceballos M, Vioque A (2007). "tRNase Z". Protein and Peptide Letters . 14 (2): 137–145. doi :10.2174/092986607779816050. PMID 17305600.
- ^ Weiner AM (octubre de 2004). "Maduración del ARNt: polimerización del ARN sin una plantilla de ácido nucleico". Current Biology . 14 (20): R883–R885. Bibcode :2004CBio...14.R883W. doi : 10.1016/j.cub.2004.09.069 . PMID 15498478.
- ^ Bratkovič T, Božič J, Rogelj B (febrero de 2020). "Diversidad funcional de pequeños ARN nucleolares". Investigación de ácidos nucleicos . 48 (4): 1627–1651. doi : 10.1093/nar/gkz1140. PMC 7038934 . PMID 31828325.
- ^ Nizami Z, Deryusheva S, Gall JG (julio de 2010). "El cuerpo de Cajal y el cuerpo del locus de histonas". Cold Spring Harbor Perspectives in Biology . 2 (7): a000653. doi :10.1101/cshperspect.a000653. PMC 2890199 . PMID 20504965.
- ^ Darzacq X, Jády BE, Verheggen C, Kiss AM, Bertrand E, Kiss T (junio de 2002). "ARN nucleares pequeños específicos del cuerpo de Cajal: una nueva clase de ARN guía de 2'-O-metilación y pseudouridilación". The EMBO Journal . 21 (11): 2746–2756. doi :10.1093/emboj/21.11.2746. PMC 126017 . PMID 12032087.
- ^ Köhler A, Hurt E (octubre de 2007). "Exportación de ARN desde el núcleo al citoplasma". Nature Reviews. Molecular Cell Biology . 8 (10): 761–773. doi :10.1038/nrm2255. PMID 17786152. S2CID 10836137.
- ^ Jambhekar A, Derisi JL (mayo de 2007). "Determinantes que actúan en cis del transporte asimétrico de ARN citoplasmático". ARN . 13 (5): 625–642. doi :10.1261/rna.262607. PMC 1852811 . PMID 17449729.
- ^ Amaral PP, Dinger ME, Mercer TR, Mattick JS (marzo de 2008). "El genoma eucariota como una máquina de ARN". Science . 319 (5871): 1787–1789. Bibcode :2008Sci...319.1787A. doi :10.1126/science.1155472. PMID 18369136. S2CID 206511756.
- ^ Hansen TM, Baranov PV, Ivanov IP, Gesteland RF, Atkins JF (mayo de 2003). "Mantenimiento del marco de lectura abierto correcto por el ribosoma". EMBO Reports . 4 (5): 499–504. doi :10.1038/sj.embor.embor825. PMC 1319180 . PMID 12717454.
- ^ Berk V, Cate JH (junio de 2007). "Información sobre la biosíntesis de proteínas a partir de las estructuras de los ribosomas bacterianos". Current Opinion in Structural Biology . 17 (3): 302–309. doi :10.1016/j.sbi.2007.05.009. PMID 17574829.
- ^ Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, et al. (mayo de 2011). "Cuantificación global del control de la expresión génica en mamíferos" (PDF) . Nature . 473 (7347): 337–342. Bibcode :2011Natur.473..337S. doi :10.1038/nature10098. PMID 21593866. S2CID 205224972.
- ^ ab Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, et al. (Marzo de 2013). "Corrección: cuantificación global del control de la expresión de genes de mamíferos". Naturaleza . 495 (7439): 126-127. Código Bib :2013Natur.495..126S. doi : 10.1038/naturaleza11848 . PMID 23407496.
- ^ Hegde RS, Kang SW (julio de 2008). "El concepto de regulación translocativa". The Journal of Cell Biology . 182 (2): 225–232. doi :10.1083/jcb.200804157. PMC 2483521 . PMID 18644895.
- ^ Alberts B , Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). "La forma y la estructura de las proteínas". Biología molecular de la célula (cuarta edición). Nueva York y Londres: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen CB (julio de 1972). "La formación y estabilización de la estructura de las proteínas". The Biochemical Journal . 128 (4): 737–749. doi :10.1042/bj1280737. PMC 1173893 . PMID 4565129.
- ^ Berg JM, Tymoczko JL, Lubert Stryer ; Contenido web de Neil D. Clarke (2002). "3. Estructura y función de las proteínas". Bioquímica . San Francisco: WH Freeman. ISBN 978-0-7167-4684-3.
- ^ Selkoe DJ (diciembre de 2003). "Plegamiento de proteínas de forma fatal". Nature . 426 (6968): 900–904. Bibcode :2003Natur.426..900S. doi :10.1038/nature02264. PMID 14685251. S2CID 6451881.
- ^ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, et al. (2010). "Estructura y función de las proteínas". Essential Cell Biology (3.ª ed.). Nueva York: Garland Science, Taylor and Francis Group, LLC. págs. 120-170. ISBN 978-0-8153-4129-1.
- ^ Hebert DN, Molinari M (octubre de 2007). "Dentro y fuera del RE: plegamiento de proteínas, control de calidad, degradación y enfermedades humanas relacionadas". Physiological Reviews . 87 (4): 1377–1408. doi :10.1152/physrev.00050.2006. PMID 17928587.
- ^ Russell R (enero de 2008). "Plegamiento incorrecto del ARN y la acción de las chaperonas". Frontiers in Bioscience . 13 (13): 1–20. doi :10.2741/2557. PMC 2610265 . PMID 17981525.
- ^ Kober L, Zehe C, Bode J (abril de 2013). "Péptidos señal optimizados para el desarrollo de líneas celulares CHO de alta expresión". Biotecnología y bioingeniería . 110 (4): 1164–1173. doi :10.1002/bit.24776. PMID 23124363. S2CID 449870.
- ^ Lu J, Wu T, Zhang B, Liu S, Song W, Qiao J, et al. (mayo de 2021). "Tipos de señales de localización nuclear y mecanismos de importación de proteínas al núcleo". Comunicación celular y señalización . 19 (1): 60. doi : 10.1186/s12964-021-00741-y . PMC 8140498 . PMID 34022911.
- ^ Lang S, Nguyen D, Bhadra P, Jung M, Helms V, Zimmermann R (11 de julio de 2022). "Características de los péptidos señal que determinan las especificidades del sustrato de los componentes de direccionamiento y translocación en la importación de proteínas del RE humano". Frontiers in Physiology . 13 : 833540. doi : 10.3389/fphys.2022.833540 . PMC 9309488 . PMID 35899032.
- ^ Murat D, Byrne M, Komeili A (octubre de 2010). "Biología celular de los orgánulos procariotas". Cold Spring Harbor Perspectives in Biology . 2 (10): a000422. doi :10.1101/cshperspect.a000422. PMC 2944366 . PMID 20739411.
- ^ ab Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002), "Del ARN a la proteína", Biología molecular de la célula. 4.ª edición , Garland Science , consultado el 10 de junio de 2024.
- ^ Moreau P, Brandizzi F, Hanton S, Chatre L, Melser S, Hawes C, et al. (2007). "La interfaz ER-Golgi de la planta: un complejo de membrana altamente estructurado y dinámico". Journal of Experimental Botany . 58 (1): 49–64. doi : 10.1093/jxb/erl135 . PMID 16990376.
- ^ Prudovsky I, Tarantini F, Landriscina M, Neivandt D, Soldi R, Kirov A, et al. (Abril de 2008). "Secreción sin Golgi". Revista de bioquímica celular . 103 (5): 1327-1343. doi :10.1002/jcb.21513. PMC 2613191 . PMID 17786931.
- ^ Zaidi SK, Young DW, Choi JY, Pratap J, Javed A, Montecino M, et al. (octubre de 2004). "Tráfico intranuclear: organización y ensamblaje de la maquinaria reguladora para el control biológico combinatorio". The Journal of Biological Chemistry . 279 (42): 43363–43366. doi : 10.1074/jbc.R400020200 . PMID 15277516.
- ^ Mattick JS, Amaral PP, Dinger ME, Mercer TR, Mehler MF (enero de 2009). "Regulación de los procesos epigenéticos por ARN". BioEssays . 31 (1): 51–59. doi : 10.1002/bies.080099 . PMID 19154003.
- ^ Martinez NJ, Walhout AJ (abril de 2009). "La interacción entre factores de transcripción y microARN en redes reguladoras a escala del genoma". BioEssays . 31 (4): 435–445. doi :10.1002/bies.200800212. PMC 3118512 . PMID 19274664.
- ^ Tomilin NV (abril de 2008). "Regulación de la expresión génica en mamíferos por retroelementos y repeticiones en tándem no codificantes". BioEssays . 30 (4): 338–348. doi : 10.1002/bies.20741 . PMID 18348251.
- ^ Lee TI, Young RA (marzo de 2013). "Regulación transcripcional y su desregulación en la enfermedad". Cell . 152 (6): 1237–1251. doi :10.1016/j.cell.2013.02.014. PMC 3640494 . PMID 23498934.
- ^ O'Connor L, Gilmour J, Bonifer C (diciembre de 2016). "El papel del factor de transcripción Sp1 expresado de forma ubicua en la regulación transcripcional específica de tejidos y en la enfermedad". The Yale Journal of Biology and Medicine . 89 (4): 513–525. PMC 5168829 . PMID 28018142.
- ^ Yesudhas D, Batool M, Anwar MA, Panneerselvam S, Choi S (agosto de 2017). "Proteínas que reconocen el ADN: singularidad estructural y versatilidad de los dominios de unión al ADN en los factores de transcripción de células madre". Genes . 8 (8): 192. doi : 10.3390/genes8080192 . PMC 5575656 . PMID 28763006.
- ^ Wang G, Wang F, Huang Q, Li Y, Liu Y, Wang Y (2015). "Comprensión de la regulación de los factores de transcripción mediante la integración de la expresión génica y los sitios hipersensibles a la DNasa I". BioMed Research International . 2015 : 757530. doi : 10.1155/2015/757530 . PMC 4573618. PMID 26425553 .
- ^ Kolovos P, Knoch TA, Grosveld FG, Cook PR, Papantonis A (enero de 2012). "Potenciadores y silenciadores: un modelo integrado y simple para su función". Epigenética y cromatina . 5 (1): 1. doi : 10.1186/1756-8935-5-1 . PMC 3281776 . PMID 22230046.
- ^ Fuda NJ, Ardehali MB, Lis JT (septiembre de 2009). "Definición de los mecanismos que regulan la transcripción de la ARN polimerasa II in vivo". Nature . 461 (7261): 186–192. Bibcode :2009Natur.461..186F. doi :10.1038/nature08449. PMC 2833331 . PMID 19741698.
- ^ Filtz TM, Vogel WK, Leid M (febrero de 2014). "Regulación de la actividad de los factores de transcripción mediante modificaciones postraduccionales interconectadas". Tendencias en ciencias farmacológicas . 35 (2): 76–85. doi :10.1016/j.tips.2013.11.005. PMC 3954851 . PMID 24388790.
- ^ Hampsey M (junio de 1998). "Genética molecular de la maquinaria transcripcional general de la ARN polimerasa II". Microbiology and Molecular Biology Reviews . 62 (2): 465–503. doi :10.1128/MMBR.62.2.465-503.1998. PMC 98922 . PMID 9618449.
- ^ Veitia RA (noviembre de 2008). "Mil y una maneras de crear potenciadores transcripcionales funcionalmente similares". BioEssays . 30 (11–12): 1052–1057. doi :10.1002/bies.20849. PMID 18937349.
- ^ Nguyen T, Nioi P, Pickett CB (mayo de 2009). "La vía de señalización del elemento de respuesta antioxidante Nrf2 y su activación por estrés oxidativo". The Journal of Biological Chemistry . 284 (20): 13291–13295. doi : 10.1074/jbc.R900010200 . PMC 2679427 . PMID 19182219.
- ^ Paul S (noviembre de 2008). "Disfunción del sistema ubiquitina-proteasoma en múltiples enfermedades: enfoques terapéuticos". BioEssays . 30 (11–12): 1172–1184. doi :10.1002/bies.20852. PMID 18937370. S2CID 29422790.
- ^ Los M, Maddika S, Erb B, Schulze-Osthoff K (mayo de 2009). "Cambio de Akt: de la señalización de supervivencia a la respuesta mortal". BioEssays . 31 (5): 492–495. doi :10.1002/bies.200900005. PMC 2954189 . PMID 19319914.
- ^ Afek A, Sela I, Musa-Lempel N, Lukatsky DB (noviembre de 2011). "La unión inespecífica del factor de transcripción y del ADN influye en la ocupación de nucleosomas en la levadura". Revista Biofísica . 101 (10): 2465–2475. arXiv : 1111.4779 . Código Bib : 2011BpJ...101.2465A. doi :10.1016/j.bpj.2011.10.012. PMC 3218343 . PMID 22098745.
- ^ Moosavi A, Motevalizadeh Ardekani A (noviembre de 2016). "El papel de la epigenética en la biología y las enfermedades humanas". Revista biomédica iraní . 20 (5): 246–258. doi :10.22045/ibj.2016.01. PMC 5075137 . PMID 27377127.
- ^ Al Aboud NM, Tupper C, Jialal I (2024). "Genética, mecanismo epigenético". StatPearls . Treasure Island (FL): StatPearls Publishing. PMID 30422591 . Consultado el 12 de junio de 2024 .
- ^ Miller JL, Grant PA (2013). "El papel de la metilación del ADN y las modificaciones de las histonas en la regulación transcripcional en humanos". Epigenética: desarrollo y enfermedad . Bioquímica subcelular. Vol. 61. págs. 289–317. doi :10.1007/978-94-007-4525-4_13. ISBN . 978-94-007-4524-7. PMC 6611551 . PMID 23150256.
- ^ Verheul TC, van Hijfte L, Perenthaler E, Barakat TS (2020). "El porqué de YY1: mecanismos de regulación transcripcional por Yin Yang 1". Frontiers in Cell and Developmental Biology . 8 : 592164. doi : 10.3389/fcell.2020.592164 . PMC 7554316 . PMID 33102493.
- ^ Spitz F, Furlong EE (septiembre de 2012). "Factores de transcripción: desde la unión de potenciadores hasta el control del desarrollo". Nature Reviews. Genetics . 13 (9): 613–626. doi :10.1038/nrg3207. PMID 22868264. S2CID 205485256.
- ^ ab Schoenfelder S, Fraser P (agosto de 2019). "Contactos promotor-potenciador de largo alcance en el control de la expresión génica". Nature Reviews. Genética . 20 (8): 437–455. doi :10.1038/s41576-019-0128-0. PMID 31086298. S2CID 152283312.
- ^ Weintraub AS, Li CH, Zamudio AV, Sigova AA, Hannett NM, Day DS, et al. (diciembre de 2017). "YY1 es un regulador estructural de los bucles potenciador-promotor". Cell . 171 (7): 1573–1588.e28. doi :10.1016/j.cell.2017.11.008. PMC 5785279 . PMID 29224777.
- ^ Lambert SA, Jolma A, Campitelli LF, Das PK, Yin Y, Albu M, et al. (febrero de 2018). "Los factores de transcripción humanos". Cell . 172 (4): 650–665. doi : 10.1016/j.cell.2018.01.029 . PMID 29425488.
- ^ Grossman SR, Engreitz J, Ray JP, Nguyen TH, Hacohen N, Lander ES (julio de 2018). "Especificidad posicional de diferentes clases de factores de transcripción dentro de potenciadores". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 115 (30): E7222–E7230. Bibcode :2018PNAS..115E7222G. doi : 10.1073/pnas.1804663115 . PMC 6065035 . PMID 29987030.
- ^ Allen BL, Taatjes DJ (marzo de 2015). "El complejo mediador: un integrador central de la transcripción". Nature Reviews. Molecular Cell Biology . 16 (3): 155–166. doi :10.1038/nrm3951. PMC 4963239 . PMID 25693131.
- ^ Mikhaylichenko O, Bondarenko V, Harnett D, Schor IE, Males M, Viales RR, et al. (enero de 2018). "El grado de actividad potenciadora o promotora se refleja en los niveles y la direccionalidad de la transcripción del ARNm". Genes & Development . 32 (1): 42–57. doi :10.1101/gad.308619.117. PMC 5828394 . PMID 29378788.
- ^ Li QJ, Yang SH, Maeda Y, Sladek FM, Sharrocks AD, Martins-Green M (enero de 2003). "La activación dependiente de la fosforilación de la quinasa MAP de Elk-1 conduce a la activación del coactivador p300". The EMBO Journal . 22 (2): 281–291. doi :10.1093/emboj/cdg028. PMC 140103 . PMID 12514134.
- ^ Carullo NV, Phillips Iii RA, Simon RC, Soto SA, Hinds JE, Salisbury AJ, et al. (septiembre de 2020). "Los ARN potenciadores predicen los vínculos reguladores entre genes potenciadores y son fundamentales para la función potenciadora en los sistemas neuronales". Nucleic Acids Research . 48 (17): 9550–9570. doi :10.1093/nar/gkaa671. PMC 7515708 . PMID 32810208.
- ^ Lövkvist C, Dodd IB, Sneppen K, Haerter JO (junio de 2016). "La metilación del ADN en los epigenomas humanos depende de la topología local de los sitios CpG". Nucleic Acids Research . 44 (11): 5123–5132. doi :10.1093/nar/gkw124. PMC 4914085 . PMID 26932361.
- ^ Jabbari K, Bernardi G (mayo de 2004). "Metilación de citosina y frecuencias de CpG, TpG (CpA) y TpA". Gene . 333 : 143–149. doi :10.1016/j.gene.2004.02.043. PMID 15177689.
- ^ Weber M, Hellmann I, Stadler MB, Ramos L, Pääbo S, Rebhan M, et al. (abril de 2007). "Distribución, potencial de silenciamiento e impacto evolutivo de la metilación del ADN del promotor en el genoma humano". Nature Genetics . 39 (4): 457–466. doi :10.1038/ng1990. PMID 17334365. S2CID 22446734.
- ^ Yang X, Han H, De Carvalho DD, Lay FD, Jones PA, Liang G (octubre de 2014). "La metilación del cuerpo del gen puede alterar la expresión génica y es un objetivo terapéutico en el cáncer". Cancer Cell . 26 (4): 577–590. doi :10.1016/j.ccr.2014.07.028. PMC 4224113 . PMID 25263941.
- ^ Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, et al. (diciembre de 2013). "Desmetilación de ADN dirigida y activación de genes endógenos utilizando proteínas de fusión TALE-TET1 programables". Nature Biotechnology . 31 (12): 1137–1142. doi :10.1038/nbt.2726. PMC 3858462 . PMID 24108092.
- ^ Kim JJ, Jung MW (2006). "Circuitos neuronales y mecanismos implicados en el condicionamiento pavloviano del miedo: una revisión crítica". Neuroscience and Biobehavioral Reviews . 30 (2): 188–202. doi :10.1016/j.neubiorev.2005.06.005. PMC 4342048 . PMID 16120461.
- ^ ab Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (julio de 2017). "Reorganización epigenómica dependiente de la experiencia en el hipocampo". Aprendizaje y memoria . 24 (7): 278–288. doi :10.1101/lm.045112.117. PMC 5473107 . PMID 28620075.
- ^ ab Bernstein C (2022). "Metilación del ADN y establecimiento de la memoria". Epigenetics Insights . 15 : 25168657211072499. doi :10.1177/25168657211072499. PMC 8793415 . PMID 35098021.
- ^ ab Keifer J (febrero de 2017). "El momento cumbre de los genes del aprendizaje". Genes . 8 (2): 69. doi : 10.3390/genes8020069 . PMC 5333058 . PMID 28208656.
- ^ Saxonov S, Berg P, Brutlag DL (enero de 2006). "Un análisis de todo el genoma de los dinucleótidos CpG en el genoma humano distingue dos clases distintas de promotores". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (5): 1412–1417. Bibcode :2006PNAS..103.1412S. doi : 10.1073/pnas.0510310103 . PMC 1345710 . PMID 16432200.
- ^ Bird A (enero de 2002). "Patrones de metilación del ADN y memoria epigenética". Genes & Development . 16 (1): 6–21. doi : 10.1101/gad.947102 . PMID 11782440.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (marzo de 2013). "Paisajes del genoma del cáncer". Science . 339 (6127): 1546–1558. Bibcode :2013Sci...339.1546V. doi :10.1126/science.1235122. PMC 3749880 . PMID 23539594.
- ^ Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, et al. (2014). "MicroARN en la red de reparación/daño del ADN y el cáncer". Revista Internacional de Genómica . 2014 : 820248. doi : 10.1155/2014/820248 . PMC 3926391 . PMID 24616890.
- ^ De Magistris P (octubre de 2021). "El gran escape: exportación de ARNm a través del complejo de poros nucleares". Revista internacional de ciencias moleculares . 22 (21): 11767. doi : 10.3390/ijms222111767 . PMC 8583845 . PMID 34769195.
- ^ Liu H, Luo M, Wen JK (mayo de 2014). "Estabilidad del ARNm en el núcleo". Revista de la Universidad de Zhejiang. Science. B . 15 (5): 444–454. doi :10.1631/jzus.B1400088. PMC 4076601 . PMID 24793762.
- ^ Yan LL, Zaher HS (octubre de 2019). "¿Cómo afrontan las células el daño del ARN y sus consecuencias?". The Journal of Biological Chemistry . 294 (41): 15158–15171. doi : 10.1074/jbc.REV119.006513 . PMC 6791314 . PMID 31439666.
- ^ Passmore LA, Coller J (febrero de 2022). "Funciones de las colas de poli(A) del ARNm en la regulación de la expresión génica eucariota". Nature Reviews. Molecular Cell Biology . 23 (2): 93–106. doi :10.1038/s41580-021-00417-y. PMC 7614307 . PMID 34594027.
- ^ Shehata SI, Watkins JM, Burke JM, Parker R (febrero de 2024). "Mecanismos y consecuencias de la desestabilización del ARNm durante las infecciones virales". Virology Journal . 21 (1): 38. doi : 10.1186/s12985-024-02305-1 . PMC 10848536 . PMID 38321453.
- ^ Tang L, Chen HY, Hao NB, Tang B, Guo H, Yong X, et al. (octubre de 2017). "Inhibidores de microARN: secuestro natural y artificial de microARN". Cancer Letters . 407 : 139–147. doi :10.1016/B978-0-444-64046-8.00282-2. ISBN 978-0-444-64047-5. PMC 7152241 . PMID 28602827.
- ^ Mayr C (octubre de 2019). "¿Qué están haciendo los 3' UTR?". Cold Spring Harbor Perspectives in Biology . 11 (10): a034728. doi :10.1101/cshperspect.a034728. PMC 6771366 . PMID 30181377.
- ^ O'Brien J, Hayder H, Zayed Y, Peng C (3 de agosto de 2018). "Descripción general de la biogénesis de microARN, mecanismos de acción y circulación". Frontiers in Endocrinology . 9 : 402. doi : 10.3389/fendo.2018.00402 . PMC 6085463 . PMID 30123182.
- ^ Mayya VK, Duchaine TF (24 de enero de 2019). "Cifras y verdugos: cómo las regiones 3' no traducidas determinan el destino de los ARN mensajeros". Frontiers in Genetics . 10 : 6. doi : 10.3389/fgene.2019.00006 . PMC 6357968 . PMID 30740123.
- ^ Nair AA, Tang X, Thompson KJ, Vedell PT, Kalari KR, Subramanian S (junio de 2020). "La frecuencia de los elementos de respuesta de microARN identifica vías de señalización patológicamente relevantes en el cáncer de mama triple negativo". iScience . 23 (6): 101249. Bibcode :2020iSci...23j1249N. doi :10.1016/j.isci.2020.101249. PMC 7322352 . PMID 32629614.
- ^ miRBase.org
- ^ ab Friedman RC, Farh KK, Burge CB, Bartel DP (enero de 2009). "La mayoría de los ARNm de mamíferos son dianas conservadas de los microARN". Genome Research . 19 (1): 92–105. doi :10.1101/gr.082701.108. PMC 2612969 . PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, et al. (febrero de 2005). "El análisis de microarrays muestra que algunos microARN regulan negativamente un gran número de ARNm objetivo". Nature . 433 (7027): 769–773. Bibcode :2005Natur.433..769L. doi :10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (septiembre de 2008). "Cambios generalizados en la síntesis de proteínas inducida por microARN". Nature . 455 (7209): 58–63. Bibcode :2008Natur.455...58S. doi :10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (septiembre de 2008). "El impacto de los microARN en la producción de proteínas". Nature . 455 (7209): 64–71. Bibcode :2008Natur.455...64B. doi :10.1038/nature07242. PMC 2745094 . PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, et al. (Julio de 2011). "Mecanismos y papel de la desregulación de microARN en la aparición y progresión del cáncer". Genética y Biología Molecular . 34 (3): 363–370. doi :10.1590/S1415-47572011000300001. PMC 3168173 . PMID 21931505.
- ^ Bernstein C, Bernstein H (mayo de 2015). "Reducción epigenética de la reparación del ADN en la progresión del cáncer gastrointestinal". Revista mundial de oncología gastrointestinal . 7 (5): 30–46. doi : 10.4251/wjgo.v7.i5.30 . PMC 4434036 . PMID 25987950.
- ^ Mellios N, Sur M (2012). "El papel emergente de los microARN en la esquizofrenia y los trastornos del espectro autista". Frontiers in Psychiatry . 3 : 39. doi : 10.3389/fpsyt.2012.00039 . PMC 3336189 . PMID 22539927.
- ^ Geaghan M, Cairns MJ (agosto de 2015). "MicroARN y desregulación postranscripcional en psiquiatría". Psiquiatría biológica . 78 (4): 231–239. doi : 10.1016/j.biopsych.2014.12.009 . hdl : 1959.13/1335073 . PMID 25636176.
- ^ Sonenberg N, Hinnebusch AG (febrero de 2009). "Regulación de la iniciación de la traducción en eucariotas: mecanismos y objetivos biológicos". Cell . 136 (4): 731–745. doi :10.1016/j.cell.2009.01.042. PMC 3610329 . PMID 19239892.
- ^ Jurėnas D, Van Melderen L (17 de abril de 2020). "La variedad en el tema común de inhibición de la traducción por sistemas de toxina-antitoxina de tipo II". Frontiers in Genetics . 11 : 262. doi : 10.3389/fgene.2020.00262 . PMC 7180214 . PMID 32362907.
- ^ Dmitriev SE, Vladimirov DO, Lashkevich KA (noviembre de 2020). "Una guía rápida sobre inhibidores de moléculas pequeñas de la síntesis de proteínas eucariotas". Bioquímica. Biokhimiia . 85 (11): 1389–1421. doi :10.1134/S0006297920110097. PMC 7689648 . PMID 33280581.
- ^ abc Walsh CT, Garneau-Tsodikova S, Gatto GJ (diciembre de 2005). "Modificaciones postraduccionales de proteínas: la química de las diversificaciones del proteoma". Angewandte Chemie . 44 (45): 7342–7372. doi :10.1002/anie.200501023. PMID 16267872. S2CID 32157563.
- ^ Khoury GA, Baliban RC, Floudas CA (septiembre de 2011). "Estadísticas de modificación postraduccional en todo el proteoma: análisis de frecuencia y conservación de la base de datos swiss-prot". Scientific Reports . 1 (90): 90. Bibcode :2011NatSR...1E..90K. doi :10.1038/srep00090. PMC 3201773 . PMID 22034591.
- ^ Mann M, Jensen ON (marzo de 2003). "Análisis proteómico de modificaciones postraduccionales". Nature Biotechnology . 21 (3): 255–261. doi :10.1038/nbt0303-255. PMID 12610572. S2CID 205266061.
- ^ Seo J, Lee KJ (enero de 2004). "Modificaciones postraduccionales y sus funciones biológicas: análisis proteómico y enfoques sistemáticos". Revista de bioquímica y biología molecular . 37 (1): 35–44. doi : 10.5483/bmbrep.2004.37.1.035 . PMID 14761301.
- ^ Rogers LD, General CM (diciembre de 2013). "Modificación postraduccional proteolítica de proteínas: herramientas proteómicas y metodología". Molecular & Cellular Proteomics . 12 (12): 3532–3542. doi : 10.1074/mcp.M113.031310 . PMC 3861706 . PMID 23887885.
- ^ Ahmad W, Gull B, Baby J, Mustafa F (junio de 2021). "Un análisis exhaustivo de los ensayos de hibridación Northern frente a hibridación líquida para ARNm, ARN pequeños y miARN utilizando un enfoque no radiomarcado". Temas actuales en biología molecular . 43 (2): 457–484. doi : 10.3390/cimb43020036 . PMC 8929067 . PMID 34206608.
- ^ Hoy MA (2013). "Capítulo 6 - Algunas herramientas adicionales para el biólogo molecular". Genética molecular de insectos (3.ª ed.). Academic Press . págs. 215–249. doi :10.1016/C2011-0-69839-4. ISBN 978-0-12-415874-0.
- ^ Petrov A, Tsa A, Puglisi JD (2013). "Análisis de ARN mediante electroforesis analítica en gel de poliacrilamida". Métodos de laboratorio en enzimología: ARN . Vol. 530. págs. 301–313. doi :10.1016/B978-0-12-420037-1.00016-6. ISBN 978-0-12-420037-1. ISSN 1557-7988. PMID 24034328.
- ^ Boyle A, Perry-O'Keefe H (mayo de 2001). "Etiquetado y detección colorimétrica de sondas no isotópicas". Protocolos actuales en biología molecular . 3 (suplemento 20): Unidad 3.18. doi : 10.1002/0471142727.mb0318s20. PMID 18265226.
- ^ Kuang J, Yan X, Genders AJ, Granata C, Bishop DJ (10 de mayo de 2018). "Una descripción general de las consideraciones técnicas al utilizar el análisis de PCR cuantitativo en tiempo real de la expresión génica en la investigación del ejercicio humano". PLOS ONE . 13 (5): e0196438. Bibcode :2018PLoSO..1396438K. doi : 10.1371/journal.pone.0196438 . PMC 5944930 . PMID 29746477.
- ^ Yang T, Zhang M, Zhang N (enero de 2022). "Protocolo de transferencia Northern modificado para la detección sencilla de ARNm en ARN total mediante sondas radiomarcadas". BMC Genomics . 23 (1): 66. doi : 10.1186/s12864-021-08275-w . PMC 8772191 . PMID 35057752.
- ^ Wesierska-Gadek J, Wang ZQ, Schmid G (enero de 1999). "Estabilidad reducida de la proteína p53 empalmada regularmente pero no empalmada alternativamente en fibroblastos de ratón deficientes en PARP". Cancer Research . 59 (1): 28–34. PMID 9892179.
- ^ Padhi BK, Singh M, Rosales M, Pelletier G, Cakmak S (mayo de 2018). "Un ensayo cuantitativo basado en PCR para la evaluación de la integridad del ARNm en muestras de ratas". Detección y cuantificación biomolecular . 15 : 18–23. doi :10.1016/j.bdq.2018.02.001. PMC 6006387 . PMID 29922590.
- ^ Kwon YM, Ricke SC (22 de marzo de 2011). Secuenciación de próxima generación de alto rendimiento. Métodos en biología molecular. Vol. 733 (1.ª ed.). Springer Science+Business Media . doi :10.1007/978-1-61779-089-8. eISSN 1940-6029. ISBN 978-1-61779-089-8. ISSN 1064-3745.
- ^ Rahmasari R, Raekiansyah M, Azallea SN, Nethania M, Bilqisthy N, Rozaliyani A, et al. (noviembre de 2022). "Ensayo RT-qPCR basado en SYBR Green de bajo costo para detectar SARS-CoV-2 en un entorno indonesio utilizando cebadores recomendados por la OMS". Heliyon . 8 (11): e11130. Bibcode :2022Heliy...811130R. doi : 10.1016/j.heliyon.2022.e11130 . PMC 9617658 . PMID 36339747.
- ^ Liang M, Cowley AW, Greene AS (enero de 2004). "Perfiles de expresión génica de alto rendimiento: un enfoque molecular para la fisiología integradora". The Journal of Physiology . 554 (Pt 1): 22–30. doi :10.1113/jphysiol.2003.049395. PMC 1664740 . PMID 14678487.
- ^ Bumgarner R (enero de 2013). "Descripción general de los microarrays de ADN: tipos, aplicaciones y su futuro". Protocolos actuales en biología molecular . Capítulo 22: Unidad 22.1. doi :10.1002/0471142727.mb2201s101. PMC 4011503. PMID 23288464 .
- ^ Byron SA, Van Keuren-Jensen KR, Engelthaler DM, Carpten JD, Craig DW (mayo de 2016). "Traducción de la secuenciación de ARN en diagnósticos clínicos: oportunidades y desafíos". Nature Reviews. Genética . 17 (5): 257–271. doi :10.1038/nrg.2016.10. PMC 7097555. PMID 26996076 .
- ^ Meyers BC, Galbraith DW, Nelson T, Agrawal V (junio de 2004). "Métodos para el perfilado transcripcional en plantas. Sé fructífero y replícate". Fisiología vegetal . 135 (2): 637–652. doi :10.1104/pp.104.040840. PMC 514100 . PMID 15173570.
- ^ Kukurba KR, Montgomery SB (abril de 2015). "Secuenciación y análisis de ARN". Protocolos de Cold Spring Harbor . 2015 (11): 951–969. doi :10.1101/pdb.top084970. PMC 4863231. PMID 25870306 .
- ^ "Resultados de la búsqueda < Atlas de expresiones < EMBL-EBI". www.ebi.ac.uk .
- ^ Moritz CP (febrero de 2020). "40 años de Western blotting: un brindis de cumpleaños científico". Journal of Proteomics . 212 : 103575. doi : 10.1016/j.jprot.2019.103575 . PMID 31706026.
- ^ Burkhart JM, Vaudel M, Gambaryan S, Radau S, Walter U, Martens L, et al. (octubre de 2012). "El primer análisis exhaustivo y cuantitativo de la composición proteica de las plaquetas humanas permite el análisis comparativo de las vías estructurales y funcionales". Blood . 120 (15): e73–e82. doi : 10.1182/blood-2012-04-416594 . PMID 22869793.
- ^ Moritz CP, Mühlhaus T, Tenzer S, Schulenborg T, Friauf E (junio de 2019). "Correlación deficiente entre transcripción y proteína en el cerebro: la correlación negativa de productos genéticos revela la polaridad neuronal como una posible causa". Journal of Neurochemistry . 149 (5): 582–604. doi :10.1111/jnc.14664. PMID 30664243. S2CID 58667771.
- ^ Banf M, Rhee SY (enero de 2017). "Inferencia computacional de redes reguladoras de genes: enfoques, limitaciones y oportunidades". Biochimica et Biophysica Acta (BBA) - Mecanismos reguladores de genes . 1860 (1): 41–52. doi : 10.1016/j.bbagrm.2016.09.003 . PMID 27641093.
- ^ Chesler EJ, Lu L, Wang J, Williams RW, Manly KF (mayo de 2004). "WebQTL: análisis exploratorio rápido de la expresión génica y las redes genéticas para el cerebro y el comportamiento". Nature Neuroscience . 7 (5): 485–486. doi :10.1038/nn0504-485. PMID 15114364. S2CID 20241963.
- ^ Song Y, Wang W, Qu X, Sun S (febrero de 2009). "Efectos del factor inducible por hipoxia-1 alfa (HIF-1 alfa) en el crecimiento y la adhesión de las células del carcinoma de células escamosas de la lengua". The Indian Journal of Medical Research . 129 (2): 154–163. PMID 19293442.
- ^ Hanriot L, Keime C, Gay N, Faure C, Dossat C, Wincker P, et al. (septiembre de 2008). "Una combinación de LongSAGE con secuenciación Solexa es adecuada para explorar la profundidad y la complejidad del transcriptoma". BMC Genomics . 9 : 418. doi : 10.1186/1471-2164-9-418 . PMC 2562395 . PMID 18796152.
- ^ Wheelan SJ, Martínez Murillo F, Boeke JD (julio de 2008). "El increíble mundo cada vez más pequeño de los microarrays de ADN". Molecular BioSystems . 4 (7): 726–732. doi :10.1039/b706237k. PMC 2535915 . PMID 18563246.
- ^ Miyakoshi M, Nishida H, Shintani M, Yamane H, Nojiri H (enero de 2009). "Mapeo de alta resolución de transcriptomas de plásmidos en diferentes bacterias hospedadoras". BMC Genomics . 10 : 12. doi : 10.1186/1471-2164-10-12 . PMC 2642839 . PMID 19134166.
- ^ Denoeud F, Aury JM, Da Silva C, Noel B, Rogier O, Delledonne M, et al. (2008). "Anotación de genomas con secuenciación de ARN a gran escala". Genome Biology . 9 (12): R175. doi : 10.1186/gb-2008-9-12-r175 . PMC 2646279 . PMID 19087247.
- ^ Clough E, Barrett T (2016). "La base de datos ómnibus de expresión genética". Genómica estadística . Métodos en biología molecular. Vol. 1418. págs. 93-110. doi :10.1007/978-1-4939-3578-9_5. ISBN 978-1-4939-3576-5. PMC 4944384 . PMID 27008011.
- ^ Kiliç S, White ER, Sagitova DM, Cornish JP, Erill I (enero de 2014). "CollecTF: una base de datos de sitios de unión de factores de transcripción validados experimentalmente en bacterias". Nucleic Acids Research . 42 (número de la base de datos): D156–D160. doi :10.1093/nar/gkt1123. PMC 3965012 . PMID 24234444.
- ^ Moretto M, Sonego P, Dierckxsens N, Brilli M, Bianco L, Ledezma-Tejeida D, et al. (Enero de 2016). "COLOMBOS v3.0: aprovechamiento de compendios de expresión génica para análisis entre especies". Investigación de ácidos nucleicos . 44 (D1): D620–D623. doi : 10.1093/nar/gkv1251. PMC 4702885 . PMID 26586805.
- ^ Faith JJ, Driscoll ME, Fusaro VA, Cosgrove EJ, Hayete B, Juhn FS, et al. (enero de 2008). "Base de datos de microarrays de muchos microbios: compendios de Affymetrix normalizados de manera uniforme con metadatos experimentales estructurados". Nucleic Acids Research . 36 (número de la base de datos): D866–D870. doi :10.1093/nar/gkm815. PMC 2238822 . PMID 17932051.
Enlaces externos
- Base de datos de factores de transcripción de plantas y plataforma de análisis y datos de regulación transcripcional de plantas

