Exón

Un exón es cualquier parte de un gen que formará parte del ARN maduro final producido por ese gen después de que se hayan eliminado los intrones mediante el empalme de ARN . El término exón se refiere tanto a la secuencia de ADN dentro de un gen como a la secuencia correspondiente en las transcripciones de ARN. En el empalme de ARN, se eliminan los intrones y los exones se unen covalentemente entre sí como parte de la generación del ARN maduro . Así como el conjunto completo de genes de una especie constituye el genoma , el conjunto completo de exones constituye el exoma .
Historia
El término exón deriva de la región expresada y fue acuñado por el bioquímico estadounidense Walter Gilbert en 1978: "La noción de cistrón ... debe ser reemplazada por la de una unidad de transcripción que contiene regiones que se perderán del mensajero maduro –que sugiero que llamemos intrones (para regiones intragénicas)– alternando con regiones que se expresarán –exones-". [1]
Esta definición se hizo originalmente para las transcripciones codificadoras de proteínas que se empalman antes de ser traducidas. El término luego llegó a incluir secuencias extraídas del ARNr [2] y ARNt [3] y otros ARNnc [4] y también se usó más tarde para las moléculas de ARN que se originan de diferentes partes del genoma y que luego se ligan mediante empalme trans. [5]
Contribución a los genomas y distribución del tamaño
Aunque los eucariotas unicelulares como la levadura no tienen intrones o tienen muy pocos, los metazoos y especialmente los genomas de vertebrados tienen una gran fracción de ADN no codificante . Por ejemplo, en el genoma humano solo el 1,1% del genoma está abarcado por exones, mientras que el 24% está en intrones, y el 75% del genoma es ADN intergénico . [6] Esto puede proporcionar una ventaja práctica en la atención médica asistida por ómicas (como la medicina de precisión ) porque hace que la secuenciación comercializada del exoma completo sea un desafío menor y menos costoso que la secuenciación comercializada del genoma completo . La gran variación en el tamaño del genoma y el valor C en las formas de vida ha planteado un desafío interesante llamado el enigma del valor C.
En todos los genes eucariotas del GenBank, había (en 2002), en promedio, 5,48 exones por gen codificador de proteínas. El exón promedio codificaba entre 30 y 36 aminoácidos . [7] Si bien el exón más largo del genoma humano tiene una longitud de 11555 pb , se ha descubierto que varios exones tienen una longitud de solo 2 pb. [8] Se ha informado de un exón de un solo nucleótido en el genoma de Arabidopsis . [9] En los humanos, al igual que el ARNm codificador de proteínas , la mayoría del ARN no codificante también contiene múltiples exones [10]
Estructura y función
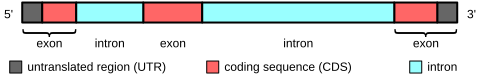
En los genes codificadores de proteínas, los exones incluyen tanto la secuencia codificante de la proteína como las regiones no traducidas (UTR) 5′ y 3′. A menudo, el primer exón incluye tanto la UTR 5′ como la primera parte de la secuencia codificante, pero en algunos genes se encuentran exones que contienen solo regiones de la UTR 5′ o (más raramente) de la UTR 3′, es decir, las UTR pueden contener intrones. [11] Algunas transcripciones de ARN no codificantes también tienen exones e intrones.
Los ARNm maduros que se originan del mismo gen no necesitan incluir los mismos exones, ya que diferentes intrones en el pre-ARNm pueden eliminarse mediante el proceso de empalme alternativo .
La exonización es la creación de un nuevo exón, como resultado de mutaciones en los intrones . [12]
Enfoques experimentales utilizando exones
La captura de exones o " captura de genes " es una técnica de biología molecular que explota la existencia del empalme intrón-exón para encontrar nuevos genes. [13] El primer exón de un gen "atrapado" se empalma en el exón que está contenido en el ADN de inserción. Este nuevo exón contiene el ORF para un gen reportero que ahora puede expresarse utilizando los potenciadores que controlan el gen objetivo. Un científico sabe que un nuevo gen ha sido atrapado cuando se expresa el gen reportero.
El empalme se puede modificar experimentalmente para que los exones objetivo se excluyan de las transcripciones de ARNm maduro al bloquear el acceso de partículas de ribonucleoproteína nuclear pequeña (snRNP) que dirigen el empalme al pre-ARNm utilizando oligos antisentido de morfolino . [14] Esto se ha convertido en una técnica estándar en biología del desarrollo . Los oligos de morfolino también se pueden dirigir para evitar que las moléculas que regulan el empalme (por ejemplo, potenciadores de empalme, supresores de empalme) se unan al pre-ARNm, alterando los patrones de empalme.
Mal uso común del término
Los usos incorrectos más comunes del término exón son que "los exones codifican proteínas", o "los exones codifican aminoácidos" o "los exones se traducen". Sin embargo, este tipo de definiciones solo cubren los genes que codifican proteínas y omiten aquellos exones que se convierten en parte de un ARN no codificante [15] o la región no traducida de un ARNm . [16] [17] Estas definiciones incorrectas aún aparecen en fuentes secundarias generalmente confiables. [18] [19]
Véase también
- DBASS3/5
- Exitrón
- Base de datos de exones e intrones
- Reorganización de exones
- Gen interrumpido
- Outron
- Twintron (Twintron)
- Región no traducida (UTR)
Referencias
- ^ Gilbert W (febrero de 1978). "¿Por qué los genes en pedazos?". Nature . 271 (5645): 501. Bibcode :1978Natur.271..501G. doi : 10.1038/271501a0 . PMID 622185.
- ^ Kister KP, Eckert WA (marzo de 1987). "Caracterización de un intermediario auténtico en el proceso de autoempalme del ARN precursor ribosómico en macronúcleos de Tetrahymena thermophila". Nucleic Acids Research . 15 (5): 1905–20. doi :10.1093/nar/15.5.1905. PMC 340607 . PMID 3645543.
- ^ Valenzuela P, Venegas A, Weinberg F, Bishop R, Rutter WJ (enero de 1978). "Estructura de los genes de fenilalanina-ARNt de levadura: un segmento de ADN intermedio dentro de la región que codifica para el ARNt". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 75 (1): 190–4. Bibcode :1978PNAS...75..190V. doi : 10.1073/pnas.75.1.190 . PMC 411211 . PMID 343104.
- ^ Khan, MR; Wellinger, RJ; Laurent, B (agosto de 2021). "Explorando el empalme alternativo de ARN largos no codificantes". Tendencias en genética . 37 (8): 695–698. doi :10.1016/j.tig.2021.03.010. PMID 33892960. S2CID 233382870.
- ^ Liu AY, Van der Ploeg LH, Rijsewijk FA, Borst P (junio de 1983). "La unidad de transposición del gen 118 de la glucoproteína de superficie variante de Trypanosoma brucei. Presencia de elementos repetidos en su borde y ausencia de secuencias asociadas al promotor". Journal of Molecular Biology . 167 (1): 57–75. doi :10.1016/S0022-2836(83)80034-5. PMID 6306255.
- ^ Venter JC ; et al. (2000). "La secuencia del genoma humano". Science . 291 (5507): 1304–51. Bibcode :2001Sci...291.1304V. doi : 10.1126/science.1058040 . PMID 11181995.
- ^ Sakharkar M, Passetti F, de Souza JE, Long M, de Souza SJ (2002). "ExInt: una base de datos de exones e intrones". Nucleic Acids Res . 30 (1): 191–4. doi :10.1093/nar/30.1.191. PMC 99089 . PMID 11752290.
- ^ Sakharkar MK; Chow VT; Kangueane P. (2004). "Distribuciones de exones e intrones en el genoma humano". In Silico Biol . 4 (4): 387–93. PMID 15217358.
- ^ Guo Lei, Liu Chun-Ming (2015). "Un exón de un solo nucleótido encontrado en Arabidopsis". Scientific Reports . 5 : 18087. Bibcode :2015NatSR...518087G. doi :10.1038/srep18087. PMC 4674806 . PMID 26657562.
- ^ Derrien, T; Johnson, R; Bussotti, G; Tanzer, A; Djebali, S; Tilgner, H; Guernec, G; Martín, D; Merkel, A; Knowles, director general; Lagarde, J; Veeravalli, L; Ruán, X; Ruán, Y; Lassmann, T; Carninci, P; Marrón, JB; Lipovich, L; González, JM; Tomás, M; Davis, California; Shiekhattar, R; Gingeras, TR; Hubbard, TJ; Notredame, C; Grada, J; Guigó, R (septiembre de 2012). "El catálogo GENCODE v7 de ARN largos no codificantes humanos: análisis de su estructura, evolución y expresión genética". Investigación del genoma . 22 (9): 1775–89. doi :10.1101/gr.132159.111. Número de modelo : PMID 22955988 .
- ^ Bicknell, AA (diciembre de 2012). "Intrones en UTR: por qué deberíamos dejar de ignorarlos". BioEssays . 34 (12): 1025–1034. doi : 10.1002/bies.201200073 . PMID 23108796. S2CID 5808466.
- ^ Sorek R (octubre de 2007). "El nacimiento de nuevos exones: mecanismos y consecuencias evolutivas". ARN . 13 (10): 1603–8. doi :10.1261/rna.682507. PMC 1986822 . PMID 17709368.
- ^ Duyk G. M; Kim SW; Myers R. M; Cox D. R (1990). "Trampa de exones: una prueba genética para identificar secuencias transcritas candidatas en ADN genómico de mamíferos clonados". Actas de la Academia Nacional de Ciencias . 87 (22): 8995–8999. Bibcode :1990PNAS...87.8995D. doi : 10.1073/pnas.87.22.8995 . PMC 55087 . PMID 2247475.
- ^ Morcos PA (junio de 2007). "Lograr una alteración cuantificable y dirigida del empalme del ARNm con oligos de morfolino". Comunicaciones de investigación bioquímica y biofísica . 358 (2): 521–7. doi :10.1016/j.bbrc.2007.04.172. PMID 17493584.
- ^ Khan, MR; Wellinger, RJ; Laurent, B (agosto de 2021). "Explorando el empalme alternativo de ARN largos no codificantes". Tendencias en genética . 37 (8): 695–698. doi :10.1016/j.tig.2021.03.010. PMID 33892960. S2CID 233382870.
- ^ Lu, J; Williams, JA; Luke, J; Zhang, F; Chu, K; Kay, MA (enero de 2017). "Un intrón diseñado que contiene un exón no codificante en el extremo 5' mejora la expresión transgénica de vectores AAV recombinantes in vivo". Terapia génica humana . 28 (1): 125–134. doi :10.1089/hum.2016.140. PMC 5278795 . PMID 27903072.
- ^ Chung, BY; Simons, C; Firth, AE; Brown, CM; Hellens, RP (19 de mayo de 2006). "Efecto de los intrones 5'UTR en la expresión génica en Arabidopsis thaliana". BMC Genomics . 7 : 120. doi : 10.1186/1471-2164-7-120 . PMC 1482700 . PMID 16712733.
- ^ "Exón". Genome.gov . Archivado desde el original el 16 de marzo de 2023 . Consultado el 23 de marzo de 2023 .
- ^ "Exón". www.nature.com . Scitable. Archivado desde el original el 23 de marzo de 2023 . Consultado el 23 de marzo de 2023 .
Bibliografía
- Zhang MQ (mayo de 1998). "Características estadísticas de los exones humanos y sus regiones flanqueantes". Genética molecular humana . 7 (5): 919–32. doi : 10.1093/hmg/7.5.919 . PMID 9536098.
- Thanaraj TA, Robinson AJ (noviembre de 2000). "Predicción de los límites exactos de los exones". Brief. Bioinform . 1 (4): 343–56. doi : 10.1093/bib/1.4.343 . PMID 11465052.
Enlaces externos
- Creador de gráficos de exones e intrones
