Edición genética CRISPR
La edición genética CRISPR (CRISPR, pronunciado /ˈkrɪspər/ "crisper", se refiere a "repeticiones palindrómicas cortas agrupadas regularmente interespaciadas " ) es una técnica de ingeniería genética en biología molecular mediante la cual se pueden modificar los genomas de organismos vivos . Se basa en una versión simplificada del sistema de defensa antiviral bacteriano CRISPR-Cas9. Al introducir la nucleasa Cas9 en una célula en combinación con un ARN guía sintético (gRNA), se puede cortar el genoma de la célula en la ubicación deseada, lo que permite eliminar genes existentes y/o agregar otros nuevos in vivo . [1]
La técnica se considera de gran importancia en biotecnología y medicina , ya que permite editar genomas in vivo de forma muy precisa, barata y sencilla. Se puede utilizar en la creación de nuevos medicamentos, productos agrícolas y organismos modificados genéticamente , o como medio para controlar patógenos y plagas . También tiene posibilidades en el tratamiento de enfermedades genéticas hereditarias , así como enfermedades derivadas de mutaciones somáticas como el cáncer. Sin embargo, su uso en la modificación genética de la línea germinal humana es muy controvertido. El desarrollo de la técnica le valió a Jennifer Doudna y Emmanuelle Charpentier el Premio Nobel de Química en 2020. [2] [3] El tercer grupo de investigadores que compartió el Premio Kavli por el mismo descubrimiento, [4] dirigido por Virginijus Šikšnys , no recibió el premio Nobel. [5] [6] [7]
La nucleasa Cas9, que funciona como una tijera genética, abre ambas hebras de la secuencia de ADN objetivo para introducir la modificación mediante uno de dos métodos. Las mutaciones knock-in, facilitadas mediante la reparación dirigida por homología (HDR), son la vía tradicional de los enfoques de edición genómica dirigida. [1] Esto permite la introducción de daños y reparaciones del ADN dirigidos . La HDR emplea el uso de secuencias de ADN similares para impulsar la reparación de la rotura mediante la incorporación de ADN exógeno para que funcione como plantilla de reparación. [1] Este método se basa en la aparición periódica y aislada de daños en el ADN en el sitio objetivo para que comience la reparación. Las mutaciones knock-out causadas por CRISPR-Cas9 son el resultado de la reparación de la rotura de doble cadena mediante unión de extremos no homólogos (NHEJ) o unión de extremos mediada por POLQ/polimerasa theta (TMEJ). Estas vías de unión de extremos a menudo pueden dar lugar a deleciones o inserciones aleatorias en el sitio de reparación, lo que puede interrumpir o alterar la funcionalidad del gen. Por lo tanto, la ingeniería genómica mediante CRISPR-Cas9 brinda a los investigadores la capacidad de generar disrupciones genéticas aleatorias y dirigidas.
Si bien la edición genómica en células eucariotas ha sido posible mediante diversos métodos desde la década de 1980, los métodos empleados habían demostrado ser ineficientes y poco prácticos para implementar a gran escala. Con el descubrimiento de CRISPR y, específicamente, la molécula de nucleasa Cas9, se hizo posible una edición eficiente y altamente selectiva. Cas9 derivada de la especie bacteriana Streptococcus pyogenes ha facilitado la modificación genómica dirigida en células eucariotas al permitir un método confiable para crear una ruptura dirigida en una ubicación específica designada por las cadenas guía crRNA y tracrRNA. [8] La facilidad con la que los investigadores pueden insertar Cas9 y ARN molde para silenciar o causar mutaciones puntuales en loci específicos ha demostrado ser invaluable para el mapeo rápido y eficiente de modelos genómicos y procesos biológicos asociados con varios genes en una variedad de eucariotas. Se han desarrollado variantes recientemente diseñadas de la nucleasa Cas9 que reducen significativamente la actividad fuera del objetivo. [9]
Las técnicas de edición genómica CRISPR-Cas9 tienen muchas aplicaciones potenciales. El uso del complejo CRISPR-Cas9-gRNA para la edición genómica [10] fue elegido por la AAAS como el Avance del Año en 2015. [11] Se han planteado muchas preocupaciones bioéticas sobre la posibilidad de utilizar CRISPR para la edición de la línea germinal , especialmente en embriones humanos . [12] En 2023, el primer fármaco que utiliza la edición genética CRISPR, Casgevy , fue aprobado para su uso en el Reino Unido para curar la anemia de células falciformes y la beta talasemia . [13] [14] Casgevy fue aprobado para su uso en los Estados Unidos el 8 de diciembre de 2023 por la Administración de Alimentos y Medicamentos . [15]
Historia
Otros métodos
A principios de la década de 2000, los investigadores alemanes comenzaron a desarrollar nucleasas de dedo de zinc (ZFN), proteínas sintéticas cuyos dominios de unión al ADN les permiten crear roturas de doble cadena en el ADN en puntos específicos. Las ZFN tienen una mayor precisión y la ventaja de ser más pequeñas que Cas9, pero las ZFN no se utilizan tan comúnmente como los métodos basados en CRISPR. En 2010, las nucleasas sintéticas llamadas nucleasas efectoras similares a activadores de la transcripción (TALEN) proporcionaron una forma más fácil de dirigir una rotura de doble cadena a una ubicación específica en la cadena de ADN. Tanto las nucleasas de dedo de zinc como las TALEN requieren el diseño y la creación de una proteína personalizada para cada secuencia de ADN objetivo, que es un proceso mucho más difícil y que requiere más tiempo que el de diseñar ARN guía. Los CRISPR son mucho más fáciles de diseñar porque el proceso requiere sintetizar solo una secuencia corta de ARN, un procedimiento que ya se usa ampliamente para muchas otras técnicas de biología molecular (por ejemplo, la creación de cebadores de oligonucleótidos ). [16]
Mientras que métodos como la interferencia de ARN (RNAi) no suprimen por completo la función de los genes, CRISPR, ZFN y TALEN proporcionan una eliminación completa e irreversible de los genes . [17] CRISPR también puede dirigirse a varios sitios de ADN simultáneamente simplemente introduciendo diferentes gRNA. Además, los costos de emplear CRISPR son relativamente bajos. [17] [18] [19]
Descubrimiento
En 2005, Alexander Bolotin del Instituto Nacional de Investigación Agrícola de Francia (INRA) descubrió un locus CRISPR que contenía nuevos genes Cas, especialmente uno que codificaba una proteína grande conocida como Cas9. [20]
En 2006, Eugene Koonin del Centro Nacional de Información Biotecnológica de Estados Unidos (NIH) propuso una explicación de cómo CRISPR actúa como un sistema inmunológico bacteriano. [20]
En 2007, Philippe Horvath de Danisco France SAS demostró experimentalmente cómo los sistemas CRISPR son un sistema inmunológico adaptativo e integran nuevo ADN de fago en la matriz CRISPR, que es como luchan contra la siguiente ola de fagos atacantes. [20]
En 2012, el equipo de investigación dirigido por la Universidad de California, Berkeley, la profesora Jennifer Doudna y la profesora de la Universidad de Umea Emmanuelle Charpentier, fueron las primeras personas en identificar, divulgar y presentar una solicitud de patente para el sistema CRISPR-Cas9 necesario para editar ADN. [20] También publicaron su hallazgo de que CRISPR- Cas9 podría programarse con ARN para editar ADN genómico, ahora considerado uno de los descubrimientos más significativos en la historia de la biología .
Patentes y comercialización
En noviembre de 2013 [actualizar], SAGE Labs (parte del grupo Horizon Discovery ) tenía derechos exclusivos de una de esas empresas para producir y vender ratas modificadas genéticamente y derechos no exclusivos para modelos de ratón y conejo. [21] En 2015 [actualizar], Thermo Fisher Scientific había obtenido la licencia de propiedad intelectual de ToolGen para desarrollar kits de reactivos CRISPR. [22]
En diciembre de 2014 [actualizar], los derechos de patente de CRISPR fueron impugnados. Se formaron varias empresas para desarrollar medicamentos y herramientas de investigación relacionados. [23] A medida que las empresas aumentaron la financiación, surgieron dudas sobre si CRISPR podría monetizarse rápidamente. [24] En 2014, Feng Zhang del Broad Institute del MIT y Harvard y otras nueve personas recibieron la patente estadounidense número 8.697.359 [25] sobre el uso de la edición genética CRISPR-Cas9 en eucariotas. Aunque a Charpentier y Doudna (conocidas como CVC) se les atribuyó la concepción de CRISPR, el Broad Institute fue el primero en lograr una "reducción a la práctica" según los jueces de patentes Sally Gardner Lane, James T. Moore y Deborah Katz. [26]
El primer conjunto de patentes fue otorgado al equipo Broad en 2015, lo que llevó a los abogados del grupo CVC a solicitar el primer procedimiento de interferencia. [27] En febrero de 2017, la Oficina de Patentes de los EE. UU. dictó sentencia sobre un caso de interferencia de patentes presentado por la Universidad de California con respecto a las patentes emitidas al Instituto Broad , y encontró que las patentes de Broad, con reclamos que cubren la aplicación de CRISPR-Cas9 en células eucariotas, eran distintas de las invenciones reclamadas por la Universidad de California. [28] [29] [30]
Poco después, la Universidad de California presentó una apelación contra esta sentencia. [31] [32] En 2019 se abrió la segunda disputa por interferencia. Esto fue en respuesta a las solicitudes de patente presentadas por CVC que requerían que la junta de apelaciones determinara el inventor original de la tecnología. La USPTO falló en marzo de 2022 en contra de la UC, declarando que el Instituto Broad fue el primero en presentar la solicitud. La decisión afectó a muchos de los acuerdos de licencia para la tecnología de edición CRISPR que fue licenciada por UC Berkeley. UC manifestó su intención de apelar la decisión de la USPTO. [33]
Eventos recientes
En marzo de 2017, la Oficina Europea de Patentes (OEP) anunció su intención de permitir reivindicaciones para la edición de todo tipo de células al Instituto Max-Planck de Berlín, la Universidad de California y la Universidad de Viena, [34] [35] y en agosto de 2017, la OEP anunció su intención de permitir reivindicaciones CRISPR en una solicitud de patente que MilliporeSigma había presentado. [34] En agosto de 2017, [actualizar]la situación de las patentes en Europa era compleja, con MilliporeSigma, ToolGen, la Universidad de Vilnius y Harvard compitiendo por reivindicaciones, junto con la Universidad de California y Broad. [36]
En julio de 2018, el TJUE dictaminó que la edición genética de plantas era una subcategoría de los alimentos transgénicos y, por lo tanto, que la técnica CRISPR en adelante estaría regulada en la Unión Europea por sus normas y reglamentos para los OGM . [37]
En febrero de 2020, un ensayo realizado en Estados Unidos demostró que la edición genética mediante CRISPR es segura en tres pacientes con cáncer. [38]
En octubre de 2020, las investigadoras Emmanuelle Charpentier y Jennifer Doudna recibieron el Premio Nobel de Química por su trabajo en este campo. [39] [40] Hicieron historia al ser las primeras dos mujeres en compartir este premio sin un colaborador masculino. [41] [5]
En junio de 2021, el primer ensayo clínico a pequeña escala de edición genética CRISPR intravenosa en humanos concluye con resultados prometedores. [42] [43]
En septiembre de 2021, el primer alimento editado con CRISPR salió a la venta en Japón. Los tomates fueron modificados genéticamente para obtener una cantidad aproximadamente cinco veces mayor de la normal de GABA , un posible calmante [44] . [45] CRISPR se aplicó por primera vez en tomates en 2014. [46]
En diciembre de 2021, se informó que el primer animal marino/ marisco editado genéticamente con CRISPR y el segundo conjunto de alimentos editados con CRISPR salieron a la venta al público en Japón: dos peces, de los cuales una especie crece hasta el doble del tamaño de los especímenes naturales debido a la alteración de la leptina , que controla el apetito, y la otra crece hasta 1,2 del tamaño medio natural con la misma cantidad de alimento debido a la miostatina desactivada , que inhibe el crecimiento muscular . [47] [48] [49]
Un estudio de 2022 ha descubierto que saber más sobre los tomates CRISPR tuvo un fuerte efecto en las preferencias de los participantes. "Casi la mitad de los 32 participantes de Alemania que son científicos demostraron elecciones constantes, mientras que la mayoría mostró una mayor disposición a comprar tomates CRISPR, en su mayoría no científicos". [50] [51]
La Universidad de California en Berkeley anunció en mayo de 2021 su intención de subastar tokens no fungibles tanto de la patente de edición genética CRISPR como de la inmunoterapia contra el cáncer . Sin embargo, en este caso, la universidad conservaría la propiedad de las patentes. [52] [53] El 85 % de los fondos obtenidos mediante la venta de la colección denominada The Fourth Pillar se utilizarían para financiar la investigación. [54] [55] Se vendió en junio de 2022 por 22 Ether, lo que equivalía a unos 54.000 dólares estadounidenses en ese momento. [56]
En noviembre de 2023, la Agencia Reguladora de Medicamentos y Productos Sanitarios (MHRA) del Reino Unido se convirtió en la primera del mundo en aprobar el uso del primer fármaco basado en la edición genética CRISPR, Casgevy, para tratar la anemia falciforme y la beta talasemia . Casgevy, o exagamglogene autotemcel , actúa directamente sobre los genes de las células madre del interior de los huesos del paciente, haciendo que produzcan glóbulos rojos sanos. Este tratamiento evita así la necesidad de realizar transfusiones de sangre periódicas y costosas. [13] [14]
En diciembre de 2023, la FDA aprobó la primera terapia génica en los EE. UU. para tratar a pacientes con enfermedad de células falciformes (ECF). La FDA aprobó dos tratamientos emblemáticos, Casgevy y Lyfgenia, que representan las primeras terapias génicas basadas en células para el tratamiento de la ECF. [57]
Ingeniería genómica

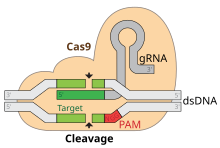
La edición genómica mediante CRISPR-Cas9 se lleva a cabo con un sistema CRISPR de tipo II . Cuando se utiliza para la edición genómica, este sistema incluye una ribonucleoproteína (RNP), que consta de Cas9 , crRNA y tracrRNA, junto con una plantilla de reparación de ADN opcional.

Componentes principales
| Componente | Función |
|---|---|
| ARNcr | Contiene el ARN guía que ubica el segmento correcto del ADN del huésped junto con una región que se une al tracrRNA (generalmente en forma de bucle de horquilla ), formando un complejo activo. |
| ARNtracr | Se une al crRNA y forma un complejo activo. |
| ARNsg | Los ARN guía únicos son un ARN combinado que consiste en un tracrRNA y al menos un crRNA . |
| Cas9 (más común) | Enzima cuya forma activa es capaz de modificar el ADN. Existen muchas variantes con diferentes funciones (es decir, corte de cadena simple, rotura de cadena doble, unión al ADN) debido a la función de reconocimiento del sitio de ADN de cada enzima. |
| Plantilla de reparación | Molécula de ADN utilizada como plantilla en el proceso de reparación del ADN de la célula huésped, permitiendo la inserción de una secuencia de ADN específica en el segmento huésped roto por Cas9. |
CRISPR-Cas9 a menudo emplea plásmidos , que codifican los componentes de RNP, para transfectar las células objetivo, o el RNP se ensambla antes de la adición a las células a través de la nucleofección. [58] Los componentes principales de este plásmido se muestran en la imagen y se enumeran en la tabla. El crRNA está diseñado exclusivamente para cada aplicación, ya que esta es la secuencia que Cas9 usa para identificar y unirse directamente a secuencias específicas dentro del ADN de la célula huésped. El crRNA debe unirse solo donde se desea la edición. La plantilla de reparación también está diseñada exclusivamente para cada aplicación, ya que debe complementar hasta cierto punto las secuencias de ADN a cada lado del corte y también contener cualquier secuencia que se desee para la inserción en el genoma huésped.
Se pueden empaquetar varios ARNcr y el ARNtracr juntos para formar un ARN guía único (ARNsg). [59] Este ARNsg se puede incluir junto con el gen que codifica la proteína Cas9 y transformarse en un plásmido para ser transfectado en células. Hay muchas herramientas en línea disponibles para ayudar a diseñar secuencias de ARNsg efectivas. [60] [61]

Alternativas a Cas9
![[icono]](http://upload.wikimedia.org/wikipedia/commons/thumb/1/1c/Wiki_letter_w_cropped.svg/20px-Wiki_letter_w_cropped.svg.png) | Esta sección necesita ser ampliada . Puedes ayudar agregándole algo. ( Octubre de 2021 ) |
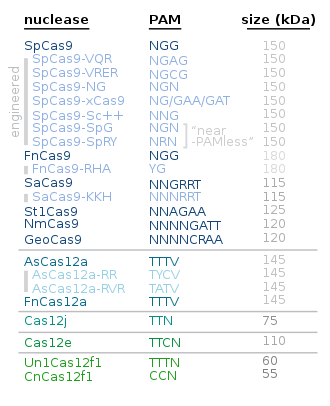
Las proteínas alternativas a Cas9 incluyen las siguientes:
| Proteína | Uso principal / características | Años |
|---|---|---|
| Cas12 | Cas12a es más pequeño y más simple que Cas9; Cas12b es útil para la ingeniería del genoma vegetal [62] [63] | |
| Cas13 | para la edición de ARN [64] | |
| Cas3 [65] [66] | Crea un espacio ancho de una sola hebra [67] | 2019 |
| CasMINI | Aproximadamente el doble de compacto que los Cas9 y Cas12a más comúnmente utilizados. [68] [69] | 2021 |
| SuperFi-Cas9 | Más preciso sin disminuir la velocidad [70] [71] | 2022 |
| Cas7-11 | Edición de ARN [72] | 2022 |
| Reparación del ADN con plantilla cromosómica | Este método sólo es aplicable a organismos cuyo cromosoma correspondiente tiene el/los gen/es deseado/s.
| 2022 |
Estructura
CRISPR-Cas9 ofrece un alto grado de fidelidad y una construcción relativamente simple. Depende de dos factores para su especificidad: la secuencia objetivo y la secuencia del motivo adyacente al protoespaciador (PAM). La secuencia objetivo tiene 20 bases de longitud como parte de cada locus CRISPR en la matriz de crRNA. [58] Una matriz de crRNA típica tiene múltiples secuencias objetivo únicas. Las proteínas Cas9 seleccionan la ubicación correcta en el genoma del huésped utilizando la secuencia para unirse con pares de bases en el ADN del huésped. La secuencia no es parte de la proteína Cas9 y, como resultado, es personalizable y se puede sintetizar de forma independiente . [75] [76]
La secuencia PAM del genoma del huésped es reconocida por Cas9. Cas9 no se puede modificar fácilmente para reconocer una secuencia PAM diferente. Sin embargo, esto no es en última instancia demasiado limitante, ya que normalmente es una secuencia muy corta y no específica que aparece con frecuencia en muchos lugares a lo largo del genoma (por ejemplo, la secuencia PAM de SpCas9 es 5'-NGG-3' y en el genoma humano aparece aproximadamente cada 8 a 12 pares de bases). [58]
Una vez que estas secuencias se han ensamblado en un plásmido y se han transfectado en las células, la proteína Cas9 con la ayuda del crRNA encuentra la secuencia correcta en el ADN de la célula huésped y, dependiendo de la variante de Cas9, crea una ruptura mono o bicatenaria en la ubicación apropiada en el ADN. [77]
Las roturas monocatenarias adecuadamente espaciadas en el ADN del huésped pueden desencadenar una reparación dirigida por homología , que es menos propensa a errores que la unión de extremos no homólogos que normalmente sigue a una rotura bicatenaria. Proporcionar una plantilla de reparación del ADN permite la inserción de una secuencia de ADN específica en una ubicación exacta dentro del genoma. La plantilla de reparación debe extenderse de 40 a 90 pares de bases más allá de la rotura del ADN inducida por Cas9. [58] El objetivo es que el proceso HDR nativo de la célula utilice la plantilla de reparación proporcionada y, por lo tanto, incorpore la nueva secuencia al genoma. Una vez incorporada, esta nueva secuencia ahora es parte del material genético de la célula y pasa a sus células hijas. La inhibición transitoria combinada de NHEJ y TMEJ por una molécula pequeña y ARNi puede aumentar la eficiencia de HDR hasta un 93% y, al mismo tiempo, prevenir la edición fuera del objetivo. [78]
Entrega
La administración de Cas9, sgRNA y complejos asociados a las células puede ocurrir a través de sistemas virales y no virales. La electroporación de ADN, ARN o ribonucleocomplejos es una técnica común, aunque puede producir efectos nocivos en las células diana. [79] También se han utilizado técnicas de transfección química que utilizan lípidos y péptidos para introducir sgRNA en complejos con Cas9 en las células. [80] [81] También se ha utilizado la administración basada en nanopartículas para la transfección. [82] Los tipos de células que son más difíciles de transfectar (por ejemplo, células madre, neuronas y células hematopoyéticas) requieren sistemas de administración más eficientes, como los basados en lentivirus (LV), adenovirus (AdV) y virus adenoasociados (AAV). [83] [84] [85]
Se ha descubierto que la eficiencia de CRISPR-Cas9 aumenta considerablemente cuando varios componentes del sistema, incluida la estructura completa de CRISPR/Cas9 hasta los complejos Cas9-gRNA, se entregan en forma ensamblada en lugar de usar transgénicos. [86] [87] Esto ha encontrado un valor particular en cultivos genéticamente modificados para comercialización masiva. [88] [89] Dado que la maquinaria de replicación del huésped no es necesaria para producir estas proteínas, la posibilidad de que la secuencia de reconocimiento del sgRNA sea casi nula, lo que disminuye la posibilidad de efectos fuera del objetivo. [82]
Edición controlada del genoma
Las mejoras y variantes posteriores del sistema CRISPR-Cas9 se han centrado en introducir un mayor control en su uso. En concreto, la investigación destinada a mejorar este sistema incluye la mejora de su especificidad, su eficiencia y la granularidad de su capacidad de edición. Las técnicas se pueden dividir y clasificar además según el componente del sistema que modifican. Entre ellas se incluyen el uso de variantes diferentes o creaciones novedosas de la proteína Cas, el uso de una proteína efectora totalmente diferente, la modificación del ARNsg o el uso de un enfoque algorítmico para identificar las soluciones óptimas existentes.
La especificidad es un aspecto importante para mejorar el sistema CRISPR-Cas9 porque los efectos fuera del objetivo que genera tienen graves consecuencias para el genoma de la célula e invocan precaución para su uso. Minimizar los efectos fuera del objetivo es, por tanto, maximizar la seguridad del sistema. Las nuevas variaciones de las proteínas Cas9 que aumentan la especificidad incluyen proteínas efectoras con una eficiencia y especificidad comparables a las del SpCas9 original que pueden dirigirse a las secuencias previamente inalcanzables y una variante que prácticamente no tiene mutaciones fuera del objetivo. [90] [91] También se han realizado investigaciones en la ingeniería de nuevas proteínas Cas9, incluidas algunas que reemplazan parcialmente los nucleótidos de ARN en crRNA con ADN y un procedimiento de generación de mutantes Cas9 guiado por la estructura que habían reducido los efectos fuera del objetivo. [92] [93] Se ha demostrado que los sgRNA truncados iterativamente y los gRNA altamente estabilizados también reducen los efectos fuera del objetivo. [94] [95] Se han utilizado métodos computacionales, incluido el aprendizaje automático, para predecir la afinidad y crear secuencias únicas para el sistema a fin de maximizar la especificidad para objetivos determinados. [96] [97]
Varias variantes de CRISPR-Cas9 permiten la activación genética o la edición del genoma con un desencadenante externo, como la luz o moléculas pequeñas. [98] [99] [100] Estos incluyen sistemas CRISPR fotoactivables desarrollados mediante la fusión de proteínas sensibles a la luz asociadas con un dominio activador y un dCas9 para la activación genética, [101] [102] o mediante la fusión de dominios similares sensibles a la luz con dos construcciones de Cas9 dividido, [103] [104] o mediante la incorporación de aminoácidos no naturales enjaulados en Cas9, [105] o mediante la modificación de los ARN guía con complementos fotoescindibles para la edición del genoma. [106]
Los métodos para controlar la edición del genoma con moléculas pequeñas incluyen un Cas9 alostérico, sin edición de fondo detectable, que activará la unión y la escisión tras la adición de 4-hidroxitamoxifeno (4-HT), [98] Cas9 ligado a inteína sensible a 4-HT , [107] o un Cas9 que responde a 4-HT cuando se fusiona a cuatro dominios ERT2. [108] El Cas9 dividido inducible por inteína permite la dimerización de fragmentos de Cas9 [109] y el sistema de Cas9 dividido inducible por rapamicina se desarrolló fusionando dos construcciones de Cas9 dividido con fragmentos de FRB y FKBP . [110] Otros estudios han podido inducir la transcripción de Cas9 con una molécula pequeña, la doxiciclina . [111] [112] Las moléculas pequeñas también se pueden utilizar para mejorar la reparación dirigida por homología, [113] a menudo inhibiendo la vía de unión de extremos no homólogos y/o la vía de unión de extremos mediada por theta. [114] [115] Se creó un sistema con la proteína efectora Cpf1 que es inducida por las moléculas pequeñas VE-822 y AZD-7762. [116] Estos sistemas permiten el control condicional de la actividad CRISPR para mejorar la precisión, la eficiencia y el control espaciotemporal. El control espaciotemporal es una forma de eliminar los efectos no deseados: solo es posible que sea necesario modificar ciertas células o partes del organismo y, por lo tanto, se pueden utilizar moléculas ligeras o pequeñas como una forma de llevar a cabo esto. La eficiencia del sistema CRISPR-Cas9 también aumenta en gran medida mediante la entrega adecuada de las instrucciones de ADN para crear las proteínas y los reactivos necesarios. [116]
CRISPR también utiliza proteínas de edición de un solo par de bases para crear ediciones específicas en una o dos bases en la secuencia objetivo. CRISPR/Cas9 se fusionó con enzimas específicas que inicialmente solo podían cambiar las mutaciones de C a T y de G a A y su reverso. Esto se logró finalmente sin requerir ninguna escisión del ADN. [117] [118] [119] Con la fusión de otra enzima, el sistema de edición de bases CRISPR-Cas9 también puede editar C a G y su reverso. [120]
Detección CRISPR
El sistema de repeticiones de palíndromo corto agrupadas y regularmente interespaciadas (CRISPR)/Cas9 es una tecnología de edición genética que puede inducir roturas de doble cadena (DSB) en cualquier lugar donde los ácidos ribonucleicos guía ( gRNA ) puedan unirse con la secuencia del motivo adyacente al protoespaciador (PAM). [121] Las muescas de cadena simple también pueden ser inducidas por mutantes del sitio activo de Cas9, [122] también conocidos como nickasas de Cas9. [123] Simplemente cambiando la secuencia de gRNA, la endonucleasa Cas9 puede ser entregada a un gen de interés e inducir DSB. [124] La eficiencia de la endonucleasa Cas9 y la facilidad con la que los genes pueden ser seleccionados llevaron al desarrollo de bibliotecas CRISPR-knockout (KO) tanto para células de ratón como humanas, que pueden cubrir conjuntos de genes específicos de interés o el genoma completo. [125] [126] El cribado CRISPR ayuda a los científicos a crear una perturbación genética sistemática y de alto rendimiento dentro de organismos modelo vivos. Esta perturbación genética es necesaria para comprender plenamente la función de los genes y la regulación epigenética. [127] La ventaja de las bibliotecas CRISPR agrupadas es que se pueden seleccionar más genes a la vez.
Las bibliotecas knock-out se crean de manera de lograr una representación y un rendimiento iguales en todos los gRNA expresados y llevan un marcador de selección antibiótico o fluorescente que se puede usar para recuperar células transducidas. [121] Hay dos sistemas de plásmidos en las bibliotecas CRISPR/Cas9. Primero, es un plásmido todo en uno, donde el sgRNA y Cas9 se producen simultáneamente en una célula transfectada. Segundo, es un sistema de dos vectores: los plásmidos sgRNA y Cas9 se entregan por separado. [127] Es importante entregar miles de vectores únicos que contienen sgRNA a un solo recipiente de células por transducción viral a baja multiplicidad de infección (MOI, típicamente a 0,1-0,6), previene la probabilidad de que un clon celular individual obtenga más de un tipo de sgRNA de lo contrario puede conducir a una asignación incorrecta de genotipo a fenotipo . [125]
Una vez preparada la biblioteca agrupada, es necesario llevar a cabo una secuenciación profunda (NGS, secuenciación de próxima generación) del ADN plasmídico amplificado por PCR para revelar la abundancia de sgRNAs. Las células de interés pueden ser infectadas por la biblioteca y luego seleccionadas según el fenotipo. Hay dos tipos de selección: negativa y positiva. Mediante la selección negativa, se detectan de manera eficiente las células muertas o de crecimiento lento. Puede identificar genes esenciales para la supervivencia, que pueden servir como candidatos para fármacos dirigidos molecularmente. Por otro lado, la selección positiva proporciona una colección de poblaciones adquiridas con ventaja de crecimiento mediante mutagénesis aleatoria. [121] Después de la selección, se recolecta y secuencia el ADN genómico mediante NGS. Se detecta la disminución o el enriquecimiento de sgRNAs y se compara con la biblioteca de sgRNA original, anotada con el gen objetivo al que corresponde el sgRNA. Luego, el análisis estadístico identifica los genes que es significativamente probable que sean relevantes para el fenotipo de interés. [125]
| Biblioteca | IDENTIFICACIÓN | Especies | PI | Genes objetivo | gRNA por gen | ARNg totales |
|---|---|---|---|---|---|---|
| Biblioteca de knockout CRISPR para ratones Bassik | 1000000121–1000000130 | Ratón | Bajo | Varía (~23.000 en total) | ~10 | Varía |
| Biblioteca de genes supresores de tumores de ratón con eliminación de genes CRISPR | 113584 Red troncal EFS 113585 Columna vertebral TBG | Ratón | Chen | 56 | ~4 | 286 |
| Biblioteca de todo el genoma del ratón Brie | 73632 (1 plásmido) 73633 (2 plásmidos) | Ratón | Doench y Root | 19.674 | 4 | 78.637 |
| Biblioteca de knockout CRISPR humana de Bassik | 101926–101934 | Humano | Bajo | Varía (~20.500 en total) | ~10 | Varía |
| Biblioteca de todo el genoma humano de Brunello | 73179 (1 plásmido) 73178 (2 plásmidos) | Humano | Doench y Root | 19.114 | 4 | 76.441 |
| Biblioteca de knockout de todo el genoma humano basada en mini-AsCpf1 humano | 130630 | Humano | Draetta | 16.977 | 3–4 | 17.032 matrices |
Además de la eliminación, también existen bibliotecas de eliminación (CRISPRi) y activación (CRISPRa), que utilizan la capacidad de las proteínas de fusión Cas9 desactivadas proteolíticamente (dCas9) para unirse al ADN objetivo, lo que significa que el gen de interés no se corta, sino que se sobreexpresa o se reprime. Esto hizo que el sistema CRISPR/Cas9 fuera aún más interesante en la edición genética. La proteína dCas9 inactiva modula la expresión genética al dirigir los represores o activadores de dCas9 hacia el promotor o los sitios de inicio de la transcripción de los genes objetivo. Para reprimir genes, Cas9 se puede fusionar al dominio efector KRAB que forma un complejo con el ARNm, mientras que CRISPRa utiliza dCas9 fusionado a diferentes dominios de activación transcripcional, que luego son dirigidos por el ARNm a las regiones promotoras para regular positivamente la expresión. [129] [130] [131]
Aplicaciones
Modelos de enfermedades
La modificación genómica de Cas9 ha permitido la generación rápida y eficiente de modelos transgénicos dentro del campo de la genética. Cas9 se puede introducir fácilmente en las células diana junto con sgRNA a través de la transfección de plásmidos para modelar la propagación de enfermedades y la respuesta y defensa de la célula frente a las infecciones. [132] La capacidad de Cas9 para introducirse in vivo permite la creación de modelos más precisos de la función genética y los efectos de las mutaciones, todo ello evitando las mutaciones fuera del objetivo que se observan típicamente con los métodos más antiguos de ingeniería genética.
La revolución de CRISPR y Cas9 en el modelado genómico no se extiende solo a los mamíferos. Los modelos genómicos tradicionales como Drosophila melanogaster , uno de los primeros organismos modelo, han visto un mayor refinamiento en su resolución con el uso de Cas9. [132] Cas9 utiliza promotores específicos de células que permiten un uso controlado de Cas9. Cas9 es un método preciso para tratar enfermedades debido a que la enzima Cas9 se dirige a ciertos tipos de células. Las células que se someten a la terapia Cas9 también se pueden eliminar y reintroducir para proporcionar efectos amplificados de la terapia. [133]
La técnica CRISPR-Cas9 se puede utilizar para editar el ADN de organismos in vivo y eliminar genes individuales o incluso cromosomas enteros de un organismo en cualquier momento de su desarrollo. Entre los cromosomas que se han eliminado con éxito in vivo mediante técnicas CRISPR se encuentran el cromosoma Y y el cromosoma X de ratones de laboratorio adultos y los cromosomas humanos 14 y 21, en líneas de células madre embrionarias y ratones aneuploides respectivamente. Este método podría ser útil para tratar trastornos genéticos causados por un número anormal de cromosomas, como el síndrome de Down y los trastornos intersexuales . [134]
Se ha demostrado que la edición genómica in vivo exitosa usando CRISPR-Cas9 se ha realizado en numerosos organismos modelo, incluidos Escherichia coli , [135] Saccharomyces cerevisiae , [136] [137] Candida albicans , Methanosarcina acetivorans , [138] [139] Caenorhabditis elegans , [140] Arabidopsis spp., [141] Danio rerio , [142] y Mus musculus . [143] [144] Se han logrado éxitos en el estudio de la biología básica, en la creación de modelos de enfermedades, [140] [145] y en el tratamiento experimental de modelos de enfermedades. [146]
Se han planteado inquietudes sobre la posibilidad de que los efectos no deseados (edición de genes distintos a los previstos) puedan confundir los resultados de un experimento de edición genética con CRISPR (es decir, el cambio fenotípico observado puede no deberse a la modificación del gen objetivo, sino a algún otro gen). Se han realizado modificaciones a CRISPR para minimizar la posibilidad de efectos no deseados. A menudo se recomiendan experimentos con CRISPR ortogonales para confirmar los resultados de un experimento de edición genética. [147] [148]
CRISPR simplifica la creación de organismos modificados genéticamente para la investigación que imitan enfermedades o muestran lo que sucede cuando un gen es eliminado o mutado. CRISPR puede usarse a nivel de la línea germinal para crear organismos en los que el gen objetivo se modifica en todas partes (es decir, en todas las células/tejidos/órganos de un organismo multicelular), o puede usarse en células que no pertenecen a la línea germinal para crear cambios locales que solo afecten a ciertas poblaciones de células dentro del organismo. [149] [150] [151]
CRISPR se puede utilizar para crear modelos celulares humanos de enfermedades. [152] Por ejemplo, cuando se aplica a células madre pluripotentes humanas , CRISPR se ha utilizado para introducir mutaciones dirigidas en genes relevantes para la enfermedad renal poliquística (PKD) y la glomeruloesclerosis focal y segmentaria (FSGS). [153] Estas células madre pluripotentes modificadas con CRISPR se cultivaron posteriormente en organoides renales humanos que exhibieron fenotipos específicos de la enfermedad. Los organoides renales de células madre con mutaciones PKD formaron estructuras quísticas grandes y translúcidas a partir de túbulos renales. Los quistes fueron capaces de alcanzar dimensiones macroscópicas, hasta un centímetro de diámetro. [154] Los organoides renales con mutaciones en un gen vinculado a FSGS desarrollaron defectos de unión entre podocitos , las células filtrantes afectadas en esa enfermedad. Esto se atribuyó a la incapacidad de los podocitos para formar microvellosidades entre células adyacentes. [155] Es importante destacar que estos fenotipos de enfermedad no estaban presentes en los organoides de control con antecedentes genéticos idénticos, pero que carecían de las modificaciones CRISPR. [153]
Se adoptó un enfoque similar para modelar el síndrome de QT largo en cardiomiocitos derivados de células madre pluripotentes. [156] Estos modelos celulares generados mediante CRISPR, con controles isogénicos, proporcionan una nueva forma de estudiar enfermedades humanas y probar medicamentos.
Biomedicina
La tecnología CRISPR-Cas se ha propuesto como un tratamiento para múltiples enfermedades humanas, especialmente aquellas con una causa genética. [157] Su capacidad para modificar secuencias de ADN específicas la convierte en una herramienta con potencial para corregir mutaciones causantes de enfermedades. Las primeras investigaciones en modelos animales sugieren que las terapias basadas en la tecnología CRISPR tienen potencial para tratar una amplia gama de enfermedades, [158] incluyendo cáncer, [159] progeria , [160] beta-talasemia, [161] [162] [163] anemia de células falciformes , [163] [164] hemofilia , [165] fibrosis quística , [166] distrofia muscular de Duchenne , [167] enfermedad de Huntington , [168] [169] amiloidosis por transtiretina [43] y enfermedades cardíacas. [170] CRISPR también se ha utilizado para curar la malaria en mosquitos, lo que podría eliminar el vector y la enfermedad en humanos. [171] CRISPR también puede tener aplicaciones en ingeniería de tejidos y medicina regenerativa, por ejemplo mediante la creación de vasos sanguíneos humanos que carecen de expresión de proteínas MHC de clase II , que a menudo causan rechazo de trasplantes. [172]
Además, los ensayos clínicos para curar la beta talasemia y la enfermedad de células falciformes en pacientes humanos utilizando la tecnología CRISPR-Cas9 han mostrado resultados prometedores. [173] [174]
Sin embargo, el uso de esta tecnología en la terapia génica sigue teniendo algunas limitaciones : la frecuencia relativamente alta de efectos fuera del objetivo , el requisito de una secuencia PAM cerca del sitio objetivo, la apoptosis mediada por p53 por roturas de doble cadena inducidas por CRISPR y la toxicidad inmunogénica debido al sistema de administración típicamente por virus . [175]
Cáncer
CRISPR también ha encontrado muchas aplicaciones en el desarrollo de inmunoterapias basadas en células. [176] El primer ensayo clínico que involucró CRISPR comenzó en 2016. Implicó tomar células inmunes de personas con cáncer de pulmón, usar CRISPR para editar el gen PD-1 expresado y luego administrar las células alteradas nuevamente a la misma persona. Otros 20 ensayos estaban en marcha o casi listos, principalmente en China, en 2017. [actualizar][ 159]
En 2016, la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) aprobó un ensayo clínico en el que se utilizaría CRISPR para alterar células T extraídas de personas con diferentes tipos de cáncer y luego administrar esas células T modificadas a las mismas personas. [177]
En noviembre de 2020, en modelos animales de ratón, se utilizó CRISPR de manera eficaz para tratar el glioblastoma (tumor cerebral de rápido crecimiento) y el cáncer de ovario metastásico , ya que son dos cánceres con algunos de los peores pronósticos en el mejor de los casos y generalmente se diagnostican durante sus últimas etapas. Los tratamientos han dado como resultado una inhibición del crecimiento tumoral y un aumento de la supervivencia en un 80% para el cáncer de ovario metastásico y la apoptosis de células tumorales , una inhibición del crecimiento tumoral en un 50% y una mejora de la supervivencia en un 30% para el glioblastoma. [178]
En octubre de 2021, CRISPR Therapeutics anunció los resultados de su ensayo de fase 1 en curso en EE. UU. para una terapia con células T alogénicas. Estas células provienen de donantes sanos y se editan para atacar las células cancerosas y evitar que el sistema inmunológico del receptor las considere una amenaza, y luego se multiplican en lotes enormes que se pueden administrar a un gran número de receptores. [179]
En diciembre de 2022, una niña británica de 13 años a la que se le había diagnosticado leucemia linfoblástica aguda de células T incurable fue curada por médicos del Great Ormond Street Hospital , en el primer uso documentado de la edición genética terapéutica para este propósito, después de someterse a seis meses de un tratamiento experimental, donde los intentos anteriores de otros tratamientos fracasaron. El procedimiento incluyó la reprogramación de una célula T sana para destruir las células T cancerosas para primero librarla de la leucemia y luego reconstruir su sistema inmunológico desde cero utilizando células inmunes sanas. [180] El equipo utilizó la edición BASE y previamente había tratado un caso de leucemia linfoblástica aguda en 2015 utilizando TALEN . [181]
Diabetes
La diabetes tipo 1 es un trastorno endocrino que resulta de la falta de células beta pancreáticas que produzcan insulina, un compuesto vital para transportar el azúcar en sangre a las células para producir energía. Los investigadores han estado tratando de trasplantar células beta sanas. Se utiliza CRISPR para editar las células con el fin de reducir la posibilidad de que el cuerpo del paciente rechace el trasplante.
El 17 de noviembre de 2021, CRISPR therapeutics y ViaCyte anunciaron que la agencia médica canadiense había aprobado su solicitud de ensayo clínico para VCTX210, una terapia con células madre editadas mediante CRISPR diseñada para tratar la diabetes tipo 1. Esto fue significativo porque fue la primera terapia editada genéticamente para la diabetes que se acercó a las clínicas. Las mismas empresas también desarrollaron un tratamiento novedoso para la diabetes tipo 1 para producir insulina a través de un pequeño implante médico que utiliza millones de células pancreáticas derivadas de células madre editadas genéticamente mediante CRISPR. [182]
En febrero de 2022, se llevó a cabo un ensayo de fase 1 en el que un paciente voluntario recibió el tratamiento. [179] [183]
VIH/SIDA
El virus de inmunodeficiencia humana o VIH es un virus que ataca el sistema inmunológico del cuerpo. Si bien existen tratamientos efectivos que pueden permitir que los pacientes vivan vidas saludables, el VIH es retroactivo, lo que significa que incorpora una versión inactiva de sí mismo en el genoma humano. CRISPR se puede utilizar para eliminar selectivamente el virus del genoma mediante el diseño de ARN guía para apuntar al genoma retroactivo del VIH. Un problema con este enfoque es que requiere la eliminación del genoma del VIH de casi todas las células, lo que puede ser difícil de lograr de manera realista. [179]
Los resultados iniciales en el tratamiento y la cura del VIH han sido bastante exitosos: en 2021, 9 de 23 ratones humanizados fueron tratados con una combinación de antirretrovirales y CRISPR/Cas-9 que hicieron que el virus se volviera indetectable, incluso después del período de rebote habitual. Ninguno de los dos tratamientos por sí solo tuvo tal efecto. [184] Los ensayos clínicos en humanos de una terapia basada en CRISPR-Cas9, EBT-101, comenzaron en 2022. [185] [186] En octubre de 2023, un estudio en etapa temprana en 3 personas de EBT-101 informó que el tratamiento parecía ser seguro y no tenía efectos secundarios importantes, pero no se revelaron datos sobre su efectividad. [187] En marzo de 2024, otra terapia CRISPR de investigadores de la Universidad de Ámsterdam informó sobre la eliminación del VIH en cultivos celulares. [188] [189]
Infección
Las "nucleasas guiadas por ARN" basadas en CRISPR-Cas se pueden utilizar para atacar factores de virulencia , genes que codifican resistencia a antibióticos y otras secuencias de interés médicamente relevantes. Por lo tanto, esta tecnología representa una nueva forma de terapia antimicrobiana y una estrategia para manipular poblaciones bacterianas. [190] [191] Estudios recientes sugieren una correlación entre la interferencia del locus CRISPR-Cas y la adquisición de resistencia a antibióticos. [192] Este sistema proporciona protección a las bacterias contra la invasión de ADN extraño, como transposones , bacteriófagos y plásmidos. Se demostró que este sistema es una fuerte presión selectiva para la adquisición de resistencia a antibióticos y factor de virulencia en patógenos bacterianos. [192]
Las terapias basadas en la tecnología de edición genética CRISPR-Cas3 administrada por bacteriófagos diseñados podrían usarse para destruir ADN específico en patógenos. [193] Cas3 es más destructivo que el más conocido Cas9. [194] [195]
Las investigaciones sugieren que la tecnología CRISPR es una forma eficaz de limitar la replicación de múltiples virus del herpes . Fue capaz de erradicar el ADN viral en el caso del virus de Epstein-Barr (VEB). Las tecnologías CRISPR contra el herpesvirus tienen aplicaciones prometedoras, como la eliminación del VEB que causa cáncer de las células tumorales, la ayuda para eliminar los invasores virales de los órganos donados para pacientes inmunodeprimidos o la prevención de brotes de herpes labial e infecciones oculares recurrentes al bloquear la reactivación del VHS-1 . En agosto de 2016 [actualizar], estas técnicas estaban pendientes de ser probadas. [196]
La tecnología CRISPR puede revivir el concepto de trasplantar órganos animales a personas. Los retrovirus presentes en los genomas animales podrían dañar a los receptores de trasplantes. En 2015, un equipo eliminó 62 copias de una secuencia particular de ADN retroviral del genoma de cerdo en una célula epitelial de riñón. [197] Recientemente, los investigadores demostraron la capacidad de generar especímenes de cerdo vivos después de eliminar estos retrovirus de su genoma utilizando CRISPR por primera vez. [198]
Trastornos neurológicos
CRISPR se puede utilizar para suprimir mutaciones que causan ganancia de función, y también para reparar mutaciones que causan pérdida de función en trastornos neurológicos. [199] La herramienta de edición genética se ha convertido en una aplicación in vivo de referencia para la asimilación de vías moleculares.
CRISPR es único en el desarrollo de soluciones para enfermedades neurológicas por varias razones. Por ejemplo, CRISPR permite a los investigadores generar rápidamente modelos de células animales y humanas. Esto les permite estudiar cómo funcionan los genes en un sistema nervioso. Al introducir mutaciones relacionadas con varias enfermedades dentro de estas células, los investigadores pueden estudiar los efectos de los cambios en el desarrollo, la función y el comportamiento del sistema nervioso. [200] Pueden descubrir los mecanismos moleculares que contribuyen a estos trastornos, lo que es esencial para desarrollar tratamientos efectivos. Esto es particularmente útil para modelar y tratar trastornos neurológicos complejos como el Alzheimer , el Parkinson y la epilepsia , entre otros.
La enfermedad de Alzheimer (EA) es una enfermedad neurodegenerativa caracterizada por la pérdida de neuronas y la acumulación de ovillos neurofibrilares intracelulares y placas amiloides extracelulares en el cerebro. [201] Se han identificado tres genes patógenos que causan la aparición temprana de EA en humanos, específicamente la proteína precursora amiloide (APP), la presenilina 1 (PSEN1) y la presenilina 2 (PSEN2). [201] Se han detectado más de 300 mutaciones en estos genes, lo que resulta en un aumento del β-amiloide total (Aβ), la relación Aβ42/40 y/o la polimerización de Aβ.
En el caso de la distrofia muscular de Duchenne , la mutación responsable de la enfermedad se produce en el gen de la distrofina. [202] Se ha utilizado CRISPR para corregir esto. De manera similar, para el síndrome de Dravet , un trastorno de epilepsia, se ha utilizado CRISPR para corregir la mutación del gen SCN1A . [203] A pesar del progreso que se ha logrado, aún existen desafíos con el uso de CRISPR, uno de los cuales es la transferencia de componentes CRISPR a través de la barrera hematoencefálica . Sin embargo, los avances recientes en sistemas de administración de nanopartículas y vectores virales han demostrado ser prometedores para superar este obstáculo [ cita requerida ] . De cara al futuro, se espera que el uso de CRISPR en neurociencia aumente a medida que evolucione la tecnología.
Ceguera
Las enfermedades oculares más comunes en todo el mundo son las cataratas y la retinitis pigmentosa (RP). Estas son causadas por una mutación sin sentido en la cadena alfa que conduce a la ceguera permanente. Un desafío para el uso de CRISPR en enfermedades oculares es que el tejido retiniano del ojo está libre de la respuesta inmune del cuerpo. El enfoque de los investigadores para usar CRISPR es embolsar el gen que codifica la proteína retiniana y editar el genoma. [204]
Amaurosis congénita de Leber
El tratamiento CRISPR para LCA10 (la variante más común de la amaurosis congénita de Leber , que es la principal causa de ceguera infantil hereditaria) modifica el gen fotorreceptor defectuoso del paciente.
En marzo de 2020, el primer paciente voluntario de este estudio con sede en EE. UU., patrocinado por Editas Medicine , recibió una dosis baja del tratamiento para comprobar su seguridad. En junio de 2021, comenzó la inscripción para una cohorte de adultos y pediátricos de dosis alta de 4 pacientes voluntarios cada uno. Se espera que la dosificación de las nuevas cohortes se complete en julio de 2022. [179] En noviembre de 2022, Editas informó de que el 20 % de los pacientes tratados habían experimentado mejoras significativas, pero también anunció que la población objetivo resultante era demasiado pequeña para sustentar un desarrollo independiente continuo. [205]
Enfermedades cardiovasculares
Se ha demostrado que la tecnología CRISPR funciona de manera eficaz en el tratamiento de enfermedades cardíacas. En el caso de la hipercolesterolemia familiar (HF), la deposición de colesterol en las paredes de las arterias provoca el bloqueo del flujo sanguíneo. Esto es causado por una mutación en los receptores de colesterol de lipoproteínas de baja densidad (LDLC) , que da como resultado una liberación excesiva de colesterol en la sangre. Esto se puede tratar mediante la eliminación de un par de bases en el exón 4 del receptor LDLC. Esta es una mutación sin sentido . [ cita requerida ]
β-Hemoglobinopatías
Esta enfermedad se enmarca dentro de los trastornos genéticos que son causados por mutaciones que ocurren en la estructura de la hemoglobina o debido a la sustitución de diferentes aminoácidos en las cadenas de globina. Debido a esto, los glóbulos rojos (RBC) causan una serie de obstáculos como insuficiencia cardíaca, obstrucción de los vasos sanguíneos, defectos en el crecimiento y problemas ópticos. [206] Para rehabilitar las β-hemoglobinopatías, las células multipotentes del paciente se transfieren a un modelo de ratones para estudiar la tasa de terapia génica ex vivo que da como resultado la expresión del ARNm y la rectificación del gen. Curiosamente, la vida media de los glóbulos rojos también aumentó.
Hemofilia
La hemofilia es una pérdida de función en la sangre en la que los factores de coagulación no funcionan correctamente. Mediante el uso de CRISPR-Cas9, se inserta un vector en las bacterias. [207] El vector utilizado es un vector adenoviral que ayuda a corregir los genes.
Agricultura
La aplicación de CRISPR en plantas se logró con éxito en el año 2013. CRISPR Cas9 se ha convertido en una herramienta influyente en la edición de genomas en cultivos. Ha dejado una marca en los sistemas de mejoramiento actuales, [208]
Para aumentar el rendimiento de los cultivos de cereales, se modifica el equilibrio de citoquinina. La citoquinina oxidasa/deshidrogenasa (CKX) es una enzima que inhibe el crecimiento de brotes en el arroz, [209] por lo que se eliminó el gen que codifica esta enzima para aumentar el rendimiento.
Los cereales tienen una gran cantidad de polisacárido amilosa. Para disminuir el contenido de amilosa, se utiliza CRISPR para alterar los aminoácidos y reducir la producción de sacáridos. Además, el trigo contiene la proteína gluten , a la que algunas personas son intolerantes y causa la enfermedad celíaca . La herramienta de edición genética se dirige a los genes que codifican el gluten, lo que da como resultado un trigo con una menor producción de gluten. [210]
Resistencia a las enfermedades
El estrés biótico de las plantas se puede reducir mediante el uso de CRISPR. Las infecciones bacterianas en el arroz provocan la activación de la transcripción de genes, cuyos productos son susceptibles a la enfermedad. Mediante el uso de CRISPR, los científicos pudieron generar resistencia hereditaria al mildiú polvoroso. [211]
Terapia génica
En la actualidad se conocen unos 6000 trastornos genéticos, la mayoría de los cuales no tienen tratamiento. El papel de CRISPR en la terapia génica es sustituir el ADN exógeno por genes defectuosos. [212] La terapia génica ha tenido un enorme impacto y ha abierto muchas nuevas posibilidades en la biotecnología médica.
Edición de base
Existen dos tipos de ediciones base:
El editor de bases de citidina es una nueva terapia en la que la citidina (C) cambia a timidina (T).
Editor de bases de adenina (ABE), [213] en este hay un cambio en los complementos de bases de adenina (A) a guanina (G).
Las mutaciones se instalaron directamente en el ADN celular, por lo que no se requiere la plantilla donante. Las ediciones de base solo pueden editar mutaciones puntuales. Además, solo pueden corregir hasta cuatro mutaciones puntuales. [214] Para abordar este problema, el sistema CRISPR introdujo una nueva técnica conocida como fusión Cas9 para aumentar la escala de genes que se pueden editar.
Silenciamiento y activación de genes
Además, la proteína CRISPR Cas9 puede modular genes ya sea activándolos o silenciándolos en función de los genes de interés. [215] Existe una nucleasa llamada dCas9 (endonucleasa) que se utiliza para silenciar o activar la expresión de genes.
Limitaciones
Los investigadores se enfrentan a muchos desafíos en la edición genética. [216] Los principales obstáculos que se presentan en las aplicaciones clínicas son las cuestiones éticas y el sistema de transporte al sitio objetivo. Como las unidades del sistema CRISPR se toman de las bacterias, cuando se transfieren a las células huésped, se produce una respuesta inmune contra ellas. Se utilizan vectores físicos, químicos y virales como vehículos para entregar el complejo al huésped. [ cita requerida ] Debido a esto, surgen muchas complicaciones, como el daño celular que conduce a la muerte celular. En el caso de los vectores virales, la capacidad del virus es pequeña y la proteína Cas9 es grande. Entonces, para superarlos, se desarrollaron nuevos métodos en los que se toman cepas más pequeñas de Cas9 de las bacterias. Finalmente, todavía se necesita una gran cantidad de trabajo para mejorar el sistema.
Como herramienta de diagnóstico


Las nucleasas asociadas a CRISPR han demostrado ser útiles como herramienta para pruebas moleculares debido a su capacidad para apuntar específicamente a secuencias de ácidos nucleicos en un alto fondo de secuencias no objetivo. [219] En 2016, la nucleasa Cas9 se utilizó para agotar secuencias de nucleótidos no deseadas en bibliotecas de secuenciación de próxima generación mientras que solo requiere 250 picogramos de entrada de ARN inicial. [220] A partir de 2017, las nucleasas asociadas a CRISPR también se utilizaron para pruebas de diagnóstico directo de ácidos nucleicos, hasta la sensibilidad de una sola molécula. [221] [222] La diversidad de CRISPR se utiliza como un objetivo de análisis para discernir la filogenia y la diversidad en bacterias, como en xantomonas por Martins et al. , 2019. [223] : 552 Las detecciones tempranas de patógenos vegetales mediante la tipificación molecular de los CRISPR del patógeno se pueden utilizar en la agricultura como lo demostraron Shen et al. , 2020. [223] : 553
Al acoplar los diagnósticos basados en CRISPR a procesos enzimáticos adicionales, es posible la detección de moléculas más allá de los ácidos nucleicos. Un ejemplo de una tecnología acoplada es el perfil de transcripción IN vitro basado en SHERLOCK (SPRINT). SPRINT se puede utilizar para detectar una variedad de sustancias, como metabolitos en muestras de pacientes o contaminantes en muestras ambientales, con alto rendimiento o con dispositivos portátiles de punto de atención. [224] También se están explorando plataformas CRISPR-Cas para la detección [225] [226] [ 218] [227] [228] e inactivación del SARS-CoV-2 , el virus que causa COVID-19 . [229] Se han identificado dos pruebas de diagnóstico integrales diferentes, AIOD-CRISPR y la prueba SHERLOCK para el SARS-CoV-2. [230] La prueba SHERLOCK se basa en un ARN reportero de prensa marcado con fluorescencia que tiene la capacidad de identificar 10 copias por microlitro. [231] La AIOD-CRISPR ayuda a la detección visual robusta y altamente sensible del ácido nucleico viral. [232]
Antropología genética
CRISPR-Cas9 se puede utilizar para investigar e identificar las diferencias genéticas de los humanos con respecto a otros simios, especialmente en el cerebro . Por ejemplo, al reintroducir variantes genéticas arcaicas en organoides cerebrales para mostrar un impacto en la neurogénesis, [233] la longitud de la metafase de los progenitores apicales del neocórtex en desarrollo, [234] o al inactivar un gen en células madre embrionarias para identificar un regulador genético que, a través de la transición temprana de la forma celular, impulsa la expansión evolutiva del prosencéfalo humano . [235] [236] Un estudio describió un impacto importante de una variante genética arcaica en el neurodesarrollo [237] [238] que puede ser un artefacto de un efecto secundario de CRISPR, [239] [240] ya que no se pudo replicar en un estudio posterior. [78]
Por técnica
Derribo/activación
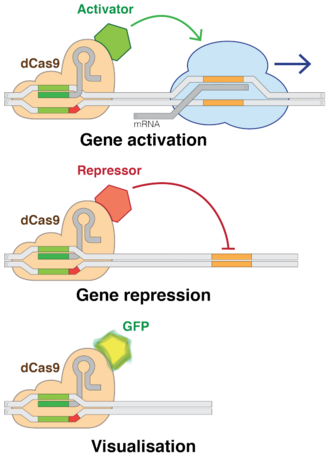
El uso de versiones "muertas" de Cas9 ( dCas9 ) elimina la capacidad de corte de ADN de CRISPR, al tiempo que preserva su capacidad de dirigirse a secuencias deseables. Múltiples grupos agregaron varios factores reguladores a dCas9s, lo que les permitió activar o desactivar casi cualquier gen o ajustar su nivel de actividad. [197] Al igual que RNAi, la interferencia CRISPR (CRISPRi) desactiva genes de manera reversible al dirigirse, pero no cortar un sitio. El sitio dirigido se metila, modificando epigenéticamente el gen. Esta modificación inhibe la transcripción. Estas modificaciones colocadas con precisión pueden luego usarse para regular los efectos sobre las expresiones genéticas y la dinámica del ADN después de la inhibición de ciertas secuencias genómicas dentro del ADN. En los últimos años, las marcas epigenéticas en diferentes células humanas se han investigado de cerca y se ha descubierto que ciertos patrones dentro de las marcas se correlacionan con todo, desde el crecimiento tumoral hasta la actividad cerebral. [10] Por el contrario, la activación mediada por CRISPR (CRISPRa) promueve la transcripción genética. [241] Cas9 es una forma eficaz de atacar y silenciar genes específicos a nivel de ADN. [242] En las bacterias, la presencia de Cas9 por sí sola es suficiente para bloquear la transcripción. Para aplicaciones en mamíferos, se añade una sección de proteína. Su ARN guía ataca secuencias de ADN reguladoras llamadas promotores que preceden inmediatamente al gen diana. [243]
Se utilizó Cas9 para transportar factores de transcripción sintéticos que activaban genes humanos específicos. La técnica logró un efecto potente al dirigir múltiples construcciones CRISPR a lugares ligeramente diferentes en el promotor del gen. [243]
Edición de ARN
En 2016, los investigadores demostraron que el CRISPR de una bacteria bucal común podía usarse para editar el ARN . Los investigadores buscaron en bases de datos que contenían cientos de millones de secuencias genéticas aquellas que se parecían a los genes CRISPR. Consideraron la fusobacteria Leptotrichia shahii . Tenía un grupo de genes que se parecían a los genes CRISPR, pero con diferencias importantes. Cuando los investigadores equiparon otras bacterias con estos genes, a las que llamaron C2c2, descubrieron que los organismos adquirían una defensa novedosa. [244] C2c2 fue posteriormente renombrado como Cas13a para adaptarse a la nomenclatura estándar de los genes Cas. [245]
Muchos virus codifican su información genética en ARN en lugar de ADN, que luego reutilizan para crear nuevos virus. El VIH y el poliovirus son algunos de ellos. Las bacterias con Cas13 crean moléculas que pueden desmembrar el ARN y destruir el virus. La adaptación de estos genes abrió cualquier molécula de ARN a la edición. [244]
Los sistemas CRISPR-Cas también se pueden emplear para editar genes de micro-ARN y ARN largo no codificante en plantas. [246]
Aplicaciones terapéuticas
La edición dirigida para corregir secuencias mutadas se propuso y demostró por primera vez en 1995. [247] Este trabajo inicial utilizó oligonucleótidos antisentido de ARN sintético complementarios a una mutación prematura del codón de terminación en una secuencia de distrofina para activar la edición de A a I del codón de terminación a un codón de lectura directa en un sistema de células modelo de xenopus. [247] Si bien esto también condujo a transiciones inadvertidas cercanas de A a I, las transiciones de A a I (leídas como G) pueden corregir los tres codones de terminación, pero no pueden crear un codón de terminación. Por lo tanto, los cambios condujeron a una corrección de >25% del codón de terminación objetivo con lectura directa a una secuencia reportera de luciferasa corriente abajo. El trabajo posterior de Rosenthal logró la edición de la secuencia de ARNm mutada en un cultivo de células de mamíferos al dirigir un oligonucleótido unido a una citidina desaminasa para corregir una secuencia de fibrosis quística mutada. [248] Más recientemente, se ha empleado CRISPR-Cas13 fusionado a desaminasas para dirigir la edición del ARNm. [249]
En 2022, se informó sobre la edición terapéutica del ARN para Cas7-11. [250] [251] Permite cortes suficientemente específicos y una versión temprana del mismo se utilizó para la edición in vitro en 2021. [252]Comparación con la edición de ADN
Impulso genético
Los impulsores genéticos pueden proporcionar una herramienta poderosa para restablecer el equilibrio de los ecosistemas mediante la eliminación de especies invasoras. Se han planteado inquietudes sobre la eficacia y las consecuencias no deseadas en las especies objetivo, así como en las especies no objetivo, en particular en relación con la posibilidad de liberación accidental de los laboratorios a la naturaleza. Los científicos han propuesto varias salvaguardas para asegurar la contención de los impulsores genéticos experimentales, incluidas las moleculares, reproductivas y ecológicas. [254] Muchos recomiendan que se desarrollen impulsores de inmunización y reversión en tándem con los impulsores genéticos para sobrescribir sus efectos si es necesario. [255] Sigue habiendo consenso en que los efectos a largo plazo deben estudiarse más a fondo, en particular en relación con el potencial de alteración ecológica que no se puede corregir con impulsores de reversión. [256]
In vitroagotamiento genético
Las bibliotecas de secuenciación no enriquecidas suelen contener abundantes secuencias no deseadas. Cas9 puede eliminar específicamente las secuencias no deseadas con rotura de doble cadena con una eficacia de hasta el 99 % y sin efectos significativos fuera del objetivo , como se observa con las enzimas de restricción . El tratamiento con Cas9 puede eliminar abundante ARNr al tiempo que aumenta la sensibilidad a los patógenos en las bibliotecas de secuenciación de ARN. [257]
Edición del epigenoma

La edición o ingeniería del epigenoma es un tipo de ingeniería genética en la que el epigenoma se modifica en sitios específicos utilizando moléculas diseñadas específicamente para esos sitios (en lugar de modificaciones del genoma completo). Mientras que la edición genética implica cambiar la secuencia de ADN en sí, la edición epigenética implica modificar y presentar secuencias de ADN a proteínas y otros factores de unión al ADN que influyen en la función del ADN. Al "editar" características epigenómicas de esta manera, los investigadores pueden determinar el papel biológico exacto de una modificación epigenética en el sitio en cuestión.
Las proteínas diseñadas que se utilizan para la edición epigenómica se componen de un dominio de unión al ADN que se dirige a secuencias específicas y un dominio efector que modifica las características epigenómicas. En la actualidad, se han utilizado predominantemente tres grupos principales de proteínas de unión al ADN para la edición epigenómica: proteínas con dedos de zinc , efectores similares a activadores de la transcripción (TALE) y fusiones Cas9 deficientes en nucleasas ( CRISPR ).Aplicaciones
La regulación dirigida de genes relacionados con enfermedades puede permitir nuevas terapias para muchas enfermedades, especialmente en casos en los que aún no se han desarrollado terapias genéticas adecuadas o son inadecuadas. [258] Si bien las consecuencias transgeneracionales y a nivel poblacional no se comprenden por completo, puede convertirse en una herramienta importante para la genómica funcional aplicada y la medicina personalizada . [259] Al igual que con la edición de ARN , no implica cambios genéticos y sus riesgos acompañantes. [258] En 2021 se describió un ejemplo de un posible uso funcional de la edición del epigenoma: la represión de la expresión del gen Na v 1.7 a través de CRISPR-dCas9 , que mostró potencial terapéutico en tres modelos de ratón de dolor crónico. [260] [261]
En 2022, una investigación evaluó su utilidad para reducir los niveles de proteína tau , regular una proteína involucrada en la enfermedad de Huntington , atacar una forma hereditaria de obesidad y el síndrome de Dravet . [262]Integrasas dirigidas por CRISPR
La combinación de CRISPR-Cas9 con integrasas permitió una técnica para realizar ediciones a gran escala sin problemas de rotura de doble cadena, como se demostró con PASTE en 2022. Los investigadores informaron que podría usarse para administrar genes de hasta 36 000 pares de bases de ADN a varios tipos de células humanas y, por lo tanto, potencialmente para tratar enfermedades causadas por una gran cantidad de mutaciones. [263] [264]
Edición de primera
La edición primaria [265] (o edición de base) es un refinamiento de CRISPR para insertar o eliminar con precisión secciones de ADN. Las ediciones CRISPR no siempre son perfectas y los cortes pueden terminar en el lugar equivocado. Ambos problemas son un problema para el uso de la tecnología en medicina. [266] La edición primaria no corta el ADN de doble cadena, sino que utiliza el aparato de orientación CRISPR para enviar una enzima adicional a una secuencia deseada, donde convierte un solo nucleótido en otro. [267] La nueva guía, llamada pegRNA, contiene una plantilla de ARN para una nueva secuencia de ADN que se agregará al genoma en la ubicación objetivo. Eso requiere una segunda proteína, unida a Cas9: una enzima transcriptasa inversa, que puede hacer una nueva cadena de ADN a partir de la plantilla de ARN e insertarla en el sitio mellado. [268] Cada uno de esos tres eventos de emparejamiento independientes brinda una oportunidad para prevenir secuencias fuera del objetivo, lo que aumenta significativamente la flexibilidad de la orientación y la precisión de la edición. [267] La edición primaria fue desarrollada por investigadores del Instituto Broad del MIT y Harvard en Massachusetts. [269] Se necesita más trabajo para optimizar los métodos. [269] [268]
Sociedad y cultura
Modificación de la línea germinal humana
En marzo de 2015, varios grupos habían anunciado investigaciones en curso con la intención de sentar las bases para aplicar CRISPR a embriones humanos para la ingeniería de la línea germinal humana , incluidos laboratorios en los EE. UU., China y el Reino Unido, así como la empresa de biotecnología estadounidense OvaScience . [270] Los científicos, incluido un codescubridor de CRISPR, instaron a una moratoria mundial sobre la aplicación de CRISPR a la línea germinal humana, especialmente para uso clínico. Dijeron que "los científicos deberían evitar incluso intentar, en jurisdicciones laxas, la modificación del genoma de la línea germinal para la aplicación clínica en humanos" hasta que las implicaciones completas "se discutan entre las organizaciones científicas y gubernamentales". [271] [272] Estos científicos apoyan una mayor investigación de bajo nivel sobre CRISPR y no ven que CRISPR esté lo suficientemente desarrollado para cualquier uso clínico en la realización de cambios hereditarios en humanos. [273]
En abril de 2015, científicos chinos informaron los resultados de un intento de alterar el ADN de embriones humanos no viables utilizando CRISPR para corregir una mutación que causa beta talasemia , un trastorno hereditario letal. [274] [275] El estudio había sido rechazado anteriormente tanto por Nature como por Science en parte debido a preocupaciones éticas. [276] Los experimentos dieron como resultado cambiar con éxito solo algunos de los genes previstos y tuvieron efectos no deseados en otros genes. Los investigadores afirmaron que CRISPR no está listo para su aplicación clínica en medicina reproductiva . [276] En abril de 2016, se informó que científicos chinos habían hecho un segundo intento fallido de alterar el ADN de embriones humanos no viables utilizando CRISPR, esta vez para alterar el gen CCR5 para hacer que el embrión sea resistente a la infección por VIH . [277]
En diciembre de 2015, se celebró en Washington una Cumbre Internacional sobre Edición Genética Humana bajo la dirección de David Baltimore . Miembros de las academias científicas nacionales de los EE. UU., el Reino Unido y China discutieron la ética de la modificación de la línea germinal. Acordaron apoyar la investigación básica y clínica bajo ciertas pautas legales y éticas. Se hizo una distinción específica entre las células somáticas , donde los efectos de las ediciones se limitan a un solo individuo, y las células de la línea germinal, donde los cambios del genoma pueden ser heredados por los descendientes. Las modificaciones hereditarias podrían tener consecuencias no deseadas y de largo alcance para la evolución humana, genéticamente (por ejemplo, interacciones gen-ambiente) y culturalmente (por ejemplo, darwinismo social ). La alteración de gametocitos y embriones para generar cambios hereditarios en humanos se definió como irresponsable. El grupo acordó iniciar un foro internacional para abordar tales preocupaciones y armonizar las regulaciones en todos los países. [278]
En febrero de 2017, el Comité de Edición Genética Humana de las Academias Nacionales de Ciencias, Ingeniería y Medicina ( NASEM ) de los Estados Unidos publicó un informe en el que se analizaban las cuestiones éticas, jurídicas y científicas de la tecnología de ingeniería genómica. La conclusión del informe afirmaba que la edición genómica hereditaria no está permitida en la actualidad, pero que podría justificarse en el caso de determinadas enfermedades; sin embargo, no justificaba el uso de CRISPR para la mejora genética. [279]
En noviembre de 2018, Jiankui He anunció que había editado dos embriones humanos para intentar desactivar el gen CCR5 , que codifica un receptor que el VIH usa para ingresar a las células. Dijo que las niñas gemelas, Lulu y Nana , habían nacido unas semanas antes. Dijo que las niñas todavía portaban copias funcionales de CCR5 junto con CCR5 desactivado ( mosaicismo ) y aún eran vulnerables al VIH. El trabajo fue ampliamente condenado como poco ético, peligroso y prematuro. [280] Un grupo internacional de científicos pidió una moratoria global sobre la edición genética de embriones humanos. [281]
Bebés de diseño
La aparición de la tecnología de edición genética CRISPR-Cas9 ha abierto la posibilidad de crear "bebés de diseño". Esta tecnología tiene la posibilidad de eliminar ciertas enfermedades genéticas o mejorar la salud al potenciar ciertos rasgos genéticos.
Barreras políticas a la ingeniería genética
Las normativas sobre el sistema CRISPR-Cas9 varían en todo el mundo. En febrero de 2016, los organismos reguladores dieron permiso a científicos británicos para modificar genéticamente embriones humanos mediante el uso de CRISPR-Cas9 y técnicas relacionadas. Sin embargo, a los investigadores se les prohibió implantar los embriones y estos debían ser destruidos después de siete días. [282]
Estados Unidos cuenta con un elaborado sistema regulatorio interdepartamental para evaluar nuevos alimentos y cultivos modificados genéticamente. Por ejemplo, la Ley de Protección de Riesgos Agrícolas de 2000 otorga al Departamento de Agricultura de los Estados Unidos la autoridad para supervisar la detección, control, erradicación, supresión, prevención o retraso de la propagación de plagas de plantas o malezas nocivas para proteger la agricultura, el medio ambiente y la economía de los Estados Unidos. La ley regula cualquier organismo modificado genéticamente que utilice el genoma de una "plaga de plantas" predefinida o cualquier planta no categorizada previamente. [283] En 2015, Yinong Yang desactivó con éxito 16 genes específicos en el champiñón blanco para que no se oscurecieran. Dado que no había agregado ADN de especies extrañas ( transgénico ) a su organismo, el hongo no podía ser regulado por el USDA según la Sección 340.2. [284] El champiñón blanco de Yang fue el primer organismo modificado genéticamente con el sistema de proteína CRISPR-Cas9 en pasar la regulación estadounidense. [285]
En 2016, el USDA patrocinó un comité para considerar la política regulatoria futura para las próximas técnicas de modificación genética. Con la ayuda de las Academias Nacionales de Ciencias, Ingeniería y Medicina de EE. UU ., grupos de intereses especiales se reunieron el 15 de abril para contemplar los posibles avances en ingeniería genética dentro de los próximos cinco años y cualquier nueva regulación que pudiera ser necesaria como resultado. [286] En 2017, la Administración de Alimentos y Medicamentos propuso una regla que clasificaría las modificaciones de ingeniería genética a los animales como "medicamentos animales", sometiéndolos a una estricta regulación si se ofrecen para la venta y reduciendo la capacidad de los individuos y las pequeñas empresas para rentabilizarlos. [287] [288]
En China, donde las condiciones sociales contrastan marcadamente con las de Occidente, las enfermedades genéticas conllevan un fuerte estigma. [289] Esto deja a China con menos barreras políticas para el uso de esta tecnología. [290] [291]
Reconocimiento
En 2012 y 2013, CRISPR fue finalista en el premio Breakthrough of the Year de la revista Science . En 2015, fue el ganador de ese premio. [197] CRISPR fue nombrado como una de las 10 tecnologías innovadoras de MIT Technology Review en 2014 y 2016. [292] [293] En 2016, Jennifer Doudna y Emmanuelle Charpentier , junto con Rudolph Barrangou, Philippe Horvath y Feng Zhang ganaron el premio Gairdner International. En 2017, Doudna y Charpentier recibieron el Premio Japón en Tokio, Japón por su revolucionaria invención de CRISPR-Cas9. En 2016, Charpentier, Doudna y Zhang ganaron el Premio Tang en Ciencia Biofarmacéutica. [294] En 2020, Charpentier y Doudna recibieron el Premio Nobel de Química , el primer premio de este tipo para un equipo formado exclusivamente por mujeres, "por el desarrollo de un método de edición del genoma". [295]
Véase también
Referencias
- ^ abc Bak RO, Gomez-Ospina N, Porteus MH (agosto de 2018). "La edición genética en el centro del escenario". Tendencias en genética . 34 (8): 600–611. doi :10.1016/j.tig.2018.05.004. PMID 29908711. S2CID 49269023.
- ^ "El Premio Nobel de Química 2020". El Premio Nobel . Consultado el 10 de diciembre de 2020 .
- ^ Cohen J (7 de octubre de 2020). "CRISPR, las revolucionarias "tijeras" genéticas, galardonadas por el Nobel de Química". Science . doi :10.1126/science.abf0540. S2CID 225116732.
- ^ Cohen J (4 de junio de 2018). «Con un prestigioso premio, un investigador eclipsado de CRISPR gana protagonismo». Ciencia | AAAS . Consultado el 2 de mayo de 2020 .
- ^ ab Owens R (8 de octubre de 2020). "Premio Nobel: ¿quién queda fuera?". The Conversation . Consultado el 13 de diciembre de 2021 .
- ^ "Los científicos lituanos no reciben el premio Nobel a pesar de haber descubierto la misma tecnología". LRT.LT . 8 de octubre de 2020.
- ^ Šikšnys V (16 de junio de 2018). "Imam genų žirkles, iškerpam klaidą, ligos nelieka". Laisvės TV / Libertad TV . 12:22 minutos. LaisvėsTV. <...>Tai mes tą savo straipsnį išsiuntėm į redakciją pirmieji, bet laimės ten daug nebuvo. Viena redakcija pasakė, kad mes net recenzentam nesiųsim. Nusiuntėm į kitą redakciją – tai jis (straipsnis) pragulėjo kažkur ant redaktoriaus stalo labai ilgai. Na ir taip galų gale išsiuntėm į trečią žurnalą ir trečias žurnalas po kelių mėnesių jį išspausdino. Bet, aišku, Berklio universiteto mokslininkams sekėsi geriau – jie išsiuntė straipsnį į žurnalą Science – jį priėmė ir išspausdino per 2 savaites. Nors iš tikro jie tą straispnį išsiuntė pora mėnesių vėliau nei mes . Consultado el 30 de junio de 2018 .
<...> Bueno, fuimos nosotros los que enviamos el artículo primero, pero no tuvimos mucha suerte.
- ^ Zhang JH, Pandey M, Kahler JF, Loshakov A, Harris B, Dagur PK, et al. (noviembre de 2014). "Mejora de la especificidad y eficacia de CRISPR/CAS9 y gRNA a través de un reportero de ADN específico de la diana". Journal of Biotechnology . 189 : 1–8. doi :10.1016/j.jbiotec.2014.08.033. PMC 4252756 . PMID 25193712.
- ^ Vakulskas CA, Dever DP, Rettig GR, Turk R, Jacobi AM, Collingwood MA, et al. (agosto de 2018). "Un mutante de Cas9 de alta fidelidad administrado como un complejo de ribonucleoproteína permite una edición genética eficiente en células madre y progenitoras hematopoyéticas humanas". Nature Medicine . 24 (8): 1216–1224. doi :10.1038/s41591-018-0137-0. PMC 6107069 . PMID 30082871.
- ^ abc Ledford H (marzo de 2016). «CRISPR: la edición genética es solo el comienzo». Nature . 531 (7593): 156–159. Bibcode :2016Natur.531..156L. doi : 10.1038/531156a . PMID 26961639.
- ^ Travis J (17 de diciembre de 2015). "Avance del año: CRISPR supera el desafío". Revista Science . Asociación Estadounidense para el Avance de la Ciencia.
- ^ Ledford H (junio de 2015). «CRISPR, el disruptor». Nature . 522 (7554): 20–24. Bibcode :2015Natur.522...20L. doi : 10.1038/522020a . PMID 26040877.
- ^ ab "Casgevy: Reino Unido aprueba un fármaco de edición genética para la anemia falciforme". BBC News . 16 de noviembre de 2023 . Consultado el 16 de noviembre de 2023 .
- ^ ab "MHRA autoriza la primera terapia génica del mundo que tiene como objetivo curar la anemia falciforme y la beta-talasemia dependiente de transfusiones". Gov.uk . 16 de noviembre de 2023 . Consultado el 16 de noviembre de 2023 .
- ^ "La FDA aprueba las primeras terapias genéticas para tratar a pacientes con anemia falciforme". Administración de Alimentos y Medicamentos . 11 de diciembre de 2023 . Consultado el 11 de diciembre de 2023 .
- ^ Young S (11 de febrero de 2014). "CRISPR y otras herramientas de edición del genoma impulsan la investigación médica y el alcance de la terapia génica". MIT Technology Review . Consultado el 13 de abril de 2014 .
- ^ ab Heidenreich M, Zhang F (enero de 2016). "Aplicaciones de los sistemas CRISPR-Cas en neurociencia". Nature Reviews. Neurociencia . 17 (1): 36–44. doi :10.1038/nrn.2015.2. PMC 4899966 . PMID 26656253.
- ^ Barrangou R, Doudna JA (septiembre de 2016). "Aplicaciones de las tecnologías CRISPR en la investigación y más allá". Nature Biotechnology . 34 (9): 933–941. doi :10.1038/nbt.3659. PMID 27606440. S2CID 21543486.
- ^ Cox DB, Platt RJ, Zhang F (febrero de 2015). "Edición terapéutica del genoma: perspectivas y desafíos". Nature Medicine . 21 (2): 121–131. doi :10.1038/nm.3793. PMC 4492683 . PMID 25654603.
- ^ abcd "Cronología CRISPR". Broad Institute . 25 de septiembre de 2015. Consultado el 8 de diciembre de 2023 .
- ^ "La locura de CRISPR". GEN . 8 de noviembre de 2013.
- ^ Staff (1 de abril de 2015). "Noticias: productos y servicios". Noticias sobre ingeniería genética y biotecnología (artículo). 35 (7): 8. doi :10.1089/gen.35.21.05.
- ^ "¿Quién posee el mayor descubrimiento biotecnológico del siglo? Existe una amarga lucha por las patentes de CRISPR, una nueva y revolucionaria forma de edición de ADN". MIT Technology Review . Consultado el 25 de febrero de 2015 .
- ^ Fye S. "Genetic Rough Draft: Editas and CRISPR". The Atlas Business Journal . Consultado el 19 de enero de 2016 .
- ^ "Sistemas CRISPR-Cas y métodos para alterar la expresión de productos genéticos". Google Patents .
- ^ Shaffer C (abril de 2022). "Broad derrota la patente CRISPR de Berkeley". Nature Biotechnology . 40 (4): 445. doi :10.1038/d41587-022-00004-2. PMID 35288688. S2CID 247453528.
- ^ "Las patentes de CRISPR van a juicio". Nature Biotechnology . 34 (2): 121. Febrero de 2016. doi : 10.1038/nbt0216-121a . PMID 26849500. S2CID 205265912.
- ^ Pollack A (15 de febrero de 2017). "Científicos de Harvard y el MIT ganan la batalla por la patente de edición genética". The New York Times .
- ^ Akst J (15 de febrero de 2017). "Broad gana el caso de interferencia de patentes de CRISPR". Revista The Scientist .
- ^ Noonan KE (16 de febrero de 2017). "PTAB decide la interferencia de CRISPR a favor del Broad Institute – Su razonamiento". Patent Docs .
- ^ Potenza A (13 de abril de 2017). «UC Berkeley impugna la decisión de que las patentes de CRISPR pertenecen al Broad Institute». The Verge . Consultado el 22 de septiembre de 2017 .
- ^ Buhr S (26 de julio de 2017). "La batalla por la patente de CRISPR ha vuelto a empezar cuando UC Berkeley presenta una apelación". TechCrunch . Consultado el 22 de septiembre de 2017 .
- ^ Westman N (1 de marzo de 2022). «UC Berkeley pierde el caso de patente CRISPR». The Verge . Consultado el 6 de marzo de 2022 .
- ^ ab Philippidis A (7 de agosto de 2017). "MilliporeSigma recibirá patente europea para tecnología CRISPR". Noticias de ingeniería genética y biotecnología . Consultado el 22 de septiembre de 2017 .
- ^ Akst J (24 de marzo de 2017). «UC Berkeley recibe patente CRISPR en Europa». The Scientist . Consultado el 22 de septiembre de 2017 .
- ^ Cohen J (4 de agosto de 2017). "La batalla por las patentes de CRISPR en Europa da un giro 'salvaje' con un actor sorprendente". Science . doi :10.1126/science.aan7211.
- ^ "Tribunal Supremo de la UE: las normas sobre los OGM cubren la técnica de edición genética de plantas". Retuers. 25 de julio de 2018.
- ^ AFP (7 de febrero de 2020). «Un ensayo en EE. UU. muestra que el genoma de 3 pacientes con cáncer se alteró de forma segura mediante CRISPR». ScienceAlert . Consultado el 9 de febrero de 2020 .
- ^ Chamary JV. "Estos científicos merecían un premio Nobel, pero no descubrieron Crispr". Forbes . Consultado el 10 de julio de 2020 .
- ^ Fischman J. "El Premio Nobel de Química se otorga al descubrimiento de unas 'tijeras genéticas' llamadas CRISPR/Cas9". Scientific American . Consultado el 24 de marzo de 2021 .
- ^ "Dos mujeres comparten el Nobel de Química en una histórica victoria para las 'tijeras genéticas'". BBC News . 2020-10-07 . Consultado el 2020-12-06 .
- ^ Kaiser J (26 de junio de 2021). «CRISPR inyectado en la sangre trata una enfermedad genética por primera vez». Ciencia | AAAS . Consultado el 11 de julio de 2021 .
- ^ ab Gillmore JD, Gane E, Taubel J, Kao J, Fontana M, Maitland ML, et al. (agosto de 2021). "Edición genética in vivo con CRISPR-Cas9 para la amiloidosis por transtiretina". The New England Journal of Medicine . 385 (6): 493–502. doi : 10.1056/NEJMoa2107454 . PMID 34215024. S2CID 235722446.
- ^ Boonstra E, de Kleijn R, Colzato LS, Alkemade A, Forstmann BU, Nieuwenhuis S (6 de octubre de 2015). "Neurotransmisores como complementos alimenticios: los efectos del GABA en el cerebro y el comportamiento". Fronteras en Psicología . 6 : 1520. doi : 10.3389/fpsyg.2015.01520 . PMC 4594160 . PMID 26500584.
- ^ "El tomate en Japón es el primer alimento editado con CRISPR del mundo que sale a la venta". IFLScience . Consultado el 18 de octubre de 2021 .
- ^ Wang T, Zhang H, Zhu H (15 de junio de 2019). "La tecnología CRISPR está revolucionando la mejora del tomate y otros cultivos frutales". Investigación en horticultura . 6 (1): 77. Bibcode :2019HorR....6...77W. doi :10.1038/s41438-019-0159-x. PMC 6570646 . PMID 31240102.
- ^ "Japón adopta el pescado editado con CRISPR". Nature Biotechnology . 40 (1): 10. enero de 2022. doi :10.1038/s41587-021-01197-8. PMID 34969964. S2CID 245593283.
- ^ "Startup espera que el pez globo modificado genéticamente sea un éxito en 2022". The Japan Times . 5 de enero de 2022. Archivado desde el original el 17 de enero de 2022 . Consultado el 17 de enero de 2022 .
- ^ "Besugo modificado genéticamente a la venta en Japón". thefishsite.com .
- ^ Götz L, Svanidze M, Tissier A, Brand A (enero de 2022). "Disposición de los consumidores a comprar tomates modificados genéticamente con CRISPR: evidencia de un estudio de caso de experimento de elección en Alemania". Sustainability . 14 (2): 971. doi : 10.3390/su14020971 . hdl : 10419/249208 .
- ^ "¿Están dispuestos los consumidores a comprar tomates CRISPR?". Actualización sobre biotecnología de cultivos . Consultado el 21 de febrero de 2022 .
- ^ Whitford E (28 de mayo de 2021). "UC Berkeley subastará NFT por dos patentes del Premio Nobel". Inside Higher Ed . Consultado el 21 de febrero de 2023 .
- ^ Sestino A, Guido G, Peluso AM (2022). Tokens no fungibles (NFT). Análisis del impacto en los consumidores y las estrategias de marketing . p. 28. doi :10.1007/978-3-031-07203-1. ISBN 978-3-031-07202-4. Número de identificación del sujeto 250238540.
- ^ Chang K (27 de mayo de 2021). "Puedes comprar una parte de un descubrimiento ganador del Premio Nobel". New York Times . Consultado el 21 de febrero de 2023 .
- ^ Trautman LJ (2022). "Arte virtual y tokens no fungibles" (PDF) . Hofstra Law Review . 50 (361): 369 y siguientes. doi :10.2139/ssrn.3814087. S2CID 234830426.
- ^ Jones N (18 de junio de 2021). "Cómo los científicos están adoptando las NFT". Nature . 594 (7864): 482. Bibcode :2021Natur.594..481J. doi : 10.1038/d41586-021-01642-3 . PMID 34145410. S2CID 235481285.
- ^ Oficina del Comisionado (8 de diciembre de 2023). «La FDA aprueba las primeras terapias genéticas para tratar a pacientes con anemia falciforme». FDA . Consultado el 14 de diciembre de 2023 .
- ^ abcde Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (noviembre de 2013). "Ingeniería genómica utilizando el sistema CRISPR-Cas9". Nature Protocols . 8 (11): 2281–2308. doi :10.1038/nprot.2013.143. hdl :1721.1/102943. PMC 3969860 . PMID 24157548.
- ^ Ly J (2013). Descubrimiento de genes responsables de enfermedades renales (Ph.D.). Universidad de Toronto . Consultado el 26 de diciembre de 2016 .
- ^ Mohr SE, Hu Y, Ewen-Campen B, Housden BE, Viswanatha R, Perrimon N (septiembre de 2016). "Diseño de ARN guía CRISPR para aplicaciones de investigación". The FEBS Journal . 283 (17): 3232–3238. doi :10.1111/febs.13777. PMC 5014588 . PMID 27276584.
- ^ Brazelton VA, Zarecor S, Wright DA, Wang Y, Liu J, Chen K, et al. (2015). "Una guía rápida para las herramientas de diseño de ARNsg CRISPR". GM Crops & Food . 6 (4): 266–276. doi :10.1080/21645698.2015.1137690. PMC 5033207 . PMID 26745836.
- ^ "Investigadores establecen un nuevo sistema CRISPR-Cas12b viable para la ingeniería del genoma vegetal". phys.org . Consultado el 6 de abril de 2020 .
- ^ Ming M, Ren Q, Pan C, He Y, Zhang Y, Liu S, et al. (marzo de 2020). "CRISPR-Cas12b permite una ingeniería genómica eficiente de las plantas". Nature Plants . 6 (3): 202–208. doi :10.1038/s41477-020-0614-6. PMID 32170285. S2CID 212643374.
- ^ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (noviembre de 2017). "Edición de ARN con CRISPR-Cas13". Science . 358 (6366): 1019–1027. Bibcode :2017Sci...358.1019C. doi :10.1126/science.aaq0180. PMC 5793859 . PMID 29070703.
- ^ "La innovación de CRISPR-Cas3 promete curas para enfermedades y avances científicos". Cornell Chronicle . Consultado el 24 de octubre de 2021 .
- ^ Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, et al. (junio de 2019). "Introducción de un espectro de deleciones genómicas de largo alcance en células madre embrionarias humanas mediante CRISPR-Cas tipo I". Molecular Cell . 74 (5): 936–950.e5. doi :10.1016/j.molcel.2019.03.014. PMC 6555677 . PMID 30975459.
- ^ Liu Z, Dong H, Cui Y, Cong L, Zhang D (septiembre de 2020). "Aplicación de diferentes tipos de sistemas basados en CRISPR/Cas en bacterias". Microbial Cell Factories . 19 (1): 172. doi : 10.1186/s12934-020-01431-z . PMC 7470686 . PMID 32883277.
- ^ "Los investigadores desarrollan un sistema de edición genómica CRISPR 'mini' diseñado". phys.org . Consultado el 18 de octubre de 2021 .
- ^ Xu X, Chemparathy A, Zeng L, Kempton HR, Shang S, Nakamura M, Qi LS (octubre de 2021). "Sistema CRISPR-Cas en miniatura diseñado para la regulación y edición del genoma de mamíferos". Molecular Cell . 81 (20): 4333–4345.e4. doi : 10.1016/j.molcel.2021.08.008 . PMID 34480847. S2CID 237417317.
- ^ Bravo JP, Liu MS, Hibshman GN, Dangerfield TL, Jung K, McCool RS, et al. (marzo de 2022). "Base estructural para la vigilancia de desajustes mediante CRISPR-Cas9". Nature . 603 (7900): 343–347. Bibcode :2022Natur.603..343B. doi :10.1038/s41586-022-04470-1. PMC 8907077 . PMID 35236982.
- ^ "Un ajuste de proteínas hace que la edición genética CRISPR sea 4000 veces menos propensa a errores". New Atlas . 2022-03-04 . Consultado el 2022-03-07 .
- ^ Kato K, Zhou W, Okazaki S, Isayama Y, Nishizawa T, Gootenberg JS, et al. (junio de 2022). "Estructura e ingeniería del complejo efector CRISPR-Cas7-11 tipo III-E". Cell . 185 (13): 2324–2337.e16. doi : 10.1016/j.cell.2022.05.003 . PMID 35643083. S2CID 249103058.
- Resumen para legos: Williams S. "Los neurocientíficos amplían el conjunto de herramientas CRISPR con la nueva enzima compacta Cas7-11". Instituto Tecnológico de Massachusetts . Consultado el 22 de junio de 2022 .
- ^ "Una forma 'más suave' de CRISPR puede editar genes con mayor precisión". New Scientist . Consultado el 21 de agosto de 2022 .
- ^ Roy S, Juste SS, Sneider M, Auradkar A, Klanseck C, Li Z, et al. (julio de 2022). "Conversión alélica inducida por Cas9/Nickase mediante reparación con plantilla de cromosoma homólogo en células somáticas de Drosophila". Science Advances . 8 (26): eabo0721. Bibcode :2022SciA....8O.721R. doi : 10.1126/sciadv.abo0721 . PMC 10883370 . PMID 35776792.
- ^ Horvath P, Barrangou R (enero de 2010). "CRISPR/Cas, el sistema inmunológico de bacterias y arqueas". Science . 327 (5962): 167–170. Bibcode :2010Sci...327..167H. doi :10.1126/science.1179555. PMID 20056882. S2CID 17960960.
- ^ Bialk P, Rivera-Torres N, Strouse B, Kmiec EB (8 de junio de 2015). "Regulación de la actividad de edición génica dirigida por oligonucleótidos monocatenarios y sistemas CRISPR/Cas9". PLOS ONE . 10 (6): e0129308. Bibcode :2015PLoSO..1029308B. doi : 10.1371/journal.pone.0129308 . PMC 4459703 . PMID 26053390.
- ^ Sander JD, Joung JK (abril de 2014). "Sistemas CRISPR-Cas para editar, regular y dirigir genomas". Nature Biotechnology . 32 (4): 347–355. doi :10.1038/nbt.2842. PMC 4022601 . PMID 24584096.
- ^ ab Riesenberg S, Kanis P, Macak D, Wollny D, Düsterhöft D, Kowalewski J, et al. (septiembre de 2023). "Edición genómica dependiente de reparación dirigida por homología de alta precisión y eficiente mediante HDRobust". Nature Methods . 20 (9): 1388–1399. doi : 10.1038/s41592-023-01949-1 . PMC 10482697 . PMID 37474806.
- ^ Lino CA, Harper JC, Carney JP, Timlin JA (noviembre de 2018). "Implementación de CRISPR: una revisión de los desafíos y enfoques". Administración de fármacos . 25 (1): 1234–1257. doi :10.1080/10717544.2018.1474964. PMC 6058482 . PMID 29801422.
- ^ Li L, Hu S, Chen X (julio de 2018). "Sistemas de administración no viral para la edición genómica basada en CRISPR/Cas9: desafíos y oportunidades". Biomateriales . 171 : 207–218. doi :10.1016/j.biomaterials.2018.04.031. PMC 5944364 . PMID 29704747.
- ^ Jain PK, Lo JH, Rananaware S, Downing M, Panda A, Tai M, et al. (noviembre de 2019). "Entrega no viral del complejo CRISPR/Cas9 usando nanocomplejos CRISPR-GPS". Nanoscale . 11 (44): 21317–21323. doi :10.1039/C9NR01786K. PMC 7709491 . PMID 31670340.
- ^ ab Yip BH (mayo de 2020). "Avances recientes en las estrategias de administración de CRISPR/Cas9". Biomolecules . 10 (6): 839. doi : 10.3390/biom10060839 . PMC 7356196 . PMID 32486234.
- ^ Bak RO, Porteus MH (julio de 2017). "Integración de casetes de genes grandes mediada por CRISPR utilizando vectores donantes AAV". Cell Reports . 20 (3): 750–756. doi :10.1016/j.celrep.2017.06.064. PMC 5568673 . PMID 28723575.
- ^ Schmidt F, Grimm D (febrero de 2015). "Ingeniería genómica CRISPR y administración de genes virales: un caso de atracción mutua". Biotechnology Journal . 10 (2): 258–272. doi :10.1002/biot.201400529. PMID 25663455. S2CID 37653318.
- ^ Waxmonsky N (24 de septiembre de 2015). «CRISPR 101: Sistemas de expresión y métodos de administración en mamíferos» . Consultado el 11 de junio de 2018 .
- ^ Mishra T, Bhardwaj V, Ahuja N, Gadgil P, Ramdas P, Shukla S, Chande A (junio de 2022). "Mejora de la edición genómica con pérdida de función mediante CRISPR-Cas9 en células humanas concomitantemente con la inhibición de la señalización de TGF-β". Terapia molecular. Ácidos nucleicos . 28 : 202–218. doi :10.1016/j.omtn.2022.03.003. PMC 8961078. PMID 35402072 .
- ^ Adli M (mayo de 2018). "El kit de herramientas CRISPR para la edición genómica y más allá". Nature Communications . 9 (1): 1911. Bibcode :2018NatCo...9.1911A. doi :10.1038/s41467-018-04252-2. PMC 5953931 . PMID 29765029.
- ^ Svitashev S, Schwartz C, Lenderts B, Young JK, Mark Cigan A (noviembre de 2016). "Edición genómica en maíz dirigida por complejos de ribonucleoproteína CRISPR-Cas9". Nature Communications . 7 (1): 13274. Bibcode :2016NatCo...713274S. doi :10.1038/ncomms13274. PMC 5116081 . PMID 27848933.
- ^ Liang Z, Chen K, Li T, Zhang Y, Wang Y, Zhao Q, et al. (enero de 2017). "Edición eficiente del genoma del trigo panificable sin ADN utilizando complejos de ribonucleoproteína CRISPR/Cas9". Nature Communications . 8 (1): 14261. Bibcode :2017NatCo...814261L. doi :10.1038/ncomms14261. PMC 5253684 . PMID 28098143. S2CID 17028472.
- ^ Cui Z, Tian R, Huang Z, Jin Z, Li L, Liu J, et al. (marzo de 2022). "FrCas9 es un sistema CRISPR/Cas9 con alta fidelidad y eficiencia de edición". Nature Communications . 13 (1): 1425. Bibcode :2022NatCo..13.1425C. doi :10.1038/s41467-022-29089-8. PMC 8931148 . PMID 35301321.
- ^ Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK (enero de 2016). "Nucleasas CRISPR-Cas9 de alta fidelidad sin efectos fuera del objetivo detectables en todo el genoma". Nature . 529 (7587): 490–495. Bibcode :2016Natur.529..490K. doi :10.1038/nature16526. PMC 4851738 . PMID 26735016.
- ^ Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (enero de 2016). "Nucleasas Cas9 diseñadas racionalmente con especificidad mejorada". Science . 351 (6268): 84–88. Bibcode :2016Sci...351...84S. doi :10.1126/science.aad5227. PMC 4714946 . PMID 26628643.
- ^ Yin H, Song CQ, Suresh S, Kwan SY, Wu Q, Walsh S, et al. (marzo de 2018). "La Cas9 guiada parcialmente por ADN permite la edición del genoma con una actividad reducida fuera del objetivo". Nature Chemical Biology . 14 (3): 311–316. doi :10.1038/nchembio.2559. PMC 5902734 . PMID 29377001.
- ^ Riesenberg S, Helmbrecht N, Kanis P, Maricic T, Pääbo S (enero de 2022). "Las estructuras secundarias mejoradas de gRNA permiten la edición de sitios diana resistentes a la escisión de CRISPR-Cas9". Nature Communications . 13 (1): 489. Bibcode :2022NatCo..13..489R. doi :10.1038/s41467-022-28137-7. PMC 8789806 . PMID 35078986. S2CID 246281892.
- ^ Fu Y, Sander JD, Reyon D, Cascio VM, Joung JK (marzo de 2014). "Mejora de la especificidad de la nucleasa CRISPR-Cas utilizando ARN guía truncados". Nature Biotechnology . 32 (3): 279–284. doi :10.1038/nbt.2808. PMC 3988262 . PMID 24463574.
- ^ Thean DG, Chu HY, Fong JH, Chan BK, Zhou P, Kwok CC, et al. (abril de 2022). "La mutagénesis combinatoria acoplada al aprendizaje automático permite la ingeniería eficiente en recursos de las actividades del editor del genoma CRISPR-Cas9". Nature Communications . 13 (1): 2219. Bibcode :2022NatCo..13.2219T. doi :10.1038/s41467-022-29874-5. PMC 9039034 . PMID 35468907.
- ^ Cho SW, Kim S, Kim Y, Kweon J, Kim HS, Bae S, Kim JS (enero de 2014). "Análisis de los efectos fuera del objetivo de las endonucleasas y nickasas guiadas por ARN derivadas de CRISPR/Cas". Genome Research . 24 (1): 132–141. doi :10.1101/gr.162339.113. PMC 3875854 . PMID 24253446.
- ^ ab Oakes BL, Nadler DC, Flamholz A, Fellmann C, Staahl BT, Doudna JA, Savage DF (junio de 2016). "El perfil de puntos críticos de ingeniería identifica un interruptor alostérico CRISPR-Cas9". Nature Biotechnology . 34 (6): 646–651. doi :10.1038/nbt.3528. PMC 4900928 . PMID 27136077.
- ^ Nuñez JK, Harrington LB, Doudna JA (marzo de 2016). "Modulación química y biofísica de Cas9 para ingeniería genómica ajustable". ACS Chemical Biology . 11 (3): 681–688. doi :10.1021/acschembio.5b01019. PMID 26857072.
- ^ Zhou W, Deiters A (abril de 2016). "Control condicional de la función CRISPR/Cas9". Angewandte Chemie . 55 (18): 5394–5399. doi : 10.1002/anie.201511441 . PMID 26996256.
- ^ Polstein LR, Gersbach CA (marzo de 2015). "Un sistema CRISPR-Cas9 inducible por luz para el control de la activación génica endógena". Nature Chemical Biology . 11 (3): 198–200. doi :10.1038/nchembio.1753. PMC 4412021 . PMID 25664691.
- ^ Nihongaki Y, Yamamoto S, Kawano F, Suzuki H, Sato M (febrero de 2015). "Sistema de transcripción fotoactivable basado en CRISPR-Cas9". Química y biología . 22 (2): 169–174. doi : 10.1016/j.chembiol.2014.12.011 . PMID 25619936.
- ^ Wright AV, Sternberg SH, Taylor DW, Staahl BT, Bardales JA, Kornfeld JE, Doudna JA (marzo de 2015). "Diseño racional de un complejo enzimático Cas9 dividido". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (10): 2984–2989. Bibcode :2015PNAS..112.2984W. doi : 10.1073/pnas.1501698112 . PMC 4364227 . PMID 25713377.
- ^ Nihongaki Y, Kawano F, Nakajima T, Sato M (julio de 2015). "CRISPR-Cas9 fotoactivable para edición optogenética del genoma". Biotecnología de la Naturaleza . 33 (7): 755–760. doi :10.1038/nbt.3245. PMID 26076431. S2CID 205281536.
- ^ Hemphill J, Borchardt EK, Brown K, Asokan A, Deiters A (mayo de 2015). "Control óptico de la edición genética mediante CRISPR/Cas9". Revista de la Sociedad Química Estadounidense . 137 (17): 5642–5645. doi :10.1021/ja512664v. PMC 4919123. PMID 25905628 .
- ^ Jain PK, Ramanan V, Schepers AG, Dalvie NS, Panda A, Fleming HE, Bhatia SN (septiembre de 2016). "Desarrollo de CRISPR activado por luz utilizando ARN guía con protectores fotoescindibles". Angewandte Chemie . 55 (40): 12440–12444. doi :10.1002/anie.201606123. PMC 5864249 . PMID 27554600.
- ^ Davis KM, Pattanayak V, Thompson DB, Zuris JA, Liu DR (mayo de 2015). "Proteína Cas9 activada por moléculas pequeñas con especificidad de edición genómica mejorada". Nature Chemical Biology . 11 (5): 316–318. doi :10.1038/nchembio.1793. PMC 4402137 . PMID 25848930.
- ^ Liu KI, Ramli MN, Woo CW, Wang Y, Zhao T, Zhang X, et al. (noviembre de 2016). "Un sistema CRISPR-Cas9 inducible químicamente para el control rápido de la edición del genoma". Nature Chemical Biology . 12 (11): 980–987. doi :10.1038/nchembio.2179. PMID 27618190. S2CID 33891039.
- ^ Truong DJ, Kühner K, Kühn R, Werfel S, Engelhardt S, Wurst W, Ortiz O (julio de 2015). "Desarrollo de un sistema Cas9 dividido mediado por inteína para terapia génica". Nucleic Acids Research . 43 (13): 6450–6458. doi :10.1093/nar/gkv601. PMC 4513872 . PMID 26082496.
- ^ Zetsche B, Volz SE, Zhang F (febrero de 2015). "Una arquitectura de Cas9 dividida para la edición inducible del genoma y la modulación de la transcripción". Nature Biotechnology . 33 (2): 139–142. doi :10.1038/nbt.3149. PMC 4503468 . PMID 25643054.
- ^ González F, Zhu Z, Shi ZD, Lelli K, Verma N, Li QV, Huangfu D (agosto de 2014). "Una plataforma iCRISPR para la edición rápida, multiplexable e inducible del genoma en células madre pluripotentes humanas". Cell Stem Cell . 15 (2): 215–226. doi :10.1016/j.stem.2014.05.018. PMC 4127112 . PMID 24931489.
- ^ Dow LE, Fisher J, O'Rourke KP, Muley A, Kastenhuber ER, Livshits G, et al. (abril de 2015). "Edición genómica in vivo inducible con CRISPR-Cas9". Nature Biotechnology . 33 (4): 390–394. doi :10.1038/nbt.3155. PMC 4390466 . PMID 25690852.
- ^ Yu C, Liu Y, Ma T, Liu K, Xu S, Zhang Y, et al. (febrero de 2015). "Pequeñas moléculas mejoran la edición del genoma mediante CRISPR en células madre pluripotentes". Cell Stem Cell . 16 (2): 142–147. doi :10.1016/j.stem.2015.01.003. PMC 4461869 . PMID 25658371.
- ^ Schimmel J, Muñoz-Subirana N, Kool H, van Schendel R, van der Vlies S, Kamp JA, et al. (febrero de 2023). "Modulación de los resultados mutacionales y mejora de la edición precisa de genes en roturas inducidas por CRISPR-Cas9 mediante inhibición química de las vías de unión de extremos". Cell Reports . 42 (2): 112019. doi : 10.1016/j.celrep.2023.112019 . hdl : 1887/3753233 . PMID 36701230. S2CID 256273893.
- ^ Maruyama T, Dougan SK, Truttmann MC, Bilate AM, Ingram JR, Ploegh HL (mayo de 2015). "Aumento de la eficiencia de la edición precisa del genoma con CRISPR-Cas9 mediante la inhibición de la unión de extremos no homólogos". Nature Biotechnology . 33 (5): 538–542. doi :10.1038/nbt.3190. PMC 4618510 . PMID 25798939.
- ^ ab Ma X, Chen X, Jin Y, Ge W, Wang W, Kong L, et al. (abril de 2018). "Pequeñas moléculas promueven la edición genómica mediada por CRISPR-Cpf1 en células madre pluripotentes humanas". Nature Communications . 9 (1): 1303. Bibcode :2018NatCo...9.1303M. doi :10.1038/s41467-018-03760-5. PMC 5880812 . PMID 29610531.
- ^ Nishida K, Arazoe T, Yachie N, Banno S, Kakimoto M, Tabata M, et al. (septiembre de 2016). "Edición dirigida de nucleótidos utilizando sistemas inmunes adaptativos híbridos procarióticos y vertebrados". Ciencia . 353 (6305): aaf8729. doi : 10.1126/ciencia.aaf8729. PMID 27492474. S2CID 5122081.
- ^ Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR (mayo de 2016). "Edición programable de una base diana en ADN genómico sin escisión de ADN de doble cadena". Nature . 533 (7603): 420–424. Bibcode :2016Natur.533..420K. doi :10.1038/nature17946. PMC 4873371 . PMID 27096365.
- ^ Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR (noviembre de 2017). "Edición de bases programable de A•T a G•C en ADN genómico sin escisión del ADN". Nature . 551 (7681): 464–471. Bibcode :2017Natur.551..464G. doi :10.1038/nature24644. PMC 5726555 . PMID 29160308.
- ^ Chen L, Park JE, Paa P, Rajakumar PD, Prekop HT, Chew YT, et al. (marzo de 2021). "Edición genómica programable de C:G a G:C con proteínas de reparación por escisión de bases dirigidas por CRISPR-Cas9". Nature Communications . 12 (1): 1384. Bibcode :2021NatCo..12.1384C. doi :10.1038/s41467-021-21559-9. PMC 7925527 . PMID 33654077.
- ^ abc Kurata M, Yamamoto K, Moriarity BS, Kitagawa M, Largaespada DA (febrero de 2018). "Detección de bibliotecas CRISPR/Cas9 para el descubrimiento de objetivos farmacológicos". Revista de genética humana . 63 (2): 179–186. doi :10.1038/s10038-017-0376-9. PMID 29158600. S2CID 3308058.
- ^ Gasiunas G, Barrangou R, Horvath P, Siksnys V (septiembre de 2012). "El complejo de ribonucleoproteína Cas9-crRNA media la escisión específica del ADN para la inmunidad adaptativa en bacterias". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (39): E2579–E2586. doi : 10.1073/pnas.1208507109 . PMC 3465414 . PMID 22949671.
- ^ Satomura A, Nishioka R, Mori H, Sato K, Kuroda K, Ueda M (mayo de 2017). "Edición precisa de bases en todo el genoma mediante el sistema CRISPR Nickase en levadura". Scientific Reports . 7 (1): 2095. Bibcode :2017NatSR...7.2095S. doi :10.1038/s41598-017-02013-7. PMC 5437071 . PMID 28522803.
- ^ Hiranniramol K, Chen Y, Liu W, Wang X (mayo de 2020). "Diseño de sgRNA generalizable para una mejor eficiencia de edición de CRISPR/Cas9". Bioinformática . 36 (9): 2684–2689. doi :10.1093/bioinformatics/btaa041. PMC 7203743 . PMID 31971562.
- ^ abc Agrotis A, Ketteler R (24 de septiembre de 2015). "Una nueva era en genómica funcional utilizando CRISPR/Cas9 en el cribado de bibliotecas en matriz". Frontiers in Genetics . 6 : 300. doi : 10.3389/fgene.2015.00300 . PMC 4585242 . PMID 26442115.
- ^ Yu JS, Yusa K (julio de 2019). "Cribado CRISPR-Cas9 de todo el genoma en células de mamíferos". Métodos . 164–165: 29–35. doi :10.1016/j.ymeth.2019.04.015. PMID 31034882. S2CID 140275157.
- ^ ab Joung J, Konermann S, Gootenberg JS, Abudayyeh OO, Platt RJ, Brigham MD, et al. (abril de 2017). "Detección de activación transcripcional y eliminación de CRISPR-Cas9 a escala genómica". Nature Protocols . 12 (4): 828–863. doi :10.1038/nprot.2017.016. PMC 5526071 . PMID 28333914.
- ^ "Addgene: Pooled Libraries" (Agregar genes: bibliotecas agrupadas). www.addgene.org . Consultado el 31 de enero de 2020 .
- ^ McDade JR, Waxmonsky NC, Swanson LE, Fan M (julio de 2016). "Consideraciones prácticas para el uso de bibliotecas CRISPR lentivirales agrupadas". Protocolos actuales en biología molecular . 115 (1): 31.5.1–31.5.13. doi :10.1002/cpmb.8. PMID 27366891. S2CID 5055878.
- ^ Cheng AW, Wang H, Yang H, Shi L, Katz Y, Theunissen TW, et al. (octubre de 2013). "Activación multiplexada de genes endógenos mediante CRISPR-on, un sistema activador transcripcional guiado por ARN". Cell Research . 23 (10): 1163–1171. doi :10.1038/cr.2013.122. PMC 3790238 . PMID 23979020.
- ^ Gilbert LA, Horlbeck MA, Adamson B, Villalta JE, Chen Y, Whitehead EH, et al. (octubre de 2014). "Control de la represión y activación génica mediado por CRISPR a escala genómica". Cell . 159 (3): 647–661. doi :10.1016/j.cell.2014.09.029. PMC 4253859 . PMID 25307932.
- ^ ab Dow LE (octubre de 2015). "Modelado de enfermedades in vivo con CRISPR/Cas9". Tendencias en medicina molecular . 21 (10): 609–621. doi :10.1016/j.molmed.2015.07.006. PMC 4592741 . PMID 26432018.
- ^ Doudna J, Mali P (2016). CRISPR-Cas: Manual de laboratorio . Cold Spring Harbor, Nueva York. ISBN 978-1-62182-130-4. OCLC 922914104.
{{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace ) - ^ Zuo E, Huo X, Yao X, Hu X, Sun Y, Yin J, et al. (noviembre de 2017). "Eliminación de cromosomas dirigida mediada por CRISPR / Cas9". Biología del genoma . 18 (1): 224. doi : 10.1186/s13059-017-1354-4 . PMC 5701507 . PMID 29178945.
- "Se utiliza CRISPR para eliminar cromosomas específicos en un nuevo estudio" . Genome Web . 27 de noviembre de 2017.
- ^ Javed MR, Sadaf M, Ahmed T, Jamil A, Nawaz M, Abbas H, Ijaz A (diciembre de 2018). "Sistema CRISPR-Cas: historia y perspectivas como herramienta de edición genómica en microorganismos". revisión. Microbiología actual . 75 (12): 1675–1683. doi :10.1007/s00284-018-1547-4. PMID 30078067. S2CID 51920661.
- ^ DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM (abril de 2013). "Ingeniería genómica en Saccharomyces cerevisiae utilizando sistemas CRISPR-Cas". Nucleic Acids Res . 41 (7): 4336–43. doi :10.1093/nar/gkt135. PMC 3627607 . PMID 23460208.
- ^ Giersch RM, Finnigan GC (diciembre de 2017). "La levadura sigue siendo una bestia: diversas aplicaciones de la tecnología de edición CRISPR/Cas en S. cerevisiae". The Yale Journal of Biology and Medicine . 90 (4): 643–651. PMC 5733842 . PMID 29259528.
- ^ Dhamad AE, Lessner DJ (octubre de 2020). Atomi H (ed.). "Un sistema CRISPRi-dCas9 para arqueas y su uso para examinar la función génica durante la fijación de nitrógeno por Methanosarcina acetivorans". Microbiología aplicada y ambiental . 86 (21): e01402–20. Código Bibliográfico :2020ApEnM..86E1402D. doi :10.1128/AEM.01402-20. PMC 7580536 . PMID 32826220.
- ^ Raschmanová H, Weninger A, Glieder A, Kovar K, Vogl T (2018). "Implementación de tecnologías CRISPR-Cas en levaduras convencionales y no convencionales: estado actual y perspectivas futuras". revisión. Avances en biotecnología . 36 (3): 641–665. doi : 10.1016/j.biotechadv.2018.01.006 . PMID 29331410.
- ^ ab Ma D, Liu F (diciembre de 2015). "Edición genómica y sus aplicaciones en organismos modelo". revisión. Genómica, proteómica y bioinformática . 13 (6): 336–344. doi :10.1016/j.gpb.2015.12.001. PMC 4747648 . PMID 26762955.
- ^ Khurshid H, Jan SA, Shinwari ZK, Jamal M, Shah SH (2018). "Una era de edición genómica de plantas mediada por CRISPR/Cas9". revisión. Temas actuales en biología molecular . 26 : 47–54. doi : 10.21775/cimb.026.047 . PMID 28879855.
- ^ Simone BW, Martínez-Gálvez G, WareJoncas Z, Ekker SC (noviembre de 2018). "Pescando por la comprensión: desbloqueando la caja de herramientas del editor de genes del pez cebra". revisión. Métodos . 150 : 3–10. doi :10.1016/j.ymeth.2018.07.012. PMC 6590056 . PMID 30076892.
- ^ Singh P, Schimenti JC, Bolcun-Filas E (enero de 2015). "Guía práctica para genetistas de ratones sobre aplicaciones de CRISPR". revisión. Genética . 199 (1): 1–15. doi :10.1534/genetics.114.169771. PMC 4286675 . PMID 25271304.
- ^ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (mayo de 2018). "La función desubiquitinasa de A20 mantiene y repara la barrera endotelial después de una lesión vascular pulmonar". Descubrimiento de muerte celular . 4 (60): 60. doi :10.1038/s41420-018-0056-3. PMC 5955943 . PMID 29796309.
- ^ Birling MC, Herault Y, Pavlovic G (agosto de 2017). "Modelado de enfermedades humanas en roedores mediante edición genómica CRISPR/Cas9". Genoma de mamíferos . 28 (7–8): 291–301. doi :10.1007/s00335-017-9703-x. PMC 5569124 . PMID 28677007.
- ^ Gao X, Tao Y, Lamas V, Huang M, Yeh WH, Pan B, et al. (enero de 2018). "Tratamiento de la pérdida auditiva autosómica dominante mediante la administración in vivo de agentes de edición genómica". Nature . 553 (7687): 217–221. Bibcode :2018Natur.553..217G. doi :10.1038/nature25164. PMC 5784267 . PMID 29258297.
- ^ Kadam US, Shelake RM, Chavhan RL, Suprasanna P (octubre de 2018). "Preocupaciones con respecto a la actividad 'fuera del objetivo' de las endonucleasas de edición del genoma". revisión. Fisiología vegetal y bioquímica . 131 : 22–30. doi :10.1016/j.plaphy.2018.03.027. PMID 29653762. S2CID 4846191.
- ^ Kimberland ML, Hou W, Alfonso-Pecchio A, Wilson S, Rao Y, Zhang S, Lu Q (octubre de 2018). "Estrategias para controlar los efectos fuera del objetivo de CRISPR/Cas9 y las variaciones biológicas en experimentos de edición del genoma de mamíferos". revisión. Journal of Biotechnology . 284 : 91–101. doi :10.1016/j.jbiotec.2018.08.007. PMID 30142414. S2CID 52078796.
- ^ van Erp PB, Bloomer G, Wilkinson R, Wiedenheft B (junio de 2015). "La historia y el impacto en el mercado de las nucleasas guiadas por ARN CRISPR". Current Opinion in Virology . 12 : 85–90. doi :10.1016/j.coviro.2015.03.011. PMC 4470805 . PMID 25914022.
- ^ Maggio I, Gonçalves MA (mayo de 2015). "Edición genómica en la encrucijada de la entrega, la especificidad y la fidelidad". Tendencias en biotecnología . 33 (5): 280–291. doi : 10.1016/j.tibtech.2015.02.011 . PMID 25819765.
- ^ Rath D, Amlinger L, Rath A, Lundgren M (octubre de 2015). "El sistema inmunológico CRISPR-Cas: biología, mecanismos y aplicaciones". Biochimie . 117 : 119–128. doi : 10.1016/j.biochi.2015.03.025 . PMID 25868999.
- ^ "¿Qué es CRISPR? ¿Cómo funciona? ¿Es edición genética?". LiveScience.Tech . 2018-04-30. Archivado desde el original el 2020-02-06 . Consultado el 2020-02-06 .
- ^ ab Freedman BS, Brooks CR, Lam AQ, Fu H, Morizane R, Agrawal V, et al. (octubre de 2015). "Modelado de la enfermedad renal con organoides renales mutantes en CRISPR derivados de esferoides epipluripotentes humanos". Nature Communications . 6 : 8715. Bibcode :2015NatCo...6.8715F. doi :10.1038/ncomms9715. PMC 4620584 . PMID 26493500.
- ^ Cruz NM, Song X, Czerniecki SM, Gulieva RE, Churchill AJ, Kim YK, et al. (noviembre de 2017). "La cistogénesis de organoides revela un papel crítico del microambiente en la enfermedad renal poliquística humana". Nature Materials . 16 (11): 1112–1119. Bibcode :2017NatMa..16.1112C. doi :10.1038/nmat4994. PMC 5936694 . PMID 28967916.
- ^ Kim YK, Refaeli I, Brooks CR, Jing P, Gulieva RE, Hughes MR, et al. (diciembre de 2017). "Los organoides renales humanos modificados genéticamente revelan mecanismos de enfermedad en el desarrollo de podocitos". Células madre . 35 (12): 2366–2378. doi :10.1002/stem.2707. PMC 5742857 . PMID 28905451.
- ^ Bellin M, Casini S, Davis RP, D'Aniello C, Haas J, Ward-van Oostwaard D, et al. (diciembre de 2013). "Pares de células madre pluripotentes humanas isogénicas revelan el papel de una mutación KCNH2 en el síndrome de QT largo". The EMBO Journal . 32 (24): 3161–3175. doi :10.1038/emboj.2013.240. PMC 3981141 . PMID 24213244.
- ^ Cai L, Fisher AL, Huang H, Xie Z (diciembre de 2016). "Edición genómica mediada por CRISPR y enfermedades humanas". Genes & Diseases . 3 (4): 244–251. doi :10.1016/j.gendis.2016.07.003. PMC 6150104 . PMID 30258895.
- ^ "Siete enfermedades que la tecnología CRISPR podría curar". Labiotech.eu . 2018-06-25 . Consultado el 2018-08-22 .
- ^ ab "CRISPR/Cas9 y cáncer". Noticias sobre inmunooncología . 27 de abril de 2018. Consultado el 18 de febrero de 2019 .
- ^ Crossley M (febrero de 2021). «La nueva tecnología CRISPR podría revolucionar la terapia genética y ofrecer nuevas esperanzas a las personas con enfermedades genéticas». The Conversation . Consultado el 3 de febrero de 2021 .
- ^ Cromer MK, Camarena J, Martin RM, Lesch BJ, Vakulskas CA, Bode NM, et al. (abril de 2021). "El reemplazo genético de α-globina por β-globina restaura el equilibrio de la hemoglobina en células madre y progenitoras hematopoyéticas derivadas de β-talasemia". Nature Medicine . 27 (4): 677–687. doi :10.1038/s41591-021-01284-y. PMC 8265212 . PMID 33737751.
- ^ Xie F, Ye L, Chang JC, Beyer AI, Wang J, Muench MO, Kan YW (septiembre de 2014). "Corrección genética perfecta de mutaciones de β-talasemia en iPSC específicas de pacientes utilizando CRISPR/Cas9 y piggyBac". Genome Research . 24 (9): 1526–1533. doi :10.1101/gr.173427.114. PMC 4158758 . PMID 25096406.
- ^ ab Frangoul H, Altshuler D, Cappellini MD, Chen YS, Domm J, Eustace BK, et al. (enero de 2021). "Edición genética CRISPR-Cas9 para la anemia de células falciformes y la β-talasemia". The New England Journal of Medicine . 384 (3): 252–260. doi : 10.1056/NEJMoa2031054 . PMID 33283989. S2CID 227521558.
- ^ Dever DP, Bak RO, Reinisch A, Camarena J, Washington G, Nicolas CE, et al. (noviembre de 2016). "Focalización del gen de la β-globina mediante CRISPR/Cas9 en células madre hematopoyéticas humanas". Nature . 539 (7629): 384–389. Bibcode :2016Natur.539..384D. doi :10.1038/nature20134. PMC 5898607 . PMID 27820943.
- ^ "Desarrollan terapia celular CRISPR 'de una sola dosis' para la hemofilia". GEN . 2018-05-02 . Consultado el 2018-08-22 .
- ^ Marangi M, Pistritto G (20 de abril de 2018). "Estrategias terapéuticas innovadoras para la fibrosis quística: avanzando hacia la técnica CRISPR". Frontiers in Pharmacology . 9 : 396. doi : 10.3389/fphar.2018.00396 . PMC 5920621 . PMID 29731717.
- ^ Bengtsson NE, Hall JK, Odom GL, Phelps MP, Andrus CR, Hawkins RD, et al. (febrero de 2017). "La edición del gen de la distrofina CRISPR/Cas9 específico del músculo mejora la patofisiología en un modelo de ratón para la distrofia muscular de Duchenne". Nature Communications . 8 : 14454. Bibcode :2017NatCo...814454B. doi :10.1038/ncomms14454. PMC 5316861 . PMID 28195574.
- ^ Eisenstein M (mayo de 2018). "CRISPR se enfrenta a la enfermedad de Huntington". Nature . 557 (7707): S42–S43. Bibcode :2018Natur.557S..42E. doi : 10.1038/d41586-018-05177-y . PMID 29844549.
- ^ Dabrowska M, Juzwa W, Krzyzosiak WJ, Olejniczak M (2018). "Extirpación precisa del tracto CAG del gen huntingtina por medio de nickasas Cas9". Frontiers in Neuroscience . 12 : 75. doi : 10.3389/fnins.2018.00075 . PMC 5834764 . PMID 29535594.
- ^ King A (marzo de 2018). "Una edición CRISPR para enfermedades cardíacas". Nature . 555 (7695): S23–S25. Bibcode :2018Natur.555.....K. doi : 10.1038/d41586-018-02482-4 . PMID 29517035.
- ^ Scudellari M (julio de 2019). "Mosquitos autodestructivos y roedores esterilizados: la promesa de los impulsores genéticos". Nature . 571 (7764): 160–162. Bibcode :2019Natur.571..160S. doi : 10.1038/d41586-019-02087-5 . PMID 31289403.
- ^ Abrahimi P, Chang WG, Kluger MS, Qyang Y, Tellides G, Saltzman WM, Pober JS (julio de 2015). "Disrupción génica eficiente en células endoteliales humanas primarias cultivadas mediante CRISPR/Cas9". Circulation Research . 117 (2): 121–128. doi :10.1161/CIRCRESAHA.117.306290. PMC 4490936 . PMID 25940550.
- ^ "Un año después, el primer paciente que se sometió a un tratamiento de edición genética para la anemia de células falciformes está prosperando". NPR.org . Consultado el 3 de febrero de 2021 .
- ^ "Tecnología CRISPR para curar la anemia falciforme". ScienceDaily . Consultado el 3 de febrero de 2021 .
- ^ Uddin F, Rudin CM, Sen T (agosto de 2020). "Terapia génica CRISPR: aplicaciones, limitaciones e implicaciones para el futuro". Frontiers in Oncology . 10 : 1387. doi : 10.3389/fonc.2020.01387 . PMC 7427626 . PMID 32850447.
- ^ Jensen TI, Axelgaard E, Bak RO (junio de 2019). "Edición genética terapéutica en trastornos hematológicos con CRISPR/Cas9". British Journal of Haematology . 185 (5): 821–835. doi : 10.1111/bjh.15851 . PMID 30864164. S2CID 76663873.
- ^ Reardon S (2016). "El primer ensayo clínico de CRISPR recibe luz verde de un panel estadounidense". Nature . doi :10.1038/nature.2016.20137. S2CID 89466280.
- ^ Rosenblum D, Gutkin A, Kedmi R, Ramishetti S, Veiga N, Jacobi AM, et al. (noviembre de 2020). "Edición genómica CRISPR-Cas9 utilizando nanopartículas lipídicas específicas para la terapia del cáncer". Science Advances . 6 (47): eabc9450. Bibcode :2020SciA....6.9450R. doi : 10.1126/sciadv.abc9450 . PMC 7673804 . PMID 33208369.
- ^ abcd "Ensayos clínicos CRISPR: una actualización de 2022". Instituto de Genómica Innovadora (IGI) . Consultado el 2 de mayo de 2022 .
- ^ "Edición básica: una terapia revolucionaria cura el cáncer incurable de una niña". BBC News . 11 de diciembre de 2022 . Consultado el 11 de diciembre de 2022 .
- ^ "Las 'células de diseño' revierten el cáncer de un niño de un año". BBC News . 2015-11-05 . Consultado el 2022-12-11 .
- ^ 12 de mayo (12 de mayo de 2022). "Investigadores de Columbia Británica lanzan un ensayo clínico para la primera terapia basada en células madre genéticamente modificadas para la diabetes tipo 1". UBC News . Consultado el 8 de diciembre de 2023 .
{{cite web}}: CS1 maint: nombres numéricos: lista de autores ( enlace ) - ^ "CRISPR Therapeutics y ViaCyte, Inc. anuncian que se administró la primera dosis a un paciente en…". CRISPR . Consultado el 2 de mayo de 2022 .
- ^ Instituto Nacional sobre el Abuso de Drogas (14 de febrero de 2020). "La terapia antirretroviral combinada con la edición genética CRISPR puede eliminar la infección por VIH en ratones". Instituto Nacional sobre el Abuso de Drogas . Consultado el 18 de abril de 2022 .
- ^ "El primer ensayo clínico de una terapia contra el VIH basada en CRISPR se basa en una investigación de vanguardia en la Facultad de Medicina Lewis Katz | Facultad de Medicina Lewis Katz de la Universidad de Temple". medicine.temple.edu . Consultado el 18 de abril de 2022 .
- ^ Excision BioTherapeutics (24 de marzo de 2022). "Estudio de fase 1/2a, de cohorte secuencial, de dosis única ascendente sobre la seguridad, tolerabilidad, biodistribución y farmacodinamia de EBT 101 en adultos avirémicos infectados por VIH-1 que reciben terapia antirretroviral estable".
- ^ "Tres personas fueron sometidas a una edición genética con el fin de curar su VIH. Se desconoce el resultado". MIT Technology Review . Consultado el 20 de marzo de 2024 .
- ^ "El VIH en cultivos celulares puede eliminarse por completo utilizando la tecnología de edición genética CRISPR-Cas, lo que aumenta las esperanzas de curación". EurekAlert! . Consultado el 20 de marzo de 2024 .
- ^ "Los científicos dicen que pueden eliminar el VIH de las células". 2024-03-20 . Consultado el 2024-03-20 .
- ^ Gomaa AA, Klumpe HE, Luo ML, Selle K, Barrangou R, Beisel CL (enero de 2014). "Eliminación programable de cepas bacterianas mediante el uso de sistemas CRISPR-Cas dirigidos al genoma". mBio . 5 (1): e00928–e00913. doi :10.1128/mBio.00928-13. PMC 3903277 . PMID 24473129.
- ^ Citorik RJ, Mimee M, Lu TK (noviembre de 2014). "Antimicrobianos específicos de secuencia que utilizan nucleasas guiadas por ARN de administración eficiente". Nature Biotechnology . 32 (11): 1141–1145. doi :10.1038/nbt.3011. hdl :1721.1/100834. PMC 4237163 . PMID 25240928.
- ^ ab Gholizadeh P, Aghazadeh M, Asgharzadeh M, Kafil HS (noviembre de 2017). "Supresión del sistema inmunitario adaptativo CRISPR/Cas en infecciones bacterianas". Revista Europea de Microbiología Clínica y Enfermedades Infecciosas . 36 (11): 2043–2051. doi :10.1007/s10096-017-3036-2. PMID 28601970. S2CID 22716314.
- ^ Gibney E (enero de 2018). «Qué esperar en 2018: la ciencia en el nuevo año». Nature . 553 (7686): 12–13. Bibcode :2018Natur.553...12G. doi : 10.1038/d41586-018-00009-5 . PMID 29300040.
- ^ Taylor P (3 de enero de 2019). "J&J adquiere participación en los antimicrobianos 'Pac-Man' basados en CRISPR de Locus". Fierce Biotech . Consultado el 27 de febrero de 2019 .
- ^ Reardon S (junio de 2017). "Los virus modificados provocan la muerte de bacterias resistentes a los antibióticos". Nature . 546 (7660): 586–587. Bibcode :2017Natur.546..586R. doi : 10.1038/nature.2017.22173 . PMID 28661508.
- ^ van Diemen FR, Kruse EM, Hooykaas MJ, Bruggeling CE, Schürch AC, van Ham PM, et al. (junio de 2016). "La edición genómica de los herpesvirus mediada por CRISPR/Cas9 limita las infecciones productivas y latentes". PLOS Pathogens . 12 (6): e1005701. doi : 10.1371/journal.ppat.1005701 . PMC 4928872 . PMID 27362483.
- "Uso de CRISPR para combatir infecciones virales: ¿una nueva forma de tratar el herpes?". PLOS Media . 4 de agosto de 2016 – vía YouTube.
- ^ Personal de abc Science News (17 de diciembre de 2015). "Y el descubrimiento científico del año es..." news.sciencemag.org . Consultado el 21 de diciembre de 2015 .
- ^ Mullin E. "El uso de CRISPR en cerdos podría hacer que sus órganos sean más seguros para el trasplante humano". MIT Technology Review . Consultado el 9 de septiembre de 2017 .
- ^ Swiech L, Heidenreich M, Banerjee A, Habib N, Li Y, Trombetta J, et al. (enero de 2015). "Interrogación in vivo de la función génica en el cerebro de los mamíferos mediante CRISPR-Cas9". Nature Biotechnology . 33 (1): 102–106. doi :10.1038/nbt.3055. PMC 4492112 . PMID 25326897.
- ^ Feijo S. "Cómo los neurocientíficos de Brown están usando CRISPR para acelerar la investigación del cerebro y más". Universidad de Brown . Consultado el 8 de diciembre de 2023 .
- ^ ab De Plano LM, Calabrese G, Conoci S, Guglielmino SP, Oddo S, Caccamo A (agosto de 2022). "Aplicaciones de CRISPR-Cas9 en la enfermedad de Alzheimer y trastornos relacionados". Revista internacional de ciencias moleculares . 23 (15): 8714. doi : 10.3390/ijms23158714 . PMC 9368966 . PMID 35955847.
- ^ Erkut E, Yokota T (febrero de 2022). "Terapéutica CRISPR para la distrofia muscular de Duchenne". Revista internacional de ciencias moleculares . 23 (3): 1832. doi : 10.3390/ijms23031832 . PMC 8836469 . PMID 35163754.
- ^ Yamagata T, Raveau M, Kobayashi K, Miyamoto H, Tatsukawa T, Ogiwara I, et al. (julio de 2020). "La activación del gen Scn1a basado en CRISPR/dCas9 en neuronas inhibidoras mejora los fenotipos epilépticos y conductuales de ratones modelo con síndrome de Dravet". Neurobiología de la enfermedad . 141 : 104954. doi : 10.1016/j.nbd.2020.104954 . PMID 32445790. S2CID 218762990.
- ^ Artero-Castro A, Long K, Bassett A, Ávila-Fernandez A, Cortón M, Vidal-Puig A, et al. (febrero de 2021). "La corrección genética recupera la fagocitosis en el epitelio pigmentario de la retina derivado de células madre pluripotentes inducidas por retinitis pigmentosa humana". Revista Internacional de Ciencias Moleculares . 22 (4): 2092. doi : 10.3390/ijms22042092 . PMC 7923278 . PMID 33672445.
- ^ "Editas hace una pausa en el programa LCA10 en busca de un socio".
- ^ "Edición genética mediada por CRISPR-Cas9: una revolución en la ingeniería genómica". Biomarcadores y aplicaciones . 1 (2). 2017-09-12. doi : 10.29011/2576-9588.100111 (inactivo 2024-03-27). Archivado desde el original el 2020-08-18 . Consultado el 2023-12-10 .
{{cite journal}}: CS1 maint: DOI inactivo a partir de marzo de 2024 ( enlace ) - ^ Huai C, Jia C, Sun R, Xu P, Min T, Wang Q, et al. (julio de 2017). "Corrección de genes somáticos y de línea germinal mediada por CRISPR/Cas9 para restaurar la hemostasia en ratones con hemofilia B". Genética humana . 136 (7): 875–883. doi :10.1007/s00439-017-1801-z. PMID 28508290. S2CID 253979773.
- ^ Zhu H, Li C, Gao C (noviembre de 2020). "Aplicaciones de CRISPR-Cas en agricultura y biotecnología vegetal". Nature Reviews. Biología celular molecular . 21 (11): 661–677. doi :10.1038/s41580-020-00288-9. PMID 32973356. S2CID 221918795.
- ^ Lu K, Wu B, Wang J, Zhu W, Nie H, Qian J, et al. (octubre de 2018). "El bloqueo del transportador de aminoácidos OsAAP3 mejora el rendimiento del grano al promover el crecimiento de brotes y aumentar el número de macollos en el arroz". Revista de biotecnología vegetal . 16 (10): 1710–1722. doi : 10.1111/pbi.12907 . PMC 6131477 . PMID 29479779. S2CID 3506253.
- ^ Sánchez-León S, Gil-Humanes J, Ozuna CV, Giménez MJ, Sousa C, Voytas DF, Barro F (abril de 2018). "Trigo no transgénico y bajo en gluten diseñado con CRISPR / Cas9". Revista de Biotecnología Vegetal . 16 (4): 902–910. doi : 10.1111/pbi.12837 . PMC 5867031 . PMID 28921815. S2CID 4376988.
- ^ Wang Y, Cheng X, Shan Q, Zhang Y, Liu J, Gao C, Qiu JL (septiembre de 2014). "La edición simultánea de tres homoeoalelos en trigo panificable hexaploide confiere resistencia hereditaria al mildiú polvoroso". Nature Biotechnology . 32 (9): 947–951. doi :10.1038/nbt.2969. PMID 25038773. S2CID 205280231.
- ^ Demirci S, Leonard A, Haro-Mora JJ, Uchida N, Tisdale JF (2019). "CRISPR/Cas9 para la enfermedad de células falciformes: aplicaciones, posibilidades futuras y desafíos". Avances en medicina y biología experimentales . 1144 . Springer International Publishing: 37–52. doi :10.1007/5584_2018_331. ISBN 978-3-030-17588-7. Número de identificación personal 30715679. Número de identificación personal 73432066.
- ^ Fortunato F, Rossi R, Falzarano MS, Ferlini A (febrero de 2021). "Enfoques terapéuticos innovadores para la distrofia muscular de Duchenne". Revista de medicina clínica . 10 (4): 820. doi : 10.3390/jcm10040820 . PMC 7922390 . PMID 33671409.
- ^ Stadtmauer EA, Fraietta JA, Davis MM, Cohen AD, Weber KL, Lancaster E, et al. (febrero de 2020). "Células T modificadas mediante ingeniería CRISPR en pacientes con cáncer refractario". Science . 367 (6481). doi : 10.1126/science.aba7365 . PMC 11249135 . PMID 32029687. S2CID 211048335.
- ^ Jiang F, Doudna JA (mayo de 2017). "Estructuras y mecanismos de CRISPR-Cas9". Revisión anual de biofísica . 46 (1): 505–529. doi : 10.1146/annurev-biophys-062215-010822 . PMID: 28375731. S2CID : 274633.
- ^ Zhang S, Shen J, Li D, Cheng Y (2021). "Estrategias en la administración de la ribonucleoproteína Cas9 para la edición del genoma mediante CRISPR/Cas9". Theranostics . 11 (2): 614–648. doi : 10.7150/thno.47007 . PMC 7738854 . PMID 33391496. S2CID 226215184.
- ^ Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, et al. (abril de 2017). "Detección de ácidos nucleicos con CRISPR-Cas13a/C2c2". Science . 356 (6336): 438–442. doi :10.1126/science.aam9321. PMC 5526198 . PMID 28408723.
- ^ ab Dhamad AE, Abdal Rhida MA (octubre de 2020). "COVID-19: métodos de detección molecular y serológica". PeerJ . 8 : e10180. doi : 10.7717/peerj.10180 . PMC 7547594 . PMID 33083156.
- ^ Reis AC, Halper SM, Vezeau GE, Cetnar DP, Hossain A, Clauer PR, Salis HM (noviembre de 2019). "Represión simultánea de múltiples genes bacterianos utilizando matrices de sgRNA extralargas no repetitivas". Nature Biotechnology . 37 (11): 1294–1301. doi :10.1038/s41587-019-0286-9. OSTI 1569832. PMID 31591552. S2CID 203852115. Archivado desde el original el 2022-09-20 . Consultado el 2023-08-13 .
- ^ Gu W, Crawford ED, O'Donovan BD, Wilson MR, Chow ED, Retallack H, DeRisi JL (marzo de 2016). "Reducción de secuencias abundantes por hibridación (DASH): uso de Cas9 para eliminar especies no deseadas de alta abundancia en bibliotecas de secuenciación y aplicaciones de conteo molecular". Genome Biology . 17 (1): 41. doi : 10.1186/s13059-016-0904-5 . PMC 4778327 . PMID 26944702.
- ^ Gootenberg JS, Abudayyeh OO, Lee JW, Essletzbichler P, Dy AJ, Joung J, Verdine V, Donghia N, Daringer NM, Freije CA, Myhrvold C, Bhattacharyya RP, Livny J, Regev A, Koonin EV, Hung DT, Sabeti PC, Collins JJ, Zhang F (abril de 2017). "Detección de ácidos nucleicos con CRISPR-Cas13a/C2c2". Science . 356 (6336): 438–442. Bibcode :2017Sci...356..438G. doi :10.1126/science.aam9321. PMC 5526198 . PMID 28408723.
- ^ Chen JS, Ma E, Harrington LB, Da Costa M, Tian X, Palefsky JM, Doudna JA (abril de 2018). "La unión a la diana CRISPR-Cas12a desencadena una actividad indiscriminada de la ADNasa monocatenaria". Science . 360 (6387): 436–439. Bibcode :2018Sci...360..436C. doi :10.1126/science.aar6245. PMC 6628903 . PMID 29449511.
- ^ ab Shami A, Mostafa M, Abd-Elsalam KA (2021). "Aplicaciones de CRISPR en bacteriología vegetal: perspectivas actuales y futuras". En Abd-Elsalam KA, Lim KT (eds.). Sistemas CRISPR y RNAi: enfoques nanobiotecnológicos para el mejoramiento y la protección de plantas . Ámsterdam : Elsevier . pp. xxxvi+804. ISBN 978-0-12-821911-9.OCLC 1240283203 .
- ^ Iwasaki RS, Batey RT (septiembre de 2020). "SPRINT: una plataforma basada en Cas13a para la detección de moléculas pequeñas". Investigación en ácidos nucleicos . 48 (17): e101. doi : 10.1093/nar/gkaa673 . PMC 7515716 . PMID 32797156.
- ^ Broughton JP, Deng X, Yu G, Fasching CL, Servellita V, Singh J, Miao X, Streithorst JA, Granados A, Sotomayor-Gonzalez A, Zorn K, Gopez A, Hsu E, Gu W, Miller S, Pan CY, Guevara H, Wadford DA, Chen JS, Chiu CY (julio de 2020). "Detección del SARS-CoV-2 basada en CRISPR-Cas12". Nature Biotechnology . 38 (7): 870–874. doi : 10.1038/s41587-020-0513-4 . PMC 9107629 . PMID 32300245.
- ^ Nguyen LT, Smith BM, Jain PK (septiembre de 2020). "La mejora de la actividad de transescisión de Cas12a con ARNcr diseñado permite la detección de ácidos nucleicos amplificados". Nature Communications . 11 (1): 4906. Bibcode :2020NatCo..11.4906N. doi : 10.1038/s41467-020-18615-1 . PMC 7528031 . PMID 32999292.
- ^ Joung J, Ladha A, Saito M, Kim NG, Woolley AE, Segel M, Barretto RP, Ranu A, Macrae RK, Faure G, Ioannidi EI, Krajeski RN, Bruneau R, Huang MW, Yu XG, Li JZ, Walker BD, Hung DT, Greninger AL, Jerome KR, Gootenberg JS, Abudayyeh OO, Zhang F (octubre de 2020). "Detección del SARS-CoV-2 con la prueba de un solo uso de SHERLOCK". The New England Journal of Medicine . 383 (15): 1492–1494. doi :10.1056/NEJMc2026172. PMC 7510942 . PMID 32937062.
- ^ Patchsung M, Jantarug K, Pattama A, Aphicho K, Suraritdechachai S, Meesawat P, Sappakhaw K, Leelahakorn N, Ruenkam T, Wongsatit T, Athipanyasilp N, Eiamthong B, Lakkanasirorat B, Phoodokmai T, Niljianskul N, Pakotiprapha D, Chanarat S, Homchan A, Tinikul R, Kamutira P, Phiwkaow K, Soithongcharoen S, Kantiwiriyawanitch C, Pongsupasa V, Trisrivirat D, Jaroensuk J, Wongnate T, Maenpuen S, Chaiyen P, Kamnerdnakta S, Swangsri J, Chuthapisith S, Sirivatanauksorn Y, Chaimayo C, Sutthent R, Kantakamalakul W, Joung J, Ladha A, Jin X, Gootenberg JS, Abudayyeh OO, Zhang F, Horthongkham N, Uttamapinant C (diciembre de 2020). "Validación clínica de un ensayo basado en Cas13 para la detección de ARN del SARS-CoV-2". Ingeniería Biomédica de la Naturaleza . 4 (12): 1140-1149. doi : 10.1038/s41551-020-00603-x . hdl : 1721.1/138450.2 . PMID 32848209.
- ^ Konwarh R (septiembre de 2020). "¿Puede la tecnología CRISPR/Cas ser una estrategia acertada contra el fiasco de la COVID-19? Perspectivas y dificultades". Frontiers in Molecular Biosciences . 7 : 557377. doi : 10.3389/fmolb.2020.557377 . PMC 7511716 . PMID 33134311.
- ^ Shademan B, Nourazarian A, Hajazimian S, Isazadeh A, Biray Avci C, Oskouee MA (11 de enero de 2022). "Tecnología CRISPR en la detección y el tratamiento del SARS-CoV-2 basados en la edición genética". Frontiers in Molecular Biosciences . 8 : 772788. doi : 10.3389/fmolb.2021.772788 . PMC 8787289 . PMID 35087864.
- ^ Kellner MJ, Koob JG, Gootenberg JS, Abudayyeh OO, Zhang F (octubre de 2019). "SHERLOCK: detección de ácidos nucleicos con nucleasas CRISPR". Nature Protocols . 14 (10): 2986–3012. doi :10.1038/s41596-019-0210-2. PMC 6956564 . PMID 31548639.
- ^ Ding X, Yin K, Li Z, Liu C (marzo de 2020). "Ensayo dual CRISPR-Cas12a todo en uno (AIOD-CRISPR): un caso de detección rápida, ultrasensible y visual del nuevo coronavirus SARS-CoV-2 y el virus del VIH". bioRxiv . doi :10.1101/2020.03.19.998724. PMC 7239053 . PMID 32511323.
- ^ Pinson A, Xing L, Namba T, Kalebic N, Peters J, Oegema CE, et al. (septiembre de 2022). "La TKTL1 humana implica una mayor neurogénesis en el neocórtex frontal de los humanos modernos que en los neandertales". Science . 377 (6611): eabl6422. doi :10.1126/science.abl6422. PMID 36074851. S2CID 252161562.
- ^ Mora-Bermúdez F, Kanis P, Macak D, Peters J, Naumann R, Xing L, et al. (julio de 2022). "Metafase más larga y menos errores de segregación cromosómica en el desarrollo cerebral de los humanos modernos que en los neandertales". Science Advances . 8 (30): eabn7702. Bibcode :2022SciA....8N7702M. doi :10.1126/sciadv.abn7702. PMC 9337762 . PMID 35905187.
- ^ "Los científicos descubren cómo los humanos desarrollan cerebros más grandes que otros simios". phys.org . Archivado desde el original el 19 de abril de 2021 . Consultado el 19 de abril de 2021 .
- ^ Benito-Kwiecinski S, Giandomenico SL, Sutcliffe M, Riis ES, Freire-Pritchett P, Kelava I, et al. (abril de 2021). "Una transición temprana de la forma de las células impulsa la expansión evolutiva del prosencéfalo humano". Celúla . 184 (8): 2084–2102.e19. doi : 10.1016/j.cell.2021.02.050 . PMC 8054913 . PMID 33765444.
 Disponible bajo CC BY 4.0 Archivado el 16 de octubre de 2017 en Wayback Machine .
Disponible bajo CC BY 4.0 Archivado el 16 de octubre de 2017 en Wayback Machine . - ^ Sawal I. «Minicerebros modificados genéticamente con CRISPR para que sean similares a los de los neandertales». New Scientist . Archivado desde el original el 10 de marzo de 2021. Consultado el 7 de marzo de 2021 .
- ^ Trujillo CA, Rice ES, Schaefer NK, Chaim IA, Wheeler EC, Madrigal AA, et al. (febrero de 2021). "La reintroducción de la variante arcaica de NOVA1 en organoides corticales altera el desarrollo neurológico". Science . 371 (6530). doi :10.1126/science.aax2537. PMC 8006534 . PMID 33574182.
- ^ Maricic T, Helmbrecht N, Riesenberg S, Macak D, Kanis P, Lackner M, et al. (octubre de 2021). "Comentario sobre "La reintroducción de la variante arcaica de NOVA1 en organoides corticales altera el desarrollo neurológico"". Ciencia . 374 (6565): eabi6060. doi :10.1126/science.abi6060. PMID 34648345. S2CID 238990790.
- ^ Herai RH, Szeto RA, Trujillo CA, Muotri AR (octubre de 2021). "Respuesta al comentario sobre "La reintroducción de la variante arcaica de NOVA1 en organoides corticales altera el neurodesarrollo"". Ciencia . 374 (6565): eabi9881. doi :10.1126/science.abi9881. PMID 34648331. S2CID 238990560.
- ^ Dominguez AA, Lim WA, Qi LS (enero de 2016). "Más allá de la edición: reutilización de CRISPR-Cas9 para la regulación y la interrogación precisas del genoma". Nature Reviews. Molecular Cell Biology . 17 (1): 5–15. doi :10.1038/nrm.2015.2. PMC 4922510 . PMID 26670017.
- ^ Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelson T, et al. (enero de 2014). "Cribado de knockout CRISPR-Cas9 a escala genómica en células humanas". Science . 343 (6166): 84–87. Bibcode :2014Sci...343...84S. doi :10.1126/science.1247005. PMC 4089965 . PMID 24336571.
- ^ ab Pennisi E (agosto de 2013). "La locura de CRISPR". News Focus. Science . 341 (6148): 833–836. Bibcode :2013Sci...341..833P. doi :10.1126/science.341.6148.833. PMID 23970676.
- ^ ab Zimmer C (3 de junio de 2016). "Los científicos encuentran una forma de edición genética CRISPR con nuevas capacidades". The New York Times . ISSN 0362-4331 . Consultado el 10 de junio de 2016 .
- ^ Pickar-Oliver A, Gersbach CA (agosto de 2019). "La próxima generación de tecnologías y aplicaciones CRISPR-Cas". Nature Reviews. Molecular Cell Biology . 20 (8): 490–507. doi :10.1038/s41580-019-0131-5. PMC 7079207 . PMID 31147612.
- ^ Basak J, Nithin C (2015). "Apuntar a ARN no codificantes en plantas con la tecnología CRISPR-Cas es un desafío que vale la pena aceptar". Frontiers in Plant Science . 6 : 1001. doi : 10.3389/fpls.2015.01001 . PMC 4652605 . PMID 26635829.
- ^ ab Woolf TM, Chase JM, Stinchcomb DT (agosto de 1995). "Hacia la edición terapéutica de secuencias de ARN mutadas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 92 (18): 8298–8302. Bibcode :1995PNAS...92.8298W. doi : 10.1073/pnas.92.18.8298 . PMC 41144 . PMID 7545300.
- ^ Montiel-Gonzalez MF, Vallecillo-Viejo I, Yudowski GA, Rosenthal JJ (noviembre de 2013). "Corrección de mutaciones en el regulador de conductancia transmembrana de la fibrosis quística mediante edición de ARN dirigida al sitio". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 110 (45): 18285–18290. Bibcode :2013PNAS..11018285M. doi : 10.1073/pnas.1306243110 . PMC 3831439 . PMID 24108353.
- ^ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (noviembre de 2017). "Edición de ARN con CRISPR-Cas13". Science . 358 (6366): 1019–1027. Bibcode :2017Sci...358.1019C. doi :10.1126/science.aaq0180. PMC 5793859 . PMID 29070703.
- ^ Williams S. "Los neurocientíficos amplían el conjunto de herramientas CRISPR con una nueva enzima compacta Cas7-11". Instituto Tecnológico de Massachusetts . Consultado el 22 de junio de 2022 .
- ^ Kato K, Zhou W, Okazaki S, Isayama Y, Nishizawa T, Gootenberg JS, et al. (junio de 2022). "Estructura e ingeniería del complejo efector CRISPR-Cas7-11 tipo III-E". Cell . 185 (13): 2324–2337.e16. doi : 10.1016/j.cell.2022.05.003 . PMID 35643083. S2CID 249103058.
- ^ Özcan A, Krajeski R, Ioannidi E, Lee B, Gardner A, Makarova KS, et al. (septiembre de 2021). "Orientación programable de ARN con el efector CRISPR de proteína única Cas7-11". Nature . 597 (7878): 720–725. Bibcode :2021Natur.597..720O. doi :10.1038/s41586-021-03886-5. PMID 34489594. S2CID 237432753.
- ^ Cross R (25 de marzo de 2019). "Cuidado, CRISPR. La carrera de edición de ARN ha comenzado". Chemical & Engineering News . 97 (12) . Consultado el 30 de septiembre de 2020 .
- ^ Akbari OS, Bellen HJ, Bier E, Bullock SL, Burt A, Church GM, et al. (agosto de 2015). "BIOSEGURIDAD. Salvaguardar los experimentos de impulso genético en el laboratorio". Science . 349 (6251): 927–929. Bibcode :2015Sci...349..927A. doi :10.1126/science.aac7932. PMC 4692367 . PMID 26229113.
- ^ Caplan AL, Parent B, Shen M, Plunkett C (noviembre de 2015). "No time to waste—the ethics challenges created by CRISPR: CRISPR/Cas, al ser una tecnología eficiente, simple y barata para editar el genoma de cualquier organismo, plantea muchos problemas éticos y regulatorios más allá de su uso para manipular células de la línea germinal humana". EMBO Reports . 16 (11): 1421–1426. doi :10.15252/embr.201541337. PMC 4641494 . PMID 26450575.
- ^ Oye KA, Esvelt K, Appleton E, Catteruccia F, Church G, Kuiken T, et al. (agosto de 2014). "Biotecnología. Regulación de impulsores genéticos". Science . 345 (6197): 626–628. Bibcode :2014Sci...345..626O. doi : 10.1126/science.1254287 . PMID 25035410.
- ^ Gu W, Crawford ED, O'Donovan BD, Wilson MR, Chow ED, Retallack H, DeRisi JL (marzo de 2016). "Reducción de secuencias abundantes por hibridación (DASH): uso de Cas9 para eliminar especies no deseadas de alta abundancia en bibliotecas de secuenciación y aplicaciones de conteo molecular". Genome Biology . 17 : 41. doi : 10.1186/s13059-016-0904-5 . PMC 4778327 . PMID 26944702.
- ^ ab Kungulovski, Goran; Jeltsch, Albert (1 de febrero de 2016). "Epigenome Editing: State of the Art, Concepts, and Perspectives". Tendencias en genética . 32 (2): 101–113. doi :10.1016/j.tig.2015.12.001. ISSN 0168-9525. PMID 26732754 . Consultado el 30 de abril de 2021 .
- ^ Laufer, Benjamin I.; Singh, Shiva M. (17 de septiembre de 2015). "Estrategias para la modulación precisa de la expresión génica mediante la edición del epigenoma: una descripción general". Epigenetics & Chromatin . 8 (1): 34. doi : 10.1186/s13072-015-0023-7 . ISSN 1756-8935. PMC 4574080 . PMID 26388942.
- ^ "La terapia génica CRISPR única ofrece un tratamiento para el dolor crónico sin opioides". New Atlas . 11 de marzo de 2021 . Consultado el 18 de abril de 2021 .
- ^ Moreno, Ana M.; Alemán, Fernando; Catroli, Glaucilene F.; Hunt, Matthew; Hu, Michael; Dailamy, Amir; Pla, Andrew; Woller, Sarah A.; Palmer, Nathan; Parekh, Udit; McDonald, Daniella; Roberts, Amanda J.; Goodwill, Vanessa; Dryden, Ian; Hevner, Robert F.; Delay, Lauriane; Santos, Gilson Gonçalves dos; Yaksh, Tony L.; Mali, Prashant (10 de marzo de 2021). "Analgesia de larga duración mediante la represión in situ dirigida de NaV1.7 en ratones". Science Translational Medicine . 13 (584): eaay9056. doi :10.1126/scitranslmed.aay9056. ISSN 1946-6234. PMC 8830379 . Número de modelo: PMID 33692134. Número de modelo: S2CID 232170826.
- ^ Kaiser, Jocelyn (1 de junio de 2022). "¿Mejor que CRISPR? Otra forma de solucionar los problemas genéticos puede ser más segura y versátil". www.science.org . Consultado el 21 de agosto de 2022 .
- ^ McDonnell S. "Nueva herramienta basada en CRISPR inserta grandes secuencias de ADN en los sitios deseados de las células". Instituto Tecnológico de Massachusetts vía phys.org . Consultado el 18 de diciembre de 2022 .
- ^ Yarnall MT, Ioannidi EI, Schmitt-Ulms C, Krajeski RN, Lim J, Villiger L, et al. (noviembre de 2022). "Inserción de secuencias grandes en el genoma mediante arrastrar y soltar sin escisión de ADN de doble cadena utilizando integrasas dirigidas por CRISPR". Nature Biotechnology . 41 (4): 500–512. bioRxiv 10.1101/2021.11.01.466786 . doi :10.1038/s41587-022-01527-4. PMC 10257351 . PMID 36424489. S2CID 253879386.
- ^ Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, et al. (diciembre de 2019). "Edición genómica de búsqueda y reemplazo sin roturas de doble cadena ni ADN donante". Nature . 576 (7785): 149–157. Bibcode :2019Natur.576..149A. doi :10.1038/s41586-019-1711-4. PMC 6907074 . PMID 31634902.
- ^ Thulin L (21 de octubre de 2019). "Una nueva herramienta de edición genética podría hacer que CRISPR sea más preciso". The Smithsonian Magazine .
- ^ ab Cohen J (21 de octubre de 2019). "Un nuevo editor de genomas 'de primera' podría superar a CRISPR". Revista Science .
- ^ ab Yasinski E (21 de octubre de 2019). "El nuevo método de "edición primaria" realiza solo cortes de ADN de cadena sencilla". The Scientist .
- ^ ab Gallagher J (21 de octubre de 2019). «Edición de primera calidad: una herramienta de ADN podría corregir el 89% de los defectos genéticos». BBC News .
- ^ Regalado A (5 de marzo de 2015). "Engineering the Perfect Baby". MIT Technology Review . Archivado desde el original el 13 de marzo de 2015.
- ^ Baltimore D, Berg P, Botchan M, Carroll D, Charo RA, Church G, et al. (abril de 2015). "Biotecnología. Un camino prudente hacia la ingeniería genómica y la modificación de genes de la línea germinal". Science . 348 (6230): 36–38. Bibcode :2015Sci...348...36B. doi :10.1126/science.aab1028. PMC 4394183 . PMID 25791083.
- ^ Lanphier E, Urnov F, Haecker SE, Werner M, Smolenski J (marzo de 2015). "No edites la línea germinal humana". Nature . 519 (7544): 410–411. Bibcode :2015Natur.519..410L. doi : 10.1038/519410a . PMID 25810189.
- ^ Wade N (19 de marzo de 2015). "Los científicos buscan la prohibición de un método de edición del genoma humano". The New York Times . Consultado el 20 de marzo de 2015.
Los biólogos que escriben en Science apoyan la continuación de la investigación de laboratorio con la técnica, y pocos científicos, si es que hay alguno, creen que esté lista para su uso clínico.
- ^ Liang P, Xu Y, Zhang X, Ding C, Huang R, Zhang Z, et al. (mayo de 2015). "Edición genética mediada por CRISPR/Cas9 en cigotos tripronucleares humanos". Protein & Cell . 6 (5): 363–372. doi :10.1007/s13238-015-0153-5. PMC 4417674 . PMID 25894090.
- ^ Kolata G (23 de abril de 2015). "Científicos chinos editan genes de embriones humanos, lo que genera inquietud". The New York Times . Consultado el 24 de abril de 2015 .
- ^ ab Cyranoski D, Reardon S (2015). "Científicos chinos modifican genéticamente embriones humanos". Nature . doi :10.1038/nature.2015.17378. S2CID 87604469.
- ^ Regalado A (8 de mayo de 2016). "Investigadores chinos experimentan con la creación de embriones a prueba de VIH". MIT Technology Review . Consultado el 10 de junio de 2016 .
- ^ "Cumbre internacional sobre edición genética". Academias Nacionales de Ciencias, Ingeniería y Medicina . 3 de diciembre de 2015. Consultado el 3 de diciembre de 2015 .
- ^ Brokowski C (abril de 2018). "¿Son suficientes las declaraciones éticas de la línea germinal CRISPR?". The CRISPR Journal . 1 (2): 115–125. doi :10.1089/crispr.2017.0024. PMC 6694771 . PMID 31021208.
- ^ Begley S (28 de noviembre de 2018). "En medio del revuelo, un científico chino defiende la creación de bebés editados genéticamente". STAT .
- ^ Muestra I (13 de marzo de 2019). «Los científicos piden una moratoria global sobre la edición genética de embriones». Theguardian.com . Consultado el 14 de marzo de 2019 .
- ^ Callaway E (febrero de 2016). "Científicos del Reino Unido obtienen licencia para editar genes en embriones humanos". Nature . 530 (7588): 18. Bibcode :2016Natur.530...18C. doi : 10.1038/nature.2016.19270 . PMID 26842037.
- ^ McHughen A, Smyth S (enero de 2008). "Sistema regulatorio de los Estados Unidos para cultivares de cultivos modificados genéticamente [organismos genéticamente modificados (OGM), ADNr o transgénicos]". Plant Biotechnology Journal . 6 (1): 2–12. doi : 10.1111/j.1467-7652.2007.00300.x . PMID 17956539. S2CID 3210837.
- ^ USDA . "Re: Solicitud de confirmación" (PDF) .
- ^ Waltz E (abril de 2016). "Un hongo modificado genéticamente mediante CRISPR escapa a la regulación estadounidense". Nature . 532 (7599): 293. Bibcode :2016Natur.532..293W. doi : 10.1038/nature.2016.19754 . PMID 27111611.
- ^ Ledford H (abril de 2016). "La edición genética aumenta a medida que Estados Unidos replantea las regulaciones". Nature . 532 (7598): 158–159. Bibcode :2016Natur.532..158L. doi : 10.1038/532158a . PMID 27075074.
- ^ Brown KV (1 de febrero de 2017). "La FDA está tomando medidas drásticas contra los ingenieros genéticos corruptos". Gizmodo . Consultado el 5 de febrero de 2017 .
- ^ "Guía para la industria n.º 187 / Regulación del ADN genómico alterado intencionalmente en animales" (PDF) . Administración de Alimentos y Medicamentos . 2020-02-11.
- ^ Cyranoski D (agosto de 2017). "La adopción por parte de China de la selección de embriones plantea cuestiones espinosas". Nature . 548 (7667): 272–274. Bibcode :2017Natur.548..272C. doi : 10.1038/548272a . PMID 28816265.
- ^ Peng Y (junio de 2016). "La moralidad y la ética que rigen las patentes de CRISPR-Cas9 en China". Nature Biotechnology . 34 (6): 616–618. doi :10.1038/nbt.3590. PMID 27281418. S2CID 38509820.
- ^ Rana P, Marcus A, Fan W (21 de enero de 2018). "China, sin trabas por las reglas, avanza a paso de tortuga en los ensayos de edición genética". Wall Street Journal . ISSN 0099-9660 . Consultado el 23 de enero de 2018 .
- ^ Talbot D (2016). «Edición genética precisa en plantas/10 tecnologías innovadoras 2016». MIT Technology review . Instituto Tecnológico de Massachusetts. Archivado desde el original el 5 de noviembre de 2017. Consultado el 18 de marzo de 2016 .
- ^ Larson C, Schaffer A (2014). "Edición del genoma/10 tecnologías innovadoras 2014". Instituto Tecnológico de Massachusetts. Archivado desde el original el 5 de diciembre de 2016. Consultado el 18 de marzo de 2016 .
- ^ "Galardonados con el premio Tang". www.tang-prize.org . Consultado el 5 de agosto de 2018 .
- ^ "Nota de prensa: El Premio Nobel de Química 2020". Fundación Nobel . Consultado el 7 de octubre de 2020 .

