Caenorhabditis elegans
| Caenorhabditis elegans | |
|---|---|
 | |
| Un gusano hermafrodita adulto C. elegans | |
| Clasificación científica | |
| Dominio: | Eucariota |
| Reino: | Animalia |
| Filo: | Nematodos |
| Clase: | Cromadorea |
| Orden: | Rabdítidos |
| Familia: | Rabdítidos |
| Género: | Caenorhabditis |
| Especies: | C. elegans |
| Nombre binomial | |
| Caenorhabditis elegans ( Maupas , 1900) [1] | |
| Subespecie | |
Caenorhabditis elegans ( / ˌs iː n oʊ r æ b ˈ d aɪ t ə s ˈ ɛ l ə ɡ æ n s / [ 6 ] ) es un nematodo transparente de vida librede aproximadamente 1 mm de longitud [7] que vive en ambientes de suelo templado. Es la especie tipo de su género. [8] El nombre es una mezcla del griego caeno- (reciente), rhabditis (similar a una varilla) [9] y el latín elegans (elegante). En 1900, Maupas lo nombró inicialmente Rhabditides elegans. Osche lo colocó en el subgénero Caenorhabditis en 1952, y en 1955, Dougherty elevó Caenorhabditis al estado de género . [10]
C. elegans es un pseudocelomado no segmentado y carece de sistemas respiratorio o circulatorio. [11] La mayoría de estos nematodos son hermafroditas y algunos son machos. [12] Los machos tienen colas especializadas para el apareamiento que incluyen espículas .
En 1963, Sydney Brenner propuso investigar sobre C. elegans, principalmente en el área del desarrollo neuronal. En 1974, comenzó a investigar la biología molecular y del desarrollo de C. elegans , que desde entonces se ha utilizado ampliamente como organismo modelo . [13] Fue el primer organismo multicelular en tener su genoma completo secuenciado , y en 2019 [14] fue el primer organismo en tener su conectoma (diagrama de cableado neuronal) completo. [15] [16] [17]
Se han ganado cuatro premios Nobel (hasta 2024) por trabajos realizados sobre C. elegans. [18]
Anatomía

C. elegans no está segmentado , es vermiforme y bilateralmente simétrico . Tiene una cutícula (una fuerte cubierta externa, como un exoesqueleto ), cuatro cordones epidérmicos principales y un pseudoceloma (cavidad corporal) lleno de líquido. También tiene algunos de los mismos sistemas de órganos que los animales más grandes. Aproximadamente uno de cada mil individuos es macho y el resto son hermafroditas. [19] La anatomía básica de C. elegans incluye una boca, faringe , intestino , gónada y cutícula colágena . Como todos los nematodos, no tienen sistema circulatorio ni respiratorio. Las cuatro bandas de músculos que recorren la longitud del cuerpo están conectadas a un sistema neural que permite que los músculos muevan el cuerpo del animal solo como flexión dorsal o ventral, pero no hacia la izquierda o la derecha, excepto la cabeza, donde los cuatro cuadrantes musculares están conectados independientemente uno del otro. Cuando una onda de contracciones musculares dorsales/ventrales avanza desde la parte posterior hacia la parte frontal del animal, este es impulsado hacia atrás. Cuando una onda de contracciones se inicia en la parte frontal y avanza posteriormente a lo largo del cuerpo, el animal es impulsado hacia adelante. Debido a esta tendencia dorsal/ventral en las curvas del cuerpo, cualquier individuo normal vivo y en movimiento tiende a recostarse sobre su lado izquierdo o sobre su lado derecho cuando se lo observa cruzando una superficie horizontal. Se cree que un conjunto de crestas en los lados laterales de la cutícula del cuerpo, las aleas, le dan al animal una tracción adicional durante estos movimientos de flexión.



En relación con el metabolismo lipídico, C. elegans no tiene tejido adiposo especializado, páncreas , hígado o incluso sangre para suministrar nutrientes en comparación con los mamíferos. Los lípidos neutros se almacenan en el intestino, la epidermis y los embriones. La epidermis corresponde a los adipocitos de los mamíferos al ser el principal depósito de triglicéridos . [20]
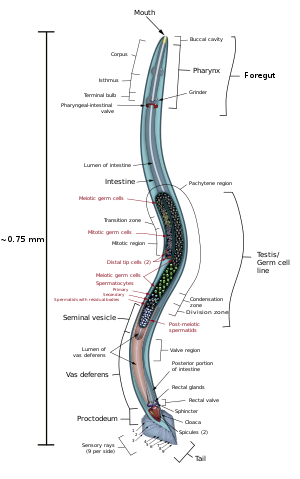
La faringe es una bomba muscular de alimentos situada en la cabeza de C. elegans , que tiene una sección transversal triangular. Esta muele los alimentos y los transporta directamente al intestino. Un conjunto de "células valvulares" conecta la faringe con el intestino, pero no se entiende cómo funciona esta válvula. Después de la digestión, el contenido del intestino se libera a través del recto, como ocurre con todos los demás nematodos. [21] No existe una conexión directa entre la faringe y el canal excretor , que funciona en la liberación de orina líquida.
Los machos tienen una gónada unilobulada, un conducto deferente y una cola especializada para el apareamiento, que incorpora espículas . Los hermafroditas tienen dos ovarios , oviductos y espermatecas , y un solo útero .
Existen 302 neuronas en C. elegans, aproximadamente un tercio de todas las células somáticas de todo el cuerpo. [22] Muchas neuronas contienen dendritas que se extienden desde la célula para recibir neurotransmisores u otras señales, y un proceso que se extiende hasta el anillo nervioso (el "cerebro") para una conexión sináptica con otras neuronas. [23] C. elegans tiene neuronas motoras colinérgicas excitatorias e inhibidoras GABAérgicas que se conectan con los músculos de la pared corporal para regular el movimiento. Además, estas neuronas y otras neuronas como las interneuronas utilizan una variedad de neurotransmisores para controlar los comportamientos. [24]
Gránulos intestinales
.jpg/440px-Nematode_Caenorhabditis_Elegans_(cropped).jpg)
En el intestino de C. elegans se encuentran numerosos gránulos intestinales , cuyas funciones aún no se conocen por completo, al igual que muchos otros aspectos de este nematodo, a pesar de los muchos años que lleva estudiándose. Estos gránulos intestinales se encuentran en todos los órdenes Rhabditida. Son muy similares a los lisosomas en que presentan un interior ácido y capacidad de endocitosis , pero son considerablemente más grandes, lo que refuerza la idea de que son orgánulos de almacenamiento. Una característica particular de los gránulos es que cuando se observan bajo luz ultravioleta , reaccionan emitiendo una intensa fluorescencia azul . Otro fenómeno observado se denomina "fluorescencia de muerte". A medida que los gusanos mueren, se emite una espectacular explosión de fluorescencia azul. Esta fluorescencia de muerte suele tener lugar en una onda de anterior a posterior que se desplaza a lo largo del intestino, y se observa tanto en gusanos jóvenes como viejos, ya sea sometidos a lesiones letales o muriendo pacíficamente de vejez.
Se han propuesto muchas teorías sobre las funciones de los gránulos intestinales, pero las primeras fueron descartadas por hallazgos posteriores. Se cree que una de sus funciones es almacenar zinc. Un análisis químico reciente ha identificado el material fluorescente azul que contienen como una forma glicosilada de ácido antranílico (AA). Se cuestiona la necesidad de las grandes cantidades de AA que contienen los numerosos gránulos intestinales. Una posibilidad es que el AA sea antibacteriano y se utilice en la defensa contra patógenos invasores. Otra posibilidad es que los gránulos proporcionen fotoprotección; las ráfagas de fluorescencia del AA implican la conversión de la dañina luz ultravioleta en luz visible relativamente inofensiva. Esto se considera un posible vínculo con los melanosomas que contienen melanina . [25]
Reproducción
El gusano hermafrodita se considera una forma especializada de hembra autofértil, ya que su soma es femenino. La línea germinal hermafrodita produce gametos masculinos primero y pone huevos a través de su útero después de la fertilización interna. Los hermafroditas producen todos sus espermatozoides en la etapa L4 (150 espermatozoides por brazo gonadal) y luego producen solo ovocitos . La gónada hermafrodita actúa como un ovotestículo con espermatozoides que se almacenan en la misma área de la gónada que los ovocitos hasta que el primer ovocito empuja el esperma hacia la espermateca (una cámara en la que los ovocitos son fertilizados por el espermatozoide). [26]
El macho puede inseminar al hermafrodita, que utilizará preferentemente esperma masculino (ambos tipos de esperma se almacenan en la espermateca).

El esperma de C. elegans es ameboide, sin flagelos ni acrosomas . [28] Cuando se autoinsemina, el gusano de tipo salvaje pone alrededor de 300 huevos. Cuando es inseminado por un macho, el número de descendientes puede superar los 1000. Los hermafroditas no suelen aparearse con otros hermafroditas. A 20 °C, la cepa de laboratorio de C. elegans (N2) tiene una vida media de alrededor de 2-3 semanas y un tiempo de generación de 3 a 4 días.
C. elegans tiene cinco pares de autosomas y un par de cromosomas sexuales . El sexo en C. elegans se basa en un sistema de determinación sexual X0 . Los hermafroditas de C. elegans tienen un par de cromosomas sexuales coincidentes (XX); los raros machos tienen solo un cromosoma sexual (X0).
Determinación del sexo
Los C. elegans son organismos en su mayoría hermafroditas, que producen tanto espermatozoides como ovocitos . [29] Los machos aparecen en la población en una tasa de aproximadamente 1 en 200 hermafroditas, pero los dos sexos están altamente diferenciados. [30] [31] Los machos se diferencian de sus contrapartes hermafroditas en que son más pequeños y se pueden identificar a través de la forma de su cola. [31] Los C.elegans se reproducen a través de un proceso llamado androdioecia . Esto significa que pueden reproducirse de dos maneras: ya sea a través de la autofecundación en hermafroditas o mediante hermafroditas que se reproducen con machos. Los machos se producen a través de la no disyunción de los cromosomas X durante la meiosis. Los gusanos que se reproducen a través de la autofecundación corren el riesgo de sufrir un alto desequilibrio de ligamiento , lo que conduce a una menor diversidad genética en las poblaciones y a un aumento en la acumulación de alelos deletéreos . [32] En C. elegans , la determinación sexual somática se atribuye al gen tra-1 . [33] El tra-1 es un gen dentro de la vía de determinación sexual del factor de transcripción TRA-1 que se regula postranscripcionalmente y funciona promoviendo el desarrollo femenino. [33] En los hermafroditas (XX), hay altos niveles de actividad tra-1 , que produce el sistema reproductor femenino e inhibe el desarrollo masculino. [29] [33] En un momento determinado de su ciclo de vida, un día antes de la edad adulta, los hermafroditas pueden identificarse mediante la adición de una vulva cerca de su cola. En los machos (XO), hay bajos niveles de actividad tra-1 , lo que resulta en un sistema reproductor masculino. [33] Investigaciones recientes han demostrado que hay otros tres genes, fem-1, fem-2 y fem-3, que regulan negativamente la vía TRA-1 y actúan como el determinante final del sexo en C. elegans . [29]
Evolución
El sistema de determinación sexual en C. elegans es un tema que ha sido de interés para los científicos durante años. [30] Dado que se utilizan como organismo modelo, cualquier información descubierta sobre la forma en que su sistema de determinación sexual podría haber evolucionado podría promover la misma investigación de biología evolutiva en otros organismos. Después de casi 30 años de investigación, los científicos han comenzado a unir las piezas de la evolución de dicho sistema. [30] Lo que han descubierto es que hay una vía compleja involucrada que tiene varias capas de regulación. [30] El organismo estrechamente relacionado Caenorhabditis briggsae ha sido estudiado extensamente y su secuencia genómica completa ha ayudado a unir las piezas faltantes en la evolución de la determinación sexual de C. elegans . [30] Se ha descubierto que dos genes se han asimilado, lo que lleva a que las proteínas XOL-1 y MIX-1 tengan un efecto en la determinación sexual también en C. elegans . [30] Las mutaciones en la vía XOL-1 conducen a la feminización en C. elegans. [34] Se sabe que el gen mix-1 hipoactiva el cromosoma X y regula la morfología de la cola masculina en C. elegans. [35] Si observamos al nematodo en su conjunto, es probable que el sexo masculino y el hermafrodita hayan evolucionado a partir de una evolución paralela. [30] La evolución paralela se define como rasgos similares que evolucionan a partir de un ancestro en condiciones similares; en pocas palabras, las dos especies evolucionan de manera similar a lo largo del tiempo. Un ejemplo de esto serían los mamíferos marsupiales y placentarios . Los científicos también han planteado la hipótesis de que la reproducción asexual hermafrodita , o "autofecundación", podría haber evolucionado de manera convergente al estudiar especies similares a C. elegans. [30] Otros estudios sobre la evolución de la determinación del sexo sugieren que los genes que involucran a los espermatozoides evolucionan a un ritmo más rápido que los genes femeninos. [36] Sin embargo, los genes de los espermatozoides en el cromosoma X tienen tasas de evolución reducidas. Los genes de los espermatozoides tienen secuencias de codificación cortas, un alto sesgo de codones y una representación desproporcionada entre los genes huérfanos . [36] Estas características de los genes de los espermatozoides pueden ser la razón de sus altas tasas de evolución y también pueden sugerir cómo evolucionaron los genes de los espermatozoides a partir de gusanos hermafroditas. En general, los científicos tienen una idea general de la vía de determinación del sexo en C. elegans , sin embargo, la evolución de cómo llegó a existir esta vía aún no está bien definida.
Desarrollo

Desarrollo embrionario
El cigoto fertilizado sufre una segmentación holoblástica rotacional .
La entrada del espermatozoide en el ovocito inicia la formación de un eje anteroposterior. [37] El centro organizador de microtúbulos del espermatozoide dirige el movimiento del pronúcleo del espermatozoide hacia el futuro polo posterior del embrión, mientras que también incita el movimiento de las proteínas PAR , un grupo de factores de determinación citoplasmática, a sus respectivas ubicaciones adecuadas. [38] Como resultado de la diferencia en la distribución de la proteína PAR, la primera división celular es altamente asimétrica . [39] La embriogénesis de C. elegans es uno de los ejemplos mejor comprendidos de división celular asimétrica. [40]
Todas las células de la línea germinal surgen de una única célula germinal primordial , llamada célula P4 , establecida temprano en la embriogénesis . [41] [42] Esta célula primordial se divide para generar dos precursores de la línea germinal que no se dividen más hasta después de la eclosión. [42]
Formación del eje
Las células hijas resultantes de la primera división celular se denominan célula AB (que contiene PAR-6 y PAR-3) y célula P1 (que contiene PAR-1 y PAR-2). Una segunda división celular produce las células ABp y ABa a partir de la célula AB, y las células EMS y P2 a partir de la célula P1. Esta división establece el eje dorso-ventral, con la célula ABp formando el lado dorsal y la célula EMS marcando el lado ventral. [43] A través de la señalización Wnt , la célula P2 instruye a la célula EMS a dividirse a lo largo del eje anteroposterior. [44] A través de la señalización Notch , la célula P2 especifica diferencialmente las células ABp y ABa, lo que define aún más el eje dorso-ventral. El eje izquierda-derecha también se hace evidente al principio de la embriogénesis, aunque no está claro exactamente cuándo se determina específicamente el eje. Sin embargo, la mayoría de las teorías del desarrollo del eje LR implican algún tipo de diferencias en las células derivadas de la célula AB. [45]
Gastrulación
La gastrulación ocurre después de que el embrión alcanza la etapa de 24 células. [46] C. elegans es una especie de protóstomos , por lo que el blastoporo finalmente forma la boca. La involución en el blastoporo comienza con el movimiento de las células del endodermo y la posterior formación del intestino, seguido por el precursor de la línea germinal P4 y, finalmente, las células del mesodermo , incluidas las células que finalmente forman la faringe. La gastrulación termina cuando la epibolia de los hipoblastos cierra el blastoporo. [47]
Desarrollo postembrionario


En condiciones ambientales favorables para la reproducción , las larvas eclosionadas se desarrollan a través de cuatro etapas larvarias - L1, L2, L3 y L4 - en solo 3 días a 20 °C. Cuando las condiciones son estresantes, como en la insuficiencia de alimentos, la densidad de población excesiva o las altas temperaturas, C. elegans puede entrar en una tercera etapa larvaria alternativa, L2d, llamada etapa dauer ( Dauer es alemán para permanente). Una feromona dauer específica regula la entrada en el estado dauer. Esta feromona está compuesta de derivados similares del azúcar 3,6-didesoxi, ascarilosa. Los ascarósidos, llamados así por la base de ascarilosa, están involucrados en muchos comportamientos sociales y específicos del sexo. [48] De esta manera, constituyen un lenguaje químico que C. elegans usa para modular varios fenotipos. Las larvas dauer son resistentes al estrés; son delgadas y sus bocas están selladas con una cutícula dauer característica y no pueden ingerir alimentos. Pueden permanecer en esta etapa durante algunos meses. [49] [50] La etapa termina cuando las condiciones mejoran y favorecen un mayor crecimiento de la larva, que ahora muda al estadio L4, aunque el desarrollo de las gónadas se detiene en el estadio L2. [51]
Cada transición de etapa está marcada por una muda de la cutícula transparente del gusano. Las transiciones a través de estas etapas están controladas por genes de la vía heterocrónica, un conjunto de factores reguladores conservados evolutivamente. [52] Muchos genes heterocrónicos codifican microARN , que reprimen la expresión de factores de transcripción heterocrónicos y otros miARN heterocrónicos. [53] Los miARN se descubrieron originalmente en C. elegans. [54] Los eventos de desarrollo importantes controlados por genes heterocrónicos incluyen la división y la eventual fusión sincitial de las células de la costura hipodérmica, y su posterior secreción de las aleas en adultos jóvenes. Se cree que la vía heterocrónica representa un predecesor conservado evolutivamente de los relojes circadianos . [55]
Algunos nematodos tienen un número fijo, determinado genéticamente, de células, un fenómeno conocido como eutelia . El hermafrodita adulto de C. elegans tiene 959 células somáticas y el macho tiene 1033 células, [56] [57] [58] aunque se ha sugerido que el número de sus células intestinales puede aumentar de una a tres en respuesta a los microbios intestinales experimentados por las madres. [59] Gran parte de la literatura describe el número de células en los machos como 1031, pero el descubrimiento de un par de neuronas MCM izquierda y derecha aumentó el número en dos en 2015. [58] El número de células no cambia después de que cesa la división celular al final del período larvario, y el crecimiento posterior se debe únicamente a un aumento en el tamaño de las células individuales. [60]
Ecología
Las diferentes especies de Caenorhabditis ocupan diversos ambientes ricos en nutrientes y bacterias. Se alimentan de las bacterias que se desarrollan en materia orgánica en descomposición ( microbívora ). Poseen receptores quimiosensoriales que permiten la detección de bacterias y metabolitos secretados por bacterias (como sideróforos de hierro), de modo que pueden migrar hacia sus presas bacterianas. [61] El suelo carece de suficiente materia orgánica para sustentar poblaciones autosuficientes. C. elegans puede sobrevivir con una dieta de una variedad de bacterias, pero su ecología salvaje es en gran parte desconocida. La mayoría de las cepas de laboratorio se tomaron de entornos artificiales como jardines y pilas de abono . Más recientemente, se ha descubierto que C. elegans prospera en otros tipos de materia orgánica, particularmente fruta podrida. [62] C. elegans también puede ingerir contaminantes, especialmente nanoplásticos diminutos, lo que podría permitir la asociación con bacterias resistentes a los antibióticos, lo que resulta en la diseminación de nanoplásticos y bacterias resistentes a los antibióticos por C. elegans a través del suelo. [63]
C. elegans también puede utilizar diferentes especies de levadura , incluyendo Cryptococcus laurentii y C. kuetzingii , como únicas fuentes de alimento. [64] Aunque es un bacterívoro , C. elegans puede ser asesinado por una serie de bacterias patógenas, incluyendo patógenos humanos como Staphylococcus aureus , [65] Pseudomonas aeruginosa , [66] Salmonella enterica o Enterococcus faecalis . [67] Las bacterias patógenas también pueden formar biopelículas, cuya matriz de exopolímero pegajosa podría impedir la motilidad de C. elegans [68] y ocultar los quimioatrayentes de detección de quórum bacteriano de la detección de depredadores. [69]
Los invertebrados como los milpiés , los insectos , los isópodos y los gasterópodos pueden transportar larvas de dauer a varios lugares adecuados. También se ha visto que las larvas se alimentan de sus huéspedes cuando mueren. [70]
Los nematodos pueden sobrevivir a la desecación y, en C. elegans , se ha demostrado que el mecanismo de esta capacidad son las proteínas abundantes en la embriogénesis tardía . [71]
C. elegans , como otros nematodos, puede ser devorado por nematodos depredadores y otros omnívoros, incluidos algunos insectos. [72]
El virus de Orsay es un virus que afecta a C. elegans , así como el virus Cer1 de Caenorhabditis elegans [73] y el virus Cer13 de Caenorhabditis elegans .
- Interacciones con hongos
Los aislamientos silvestres de Caenorhabditis elegans se encuentran regularmente con infecciones por hongos Microsporidia . Una de estas especies, Nematocida parisii , se replica en los intestinos de C. elegans . [74]
Arthrobotrys oligospora es el organismo modelo para las interacciones entre hongos y nematodos. [75] Es el hongo que captura nematodos más común y extendido.
Utilizar como organismo modelo
En 1963, Sydney Brenner propuso utilizar C. elegans como organismo modelo para la investigación principalmente del desarrollo neuronal en animales. Es uno de los organismos más simples con un sistema nervioso . Las neuronas no disparan potenciales de acción y no expresan ningún canal de sodio dependiente del voltaje . [76] En el hermafrodita, este sistema comprende 302 neuronas [77] cuyo patrón ha sido mapeado exhaustivamente, [14] en lo que se conoce como un conectoma , [78] y se ha demostrado que es una red de mundo pequeño . [79]
La investigación ha explorado los mecanismos neuronales y moleculares que controlan varios comportamientos de C. elegans , incluyendo la quimiotaxis , la termotaxis , la mecanotransducción , el aprendizaje , la memoria y el comportamiento de apareamiento . [80] En 2019 se publicó el conectoma del macho utilizando una técnica distinta a la utilizada para el hermafrodita. El mismo artículo utilizó la nueva técnica para rehacer el conectoma del hermafrodita, encontrando 1.500 nuevas sinapsis. [81]
Se ha utilizado como organismo modelo para estudiar los mecanismos moleculares de las enfermedades metabólicas. [82] Brenner también lo eligió porque es fácil de cultivar en poblaciones a granel y es conveniente para el análisis genético. [83] Es un organismo eucariota multicelular , pero lo suficientemente simple como para ser estudiado en gran detalle. La transparencia de C. elegans facilita el estudio de la diferenciación celular y otros procesos de desarrollo en el organismo intacto. Las espículas en el macho distinguen claramente a los machos de las hembras. Las cepas son baratas de criar y se pueden congelar. Cuando se descongelan posteriormente, siguen siendo viables, lo que permite el almacenamiento a largo plazo. [13] El mantenimiento es fácil en comparación con otros organismos modelo multicelulares. Se pueden mantener unos pocos cientos de nematodos en una sola placa de agar y un medio de crecimiento adecuado. Brenner describió el uso de un mutante de E. coli : OP50. OP50 es un organismo que requiere uracilo y su deficiencia en la placa evita el crecimiento excesivo de bacterias que oscurecerían a los gusanos. [84] El uso de OP50 no exige ninguna medida de seguridad de laboratorio importante, ya que no es patógeno y se cultiva fácilmente en medios Luria-Bertani (LB) durante la noche. [85]
Mapeo del linaje celular
Se ha mapeado el destino del desarrollo de cada célula somática (959 en el hermafrodita adulto; 1031 en el macho adulto). [86] [87] Estos patrones de linaje celular son en gran medida invariables entre individuos, mientras que en los mamíferos, el desarrollo celular depende más de las señales celulares del embrión.
Como se mencionó anteriormente, las primeras divisiones celulares de la embriogénesis temprana en C. elegans se encuentran entre los ejemplos mejor comprendidos de divisiones celulares asimétricas , y el gusano es un sistema modelo muy popular para estudiar la biología del desarrollo. [40]
Muerte celular programada
La muerte celular programada ( apoptosis ) elimina muchas células adicionales (131 en el hermafrodita, la mayoría de las cuales de otro modo se convertirían en neuronas ); esta "predicibilidad apoptótica" ha contribuido a la elucidación de algunos genes apoptóticos . Se han identificado genes promotores de la muerte celular y un único inhibidor de la muerte celular. [88]
Interferencia de ARN y silenciamiento de genes

La interferencia de ARN (RNAi) es un método relativamente sencillo para alterar la función de genes específicos. Silenciar la función de un gen a veces permite a un investigador inferir su posible función. El nematodo puede empaparse, inyectarse [89] o alimentarse con bacterias transformadas genéticamente que expresen el ARN bicatenario de interés, cuya secuencia complementa la secuencia del gen que el investigador desea desactivar. [90] La interferencia de ARN ha surgido como una herramienta poderosa en el estudio de la genómica funcional. C. elegans se ha utilizado para analizar las funciones de los genes y reivindicar la promesa de futuros hallazgos en las interacciones genéticas sistemáticas. [91]
La absorción de ARN ambiental es mucho peor en otras especies de gusanos del género Caenorhabditis . Aunque la inyección de ARN en la cavidad corporal del animal induce el silenciamiento genético en la mayoría de las especies, solo C. elegans y algunos otros nematodos distantemente relacionados pueden absorber ARN de las bacterias que comen para el ARNi. [92] Esta capacidad se ha atribuido a un solo gen, sid-2 , que, cuando se inserta como transgén en otras especies, les permite absorber ARN para el ARNi como lo hace C. elegans . [93]
División celular y ciclo celular
La investigación sobre la meiosis se ha simplificado considerablemente, ya que cada núcleo de célula germinal se encuentra en la misma posición a medida que desciende por la gónada, por lo que se encuentra en la misma etapa de la meiosis. En una fase temprana de la meiosis, los ovocitos se vuelven extremadamente resistentes a la radiación y esta resistencia depende de la expresión de los genes rad51 y atm que tienen papeles clave en la reparación recombinatoria. [94] [95] El gen mre-11 también juega un papel crucial en la reparación recombinatoria del daño del ADN durante la meiosis. [96] Un estudio de la frecuencia de cruzamiento externo en poblaciones naturales mostró que la autofecundación es el modo predominante de reproducción en C. elegans , pero que los eventos de cruzamiento externo poco frecuentes ocurren a una tasa de alrededor del 1%. [97] Es poco probable que las meiosis que resultan en autofecundación contribuyan significativamente a la variabilidad genética beneficiosa, pero estas meiosis pueden proporcionar el beneficio adaptativo de la reparación recombinatoria de los daños del ADN que surgen, especialmente en condiciones estresantes. [98]
Abuso y adicción a las drogas
La dependencia de la nicotina también se puede estudiar utilizando C. elegans porque muestra respuestas conductuales a la nicotina que son paralelas a las de los mamíferos. Estas respuestas incluyen respuesta aguda, tolerancia, abstinencia y sensibilización. [99]
Bases de datos biológicas
Como ocurre con la mayoría de los organismos modelo, los científicos que trabajan en este campo mantienen una base de datos en línea dedicada a C. elegans , y WormBase es la única que se ocupa de esta especie . WormBase intenta recopilar toda la información publicada sobre C. elegans y otros nematodos relacionados. La información sobre C. elegans se incluye con los datos sobre otros organismos modelo en la Alianza de Recursos Genómicos. [100]
Envejecimiento
C. elegans ha sido un organismo modelo para la investigación sobre el envejecimiento ; por ejemplo, se ha demostrado que la inhibición de una vía de señalización del factor de crecimiento similar a la insulina aumenta la esperanza de vida adulta tres veces; [101] [102] mientras que la alimentación con glucosa promueve el estrés oxidativo y reduce la esperanza de vida adulta a la mitad. [82] De manera similar, la degradación inducida de un receptor de insulina/IGF-1 en etapas tardías de la vida extendió la esperanza de vida de los gusanos dramáticamente. [103] Se demostró que los mutantes de larga vida de C. elegans eran resistentes al estrés oxidativo y a la luz ultravioleta . [104] Estos mutantes de larga vida tenían una mayor capacidad de reparación del ADN que el C. elegans de tipo salvaje . [104] La eliminación del gen de reparación por escisión de nucleótidos Xpa-1 aumentó la sensibilidad a los rayos UV y redujo la esperanza de vida de los mutantes de larga vida. Estos hallazgos indican que la capacidad de reparación del ADN subyace a la longevidad .
La capacidad de reparar el daño del ADN mediante el proceso de reparación por escisión de nucleótidos disminuye con la edad. [105]
Los C. elegans expuestos a 5 mM de cloruro de litio (LiCl) mostraron una mayor expectativa de vida. [106] Cuando se expusieron a 10 μM de LiCl, se observó una mortalidad reducida, pero no con 1 μM. [107]
C. elegans ha sido fundamental en la identificación de las funciones de los genes implicados en la enfermedad de Alzheimer , como la presenilina . [108] Además, una amplia investigación sobre C. elegans ha identificado las proteínas de unión al ARN como factores esenciales durante la línea germinal y el desarrollo embrionario temprano. [109]
Los telómeros , cuya longitud se ha demostrado que se correlaciona con una mayor esperanza de vida y un inicio retardado de la senescencia en una multitud de organismos, desde C. elegans [110] [111] hasta los humanos, [112] muestran un comportamiento interesante en C. elegans. Mientras que C. elegans mantiene sus telómeros de una manera canónica similar a otros eucariotas, en contraste, Drosophila melanogaster es notable en su uso de retrotransposones para mantener sus telómeros, [113] durante la eliminación de la subunidad catalítica de la telomerasa ( trt-1 ) C. elegans puede obtener la capacidad de alargamiento alternativo de telómeros (ALT). C. elegans fue el primer eucariota en obtener la funcionalidad de ALT después de la eliminación de la vía canónica de la telomerasa . [114] ALT también se observa en aproximadamente el 10-15% de todos los cánceres clínicos. [115] Por lo tanto, C. elegans es un candidato principal para la investigación de ALT. [116] [117] [118] Bayat et al. demostraron el acortamiento paradójico de los telómeros durante la sobreexpresión de trt-1 que conduce a una esterilidad casi total, mientras que los gusanos incluso exhibieron un ligero aumento en la esperanza de vida, a pesar de los telómeros acortados. [119]
Dormir
En los estudios sobre el sueño animal, C. elegans es el organismo más primitivo que muestra estados similares al sueño. En C. elegans , una fase de letargo ocurre poco antes de cada muda . [120] También se ha demostrado que C. elegans duerme después de la exposición al estrés físico, incluido el choque térmico, la radiación ultravioleta y las toxinas bacterianas. [121]
Biología sensorial
Aunque el gusano no tiene ojos, se ha descubierto que es sensible a la luz debido a un tercer tipo de proteína fotorreceptora animal sensible a la luz , LITE-1 , que es de 10 a 100 veces más eficiente para absorber la luz que los otros dos tipos de fotopigmentos ( opsinas y criptocromos ) que se encuentran en el reino animal. [122]
El C. elegans es notablemente capaz de tolerar la aceleración. Puede soportar 400.000 g , según los genetistas de la Universidad de São Paulo en Brasil. En un experimento, el 96% de ellos seguían vivos sin efectos adversos después de una hora en una ultracentrífuga. [123]
Investigación de vuelos espaciales
C. elegans fue noticia cuando se descubrió que especímenes habían sobrevivido al desastre del transbordador espacial Columbia en febrero de 2003. [124] Más tarde, en enero de 2009, se anunció que muestras vivas de C. elegans de la Universidad de Nottingham pasarían dos semanas en la Estación Espacial Internacional ese octubre, en un proyecto de investigación espacial para explorar los efectos de la gravedad cero en el desarrollo y la fisiología muscular. La investigación se centró principalmente en la base genética de la atrofia muscular , que se relaciona con los vuelos espaciales o el postramiento en cama, la edad geriátrica o la diabetes . [125] Los descendientes de los gusanos a bordo del Columbia en 2003 fueron lanzados al espacio en el Endeavour para la misión STS-134 . [126] A partir de 2018 se llevaron a cabo experimentos adicionales sobre distrofia muscular durante los vuelos espaciales a bordo de la ISS. [127] Se demostró que los genes que afectan a la unión de los músculos se expresaban menos en el espacio. Sin embargo, aún está por verse si esto afecta a la fuerza muscular.
Genética
Genoma
 Cariotipo de C. elegans Explicación de los colores Cromosomas mitóticos de C. elegans . ADN (rojo)/ cinetocoros (verde). Los organismos holocéntricos , incluido C. elegans , ensamblan cinetocoros difusos a lo largo de toda la cara polar de cada cromátida hermana. | |
| Identificación genómica del NCBI | 41 |
|---|---|
| Ploidía | diploide |
| Tamaño del genoma | 101,169 Mb (haploide) |
| Número de cromosomas | 5 pares de autosomas (I, II, III, IV y V) + 1 o 2 cromosomas sexuales (X [128] ) |
| Año de finalización | 1998 |
| Organelo secuenciado | mitocondria |
| Tamaño de los orgánulos | 0,01 Mb |

C. elegans fue el primer organismo multicelular cuyo genoma completo fue secuenciado . La secuencia se publicó en 1998, [129] aunque había algunas pequeñas lagunas; la última laguna se terminó en octubre de 2002. [ cita requerida ] En el período previo al genoma completo, el Consorcio de Secuenciación de C. elegans / Proyecto Genoma de C. elegans publicó varios escaneos parciales, incluido el de Wilson et al. 1994. [130] [131] [132]
Tamaño y contenido genético
El genoma de C. elegans tiene una longitud de unos 100 millones de pares de bases y consta de seis pares de cromosomas en hermafroditas o cinco pares de autosomas con cromosoma XO en C. elegans macho y un genoma mitocondrial . Su densidad genética es de aproximadamente un gen por cada cinco kilopares de bases . Los intrones constituyen el 26% y las regiones intergénicas el 47% del genoma. Muchos genes están dispuestos en grupos y no está claro cuántos de estos son operones . [133] C. elegans y otros nematodos se encuentran entre los pocos eucariotas que actualmente se sabe que tienen operones; estos incluyen tripanosomas , gusanos planos (notablemente el trematodo Schistosoma mansoni ) y un tunicado cordado primitivo Oikopleura dioica . Es probable que se demuestre que muchos más organismos tienen estos operones. [134]
El genoma contiene aproximadamente 20.470 genes codificadores de proteínas . [135] Alrededor del 35% de los genes de C. elegans tienen homólogos humanos . Sorprendentemente, se ha demostrado repetidamente que los genes humanos reemplazan a sus homólogos de C. elegans cuando se introducen en C. elegans . Por el contrario, muchos genes de C. elegans pueden funcionar de manera similar a los genes de los mamíferos. [49]
La cantidad de genes de ARN conocidos en el genoma ha aumentado considerablemente debido al descubrimiento en 2006 de una nueva clase llamada genes 21U-ARN , [136] y ahora se cree que el genoma contiene más de 16.000 genes de ARN, frente a tan solo 1.300 en 2005. [137]
Los curadores científicos continúan evaluando el conjunto de genes conocidos; se siguen añadiendo nuevos modelos genéticos y se siguen modificando o eliminando los incorrectos.
La secuencia de referencia del genoma de C. elegans continúa cambiando a medida que nuevas evidencias revelan errores en la secuenciación original. La mayoría de los cambios son menores y sólo agregan o eliminan unos pocos pares de bases de ADN. Por ejemplo, la versión WS202 de WormBase (abril de 2009) agregó dos pares de bases a la secuencia del genoma. [138] A veces, se realizan cambios más extensos, como se señaló en la versión WS197 de diciembre de 2008, que agregó una región de más de 4300 pb a la secuencia. [139] [140]
El Proyecto Genoma de C. elegans , realizado por Wilson et al. en 1994, encontró CelVav [132] y un dominio del factor A de von Willebrand [130] y, junto con Wilson et al. en 1998, proporciona la primera evidencia creíble de un homólogo del receptor de hidrocarburos arílicos (AHR) fuera de los vertebrados. [131] 2
Genomas relacionados
En 2003, también se determinó la secuencia del genoma del nematodo relacionado C. briggsae , lo que permitió a los investigadores estudiar la genómica comparativa de estos dos organismos. [141] Las secuencias del genoma de más nematodos del mismo género , por ejemplo, C. remanei , [142] C. japonica [143] y C. brenneri (llamado así por Brenner), también se han estudiado utilizando la técnica de secuenciación de escopeta . [144] Estas secuencias ahora se han completado. [145] [146]
Otros estudios genéticos

A partir de 2014, C. elegans es la especie más basal del grupo 'Elegans' (10 especies) del supergrupo 'Elegans' (17 especies) en estudios filogenéticos. Forma una rama propia distinta de cualquier otra especie del grupo. [147]
El transposón Tc1 es un transposón de ADN activo en C. elegans .
Comunidad científica
Varios científicos han ganado el Premio Nobel de Fisiología o Medicina por descubrimientos científicos realizados trabajando con C. elegans . Fue otorgado en 2002 a Sydney Brenner , H. Robert Horvitz y John Sulston por su trabajo sobre la genética del desarrollo de órganos y la muerte celular programada , en 2006 a Andrew Fire y Craig C. Mello por su descubrimiento de la interferencia del ARN , y en 2024 a Victor Ambros y Gary Ruvkun por su descubrimiento del microARN y su papel en la regulación genética. [148] [149] En 2008, Martin Chalfie compartió un Premio Nobel de Química por su trabajo sobre la proteína fluorescente verde ; algunas de las investigaciones involucraron el uso de C. elegans .
Muchos científicos que investigan C. elegans tienen una estrecha relación con Sydney Brenner, con quien casi toda la investigación en este campo comenzó en la década de 1970; han trabajado como investigadores posdoctorales o de posgrado en el laboratorio de Brenner o en el laboratorio de alguien que trabajó previamente con Brenner. La mayoría de los que trabajaron en su laboratorio establecieron posteriormente sus propios laboratorios de investigación de gusanos, creando así un "linaje" bastante bien documentado de científicos de C. elegans , que se registró en la base de datos WormBase con cierto detalle en la Reunión Internacional sobre Gusanos de 2003. [150]
Véase también
- Experimentación con animales invertebrados
- Bioluminiscencia
- Eileen Southgate
- Intronerador
- Gusano abierto
- Libro de gusanos
Referencias
- ^ Maupas, É (1900). "Modos y formas de reproducción de nematodos". Archivos de Zoología Experimental y General . 8 : 463–624.
- ^ Nigón V (1949). "Las modalidades de la reproducción y el déterminismo del sexo en los quelques nematodes libres". Ana. Ciencia. Nat. Zoológico. Biol. Ánimo . 11 : 1–132.
- ^ Moerman DG, Waterston RH (diciembre de 1984). "Mutaciones espontáneas inestables de unc-22 IV en C. elegans var. Bergerac". Genética . 108 (4): 859–77. doi :10.1093/genetics/108.4.859. PMC 1224270 . PMID 6096205.
- ^ Babity JM, Starr TV, Rose AM (junio de 1990). "Transposición de Tc1 y actividad mutadora en una cepa Bristol de Caenorhabditis elegans ". Genética molecular y general . 222 (1): 65–70. doi :10.1007/bf00283024. PMID 1978238. S2CID 11275388.
- ^ Harris LJ, Rose AM (julio de 1989). "Análisis estructural de elementos Tc1 en Caenorhabditis elegans var. Bristol (cepa N2)". Plásmido . 22 (1): 10–21. doi :10.1016/0147-619x(89)90031-0. PMID 2550981.
- ^ "Caenorhabditis". Diccionario Merriam-Webster.com . Merriam-Webster.
- ^ Wood, WB (1988). El nematodo Caenorhabditis elegans. Prensa de laboratorio de Cold Spring Harbor . pág. 1. ISBN 978-0-87969-433-3.
- ^ Sudhaus W, Kiontke K (2009). "Filogenia del subgénero Caenorhabditis de Rhabditis (Rhabditidae, Nematoda)". Revista de Sistemática Zoológica e Investigación Evolutiva . 34 (4): 217–233. doi : 10.1111/j.1439-0469.1996.tb00827.x .
- ^ καινός (caenos) = nuevo, reciente; ῥάβδος (rhabdos) = vara, vara.
- ^ Ferris, H (30 de noviembre de 2013). «Caenorhabditis elegans». Universidad de California, Davis . Archivado desde el original el 9 de diciembre de 2013. Consultado el 19 de noviembre de 2013 .
- ^ Wallace RL, Ricci C, Melone G (1996). "Un análisis cladístico de la morfología de los pseudocelomados (aschelmintos)". Biología de invertebrados . 115 (2): 104–112. doi :10.2307/3227041. JSTOR 3227041.
- ^ "Introducción a la determinación sexual". www.wormbook.org . Consultado el 15 de marzo de 2017 .
- ^ ab Brenner S (mayo de 1974). "La genética de Caenorhabditis elegans". Genética . 77 (1): 71–94. doi :10.1093/genetics/77.1.71. PMC 1213120 . PMID 4366476.
- ^ ab Cook, SJ; Jarrell, TA; Brittin, CA; Wang, Y; Bloniarz, AE; Yakovlev, MA; Nguyen, KCQ; Tang, Lt-H; Bayer, EA; Duerr, JS; Bulow, HE; Hobert, O; Hall, DH; Emmons, SW (3 de diciembre de 2019). "Conectomas de animales completos de ambos sexos de Caenorhabditis elegans". Nature . 571 (7763). Biblioteca Nacional de Medicina de EE. UU., Institutos Nacionales de Salud: 63–71. Bibcode :2019Natur.571...63C. doi :10.1038/s41586-019-1352-7. PMC 6889226 . PMID 31270481.
- ^ White JG, Southgate E, Thomson JN, Brenner S (noviembre de 1986). "La estructura del sistema nervioso del nematodo Caenorhabditis elegans". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 314 (1165): 1–340. Bibcode :1986RSPTB.314....1W. doi : 10.1098/rstb.1986.0056 . PMID 22462104.
- ^ White JG (junio de 2013). "Cómo adentrarse en la mente de un gusano: una visión personal". WormBook : 1–10. doi :10.1895/wormbook.1.158.1. PMC 4781474 . PMID 23801597.
- ^ Jabr F (2 de octubre de 2012). "El debate sobre el conectoma: ¿Vale la pena mapear la mente de un gusano?". Scientific American . Consultado el 18 de enero de 2014 .
- ^ "Estos pequeños gusanos son responsables de al menos cuatro premios Nobel", The New York Times , 17 de octubre de 2024
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2007). Biología molecular de la célula (5.ª ed.). Garland Science . pág. 1321. ISBN 978-0-8153-4105-5.
- ^ Lemieux GA, Ashrafi K (agosto de 2016). "Investigación de las conexiones entre el metabolismo, la longevidad y el comportamiento en Caenorhabditis elegans". Tendencias en endocrinología y metabolismo . 27 (8): 586–596. doi :10.1016/j.tem.2016.05.004. PMC 4958586. PMID 27289335 .
- ^ "La faringe de C. elegans: un modelo de organogénesis". www.wormbook.org . Consultado el 15 de marzo de 2017 .
- ^ Soares FA, Fagundez DA, Avila DS (2017). "Neurodegeneración inducida por metales en Caenorhabditis elegans". Neurotoxicidad de los metales . Avances en neurobiología. Vol. 18. págs. 55–383. doi :10.1007/978-3-319-60189-2_18. ISBN . 978-3-319-60188-5. Número de identificación personal 28889277.
- ^ Nonet, M. (2004) Acerca del nematodo Caenorhabdtis elegans
- ^ Hobert, Oliver (2005). "Especificación del sistema nervioso". WormBook : 1–19. doi :10.1895/wormbook.1.12.1. PMC 4781215 . PMID 18050401.
- ^ Coburn C, Gems D (2013). "El misterioso caso del gránulo intestinal de C. elegans: fluorescencia de muerte, ácido antranílico y la vía de la quinurenina". Frontiers in Genetics . 4 : 151. doi : 10.3389/fgene.2013.00151 . PMC 3735983 . PMID 23967012.
- ^ Nayak S, Goree J, Schedl T (enero de 2005). "fog-2 y la evolución del hermafroditismo autofértil en Caenorhabditis". PLOS Biology . 3 (1): e6. doi : 10.1371/journal.pbio.0030006 . PMC 539060 . PMID 15630478.
- ^ Loer CM, Kenyon CJ (diciembre de 1993). "Mutantes deficientes en serotonina y comportamiento de apareamiento masculino en el nematodo Caenorhabditis elegans". The Journal of Neuroscience . 13 (12): 5407–17. doi : 10.1523/jneurosci.13-12-05407.1993 . PMC 6576401 . PMID 8254383.
- ^ Ma X, Zhao Y, Sun W, Shimabukuro K, Miao L (octubre de 2012). "Transformación: ¿cómo se activan y se arrastran los espermatozoides de los nematodos?". Protein & Cell . 3 (10): 755–61. doi :10.1007/s13238-012-2936-2. PMC 4875351 . PMID 22903434.
- ^ abc Starostina NG, Lim JM, Schvarzstein M, Wells L, Spence AM, Kipreos ET (julio de 2007). "Una ligasa de ubiquitina CUL-2 que contiene tres proteínas FEM degrada TRA-1 para regular la determinación sexual de C. elegans". Developmental Cell . 13 (1): 127–39. doi :10.1016/j.devcel.2007.05.008. PMC 2064902 . PMID 17609115.
- ^ abcdefgh Haag ES (diciembre de 2005). "La evolución de la determinación sexual de los nematodos: C. elegans como punto de referencia para la biología comparada". WormBook: The Online Review of C. Elegans Biology : 1–14. doi :10.1895/wormbook.1.120.1. PMC 4781019 . PMID 18050417.
- ^ ab "Manual - Introducción masculina". www.wormatlas.org . Consultado el 30 de marzo de 2021 .
- ^ Frézal L, Félix MA (marzo de 2015). "C. elegans fuera de la placa de Petri". eLife . 4 : e05849. doi : 10.7554/eLife.05849 . PMC 4373675 . PMID 25822066.
- ^ abcd Conradt, Barbara; Horvitz, H. Robert (6 de agosto de 1999). "La proteína de determinación sexual TRA-1A de C. elegans regula la muerte celular sexualmente dimórfica reprimiendo el gen activador de muerte celular egl-1". Cell . 98 (3): 317–327. doi : 10.1016/S0092-8674(00)81961-3 . ISSN 0092-8674. PMID 10458607. S2CID 14951719.
- ^ Miller LM, Plenefisch JD, Casson LP, Meyer BJ (octubre de 1988). "xol-1: un gen que controla los modos masculinos de determinación sexual y compensación de dosis del cromosoma X en C. elegans". Cell . 55 (1): 167–83. doi :10.1016/0092-8674(88)90019-0. PMID 3167975. S2CID 5005906.
- ^ "mix-1 (gen) - WormBase: recurso de información sobre nematodos". wormbase.org . Consultado el 23 de abril de 2021 .
- ^ ab Cutter AD, Ward S (enero de 2005). "Dinámica sexual y temporal de la evolución molecular en el desarrollo de C. elegans". Biología molecular y evolución . 22 (1): 178–88. doi : 10.1093/molbev/msh267 . PMID 15371532.
- ^ Goldstein B, Hird SN (mayo de 1996). "Especificación del eje anteroposterior en Caenorhabditis elegans". Desarrollo . 122 (5): 1467–74. doi :10.1242/dev.122.5.1467. PMID 8625834.
- ^ Gilbert SF (2016). Biología del desarrollo (11.ª ed.). Sinauer. pág. 268. ISBN 9781605354705.
- ^ Guo S, Kemphues KJ (mayo de 1995). "par-1, un gen necesario para establecer la polaridad en los embriones de C. elegans, codifica una supuesta quinasa Ser/Thr que se distribuye asimétricamente". Cell . 81 (4): 611–20. doi : 10.1016/0092-8674(95)90082-9 . PMID 7758115.
- ^ ab Gönczy P, Rose LS (octubre de 2005). "División celular asimétrica y formación del eje en el embrión". WormBook : 1–20. doi :10.1895/wormbook.1.30.1. PMC 4780927 . PMID 18050411.
- ^ Kimble J, Crittenden SL. Proliferación de la línea germinal y su control. 15 de agosto de 2005. En: WormBook: The Online Review of C. elegans Biology [Internet]. Pasadena (CA): WormBook; 2005-. Disponible en: https://www.ncbi.nlm.nih.gov/books/NBK19769/
- ^ ab "WBbt:0006773 (término anatómico)". WormBase (WS242 ed.). 14 de mayo de 2014. WBbt:0006773.
- ^ Gilbert SF (2016). Biología del desarrollo (11.ª ed.). Sinauer. pág. 272. ISBN 9781605354705.
- ^ Thorpe CJ, Schlesinger A, Carter JC, Bowerman B (agosto de 1997). "La señalización de Wnt polariza un blastómero temprano de C. elegans para distinguir el endodermo del mesodermo". Cell . 90 (4): 695–705. doi : 10.1016/s0092-8674(00)80530-9 . PMID 9288749.
- ^ Pohl C, Bao Z (septiembre de 2010). "Las fuerzas quirales organizan los patrones de izquierda a derecha en C. elegans desacoplando la línea media y el eje anteroposterior". Developmental Cell . 19 (3): 402–12. doi :10.1016/j.devcel.2010.08.014. PMC 2952354 . PMID 20833362. Villares JC, Carlini EA (1988). "[Cuantificación de la excreción sebácea en voluntarios: influencia de la edad cronológica, sexo y raza]". Med Cutan Ibero Lat Am . 16 (6): 439–44. PMID 3073266. Gilbert SF (2016). Biología del desarrollo (11.ª ed.). Sinauer. pág. 269. ISBN 9781605354705.
- ^ Skiba F, Schierenberg E (junio de 1992). "Linajes celulares, cronología del desarrollo y formación de patrones espaciales en embriones de nematodos del suelo de vida libre". Biología del desarrollo . 151 (2): 597–610. doi :10.1016/0012-1606(92)90197-o. PMID 1601187.
- ^ Gilbert SF (2016). Biología del desarrollo (11.ª ed.). Sinauer. pág. 273. ISBN 9781605354705.
- ^ Ludewig, Andreas H.; Schroeder, Frank C. (18 de enero de 2013). "Señalización de ascarósidos en C. elegans". WormBook : 1–22. doi :10.1895/wormbook.1.155.1. ISSN 1551-8507. PMC 3758900 . PMID 23355522.
- ^ ab "Introducción a C. Elegans". C. Elegans como organismo modelo . Universidad Rutgers. Archivado desde el original el 18 de agosto de 2002. Consultado el 15 de agosto de 2014 .
- ^ "Manual - Introducción".
- ^ "Dauer". www.wormbook.org . Consultado el 27 de septiembre de 2018 .
- ^ Resnick TD, McCulloch KA, Rougvie AE (mayo de 2010). "Los miRNAs le dan a los gusanos el mejor momento de sus vidas: ARN pequeños y control temporal en Caenorhabditis elegans". Dinámica del desarrollo . 239 (5): 1477–89. doi :10.1002/dvdy.22260. PMC 4698981 . PMID 20232378.
- ^ Rougvie AE, Moss EG (2013). "Transiciones del desarrollo en los estadios larvarios de C. elegans". Transiciones del desarrollo en los estadios larvarios de C. elegans. Tiempos de desarrollo. Vol. 105. Academic Press. págs. 153–80. doi :10.1016/B978-0-12-396968-2.00006-3. ISBN 9780123969682. Número de identificación personal 23962842.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Lee RC, Feinbaum RL, Ambros V (diciembre de 1993). "El gen heterocrónico lin-4 de C. elegans codifica ARN pequeños con complementariedad antisentido con lin-14". Cell . 75 (5): 843–54. doi : 10.1016/0092-8674(93)90529-y . PMID 8252621.
- ^ Banerjee D, Kwok A, Lin SY, Slack FJ (febrero de 2005). "El ritmo de desarrollo en C. elegans está regulado por kin-20 y tim-1, homólogos de los genes centrales del reloj circadiano". Developmental Cell . 8 (2): 287–95. doi : 10.1016/j.devcel.2004.12.006 . PMID 15691769.
- ^ Sulston, JE; Horvitz, HR (marzo de 1977). "Líneas celulares postembrionarias del nematodo Caenorhabditis elegans". Biología del desarrollo . 56 (1): 110–156. doi :10.1016/0012-1606(77)90158-0. PMID 838129.
- ^ Sulston, JE; Schierenberg, E.; White, JG; Thomson, JN (noviembre de 1983). "El linaje celular embrionario del nematodo Caenorhabditis elegans". Biología del desarrollo . 100 (1): 64–119. doi :10.1016/0012-1606(83)90201-4. PMID 6684600.
- ^ ab Sammut, Michele; Cook, Steven J.; Nguyen, Ken CQ; Felton, Terry; Hall, David H.; Emmons, Scott W.; Poole, Richard J.; Barrios, Arantza (octubre de 2015). "Las neuronas derivadas de la glía son necesarias para el aprendizaje específico del sexo en C. elegans". Nature . 526 (7573): 385–390. Bibcode :2015Natur.526..385S. doi :10.1038/nature15700. ISSN 0028-0836. PMC 4650210 . PMID 26469050.
- ^ Ohno, Hayao; Bao, Zhirong (14 de noviembre de 2020). "Los ARN pequeños acoplan programas de desarrollo embrionario a microbios intestinales". bioRxiv . doi :10.1101/2020.11.13.381830. S2CID 227060212.
- ^ Ruppert EE, Fox RS, Barnes RD (2004). Zoología de invertebrados (7.ª ed.). Cengage Learning. pág. 753. ISBN 978-81-315-0104-7.
- ^ Hu, Minqi; Ma, Yeping; Chua, Song Lin (16 de enero de 2024). "Los nematodos bacteriófagos descifran sideróforos de hierro microbianos como señal de presa en interacciones depredador-presa". Actas de la Academia Nacional de Ciencias . 121 (3): e2314077121. Bibcode :2024PNAS..12114077H. doi : 10.1073/pnas.2314077121 . ISSN 0027-8424. PMC 10801909 . PMID 38190542.
- ^ Félix MA, Braendle C (noviembre de 2010). "La historia natural de Caenorhabditis elegans". Current Biology . 20 (22): R965–9. Bibcode :2010CBio...20.R965F. doi : 10.1016/j.cub.2010.09.050 . PMID 21093785.
- ^ Chan, Shepherd Yuen; Liu, Sylvia Yang; Wu, Rongben; Wei, Wei; Fang, James Kar-Hei; Chua, Song Lin (2023-06-02). "Difusión simultánea de nanoplásticos y resistencia a los antibióticos por mensajeros nematodos". Ciencia y tecnología medioambiental . 57 (23): 8719–8727. Bibcode :2023EnST...57.8719C. doi :10.1021/acs.est.2c07129. ISSN 0013-936X. PMID 37267481. S2CID 259047038.
- ^ Mylonakis E, Ausubel FM, Perfect JR, Heitman J, Calderwood SB (noviembre de 2002). "Muerte de Caenorhabditis elegans por Cryptococcus neoformans como modelo de patogénesis de levaduras". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (24): 15675–80. Bibcode :2002PNAS...9915675M. doi : 10.1073/pnas.232568599 . PMC 137775 . PMID 12438649.
- ^ Sifri CD, Begun J, Ausubel FM, Calderwood SB (abril de 2003). "Caenorhabditis elegans como huésped modelo para la patogénesis de Staphylococcus aureus". Infección e inmunidad . 71 (4): 2208–17. doi :10.1128/IAI.71.4.2208-2217.2003. PMC 152095 . PMID 12654843.
- ^ Tan MW, Mahajan-Miklos S, Ausubel FM (enero de 1999). "Eliminación de Caenorhabditis elegans por Pseudomonas aeruginosa utilizada para modelar la patogénesis bacteriana en mamíferos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 96 (2): 715–20. Bibcode :1999PNAS...96..715T. doi : 10.1073/pnas.96.2.715 . PMC 15202 . PMID 9892699.
- ^ Garsin DA, Villanueva JM, Begun J, Kim DH, Sifri CD, Calderwood SB, et al. (junio de 2003). "Los mutantes daf-2 de C. elegans de larga vida son resistentes a patógenos bacterianos". Science . 300 (5627): 1921. doi :10.1126/science.1080147. PMID 12817143. S2CID 37703980.
- ^ Chan, Shepherd Yuen; Liu, Sylvia Yang; Seng, Zijing; Chua, Song Lin (enero de 2021). "La matriz de biopelícula altera la motilidad y el comportamiento depredador de los nematodos". The ISME Journal . 15 (1): 260–269. Bibcode :2021ISMEJ..15..260C. doi :10.1038/s41396-020-00779-9. ISSN 1751-7370. PMC 7852553 . PMID 32958848.
- ^ Li, Shaoyang; Liu, Sylvia Yang; Chan, Shepherd Yuen; Chua, Song Lin (mayo de 2022). "La matriz de biopelícula oculta los quimioatrayentes de detección de quórum bacteriano para evitar la detección de depredadores". The ISME Journal . 16 (5): 1388–1396. Bibcode :2022ISMEJ..16.1388L. doi :10.1038/s41396-022-01190-2. ISSN 1751-7370. PMC 9038794 . PMID 35034106.
- ^ Kiontke K, Sudhaus W (enero de 2006). "Ecología de las especies de Caenorhabditis". Libro de gusanos : 1–14. doi : 10.1895/wormbook.1.37.1. PMC 4780885 . PMID 18050464.
- ^ Gal TZ, Glazer I, Koltai H (noviembre de 2004). "Un miembro de la familia LEA grupo 3 está involucrado en la supervivencia de C. elegans durante la exposición al estrés". FEBS Letters . 577 (1–2): 21–6. Bibcode :2004FEBSL.577...21G. doi : 10.1016/j.febslet.2004.09.049 . PMID 15527756. S2CID 21960486.
- ^ Elaine R. Ingham Introducción a la biología del suelo USDA
- ^ Bowen, NJ (1999). "El análisis genómico de Caenorhabditis elegans revela antiguas familias de elementos similares a los retrovirales". Genome Research . 9 (10): 924–935. doi : 10.1101/gr.9.10.924 . PMID 10523521.
- ^ Cuomo CA, Desjardins CA, Bakowski MA, Goldberg J, Ma AT, Becnel JJ, Didier ES, Fan L, Heiman DI, Levin JZ, Young S, Zeng Q, Troemel ER (diciembre de 2012). "El análisis del genoma de los microsporidios revela estrategias evolutivas para el crecimiento intracelular obligado". Genome Research . 22 (12): 2478–88. doi :10.1101/gr.142802.112. PMC 3514677 . PMID 22813931.
- ^ Niu XM, Zhang KQ (2011). "Arthrobotrys oligospora: Un organismo modelo para comprender la interacción entre hongos y nematodos". Micología . 2 (2): 59–78. doi : 10.1080/21501203.2011.562559 .
- ^ Clare JJ, Tate SN, Nobbs M, Romanos MA (noviembre de 2000). "Canales de sodio dependientes de voltaje como objetivos terapéuticos". Drug Discovery Today . 5 (11): 506–520. doi :10.1016/S1359-6446(00)01570-1. PMID 11084387.
- ^ Kosinski RA, Zaremba M (2007). "Dinámica del modelo de la red neuronal de Caenorhabditis Elegans". Acta Physica Polonica B . 38 (6): 2201. Código Bibliográfico :2007AcPPB..38.2201K.
- ^ Brouillette, Monique (21 de abril de 2022). "Mapear el cerebro para comprender la mente". Revista Knowable | Reseñas anuales . doi : 10.1146/knowable-042122-1 .
- ^ Watts DJ, Strogatz SH (junio de 1998). "Dinámica colectiva de redes de 'mundo pequeño'". Nature . 393 (6684): 440–2. Bibcode :1998Natur.393..440W. doi :10.1038/30918. PMID 9623998. S2CID 4429113.
- ^ Schafer WR (septiembre de 2005). "Descifrando los mecanismos neuronales y moleculares del comportamiento de C. elegans". Current Biology . 15 (17): R723–9. Bibcode :2005CBio...15.R723S. doi : 10.1016/j.cub.2005.08.020 . PMID 16139205.
- ^ Cook SJ, Jarrell TA, Brittin CA, Wang Y, Bloniarz AE, Yakovlev MA, et al. (julio de 2019). "Conectomas de animales completos de ambos sexos de Caenorhabditis elegans". Naturaleza . 571 (7763): 63–71. Código Bib :2019Natur.571...63C. doi :10.1038/s41586-019-1352-7. PMC 6889226 . PMID 31270481.
- ^ ab Alcántar-Fernández J, Navarro RE, Salazar-Martínez AM, Pérez-Andrade ME, Miranda-Ríos J (2018). "Caenorhabditis elegans responde a dietas ricas en glucosa a través de una red de factores de transcripción que responden al estrés". MÁS UNO . 13 (7): e0199888. Código Bib : 2018PLoSO..1399888A. doi : 10.1371/journal.pone.0199888 . PMC 6039004 . PMID 29990370.
- ^ Avery, L. "Sydney Brenner". Southwestern Medical Center . Archivado desde el original el 15 de agosto de 2011.URL alternativa archivada el 8 de diciembre de 2013 en Wayback Machine
- ^ Brenner, S (1974). "La genética de Caenorhabditis elegans". Genética . 77 (1): 71–94. doi :10.1093/genetics/77.1.71. PMC 1213120 . PMID 4366476.
- ^ "Comportamiento". www.wormbook.org . Consultado el 26 de septiembre de 2018 .
- ^ Sulston JE, Horvitz HR (marzo de 1977). "Líneas celulares postembrionarias del nematodo Caenorhabditis elegans". Biología del desarrollo . 56 (1): 110–56. doi :10.1016/0012-1606(77)90158-0. PMID 838129.
- ^ Kimble J, Hirsh D (junio de 1979). "Los linajes celulares postembrionarios de las gónadas hermafroditas y masculinas en Caenorhabditis elegans". Biología del desarrollo . 70 (2): 396–417. doi :10.1016/0012-1606(79)90035-6. PMID 478167.
- ^ Peden E, Killian DJ, Xue D (agosto de 2008). "Especificación de muerte celular en C. elegans". Ciclo celular . 7 (16): 2479–84. doi :10.4161/cc.7.16.6479. PMC 2651394 . PMID 18719375.
- ^ NIDDK, Instituto Nacional de Diabetes y Enfermedades Digestivas y Renales (5 de marzo de 2015). "Inyección de gónadas de C. elegans". YouTube . Archivado desde el original el 17 de noviembre de 2021. Consultado el 21 de marzo de 2020 .
- ^ Kamath RS, Fraser AG, Dong Y, Poulin G, Durbin R, Gotta M, Kanapin A, Le Bot N, Moreno S, Sohrmann M, Welchman DP, Zipperlen P, Ahringer J (enero de 2003). "Análisis funcional sistemático del genoma de Caenorhabditis elegans utilizando RNAi". Nature . 421 (6920): 231–7. Bibcode :2003Natur.421..231K. doi :10.1038/nature01278. hdl :10261/63159. PMID 12529635. S2CID 15745225.
- ^ Fortunato A, Fraser AG (2005). "Descubrimiento de interacciones genéticas en Caenorhabditis elegans mediante interferencia de ARN". Bioscience Reports . 25 (5–6): 299–307. doi :10.1007/s10540-005-2892-7. PMID 16307378. S2CID 6983519.
- ^ Félix MA (noviembre de 2008). "Interferencia de ARN en nematodos y la probabilidad que favoreció a Sydney Brenner". Journal of Biology . 7 (9): 34. doi : 10.1186/jbiol97 . PMC 2776389 . PMID 19014674.
- ^ Winston WM, Sutherlin M, Wright AJ, Feinberg EH, Hunter CP (junio de 2007). "Se requiere SID-2 de Caenorhabditis elegans para la interferencia del ARN ambiental". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (25): 10565–70. Bibcode :2007PNAS..10410565W. doi : 10.1073/pnas.0611282104 . PMC 1965553 . PMID 17563372.
- ^ Takanami T, Mori A, Takahashi H, Higashitani A (noviembre de 2000). "Hiperresistencia de las células meióticas a la radiación debido a una fuerte expresión de un único gen similar a recA en Caenorhabditis elegans". Nucleic Acids Research . 28 (21): 4232–6. doi :10.1093/nar/28.21.4232. PMC 113154 . PMID 11058122.
- ^ Takanami T, Zhang Y, Aoki H, Abe T, Yoshida S, Takahashi H, Horiuchi S, Higashitani A (septiembre de 2003). "Reparación eficiente del daño del ADN inducido por partículas de iones pesados en núcleos de profase meiótica I de Caenorhabditis elegans". Revista de investigación sobre radiación . 44 (3): 271–6. Código Bib : 2003JRadR..44..271T. doi : 10.1269/jrr.44.271 . PMID 14646232.
- ^ Chin GM, Villeneuve AM (marzo de 2001). "El mre-11 de C. elegans es necesario para la recombinación meiótica y la reparación del ADN, pero es prescindible para el punto de control de daño del ADN G(2) meiótico". Genes & Development . 15 (5): 522–34. doi :10.1101/gad.864101. PMC 312651 . PMID 11238374.
- ^ Barrière A, Félix MA (julio de 2005). "Alta diversidad genética local y baja tasa de cruzamiento en poblaciones naturales de Caenorhabditis elegans". Current Biology . 15 (13): 1176–84. arXiv : q-bio/0508003 . Bibcode :2005CBio...15.1176B. doi :10.1016/j.cub.2005.06.022. PMID 16005289. S2CID 2229622.
- ^ Bernstein H, Bernstein C (julio de 2010) "Origen evolutivo de la recombinación durante la meiosis", BioScience 60(7), 498-505. https://doi.org/10.1525/bio.2010.60.7.5
- ^ Feng Z, Li W, Ward A, Piggott BJ, Larkspur ER, Sternberg PW, Xu XZ (noviembre de 2006). "Un modelo de C. elegans de la conducta dependiente de la nicotina: regulación por los canales de la familia TRP". Cell . 127 (3): 621–33. doi :10.1016/j.cell.2006.09.035. PMC 2859215 . PMID 17081982.
- ^ "Foro de la comunidad de la Alianza de Recursos Genómicos". Foro de la comunidad de la Alianza de Recursos Genómicos . Consultado el 1 de agosto de 2024 .
- ^ Wolkow CA, Kimura KD, Lee MS, Ruvkun G (octubre de 2000). "Regulación de la longevidad de C. elegans mediante señalización similar a la insulina en el sistema nervioso". Science . 290 (5489): 147–50. Bibcode :2000Sci...290..147W. doi :10.1126/science.290.5489.147. PMID 11021802.
- ^ Ewald CY, Landis JN, Porter Abate J, Murphy CT, Blackwell TK (marzo de 2015). "La señalización de insulina/IGF-1 independiente de Dauer implica la remodelación del colágeno en la longevidad". Nature . 519 (7541): 97–101. Bibcode :2015Natur.519...97E. doi :10.1038/nature14021. PMC 4352135 . PMID 25517099.
- ^ Venz, Richard; Pekec, Tina; Katic, Iskra; Ciosk, Rafal; Ewald, Collin Yvès (10 de septiembre de 2021). Leiser, Scott F; Kaeberlein, Matt; Alcedo, Joy (eds.). "La degradación dirigida al final de la vida del receptor de insulina/IGF-1 DAF-2 promueve la longevidad libre de patologías relacionadas con el crecimiento". eLife . 10 : e71335. doi : 10.7554/eLife.71335 . ISSN 2050-084X. PMC 8492056 . PMID 34505574.
- ^ ab Hyun, Moonjung; Lee, Jihyun; Lee, Kyungjin; May, Alfred; Bohr, Vilhelm A.; Ahn, Byungchan (2008). "La longevidad y la resistencia al estrés se correlacionan con la capacidad de reparación del ADN en Caenorhabditis elegans". Nucleic Acids Research . 36 (4): 1380–1389. doi :10.1093/nar/gkm1161. PMC 2275101 . PMID 18203746.
- ^ Meyer, Joel N.; Boyd, Windy A.; Azzam, Gregory A.; Haugen, Astrid C.; Freedman, Jonathan H.; Van Houten, Bennett (2007). "Disminución de la capacidad de reparación por escisión de nucleótidos en el envejecimiento de Caenorhabditis elegans". Genome Biology . 8 (5): R70. doi : 10.1186/gb-2007-8-5-r70 . PMC 1929140 . PMID 17472752.
- ^ McColl G, Killilea DW, Hubbard AE, Vantipalli MC, Melov S, Lithgow GJ (enero de 2008). "Análisis farmacogenético del envejecimiento retardado inducido por litio en Caenorhabditis elegans". The Journal of Biological Chemistry . 283 (1): 350–7. doi : 10.1074/jbc.M705028200 . PMC 2739662 . PMID 17959600.
- ^ Zarse K, Terao T, Tian J, Iwata N, Ishii N, Ristow M (agosto de 2011). "La absorción de litio en dosis bajas promueve la longevidad en humanos y metazoos". Revista Europea de Nutrición . 50 (5): 387–9. doi :10.1007/s00394-011-0171-x. PMC 3151375 . PMID 21301855.
- ^ Ewald CY, Li C (marzo de 2010). "Comprensión de la base molecular de la enfermedad de Alzheimer utilizando un sistema modelo de Caenorhabditis elegans". Estructura y función cerebral . 214 (2–3): 263–83. doi :10.1007/s00429-009-0235-3. PMC 3902020 . PMID 20012092.
- ^ Hanazawa M, Yonetani M, Sugimoto A (marzo de 2011). "Las proteínas PGL se autoasocian y se unen a las RNP para mediar el ensamblaje de los gránulos germinales en C. elegans". The Journal of Cell Biology . 192 (6): 929–37. doi :10.1083/jcb.201010106. PMC 3063142 . PMID 21402787.
- ^ Coutts, Fiona; Palmos, Alish B.; Duarte, Rodrigo RR; de Jong, Simone; Lewis, Cathryn M.; Dima, Danai; Powell, Timothy R. (marzo de 2019). "La naturaleza poligénica de la longitud de los telómeros y las propiedades antienvejecimiento del litio". Neuropsicofarmacología . 44 (4): 757–765. doi :10.1038/s41386-018-0289-0. ISSN 1740-634X. PMC 6372618 . PMID 30559463.
- ^ Raices, Marcela; Maruyama, Hugo; Dillin, Andrew; Karlseder, Jan (septiembre de 2005). "Desacoplamiento de la longevidad y la longitud de los telómeros en C. elegans". PLOS Genetics . 1 (3): e30. doi : 10.1371/journal.pgen.0010030 . ISSN 1553-7404. PMC 1200426 . PMID 16151516.
- ^ Lulkiewicz, M.; Bajsert, J.; Kopczynski, P.; Barczak, W.; Rubis, B. (septiembre de 2020). "Longitud de los telómeros: cómo la longitud marca la diferencia". Molecular Biology Reports . 47 (9): 7181–7188. doi :10.1007/s11033-020-05551-y. ISSN 1573-4978. PMC 7561533 . PMID 32876842.
- ^ Pardue, Mary-Lou; DeBaryshe, PG (2011-12-20). "Retrotransposones que mantienen los extremos de los cromosomas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (51): 20317–20324. doi : 10.1073/pnas.1100278108 . ISSN 1091-6490. PMC 3251079 . PMID 21821789.
- ^ Meier, Bettina; Clejan, Iuval; Liu, Yan; Lowden, Mia; Gartner, Anton; Hodgkin, Jonathan; Ahmed, Shawn (febrero de 2006). "trt-1 es la subunidad catalítica de la telomerasa de Caenorhabditis elegans". PLOS Genetics . 2 (2): e18. doi : 10.1371/journal.pgen.0020018 . ISSN 1553-7404. PMC 1361356 . PMID 16477310.
- ^ Cesare, Anthony J.; Reddel, Roger R. (mayo de 2010). "Alargamiento alternativo de los telómeros: modelos, mecanismos e implicaciones". Nature Reviews. Genética . 11 (5): 319–330. doi :10.1038/nrg2763. ISSN 1471-0064. PMID 20351727. S2CID 19224032.
- ^ Ijomone, Omamuyovwi M.; Miah, Mahfuzur R.; Peres, Tanara V.; Nwoha, Polycarp U.; Aschner, Michael (diciembre de 2016). "Los mutantes de alelo nulo de trt-1, la subunidad catalítica de la telomerasa en Caenorhabditis elegans, son menos sensibles a la toxicidad inducida por Mn y a la degeneración DAérgica". Neurotoxicología . 57 : 54–60. Bibcode :2016NeuTx..57...54I. doi :10.1016/j.neuro.2016.08.016. ISSN 1872-9711. PMID 27593554.
- ^ Shtessel, Ludmila; Lowden, Mia Rochelle; Cheng, Chen; Simon, Matt; Wang, Kyle; Ahmed, Shawn (febrero de 2013). "POT-1 y POT-2 de Caenorhabditis elegans reprimen las vías de mantenimiento de los telómeros". G3: Genes, Genomas, Genética . 3 (2): 305–313. doi :10.1534/g3.112.004440. ISSN 2160-1836. PMC 3564990 . PMID 23390606.
- ^ Kwon, Mi-Sun; Min, Jaewon; Jeon, Hee-Yeon; Hwang, Kwangwoo; Kim, Chuna; Lee, Junho; Joung, Je-Gun; Park, Woong-Yang; Lee, Hyunsook (octubre de 2016). "Retraso paradójico de la senescencia tras el agotamiento de BRCA2 en gusanos deficientes en telomerasa". FEBS Open Bio . 6 (10): 1016–1024. doi :10.1002/2211-5463.12109. ISSN 2211-5463. PMC 5055038 . PMID 27761361.
- ^ Bayat, Melih; Tanny, Robyn E.; Wang, Ye; Herden, Carla; Daniel, Jens; Andersen, Erik C.; Liebau, Eva; Waschk, Daniel EJ (30 de marzo de 2020). "Efectos de la sobreexpresión de la telomerasa en el organismo modelo Caenorhabditis elegans". Gene . 732 : 144367. doi :10.1016/j.gene.2020.144367. ISSN 1879-0038. PMID 31954861. S2CID 210829489.
- ^ Iwanir S, Tramm N, Nagy S, Wright C, Ish D, Biron D (marzo de 2013). "La microarquitectura del comportamiento de C. elegans durante el letargo: dinámica de ataques homeostáticos, una postura corporal típica y regulación por una neurona central". Sleep . 36 (3): 385–95. doi :10.5665/Sleep.2456. PMC 3571756 . PMID 23449971.
- ^ Hill AJ, Mansfield R, Lopez JM, Raizen DM, Van Buskirk C (octubre de 2014). "El estrés celular induce un estado protector similar al sueño en C. elegans". Current Biology . 24 (20): 2399–405. Bibcode :2014CBio...24.2399H. doi :10.1016/j.cub.2014.08.040. PMC 4254280 . PMID 25264259.
- ^ Gusanos diminutos y sin ojos tienen células detectoras de luz completamente nuevas
- ^ Scientific American, agosto de 2018, página 14
- ^ "Los gusanos sobrevivieron al desastre del Columbia". BBC News . 1 de mayo de 2003 . Consultado el 11 de julio de 2008 .
- ^ "La universidad envía gusanos al espacio". BBC News . 17 de enero de 2009 . Consultado el 9 de julio de 2009 .
- ^ Klotz, I (16 de mayo de 2011). "Gusanos espaciales heredados volando en el transbordador". Discovery News . Archivado desde el original el 16 de junio de 2012. Consultado el 17 de mayo de 2011 .
- ^ Soni, Purushottam; Anupom, Taslim; Lesanpezeshki, Leila; Rahman, Mizanur; Hewitt, Jennifer E.; Vellone, Matthew; Stodieck, Louis; Blawzdziewicz, Jerzy; Szewczyk, Nathaniel J.; Vanapalli, Siva A. (7 de noviembre de 2022). "Hardware de vuelo espacial integrado con microfluidos para medir la fuerza muscular de Caenorhabditis elegans en la Estación Espacial Internacional". npj Microgravity . 8 (1): 50. Bibcode :2022npjMG...8...50S. doi :10.1038/s41526-022-00241-4. ISSN 2373-8065. PMC 9640571 . PMID 36344513.
- ^ Strome S, Kelly WG, Ercan S, Lieb JD (marzo de 2014). "Regulación de los cromosomas X en Caenorhabditis elegans". Cold Spring Harbor Perspectives in Biology . 6 (3): a018366. doi :10.1101/cshperspect.a018366. PMC 3942922 . PMID 24591522.
- ^ El Consorcio de Secuenciación de C. elegans (diciembre de 1998). "Secuencia del genoma del nematodo C. elegans : una plataforma para investigar la biología". Science . 282 (5396): 2012–8. Bibcode :1998Sci...282.2012.. doi :10.1126/science.282.5396.2012. PMID 9851916.
- ^ ab Sadler, J. Evan (1998). "Bioquímica y genética del factor de von Willebrand". Revisión anual de bioquímica . 67 (1). Revisiones anuales : 395–424. doi : 10.1146/annurev.biochem.67.1.395 . ISSN 0066-4154. PMID 9759493.
- ^ ab Hahn, Mark E. (2002). "Receptores de hidrocarburos arílicos: diversidad y evolución". Interacciones químico-biológicas . 141 (1–2). Elsevier : 131–160. Bibcode :2002CBI...141..131H. doi :10.1016/s0009-2797(02)00070-4. ISSN 0009-2797. PMID 12213389.
- ^ ab Bustelo, Xosé R. (2000). "Propiedades reguladoras y de señalización de la familia Vav". Biología molecular y celular . 20 (5). Sociedad Americana de Microbiología : 1461–1477. doi :10.1128/mcb.20.5.1461-1477.2000. ISSN 0270-7306. PMC 85310 . PMID 10669724.
- ^ Blumenthal T, Evans D, Link CD, Guffanti A, Lawson D, Thierry-Mieg J, Thierry-Mieg D, Chiu WL, Duke K, Kiraly M, Kim SK (junio de 2002). "Un análisis global de los operones de Caenorhabditis elegans ". Nature . 417 (6891): 851–4. Código Bibliográfico :2002Natur.417..851B. doi :10.1038/nature00831. PMID 12075352. S2CID 4351788.
- ^ Blumenthal T (noviembre de 2004). "Operones en eucariotas". Briefings in Functional Genomics & Proteomics . 3 (3): 199–211. doi : 10.1093/bfgp/3.3.199 . PMID 15642184.
- ^ "WS227 Release Letter". WormBase . 10 de agosto de 2011. Archivado desde el original el 28 de noviembre de 2013 . Consultado el 19 de noviembre de 2013 .
- ^ Ruby JG, Jan C, Player C, Axtell MJ, Lee W, Nusbaum C, Ge H, Bartel DP (diciembre de 2006). "La secuenciación a gran escala revela 21U-RNA y microRNA adicionales y siRNA endógenos en C. elegans". Cell . 127 (6): 1193–207. doi : 10.1016/j.cell.2006.10.040 . PMID 17174894.
- ^ Stricklin SL, Griffiths-Jones S, Eddy SR (junio de 2005). "Genes de ARN no codificantes de C. elegans". WormBook : 1–7. doi :10.1895/wormbook.1.1.1. PMC 4781554 . PMID 18023116.
- ^ "WS202 Release Letter". WormBase . 29 de mayo de 2009. Archivado desde el original el 1 de diciembre de 2013 . Consultado el 19 de noviembre de 2013 .
- ^ "WS197 Release Letter". WormBase . 27 de noviembre de 2008. Archivado desde el original el 17 de octubre de 2019 . Consultado el 19 de noviembre de 2013 .
- ^ "Cambios en la secuencia del genoma". WormBase . 15 de junio de 2011. Archivado desde el original el 17 de octubre de 2019. Consultado el 13 de agosto de 2011 .
- ^ Stein LD, Bao Z, Blasiar D, Blumenthal T, Brent MR, Chen N, Chinwalla A, Clarke L, Clee C, Coghlan A, Coulson A, D'Eustachio P, Fitch DH, Fulton LA, Fulton RE, Griffiths- Jones S, Harris TW, Hillier LW, Kamath R, Kuwabara PE, Mardis ER, Marra MA, Miner TL, Minx P, Mullikin JC, Plumb RW, Rogers J, Schein JE, Sohrmann M, Spieth J, Stajich JE, Wei C , Willey D, Wilson RK, Durbin R, Waterston RH (noviembre de 2003). "La secuencia del genoma de Caenorhabditis briggsae: una plataforma para la genómica comparada". Más biología . 1 (2): E45. doi : 10.1371/journal.pbio.0000045 . PMC 261899. PMID 14624247 .
- ^ Centro de secuenciación del genoma. «Caenorhabditis remanei: antecedentes». Facultad de Medicina de la Universidad de Washington . Archivado desde el original el 16 de junio de 2008. Consultado el 11 de julio de 2008 .
- ^ Centro de secuenciación del genoma. «Caenorhabditis japonica: antecedentes». Facultad de Medicina de la Universidad de Washington . Archivado desde el original el 26 de junio de 2008. Consultado el 11 de julio de 2008 .
- ^ Staden R (junio de 1979). "Una estrategia de secuenciación de ADN empleando programas informáticos". Nucleic Acids Research . 6 (7): 2601–10. doi :10.1093/nar/6.7.2601. PMC 327874 . PMID 461197.
- ^ "Navegador de genomas de la UCSC" . Consultado el 8 de julio de 2014 .
- ^ Kuhn RM, Karolchik D, Zweig AS, Wang T, Smith KE, Rosenbloom KR, Rhead B, Raney BJ, Pohl A, Pheasant M, Meyer L, Hsu F, Hinrichs AS, Harte RA, Giardine B, Fujita P, Diekhans M, Dreszer T, Clawson H, Barber GP, Haussler D, Kent WJ (enero de 2009). "Base de datos del navegador del genoma de la UCSC: actualización de 2009". Nucleic Acids Research . 37 (número de la base de datos): D755–61. doi :10.1093/nar/gkn875. PMC 2686463 . PMID 18996895.
- ^ Félix MA, Braendle C, Cutter AD (2014). "Un sistema simplificado para el diagnóstico de especies en Caenorhabditis (Nematoda: Rhabditidae) con designaciones de nombres para 15 especies biológicas distintas". PLOS ONE . 9 (4): e94723. Bibcode :2014PLoSO...994723F. doi : 10.1371/journal.pone.0094723 . PMC 3984244 . PMID 24727800.
- ^ Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC (febrero de 1998). "Interferencia genética potente y específica por ARN bicatenario en Caenorhabditis elegans". Nature . 391 (6669): 806–11. Bibcode :1998Natur.391..806F. doi :10.1038/35888. PMID 9486653. S2CID 4355692.
- ^ Callaway, Ewen; Sanderson, Katharine (7 de octubre de 2024). «Nobel de medicina otorgado a los microARN reguladores de genes». Nature Publishing Group . Consultado el 8 de octubre de 2024 .
- ^ Harris TW, Antoshechkin I, Bieri T, Blasiar D, Chan J, Chen WJ, et al. (enero de 2010). "WormBase: un recurso completo para la investigación sobre nematodos". Nucleic Acids Research . 38 (número de la base de datos): D463-7. doi :10.1093/nar/gkp952. PMC 2808986 . PMID 19910365.
Lectura adicional
- Bird J, Bird AC (1991). La estructura de los nematodos . Academic Press . Págs. 1, 69–70, 152–153, 165, 224–225. ISBN. 978-0-12-099651-3.
- Esperanza, IA (1999).C. elegans : un enfoque práctico . Oxford University Press . Págs. 1–6. ISBN. 978-0-19-963738-6.
- Riddle DL, Blumenthal T, Meyer RJ, Priess JR (1997). C. elegans II. Cold Spring Harbor Laboratory Press . págs. 1–4, 679–683. ISBN 978-0-87969-532-3. Número de identificación personal 21413221.
Enlaces externos
Caenorhabditis elegans (categoría)
- Brenner S (2002) El regalo de la naturaleza a la ciencia. En http://nobelprize.org/nobel_prizes/medicine/laureates/2002/brenner-lecture.pdf (también conferencias de Horvitz y Sulston)
- WormBase: una extensa base de datos en línea que cubre la biología y genómica de C. elegans y otros nematodos
- WormAtlas: base de datos en línea sobre todos los aspectos de la anatomía de C. elegans con explicaciones detalladas e imágenes de alta calidad
- WormBook: revisión en línea de la biología de C. elegans
- AceView WormGenes: otra base de datos del genoma de C. elegans , mantenida en el NCBI
- C. elegans II – un libro de texto gratuito en línea.
- Red neuronal WormWeb: una herramienta en línea para visualizar y navegar por el conectoma de C. elegans
- Películas de C. elegans: una introducción visual a C. elegans
- Vea el ensamblaje del genoma ce11 en el navegador de genoma de la UCSC .
- Caenorhabditis elegans en eppo.int ( código EPPO CAEOEL)
- Cylinder, Drew (2022). "Uso de C. elegans para estudiar la anestesia". doi :10.6084/m9.figshare.20963590.v1.
{{cite journal}}: Requiere citar revista|journal=( ayuda )


