Ribonucleasa H
| ribonucleasa H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura cristalográfica de la ARNasa HI de E. coli . [1] | |||||||||
| Identificadores | |||||||||
| N.º CE | 3.1.26.4 | ||||||||
| N.º CAS | 9050-76-4 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| Ontología genética | AmiGO / QuickGO | ||||||||
| |||||||||
| ribonucleasa retroviral H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| N.º CE | 3.1.26.13 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| |||||||||
La ribonucleasa H (abreviada como ARNasa H o RNH ) es una familia de enzimas endonucleasas no específicas de secuencia que catalizan la escisión del ARN en un sustrato de ARN/ ADN a través de un mecanismo hidrolítico . Los miembros de la familia de las ARNasas H se pueden encontrar en casi todos los organismos, desde bacterias hasta arqueas y eucariotas .
La familia se divide en grupos relacionados evolutivamente con preferencias de sustrato ligeramente diferentes , ampliamente designados ribonucleasa H1 y H2. [2] El genoma humano codifica tanto H1 como H2. La ribonucleasa humana H2 es un complejo heterotrimérico compuesto por tres subunidades, las mutaciones en cualquiera de las cuales se encuentran entre las causas genéticas de una enfermedad rara conocida como síndrome de Aicardi-Goutières . [3] Un tercer tipo, estrechamente relacionado con H2, se encuentra solo en unos pocos procariotas , [4] mientras que H1 y H2 ocurren en todos los dominios de la vida . [4] Además, los dominios H de ribonucleasa retroviral similares a la ARNasa H1 ocurren en proteínas de transcriptasa inversa multidominio , que están codificadas por retrovirus como el VIH y son necesarias para la replicación viral. [5] [6]
En los eucariotas, la ribonucleasa H1 está involucrada en la replicación del ADN del genoma mitocondrial . Tanto la H1 como la H2 están involucradas en tareas de mantenimiento del genoma, como el procesamiento de las estructuras de bucle R. [2] [7]
Clasificación y nomenclatura
La ribonucleasa H es una familia de enzimas endonucleasas con una especificidad de sustrato compartida para la cadena de ARN de los dúplex ARN - ADN . Por definición, las ARNasas H escinden los enlaces fosfodiéster de la cadena principal del ARN para dejar un grupo hidroxilo 3' y un grupo fosfato 5' . [7] Las ARNasas H se han propuesto como miembros de una superfamilia relacionada evolutivamente que abarca otras nucleasas y enzimas de procesamiento de ácidos nucleicos como las integrasas retrovirales , las transposasas de ADN , las resolvasas de unión de Holliday , las proteínas Piwi y Argonaute , varias exonucleasas y la proteína espliceosómica Prp8 . [8] [9]
Las ARNasas H pueden dividirse en dos subtipos, H1 y H2, que por razones históricas reciben designaciones con números arábigos en eucariotas y designaciones con números romanos en procariotas . Por lo tanto, la ARNasa HI de Escherichia coli es un homólogo de la ARNasa H1 de Homo sapiens . [2] [7] En E. coli y muchos otros procariotas, el gen rnhA codifica HI y el gen rnhB codifica HII. Una tercera clase relacionada, llamada HIII, se presenta en algunas bacterias y arqueas ; está estrechamente relacionada con las enzimas HII procariotas. [4]
Estructura
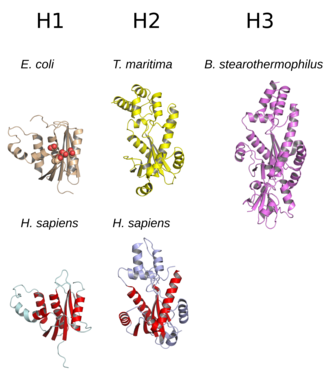
La estructura de la ARNasa H consiste comúnmente en una lámina β de cinco cadenas rodeada por una distribución de hélices α . [10] Todas las ARNasas H tienen un sitio activo centrado en un motivo de secuencia conservada compuesto por residuos de aspartato y glutamato , a menudo denominado motivo DEDD. Estos residuos interactúan con iones de magnesio necesarios catalíticamente. [7] [5]
Las ARNasas H2 son más grandes que las H1 y suelen tener hélices adicionales. La organización de los dominios de las enzimas varía; algunos miembros procariotas y la mayoría de los eucariotas del grupo H1 tienen un pequeño dominio adicional en el extremo N-terminal conocido como "dominio de unión híbrido", que facilita la unión a los dúplex híbridos ARN:ADN y, a veces, confiere una mayor procesividad . [2] [7] [11] Si bien todos los miembros del grupo H1 y los miembros procariotas del grupo H2 funcionan como monómeros, las enzimas H2 eucariotas son heterotrímeros obligados . [2] [7] Las enzimas HIII procariotas son miembros del grupo H2 más amplio y comparten la mayoría de las características estructurales con H2, con la adición de un dominio de unión de caja TATA N-terminal . [7] Los dominios H de ARNasa retroviral que se encuentran en las proteínas de transcriptasa inversa multidominio tienen estructuras que se parecen mucho al grupo H1. [5]
Las ARNasas H1 se han estudiado ampliamente para explorar las relaciones entre la estructura y la actividad enzimática. También se utilizan, especialmente el homólogo de E. coli , como sistemas modelo para estudiar el plegamiento de proteínas . [12] [13] [14] Dentro del grupo H1, se ha identificado una relación entre una mayor afinidad de unión al sustrato y la presencia de elementos estructurales que consisten en una hélice y un bucle flexible que proporcionan una superficie de unión al sustrato más grande y más básica . La hélice C tiene una distribución taxonómica dispersa; está presente en los homólogos de la ARNasa H1 de E. coli y humana y ausente en el dominio H de la ARNasa del VIH, pero existen ejemplos de dominios retrovirales con hélices C. [15] [16]
Función
Las enzimas ribonucleasas H escinden los enlaces fosfodiéster del ARN en un híbrido ARN:ADN de doble cadena, dejando un hidroxilo 3' y un grupo fosfato 5' en cada extremo del sitio de corte con un mecanismo de catálisis de dos iones metálicos, en el que dos cationes divalentes, como Mg2+ y Mn2+, participan directamente en la función catalítica. [17] Dependiendo de las diferencias en sus secuencias de aminoácidos, estas ARNasas H se clasifican en ARNasas H de tipo 1 y tipo 2. [7] [18] Las ARNasas H de tipo 1 tienen ARNasas H1 procariotas y eucariotas y ARNasa H retroviral. Las ARNasas H de tipo 2 tienen ARNasas H2 procariotas y eucariotas y ARNasa H3 bacteriana. Estas ARNasas H existen en forma monomérica, excepto las ARNasas H2 eucariotas, que existen en forma heterotrimérica. [19] [20] Las ARNasas H1 y H2 tienen preferencias de sustrato distintas y funciones distintas pero superpuestas en la célula. En procariotas y eucariotas inferiores, ninguna enzima es esencial , mientras que se cree que ambas son esenciales en eucariotas superiores. [2] La actividad combinada de las enzimas H1 y H2 está asociada con el mantenimiento de la estabilidad del genoma debido a la degradación por parte de las enzimas del componente de ARN de los bucles R. [21] [22]
Ribonucleasa H1
| Identificadores | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Símbolo | ARNasa H | ||||||||
| Pfam | PF00075 | ||||||||
| Clan Pfam | CL0219 | ||||||||
| Interprofesional | IPR002156 | ||||||||
| PROSITIO | PS50879 | ||||||||
| |||||||||
Las enzimas ribonucleasas H1 requieren al menos cuatro pares de bases que contengan ribonucleótidos en un sustrato y no pueden eliminar un solo ribonucleótido de una cadena que de otro modo estaría compuesta de desoxirribonucleótidos. Por esta razón, se considera poco probable que las enzimas RNasa H1 estén involucradas en el procesamiento de cebadores de ARN a partir de fragmentos de Okazaki durante la replicación del ADN . [2] La RNasa H1 no es esencial en los organismos unicelulares donde se ha investigado; en E. coli , los knockouts de RNasa H1 confieren un fenotipo sensible a la temperatura, [7] y en S. cerevisiae , producen defectos en la respuesta al estrés. [23]
En muchos eucariotas, incluidos los mamíferos , los genes de la ARNasa H1 incluyen una secuencia de orientación mitocondrial , lo que conduce a la expresión de isoformas con y sin el MTS presente. Como resultado, la ARNasa H1 se localiza tanto en las mitocondrias como en el núcleo . En modelos de ratones knockout , los mutantes nulos de la ARNasa H1 son letales durante la embriogénesis debido a defectos en la replicación del ADN mitocondrial . [2] [24] [25] Los defectos en la replicación del ADN mitocondrial inducidos por la pérdida de la ARNasa H1 probablemente se deban a defectos en el procesamiento del bucle R. [22]
Ribonucleasa H2
| Identificadores | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Símbolo | ARNasa HII | ||||||||
| Pfam | PF01351 | ||||||||
| Clan Pfam | CL0219 | ||||||||
| Interprofesional | IPR024567 | ||||||||
| |||||||||
En los procariotas, la ARNasa H2 es enzimáticamente activa como proteína monomérica. En los eucariotas, es un heterotrímero obligado compuesto por una subunidad catalítica A y subunidades estructurales B y C. Mientras que la subunidad A es estrechamente homóloga a la ARNasa H2 procariota, las subunidades B y C no tienen homólogos aparentes en los procariotas y están poco conservadas a nivel de secuencia incluso entre los eucariotas. [26] [27] La subunidad B media las interacciones proteína-proteína entre el complejo H2 y PCNA , que localiza H2 en focos de replicación . [28]
Tanto las enzimas procariotas como las eucariotas H2 pueden escindir ribonucleótidos individuales en una hebra. [2] Sin embargo, tienen patrones de escisión y preferencias de sustrato ligeramente diferentes: las enzimas procariotas tienen menor procesividad e hidrolizan ribonucleótidos sucesivos de manera más eficiente que los ribonucleótidos con un desoxirribonucleótido 5' , mientras que las enzimas eucariotas son más procesivas e hidrolizan ambos tipos de sustrato con una eficiencia similar. [2] [27] La especificidad del sustrato de la ARNasa H2 le otorga un papel en la reparación por escisión de ribonucleótidos , eliminando ribonucleótidos mal incorporados del ADN, además del procesamiento del bucle R. [29] [30] [28] Aunque tanto H1 como H2 están presentes en el núcleo de la célula de los mamíferos , H2 es la fuente dominante de actividad de la ARNasa H allí y es importante para mantener la estabilidad del genoma. [28]
Algunos procariotas poseen un gen adicional de tipo H2 denominado ARNasa HIII en la nomenclatura de números romanos utilizada para los genes procariotas. Las proteínas HIII están más estrechamente relacionadas con el grupo H2 por identidad de secuencia y similitud estructural, pero tienen preferencias de sustrato que se asemejan más a las H1. [7] [31] A diferencia de HI y HII, que están ampliamente distribuidas entre los procariotas, HIII se encuentra solo en unos pocos organismos con una distribución taxonómica dispersa; es algo más común en las arqueas y rara vez o nunca se encuentra en el mismo genoma procariota que HI. [32]
Mecanismo

El sitio activo de casi todas las ARNasas H contiene cuatro residuos de aminoácidos cargados negativamente, conocidos como motivo DEDD; a menudo también está presente una histidina, por ejemplo, en el VIH-1, el ser humano o E. coli. [2] [7]
Los residuos cargados se unen a dos iones metálicos que son necesarios para la catálisis; en condiciones fisiológicas, estos son iones de magnesio , pero el manganeso también suele favorecer la actividad enzimática, [2] [7] mientras que el calcio o una alta concentración de Mg2+ inhibe la actividad. [11] [33] [34]
Basándose en evidencia experimental y simulaciones por computadora, la enzima activa una molécula de agua unida a uno de los iones metálicos con la histidina conservada. [33] [35] El estado de transición es asociativo por naturaleza [17] y forma un intermedio con fosfato protonado y grupo saliente alcóxido desprotonado. [35] El grupo saliente se protona a través del glutamato que tiene un pKa elevado y es probable que esté protonado. El mecanismo es similar al de la ARNasa T y la subunidad RuvC en la enzima Cas9 , que también utilizan un mecanismo de histidina y dos iones metálicos.
El mecanismo de liberación del producto escindido aún no se ha resuelto. La evidencia experimental obtenida mediante cristalografía con resolución temporal y nucleasas similares apunta a un papel de un tercer ion en la reacción reclutado en el sitio activo. [36] [37]
En biología humana
El genoma humano contiene cuatro genes que codifican la ARNasa H:
- RNASEH1 , un ejemplo del subtipo H1 (monomérico)
- RNASEH2A , la subunidad catalítica del complejo trimérico H2
- RNASEH2B , una subunidad estructural del complejo trimérico H2
- RNASEH2C , una subunidad estructural del complejo trimérico H2
Además, el material genético de origen retroviral aparece con frecuencia en el genoma, lo que refleja la integración de los genomas de los retrovirus endógenos humanos . Dichos eventos de integración dan como resultado la presencia de genes que codifican la transcriptasa inversa retroviral , que incluye un dominio de ARNasa H. Un ejemplo es ERVK6 . [38] Los retrotransposones de repetición terminal larga (LTR) y de repetición terminal no larga (no LTR) también son comunes en el genoma y a menudo incluyen sus propios dominios de ARNasa H, con una historia evolutiva compleja. [39] [40] [41]
Papel en la enfermedad

En estudios pequeños, las mutaciones en la ARNasa H1 humana se han asociado con oftalmoplejía externa progresiva crónica , una característica común de la enfermedad mitocondrial . [25]
Las mutaciones en cualquiera de las tres subunidades de la ARNasa H2 están bien establecidas como causas de un trastorno genético raro conocido como síndrome de Aicardi-Goutières (AGS), [3] que se manifiesta como síntomas neurológicos y dermatológicos a una edad temprana. [43] Los síntomas del AGS se parecen mucho a los de la infección viral congénita y están asociados con una regulación positiva inapropiada del interferón tipo I. El AGS también puede ser causado por mutaciones en otros genes: TREX1 , SAMHD1 , ADAR y MDA5 /IFIH1, todos los cuales están involucrados en el procesamiento de ácidos nucleicos. [44] La caracterización de la distribución mutacional en una población de pacientes con AGS encontró un 5% de todas las mutaciones de AGS en RNASEH2A, un 36% en 2B y un 12% en 2C. [45] Las mutaciones en 2B se han asociado con un deterioro neurológico algo más leve [46] y con una ausencia de regulación positiva de genes inducida por interferón que se puede detectar en pacientes con otros genotipos asociados a AGS. [44]
En los virus

Dos grupos de virus utilizan la transcripción inversa como parte de sus ciclos de vida: los retrovirus , que codifican sus genomas en ARN monocatenario y se replican a través de un intermediario de ADN bicatenario; y los virus dsDNA-RT , que replican sus genomas de ADN bicatenario a través de un intermediario de ARN "pregenoma". Los ejemplos patógenos incluyen el virus de la inmunodeficiencia humana y el virus de la hepatitis B , respectivamente. Ambos codifican grandes proteínas multifuncionales de transcriptasa inversa (RT) que contienen dominios de ARNasa H. [48] [49]
Las proteínas RT retrovirales del VIH-1 y del virus de la leucemia murina son los miembros de la familia mejor estudiados. [50] [51] La RT retroviral es responsable de convertir el genoma de ARN monocatenario del virus en ADN bicatenario. Este proceso requiere tres pasos: primero, la actividad de la ADN polimerasa dependiente del ARN produce ADN de cadena negativa a partir de la plantilla de ARN de cadena positiva, generando un intermedio híbrido ARN:ADN; segundo, se destruye la cadena de ARN; y tercero, la actividad de la ADN polimerasa dependiente del ADN sintetiza ADN de cadena positiva, generando ADN bicatenario como producto final. El segundo paso de este proceso lo lleva a cabo un dominio H de la ARNasa ubicado en el extremo C de la proteína RT. [5] [6] [52] [53]
La ARNasa H realiza tres tipos de acciones de escisión: degradación no específica del genoma de ARN de cadena positiva, eliminación específica del cebador de ARNt de cadena negativa y eliminación del cebador del tracto de polipurina (PPT) rico en purina de cadena positiva. [54] La ARNasa H desempeña un papel en la preparación de la cadena positiva, pero no en el método convencional de sintetizar una nueva secuencia de cebador. En cambio, la ARNasa H crea un "cebador" a partir del PPT que es resistente a la escisión de la ARNasa H. Al eliminar todas las bases excepto el PPT, el PPT se utiliza como marcador para el final de la región U3 de su repetición terminal larga . [53]
Debido a que la actividad de la ARNasa H es necesaria para la proliferación viral, este dominio se ha considerado un objetivo farmacológico para el desarrollo de fármacos antirretrovirales utilizados en el tratamiento del VIH/SIDA y otras afecciones causadas por retrovirus. Se han identificado inhibidores de la ARNasa H retroviral de varios quimiotipos diferentes, muchos de los cuales tienen un mecanismo de acción basado en la quelación de los cationes del sitio activo. [55] Los inhibidores de la transcriptasa inversa que inhiben específicamente la función de la polimerasa de la RT se utilizan ampliamente en la clínica, pero no los inhibidores de la función de la ARNasa H; es la única función enzimática codificada por el VIH que aún no es el objetivo de los fármacos en uso clínico. [52] [56]
Evolución
Las ARNasas H están ampliamente distribuidas y se encuentran en todos los dominios de la vida . La familia pertenece a una superfamilia más grande de enzimas nucleasas [8] [9] y se considera evolutivamente antigua. [57] En los genomas procariotas, a menudo están presentes múltiples genes de ARNasa H, pero hay poca correlación entre la aparición de genes HI, HII y HIII y las relaciones filogenéticas generales , lo que sugiere que la transferencia horizontal de genes puede haber jugado un papel en el establecimiento de la distribución de estas enzimas. Las ARNasas HI y HIII rara vez o nunca aparecen en el mismo genoma procariota. Cuando el genoma de un organismo contiene más de un gen de ARNasa H, a veces tienen diferencias significativas en el nivel de actividad. Se ha sugerido que estas observaciones reflejan un patrón evolutivo que minimiza la redundancia funcional entre los genes de ARNasa H. [7] [32] La ARNasa HIII, que es exclusiva de los procariotas, tiene una distribución taxonómica dispersa y se encuentra tanto en bacterias como en arqueas ; [32] Se cree que se separó de HII bastante temprano. [58]
La trayectoria evolutiva de la ARNasa H2 en eucariotas, especialmente el mecanismo por el cual los homólogos eucariotas se convirtieron en heterotrímeros obligados, no está clara; las subunidades B y C no tienen homólogos aparentes en procariotas. [2] [27]
Aplicaciones
Debido a que la ARNasa H degrada específicamente solo el ARN en híbridos ARN:ADN de doble cadena, se utiliza comúnmente como reactivo de laboratorio en biología molecular . Las preparaciones purificadas de ARNasa HI y HII de E. coli están disponibles comercialmente. La ARNasa HI se utiliza a menudo para destruir la plantilla de ARN después de la síntesis de ADN complementario de primera cadena (ADNc) por transcripción inversa . También se puede utilizar para escindir secuencias de ARN específicas en presencia de segmentos complementarios cortos de ADN. [59] Se pueden utilizar técnicas altamente sensibles como la resonancia de plasmón de superficie para la detección. [60] [61] La ARNasa HII se puede utilizar para degradar el componente cebador de ARN de un fragmento de Okazaki o para introducir mellas monocatenarias en posiciones que contienen un ribonucleótido. [59] Se ha descrito una variante de PCR de inicio en caliente , conocida como PCR dependiente de ARNasa H o rhPCR, que utiliza una ARNasa HII termoestable de la arquea hipertermófila Pyrococcus abyssi . [62] Cabe destacar que la proteína inhibidora de ribonucleasa comúnmente utilizada como reactivo no es eficaz para inhibir la actividad de HI o HII. [59]
Historia
Las ribonucleasas H se descubrieron por primera vez en el laboratorio de Peter Hausen cuando los investigadores encontraron actividad endonucleasa híbrida ARN:ADN en timo de ternera en 1969 y le dieron el nombre de "ribonucleasa H " para designar su especificidad híbrida . [26] [63] [64] La actividad de la ARNasa H se descubrió posteriormente en E. coli [65] y en una muestra de oncovirus con genomas de ARN durante los primeros estudios de transcripción inversa viral . [66] [67] Más tarde quedó claro que el extracto de timo de ternera contenía más de una proteína con actividad de ARNasa H [68] y que E. coli contenía dos genes de ARNasa H. [69] [70] Originalmente, la enzima ahora conocida como ARNasa H2 en eucariotas se designaba H1 y viceversa, pero los nombres de las enzimas eucariotas se cambiaron para que coincidieran con los de E. coli para facilitar el análisis comparativo, lo que produjo la nomenclatura moderna en la que las enzimas procariotas se designan con números romanos y las enzimas eucariotas con números arábigos. [2] [26] [31] [71] La ARNasa HIII procariota, informada en 1999, fue el último subtipo de ARNasa H en ser identificado. [31]
La caracterización de la ARNasa H2 eucariota ha sido históricamente un desafío, en parte debido a su baja abundancia. [2] Los esfuerzos cuidadosos de purificación de la enzima sugirieron que, a diferencia de la ARNasa H2 de E. coli , la enzima eucariota tenía múltiples subunidades. [72] El homólogo de S. cerevisiae de la proteína de E. coli (es decir, la subunidad H2A) fue fácilmente identificable por bioinformática cuando se secuenció el genoma de la levadura, [73] pero se encontró que la proteína correspondiente no tenía actividad enzimática de forma aislada. [2] [23] Finalmente, las subunidades B y C de la levadura se aislaron por copurificación y se encontró que eran necesarias para la actividad enzimática. [74] Sin embargo, las subunidades B y C de la levadura tienen una identidad de secuencia muy baja con sus homólogos en otros organismos, y las proteínas humanas correspondientes se identificaron de manera concluyente solo después de que se encontró que las mutaciones en las tres causaban el síndrome de Aicardi-Goutières . [2] [3]
Referencias
- ^ PDB : 1JL1 ; Goedken ER, Marqusee S (diciembre de 2001). "Energética del estado nativo de una variante termoestabilizada de la ribonucleasa HI". Journal of Molecular Biology . 314 (4): 863–71. doi :10.1006/jmbi.2001.5184. PMID 11734003.
- ^ abcdefghijklmnopq Cerritelli SM, Crouch RJ (marzo de 2009). "Ribonucleasa H: las enzimas en eucariotas". Revista FEBS . 276 (6): 1494–505. doi :10.1111/j.1742-4658.2009.06908.x. PMC 2746905 . PMID 19228196.
- ^ abc Crow YJ, Leitch A, Hayward BE, Garner A, Parmar R, Griffith E, et al. (agosto de 2006). "Las mutaciones en los genes que codifican las subunidades H2 de la ribonucleasa causan el síndrome de Aicardi-Goutières y simulan una infección cerebral viral congénita". Nature Genetics . 38 (8): 910–6. doi :10.1038/ng1842. PMID 16845400. S2CID 8076225.
- ^ abc Figiel M, Nowotny M (agosto de 2014). "La estructura cristalina del complejo de sustrato-RNasa H3 revela una evolución paralela del reconocimiento híbrido ARN/ADN". Nucleic Acids Research . 42 (14): 9285–94. doi :10.1093/nar/gku615. PMC 4132731 . PMID 25016521.
- ^ abcd Davies JF, Hostomska Z, Hostomsky Z, Jordan SR, Matthews DA (abril de 1991). "Estructura cristalina del dominio H de la ribonucleasa de la transcriptasa inversa del VIH-1". Science . 252 (5002): 88–95. Bibcode :1991Sci...252...88D. doi :10.1126/science.1707186. PMID 1707186.
- ^ ab Hansen J, Schulze T, Mellert W, Moelling K (enero de 1988). "Identificación y caracterización de la ARNasa H específica del VIH mediante anticuerpos monoclonales". The EMBO Journal . 7 (1): 239–43. doi :10.1002/j.1460-2075.1988.tb02805.x. PMC 454263 . PMID 2452083.
- ^ abcdefghijklm Tadokoro T, Kanaya S (marzo de 2009). "Ribonucleasa H: diversidad molecular, dominios de unión al sustrato y mecanismo catalítico de las enzimas procariotas". The FEBS Journal . 276 (6): 1482–93. doi :10.1111/j.1742-4658.2009.06907.x. PMID 19228197. S2CID 29008571.
- ^ ab Majorek KA, Dunin-Horkawicz S, Steczkiewicz K, Muszewska A, Nowotny M, Ginalski K, Bujnicki JM (abril de 2014). "La superfamilia de ARNasas tipo H: nuevos miembros, análisis estructural comparativo y clasificación evolutiva". Investigación de ácidos nucleicos . 42 (7): 4160–79. doi :10.1093/nar/gkt1414. PMC 3985635 . PMID 24464998.
- ^ ab Rice P, Craigie R, Davies DR (febrero de 1996). "Integrasas retrovirales y sus primas". Current Opinion in Structural Biology . 6 (1): 76–83. doi :10.1016/s0959-440x(96)80098-4. PMID 8696976.
- ^ Schmitt TJ, Clark JE, Knotts TA (diciembre de 2009). "Plegamiento multiestatal térmico y mecánico de la ribonucleasa H". The Journal of Chemical Physics . 131 (23): 235101. Bibcode :2009JChPh.131w5101S. doi :10.1063/1.3270167. PMID 20025349.
- ^ ab Nowotny M, Cerritelli SM, Ghirlando R, Gaidamakov SA, Crouch RJ, Yang W (abril de 2008). "Reconocimiento específico de híbridos ARN/ADN y mejora de la actividad de la ARNasa H1 humana mediante HBD". The EMBO Journal . 27 (7): 1172–81. doi :10.1038/emboj.2008.44. PMC 2323259 . PMID 18337749.
- ^ Cecconi C, Shank EA, Bustamante C, Marqusee S (septiembre de 2005). "Observación directa del plegamiento en tres estados de una única molécula de proteína". Science . 309 (5743): 2057–60. Bibcode :2005Sci...309.2057C. doi :10.1126/science.1116702. PMID 16179479. S2CID 43823877.
- ^ Hollien J, Marqusee S (marzo de 1999). "Una comparación termodinámica de las ribonucleasas mesófilas y termófilas H". Bioquímica . 38 (12): 3831–6. doi :10.1021/bi982684h. PMID 10090773.
- ^ Raschke TM, Marqusee S (abril de 1997). "El intermediario del plegamiento cinético de la ribonucleasa H se asemeja al glóbulo fundido ácido y a las moléculas parcialmente desplegadas detectadas en condiciones nativas". Nature Structural Biology . 4 (4): 298–304. doi :10.1038/nsb0497-298. PMID 9095198. S2CID 33673059.
- ^ Schultz SJ, Champoux JJ (junio de 2008). "Actividad de la ARNasa H: estructura, especificidad y función en la transcripción inversa". Virus Research . 134 (1–2): 86–103. doi :10.1016/j.virusres.2007.12.007. PMC 2464458 . PMID 18261820.
- ^ Champoux JJ, Schultz SJ (marzo de 2009). "Ribonucleasa H: propiedades, especificidad de sustrato y funciones en la transcripción inversa retroviral". The FEBS Journal . 276 (6): 1506–16. doi :10.1111/j.1742-4658.2009.06909.x. PMC 2742777 . PMID 19228195.
- ^ ab Yang W, Lee JY, Nowotny M (abril de 2006). "Fabricación y descomposición de ácidos nucleicos: catálisis de dos iones Mg2+ y especificidad del sustrato". Molecular Cell . 22 (1): 5–13. doi : 10.1016/j.molcel.2006.03.013 . PMID 16600865.
- ^ Ohtani N, Haruki M, Morikawa M, Kanaya S (enero de 1999). "Diversidades moleculares de las ARNasas H". Revista de biociencias y bioingeniería . 88 (1): 12–9. doi :10.1016/s1389-1723(99)80168-6. PMID 16232566.
- ^ Bubeck D, Reijns MA, Graham SC, Astell KR, Jones EY, Jackson AP (mayo de 2011). "PCNA dirige la actividad de la ARNasa H de tipo 2 en sustratos de replicación y reparación del ADN". Nucleic Acids Research . 39 (9): 3652–66. doi :10.1093/nar/gkq980. PMC 3089482 . PMID 21245041.
- ^ Figiel M, Chon H, Cerritelli SM, Cybulska M, Crouch RJ, Nowotny M (marzo de 2011). "La caracterización estructural y bioquímica del complejo humano RNasa H2 revela la base molecular para el reconocimiento del sustrato y los defectos del síndrome de Aicardi-Goutières". The Journal of Biological Chemistry . 286 (12): 10540–50. doi : 10.1074/jbc.M110.181974 . PMC 3060507 . PMID 21177858.
- ^ Amon JD, Koshland D (diciembre de 2016). "La ARNasa H permite una reparación eficiente del daño del ADN inducido por el bucle R". eLife . 5 : e20533. doi : 10.7554/eLife.20533 . PMC 5215079 . PMID 27938663.
- ^ ab Lima WF, Murray HM, Damle SS, Hart CE, Hung G, De Hoyos CL, et al. (junio de 2016). "Los ratones knock-out viables de RNaseH1 muestran que RNaseH1 es esencial para el procesamiento del bucle R, la función mitocondrial y hepática". Nucleic Acids Research . 44 (11): 5299–312. doi :10.1093/nar/gkw350. PMC 4914116 . PMID 27131367.
- ^ ab Arudchandran A, Cerritelli S, Narimatsu S, Itaya M, Shin DY, Shimada Y, Crouch RJ (octubre de 2000). "La ausencia de ribonucleasa H1 o H2 altera la sensibilidad de Saccharomyces cerevisiae a la hidroxiurea, la cafeína y el metanosulfonato de etilo: implicaciones para las funciones de las ARNasas H en la replicación y reparación del ADN". Genes to Cells . 5 (10): 789–802. doi : 10.1046/j.1365-2443.2000.00373.x . PMID 11029655.
- ^ Cerritelli SM, Frolova EG, Feng C, Grinberg A, Love PE, Crouch RJ (marzo de 2003). "La incapacidad de producir ADN mitocondrial da como resultado letalidad embrionaria en ratones sin Rnaseh1". Molecular Cell . 11 (3): 807–15. doi : 10.1016/s1097-2765(03)00088-1 . PMID 12667461.
- ^ ab Reyes A, Melchionda L, Nasca A, Carrara F, Lamantea E, Zanolini A, et al. (julio de 2015). "Las mutaciones de RNASEH1 perjudican la replicación del ADNmt y causan encefalomiopatía mitocondrial de inicio en la edad adulta". American Journal of Human Genetics . 97 (1): 186–93. doi :10.1016/j.ajhg.2015.05.013. PMC 4572567 . PMID 26094573.
- ^ abc Hollis T, Shaban NM (1 de enero de 2011). "Estructura y función de las enzimas ARNasa H". En Nicholson AW (ed.). Ribonucleasas . Ácidos nucleicos y biología molecular. Springer Berlin Heidelberg. págs. 299–317. doi :10.1007/978-3-642-21078-5_12. ISBN 978-3-642-21077-8.
- ^ abc Chon H, Vassilev A, DePamphilis ML, Zhao Y, Zhang J, Burgers PM, et al. (enero de 2009). "Contribuciones de las dos subunidades accesorias, RNASEH2B y RNASEH2C, a la actividad y propiedades del complejo ARNasa H2 humano". Nucleic Acids Research . 37 (1): 96–110. doi :10.1093/nar/gkn913. PMC 2615623 . PMID 19015152.
- ^ abc Reijns MA, Jackson AP (agosto de 2014). "Ribonucleasa H2 en la salud y la enfermedad". Biochemical Society Transactions . 42 (4): 717–25. doi :10.1042/BST20140079. PMID 25109948.
- ^ Wahba L, Amon JD, Koshland D, Vuica-Ross M (diciembre de 2011). "La ARNasa H y múltiples factores de biogénesis del ARN cooperan para evitar que los híbridos ARN:ADN generen inestabilidad genómica". Molecular Cell . 44 (6): 978–88. doi :10.1016/j.molcel.2011.10.017. PMC 3271842 . PMID 22195970.
- ^ Kim N, Huang SN, Williams JS, Li YC, Clark AB, Cho JE, et al. (junio de 2011). "Procesamiento mutagénico de ribonucleótidos en ADN por la topoisomerasa I de levadura". Science . 332 (6037): 1561–4. Bibcode :2011Sci...332.1561K. doi :10.1126/science.1205016. PMC 3380281 . PMID 21700875.
- ^ abc Ohtani N, Haruki M, Morikawa M, Crouch RJ, Itaya M, Kanaya S (enero de 1999). "Identificación de los genes que codifican la ARNasa HII dependiente de Mn2+ y la ARNasa HIII dependiente de Mg2+ de Bacillus subtilis: clasificación de las ARNasas H en tres familias". Bioquímica . 38 (2): 605–18. doi :10.1021/bi982207z. PMID 9888800.
- ^ abc Kochiwa H, Tomita M, Kanai A (julio de 2007). "Evolución de los genes de la ribonucleasa H en procariotas para evitar la herencia de genes redundantes". BMC Evolutionary Biology . 7 (1): 128. Bibcode :2007BMCEE...7..128K. doi : 10.1186/1471-2148-7-128 . PMC 1950709 . PMID 17663799.
- ^ ab Alla NR, Nicholson AW (diciembre de 2012). "Evidencia de un papel funcional dual de una histidina conservada en la escisión del heterodúplex ARN·ADN por la ARNasa H1 humana". FEBS Journal . 279 (24): 4492–500. doi :10.1111/febs.12035. PMC 3515698 . PMID 23078533.
- ^ Rosta E, Yang W, Hummer G (febrero de 2014). "Inhibición de calcio de la catálisis de iones de dos metales de la ribonucleasa H1". Journal of the American Chemical Society . 136 (8): 3137–44. doi :10.1021/ja411408x. PMC 3985467 . PMID 24499076.
- ^ ab Dürr S, Bohusewicz O, Berta D, Suardiaz R, Peter C, Jambrina PG, Peter C, Shao Y, Rosta E (16 de junio de 2021). "El papel de los residuos conservados en el motivo DEDDh: el mecanismo de transferencia de protones de la ARNasa H del VIH-1". Catálisis ACS . 11 (13): 7915–7927. doi :10.1021/acscatal.1c01493. S2CID 236285134.
- ^ Gan J, Shaw G, Tropea JE, Waugh DS, Court DL, Ji X (enero de 2008). "Un modelo escalonado para el procesamiento de ARN bicatenario por ribonucleasa III". Mol Microbiol . 67 (1): 143–54. doi : 10.1111/j.1365-2958.2007.06032.x . PMID 18047582.
- ^ Samara NL, Yang W (agosto de 2019). "El tráfico de cationes impulsa la hidrólisis del ARN". Nature Structural & Molecular Biology . 25 (8): 715–721. doi :10.1038/s41594-018-0099-4. PMC 6110950 . PMID 30076410.
- ^ Reus K, Mayer J, Sauter M, Scherer D, Müller-Lantzsch N, Meese E (marzo de 2001). "Organización genómica del retrovirus endógeno humano HERV-K(HML-2.HOM) (ERVK6) en el cromosoma 7". Genómica . 72 (3): 314–20. doi :10.1006/geno.2000.6488. PMID 11401447.
- ^ Ustyantsev K, Blinov A, Smyshlyaev G (14 de marzo de 2017). "Convergencia de retrotransposones en oomicetos y plantas". ADN móvil . 8 (1): 4. doi : 10.1186/s13100-017-0087-y . PMC 5348765 . PMID 28293305.
- ^ Ustyantsev K, Novikova O, Blinov A, Smyshlyaev G (mayo de 2015). "Evolución convergente de la ribonucleasa h en retrotransposones LTR y retrovirus". Biología molecular y evolución . 32 (5): 1197–207. doi :10.1093/molbev/msv008. PMC 4408406 . PMID 25605791.
- ^ Malik HS (2005). "Evolución de la ribonucleasa H en elementos retrotransponibles". Investigación citogenética y genómica . 110 (1–4): 392–401. doi :10.1159/000084971. PMID 16093691. S2CID 7481781.
- ^ Figiel M, Chon H, Cerritelli SM, Cybulska M, Crouch RJ, Nowotny M (marzo de 2011). "La caracterización estructural y bioquímica del complejo humano RNasa H2 revela la base molecular para el reconocimiento del sustrato y los defectos del síndrome de Aicardi-Goutières". The Journal of Biological Chemistry . 286 (12): 10540–50. doi : 10.1074/jbc.M110.181974 . PMC 3060507 . PMID 21177858.
- ^ Orcesi S, La Piana R, Fazzi E (2009). "Síndrome de Aicardi-Goutieres". Boletín médico británico . 89 : 183-201. doi : 10.1093/bmb/ldn049 . PMID 19129251.
- ^ ab Crow YJ, Manel N (julio de 2015). "Síndrome de Aicardi-Goutières y las interferonopatías de tipo I". Nature Reviews. Inmunología . 15 (7): 429–40. doi :10.1038/nri3850. PMID 26052098. S2CID 34259643.
- ^ Crow YJ, Chase DS, Lowenstein Schmidt J, Szynkiewicz M, Forte GM, Gornall HL, et al. (febrero de 2015). "Caracterización de fenotipos de enfermedades humanas asociadas con mutaciones en TREX1, RNASEH2A, RNASEH2B, RNASEH2C, SAMHD1, ADAR e IFIH1". American Journal of Medical Genetics. Parte A. 167A ( 2): 296–312. doi :10.1002/ajmg.a.36887. PMC 4382202. PMID 25604658 .
- ^ Rice G, Patrick T, Parmar R, Taylor CF, Aeby A, Aicardi J, et al. (octubre de 2007). "Fenotipo clínico y molecular del síndrome de Aicardi-Goutières". Revista Estadounidense de Genética Humana . 81 (4): 713–25. doi :10.1086/521373. PMC 2227922 . PMID 17846997.
- ^ Sarafianos SG, Das K, Tantillo C, Clark AD, Ding J, Whitcomb JM, et al. (marzo de 2001). "Estructura cristalina de la transcriptasa inversa del VIH-1 en un complejo con un tracto de polipurina ARN:ADN". The EMBO Journal . 20 (6): 1449–61. doi :10.1093/emboj/20.6.1449. PMC 145536 . PMID 11250910.
- ^ Seeger C, Mason WS (mayo de 2015). "Biología molecular de la infección por el virus de la hepatitis B". Virology . 479–480: 672–86. doi :10.1016/j.virol.2015.02.031. PMC 4424072 . PMID 25759099.
- ^ Moelling K, Broecker F, Kerrigan JE (1 de enero de 2014). "RNasa H: especificidad, mecanismos de acción y objetivo antiviral". En Vicenzi E, Poli G (eds.). Retrovirus humanos . Métodos en biología molecular. Vol. 1087. Humana Press. págs. 71–84. doi :10.1007/978-1-62703-670-2_7. ISBN 978-1-62703-669-6. Número de identificación personal 24158815.
- ^ Mizuno M, Yasukawa K, Inouye K (febrero de 2010). "Información sobre el mecanismo de estabilización de la transcriptasa inversa del virus de la leucemia murina de Moloney mediante la eliminación de la actividad de la ARNasa H". Biociencia, biotecnología y bioquímica . 74 (2): 440–2. doi :10.1271/bbb.90777. PMID 20139597. S2CID 28110533.
- ^ Coté ML, Roth MJ (junio de 2008). "Transcriptasa inversa del virus de la leucemia murina: comparación estructural con la transcriptasa inversa del VIH-1". Virus Research . 134 (1–2): 186–202. doi :10.1016/j.virusres.2008.01.001. PMC 2443788 . PMID 18294720.
- ^ ab Nowotny M, Figiel M (1 de enero de 2013). "El dominio H de la ARNasa: estructura, función y mecanismo". En LeGrice S, Gotte M (eds.). Transcriptasa inversa del virus de inmunodeficiencia humana . Springer Nueva York. págs. 53–75. doi :10.1007/978-1-4614-7291-9_3. ISBN 978-1-4614-7290-2.
- ^ ab Beilhartz GL, Götte M (abril de 2010). "Ribonucleasa H del VIH-1: estructura, mecanismo catalítico e inhibidores". Viruses . 2 (4): 900–26. doi : 10.3390/v2040900 . PMC 3185654 . PMID 21994660.
- ^ Klarmann GJ, Hawkins ME, Le Grice SF (2002). "El descubrimiento de las complejidades de la ribonucleasa H retroviral revela su potencial como objetivo terapéutico". AIDS Reviews . 4 (4): 183–94. PMID 12555693.
- ^ Tramontano E, Di Santo R (2010). "Inhibidores de la función de la ARNasa H asociada a la RT del VIH-1: avances recientes en el desarrollo de fármacos". Química medicinal actual . 17 (26): 2837–53. doi :10.2174/092986710792065045. PMID 20858167.
- ^ Cao L, Song W, De Clercq E, Zhan P, Liu X (junio de 2014). "Progresos recientes en la investigación de inhibidores de la ARNasa H del VIH-1 de moléculas pequeñas". Química medicinal actual . 21 (17): 1956–67. doi :10.2174/0929867321666140120121158. PMID 24438523.
- ^ Ma BG, Chen L, Ji HF, Chen ZH, Yang FR, Wang L, et al. (febrero de 2008). "Características de proteínas muy antiguas". Comunicaciones de investigación bioquímica y biofísica . 366 (3): 607–11. doi :10.1016/j.bbrc.2007.12.014. PMID 18073136.
- ^ Brindefalk B, Dessailly BH, Yeats C, Orengo C, Werner F, Poole AM (marzo de 2013). "Historia evolutiva de la superfamilia del dominio TBP". Nucleic Acids Research . 41 (5): 2832–45. doi :10.1093/nar/gkt045. PMC 3597702 . PMID 23376926.
- ^ abc Nichols NM, Yue D (1 de enero de 2001). Ribonucleasas . Vol. Capítulo 3. John Wiley & Sons, Inc. pp. Unidad 3.13. doi :10.1002/0471142727.mb0313s84. ISBN 978-0-471-14272-0. Número de identificación personal 18972385. Número de identificación personal 221604377.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Loo JF, Wang SS, Peng F, He JA, He L, Guo YC, et al. (julio de 2015). "Una plataforma SPR sin PCR que utiliza ARNasa H para detectar microARN 29a-3p a partir de hisopados de garganta de sujetos humanos con infección por el virus de la influenza A H1N1". The Analyst . 140 (13): 4566–75. Bibcode :2015Ana...140.4566L. doi :10.1039/C5AN00679A. PMID 26000345. S2CID 28974459.
- ^ Goodrich TT, Lee HJ, Corn RM (abril de 2004). "Detección directa de ADN genómico mediante mediciones de imágenes SPR amplificadas enzimáticamente de microarreglos de ARN". Journal of the American Chemical Society . 126 (13): 4086–7. CiteSeerX 10.1.1.475.1922 . doi :10.1021/ja039823p. PMID 15053580.
- ^ Dobosy JR, Rose SD, Beltz KR, Rupp SM, Powers KM, Behlke MA, Walder JA (agosto de 2011). "PCR dependiente de ARNasa H (rhPCR): especificidad mejorada y detección de polimorfismos de un solo nucleótido utilizando cebadores escindibles bloqueados". BMC Biotechnology . 11 : 80. doi : 10.1186/1472-6750-11-80 . PMC 3224242 . PMID 21831278.
- ^ Stein H, Hausen P (octubre de 1969). "Enzima del timo de ternera que degrada la fracción de ARN de los híbridos ADN-ARN: efecto sobre la ARN polimerasa dependiente del ADN". Science . 166 (3903): 393–5. Bibcode :1969Sci...166..393S. doi :10.1126/science.166.3903.393. PMID 5812039. S2CID 43683241.
- ^ Hausen P, Stein H (junio de 1970). "Ribonucleasa H. Una enzima que degrada la fracción de ARN de los híbridos ADN-ARN". Revista Europea de Bioquímica . 14 (2): 278–83. doi : 10.1111/j.1432-1033.1970.tb00287.x . PMID 5506170.
- ^ Miller HI, Riggs AD, Gill GN (abril de 1973). "Ribonucleasa H (híbrida) en Escherichia coli. Identificación y caracterización". The Journal of Biological Chemistry . 248 (7): 2621–4. doi : 10.1016/S0021-9258(19)44152-5 . PMID 4572736.
- ^ Mölling K, Bolognesi DP, Bauer H, Büsen W, Plassmann HW, Hausen P (diciembre de 1971). "Asociación de la transcriptasa inversa viral con una enzima que degrada la fracción de ARN de los híbridos ARN-ADN". Nature . 234 (51): 240–3. doi :10.1038/newbio234240a0. PMID 4331605.
- ^ Grandgenett DP, Gerard GF, Green M (diciembre de 1972). "Ribonucleasa H: una actividad ubicua en viriones de virus tumorales de ácido ribonucleico". Journal of Virology . 10 (6): 1136–42. doi :10.1128/jvi.10.6.1136-1142.1972. PMC 356594 . PMID 4118867.
- ^ Büsen W, Hausen P (marzo de 1975). "Actividades distintas de la ribonucleasa H en el timo de ternera". Revista Europea de Bioquímica . 52 (1): 179–90. doi : 10.1111/j.1432-1033.1975.tb03985.x . PMID 51794.
- ^ Kanaya S, Crouch RJ (enero de 1983). "Secuencia de ADN del gen que codifica la ribonucleasa H de Escherichia coli". The Journal of Biological Chemistry . 258 (2): 1276–81. doi : 10.1016/S0021-9258(18)33189-2 . PMID 6296074.
- ^ Itaya M (noviembre de 1990). "Aislamiento y caracterización de una segunda ARNasa H (ARNasa HII) de Escherichia coli K-12 codificada por el gen rnhB". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 87 (21): 8587–91. Bibcode :1990PNAS...87.8587I. doi : 10.1073/pnas.87.21.8587 . PMC 55002 . PMID 2172991.
- ^ Crouch RJ, Arudchandran A, Cerritelli SM (1 de enero de 2001). "RNasa H1 de Saccharomyces cerevisiae: métodos y nomenclatura". Ribonucleasas - Parte A. Métodos en enzimología. Vol. 341. págs. 395–413. doi :10.1016/s0076-6879(01)41166-9. ISBN 978-0-12-182242-2. Número de identificación personal 11582793.
- ^ Frank P, Braunshofer-Reiter C, Wintersberger U, Grimm R, Büsen W (octubre de 1998). "Clonación del ADNc que codifica la subunidad grande de la ARNasa HI humana, un homólogo de la ARNasa HII procariota". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 95 (22): 12872–7. Bibcode :1998PNAS...9512872F. doi : 10.1073/pnas.95.22.12872 . PMC 23637 . PMID 9789007.
- ^ Frank P, Braunshofer-Reiter C, Wintersberger U (enero de 1998). "La ARNasa H(35) de levadura es la contraparte de la ARNasa HI de los mamíferos y está relacionada evolutivamente con la ARNasa HII procariota". FEBS Letters . 421 (1): 23–6. doi :10.1016/s0014-5793(97)01528-7. PMID 9462832.
- ^ Jeong HS, Backlund PS, Chen HC, Karavanov AA, Crouch RJ (1 de enero de 2004). "La ARNasa H2 de Saccharomyces cerevisiae es un complejo de tres proteínas". Investigación de ácidos nucleicos . 32 (2): 407–14. doi :10.1093/nar/gkh209. PMC 373335 . PMID 14734815.
Enlaces externos
- Entrada de GeneReviews/NCBI/NIH/UW sobre el síndrome de Aicardi-Goutières
- RNasa+H en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
