Ribonucleasa H retroviral
| Ribonucleasa H retroviral | |||||||||
|---|---|---|---|---|---|---|---|---|---|
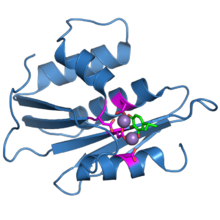 El dominio H de la ribonucleasa de la proteína transcriptasa inversa del VIH-1. Los cuatro residuos de carboxilato del sitio activo se muestran en magenta. Dos iones de manganeso unidos se muestran como esferas moradas. Una molécula inhibidora unida, beta-tuyaplicinol, se muestra en verde. [1] | |||||||||
| Identificadores | |||||||||
| N.º CE | 3.1.26.13 | ||||||||
| N.º CAS | 9050-76-4 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| |||||||||
La ribonucleasa H retroviral (RNasa H retroviral) es un dominio catalítico de la enzima transcriptasa inversa (RT) retroviral. La enzima RT se utiliza para generar ADN complementario ( ADNc ) a partir del genoma de ARN retroviral . Este proceso se llama transcripción inversa . Para completar este complejo proceso, las enzimas RT retrovirales deben adoptar una naturaleza multifuncional. Por lo tanto, poseen 3 de las siguientes actividades bioquímicas : ADN polimerasa dependiente de ARN , ribonucleasa H y actividades de ADN polimerasa dependiente de ADN . [2] Como todas las enzimas ARNasa H, el dominio ARNasa H retroviral escinde dúplex de ADN/ARN y no degradará ADN o ARN no hibridado.
Estructura


Aunque las estructuras RT de los retrovirus humanos , murinos y aviares muestran diferentes subunidades, los tamaños relativos, la orientación y la conexión de los dominios de la ADN polimerasa y la ARNasa H son sorprendentemente similares. El dominio de la ARNasa H ocupa ~25% del extremo C-terminal de la proteína RT . El dominio de la ADN polimerasa ocupa ~55% del extremo N-terminal de la proteína RT. [5] Los dominios de la ARNasa H de las enzimas RT de MMLV y VIH-1 son estructuralmente muy similares a las ARNasas H de Escherichia coli y Bacillus halodurans , así como a la ARNasa H1 humana . [6 ] [7] [8] [9] [10] En general, las estructuras plegadas de los dominios de la ARNasa H retroviral toman la forma de láminas beta mixtas de 5 hebras flanqueadas por cuatro hélices alfa en una distribución asimétrica. Una diferencia notable entre las distintas proteínas ARNasa H es la presencia o ausencia de la hélice C (presente en E. coli, MLV y ARNasas H humanas, ausente en VIH-1, B. halodurans y ARNasas H ASLV), una hélice alfa con carga positiva también conocida como bucle básico o protrusión. [10] Se cree que tiene un papel en la unión del sustrato. [10]

Función
Durante la transcripción inversa del ARN genómico viral en ADNc, se crea un híbrido ARN/ADN. Luego, la cadena de ARN es hidrolizada por el dominio ARNasa H para permitir la síntesis de la segunda cadena de ADN por la función ADN polimerasa de la enzima RT. [5] Además, los viriones retrovirales empaquetan una sola molécula de ARNt que utilizan como cebador durante la transcripción inversa del ARN genómico viral. [11] La ARNasa H retroviral es necesaria para digerir la molécula de ARNt cuando ya no es necesaria. Estos procesos ocurren de manera dependiente de Mg2+. [12] [13]
Las ARNasas retrovirales H escinden sus sustratos a través de tres modos diferentes:
- Escisión interna específica de secuencia del ARN [1-4]. Las enzimas del virus de inmunodeficiencia humana tipo 1 y del virus de la leucemia murina de Moloney prefieren escindir la cadena de ARN a un nucleótido de distancia de la unión ARN-ADN.
- Escisión dirigida del extremo 5' del ARN a 13-19 nucleótidos del extremo del ARN.
- Escisión dirigida del extremo 3' del ADN a 15-20 nucleótidos del extremo del cebador.
Los dos modos dirigidos al extremo son exclusivos de las ARNasas retrovirales H debido a una serie de efectos del dominio de polimerasa asociado de la RT retroviral. [6] En el modo de escisión interna más universal, las ARNasas H se comportan como endonucleasas típicas y escinden el ARN a lo largo de la longitud de un sustrato híbrido de ADN/ARN en ausencia de cualquier efecto "extremo". [14] [15] [16] [17]
Referencias
- ^ ab Himmel DM, Maegley KA, Pauly TA, Bauman JD, Das K, Dharia C, Clark AD, Ryan K, Hickey MJ, Love RA, Hughes SH, Bergqvist S, Arnold E (diciembre de 2009). "Estructura de la transcriptasa inversa del VIH-1 con el inhibidor beta-Thujaplicinol unido al sitio activo de la ARNasa H". Estructura . 17 (12): 1625–1635. doi :10.1016/j.str.2009.09.016. PMC 3365588 . PMID 20004166.
- ^ Worthington, Von (1993). Manual de enzimas de Worthington . Worthington. pág. 280.
- ^ Katayanagi K, Miyagawa M, Matsushima M, Ishikawa M, Kanaya S, Nakamura H, Ikehara M, Matsuzaki T, Morikawa K (febrero de 1992). "Detalles estructurales de la ribonucleasa H de Escherichia coli refinados hasta una resolución atómica". Journal of Molecular Biology . 223 (4): 1029–52. doi :10.1016/0022-2836(92)90260-q. PMID 1311386.
- ^ Sarafianos SG, Das K, Tantillo C, Clark AD, Ding J, Whitcomb JM, Boyer PL, Hughes SH, Arnold E (marzo de 2001). "Estructura cristalina de la transcriptasa inversa del VIH-1 en un complejo con un tracto de polipurina ARN:ADN". The EMBO Journal . 20 (6): 1449–61. doi :10.1093/emboj/20.6.1449. PMC 145536 . PMID 11250910.
- ^ ab Beilhartz GL, Götte M (abril de 2010). "Ribonucleasa H del VIH-1: estructura, mecanismo catalítico e inhibidores". Viruses . 2 (4): 900–26. doi : 10.3390/v2040900 . PMC 3185654 . PMID 21994660.
- ^ ab Lim D, Gregorio GG, Bingman C, Martinez-Hackert E, Hendrickson WA, Goff SP (septiembre de 2006). "Estructura cristalina del dominio H de la ARNasa del virus de la leucemia murina de Moloney". Journal of Virology . 80 (17): 8379–89. doi :10.1128/jvi.00750-06. PMC 1563865 . PMID 16912289.
- ^ Katayanagi K, Miyagawa M, Matsushima M, Ishikawa M, Kanaya S, Ikehara M, Matsuzaki T, Morikawa K (septiembre de 1990). "Estructura tridimensional de la ribonucleasa H de E. coli". Naturaleza . 347 (6290): 306–9. Código Bib :1990Natur.347..306K. doi :10.1038/347306a0. PMID 1698262. S2CID 4234320.
- ^ Yang W, Hendrickson WA, Crouch RJ, Satow Y (septiembre de 1990). "Estructura de la ribonucleasa H en fase con una resolución de 2 A mediante análisis MAD de la proteína selenometionil". Science . 249 (4975): 1398–405. doi :10.1126/science.2169648. PMID 2169648.
- ^ Nowotny M, Gaidamakov SA, Crouch RJ, Yang W (julio de 2005). "Estructuras cristalinas de la ARNasa H unida a un híbrido ARN/ADN: especificidad del sustrato y catálisis dependiente del metal". Cell . 121 (7): 1005–16. doi : 10.1016/j.cell.2005.04.024 . PMID 15989951.
- ^ abc Leo B, Schweimer K, Rösch P, Hartl MJ, Wöhrl BM (septiembre de 2012). "La estructura en solución del dominio H de la ARNasa del virus espumoso prototipo indica un papel importante del bucle básico en la unión del sustrato". Retrovirology . 9 (73): 73. doi : 10.1186/1742-4690-9-73 . PMC 3443672 . PMID 22962864.
- ^ Fu, Tie-Bo; John Taylor (27 de marzo de 1992). "Cuando las transcriptasas inversas retrovirales alcanzan el final de sus plantillas de ARN". Journal of Virology . 66 (7): 4271–4278. doi : 10.1128/JVI.66.7.4271-4278.1992 . PMC 241232 . PMID 1376369.
- ^ Taylor JM (marzo de 1977). "Análisis del papel de las especies de ARNt como iniciadores para la transcripción en ADN de genomas de virus tumorales de ARN". Biochimica et Biophysica Acta (BBA) - Reseñas sobre el cáncer . 473 (1): 57–71. doi :10.1016/0304-419x(77)90007-5. PMID 66067.
- ^ Talele TT, Upadhyay A, Pandey VN (octubre de 2009). "Influencia del dominio H de la ARNasa de las transcriptasas inversas retrovirales en la especificidad del metal y la selección del sustrato de sus dominios de polimerasa". Virology Journal . 6 (159): 159. doi : 10.1186/1743-422x-6-159 . PMC 2765437 . PMID 19814799.
- ^ Schultz SJ, Zhang M, Champoux JJ (noviembre de 2004). "Reconocimiento de sitios de escisión internos por ARNasas retrovirales H". Journal of Molecular Biology . 344 (3): 635–52. doi :10.1016/j.jmb.2004.09.081. PMID 15533434.
- ^ Krug MS, Berger SL (mayo de 1989). "Las actividades de la ribonucleasa H asociadas con las transcriptasas inversas virales son endonucleasas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 86 (10): 3539–43. Bibcode :1989PNAS...86.3539K. doi : 10.1073/pnas.86.10.3539 . PMC 287173 . PMID 2471188.
- ^ Champoux JJ, Schultz SJ (marzo de 2009). "Ribonucleasa H: propiedades, especificidad de sustrato y funciones en la transcripción inversa retroviral". The FEBS Journal . 276 (6): 1506–16. doi :10.1111/j.1742-4658.2009.06909.x. PMC 2742777 . PMID 19228195.
- ^ Schultz SJ, Champoux JJ (junio de 2008). "Actividad de la ARNasa H: estructura, especificidad y función en la transcripción inversa". Virus Research . 134 (1–2): 86–103. doi :10.1016/j.virusres.2007.12.007. PMC 2464458 . PMID 18261820.
Enlaces externos
- Retroviral+ribonucleasa+H en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
