Ribonucleasa T
| Ribonucleasa T | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Un dímero de ribonucleasa T en complejo con ADN (naranja), de PDB ID 3NH1. [1] | |||||||||
| Identificadores | |||||||||
| Símbolo | RNT-R ... | ||||||||
| Pfam | PF00929 | ||||||||
| Interprofesional | IPR013520 | ||||||||
| ELEGANTE | SM00479 | ||||||||
| |||||||||
La ribonucleasa T ( RNasa T , exonucleasa T , exo T ) es una enzima ribonucleasa involucrada en la maduración del ARN de transferencia y ARN ribosómico en bacterias , [2] así como en las vías de reparación del ADN . [3] Es un miembro de la familia DnaQ de exonucleasas y actúa de manera no procesiva en el extremo 3' de los ácidos nucleicos monocatenarios . La RNasa T es capaz de escindir tanto el ADN como el ARN , con una especificidad de secuencia extrema que discrimina contra la citosina en el extremo 3' del sustrato . [1] [2]
Estructura y mecanismo
La ARNasa T cataliza la eliminación de nucleótidos del extremo 3' tanto del ARN como del ADN. Es inhibida tanto por el ADN y el ARN de doble cadena como por los residuos de citosina en el extremo 3' del ARN. Dos citosinas en el extremo 3' del ARN parecen eliminar por completo la actividad de la ARNasa T. [3] Sin embargo, este efecto de la citosina se observa menos con el ADN monocatenario. Esta falta de especificidad de secuencia en el ADN monocatenario, combinada con su capacidad para actuar sobre el ADN monocatenario cerca de una región dúplex, ha llevado a su uso en la creación de extremos romos para la clonación de ADN. [4] Estructuralmente, la ARNasa T existe como un dímero antiparalelo [5] [6] y requiere un catión divalente para funcionar. [7]
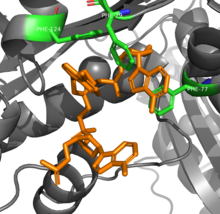
La ARNasa T es capaz de lograr su especificidad de secuencia en la digestión del ARN a través de varios residuos aromáticos que se intercalan entre las nucleobases. Las interacciones π -π entre los cuatro residuos de fenilalanina y los dos nucleótidos en el extremo 3' son diferentes según la identidad de los nucleótidos, lo que cambia la conformación y, por lo tanto, la actividad de la enzima. [8] Un residuo adicional de ácido glutámico gira para unirse a la citosina mediante un enlace de hidrógeno, no otras bases, lo que aumenta aún más la especificidad. [9]
Función
La ARNasa T, miembro de la gran familia de exorribonucleasas DEDD, desempeña un papel clave en la maduración del ARNt [10] , así como en la maduración de los dominios 5S [11] y 23S [12] del ARNr. En concreto, la ARNasa T escinde el residuo AMP 3' de las secuencias CCA 3' en el extremo del ARNt, lo que explica la especificidad de secuencia de la ARNasa T para detenerse en la secuencia CC 3'. [13] Además, la ARNasa T puede desempeñar un papel en la reparación del ADN escindiendo el extremo 3' del ADN de protuberancia. [3]
Aunque E. coli puede sobrevivir sin la ARNasa T, su ausencia conduce a ciclos de vida más lentos y una respuesta debilitada a la inanición. [14] Además, la presencia de la ARNasa T en E. coli está relacionada con una mayor resistencia al daño UV . [15] Se ha teorizado que, mientras que otras ribonucleasas pueden realizar la función de la ARNasa T, el hecho de que la ARNasa T sea más eficaz para escindir el ADN y el ARN cerca de regiones de doble cadena significa que las alternativas son menos efectivas. [16] A pesar de la aparente utilidad de la ARNasa T, la enzima solo se encuentra en gammaproteobacteria . [17]
En E. coli, la ARNasa T está codificada por el gen rnt y se plantea la hipótesis de que se separó de las subunidades correctoras de la polimerasa III durante la aparición de las gammaproteobacterias. [16] [17]
Referencias
- ^ ab Hsiao YY, Duh Y, Chen YP, Wang YT, Yuan HS (septiembre de 2012). "Cómo decide una exonucleasa dónde detenerse en el recorte de ácidos nucleicos: estructuras cristalinas de complejos de productos de la ARNasa T". Nucleic Acids Research . 40 (16): 8144–54. doi :10.1093/nar/gks548. PMC 3439924 . PMID 22718982.
- ^ ab Zuo Y, Deutscher MP (agosto de 2002). "El papel fisiológico de la ARNasa T puede explicarse por su inusual especificidad de sustrato". The Journal of Biological Chemistry . 277 (33): 29654–61. doi : 10.1074/jbc.M204252200 . PMID 12050169.
- ^ abc Hsiao YY, Fang WH, Lee CC, Chen YP, Yuan HS (marzo de 2014). "Información estructural sobre la reparación del ADN por la ARNasa T, una exonucleasa que procesa el extremo 3' del ADN estructurado en las vías de reparación". PLOS Biology . 12 (3): e1001803. doi : 10.1371/journal.pbio.1001803 . PMC 3942315 . PMID 24594808.
- ^ Zuo Y, Deutscher MP (octubre de 1999). "La actividad de la RNasa T y su aplicación a la clonación de ADN". Nucleic Acids Research . 27 (20): 4077–82. doi :10.1093/nar/27.20.4077. PMC 148676 . PMID 10497273.
- ^ Li Z, Zhan L, Deutscher MP (enero de 1996). "Escherichia coli RNase T funciona in vivo como un dímero dependiente de la cisteína 168". The Journal of Biological Chemistry . 271 (2): 1133–7. doi : 10.1074/jbc.271.2.1133 . PMID 8557641.
- ^ Zuo Y, Zheng H, Wang Y, Chruszcz M, Cymborowski M, Skarina T, et al. (abril de 2007). "Estructura cristalina de la ARNasa T, una exorribonucleasa implicada en la maduración y el recambio final del ARNt". Structure . 15 (4): 417–28. doi :10.1016/j.str.2007.02.004. PMC 1907377 . PMID 17437714.
- ^ Deutscher MP, Marlor CW (junio de 1985). "Purificación y caracterización de la ARNasa T de Escherichia coli". The Journal of Biological Chemistry . 260 (11): 7067–71. doi : 10.1016/S0021-9258(18)88888-3 . PMID 3888994.
- ^ Duh Y, Hsiao YY, Li CL, Huang JC, Yuan HS (diciembre de 2015). "Residuos aromáticos en la ARNasa T se apilan con nucleobases para guiar el reconocimiento y la escisión específicos de secuencia de los ácidos nucleicos". Protein Science . 24 (12): 1934–41. doi :10.1002/pro.2800. PMC 4815224 . PMID 26362012.
- ^ Hsiao YY, Yang CC, Lin CL, Lin JL, Duh Y, Yuan HS (abril de 2011). "Base estructural del recorte de ARN por la ARNasa T en la maduración estable del extremo 3' del ARN". Nature Chemical Biology . 7 (4): 236–43. doi :10.1038/nchembio.524. PMID 21317904.
- ^ Li Z, Deutscher MP (agosto de 1996). "Vías de maduración de los precursores de ARNt de E. coli: un proceso multienzimático aleatorio in vivo". Cell . 86 (3): 503–12. doi : 10.1016/s0092-8674(00)80123-3 . PMID 8756732.
- ^ Li Z, Deutscher MP (julio de 1995). "La enzima de procesamiento de ARNt, ARNasa T, es esencial para la maduración del ARN 5S". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 92 (15): 6883–6. Bibcode :1995PNAS...92.6883L. doi : 10.1073/pnas.92.15.6883 . PMC 41434 . PMID 7542780.
- ^ Li Z, Pandit S, Deutscher MP (enero de 1999). "La maduración del ARN ribosómico 23S requiere la exorribonucleasa ARNasa T". ARN . 5 (1): 139–46. doi :10.1017/s1355838299981669. PMC 1369746 . PMID 9917073.
- ^ Deutscher MP, Marlor CW, Zaniewski R (julio de 1984). "Ribonucleasa T: nueva exorribonucleasa posiblemente implicada en el recambio final del ARNt". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 81 (14): 4290–3. Bibcode :1984PNAS...81.4290D. doi : 10.1073/pnas.81.14.4290 . PMC 345573 . PMID 6379642.
- ^ Kelly KO, Deutscher MP (octubre de 1992). "La presencia de sólo una de las cinco exorribonucleasas es suficiente para sustentar el crecimiento de Escherichia coli". Journal of Bacteriology . 174 (20): 6682–4. doi :10.1128/jb.174.20.6682-6684.1992. PMC 207653 . PMID 1400219.
- ^ Viswanathan M, Lanjuin A, Lovett ST (marzo de 1999). "Identificación de la ARNasa T como un supresor de alto número de copias de la sensibilidad a los rayos UV asociada con la deficiencia de exonucleasa de ADN monocatenario en Escherichia coli". Genética . 151 (3): 929–34. doi :10.1093/genetics/151.3.929. PMC 1460521 . PMID 10049912.
- ^ ab Bechhofer DH, Deutscher MP (junio de 2019). "Ribonucleasas bacterianas y sus funciones en el metabolismo del ARN". Critical Reviews in Biochemistry and Molecular Biology . 54 (3): 242–300. doi :10.1080/10409238.2019.1651816. PMC 6776250 . PMID 31464530.
- ^ ab Zuo Y, Deutscher MP (marzo de 2001). "Superfamilias de exorribonucleasas: análisis estructural y distribución filogenética". Nucleic Acids Research . 29 (5): 1017–26. doi :10.1093/nar/29.5.1017. PMC 56904 . PMID 11222749.
