Virus de ARN
Partes de este artículo (las relacionadas con la taxonomía en las secciones de Baltimore [lanzamiento de ICTV 2018b→2019]) necesitan ser actualizadas . ( Enero de 2021 ) |

Un virus ARN es un virus —distinto de un retrovirus— que tiene ácido ribonucleico ( ARN ) como material genético . [1] El ácido nucleico suele ser ARN monocatenario ( ssRNA ), pero puede ser bicatenario (dsRNA). [2] Las enfermedades humanas notables causadas por virus ARN incluyen el resfriado común , la influenza , el SARS , el MERS , la COVID-19 , el virus del dengue , la hepatitis C , la hepatitis E , la fiebre del Nilo Occidental , la enfermedad por el virus del Ébola , la rabia , la poliomielitis , las paperas y el sarampión .
El Comité Internacional de Taxonomía de Virus (ICTV) clasifica a los virus ARN como aquellos que pertenecen al Grupo III , Grupo IV o Grupo V del sistema de clasificación de Baltimore . Esta categoría excluye al Grupo VI , virus con material genético ARN pero que utilizan intermediarios de ADN en su ciclo de vida : estos son los llamados retrovirus , [3] incluyendo el VIH-1 y el VIH-2 que causan el SIDA .
A partir de mayo de 2020, se cree que todos los virus de ARN conocidos que codifican una ARN polimerasa dirigida por ARN forman un grupo monofilético, conocido como el reino Riboviria . [4] La mayoría de estos virus de ARN caen en el reino Orthornavirae y el resto tiene un posicionamiento aún no definido . [5] El reino no contiene todos los virus de ARN: Deltavirus , Avsunviroidae y Pospiviroidae son taxones de virus de ARN que se incluyeron por error en 2019, [a] pero se corrigieron en 2020. [6]
Características
Virus de ARN monocatenario y ARN sentido
Los virus de ARN pueden clasificarse además según el sentido o polaridad de su ARN en virus de ARN de sentido negativo y de sentido positivo , o ambisentidos. El ARN viral de sentido positivo es similar al ARNm y, por lo tanto, puede ser traducido inmediatamente por la célula huésped. El ARN viral de sentido negativo es complementario al ARNm y, por lo tanto, debe convertirse en ARN de sentido positivo mediante una ARN polimerasa dependiente de ARN antes de la traducción. El ARN purificado de un virus de sentido positivo puede causar infección directamente, aunque puede ser menos infeccioso que la partícula viral completa. Por el contrario, el ARN purificado de un virus de sentido negativo no es infeccioso por sí mismo, ya que necesita ser transcrito en ARN de sentido positivo; cada virión puede transcribirse en varios ARN de sentido positivo. Los virus de ARN ambisentidos se parecen a los virus de ARN de sentido negativo, excepto que traducen genes de sus cadenas negativa y positiva. [7]
Virus de ARN bicatenario
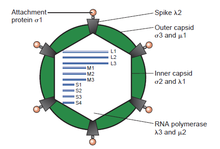
Los virus de doble cadena (ds)ARN representan un grupo diverso de virus que varían ampliamente en el rango de hospedadores (humanos, animales, plantas, hongos [ b] y bacterias ), número de segmentos del genoma (uno a doce) y organización del virión ( número de triangulación , capas de la cápside , picos, torretas, etc.). Los miembros de este grupo incluyen los rotavirus , que son la causa más común de gastroenteritis en niños pequeños, y los picobirnavirus , que son los virus más comunes en muestras fecales de humanos y animales con o sin signos de diarrea. El virus de la lengua azul es un patógeno económicamente importante que infecta al ganado vacuno y ovino. En los últimos años, se ha avanzado en la determinación de las estructuras de resolución atómica y subnanómetro de varias proteínas virales clave y cápsides de virión de varios virus dsARN, lo que resalta los paralelos significativos en la estructura y los procesos replicativos de muchos de estos virus. [2] [ página necesaria ]
Tasas de mutación
Los virus de ARN generalmente tienen tasas de mutación muy altas en comparación con los virus de ADN , [9] porque las polimerasas de ARN virales carecen de la capacidad de corrección de las polimerasas de ADN . [10] La diversidad genética de los virus de ARN es una de las razones por las que es difícil fabricar vacunas efectivas contra ellos. [11] Los retrovirus también tienen una alta tasa de mutación a pesar de que su intermedio de ADN se integra en el genoma del huésped (y, por lo tanto, está sujeto a la corrección de ADN del huésped una vez integrado), porque los errores durante la transcripción inversa se incrustan en ambas cadenas de ADN antes de la integración. [12] Algunos genes del virus de ARN son importantes para los ciclos de replicación viral y no se toleran las mutaciones. Por ejemplo, la región del genoma del virus de la hepatitis C que codifica la proteína central está altamente conservada , [13] porque contiene una estructura de ARN involucrada en un sitio de entrada interno al ribosoma . [14]
Complejidad de secuencia
En promedio, los virus dsRNA muestran una redundancia de secuencia menor en relación con los virus ssRNA. Por el contrario, los virus dsDNA contienen las secuencias genómicas más redundantes, mientras que los virus ssDNA tienen la menor cantidad. [15] Se ha demostrado que la complejidad de la secuencia de los virus es una característica clave para una clasificación viral precisa sin referencias. [15]
Replicación
Los virus ARN animales están clasificados por el ICTV. Existen tres grupos distintos de virus ARN según su genoma y modo de replicación:
- Los virus de ARN bicatenario (Grupo III) contienen de una a una docena de moléculas de ARN diferentes, cada una de las cuales codifica una o más proteínas virales.
- Los virus ssRNA de sentido positivo (Grupo IV) tienen su genoma utilizado directamente como ARNm, y los ribosomas del huésped lo traducen en una sola proteína que es modificada por las proteínas del huésped y del virus para formar las distintas proteínas necesarias para la replicación. Una de ellas incluye la ARN polimerasa dependiente de ARN (ARN replicasa), que copia el ARN viral para formar una forma replicativa de doble cadena. A su vez, este dsRNA dirige la formación de nuevo ARN viral.
- Los virus ssRNA de sentido negativo (grupo V) deben tener su genoma copiado por una ARN replicasa para formar ARN de sentido positivo. Esto significa que el virus debe llevar consigo la enzima ARN replicasa. La molécula de ARN de sentido positivo actúa entonces como ARNm viral, que es traducido en proteínas por los ribosomas del huésped.
Los retrovirus (Grupo VI) tienen un genoma de ARN monocatenario pero, en general, no se consideran virus de ARN porque utilizan intermediarios de ADN para replicarse. La transcriptasa inversa , una enzima viral que proviene del propio virus después de que este se desenrolla, convierte el ARN viral en una cadena complementaria de ADN, que se copia para producir una molécula de ADN viral de doble cadena. Después de que este ADN se integra en el genoma del huésped utilizando la enzima viral integrasa , la expresión de los genes codificados puede conducir a la formación de nuevos viriones.
Recombinación
Numerosos virus de ARN son capaces de recombinación genética cuando al menos dos genomas virales están presentes en la misma célula huésped. [16] Muy raramente el ARN viral puede recombinarse con el ARN del huésped. [17] La recombinación de ARN parece ser una fuerza impulsora importante en la determinación de la arquitectura del genoma y el curso de la evolución viral entre Picornaviridae ( (+)ssRNA ), p. ej. poliovirus . [18] En Retroviridae ((+)ssRNA), p. ej. VIH , el daño en el genoma de ARN parece evitarse durante la transcripción inversa mediante el cambio de cadena, una forma de recombinación. [19] [20] [21] La recombinación también ocurre en Reoviridae (dsRNA), p. ej. reovirus; Orthomyxoviridae ((-)ssRNA), p. ej. virus de la influenza ; [21] y Coronaviridae ((+)ssRNA), p. ej. SARS . [22] La recombinación en virus de ARN parece ser una adaptación para hacer frente al daño del genoma. [16] La recombinación puede ocurrir con poca frecuencia entre virus animales de la misma especie pero de linajes divergentes. Los virus recombinantes resultantes pueden causar a veces un brote de infección en humanos. [22]
Clasificación
Es posible que sea necesario limpiar esta sección para cumplir con los estándares de calidad de Wikipedia . El problema específico es que está desactualizada y es redundante con el artículo sobre riboviria , así como con textos posteriores; consulte Virus de ADN para una integración limpia entre ICTV de orden superior y Baltimore. ( Enero de 2021 ) |
La clasificación se basa principalmente en el tipo de genoma (bicatenario, monocatenario negativo o positivo) y en el número y organización de los genes. Actualmente, se reconocen 5 órdenes y 47 familias de virus ARN. También hay muchas especies y géneros sin asignar.
Los viroides y los virus satélite de ARN están relacionados con los virus de ARN, pero son distintos de ellos . Estos no están clasificados actualmente como virus de ARN y se describen en sus propias páginas.
Un estudio de varios miles de virus ARN ha demostrado la presencia de al menos cinco taxones principales: un grupo de levivirus y parientes; un supergrupo de picornavirus; un supergrupo de alfavirus más un supergrupo de flavivirus; los virus dsRNA; y los virus de cadena -ve. [23] El grupo de lentivirus parece ser basal para todos los virus ARN restantes. La siguiente división principal se encuentra entre el supragrupo picorna y los virus restantes. Los virus dsRNA parecen haber evolucionado a partir de un ancestro de ARN +ve y los virus de ARN -ve de dentro de los virus dsRNA. La relación más cercana a los virus de ARN de cadena -ve es Reoviridae .
Este es el grupo más grande de virus ARN [24] y ha sido organizado por el ICTV en los filos Kitrinoviricota , Lenarviricota y Pisuviricota en el reino Orthornavirae y el reino Riboviria . [25]
Los virus de ARN de cadena positiva también pueden clasificarse en función de la ARN polimerasa dependiente de ARN. Se han reconocido tres grupos: [26]
- Bymovirus, comovirus, nepovirus, nodavirus, picornavirus, potyvirus, sobemovirus y un subconjunto de luteovirus (virus del amarillamiento occidental de la remolacha y virus del enrollamiento de la hoja de la papa), el grupo similar a los picorna (Picornavirata).
- Carmovirus, dianthovirus, flavivirus, pestivirus, estatovirus, tombusvirus, bacteriófagos de ARN monocatenario, virus de la hepatitis C y un subconjunto de luteovirus (virus del enanismo amarillo de la cebada): el grupo similar a flavi (Flavivirata).
- Alfavirus, carlavirus, furovirus, hordeivirus, potexvirus, rubivirus, tobravirus, tricornavirus, timovirus, virus de la mancha clorótica de la hoja del manzano, virus del amarillamiento de la remolacha y virus de la hepatitis E (el grupo similar a alfa, Rubivirata).
Se ha propuesto una división del supergrupo de tipo alfa (similar a Sindbis) sobre la base de un nuevo dominio ubicado cerca de los extremos N de las proteínas involucradas en la replicación viral. [27] Los dos grupos propuestos son: el grupo de los "altovirus" (alfavirus, furovirus, virus de la hepatitis E, hordeivirus, tobamovirus, tobravirus, tricornavirus y probablemente rubivirus); y el grupo de los "tipovirus" (virus de la mancha clorotica de la hoja del manzano, carlavirus, potexvirus y timovirus).
El supergrupo tipo alfa se puede dividir en tres clados : los virus tipo rubí, tipo tobamo y tipo tymo. [28]
Trabajos adicionales han identificado cinco grupos de virus de ARN de cadena positiva que contienen cuatro, tres, tres, tres y un orden(es), respectivamente. [29] Estos catorce órdenes contienen 31 familias de virus (incluidas 17 familias de virus de plantas) y 48 géneros (incluidos 30 géneros de virus de plantas). Este análisis sugiere que los alfavirus y flavivirus pueden separarse en dos familias (Togaviridae y Flaviridae, respectivamente), pero sugiere que otras asignaciones taxonómicas, como los pestivirus, el virus de la hepatitis C, los rubivirus, el virus de la hepatitis E y los arterivirus, pueden ser incorrectas. Los coronavirus y los torovirus parecen ser familias distintas en órdenes distintos y no géneros distintos de la misma familia como se clasifica actualmente. Los luteovirus parecen ser dos familias en lugar de una, y el virus de la mancha foliar clorótica del manzano parece no ser un closterovirus sino un nuevo género de Potexviridae.
Evolución
La evolución de los picornavirus basada en un análisis de sus ARN polimerasas y helicasas parece remontarse a la divergencia de los eucariotas . [30] Sus supuestos ancestros incluyen los retroelementos del grupo II bacteriano , la familia de proteasas HtrA y los bacteriófagos de ADN .
Los partitivirus están relacionados con un ancestro totivirus y pueden haber evolucionado a partir de él. [31]
Los hipovirus y los barnavirus parecen compartir una ascendencia con los linajes potyvirus y sobemovirus respectivamente. [31]
Virus de ARN bicatenario
Este análisis también sugiere que los virus dsRNA no están estrechamente relacionados entre sí, sino que pertenecen a cuatro clases adicionales (Birnaviridae, Cystoviridae, Partitiviridae y Reoviridae) y a un orden adicional (Totiviridae) de una de las clases de virus ssRNA positivos en el mismo subfilo que los virus de ARN de cadena positiva.
Un estudio ha sugerido que hay dos grandes clados: uno incluye las familias Caliciviridae , Flaviviridae y Picornaviridae y un segundo que incluye las familias Alphatetraviridae , Birnaviridae , Cystoviridae , Nodaviridae y Permutotretraviridae . [32]
Virus de ARN de cadena negativa
Estos virus tienen múltiples tipos de genomas que van desde una única molécula de ARN hasta ocho segmentos. A pesar de su diversidad, parece que pueden haberse originado en artrópodos y haberse diversificado a partir de allí. [33]
Virus satelitales
También se conocen varios virus satélite (virus que requieren la asistencia de otro virus para completar su ciclo de vida). Su taxonomía aún está por determinar. Se han propuesto los siguientes cuatro géneros para los virus satélite de ARN monocatenario de sentido positivo que infectan plantas: Albetovirus , Aumaivirus , Papanivirus y Virtovirus . [34] Se ha propuesto una familia (Sarthroviridae, que incluye el género Macronovirus ) para los virus satélite de ARN monocatenario de sentido positivo que infectan a los artrópodos .
Grupo III – Virus dsRNA
Hay doce familias y varios géneros y especies no asignados reconocidos en este grupo. [10]
- Familia Amalgaviridae
- Familia Birnaviridae
- Familia Chrysoviridae
- Familia Cystoviridae
- Familia Endornaviridae
- Familia Hypoviridae
- Familia Megabirnaviridae
- Familia Partitiviridae
- Familia Picobirnaviridae
- Familia Reoviridae – incluye Rotavirus
- Familia Totiviridae
- Familia Quadriviridae
- Género Botybirnavirus
- Especies no asignadas
- Virus ARN de Botrytis porri 1
- Virus del circulifer tenellus 1
- Virus filamentoso Colletotrichum camelliae 1
- Virus asociado al amarillamiento de las cucurbitáceas
- Virus asociado a la debilitación por Sclerotinia sclerotiorum
- Virus 1 de Spissistilus festinus
Grupo IV – virus ssRNA de sentido positivo
En este grupo se reconocen tres órdenes y 34 familias. Además, hay varias especies y géneros sin clasificar.
- Orden Nidovirales
- Familia Arteriviridae
- Familia Coronaviridae : incluye coronavirus humanos (virus del resfriado común HCoV-229E , HCoV-HKU1 , HCoV-NL63 y HCoV-OC43 ), MERS-CoV , SARS-CoV-1 y SARS-CoV-2
- Familia Mesoniviridae
- Familia Roniviridae
- Orden Picornavirales
- Familia Dicistroviridae
- Familia Iflaviridae
- Familia Marnaviridae
- Familia Picornaviridae : incluye poliovirus , rinovirus (un virus del resfriado común) y virus de la hepatitis A.
- La familia Secoviridae incluye la subfamilia Comovirinae.
- Género Bacillariornavirus
- Especies Virus de la mosca de las algas
- Ordenar Tymovirales
- Familia Alphaflexiviridae
- Familia Betaflexiviridae
- Familia Gammaflexiviridae
- Familia Tymoviridae
- Sin asignar
- Familia Alphatetraviridae
- Familia Alvernaviridae
- Familia Astroviridae
- Familia Barnaviridae
- Familia Benyviridae
- Familia Botourmiaviridae
- Familia Bromoviridae
- Familia Caliciviridae : incluye el virus Norwalk
- Familia Carmotetraviridae
- Familia Closteroviridae
- Familia Flaviviridae : incluye el virus de la fiebre amarilla , el virus del Nilo Occidental , el virus de la hepatitis C , el virus del dengue y el virus del Zika.
- Familia Fusariviridae
- Familia Hepeviridae
- Familia Hypoviridae
- Familia Leviviridae
- Familia Luteoviridae : incluye el virus del enanismo amarillo de la cebada
- Familia Polycipiviridae
- Familia Narnaviridae
- Familia Nodaviridae
- Familia Permutotetraviridae
- Familia Potyviridae
- Familia Sarthroviridae
- Familia de estatovirus
- Familia Togaviridae : incluye el virus de la rubéola , el virus del río Ross , el virus Sindbis y el virus Chikungunya.
- Familia Tombusviridae
- Familia Virgaviridae [35]
- Géneros no asignados
- Género Blunervirus
- Género Cilevirus
- Género Higrevirus
- Género Idaeovirus
- Género Negevirus
- Género Ourmiavirus
- Género Polemovirus
- Género Sinaivirus
- Género Sobemovirus
- Especies no asignadas
- Virus del acyrthosiphon pisum
- Bastrovirus
- Virus de Blackford
- Virus de la mancha anular necrótica del arándano
- Cadicistrovirus
- Virus Chara australis
- Virus extra pequeño
- Virus de la clorosis de las bayas de Goji
- Virus 1 de Harmonia axyridis
- Hepelivirus
- Virus de la garrapata Jingmen
- Virus Le Blanc
- Nedicistrovirus
- Virus 1 de Nesidiocoris tenuis
- Niflavirus
- Virus 1 de Nylanderia fulva
- Virus de Orsay
- Virus ARN tipo 1 de Osedax japonicus
- Virus de picali
- Nidovirus de células secretoras de planarias
- Virus Plasmopara halstedii
- Fusarivirus 1 de Rosellinia necatrix
- Virus Santeuil
- Secalivirus
- Virus Solenopsis invicta 3
- Virus del gusano redondo del cerdo grande de Wuhan
Virus satelitales
- Familia Sarthroviridae
- Género Albetovirus
- Género Aumaivirus
- Género Papanivirus
- Género Virtovirus
- Virus de la parálisis crónica de las abejas
También se ha descrito un virus similar a un astrovirus/hepevirus no clasificado. [36]
Grupo V – virus ssRNA de sentido negativo
Con excepción del virus de la hepatitis D , este grupo de virus se ha incluido en un único filo: Negarnaviricota . Este filo se ha dividido en dos subfilos: Haploviricotina y Polyploviricotina . Dentro del subfilo Haploviricotina, actualmente se reconocen cuatro clases: Chunqiuviricetes , Milneviricetes , Monjiviricetes y Yunchangviricetes . En el subfilo Polyploviricotina, se reconocen dos clases: Ellioviricetes e Insthoviricetes .
Actualmente se reconocen en este grupo seis clases, siete órdenes y veinticuatro familias. Aún quedan por clasificar varias especies y géneros no asignados. [10]
- Filo Negarnaviricota [37]
- Subfilo Haploviricotina
- Clase Chunqiuviricetes
- Ordenar Muvirales
- Familia Qinviridae
- Ordenar Muvirales
- Clase Milneviricetes
- Orden Serpentovirales
- Familia Aspiviridae
- Orden Serpentovirales
- Clase Monjiviricetes
- Orden Jingchuvirales
- Familia Chuviridae
- Ordenar Mononegavirales
- Familia Bornaviridae – Virus de la enfermedad de Borna
- Familia Filoviridae : incluye el virus del Ébola y el virus de Marburgo.
- Familia Mymonaviridae
- Familia Nyamiviridae [38]
- Familia Paramyxoviridae : incluye el virus del sarampión , el virus de las paperas , el virus Nipah , el virus Hendra y el NDV.
- Familia Pneumoviridae : incluye el virus respiratorio sincitial y el metapneumovirus
- Familia Rhabdoviridae : incluye el virus de la rabia
- Familia Sunviridae
- Género Anphevirus
- Género Arlivirus
- Género Chengtivirus
- Género Crustavirus
- Género Wastrivirus
- Orden Jingchuvirales
- Clase Yunchangviricetes
- Orden Goujianvirales
- Familia Yueviridae
- Orden Goujianvirales
- Clase Chunqiuviricetes
- Subfilo Polyploviricotina
- Clase Ellioviricetes
- Ordenar Bunyavirales
- Familia Arenaviridae – incluye el virus Lassa
- Familia Cruliviridae
- Familia Feraviridae
- Familia Fimoviridae
- Familia Hantaviridae
- Familia Jonviridae
- Familia Nairoviridae
- Familia Peribunyaviridae
- Familia Phasmaviridae
- Familia Phenuiviridae
- Familia Tospoviridae
- Género Tilapineviridae
- Ordenar Bunyavirales
- Clase Insthoviricetes
- Ordenar Articulavirales
- Familia Amnoonviridae : incluye el virus Taastrup
- Familia Orthomyxoviridae : incluye los virus de la influenza
- Ordenar Articulavirales
- Clase Ellioviricetes
- Subfilo Haploviricotina
- Géneros no asignados:
- Género Deltavirus : incluye el virus de la hepatitis D (no es un virus verdadero, sino un agente subviral )
Galería
- Rabia ( Rhabdoviridae )
Véase también
- Clasificación de virus
- Lista de virus
- Replicación viral
- Sentido positivo/negativo
- Virus animales
- Virus de ARN bicatenario
- Retrovirus
- Virus de ADN
- Elemento de replicación que actúa en cis del norovirus
- Viroide
Notas
- ^ Esta inclusión se debió a la TaxoProp 2017.006G, que propuso Riboviria . La confusión podría deberse a la referencia de la TaxoProp a una "monofilia de todos los virus ARN", incorrectamente denominada ya que solo se demostró con RdRP. Por otro lado, la definición propuesta de Riboviria sí mencionó correctamente a RdRP.
- ^ La mayoría de los virus fúngicos son virus de ARN de doble cadena. Se ha descrito una pequeña cantidad de virus de ARN de cadena positiva. Un informe ha sugerido la posibilidad de un virus de cadena negativa. [8]
Referencias
- ^ Wagner, Edward K.; Hewlett, Martinez J. (1999). Virología básica. Malden, MA: Blackwell Science, Inc. p. 249. ISBN 0-632-04299-0. Recuperado el 30 de marzo de 2020 .
- ^ "Listado en orden taxonómico – Índice de listas de especies del ICTV" . Consultado el 11 de abril de 2008 .
- ^ Comité Ejecutivo del Comité Internacional de Taxonomía de Virus (mayo de 2020). "El nuevo alcance de la taxonomía de virus: dividiendo la virosfera en 15 rangos jerárquicos". Nature Microbiology . 5 (5): 668–674. doi : 10.1038/s41564-020-0709-x . PMC 7186216 . PMID 32341570.
- ^ Taxonomía 2019.006G
- ^ Taxonomía 2019.009G
- ^ Nguyen M, Haenni AL (junio de 2003). "Estrategias de expresión de virus ambisense". Virus Research . 93 (2): 141–50. doi :10.1016/S0168-1702(03)00094-7. PMID 12782362.
- ^ Kondo H, Chiba S, Toyoda K, Suzuki N (enero de 2013). "Evidencia de infección por virus de ARN de cadena negativa en hongos". Virology . 435 (2): 201–09. doi : 10.1016/j.virol.2012.10.002 . PMID 23099204.
- ^ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (octubre de 2010). "Tasas de mutación viral". Revista de Virología . 84 (19): 9733–48. doi :10.1128/JVI.00694-10. PMC 2937809 . PMID 20660197.
- ^ abc Klein DW, Prescott LM, Harley J (1993). Microbiología . Dubuque, Iowa: Wm. C. Brown. ISBN 978-0-697-01372-9.
- ^ Steinhauer DA, Holland JJ (1987). "Evolución rápida de los virus ARN". Revista anual de microbiología . 41 : 409–33. doi :10.1146/annurev.mi.41.100187.002205. PMID 3318675.
- ^ Boutwell CL, Rolland MM, Herbeck JT, Mullins JI, Allen TM (octubre de 2010). "Evolución viral y escape durante la infección aguda por VIH-1". The Journal of Infectious Diseases . 202 (Supl. 2): S309–14. doi :10.1086/655653. PMC 2945609 . PMID 20846038.
- ^ Bukh J, Purcell RH, Miller RH (agosto de 1994). "Análisis de secuencia del gen central de 14 genotipos del virus de la hepatitis C". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (17): 8239–43. Bibcode :1994PNAS...91.8239B. doi : 10.1073/pnas.91.17.8239 . PMC 44581 . PMID 8058787.
- ^ Tuplin A, Evans DJ, Simmonds P (octubre de 2004). "Mapeo detallado de las estructuras secundarias del ARN en las secuencias de la región central y codificante de NS5B del virus de la hepatitis C mediante escisión por ARNasa y nuevos métodos de predicción bioinformática". The Journal of General Virology . 85 (Pt 10): 3037–47. doi : 10.1099/vir.0.80141-0 . PMID 15448367.
- ^ ab Silva JM, Pratas D, Caetano T, Matos D (agosto de 2022). "El panorama complejo de los genomas virales". GigaCiencia . 11 : 1–16. doi : 10.1093/gigascience/giac079. PMC 9366995 . PMID 35950839.
- ^ ab Barr JN, Fearns R (junio de 2010). "Cómo los virus de ARN mantienen la integridad de su genoma". The Journal of General Virology . 91 (Pt 6): 1373–87. doi : 10.1099/vir.0.020818-0 . PMID 20335491.
- ^ Stedman, Kenneth M. (2015). "Recombinación profunda: genes de virus de ARN y ADNmc en virus de ADN y genomas hospedadores". Revisión anual de virología . 2 (1): 203–217. doi : 10.1146/annurev-virology-100114-055127 . ISSN 2327-0578. PMID 26958913. S2CID 207745438.
- ^ Muslin C, Mac Kain A, Bessaud M, Blondel B, Delpeyroux F (septiembre de 2019). "Recombinación en enterovirus, un proceso evolutivo modular de múltiples pasos". Viruses . 11 (9): 859. doi : 10.3390/v11090859 . PMC 6784155 . PMID 31540135.
- ^ Hu WS, Temin HM (noviembre de 1990). "Recombinación retroviral y transcripción inversa". Science . 250 (4985): 1227–33. Bibcode :1990Sci...250.1227H. doi :10.1126/science.1700865. PMID 1700865.
- ^ Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (noviembre de 2018). "La recombinación es necesaria para la replicación eficiente del VIH-1 y el mantenimiento de la integridad del genoma viral". Nucleic Acids Research . 46 (20): 10535–45. doi :10.1093/nar/gky910. PMC 6237782 . PMID 30307534.
- ^ ab Bernstein H, Bernstein C, Michod RE (enero de 2018). "Sexo en patógenos microbianos". Infección, genética y evolución . 57 : 8–25. doi : 10.1016/j.meegid.2017.10.024 . PMID 29111273.
- ^ ab Su S, Wong G, Shi W, Liu J, Lai AC, Zhou J, et al. (junio de 2016). "Epidemiología, recombinación genética y patogénesis de los coronavirus". Tendencias en microbiología . 24 (6): 490–502. doi : 10.1016/j.tim.2016.03.003 . PMC 7125511 . PMID 27012512.
- ^ Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Koonin EV (noviembre de 2018). "Orígenes y evolución del viroma de ARN global". mBio . 9 (6). doi :10.1128/mBio.02329-18. PMC 6282212 . PMID 30482837.
- ^ Francki RI, Fauquet CM, Knudson DL, Brown F (1991). Clasificación y nomenclatura de virus. Quinto informe del Comité Internacional de Taxonomía de Virus, Archives of Virology (Supl. 2) . Springer. ISBN 978-3-7091-9163-7.
- ^ "Actual publicación de la taxonomía ICTV | ICTV". ictv.global . Consultado el 3 de abril de 2023 .
- ^ Koonin EV (septiembre de 1991). "La filogenia de las ARN polimerasas dependientes de ARN de virus de ARN de cadena positiva". The Journal of General Virology . 72 (Pt 9): 2197–206. doi : 10.1099/0022-1317-72-9-2197 . PMID 1895057.
- ^ Rozanov MN, Koonin EV, Gorbalenya AE (agosto de 1992). "Conservación del supuesto dominio de la metiltransferasa: un sello distintivo del supergrupo 'similar a Sindbis' de virus de ARN de cadena positiva". The Journal of General Virology . 73 (Pt 8): 2129–34. CiteSeerX 10.1.1.532.7367 . doi :10.1099/0022-1317-73-8-2129. PMID 1645151.
- ^ Koonin EV, Dolja VV (1993). "Evolución y taxonomía de virus de ARN de cadena positiva: implicaciones del análisis comparativo de secuencias de aminoácidos". Critical Reviews in Biochemistry and Molecular Biology . 28 (5): 375–430. doi :10.3109/10409239309078440. PMID 8269709.
- ^ Ward CW (1993). "Progreso hacia una taxonomía superior de los virus". Investigación en Virología . 144 (6): 419–53. doi :10.1016/S0923-2516(06)80059-2. PMC 7135741 . PMID 8140287.
- ^ Koonin EV, Wolf YI, Nagasaki K, Dolja VV (diciembre de 2008). "El Big Bang de la evolución de virus similares a picorna antecede a la radiación de supergrupos eucariotas". Nature Reviews. Microbiology . 6 (12): 925–39. doi : 10.1038/nrmicro2030 . PMID 18997823.
- ^ ab Ghabrial SA (1998). "Origen, adaptación y vías evolutivas de los virus fúngicos". Virus Genes . 16 (1): 119–31. doi :10.1023/a:1007966229595. PMC 7089520 . PMID 9562896.
- ^ Gibrat JF, Mariadassou M, Boudinot P, Delmas B (julio de 2013). "Análisis de la radiación de los birnavirus de diversos filos hospedadores y de sus afinidades evolutivas con otros virus de ARN de doble cadena y de ARN de cadena positiva utilizando alineaciones de secuencias múltiples basadas en la estructura robusta y métodos filogenéticos avanzados". BMC Evolutionary Biology . 13 (1): 154. Bibcode :2013BMCEE..13..154G. doi : 10.1186/1471-2148-13-154 . PMC 3724706 . PMID 23865988.
- ^ Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, et al. (enero de 2015). "La diversidad genómica sin precedentes de los virus de ARN en artrópodos revela la ascendencia de los virus de ARN de sentido negativo". eLife . 4 . doi : 10.7554/eLife.05378 . PMC 4384744 . PMID 25633976.
- ^ Krupovic M, Kuhn JH, Fischer MG (enero de 2016). "Un sistema de clasificación para virófagos y virus satélite". Archivos de Virología . 161 (1): 233–47. doi : 10.1007/s00705-015-2622-9 . hdl : 11858/00-001M-0000-0028-DC34-F . PMID 26446887.
- ^ Adams MJ, Antoniw JF, Kreuze J (2009). "Virgaviridae: una nueva familia de virus vegetales con forma de bastón". Archivos de Virología . 154 (12): 1967–72. doi : 10.1007/s00705-009-0506-6 . PMID 19862474.
- ^ Pankovics P, Boros Á, Kiss T, Engelmann P, Reuter G (2019) Virus de ARN genéticamente altamente divergente con organización genómica similar a la de astrovirus (extremo 5') y hepevirus (extremo 3') en aves carnívoras, la carraca europea ( Coracias garrulus ). Infect Genet Evol
- ^ "Taxonomía de virus: publicación de 2018". Comité Internacional de Taxonomía de Virus . Consultado el 13 de noviembre de 2018 .
- ^ Mihindukulasuriya KA, Nguyen NL, Wu G, Huang HV, da Rosa AP, Popov VL, et al. (mayo de 2009). "Los virus Nyamanini y Midway definen un nuevo taxón de virus de ARN en el orden Mononegavirales". Revista de Virología . 83 (10): 5109–16. doi :10.1128/JVI.02667-08. PMC 2682064 . PMID 19279111.
Enlaces externos
- Virus de ARN en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Virus animales










_EM_PHIL_2175_lores.jpg/440px-Respiratory_Syncytial_Virus_(RSV)_EM_PHIL_2175_lores.jpg)


_EM_18_lores.jpg/440px-Vesicular_stomatitis_virus_(VSV)_EM_18_lores.jpg)
