Enzima desubiquitinante
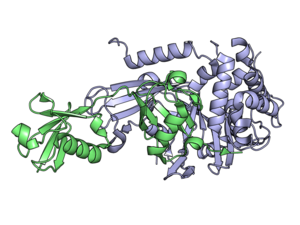

Las enzimas desubiquitinantes ( DUB ), también conocidas como peptidasas desubiquitinantes , isopeptidasas desubiquitinantes , deubiquitinasas , ubiquitina proteasas , ubiquitina hidrolasas o ubiquitina isopeptidasas , son un gran grupo de proteasas [1] que escinden la ubiquitina de las proteínas. [2] La ubiquitina se une a las proteínas para regular la degradación de proteínas a través del proteasoma y el lisosoma ; coordinar la localización celular de las proteínas; activar e inactivar proteínas; y modular las interacciones proteína-proteína . [3] [4] [5] Las DUB pueden revertir estos efectos al escindir el enlace peptídico o isopeptídico entre la ubiquitina y su proteína sustrato. En los humanos hay casi 100 genes DUB, que se pueden clasificar en dos clases principales: cisteína proteasas y metaloproteasas . Las proteasas de cisteína incluyen las proteasas específicas de ubiquitina (USP), las hidrolasas de ubiquitina C-terminal (UCH), las proteasas de dominio Machado-Josephin (MJD) y las proteasas de tumores ováricos (OTU). El grupo de metaloproteasas contiene solo las proteasas de dominio N-terminal+ (MPN+) (JAMM) de Jab1/Mov34/Mpr1 Pad1. [2]
Clases
En los humanos hay 102 genes DUB putativos, que pueden clasificarse en dos clases principales: cisteína proteasas y metaloproteasas , que consisten en 58 proteasas específicas de ubiquitina (USP), 4 hidrolasas C-terminales de ubiquitina (UCH), 5 proteasas del dominio Machado-Josephin (MJD), 14 proteasas de tumores ováricos (OTU) y 14 genes que contienen el dominio Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM). Se predice que 11 de estas proteínas no son funcionales, dejando 79 enzimas funcionales. [6] En la levadura, las USP se conocen como proteasas de procesamiento específicas de ubiquitina (UBP).
Proteasas de cisteína
Hay seis superfamilias principales de cisteína proteasas DUB: [7]
- la superfamilia de proteasas específicas de ubiquitina (USP/UBP); ( USP1 , USP2 , USP3, USP4 , USP5 , USP6 , USP7 , USP8 , USP9X , USP9Y , USP10 , USP11 , USP12, USP13 , USP14 , USP15 , USP16 , USP17, USP17L2, USP17L3, USP17L4, USP17L5, USP17L7, USP17L8, USP18 , USP19 , USP20 , USP21 , USP22, USP23 , USP24 , USP25 , USP26 , USP27X , USP28, USP29, USP30, USP31, USP32, USP33 , USP34 , USP35, USP36 , USP37 , USP38, USP39 , USP40 , USP41, USP42 , USP43, USP44 , USP45, USP46)
- la superfamilia de tumores ováricos (OTU) ( OTUB1 , OTUB2 );
- y la superfamilia del dominio Machado-Josephin (MJD). ( ATXN3 , ATXN3L)
- la superfamilia de la hidrolasa C-terminal de ubiquitina (UCH); ( BAP1 , UCHL1 , UCHL3 , UCHL5 )
- la familia MINDY de desubiquitinasas específicas de K48; ( MINDY1 , MINDY2 , MINDY3 , MINDY4 ) [8]
- La familia ZUFSP, recientemente descubierta, actualmente representada únicamente por ZUP1 [9] [10]
| UCI | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 USP2 en complejo con ubiquitina. | |||||||||
| Identificadores | |||||||||
| Símbolo | UCI | ||||||||
| Pfam | PF00443 | ||||||||
| Clan Pfam | CL0125 | ||||||||
| Interprofesional | IPR001394 | ||||||||
| PROSITIO | PDOC00750 | ||||||||
| MEROPS | C19 | ||||||||
| SCOP2 | 1nb8 / ALCANCE / SUPFAM | ||||||||
| |||||||||
También existe un grupo putativo poco conocido de DUB llamado superfamilia de peptidasas de pliegue de papaína permutadas de virus dsADN y eucariotas (PPPDE), que, si se demuestra que son DUB genuinos, serían los séptimos en la clase de cisteína proteasas. [11]
Metaloproteasas
Las proteínas de la superfamilia del dominio Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM) se unen al zinc y, por lo tanto, son metaloproteasas. [7]
Papel de las enzimas desubiquitinantes
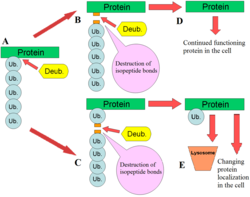
Las DUB desempeñan varias funciones en la vía de la ubiquitina. Una de las funciones mejor caracterizadas de las DUB es la eliminación de cadenas de monoubiquitina y poliubiquitina de las proteínas. Estas modificaciones son una modificación postraduccional (adición a una proteína después de que se ha creado) donde las proteínas ubiquitina individuales o cadenas de ubiquitina se agregan a las lisinas de una proteína sustrato. Estas modificaciones de ubiquitina se agregan a las proteínas mediante la maquinaria de ubiquitinación; enzimas activadoras de ubiquitina (E1), enzimas conjugadoras de ubiquitina (E2) y ligasas de ubiquitina (E3). El resultado es ubiquitina unida a residuos de lisina a través de un enlace isopeptídico . [12] Las proteínas se ven afectadas por estas modificaciones de varias maneras: regulan la degradación de proteínas a través del proteasoma y el lisosoma ; coordinan la localización celular de las proteínas; activan e inactivan proteínas; y modulan las interacciones proteína-proteína . [3] [4] [5] Los DUB desempeñan un papel antagónico en este eje al eliminar estas modificaciones, revirtiendo así el destino de las proteínas. [2] Además, un papel menos comprendido de los DUB es la escisión de proteínas similares a la ubiquitina, como SUMO y NEDD8 . Algunos DUB pueden tener la capacidad de escindir enlaces isopeptídicos entre estas proteínas y proteínas sustrato. [13]
Activan la ubiquitina mediante la proteólisis (descomposición) de las formas inactivas expresadas de ubiquitina. La ubiquitina está codificada en mamíferos por 4 genes diferentes: UBA52 , RPS27A , UBB y UBC . Un conjunto similar de genes se encuentra en otros eucariotas como la levadura. Los genes UBA52 y RPS27A producen ubiquitina que se fusiona a proteínas ribosómicas y los genes UBB y UBC producen poliubiquitina (una cadena de ubiquitina unida por sus extremos C y N ). [14] [15] Los DUB escinden la ubiquitina de estas proteínas, produciendo unidades individuales activas de ubiquitina. [2]
Las DUB también escinden proteínas ubiquitinas individuales que pueden haber tenido sus colas C-terminales unidas accidentalmente a pequeños nucleófilos celulares . [2] Estas ubiquitinamidas y ubiquitintioésteres pueden formarse durante reacciones de ubiquitinación estándar por la cascada E1-E2-E3. El glutatión y las poliaminas son dos nucleófilos que podrían atacar el enlace tioléster entre la ubiquitina y estas enzimas. La hidrolasa C-terminal de la ubiquitina es un ejemplo de DUB que hidroliza estos enlaces con amplia especificidad. [13] [16]
Las cadenas de poliubiquitina libre son escindidas por los DUB para producir monoubiquitina. Las cadenas pueden ser producidas por la maquinaria E1-E2-E3 en la célula libre de cualquier proteína sustrato. Otra fuente de poliubiquitina libre es el producto de la escisión de la ubiquitina con el sustrato. Si los DUB escinden la base de la cadena de poliubiquitina que está unida a una proteína, toda la cadena quedará libre y deberá ser reciclada por los DUB. [2]
Dominios

Los DUB suelen contener un dominio catalítico rodeado de uno o más dominios accesorios, algunos de los cuales contribuyen al reconocimiento del objetivo. Estos dominios adicionales incluyen el dominio presente en las proteasas específicas de ubiquitina (DUSP); el dominio similar a la ubiquitina (UBL); el dominio de homología de meprina y TRAF (MATH); el dominio de la proteasa específica de ubiquitina de dedo de zinc (ZnF-UBP); el dominio mieloide, nervioso y DEAF1 de dedo de zinc (ZnF-MYND); el dominio asociado a la ubiquitina (UBA); el dominio CHORD-SGT1 (CS); el dominio de interacción y tráfico de microtúbulos (MIT); el dominio similar a la rodenasa; el dominio TBC/RABGAP; y el dominio B-box. [6] [17]
Dominio catalítico
El dominio catalítico de las DUB es lo que las clasifica en grupos particulares; USP, OTU, MJD, UCH y MPN+/JAMM. Los primeros 4 grupos son cisteína proteasas , mientras que el último es una metaloproteasa de zinc . Las DUB de cisteína proteasa son similares a la papaína y, por lo tanto, tienen un mecanismo de acción similar. Utilizan díadas o tríadas catalíticas (ya sea de dos o tres aminoácidos ) para catalizar la hidrólisis de los enlaces amida entre la ubiquitina y el sustrato. Los residuos del sitio activo que contribuyen a la actividad catalítica de las DUB de cisteína proteasa son cisteína (díada/tríada), histidina (díada/tríada) y aspartato o asparagina (solo tríada). La histidina es polarizada por el aspartato o la asparagina en tríadas catalíticas o por otras formas en díadas. Este residuo polarizado reduce el pKa de la cisteína, lo que le permite realizar un ataque nucleofílico sobre el enlace isopeptídico entre el extremo C de la ubiquitina y el sustrato lisina . Las metaloproteasas coordinan los iones de zinc con residuos de histidina, aspartato y serina , que activan las moléculas de agua y les permiten atacar el enlace isopeptídico. [18] [19]
Universidad de Blenheim
Los dominios similares a la ubiquitina (UBL) tienen una estructura (pliegue) similar a la ubiquitina, excepto que carecen de los residuos terminales de glicina. Se propone que 18 USP tienen dominios UBL. Solo otros 2 DUB tienen UBL fuera del grupo USP: OTU1 y VCPIP1 . USP4, USP7, USP11, USP15, USP32, USP40 y USP47 tienen múltiples dominios UBL. A veces, los dominios UBL están en tándem, como en USP7, donde están presentes 5 dominios UBL C-terminales en tándem . USP4, USP6, USP11, USP15, USP19, USP31, USP32 y USP43 tienen dominios UBL insertados en el dominio catalítico. Las funciones de los dominios UBL son diferentes entre USP, pero comúnmente regulan la actividad catalítica de USP. Pueden coordinar la localización en el proteasoma (USP14); regulan negativamente las USP al competir por el sitio catalítico de la USP (USP4) e inducen cambios conformacionales para aumentar la actividad catalítica (USP7). [17] [20] [21] Al igual que otros dominios UBL, la estructura de los dominios UBL de la USP muestra un pliegue de agarre β. [22] [23]
POLVO
| Dominio DUSP | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura de la solución del dominio DUSP de HUSP15. | |||||||||
| Identificadores | |||||||||
| Símbolo | POLVO | ||||||||
| Pfam | PF06337 | ||||||||
| Interprofesional | IPR006615 | ||||||||
| MEROPS | C19 | ||||||||
| |||||||||
Se encuentran dominios DUSP en tándem simples o múltiples de aproximadamente 120 residuos en seis USP. La función del dominio DUSP es actualmente desconocida, pero puede desempeñar un papel en la interacción proteína-proteína , en particular en el reconocimiento del sustrato de los DUB. Esto se predice debido a la hendidura hidrofóbica presente en el dominio DUSP de USP15 y a que algunas interacciones de proteínas con USP que contienen DUSP no ocurren sin estos dominios. El dominio DUSP muestra un nuevo pliegue tipo trípode que comprende tres hélices y una lámina beta antiparalela hecha de tres hebras. Este pliegue se asemeja a las patas (hélices) y al asiento (lámina beta) del trípode. Dentro de la mayoría de los dominios DUSP en USP hay una secuencia conservada de aminoácidos conocida como motivo PGPI . Esta es una secuencia de cuatro aminoácidos; prolina , glicina , prolina e isoleucina , que se empaqueta contra el haz de tres hélices y está altamente ordenada. [6] [24]
Papel en la enfermedad
Aún queda por dilucidar el alcance total del papel de las DUB en las enfermedades. Su participación en las enfermedades se predice debido a funciones conocidas en los procesos fisiológicos que intervienen en los estados patológicos, incluidos el cáncer y los trastornos neurológicos. [25]
La enzima USP28 se encuentra sobreexpresada en diferentes tipos de cáncer como el de colon o pulmón. Además, USP28 desubiquitina y estabiliza oncogenes importantes como c-Myc , Notch1 , c-jun o ΔNp63 . [26] [27] [28] En tumores escamosos, USP28 regula la resistencia a la quimioterapia regulando la reparación del ADN a través del eje de la vía ΔNp63 -anemia de Fanconia. [29]
Las enzimas desubiquitinantes UCH-L3 y YUH1 son capaces de hidrolizar la ubiquitina mutante UBB+1 a pesar del hecho de que la glicina en la posición 76 está mutada. [30]
Los niveles de UCH-L1 son altos en varios tipos de neoplasias malignas ( cáncer ). [31]
Papel en el ciclo celular
Las DUB desempeñan un papel activo en la modulación del ciclo celular. La proteasa de procesamiento específico de ubiquitina (USP) es una familia de enzimas desubiquitinantes que desempeñan un papel crucial en la regulación del ciclo celular. [32] Dos de estas enzimas son la USP17 y la USP44. La USP17 regula las vías responsables del progreso de las células a través del ciclo celular. [33] Sus objetivos incluyen reguladores de Ras, CDK2 y ciclina A. [34] La USP44 desempeña un papel importante en la iniciación de la anafase. [35] Una nueva investigación sobre el punto de control mitótico ha revelado un nuevo papel de la USP44 en la regulación de la progresión del ciclo celular. [35]
Regulación USP de Ras
La vía ERK permite la transducción de señales mitogénicas externas en señales intracelulares que promueven la proliferación celular. Uno de los reguladores clave de esta vía es Ras, una GTPasa que, al activarse, se une a GTP para "activar" la cascada de señalización posterior. La enzima convertidora de Ras 1 (RCE1) escinde postraduccionalmente los 3 residuos en el extremo C de Ras, lo que permite que Ras se localice correctamente en la membrana plasmática. [36]
La USP17 actúa para desubiquitinar los dominios de ubiquitina K63 en RCE1. [34] Dicha estabilización de RCE1 permite la localización adecuada de Ras, promoviendo así la proliferación tras la activación de los receptores tempranos en la vía ERK. La hiperactividad de Ras puede provocar una desregulación del ciclo celular. [37] Por lo tanto, la regulación de Ras a través de la USP17 actúa como otro punto en la regulación de Ras.
Regulación de la USP de la transición G1-S
Las quinasas dependientes de ciclina (CDK) son una familia de enzimas que fosforilan residuos de serina y treonina para impulsar la célula a través del ciclo celular. La activación de CDK2 es fundamental para la transición G1-S. Para que CDK2 se active, la ciclina A debe unirse al complejo de quinasa dependiente de ciclina (CDKC). El ciclo de división celular 25A (CDC25A) es una fosfatasa que elimina un grupo fosfato inhibidor de CDK2. [38] Mientras que la ubiquitinación marcaría CDC25A para degradación, bloqueando así la progresión a la fase S, USP17 desubiquitina CDC25A. [34] Un aumento en la estabilidad de CDC25A promueve la actividad de CDKC, impulsando así la célula a través de la transición G1-S.
La USP17 también regula la progresión del ciclo celular al actuar sobre SETD8 para regular negativamente la transcripción del inhibidor de la quinasa dependiente de ciclina 1 (CDKN1A), también conocido como p21. [34] La CDKN1A se une a la CDK2 y la inhibe utilizando su dominio de unión N-terminal, bloqueando así la progresión a través de la transición G1-S. La SETD8, una metiltransferasa, utiliza S-adenosil metionina para metilar el residuo Lys20 de la histona 4, lo que da como resultado la condensación de los cromosomas. [39] Esta compactación del ADN regula negativamente la transcripción de CDKN1A. La USP17 desubiquitina a SETD8, reduciendo así su propensión a la degradación y aumentando su estabilidad intracelular. [34] La regulación negativa resultante en la transcripción de CDKN1A promueve la actividad de CDK2, lo que permite que la célula progrese a través de la transición G1-S. Véase el esquema del papel de las DUB en la regulación del ciclo celular. [34]

USP44 en la iniciación de la anafase
El punto de control del huso (también conocido como punto de control mitótico) garantiza la separación adecuada de los cromosomas. En términos generales, el punto de control mitótico promueve la fidelidad en la segregación cromosómica, lo que aumenta la probabilidad de que cada célula hija reciba solo un cromosoma duplicado. [40] Este mecanismo es crucial, ya que los errores en la separación cromosómica se han relacionado con el cáncer, los defectos de nacimiento y la resistencia a los antibióticos en los patógenos. [41] Una de las proteínas reguladoras centrales es el complejo promotor de anafase (APC/C). APC/C ubiquitina la securina. [42] La destrucción resultante de la separasa de liberación de seguridad, [40] que hidroliza la cohesión, la proteína que une las cromátidas hermanas.
Una nueva investigación de Stegmeier y colegas [35] publicada en la revista Nature demuestra un papel crucial para USP44 en la regulación del punto de control del huso. Usando un cribado de shRNA, se identificó que USP44 estabiliza la inhibición de APC/C [35] La unión de CDC20 a APC/C es necesaria para la ubiquitinación de la securina. [43] Una proteína llamada hMAD2 puede formar un trímero inactivo con APC y CDC20, formando el complejo hMAD2-CDC-APC. [43] Tras la ubiquitinación de CDC20 por UbcH10, hMAD2 se disocia y APC/C se activa. [44] Es importante señalar que la ubiquitinación de CDC20 no sirve para marcarlo para la degradación, sino que promueve la disociación de hMAD2 del complejo hMAD2-CDC-APC. La USP44, una proteasa que procesa específicamente la ubiquitina, puede estabilizar el complejo inactivo hMAD2-CDC-APC al contrarrestar la ubiquitinación de UbcH10. Esto bloquea la disociación de hMAD2 y permite la regulación adecuada de APC/C, manteniéndolo inactivo hasta que se adhiera correctamente al huso mitótico. Tras la unión adecuada, el comportamiento similar a un interruptor permite la activación de APC/C. [35] Esto da como resultado la escisión de la cohesión, lo que permite la separación de las cromátidas hermanas.
Papel en la reparación del daño del ADN mediada por p53
Los daños en el ADN pueden resultar catastróficos para un organismo. Los mecanismos de mutación del ADN incluyen estrés oxidativo, errores de replicación del ADN, carcinógenos exógenos, radiación y mutación espontánea de bases. Cuando se produce un daño en el ADN, se detiene la progresión del ciclo celular para evitar la propagación de la mutación. El gen TP53 (también conocido como p53) es crucial para garantizar la conservación del genoma. [45] Las enzimas desubiquitinantes desempeñan un papel fundamental en el mantenimiento de la función del p53.
En las células sanas, p53 activa la ligasa de ubiquitina E3 MDM2, que a su vez ubiquitina p53. Esto crea un ciclo de retroalimentación negativa , por el cual la degradación de p53 permite que las células fluyan a través del ciclo celular. Cuando se daña el ADN, la proteasa de procesamiento específico de ubiquitina 7 (USP7) estabiliza p53 al escindir la ubiquitina. [46] Para que USP7 desubiquitine p53, debe localizarse en el núcleo. Sin embargo, no se ha encontrado ninguna secuencia de localización nuclear (NLS). [47] A pesar de que no se conoce ninguna NLS, un estudio mostró que, tras la eliminación del extremo N de USP7, no se produjo localización nuclear. [47] Es posible que otras proteínas faciliten la entrada nuclear de USP7.
Una vez estabilizado, el p53 puede ejercer su función de supresión tumoral. Las vías descendentes del p53 actúan para detener la progresión del ciclo celular en las fases G1 o G2 del ciclo celular [48] o promover la muerte celular, dependiendo de la gravedad del daño del ADN. [49] Ver esquema del papel de USP7 en la vía dependiente del p53. [48] o promover la muerte celular, dependiendo de la gravedad del daño del ADN. [49] Ver esquema del papel de USP7 en la vía dependiente del p53. [49]

Referencias
- ^ Wilkinson KD (diciembre de 1997). "Regulación de los procesos dependientes de la ubiquitina mediante enzimas desubiquitinantes". FASEB Journal . 11 (14): 1245–56. doi : 10.1096/fasebj.11.14.9409543 . PMID 9409543. S2CID 11788220.
- ^ abcdef Reyes-Turcu FE, Ventii KH, Wilkinson KD (2009). "Regulación y funciones celulares de las enzimas desubiquitinantes específicas de la ubiquitina". Revisión anual de bioquímica . 78 : 363–97. doi :10.1146/annurev.biochem.78.082307.091526. PMC 2734102 . PMID 19489724.
- ^ ab Glickman MH, Ciechanover A (abril de 2002). "La vía proteolítica ubiquitina-proteasoma: destrucción en aras de la construcción". Physiological Reviews . 82 (2): 373–428. doi :10.1152/physrev.00027.2001. PMID 11917093.
- ^ ab Mukhopadhyay D, Riezman H (enero de 2007). "Funciones independientes del proteasoma de la ubiquitina en la endocitosis y la señalización". Science . 315 (5809): 201–5. Bibcode :2007Sci...315..201M. doi :10.1126/science.1127085. PMID 17218518. S2CID 35434448.
- ^ ab Schnell JD, Hicke L (septiembre de 2003). "Funciones no tradicionales de la ubiquitina y de las proteínas de unión a la ubiquitina". The Journal of Biological Chemistry . 278 (38): 35857–60. doi : 10.1074/jbc.R300018200 . PMID 12860974.
- ^ abc Nijman SM, Luna-Vargas MP, Velds A, Brummelkamp TR, Dirac AM, Sixma TK, Bernards R (diciembre de 2005). "Un inventario genómico y funcional de enzimas desubiquitinantes". Celúla . 123 (5): 773–86. doi :10.1016/j.cell.2005.11.007. hdl : 1874/20959 . PMID 16325574. S2CID 15575576.
- ^ ab Amerik AY, Hochstrasser M (noviembre de 2004). "Mecanismo y función de las enzimas desubiquitinantes". Biochimica et Biophysica Acta (BBA) - Investigación de células moleculares . 1695 (1–3): 189–207. doi : 10.1016/j.bbamcr.2004.10.003 . PMID 15571815.
- ^ Abdul Rehman SA, Kristariyanto YA, Choi SY, Nkosi PJ, Weidlich S, Labib K, et al. (julio de 2016). "MINDY-1 es miembro de una nueva familia de enzimas desubiquitinantes conservadas evolutivamente y estructuralmente distintas". Molecular Cell . 63 (1): 146–55. doi :10.1016/j.molcel.2016.05.009. PMC 4942677 . PMID 27292798.
- ^ Kwasna D, Abdul Rehman SA, Natarajan J, Matthews S, Madden R, De Cesare V, et al. (Abril de 2018). "Descubrimiento y caracterización de ZUFSP/ZUP1, una clase distinta de deubiquitinasa importante para la estabilidad del genoma". Célula molecular . 70 (1): 150–164.e6. doi :10.1016/j.molcel.2018.02.023. PMC 5896202 . PMID 29576527.
- ^ Hermanns, Thomas; Pichlo, Christian; Woiwode, Ilka; Klopffleisch, Karsten; Witting, Katharina F.; Ovaa, Huib; Baumann, Ulrich; Hofmann, Kay (23 de febrero de 2018). "Una familia de desubiquitinasas no convencionales con determinantes de especificidad de cadena modular". Nature Communications . 9 (1): 799. Bibcode :2018NatCo...9..799H. doi :10.1038/s41467-018-03148-5. ISSN 2041-1723. PMC 5824887 . PMID 29476094.
- ^ Iyer LM, Koonin EV, Aravind L (noviembre de 2004). "Nuevas peptidasas predichas con un papel potencial en la vía de señalización de la ubiquitina". Cell Cycle . 3 (11): 1440–50. doi :10.4161/cc.3.11.1206. PMID 15483401.
- ^ Kerscher O, Felberbaum R, Hochstrasser M (2006). "Modificación de proteínas por ubiquitina y proteínas similares a la ubiquitina". Revisión anual de biología celular y del desarrollo . 22 : 159–80. doi :10.1146/annurev.cellbio.22.010605.093503. PMID 16753028. S2CID 17584645.
- ^ ab Wing SS (mayo de 2003). "Enzimas desubiquitinantes: la importancia de conducir en sentido inverso a lo largo de la vía ubiquitina-proteasoma". The International Journal of Biochemistry & Cell Biology . 35 (5): 590–605. doi :10.1016/s1357-2725(02)00392-8. PMID 12672452.
- ^ Kimura Y, Tanaka K (junio de 2010). "Mecanismos reguladores implicados en el control de la homeostasis de la ubiquitina". Journal of Biochemistry . 147 (6): 793–8. doi : 10.1093/jb/mvq044 . PMID 20418328.
- ^ Ozkaynak E, Finley D, Solomon MJ, Varshavsky A (mayo de 1987). "Los genes de la ubiquitina de la levadura: una familia de fusiones génicas naturales". The EMBO Journal . 6 (5): 1429–39. doi :10.1002/j.1460-2075.1987.tb02384.x. PMC 553949 . PMID 3038523.
- ^ Pickart CM, Rose IA (julio de 1985). "La hidrolasa carboxilo-terminal de la ubiquitina actúa sobre las amidas carboxilo-terminales de la ubiquitina". The Journal of Biological Chemistry . 260 (13): 7903–10. doi : 10.1016/S0021-9258(17)39538-8 . PMID 2989266.
- ^ ab Komander D, Clague MJ, Urbé S (agosto de 2009). "Rompiendo las cadenas: estructura y función de las desubiquitinasas". Nature Reviews. Molecular Cell Biology . 10 (8): 550–63. doi :10.1038/nrm2731. PMID 19626045. S2CID 19149247.
- ^ Komander D (2010). "Mecanismo, especificidad y estructura de las desubiquitinasas". Conjugación y desconjugación de modificadores de la familia de la ubiquitina . Bioquímica subcelular. Vol. 54. págs. 69–87. doi :10.1007/978-1-4419-6676-6_6. ISBN 978-1-4419-6675-9. Número de identificación personal 21222274.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Chapman HA, Riese RJ, Shi GP (1997). "Funciones emergentes de las proteasas de cisteína en la biología humana". Revisión anual de fisiología . 59 : 63–88. doi :10.1146/annurev.physiol.59.1.63. PMID 9074757.
- ^ Faesen AC, Luna-Vargas MP, Sixma TK (junio de 2012). "El papel de los dominios UBL en las proteasas específicas de la ubiquitina". Biochemical Society Transactions . 40 (3): 539–45. doi :10.1042/BST20120004. PMID 22616864.
- ^ Ye Y, Scheel H, Hofmann K, Komander D (diciembre de 2009). "La disección de los dominios catalíticos de la USP revela cinco puntos de inserción comunes". Molecular BioSystems . 5 (12): 1797–808. doi :10.1039/b907669g. PMID 19734957.
- ^ Elliott PR, Liu H, Pastok MW, Grossmann GJ, Rigden DJ, Clague MJ, et al. (noviembre de 2011). "Variabilidad estructural de los dominios dobles DUSP-UBL de la proteasa específica de ubiquitina". FEBS Letters . 585 (21): 3385–90. doi :10.1016/j.febslet.2011.09.040. PMID 22001210. S2CID 5312371.
- ^ Harper S, Besong TM, Emsley J, Scott DJ, Dreveny I (septiembre de 2011). "Estructura de los dominios N-terminales de USP15: una horquilla β media la asociación estrecha entre los dominios DUSP y UBL". Biochemistry . 50 (37): 7995–8004. doi :10.1021/bi200726e. PMID 21848306.
- ^ de Jong RN, Ab E, Diercks T, Truffault V, Daniëls M, Kaptein R, Folkers GE (febrero de 2006). "Estructura de la solución del dominio DUSP de la proteasa 15 específica de la ubiquitina humana". The Journal of Biological Chemistry . 281 (8): 5026–31. doi : 10.1074/jbc.M510993200 . PMID 16298993.
- ^ Singhal S, Taylor MC, Baker RT (octubre de 2008). "Enzimas desubiquitilantes y enfermedades". BMC Biochemistry . 9 (Supl 1): S3. doi : 10.1186/1471-2091-9-S1-S3 . PMC 2582804 . PMID 19007433.
- ^ Diefenbacher ME, Popov N, Blake SM, Schülein-Völk C, Nye E, Spencer-Dene B, et al. (agosto de 2014). "La deubiquitinasa USP28 controla la homeostasis intestinal y promueve el cáncer colorrectal". The Journal of Clinical Investigation . 124 (8): 3407–18. doi :10.1172/JCI73733. PMC 4109555 . PMID 24960159.
- ^ Prieto-García C, Hartmann O, Reissland M, Braun F, Fischer T, Walz S, et al. (Junio de 2019). "El eje USP28-∆Np63 es una vulnerabilidad de los tumores escamosos". bioRxiv 10.1101/683508 .
- ^ Prieto-García C, Hartmann O, Reissland M, Braun F, Fischer T, Walz S, et al. (abril de 2020). "Las células de cáncer escamoso requieren mantener la estabilidad proteica de ∆Np63 a través de USP28". EMBO Molecular Medicine . 12 (4): e11101. doi :10.15252/emmm.201911101. PMC 7136964 . PMID 32128997.
- ^ Prieto-García C, Hartmann O, Reissland M, Fischer T, Maier CR, Rosenfeldt M, et al. (septiembre de 2020). "La inhibición de USP28 supera la resistencia al cisplatino de los tumores escamosos mediante la supresión de la vía de la anemia de Fanconi". bioRxiv 10.1101/2020.09.10.291278 .
- ^ Dennissen FJ, Kholod N, Hermes DJ, Kemmerling N, Steinbusch HW, Dantuma NP, van Leeuwen FW (agosto de 2011). "La ubiquitina mutante (UBB+1) asociada con trastornos neurodegenerativos es hidrolizada por la hidrolasa L3 del extremo C de la ubiquitina (UCH-L3)". FEBS Letters . 585 (16): 2568–74. doi : 10.1016/j.febslet.2011.06.037 . PMID 21762696. S2CID 28207136.
- ^ Fang Y, Fu D, Shen XZ (agosto de 2010). "El papel potencial de las hidrolasas de la ubiquitina c-terminal en la oncogénesis". Biochimica et Biophysica Acta (BBA) - Reseñas sobre el cáncer . 1806 (1): 1–6. doi :10.1016/j.bbcan.2010.03.001. PMID 20302916.
- ^ Valles GJ, Bezsonova I, Woodgate R, Ashton NW (agosto de 2020). "USP7 es un regulador maestro de la estabilidad del genoma". Frontiers in Cell and Developmental Biology . 8 (717): 717. doi : 10.3389/fcell.2020.00717 . PMC 7419626 . PMID 32850836.
- ^ Fukuura K, Inoue Y, Miyajima C, Watanabe S, Tokugawa M, Morishita D, et al. (noviembre de 2019). "La proteasa específica de ubiquitina USP17 previene la senescencia celular al estabilizar la metiltransferasa SET8 y reprimir transcripcionalmente p21". The Journal of Biological Chemistry . 294 (44): 16429–16439. doi : 10.1074/jbc.RA119.009006 . PMC 6827320 . PMID 31533987.
- ^ abcdef Ducker C, Shaw PE (enero de 2021). "Desubiquitinación mediada por USP17 y cáncer: los clientes se agrupan alrededor del ciclo celular" (PDF) . The International Journal of Biochemistry & Cell Biology . 130 (130): 105886. doi : 10.1016/j.biocel.2020.105886 . ISSN 1357-2725. PMID 33227393. S2CID 227158810.
- ^ abcde Stegmeier F, Rape M, Draviam VM, Nalepa G, Sowa ME, Ang XL, et al. (Abril de 2007). "El inicio de la anafase está regulado por actividades antagonistas de ubiquitinación y desubiquitinación". Naturaleza . 446 (7138): 876–81. Código Bib :2007Natur.446..876S. doi : 10.1038/naturaleza05694. PMID 17443180. S2CID 4321083.
- ^ Karlsson C, Akula MK, Staffas A, Cisowski J, Sayin VI, Ibrahim MX, et al. (febrero de 2021). "La inactivación de la endoproteasa RAS RCE1 acelera la leucemia mieloide al regular negativamente GADD45b". Leucemia . 35 (2): 606–609. doi :10.1038/s41375-020-0859-0. PMID 32398789. S2CID 218605961.
- ^ Braun BS, Shannon K (abril de 2008). "Ataque a Ras en leucemias mieloides". Clinical Cancer Research . 14 (8): 2249–52. doi : 10.1158/1078-0432.CCR-07-1005 . PMID 18413813.
- ^ Shen T, Huang S (julio de 2012). "El papel de Cdc25A en la regulación de la proliferación celular y la apoptosis". Agentes anticáncer en química medicinal . 12 (6): 631–9. doi :10.2174/187152012800617678. PMC 3544488. PMID 22263797 .
- ^ David R (diciembre de 2010). "Ciclo celular: eliminación de SETD8". Nature Reviews. Molecular Cell Biology . 11 (12): 819. doi : 10.1038/nrm3020 . PMID 21102605. S2CID 30122715.
- ^ ab Liu ST, Zhang H (octubre de 2016). "El complejo de puntos de control mitótico (MCC): una mirada atrás y atrás después de 15 años". AIMS Molecular Science . 3 (4): 597–634. doi :10.3934/molsci.2016.4.597. PMC 5597056 . PMID 28920074.
- ^ Potapova T, Gorbsky GJ (febrero de 2017). "Las consecuencias de los errores de segregación cromosómica en la mitosis y la meiosis". Biology . 6 (1): 12. doi : 10.3390/biology6010012 . PMC 5372005 . PMID 28208750.
- ^ Wirth KG, Ricci R, Giménez-Abián JF, Taghybeeglu S, Kudo NR, Jochum W, et al. (Enero de 2004). "La pérdida del complejo promotor de la anafase en las células inactivas provoca una proliferación no programada de hepatocitos". Genes y desarrollo . 18 (1): 88–98. doi :10.1101/gad.285404. PMC 314282 . PMID 14724179.
- ^ ab Fang G, Yu H, Kirschner MW (junio de 1998). "La proteína de punto de control MAD2 y el regulador mitótico CDC20 forman un complejo ternario con el complejo promotor de la anafase para controlar la iniciación de la anafase". Genes & Development . 12 (12): 1871–83. doi :10.1101/gad.12.12.1871. PMC 316912 . PMID 9637688.
- ^ Peters JM (abril de 2007). "Biología celular: el freno del punto de control aliviado". Nature . 446 (7138): 868–9. Bibcode :2007Natur.446..868P. doi : 10.1038/446868a . PMID 17443175.
- ^ "Gen TP53". MedlinePlus . Biblioteca Nacional de Medicina de EE. UU . . Consultado el 18 de mayo de 2021 .
- ^ Li M, Chen D, Shiloh A, Luo J, Nikolaev AY, Qin J, Gu W (abril de 2002). "La desubiquitinación de p53 por HAUSP es una vía importante para la estabilización de p53". Nature . 416 (6881): 648–53. Bibcode :2002Natur.416..648L. doi :10.1038/nature737. PMID 11923872. S2CID 4389394.
- ^ ab Fernández-Montalván A, Bouwmeester T, Joberty G, Mader R, Mahnke M, Pierrat B, et al. (agosto de 2007). "La caracterización bioquímica de USP7 revela sitios de modificación postraduccional y requisitos estructurales para el procesamiento del sustrato y la localización subcelular". The FEBS Journal . 274 (16): 4256–70. doi : 10.1111/j.1742-4658.2007.05952.x . PMID 17651432.
- ^ ab Donehower LA (mayo de 2014). "Las fosfatasas revierten los puntos de control del ciclo celular mediados por p53". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 111 (20): 7172–3. Bibcode :2014PNAS..111.7172D. doi : 10.1073/pnas.1405663111 . PMC 4034213 . PMID 24808140.
- ^ abc Chen L, Liu S, Tao Y (junio de 2020). "Regulación de genes supresores de tumores: modificaciones postraduccionales". Transducción de señales y terapia dirigida . 5 (1): 90. doi :10.1038/s41392-020-0196-9. PMC 7293209. PMID 32532965 .