Enzima conjugadora de ubiquitina
| Ubiquitina: proteína ligasa | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| N.º CE | 6.3.2.19 | ||||||||
| N.º CAS | 74812-49-0 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| Ontología genética | AmiGO / QuickGO | ||||||||
| |||||||||
| Enzima conjugadora de ubiquitina, E2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | UBQ-conjugado_E2 | ||||||||
| Pfam | PF00179 | ||||||||
| Interprofesional | IPR000608 | ||||||||
| ELEGANTE | SM00212 | ||||||||
| PROSITIO | PDOC00163 | ||||||||
| Membranoma | 241 | ||||||||
| |||||||||
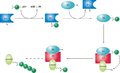
Las enzimas conjugadoras de ubiquitina , también conocidas como enzimas E2 y más raramente como enzimas transportadoras de ubiquitina , realizan el segundo paso en la reacción de ubiquitinación que selecciona una proteína para su degradación a través del proteasoma . El proceso de ubiquitinación une covalentemente la ubiquitina , una proteína corta de 76 aminoácidos , a un residuo de lisina en la proteína objetivo. Una vez que una proteína ha sido marcada con una molécula de ubiquitina, rondas adicionales de ubiquitinación forman una cadena de poliubiquitina que es reconocida por la partícula reguladora 19S del proteasoma, lo que desencadena el desdoblamiento dependiente de ATP de la proteína objetivo que permite el paso a la partícula central 20S del proteasoma, donde las proteasas degradan el objetivo en fragmentos de péptidos cortos para su reciclaje por la célula . [1]
Relaciones
Una enzima activadora de ubiquitina , o E1, primero activa la ubiquitina uniendo covalentemente la molécula a su residuo de cisteína del sitio activo . Luego, la ubiquitina activada se transfiere a una cisteína E2. Una vez conjugada con la ubiquitina, la molécula E2 se une a una de varias ligasas de ubiquitina o E3 a través de una región de unión estructuralmente conservada . La molécula E3 es responsable de unirse al sustrato de la proteína objetivo y transferir la ubiquitina desde la cisteína E2 a un residuo de lisina en la proteína objetivo. [1]
- Diagrama esquemático del sistema de ubiquitinación.
Una célula particular normalmente contiene sólo unos pocos tipos de moléculas E1, una mayor diversidad de E2 y una gran variedad de E3. En los seres humanos, hay alrededor de 30 E2 que pueden unirse a una de las más de 600 E3. [2] Las moléculas E3 responsables de la identificación y unión del sustrato son, por lo tanto, los mecanismos de especificidad del sustrato en la degradación proteasomal. Cada tipo de E2 puede asociarse con muchos E3. [3]
Las E2 también se pueden utilizar para estudiar los mecanismos de plegamiento de proteínas. Dado que el sistema de ubiquitinación es compartido por todos los organismos, los estudios pueden utilizar proteínas E2 modificadas para comprender el sistema general de cómo todos los organismos procesan las proteínas. [4] También hay algunas proteínas que pueden actuar como E2 y como dominios que contienen E3 que cubren tanto la funcionalidad de E2 como la de E3. [5]
Isoenzimas
Los siguientes genes humanos codifican enzimas conjugadoras de ubiquitina:
- UBE2A
- UBE2B
- UBE2C
- UBE2D1 , UBE2D2 , UBE2D3 , UBE2D4 (este último putativo)
- UBE2E1 , UBE2E2 , UBE2E3
- UBE2F (putativo)
- UBE2G1 , UBE2G2
- UBE2H
- UBE2I
- UBE2J1 , UBE2J2
- UBE2K
- UBE2L3 , UBE2L6 ; (UBE2L1, UBE2L2, UBE2L4 son pseudogenes)
- UBE2M
- UBE2N
- UBE2O
- UBE2T1, UBE2T2
- UBE2R1 (CDC34), UBE2R2
- UBE2S
- UBE2T (putativo)
- UBE2U (putativo)
- UBE2V1 , UBE2V2
- UBE2W (putativo)
- UBE2Z
- ATG3
- BIRC6
- UFC 1
Véase también
Referencias
- ^ ab Nandi D, Tahiliani P, Kumar A, Chandu D (marzo de 2006). "El sistema ubiquitina-proteasoma". Revista de biociencias . 31 (1): 137–155. doi :10.1007/BF02705243. PMID 16595883. S2CID 21603835.
- ^ Burge RJ, Damianou A, Wilkinson AJ, Rodenko B, Mottram JC (octubre de 2020). "La diferenciación de Leishmania requiere la conjugación de ubiquitina mediada por un complejo UBC2-UEV1 E2". PLOS Pathogens . 16 (10): e1008784. doi : 10.1371/journal.ppat.1008784 . PMC 7647121 . PMID 33108402.
- ^ Risseeuw EP, Daskalchuk TE, Banks TW, Liu E, Cotelesage J, Hellmann H, et al. (junio de 2003). "Análisis de interacción de proteínas de subunidades de ligasa de ubiquitina E3 SCF de Arabidopsis". El diario de las plantas . 34 (6): 753–767. doi :10.1046/j.1365-313X.2003.01768.x. PMID 12795696.
- ^ Henneberg LT, Schulman BA (julio de 2021). "Decodificación de la mensajería del sistema de ubiquitina mediante sondas químicas y proteicas". Biología química celular . 28 (7): 889–902. doi :10.1016/j.chembiol.2021.03.009. PMC 7611516 . PMID 33831368.
- ^ Chang SC, Zhang BX, Ding JL (marzo de 2022). "Emparejamiento de enzimas ubiquitinas E2-E3: asociación para provocar o mitigar cánceres". Biochimica et Biophysica Acta (BBA) - Reseñas sobre el cáncer . 1877 (2): 188679. doi : 10.1016/j.bbcan.2022.188679 . PMID 35074437. S2CID 246224363.
Enlaces externos
- Recurso de motivo lineal eucariota de clase MOD_SUMO
- Enzimas conjugadoras de ubiquitina en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.