Proteína similar a la ubiquitina
| Familia de ubiquitinas | |||||||||
|---|---|---|---|---|---|---|---|---|---|
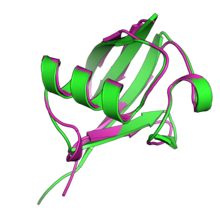 Superposición de las estructuras de la ubiquitina ( PDB : 1UBQ , verde) y NEDD8 ( PDB : 1NDD , magenta) | |||||||||
| Identificadores | |||||||||
| Símbolo | Ubiquitina | ||||||||
| Pfam | PF00240 | ||||||||
| Interprofesional | IPR029071 | ||||||||
| ELEGANTE | SM00213 | ||||||||
| |||||||||
Las proteínas similares a la ubiquitina (UBL) son una familia de proteínas pequeñas involucradas en la modificación postraduccional de otras proteínas en una célula , generalmente con una función reguladora . La familia de proteínas UBL deriva su nombre del primer miembro de la clase que se descubrió, la ubiquitina (Ub), mejor conocida por su papel en la regulación de la degradación de proteínas a través de la modificación covalente de otras proteínas. Después del descubrimiento de la ubiquitina, se describieron muchos miembros adicionales del grupo relacionados evolutivamente, que involucran procesos reguladores paralelos y una química similar. Las UBL están involucradas en una amplia gama de funciones celulares que incluyen autofagia , tráfico de proteínas , inflamación y respuestas inmunes , transcripción , reparación de ADN , empalme de ARN y diferenciación celular . [1] [2] [3]
Descubrimiento
La ubiquitina en sí fue descubierta por primera vez en la década de 1970 y originalmente llamada "polipéptido inmunopoyético ubicuo". [4] Posteriormente, otras proteínas con similitud de secuencia con la ubiquitina fueron reportadas ocasionalmente en la literatura, pero la primera que mostró compartir la característica clave de la modificación proteica covalente fue ISG15, descubierta en 1987. [5] Una sucesión de informes a mediados de la década de 1990 se reconoce como un punto de inflexión en el campo, [6] con el descubrimiento de SUMO (small u biquitin - like mo difier , también conocido como Sentrin o SENP1 ) reportado aproximadamente al mismo tiempo por una variedad de investigadores en 1996, [7] NEDD8 en 1997, [8] y Apg12 en 1998. [9] Desde entonces, una encuesta sistemática ha identificado más de 10,000 genes distintos para ubiquitina o proteínas similares a ubiquitina representadas en genomas eucariotas . [10]
Estructura y clasificación
Los miembros de la familia UBL son proteínas pequeñas, no enzimáticas que comparten una estructura común ejemplificada por la ubiquitina, que tiene 76 residuos de aminoácidos dispuestos en un pliegue proteico de "agarre beta" que consiste en una lámina beta antiparalela de cinco hebras que rodea una hélice alfa . [1] [11] [12] El pliegue de agarre beta está ampliamente distribuido en otras proteínas de origen tanto eucariota como procariota. [13] En conjunto, la ubiquitina y las proteínas similares a la ubiquitina a veces se denominan "ubiquitones". [3]
Los UBL se pueden dividir en dos categorías dependiendo de su capacidad para conjugarse covalentemente con otras moléculas. Los UBL que son capaces de conjugarse (a veces conocidos como Tipo I) tienen un motivo de secuencia característico que consiste en uno o dos residuos de glicina en el extremo C , a través del cual ocurre la conjugación covalente. Normalmente, los UBL se expresan como precursores inactivos y deben activarse mediante proteólisis del extremo C para exponer la glicina activa. [1] [12] Casi todos estos UBL están finalmente unidos a otra proteína, pero hay al menos una excepción; ATG8 está unido a fosfatidiletanolamina . [1] Los UBL que no exhiben conjugación covalente (Tipo II) a menudo ocurren como dominios proteicos fusionados genéticamente a otros dominios en una única cadena polipeptídica más grande, y pueden procesarse proteolíticamente para liberar el dominio UBL [1] o pueden funcionar como dominios de interacción proteína-proteína . [11] Los dominios UBL de proteínas más grandes a veces se conocen como dominios UBX . [14]
Distribución
La ubiquitina es, como su nombre lo sugiere, ubicua en eucariotas ; tradicionalmente se considera que está ausente en bacterias y arqueas , [11] aunque se han descrito algunos ejemplos en arqueas . [15] Las UBL también están ampliamente distribuidas en eucariotas, pero su distribución varía entre linajes; por ejemplo, ISG15 , involucrada en la regulación del sistema inmunológico , no está presente en eucariotas inferiores. [1] Otras familias exhiben diversificación en algunos linajes; un solo miembro de la familia SUMO se encuentra en el genoma de la levadura , pero hay al menos cuatro en los genomas de vertebrados , que muestran cierta redundancia funcional, [1] [2] y hay al menos ocho en el genoma de la planta modelo Arabidopsis thaliana . [16]
En los humanos
El genoma humano codifica al menos ocho familias de UBL, sin incluir la ubiquitina en sí, que se consideran UBL de tipo I y se sabe que modifican covalentemente otras proteínas: SUMO , NEDD8 , ATG8 , ATG12 , URM1 , UFM1 , FAT10 e ISG15 . [1] Una proteína adicional, conocida como FUBI, está codificada como una proteína de fusión en el gen FAU y se procesa proteolíticamente para generar un extremo C de glicina libre, pero no se ha demostrado experimentalmente que forme modificaciones proteicas covalentes. [1]
En las plantas
Se sabe que los genomas de las plantas codifican al menos siete familias de UBL además de la ubiquitina: SUMO , RUB (el homólogo vegetal de NEDD8 ), ATG8 , ATG12 , MUB, UFM1 y HUB1, así como una serie de UBL de tipo II. [17] Algunas familias de UBL y sus proteínas reguladoras asociadas en las plantas han experimentado una expansión espectacular, probablemente debido tanto a la duplicación del genoma completo como a otras formas de duplicación genética ; se ha estimado que las familias de ubiquitina, SUMO, ATG8 y MUB representan casi el 90% de los genes UBL de las plantas. [18] Las proteínas asociadas con la señalización de ubiquitina y SUMO están altamente enriquecidas en los genomas de los embriofitos . [15]
En procariotas

En comparación con los eucariotas, las proteínas procariotas con relaciones con las UBL están restringidas filogenéticamente. [19] [20] La proteína similar a la ubiquitina procariota (Pup) se presenta en algunas actinobacterias y tiene funciones muy análogas a la ubiquitina en el etiquetado de proteínas para la degradación proteasomal ; sin embargo, está intrínsecamente desordenada y su relación evolutiva con las UBL no está clara. [19] Recientemente se ha descrito una proteína relacionada, UBact, en algunos linajes gramnegativos . [21] Por el contrario, la proteína TtuB en las bacterias del género Thermus comparte el pliegue de agarre beta con las UBL eucariotas; se informa que tiene funciones duales como proteína transportadora de azufre y como modificación de proteína conjugada covalentemente. [19] En las arqueas , las pequeñas proteínas modificadoras arqueales (SAMP) comparten el pliegue de agarre beta y se ha demostrado que desempeñan un papel similar a la ubiquitina en la degradación de proteínas. [19] [20] Recientemente, en 2011 se identificó un conjunto aparentemente completo de genes correspondientes a una vía de ubiquitina similar a la eucariota en una arquea no cultivada , [22] [23] [24] y se cree que al menos tres linajes de arqueas—" Euryarchaeota ", Thermoproteota (anteriormente Crenarchaeota) y " Aigarchaeota "— poseen tales sistemas. [15] [25] [26] Además, algunas bacterias patógenas han desarrollado proteínas que imitan las de las vías UBL eucariotas e interactúan con las UBL en la célula huésped , interfiriendo con su función de señalización. [27] [28]
Regulación

La regulación de las UBL que son capaces de conjugación covalente en eucariotas es elaborada pero típicamente paralela para cada miembro de la familia, mejor caracterizada para la ubiquitina misma. El proceso de ubiquitinación es una secuencia de tres pasos estrechamente regulada: activación, realizada por enzimas activadoras de ubiquitina (E1); conjugación, realizada por enzimas conjugadoras de ubiquitina (E2); y ligadura, realizada por ligasas de ubiquitina (E3). El resultado de este proceso es la formación de un enlace covalente entre el extremo C de la ubiquitina y un residuo (típicamente una lisina ) en la proteína objetivo. Muchas familias de UBL tienen un proceso similar de tres pasos catalizado por un conjunto distinto de enzimas específicas de esa familia. [1] [29] [30] La desubiquitinación o desconjugación, es decir, la eliminación de la ubiquitina de un sustrato proteico, es realizada por enzimas desubiquitinantes (DUB); Las UBL también pueden degradarse mediante la acción de las proteasas específicas de la ubiquitina (ULP). [31] La gama de UBL sobre las que pueden actuar estas enzimas es variable y puede ser difícil de predecir. Algunas UBL, como SUMO y NEDD8, tienen DUB y ULP específicos de la familia. [32]
La ubiquitina es capaz de formar cadenas poliméricas, con moléculas de ubiquitina adicionales unidas covalentemente a la primera, que a su vez está unida a su sustrato proteico. Estas cadenas pueden ser lineales o ramificadas, y las diferencias en la longitud y ramificación de la cadena de ubiquitina pueden enviar diferentes señales reguladoras. [31] Aunque no se sabe que todas las familias de UBL formen cadenas, se han detectado experimentalmente cadenas SUMO, NEDD8 y URM1. [1] Además, la ubiquitina puede ser modificada por las UBL, como se sabe que ocurre con SUMO y NEDD8. [31] [33] Las intersecciones mejor caracterizadas entre distintas familias de UBL involucran a la ubiquitina y SUMO. [34] [35]
Funciones celulares
Las UBL como clase están involucradas en una gran variedad de procesos celulares. Además, las familias individuales de UBL varían en el alcance de sus actividades y la diversidad de las proteínas a las que están conjugadas. [1] La función más conocida de la ubiquitina es identificar las proteínas que serán degradadas por el proteasoma , pero la ubiquitinación puede desempeñar un papel en otros procesos como la endocitosis y otras formas de tráfico de proteínas , la transcripción y regulación de factores de transcripción , la señalización celular , la modificación de histonas y la reparación del ADN . [11] [12] [36] La mayoría de las otras UBL tienen funciones similares en la regulación de procesos celulares, generalmente con un rango conocido más restringido que el de la propia ubiquitina. Las proteínas SUMO tienen la variedad más amplia de objetivos proteicos celulares después de la ubiquitina [1] y están involucradas en procesos que incluyen la transcripción , la reparación del ADN y la respuesta al estrés celular . [33] NEDD8 es mejor conocido por su papel en la regulación de las proteínas cullina , que a su vez regulan la degradación de proteínas mediada por ubiquitina, [2] aunque probablemente también tenga otras funciones. [37] Dos UBL, ATG8 y ATG12 , están involucrados en el proceso de autofagia ; [38] ambos son inusuales en que ATG12 tiene solo dos sustratos proteicos conocidos y ATG8 está conjugado no a una proteína sino a un fosfolípido , la fosfatidiletanolamina . [1]
Evolución

La evolución de las UBL y sus conjuntos asociados de proteínas reguladoras ha sido de interés desde poco después de que fueron reconocidas como una familia. [39] Los estudios filogenéticos de la superfamilia de plegamientos de proteínas beta-grasp sugieren que las UBL eucariotas son monofiléticas , lo que indica un origen evolutivo compartido. [13] Se cree que los sistemas reguladores de las UBL, incluidos los propios UBL y la cascada de enzimas que interactúan con ellos, comparten un origen evolutivo común con las vías de biosíntesis procariota para los cofactores tiamina y molibdopterina ; las proteínas bacterianas de transferencia de azufre ThiS y MoaD de estas vías comparten el pliegue beta-grasp con las UBL, mientras que la similitud de secuencia y un mecanismo catalítico común vinculan a los miembros de la vía ThiF y MoeB con las enzimas activadoras de la ubiquitina . [13] [17] [11] Curiosamente, la proteína eucariota URM1 funciona como una UBL y una proteína transportadora de azufre, y ha sido descrita como un fósil molecular que establece este vínculo evolutivo. [11] [40]
Los estudios genómicos comparativos de las familias UBL y las proteínas relacionadas sugieren que la señalización UBL ya estaba bien desarrollada en el último ancestro común eucariota y, en última instancia, se origina en las arqueas ancestrales , [15] una teoría respaldada por la observación de que algunos genomas arqueológicos poseen los genes necesarios para una vía de ubiquitinación en pleno funcionamiento. [25] [18] Se han identificado dos eventos de diversificación diferentes dentro de la familia UBL en linajes eucariotas, que corresponden al origen de la multicelularidad tanto en linajes animales como vegetales. [15]
Referencias
- ^ abcdefghijklmn Cappadocia L, Lima CD (febrero de 2018). "Conjugación de proteínas similares a la ubiquitina: estructuras, química y mecanismo". Chemical Reviews . 118 (3): 889–918. doi :10.1021/acs.chemrev.6b00737. PMC 5815371 . PMID 28234446.
- ^ abc van der Veen AG, Ploegh HL (7 de julio de 2012). "Proteínas similares a la ubiquitina". Revisión anual de bioquímica . 81 (1): 323–57. doi :10.1146/annurev-biochem-093010-153308. PMID 22404627.
- ^ ab Welchman RL, Gordon C, Mayer RJ (agosto de 2005). "Ubiquitina y proteínas similares a la ubiquitina como señales multifuncionales". Nature Reviews. Molecular Cell Biology . 6 (8): 599–609. doi :10.1038/nrm1700. PMID 16064136. S2CID 7373421.
- ^ Goldstein G, Scheid M, Hammerling U, Schlesinger DH, Niall HD, Boyse EA (enero de 1975). "Aislamiento de un polipéptido que tiene propiedades de diferenciación de linfocitos y que probablemente está representado universalmente en células vivas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 72 (1): 11–5. Bibcode :1975PNAS...72...11G. doi : 10.1073/pnas.72.1.11 . PMC 432229 . PMID 1078892.
- ^ Haas AL, Ahrens P, Bright PM, Ankel H (agosto de 1987). "El interferón induce una proteína de 15 kilodalton que exhibe una marcada homología con la ubiquitina". The Journal of Biological Chemistry . 262 (23): 11315–23. doi : 10.1016/S0021-9258(18)60961-5 . PMID 2440890.
- ^ Yeh ET, Gong L, Kamitani T (mayo de 2000). "Proteínas similares a la ubiquitina: nuevos vinos en nuevas botellas". Gene . 248 (1–2): 1–14. doi :10.1016/S0378-1119(00)00139-6. PMID 10806345.
- ^ Saitoh, Hisato; Pu, Robert T.; Dasso, Mary (octubre de 1997). "SUMO-1: luchando con un nuevo modificador relacionado con la ubiquitina". Tendencias en ciencias bioquímicas . 22 (10): 374–376. doi :10.1016/S0968-0004(97)01102-X. PMID 9357311.
- ^ Kamitani T, Kito K, Nguyen HP, Yeh ET (noviembre de 1997). "Caracterización de NEDD8, una proteína similar a la ubiquitina regulada a la baja durante el desarrollo". The Journal of Biological Chemistry . 272 (45): 28557–62. doi : 10.1074/jbc.272.45.28557 . PMID 9353319.
- ^ Mizushima N, Noda T, Yoshimori T, Tanaka Y, Ishii T, George MD, Klionsky DJ, Ohsumi M, Ohsumi Y (septiembre de 1998). "Un sistema de conjugación de proteínas esencial para la autofagia". Nature . 395 (6700): 395–8. Bibcode :1998Natur.395..395M. doi :10.1038/26506. PMID 9759731. S2CID 204997310.
- ^ Zhou J, Xu Y, Lin S, Guo Y, Deng W, Zhang Y, Guo A, Xue Y (enero de 2018). "iUUCD 2.0: una actualización con anotaciones enriquecidas para ubiquitina y conjugaciones similares a la ubiquitina". Investigación de ácidos nucleicos . 46 (D1): D447–D453. doi :10.1093/nar/gkx1041. PMC 5753239 . PMID 29106644.
- ^ abcdef Hochstrasser M (marzo de 2009). "Origen y función de las proteínas similares a la ubiquitina". Nature . 458 (7237): 422–9. Bibcode :2009Natur.458..422H. doi :10.1038/nature07958. PMC 2819001 . PMID 19325621.
- ^ abc Kerscher O, Felberbaum R, Hochstrasser M (noviembre de 2006). "Modificación de proteínas por ubiquitina y proteínas similares a la ubiquitina". Revisión anual de biología celular y del desarrollo . 22 (1): 159–80. doi :10.1146/annurev.cellbio.22.010605.093503. PMID 16753028.
- ^ abc Burroughs AM, Balaji S, Iyer LM, Aravind L (julio de 2007). "Pequeño pero versátil: la extraordinaria diversidad funcional y estructural del pliegue de agarre beta". Biology Direct . 2 (1): 18. doi : 10.1186/1745-6150-2-18 . PMC 1949818 . PMID 17605815.
- ^ Buchberger A, Howard MJ, Proctor M, Bycroft M (marzo de 2001). "El dominio UBX: un módulo similar a la ubiquitina muy extendido". Journal of Molecular Biology . 307 (1): 17–24. doi :10.1006/jmbi.2000.4462. PMID 11243799.
- ^ abcde Grau-Bové X, Sebé-Pedrós A, Ruiz-Trillo I (marzo de 2015). "El ancestro eucariota tenía un complejo sistema de señalización de ubiquitina de origen arqueal". Biología Molecular y Evolución . 32 (3): 726–39. doi :10.1093/molbev/msu334. PMC 4327156 . PMID 25525215.
- ^ Miura K, Hasegawa PM (abril de 2010). "Sumoilación y otras modificaciones postraduccionales similares a la ubiquitina en plantas". Tendencias en biología celular . 20 (4): 223–32. doi :10.1016/j.tcb.2010.01.007. PMID 20189809.
- ^ ab Vierstra RD (septiembre de 2012). "El universo en expansión de la ubiquitina y los modificadores similares a la ubiquitina". Fisiología vegetal . 160 (1): 2–14. doi :10.1104/pp.112.200667. PMC 3440198 . PMID 22693286.
- ^ ab Hua Z, Doroodian P, Vu W (julio de 2018). "Los patrones de duplicación contrastantes reflejan diversidades funcionales de la ubiquitina y modificadores de proteínas similares a la ubiquitina en plantas". The Plant Journal . 95 (2): 296–311. doi : 10.1111/tpj.13951 . PMID 29738099.
- ^ abcd Maupin-Furlow JA (2014). "Modificación de proteínas procariotas similares a la ubiquitina". Revisión anual de microbiología . 68 : 155–75. doi :10.1146/annurev-micro-091313-103447. PMC 4757901 . PMID 24995873.
- ^ ab Ganguli, S; Ratna Prabha, C (2017). "Cachorros, SAMP y proteosomas procarióticos". En Chakraborti, S; Dhalla, N (eds.). Proteasas en fisiología y patología . Saltador. ISBN 978-981-10-2512-9.
- ^ Lehmann G, Udasin RG, Livneh I, Ciechanover A (febrero de 2017). "Identificación de UBact, una proteína similar a la ubiquitina, junto con otros componentes homólogos de un sistema de conjugación y el proteasoma en diferentes bacterias gramnegativas". Comunicaciones de investigación bioquímica y biofísica . 483 (3): 946–950. doi :10.1016/j.bbrc.2017.01.037. PMID 28087277.
- ^ Nunoura T, Takaki Y, Kakuta J, Nishi S, Sugahara J, Kazama H, Chee GJ, Hattori M, Kanai A, Atomi H, Takai K, Takami H (abril de 2011). "Conocimientos sobre la evolución de Archaea y los sistemas modificadores de proteínas eucariotas revelados por el genoma de un nuevo grupo de arqueas". Investigación de ácidos nucleicos . 39 (8): 3204–23. doi : 10.1093/nar/gkq1228. PMC 3082918 . PMID 21169198.
- ^ Hennell James R, Caceres EF, Escasinas A, Alhasan H, Howard JA, Deery MJ, Ettema TJ, Robinson NP (octubre de 2017). "Reconstrucción funcional de una cascada de ubiquitilación E1 / E2 / (RING) E3 de tipo eucariota de un arqueón no cultivado". Comunicaciones de la naturaleza . 8 (1): 1120. Código Bib : 2017NatCo...8.1120H. doi :10.1038/s41467-017-01162-7. PMC 5654768 . PMID 29066714.
- ^ Fuchs AC, Maldoner L, Wojtynek M, Hartmann MD, Martin J (julio de 2018). "Procesamiento de ubiquitina mediado por Rpn11 en un sistema de ubiquitinación arqueal ancestral". Nature Communications . 9 (1): 2696. Bibcode :2018NatCo...9.2696F. doi :10.1038/s41467-018-05198-1. PMC 6043591 . PMID 30002364.
- ^ ab Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, Seitz KW, Anantharaman K, Starnawski P, Kjeldsen KU, Stott MB, Nunoura T, Banfield JF, Schramm A, Baker BJ, Spang A, Ettema TJ (enero de 2017). "Asgard archaea ilumina el origen de la complejidad celular eucariota". Naturaleza . 541 (7637): 353–358. Código Bib :2017Natur.541..353Z. doi : 10.1038/naturaleza21031. OSTI 1580084. PMID 28077874. S2CID 4458094.
- ^ Hua ZS, Qu YN, Zhu Q, Zhou EM, Qi YL, Yin YR, Rao YZ, Tian Y, Li YX, Liu L, Castelle CJ, Hedlund BP, Shu WS, Knight R, Li WJ (julio de 2018). "Inferencia genómica del metabolismo y la evolución del filo arqueal Aigarchaeota". Nature Communications . 9 (1): 2832. Bibcode :2018NatCo...9.2832H. doi :10.1038/s41467-018-05284-4. PMC 6053391 . PMID 30026532.
- ^ Zhou Y, Zhu Y (enero de 2015). "Diversidad de la manipulación bacteriana de las vías de ubiquitina del huésped". Microbiología celular . 17 (1): 26–34. doi :10.1111/cmi.12384. PMID 25339545. S2CID 33328949.
- ^ Ribet D, Cossart P (noviembre de 2018). "Ubiquitina, SUMO y NEDD8: objetivos clave de los patógenos bacterianos" (PDF) . Tendencias en biología celular . 28 (11): 926–940. doi :10.1016/j.tcb.2018.07.005. PMC 7028394 . PMID 30107971.
- ^ Streich FC, Lima CD (6 de mayo de 2014). "Información estructural y funcional sobre la conjugación de proteínas similares a la ubiquitina". Revisión anual de biofísica . 43 (1): 357–79. doi :10.1146/annurev-biophys-051013-022958. PMC 4118471. PMID 24773014 .
- ^ Schulman BA, Harper JW (mayo de 2009). "Activación de proteínas similares a la ubiquitina por enzimas E1: el punto culminante de las vías de señalización descendentes". Nature Reviews. Molecular Cell Biology . 10 (5): 319–31. doi :10.1038/nrm2673. PMC 2712597 . PMID 19352404.
- ^ abc Mevissen TE, Komander D (junio de 2017). "Mecanismos de regulación y especificidad de la deubiquitinasa". Revisión anual de bioquímica . 86 (1): 159–192. doi : 10.1146/annurev-biochem-061516-044916 . PMID 28498721.
- ^ Ronau JA, Beckmann JF, Hochstrasser M (abril de 2016). "Especificidad de sustrato de las proteasas ubiquitina y Ubl". Cell Research . 26 (4): 441–56. doi :10.1038/cr.2016.38. PMC 4822132 . PMID 27012468.
- ^ ab Swatek KN, Komander D (abril de 2016). "Modificaciones de la ubiquitina". Cell Research . 26 (4): 399–422. doi :10.1038/cr.2016.39. PMC 4822133 . PMID 27012465.
- ^ Denuc A, Marfany G (febrero de 2010). "Los caminos de SUMO y ubiquitina convergen". Transacciones de la sociedad bioquímica . 38 (Parte 1): 34–9. doi :10.1042/BST0380034. PMID 20074031.
- ^ Wilkinson KA, Henley JM (mayo de 2010). "Mecanismos, regulación y consecuencias de la sumoilación de proteínas". The Biochemical Journal . 428 (2): 133–45. doi :10.1042/BJ20100158. PMC 3310159 . PMID 20462400.
- ^ Mukhopadhyay D, Riezman H (enero de 2007). "Funciones independientes del proteasoma de la ubiquitina en la endocitosis y la señalización". Science . 315 (5809): 201–5. Bibcode :2007Sci...315..201M. doi :10.1126/science.1127085. PMID 17218518. S2CID 35434448.
- ^ Enchev RI, Schulman BA, Peter M (enero de 2015). "Neddilación de proteínas: más allá de las ligasas cullin-RING". Nature Reviews. Molecular Cell Biology . 16 (1): 30–44. doi :10.1038/nrm3919. PMC 5131867 . PMID 25531226.
- ^ Shpilka T, Mizushima N, Elazar Z (mayo de 2012). "Proteínas similares a la ubiquitina y autofagia de un vistazo". Journal of Cell Science . 125 (Pt 10): 2343–8. doi : 10.1242/jcs.093757 . PMID 22736434.
- ^ Hochstrasser M (agosto de 2000). "Evolución y función de los sistemas de conjugación de proteínas similares a la ubiquitina". Nature Cell Biology . 2 (8): E153-7. doi :10.1038/35019643. PMID 10934491. S2CID 29557235.
- ^ Wang F, Liu M, Qiu R, Ji C (agosto de 2011). "El papel dual de la proteína similar a la ubiquitina Urm1 como modificador de proteínas y transportador de azufre". Protein & Cell . 2 (8): 612–9. doi :10.1007/s13238-011-1074-6. PMC 4875326 . PMID 21904977.