Bacterias grampositivas
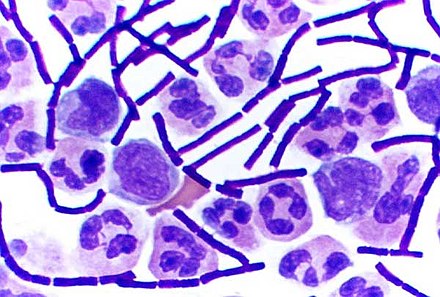

En bacteriología , las bacterias grampositivas son bacterias que dan un resultado positivo en la prueba de tinción de Gram , que se utiliza tradicionalmente para clasificar rápidamente las bacterias en dos grandes categorías según su tipo de pared celular .
Los microbiólogos utilizan la tinción de Gram para clasificar las bacterias en dos categorías principales: grampositivas (+) y gramnegativas (-). Las bacterias grampositivas tienen una capa gruesa de peptidoglicano dentro de la pared celular, mientras que las gramnegativas tienen una capa fina de peptidoglicano.
Las bacterias grampositivas absorben la tinción de violeta cristal utilizada en la prueba y, luego, parecen tener un color púrpura cuando se observan a través de un microscopio óptico . Esto se debe a que la capa gruesa de peptidoglicano en la pared celular bacteriana retiene la tinción después de que se elimina del resto de la muestra, en la etapa de decoloración de la prueba.
Por el contrario, las bacterias gramnegativas no pueden retener la tinción violeta después del paso de decoloración; el alcohol utilizado en esta etapa degrada la membrana externa de las células gramnegativas, haciendo que la pared celular sea más porosa e incapaz de retener la tinción violeta cristal. Su capa de peptidoglicano es mucho más delgada y está intercalada entre una membrana celular interna y una membrana externa bacteriana , lo que hace que absorban la contratinción ( safranina o fucsina ) y se vean rojas o rosadas.
A pesar de su capa de peptidoglicano más gruesa, las bacterias grampositivas son más receptivas a ciertos antibióticos dirigidos a la pared celular que las bacterias gramnegativas, debido a la ausencia de la membrana externa. [1]
Características


En general, en las bacterias grampositivas están presentes las siguientes características: [2]
- Membrana lipídica citoplasmática
- Capa gruesa de peptidoglicano
- Están presentes ácidos teicoicos y lipoides, formando ácidos lipoteicoicos , que sirven como agentes quelantes , y también para ciertos tipos de adherencia.
- Las cadenas de peptidoglicano se reticulan para formar paredes celulares rígidas mediante una enzima bacteriana DD -transpeptidasa .
- Un volumen de periplasma mucho menor que el de las bacterias gramnegativas.
Solo algunas especies tienen una cápsula , que generalmente consiste en polisacáridos . Además, solo algunas especies son flageladas , y cuando tienen flagelos , solo tienen dos anillos corporales basales para sostenerlos, mientras que las gramnegativas tienen cuatro. Tanto las bacterias grampositivas como las gramnegativas suelen tener una capa superficial llamada capa S. En las bacterias grampositivas, la capa S está unida a la capa de peptidoglicano. La capa S de las bacterias gramnegativas está unida directamente a la membrana externa . Específica de las bacterias grampositivas es la presencia de ácidos teicoicos en la pared celular. Algunos de estos son ácidos lipoteicoicos, que tienen un componente lipídico en la membrana celular que puede ayudar a anclar el peptidoglicano. [3]
Clasificación
Junto con la forma celular , la tinción de Gram es un método rápido que se utiliza para diferenciar especies bacterianas. Esta tinción, junto con las pruebas de requerimiento de crecimiento y sensibilidad a los antibióticos y otras pruebas macroscópicas y fisiológicas, forman una base para la clasificación y subdivisión práctica de las bacterias (p. ej., consulte la figura y las versiones anteriores a 1990 del Manual de bacteriología sistemática de Bergey ). [ cita requerida ]

Históricamente , el reino Monera se dividió en cuatro divisiones basadas principalmente en la tinción de Gram: Bacillota (tinción positiva), Gracilicutes (tinción negativa), Mollicutes (tinción neutra) y Mendocutes (tinción variable). [4] Con base en estudios filogenéticos del ARN ribosómico 16S del difunto microbiólogo Carl Woese y colaboradores y colegas de la Universidad de Illinois , se cuestionó la monofilia de las bacterias grampositivas, [5] con importantes implicaciones para el estudio terapéutico y general de estos organismos. Con base en estudios moleculares de las secuencias 16S, Woese reconoció doce filos bacterianos . Dos de estos eran grampositivos y se dividieron según la proporción del contenido de guanina y citosina en su ADN . El filo alto en G + C estaba formado por Actinobacteria , y el filo bajo en G + C contenía Firmicutes . [5] Los Actinomycetota incluyen los géneros Corynebacterium , Mycobacterium , Nocardia y Streptomyces . Los Bacillota (bajo en G + C) tienen un contenido de GC de 45-60%, pero este es menor que el de los Actinomycetota. [2]
Importancia de la membrana celular externa en la clasificación bacteriana
Se ha sugerido que esta sección se divida en otro artículo titulado Tinción de Gram . ( Discutir ) (noviembre de 2023) |

Aunque las bacterias se dividen tradicionalmente en dos grupos principales, gram-positivas y gram-negativas, en función de su propiedad de retención de la tinción de Gram, este sistema de clasificación es ambiguo ya que se refiere a tres aspectos distintos (resultado de la tinción, organización de la envoltura, grupo taxonómico), que no necesariamente coinciden para algunas especies bacterianas. [6] [7] [8] [9] La respuesta de tinción de gram-positivas y gram-negativas tampoco es una característica confiable ya que estos dos tipos de bacterias no forman grupos filogenéticamente coherentes. [6] Sin embargo, aunque la respuesta de tinción de Gram es un criterio empírico, su base radica en las marcadas diferencias en la ultraestructura y la composición química de la pared celular bacteriana, marcadas por la ausencia o presencia de una membrana lipídica externa. [6] [10]
Todas las bacterias grampositivas están delimitadas por una membrana lipídica de una sola unidad y, en general, contienen una capa gruesa (20–80 nm) de peptidoglicano responsable de retener la tinción de Gram. Varias otras bacterias, que están delimitadas por una sola membrana, pero se tiñen como gramnegativas debido a la falta de la capa de peptidoglicano, como en los micoplasmas , o a su incapacidad para retener la tinción de Gram debido a la composición de su pared celular, también muestran una relación estrecha con las bacterias grampositivas. Para las células bacterianas delimitadas por una sola membrana celular, se ha propuesto el término bacterias monodérmicas . [6] [10]
A diferencia de las bacterias grampositivas, todas las bacterias gramnegativas típicas están delimitadas por una membrana citoplasmática y una membrana celular externa; contienen solo una fina capa de peptidoglicano (2-3 nm) entre estas membranas. La presencia de membranas celulares internas y externas define un nuevo compartimento en estas células: el espacio periplásmico o el compartimento periplásmico. Estas bacterias han sido designadas como bacterias didermas . [6] [10] La distinción entre las bacterias monodermas y didermas está respaldada por indeles de firma conservados en varias proteínas importantes (a saber, DnaK, GroEL). [6] [7] [10] [11] De estos dos grupos de bacterias estructuralmente distintos, se indica que los monodermos son ancestrales. Basándose en una serie de observaciones, incluyendo que las bacterias gram-positivas son las principales productoras de antibióticos y que, en general, las bacterias gram-negativas son resistentes a ellos, se ha propuesto que la membrana celular externa en bacterias gram-negativas (didermas) ha evolucionado como un mecanismo de protección contra la presión de selección de antibióticos . [6] [7] [10] [11] Algunas bacterias, como Deinococcus , que se tiñen de gram-positivas debido a la presencia de una gruesa capa de peptidoglicano y también poseen una membrana celular externa se sugieren como intermediarios en la transición entre bacterias monodérmicas (gram-positivas) y didérmicas (gram-negativas). [6] [11] Las bacterias didérmicas también pueden diferenciarse entre didérmicas simples que carecen de lipopolisacárido, las bacterias didérmicas arquetípicas donde la membrana celular externa contiene lipopolisacárido, y las bacterias didérmicas donde la membrana celular externa está formada por ácido micólico . [8] [11] [12]
Excepciones
En general, las bacterias grampositivas son monodermas y tienen una sola bicapa lipídica , mientras que las bacterias gramnegativas son didermas y tienen dos bicapas. Las excepciones incluyen:
- Algunos taxones carecen de peptidoglicano (como la clase Mollicutes , algunos miembros de Rickettsiales y los insectos endosimbiontes de Enterobacteriales ) y son gram-indeterminados .
- Los Deinococcota tienen bacterias grampositivas, aunque estructuralmente son similares a las bacterias gramnegativas con dos capas.
- Los Chloroflexota tienen una sola capa, pero (con algunas excepciones [13] ) se tiñen de forma negativa. [14] Dos filos relacionados con los Chloroflexi, el clado TM7 y los Ktedonobacteria, también son monodermos. [15] [16]
Algunas especies de Bacillota no son grampositivas. La clase Negativicutes, que incluye a Selenomonas , es didermal y se tiñe de gramnegativa. [12] Además, se ha descubierto que varios taxones bacterianos (a saber, Negativicutes , Fusobacteriota , Synergistota y Elusimicrobiota ) que forman parte del filo Bacillota o se ramifican en su proximidad poseen una estructura celular didermal. [9] [11] [12] Sin embargo, una firma indel conservada (CSI) en la proteína HSP60 ( GroEL ) distingue a todos los filos tradicionales de bacterias gramnegativas (por ejemplo, Pseudomonadota , Aquificota , Chlamydiota , Bacteroidota , Chlorobiota , " Cianobacteria ", Fibrobacterota , Verrucomicrobiota , Planctomycetota , Spirochaetota , Acidobacteriota , etc.) de estas otras bacterias didermicas atípicas, así como otros filos de bacterias monodermicas (por ejemplo, Actinomycetota , Bacillota , Thermotogota , Chloroflexota , etc.). [11] La presencia de este CSI en todas las especies secuenciadas de filos bacterianos gramnegativos que contienen LPS ( lipopolisacáridos ) convencionales proporciona evidencia de que estos filos de bacterias forman un clado monofilético y que no ha ocurrido ninguna pérdida de la membrana externa de ninguna especie de este grupo. [11]
Patogenicidad

En el sentido clásico, seis géneros grampositivos son típicamente patógenos en humanos. Dos de estos, Streptococcus y Staphylococcus , son cocos (con forma de esfera). Los organismos restantes son bacilos (con forma de varilla) y pueden subdividirse en función de su capacidad para formar esporas . Los que no forman esporas son Corynebacterium y Listeria (un cocobacilo), mientras que Bacillus y Clostridium producen esporas. [17] Las bacterias formadoras de esporas pueden dividirse nuevamente en función de su respiración : Bacillus es un anaerobio facultativo , mientras que Clostridium es un anaerobio obligado . [18] Además, Rathybacter , Leifsonia y Clavibacter son tres géneros grampositivos que causan enfermedades en las plantas. Las bacterias grampositivas son capaces de causar infecciones graves y, a veces, fatales en los recién nacidos. [19] Las nuevas especies de bacterias grampositivas clínicamente relevantes también incluyen Catabacter hongkongensis , que es un patógeno emergente perteneciente a Bacillota . [20]
Transformación bacteriana
La transformación es uno de los tres procesos de transferencia horizontal de genes , en el que el material genético exógeno pasa de una bacteria donante a una bacteria receptora; los otros dos procesos son la conjugación (transferencia de material genético entre dos células bacterianas en contacto directo) y la transducción (inyección de ADN bacteriano donante por un virus bacteriófago en una bacteria huésped receptora). [21] [22] En la transformación, el material genético pasa a través del medio intermedio y la captación depende completamente de la bacteria receptora. [21]
En 2014 se sabía que alrededor de 80 especies de bacterias eran capaces de transformarse, divididas aproximadamente de manera uniforme entre bacterias grampositivas y gramnegativas ; el número podría ser una sobreestimación ya que varios de los informes están respaldados por artículos individuales. [21] La transformación entre bacterias grampositivas se ha estudiado en especies de importancia médica como Streptococcus pneumoniae , Streptococcus mutans , Staphylococcus aureus y Streptococcus sanguinis y en bacterias grampositivas del suelo Bacillus subtilis y Bacillus cereus . [23]
Nota ortográfica
Los adjetivos grampositivo y gramnegativo derivan del apellido de Hans Christian Gram ; como adjetivos epónimos , su letra inicial puede ser G mayúscula o g minúscula , dependiendo de qué guía de estilo (por ejemplo, la de los CDC ), si la hay, rija el documento que se está escribiendo. [24]
Referencias
- ^ Biología básica (18 de marzo de 2016). "Bacterias".
- ^ ab Madigan, Michael T.; Martinko, John M. (2006). Brock Biología de microorganismos (11.ª ed.). Pearson Prentice Hall. ISBN 978-0131443297.
- ^ Brown, Stephanie; Santa Maria, John P.; Walker, Suzanne (8 de septiembre de 2013). "Ácidos teicoicos de pared de bacterias grampositivas". Revisión anual de microbiología . 67 (1): 313–336. doi :10.1146/annurev-micro-092412-155620. ISSN 0066-4227. PMC 3883102 . PMID 24024634.
- ^ Gibbons, NE; Murray, RGE (1978). "Propuestas relativas a los taxones superiores de bacterias". Revista internacional de microbiología sistemática y evolutiva . 28 (1): 1–6. doi : 10.1099/00207713-28-1-1 .
- ^ ab Woese, CR (1987). "Evolución bacteriana". Microbiological Reviews . 51 (2): 221–271. doi :10.1128/MMBR.51.2.221-271.1987. PMC 373105 . PMID 2439888.
- ^ abcdefgh Gupta, RS (1998). "Filogenias de proteínas y secuencias distintivas: una reevaluación de las relaciones evolutivas entre arqueobacterias, eubacterias y eucariotas". Microbiology and Molecular Biology Reviews . 62 (4): 1435–1491. doi :10.1128/MMBR.62.4.1435-1491.1998. PMC 98952 . PMID 9841678.
- ^ abc Gupta, RS (2000). "Las relaciones evolutivas naturales entre procariotas" (PDF) . Critical Reviews in Microbiology . 26 (2): 111–131. CiteSeerX 10.1.1.496.1356 . doi :10.1080/10408410091154219. PMID 10890353. S2CID 30541897. Archivado (PDF) desde el original el 25 de junio de 2013.
- ^ ab Desvaux, M.; Hébraud, M.; Talon, R.; Henderson, IR (2009). "Secreción y localizaciones subcelulares de proteínas bacterianas: un problema de conciencia semántica". Tendencias en microbiología . 17 (4): 139–145. doi :10.1016/j.tim.2009.01.004. PMID 19299134.
- ^ ab Sutcliffe, IC (2010). "Una perspectiva a nivel de filo sobre la arquitectura de la envoltura celular bacteriana". Tendencias en microbiología . 18 (10): 464–470. doi :10.1016/j.tim.2010.06.005. PMID 20637628.
- ^ abcde Gupta, RS (1998). "¿Qué son las arqueobacterias: el tercer dominio de la vida o procariotas monodérmicos relacionados con las bacterias Gram-positivas? Una nueva propuesta para la clasificación de los organismos procariotas". Microbiología molecular . 29 (3): 695–707. doi :10.1046/j.1365-2958.1998.00978.x. PMID 9723910. S2CID 41206658.
- ^ abcdefg Gupta, RS (2011). "Origen de las bacterias didermicas (gramnegativas): la presión de selección antibiótica en lugar de la endosimbiosis probablemente condujo a la evolución de células bacterianas con dos membranas". Antonie van Leeuwenhoek . 100 (2): 171–182. doi :10.1007/s10482-011-9616-8. PMC 3133647 . PMID 21717204.
- ^ abc Marchandin, H.; Teyssier, C.; Campos, J.; Jean-Pierre, H.; Roger, F.; Gay, B.; Carlier, J.-P.; Jumas-Bilak, E. (2009). "Negativicoccus succinicivorans gen. Nov., sp. Nov., aislado de muestras clínicas humanas, descripción enmendada de la familia Veillonellaceae y descripción de Negativicutes classis nov., Selenomonadales ord. nov. y Acidaminococcaceae fam. nov. en el filo bacteriano Firmicutes". Revista internacional de microbiología sistemática y evolutiva . 60 (6): 1271–1279. doi : 10.1099/ijs.0.013102-0 . PMID 19667386.
- ^ Yabe, S.; Aiba, Y.; Sakai, Y.; Hazaka, M.; Yokota, A. (2010). "Thermogemmatispora onikobensis gen. nov., sp. nov. y Thermogemmatispora foliorum sp. nov., aisladas de hojas caídas en suelos geotérmicos, y descripción de Thermogemmatisporaceae fam. nov. y Thermogemmatisporales ord. Nov. dentro de la clase Ktedonobacteria". Revista Internacional de Microbiología Sistemática y Evolutiva . 61 (4): 903–910. doi : 10.1099/ijs.0.024877-0 . PMID 20495028.
- ^ Sutcliffe, IC (2011). "Arquitectura de la envoltura celular en los cloroflexos: una línea de frente cambiante en una guerra territorial filogenética". Microbiología ambiental . 13 (2): 279–282. Bibcode :2011EnvMi..13..279S. doi :10.1111/j.1462-2920.2010.02339.x. PMID 20860732.
- ^ Hugenholtz, P.; Tyson, GW; Webb, RI; Wagner, AM; Blackall, LL (2001). "Investigación de la división candidata TM7, un linaje principal recientemente reconocido de las bacterias del dominio sin representantes conocidos en cultivos puros". Microbiología aplicada y ambiental . 67 (1): 411–419. Bibcode :2001ApEnM..67..411H. doi :10.1128/AEM.67.1.411-419.2001. PMC 92593 . PMID 11133473.
- ^ Cavaletti, L.; Monciardini, P.; Bamonte, R.; Schumann, P.; Rohde, M.; Sosio, M.; Donadio, S. (2006). "Nuevo linaje de bacterias filamentosas, formadoras de esporas y grampositivas del suelo". Applied and Environmental Microbiology . 72 (6): 4360–4369. Bibcode :2006ApEnM..72.4360C. doi :10.1128/AEM.00132-06. PMC 1489649 . PMID 16751552.
- ^ Gladwin, Mark; Trattler, Bill (2007). Microbiología clínica simplificada hasta lo ridículo . Miami, FL: MedMaster. pp. 4–5. ISBN 978-0-940780-81-1.
- ^ Sahebnasagh, R.; Saderi, H.; Owlia, P. (4–7 de septiembre de 2011). Detección de cepas de Staphylococcus aureus resistentes a la meticilina a partir de muestras clínicas en Teherán mediante la detección de los genes mecA y nuc . Primer Congreso Internacional Iraní de Bacteriología Médica. Tabriz, Irán.
- ^ MacDonald, Mhairi (2015). Neonatología de Avery: fisiopatología y tratamiento del recién nacido . Filadelfia, PA: Wolters Kluwer. ISBN 9781451192681.Acceso proporcionado por la Universidad de Pittsburgh.
- ^ Lau, SKP; McNabb, A.; Woo, GKS; Hoang, L.; Fung, AMY; Chung, LMW; Woo, PCY; Yuen, K.-Y. (22 de noviembre de 2006). "Catabacter hongkongensis gen. nov., sp. nov., aislado de hemocultivos de pacientes de Hong Kong y Canadá". Revista de microbiología clínica . 45 (2): 395–401. doi : 10.1128/jcm.01831-06 . ISSN 0095-1137. PMC 1829005 . PMID 17122022.
- ^ abc Johnston, C.; Martin, B.; Fichant, G.; Polard, P; Claverys, JP (2014). "Transformación bacteriana: distribución, mecanismos compartidos y control divergente". Nature Reviews. Microbiología . 12 (3): 181–196. doi :10.1038/nrmicro3199. PMID 24509783. S2CID 23559881.
- ^ Korotetskiy I, Shilov S, Kuznetsova T, Kerimzhanova B, Korotetskaya N, Ivanova L, Zubenko N, Parenova R, Reva O (2023). "Análisis de secuencias de genoma completo de aislamientos patógenos grampositivos y gramnegativos del mismo entorno hospitalario para investigar tendencias evolutivas comunes asociadas con el intercambio horizontal de genes, mutaciones y patrones de metilación del ADN". Microorganismos . 11 (2): 323. doi : 10.3390/microorganisms11020323 . PMC 9961978 . PMID 36838287.
- ^ Michod, RE; Bernstein, H.; Nedelcu, AM (2008). "Valor adaptativo del sexo en patógenos microbianos". Infección, genética y evolución . 8 (3): 267–285. doi :10.1016/j.meegid.2008.01.002. PMID 18295550.
- ^ "Guía de estilo para revistas sobre enfermedades infecciosas emergentes". CDC.gov . Centros para el Control y la Prevención de Enfermedades.
Enlaces externos
 Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.
Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.- Estructuras 3D de proteínas asociadas a la membrana plasmática de bacterias grampositivas
- Estructuras 3D de proteínas asociadas a la membrana externa de bacterias grampositivas
