Gene
| Parte de una serie sobre |
| Genética |
|---|
 |
Un cromosoma y su larga cadena de ADN empaquetada desenredada. Los pares de bases del ADN codifican genes, que proporcionan funciones. Un ADN humano puede tener hasta 500 millones de pares de bases con miles de genes. |
En biología , la palabra gen tiene dos significados. El gen mendeliano es una unidad básica de la herencia . El gen molecular es una secuencia de nucleótidos en el ADN que se transcribe para producir un ARN funcional . Existen dos tipos de genes moleculares: los genes codificantes de proteínas y los genes no codificantes. [1] [2] [3]
Durante la expresión genética (la síntesis de ARN o proteínas a partir de un gen), el ADN se copia primero en ARN . El ARN puede ser directamente funcional o ser la plantilla intermedia para la síntesis de una proteína.
La transmisión de genes a la descendencia de un organismo es la base de la herencia de los rasgos fenotípicos de una generación a la siguiente. Estos genes forman diferentes secuencias de ADN, llamadas en conjunto genotipo , que es específico de cada individuo, dentro del acervo genético de la población de una especie determinada . El genotipo, junto con factores ambientales y de desarrollo, determina en última instancia el fenotipo del individuo.
La mayoría de los rasgos biológicos se producen bajo la influencia combinada de poligenes (un conjunto de genes diferentes) e interacciones entre genes y ambiente . Algunos rasgos genéticos son visibles al instante, como el color de los ojos o el número de extremidades, otros no lo son, como el tipo de sangre , el riesgo de enfermedades específicas o los miles de procesos bioquímicos básicos que constituyen la vida .
Un gen puede adquirir mutaciones en su secuencia , dando lugar a diferentes variantes, conocidas como alelos , en la población . Estos alelos codifican versiones ligeramente diferentes de un gen, lo que puede causar diferentes rasgos fenotípicos . [4] Los genes evolucionan debido a la selección natural o la supervivencia del más apto y la deriva genética de los alelos.
El término gen fue introducido por el botánico, fisiólogo vegetal y genetista danés Wilhelm Johannsen en 1909. [5] Está inspirado en el griego antiguo : γόνος, gonos , que significa descendencia y procreación.
Definiciones
Hay muchas formas diferentes de utilizar el término "gen" en función de diferentes aspectos de su herencia, selección, función biológica o estructura molecular, pero la mayoría de estas definiciones se dividen en dos categorías: el gen mendeliano o el gen molecular. [1] [6] [7] [8] [9]
El gen mendeliano es el gen clásico de la genética y se refiere a cualquier rasgo hereditario. Este es el gen descrito en El gen egoísta . [10] Se pueden encontrar análisis más detallados de esta versión de un gen en los artículos Genética y Visión de la evolución centrada en los genes .
La definición de gen molecular es la más utilizada en bioquímica, biología molecular y la mayor parte de la genética: el gen que se describe en términos de secuencia de ADN. [1] Existen muchas definiciones diferentes de este gen, algunas de las cuales son engañosas o incorrectas. [6] [11]
Los primeros trabajos en el campo que se convirtió en genética molecular sugirieron el concepto de que un gen produce una proteína (originalmente "un gen - una enzima"). [12] [13] Sin embargo, los genes que producen ARN represores se propusieron en la década de 1950 [14] y en la década de 1960, los libros de texto usaban definiciones de genes moleculares que incluían aquellas que especificaban moléculas de ARN funcionales como el ARN ribosómico y el ARNt (genes no codificantes), así como genes codificadores de proteínas. [15]
Esta idea de dos tipos de genes todavía forma parte de la definición de gen en la mayoría de los libros de texto. Por ejemplo,
La función principal del genoma es producir moléculas de ARN. Se copian porciones seleccionadas de la secuencia de nucleótidos del ADN en una secuencia de nucleótidos del ARN correspondiente, que codifica una proteína (si es un ARNm) o forma un ARN "estructural", como una molécula de ARN de transferencia (ARNt) o ARN ribosómico (ARNr). Cada región de la hélice del ADN que produce una molécula de ARN funcional constituye un gen. [16]
Definimos un gen como una secuencia de ADN que se transcribe. Esta definición incluye genes que no codifican proteínas (no todas las transcripciones son ARN mensajero). La definición normalmente excluye regiones del genoma que controlan la transcripción pero que no se transcriben. Encontraremos algunas excepciones a nuestra definición de gen; sorprendentemente, no hay ninguna definición que sea completamente satisfactoria. [17]
Un gen es una secuencia de ADN que codifica un producto difundible. Este producto puede ser una proteína (como es el caso de la mayoría de los genes) o puede ser ARN (como es el caso de los genes que codifican ARNt y ARNr). La característica crucial es que el producto se difunde fuera de su sitio de síntesis para actuar en otro lugar. [18]
Las partes importantes de estas definiciones son: (1) que un gen corresponde a una unidad de transcripción; (2) que los genes producen ARNm y ARN no codificante; y (3) las secuencias reguladoras controlan la expresión génica pero no son parte del gen en sí. Sin embargo, hay otra parte importante de la definición que se destaca en el libro de Kostas Kampourakis Making Sense of Genes .
Por ello, en este libro consideraré los genes como secuencias de ADN que codifican información para productos funcionales, ya sean proteínas o moléculas de ARN. Con "codificación de información" me refiero a que la secuencia de ADN se utiliza como plantilla para la producción de una molécula de ARN o una proteína que realiza alguna función. [6]
El énfasis en la función es esencial porque hay tramos de ADN que producen transcripciones no funcionales y no se califican como genes. Entre ellos se incluyen ejemplos obvios como pseudogenes transcritos, así como ejemplos menos obvios como el ARN basura producido como ruido debido a errores de transcripción. Para calificar como un verdadero gen, según esta definición, uno tiene que demostrar que la transcripción tiene una función biológica. [6]
Las primeras especulaciones sobre el tamaño de un gen típico se basaban en el mapeo genético de alta resolución y en el tamaño de las proteínas y las moléculas de ARN. Una longitud de 1500 pares de bases parecía razonable en ese momento (1965). [15] Esto se basaba en la idea de que el gen era el ADN que era directamente responsable de la producción del producto funcional. El descubrimiento de los intrones en la década de 1970 significó que muchos genes eucariotas eran mucho más grandes de lo que implicaría el tamaño del producto funcional. Los genes codificadores de proteínas típicos de mamíferos, por ejemplo, tienen una longitud de aproximadamente 62.000 pares de bases (región transcrita) y, dado que hay alrededor de 20.000 de ellos, ocupan aproximadamente el 35-40% del genoma de los mamíferos (incluido el genoma humano). [19] [20] [21]
A pesar de que hace más de 50 años que se conocen tanto los genes codificadores de proteínas como los genes no codificantes, todavía hay numerosos libros de texto, sitios web y publicaciones científicas que definen un gen como una secuencia de ADN que especifica una proteína. En otras palabras, la definición se limita a los genes codificadores de proteínas. A continuación se incluye un ejemplo extraído de un artículo reciente de American Scientist.
... para evaluar verdaderamente la importancia potencial de los genes de novo, nos basamos en una definición estricta de la palabra "gen" con la que casi todos los expertos pueden estar de acuerdo. En primer lugar, para que una secuencia de nucleótidos se considere un gen verdadero, debe estar presente un marco de lectura abierto (ORF). El ORF puede considerarse como el "gen en sí mismo"; comienza con una marca de inicio común para todos los genes y termina con una de tres posibles señales de meta. Una de las enzimas clave en este proceso, la ARN polimerasa, se desplaza a lo largo de la cadena de ADN como un tren en un monorraíl, transcribiéndola en su forma de ARN mensajero. Este punto nos lleva a nuestro segundo criterio importante: un gen verdadero es aquel que se transcribe y se traduce. Es decir, un gen verdadero se utiliza primero como plantilla para fabricar ARN mensajero transitorio, que luego se traduce en una proteína. [22]
Esta definición restringida es tan común que ha dado lugar a muchos artículos recientes que critican esta "definición estándar" y piden una nueva definición ampliada que incluya los genes no codificantes. Sin embargo, algunos autores modernos todavía no reconocen los genes no codificantes, aunque esta denominada "nueva" definición se reconoce desde hace más de medio siglo. [23] [24] [25]
Aunque algunas definiciones pueden ser más ampliamente aplicables que otras, la complejidad fundamental de la biología significa que ninguna definición de un gen puede capturar todos los aspectos a la perfección. No todos los genomas son ADN (por ejemplo, los virus de ARN ), [26] los operones bacterianos son múltiples regiones codificantes de proteínas transcritas en ARNm grandes únicos, el empalme alternativo permite que una sola región genómica codifique múltiples productos de distrito y el trans-empalme concatena ARNm de secuencias codificantes más cortas a lo largo del genoma. [27] [28] [29] Dado que las definiciones moleculares excluyen elementos como intrones, promotores y otras regiones reguladoras , estos se consideran en cambio "asociados" con el gen y afectan su función.
A veces se utiliza una definición operativa aún más amplia para abarcar la complejidad de estos diversos fenómenos, en la que un gen se define como una unión de secuencias genómicas que codifican un conjunto coherente de productos funcionales potencialmente superpuestos. [30] Esta definición clasifica a los genes por sus productos funcionales (proteínas o ARN) en lugar de sus loci de ADN específicos, y los elementos reguladores se clasifican como regiones asociadas a genes . [30]
Historia
Descubrimiento de unidades hereditarias discretas

La existencia de unidades heredables discretas fue sugerida por primera vez por Gregor Mendel (1822-1884). [31] De 1857 a 1864, en Brno , Imperio austríaco (hoy República Checa), estudió los patrones de herencia en 8000 plantas de guisantes comestibles comunes , rastreando rasgos distintivos de los padres a la descendencia. Los describió matemáticamente como 2 n combinaciones donde n es el número de características diferentes en los guisantes originales. Aunque no utilizó el término gen , explicó sus resultados en términos de unidades hereditarias discretas que dan lugar a características físicas observables. Esta descripción prefiguró la distinción de Wilhelm Johannsen entre genotipo (el material genético de un organismo) y fenotipo (los rasgos observables de ese organismo). Mendel también fue el primero en demostrar el surtido independiente , la distinción entre rasgos dominantes y recesivos , la distinción entre un heterocigoto y un homocigoto y el fenómeno de la herencia discontinua.
Antes del trabajo de Mendel, la teoría dominante de la herencia era la de la herencia combinada , [32] que sugería que cada progenitor aportaba fluidos al proceso de fertilización y que los rasgos de los progenitores se combinaban y mezclaban para producir la descendencia. Charles Darwin desarrolló una teoría de la herencia que denominó pangénesis , del griego pan ("todo, completo") y génesis ("nacimiento") / genos ("origen"). [33] [34] Darwin utilizó el término gémula para describir partículas hipotéticas que se mezclarían durante la reproducción.
El trabajo de Mendel pasó en gran medida desapercibido después de su primera publicación en 1866, pero fue redescubierto a fines del siglo XIX por Hugo de Vries , Carl Correns y Erich von Tschermak , quienes (afirmaron haber) llegado a conclusiones similares en su propia investigación. [35] Específicamente, en 1889, Hugo de Vries publicó su libro Intracellular Pangenesis , [36] en el que postuló que diferentes caracteres tienen portadores hereditarios individuales y que la herencia de rasgos específicos en los organismos viene en partículas. De Vries llamó a estas unidades "pangenes" ( Pangens en alemán), en honor a la teoría de la pangénesis de Darwin de 1868.
Veinte años después, en 1909, Wilhelm Johannsen introdujo el término "gen" [5] y, en 1906, William Bateson , el de " genética " [37] [30] mientras que Eduard Strasburger , entre otros, todavía utilizaba el término "pangen" para la unidad física y funcional fundamental de la herencia. [36] : Prefacio del traductor, viii
Descubrimiento del ADN
Los avances en la comprensión de los genes y la herencia continuaron durante todo el siglo XX. Se demostró que el ácido desoxirribonucleico (ADN) es el depósito molecular de la información genética mediante experimentos realizados en los años 1940 y 1950. [38] [39] La estructura del ADN fue estudiada por Rosalind Franklin y Maurice Wilkins utilizando cristalografía de rayos X , lo que llevó a James D. Watson y Francis Crick a publicar un modelo de la molécula de ADN bicatenario cuyas bases de nucleótidos apareadas indicaban una hipótesis convincente para el mecanismo de replicación genética. [40] [41]
A principios de la década de 1950, la opinión predominante era que los genes de un cromosoma actuaban como entidades discretas dispuestas como cuentas en un collar. Los experimentos de Benzer con mutantes defectuosos en la región rII del bacteriófago T4 (1955-1959) demostraron que los genes individuales tienen una estructura lineal simple y es probable que sean equivalentes a una sección lineal de ADN. [42] [43]
En conjunto, este conjunto de investigaciones estableció el dogma central de la biología molecular , que afirma que las proteínas se traducen a partir del ARN , que se transcribe a partir del ADN . Desde entonces se ha demostrado que este dogma tiene excepciones, como la transcripción inversa en los retrovirus . El estudio moderno de la genética a nivel del ADN se conoce como genética molecular .
En 1972, Walter Fiers y su equipo fueron los primeros en determinar la secuencia de un gen: el de la proteína de la cubierta del bacteriófago MS2 . [44] El desarrollo posterior de la secuenciación de ADN de terminación de cadena en 1977 por Frederick Sanger mejoró la eficiencia de la secuenciación y la convirtió en una herramienta de laboratorio de rutina. [45] Una versión automatizada del método de Sanger se utilizó en las primeras fases del Proyecto Genoma Humano . [46]
La síntesis moderna y sus sucesoras
Las teorías desarrolladas a principios del siglo XX para integrar la genética mendeliana con la evolución darwiniana se denominan síntesis moderna , un término introducido por Julian Huxley . [47]
Esta visión de la evolución fue enfatizada por la visión centrada en los genes de la evolución de George C. Williams . Propuso que el gen mendeliano es una unidad de selección natural con la definición: "aquello que se segrega y recombina con una frecuencia apreciable". [48] : 24 Richard Dawkins popularizó ideas relacionadas que enfatizaban la centralidad de los genes mendelianos y la importancia de la selección natural en la evolución . [10] [49]
El desarrollo de la teoría neutral de la evolución a finales de los años 1960 condujo al reconocimiento de que la deriva genética aleatoria es un factor importante en la evolución y que la teoría neutral debería ser la hipótesis nula de la evolución molecular. [50] Esto condujo a la construcción de árboles filogenéticos y al desarrollo del reloj molecular , que es la base de todas las técnicas de datación que utilizan secuencias de ADN. Estas técnicas no se limitan a las secuencias de genes moleculares, sino que se pueden utilizar en todos los segmentos de ADN del genoma.
Base molecular

ADN
La gran mayoría de los organismos codifican sus genes en largas cadenas de ADN (ácido desoxirribonucleico). El ADN consiste en una cadena formada por cuatro tipos de subunidades de nucleótidos , cada una compuesta por: un azúcar de cinco carbonos ( 2-desoxirribosa ), un grupo fosfato y una de las cuatro bases adenina , citosina , guanina y timina . [51] : 2.1
Dos cadenas de ADN se enroscan entre sí para formar una doble hélice de ADN con la cadena principal de fosfato y azúcar en espiral alrededor del exterior y las bases apuntando hacia adentro, con la adenina apareándose con la timina y la guanina con la citosina. La especificidad del apareamiento de bases se produce porque la adenina y la timina se alinean para formar dos enlaces de hidrógeno , mientras que la citosina y la guanina forman tres enlaces de hidrógeno. Por lo tanto, las dos hebras de una doble hélice deben ser complementarias , con su secuencia de bases coincidente de modo que las adeninas de una hebra se apareen con las timinas de la otra hebra, y así sucesivamente. [51] : 4.1
Debido a la composición química de los residuos de pentosa de las bases, las cadenas de ADN tienen direccionalidad. Un extremo de un polímero de ADN contiene un grupo hidroxilo expuesto en la desoxirribosa ; esto se conoce como el extremo 3' de la molécula. El otro extremo contiene un grupo fosfato expuesto ; este es el extremo 5' . Las dos cadenas de una doble hélice corren en direcciones opuestas. La síntesis de ácidos nucleicos, incluida la replicación y transcripción del ADN , ocurre en la dirección 5'→3', porque se agregan nuevos nucleótidos a través de una reacción de deshidratación que utiliza el hidroxilo 3' expuesto como nucleófilo . [52] : 27.2
La expresión de los genes codificados en el ADN comienza con la transcripción del gen en ARN , un segundo tipo de ácido nucleico que es muy similar al ADN, pero cuyos monómeros contienen el azúcar ribosa en lugar de desoxirribosa . El ARN también contiene la base uracilo en lugar de timina . Las moléculas de ARN son menos estables que el ADN y suelen ser monocatenarias. Los genes que codifican proteínas están compuestos por una serie de secuencias de tres nucleótidos llamadas codones , que sirven como las "palabras" en el "lenguaje" genético. El código genético especifica la correspondencia durante la traducción de proteínas entre codones y aminoácidos . El código genético es casi el mismo para todos los organismos conocidos. [51] : 4.1
Cromosomas


El conjunto total de genes de un organismo o célula se conoce como su genoma , que puede estar almacenado en uno o más cromosomas . Un cromosoma consiste en una única hélice de ADN muy larga en la que se codifican miles de genes. [51] : 4.2 La región del cromosoma en la que se encuentra un gen en particular se denomina su locus . Cada locus contiene un alelo de un gen; sin embargo, los miembros de una población pueden tener diferentes alelos en el locus, cada uno con una secuencia genética ligeramente diferente.
La mayoría de los genes eucariotas se almacenan en un conjunto de cromosomas grandes y lineales. Los cromosomas se empaquetan dentro del núcleo en complejo con proteínas de almacenamiento llamadas histonas para formar una unidad llamada nucleosoma . El ADN empaquetado y condensado de esta manera se llama cromatina . [51] : 4.2 La forma en que el ADN se almacena en las histonas, así como las modificaciones químicas de la propia histona, regulan si una región particular del ADN es accesible para la expresión génica . Además de los genes, los cromosomas eucariotas contienen secuencias implicadas en asegurar que el ADN se copie sin degradación de las regiones finales y se clasifique en células hijas durante la división celular: orígenes de replicación , telómeros y el centrómero . [51] : 4.2 Los orígenes de replicación son las regiones de secuencia donde se inicia la replicación del ADN para hacer dos copias del cromosoma. Los telómeros son largos tramos de secuencias repetitivas que tapan los extremos de los cromosomas lineales y evitan la degradación de las regiones codificantes y reguladoras durante la replicación del ADN . La longitud de los telómeros disminuye cada vez que se replica el genoma y se la ha relacionado con el proceso de envejecimiento . [54] El centrómero es necesario para unir las fibras del huso para separar las cromátidas hermanas en células hijas durante la división celular . [51] : 18.2
Los procariotas ( bacterias y arqueas ) suelen almacenar sus genomas en un único cromosoma circular grande . De forma similar, algunos orgánulos eucariotas contienen un cromosoma circular remanente con una pequeña cantidad de genes. [51] : 14.4 Los procariotas a veces complementan su cromosoma con pequeños círculos adicionales de ADN llamados plásmidos , que normalmente codifican sólo unos pocos genes y son transferibles entre individuos. Por ejemplo, los genes de resistencia a los antibióticos suelen estar codificados en plásmidos bacterianos y pueden transmitirse entre células individuales, incluso las de especies diferentes, mediante transferencia horizontal de genes . [55]
Mientras que los cromosomas de los procariotas son relativamente densos en genes, los de los eucariotas a menudo contienen regiones de ADN que no cumplen ninguna función obvia. Los eucariotas unicelulares simples tienen cantidades relativamente pequeñas de dicho ADN, mientras que los genomas de los organismos multicelulares complejos , incluidos los humanos, contienen una mayoría absoluta de ADN sin una función identificada. [56] A menudo se ha hecho referencia a este ADN como " ADN basura ". Sin embargo, análisis más recientes sugieren que, aunque el ADN codificador de proteínas constituye apenas el 2% del genoma humano , alrededor del 80% de las bases del genoma pueden expresarse, por lo que el término "ADN basura" puede ser un nombre inapropiado. [27]
Estructura y función
Estructura
|
La estructura de un gen codificador de proteínas consta de muchos elementos de los cuales la secuencia codificadora de proteínas propiamente dicha suele ser solo una pequeña parte. Entre ellos se incluyen los intrones y las regiones no traducidas del ARNm maduro. Los genes no codificantes también pueden contener intrones que se eliminan durante el procesamiento para producir el ARN funcional maduro.
Todos los genes están asociados con secuencias reguladoras que son necesarias para su expresión. En primer lugar, los genes requieren una secuencia promotora . El promotor es reconocido y unido por factores de transcripción que reclutan y ayudan a la ARN polimerasa a unirse a la región para iniciar la transcripción. [51] : 7.1 El reconocimiento ocurre típicamente como una secuencia de consenso como la caja TATA . Un gen puede tener más de un promotor, lo que resulta en ARN mensajeros ( ARNm ) que difieren en qué tan lejos se extienden en el extremo 5'. [58] Los genes altamente transcritos tienen secuencias promotoras "fuertes" que forman fuertes asociaciones con factores de transcripción, iniciando así la transcripción a una tasa alta. Otros genes tienen promotores "débiles" que forman asociaciones débiles con factores de transcripción e inician la transcripción con menor frecuencia. [51] : 7.2 Las regiones promotoras eucariotas son mucho más complejas y difíciles de identificar que los promotores procariotas . [51] : 7.3
Además, los genes pueden tener regiones reguladoras muchas kilobases antes o después del gen que alteran la expresión. Estas actúan uniéndose a factores de transcripción que luego hacen que el ADN se enrolle de manera que la secuencia reguladora (y el factor de transcripción unido) se acerque al sitio de unión de la ARN polimerasa. [59] Por ejemplo, los potenciadores aumentan la transcripción uniéndose a una proteína activadora que luego ayuda a reclutar la ARN polimerasa al promotor; por el contrario, los silenciadores se unen a proteínas represoras y hacen que el ADN esté menos disponible para la ARN polimerasa. [60]
El ARN mensajero maduro producido a partir de genes codificadores de proteínas contiene regiones no traducidas en ambos extremos que contienen sitios de unión para ribosomas , proteínas de unión a ARN , miARN , así como codones de terminación , inicio y parada . [61] Además, la mayoría de los marcos de lectura abiertos eucariotas contienen intrones no traducidos , que se eliminan, y exones , que se conectan entre sí en un proceso conocido como empalme de ARN . Finalmente, los extremos de las transcripciones de genes están definidos por sitios de escisión y poliadenilación (CPA) , donde el pre-ARNm recién producido se escinde y se agrega una cadena de ~200 monofosfatos de adenosina en el extremo 3'. La cola de poli(A) protege al ARNm maduro de la degradación y tiene otras funciones, que afectan la traducción, la localización y el transporte de la transcripción desde el núcleo. El empalme, seguido de CPA, genera el ARNm maduro final , que codifica la proteína o el producto de ARN. [62]
Muchos genes no codificantes en eucariotas tienen diferentes mecanismos de terminación de la transcripción y no tienen colas poli(A).
Muchos genes procariotas están organizados en operones , con múltiples secuencias codificantes de proteínas que se transcriben como una unidad. [63] [64] Los genes de un operón se transcriben como un ARN mensajero continuo , denominado ARNm policistrónico . El término cistrón en este contexto es equivalente a gen. La transcripción del ARNm de un operón suele estar controlada por un represor que puede producirse en estado activo o inactivo dependiendo de la presencia de metabolitos específicos. [65] Cuando está activo, el represor se une a una secuencia de ADN al principio del operón, llamada región operadora , y reprime la transcripción del operón ; cuando el represor está inactivo, puede producirse la transcripción del operón (véase, por ejemplo, el operón Lac ). Los productos de los genes del operón suelen tener funciones relacionadas y están implicados en la misma red reguladora . [51] : 7.3
Complejidad
Aunque muchos genes tienen estructuras simples, como ocurre con gran parte de la biología, otros pueden ser bastante complejos o representar casos extremos inusuales. Los genes eucariotas a menudo tienen intrones que suelen ser mucho más grandes que sus exones, [66] [67] y esos intrones pueden incluso tener otros genes anidados dentro de ellos . [68] Los potenciadores asociados pueden estar a muchos kilobases de distancia, o incluso en cromosomas completamente diferentes que operan a través del contacto físico entre dos cromosomas. [69] [70] Un solo gen puede codificar múltiples productos funcionales diferentes mediante empalme alternativo y, a la inversa, un gen puede dividirse entre cromosomas, pero esas transcripciones se concatenan nuevamente en una secuencia funcional mediante empalme trans . [71] También es posible que los genes superpuestos compartan parte de su secuencia de ADN, ya sea en cadenas opuestas o en la misma cadena (en un marco de lectura diferente, o incluso en el mismo marco de lectura). [72]
Expresión genética
En todos los organismos, se requieren dos pasos para leer la información codificada en el ADN de un gen y producir la proteína que especifica. Primero, el ADN del gen se transcribe a ARN mensajero ( ARNm ). [51] : 6.1 Segundo, ese ARNm se traduce a proteína. [51] : 6.2 Los genes codificadores de ARN aún deben pasar por el primer paso, pero no se traducen a proteína. [73] El proceso de producir una molécula biológicamente funcional de ARN o proteína se llama expresión génica , y la molécula resultante se llama producto génico .
Código genético

La secuencia de nucleótidos del ADN de un gen especifica la secuencia de aminoácidos de una proteína a través del código genético . Los conjuntos de tres nucleótidos, conocidos como codones , corresponden cada uno a un aminoácido específico. [51] : 6 El principio de que tres bases secuenciales de ADN codifican cada aminoácido se demostró en 1961 utilizando mutaciones de cambio de marco en el gen rIIB del bacteriófago T4 [74] (ver experimento de Crick, Brenner et al. ).
Además, un " codón de inicio " y tres " codones de terminación " indican el comienzo y el final de la región codificante de la proteína . Hay 64 codones posibles (cuatro nucleótidos posibles en cada una de las tres posiciones, por lo tanto, 4 3 codones posibles) y solo 20 aminoácidos estándar; por lo tanto, el código es redundante y múltiples codones pueden especificar el mismo aminoácido. La correspondencia entre codones y aminoácidos es casi universal entre todos los organismos vivos conocidos. [75]
Transcripción
La transcripción produce una molécula de ARN monocatenario conocida como ARN mensajero , cuya secuencia de nucleótidos es complementaria al ADN del que se transcribió. [51] : 6.1 El ARNm actúa como intermediario entre el gen de ADN y su producto proteico final. El ADN del gen se utiliza como plantilla para generar un ARNm complementario . El ARNm coincide con la secuencia de la cadena codificante de ADN del gen porque se sintetiza como complemento de la cadena plantilla . La transcripción la realiza una enzima llamada ARN polimerasa , que lee la cadena plantilla en la dirección 3' a 5' y sintetiza el ARN de 5' a 3' . Para iniciar la transcripción, la polimerasa primero reconoce y se une a una región promotora del gen. Por lo tanto, un mecanismo principal de regulación génica es el bloqueo o secuestro de la región promotora, ya sea mediante una unión estrecha por moléculas represoras que bloquean físicamente la polimerasa o mediante la organización del ADN de modo que la región promotora no sea accesible. [51] : 7
En los procariotas , la transcripción ocurre en el citoplasma ; para transcripciones muy largas, la traducción puede comenzar en el extremo 5' del ARN mientras el extremo 3' aún se está transcribiendo. En los eucariotas , la transcripción ocurre en el núcleo, donde se almacena el ADN de la célula. La molécula de ARN producida por la polimerasa se conoce como transcripción primaria y sufre modificaciones postranscripcionales antes de ser exportada al citoplasma para su traducción. Una de las modificaciones realizadas es el empalme de intrones que son secuencias en la región transcrita que no codifican una proteína. Los mecanismos de empalme alternativo pueden dar como resultado transcripciones maduras del mismo gen que tienen secuencias diferentes y, por lo tanto, codifican proteínas diferentes. Esta es una forma importante de regulación en las células eucariotas y también ocurre en algunos procariotas. [51] : 7.5 [76]
Traducción

La traducción es el proceso por el cual una molécula de ARNm madura se utiliza como plantilla para sintetizar una nueva proteína . [51] : 6.2 La traducción la llevan a cabo los ribosomas , grandes complejos de ARN y proteína responsables de llevar a cabo las reacciones químicas para agregar nuevos aminoácidos a una cadena polipeptídica en crecimiento mediante la formación de enlaces peptídicos . El código genético se lee de tres en tres nucleótidos a la vez, en unidades llamadas codones , a través de interacciones con moléculas de ARN especializadas llamadas ARN de transferencia (ARNt). Cada ARNt tiene tres bases desapareadas conocidas como anticodón que son complementarias al codón que lee en el ARNm. El ARNt también está unido covalentemente al aminoácido especificado por el codón complementario. Cuando el ARNt se une a su codón complementario en una cadena de ARNm, el ribosoma une su carga de aminoácidos a la nueva cadena polipeptídica, que se sintetiza desde el extremo amino hasta el extremo carboxilo . Durante y después de la síntesis, la mayoría de las proteínas nuevas deben plegarse a su estructura tridimensional activa antes de poder llevar a cabo sus funciones celulares. [51] : 3
Regulación
Los genes se regulan para que se expresen solo cuando se necesita el producto, ya que la expresión se basa en recursos limitados. [51] : 7 Una célula regula su expresión genética dependiendo de su entorno externo (por ejemplo, nutrientes disponibles , temperatura y otras tensiones ), su entorno interno (por ejemplo, ciclo de división celular , metabolismo , estado de infección ) y su papel específico si se trata de un organismo multicelular . La expresión genética se puede regular en cualquier paso: desde la iniciación de la transcripción , hasta el procesamiento del ARN , pasando por la modificación postraduccional de la proteína. La regulación de los genes del metabolismo de la lactosa en E. coli ( operón lac ) fue el primer mecanismo de este tipo que se describió en 1961. [77]
Genes de ARN
Un gen típico codificador de proteínas se copia primero en ARN como intermediario en la fabricación del producto proteico final. [51] : 6.1 En otros casos, las moléculas de ARN son los productos funcionales reales, como en la síntesis de ARN ribosómico y ARN de transferencia . Algunos ARN conocidos como ribozimas son capaces de función enzimática , mientras que otros como los microARN y los riboswitches tienen funciones reguladoras. Las secuencias de ADN a partir de las cuales se transcriben dichos ARN se conocen como genes de ARN no codificante . [73]
Algunos virus almacenan sus genomas completos en forma de ARN y no contienen ADN en absoluto. [78] [79] Debido a que utilizan ARN para almacenar genes, sus huéspedes celulares pueden sintetizar sus proteínas tan pronto como son infectados y sin la demora de esperar la transcripción. [80] Por otro lado, los retrovirus de ARN , como el VIH , requieren la transcripción inversa de su genoma de ARN a ADN antes de que sus proteínas puedan sintetizarse.
Herencia

Los organismos heredan sus genes de sus progenitores. Los organismos asexuales simplemente heredan una copia completa del genoma de sus progenitores. Los organismos sexuales tienen dos copias de cada cromosoma porque heredan un conjunto completo de cada progenitor. [51] : 1
Herencia mendeliana
Según la herencia mendeliana , las variaciones en el fenotipo de un organismo (características físicas y conductuales observables) se deben en parte a variaciones en su genotipo (conjunto particular de genes). Cada gen especifica un rasgo particular con una secuencia diferente de un gen ( alelos ) dando lugar a diferentes fenotipos. La mayoría de los organismos eucariotas (como las plantas de guisantes en las que trabajó Mendel) tienen dos alelos para cada rasgo, uno heredado de cada progenitor. [51] : 20
Los alelos de un locus pueden ser dominantes o recesivos ; los alelos dominantes dan lugar a sus fenotipos correspondientes cuando se combinan con cualquier otro alelo para el mismo rasgo, mientras que los alelos recesivos dan lugar a su fenotipo correspondiente solo cuando se combinan con otra copia del mismo alelo. Si conoce los genotipos de los organismos, puede determinar qué alelos son dominantes y cuáles son recesivos. Por ejemplo, si el alelo que especifica tallos altos en plantas de guisantes es dominante sobre el alelo que especifica tallos cortos, entonces las plantas de guisantes que heredan un alelo alto de un progenitor y un alelo corto del otro progenitor también tendrán tallos altos. El trabajo de Mendel demostró que los alelos se clasifican de forma independiente en la producción de gametos o células germinales , lo que garantiza la variación en la siguiente generación. Aunque la herencia mendeliana sigue siendo un buen modelo para muchos rasgos determinados por genes individuales (incluidos varios trastornos genéticos bien conocidos ), no incluye los procesos físicos de replicación del ADN y división celular. [81] [82]
Replicación del ADN y división celular
El crecimiento, desarrollo y reproducción de los organismos dependen de la división celular , el proceso por el cual una sola célula se divide en dos células hijas, generalmente idénticas . Esto requiere primero hacer una copia duplicada de cada gen en el genoma en un proceso llamado replicación del ADN . [51] : 5.2 Las copias son hechas por enzimas especializadas conocidas como ADN polimerasas , que "leen" una hebra del ADN de doble hélice, conocida como la hebra molde, y sintetizan una nueva hebra complementaria. Debido a que la doble hélice del ADN se mantiene unida por apareamiento de bases , la secuencia de una hebra especifica completamente la secuencia de su complemento; por lo tanto, solo una hebra necesita ser leída por la enzima para producir una copia fiel. El proceso de replicación del ADN es semiconservativo ; es decir, la copia del genoma heredado por cada célula hija contiene una hebra original y una nueva hebra de ADN sintetizada. [51] : 5.2
La tasa de replicación del ADN en células vivas se midió primero como la tasa de elongación del ADN del fago T4 en E. coli infectada con fagos y se encontró que era impresionantemente rápida. [83] Durante el período de aumento exponencial del ADN a 37 °C, la tasa de elongación fue de 749 nucleótidos por segundo.
Una vez completada la replicación del ADN, la célula debe separar físicamente las dos copias del genoma y dividirse en dos células distintas unidas por membranas. [51] : 18.2 En los procariotas ( bacterias y arqueas ) esto ocurre generalmente a través de un proceso relativamente simple llamado fisión binaria , en el que cada genoma circular se adhiere a la membrana celular y se separa en las células hijas a medida que la membrana se invagina para dividir el citoplasma en dos porciones unidas por membrana. La fisión binaria es extremadamente rápida en comparación con las tasas de división celular en eucariotas . La división celular eucariota es un proceso más complejo conocido como ciclo celular ; la replicación del ADN ocurre durante una fase de este ciclo conocida como fase S , mientras que el proceso de segregación de cromosomas y división del citoplasma ocurre durante la fase M. [51] : 18.1
Herencia molecular
La duplicación y transmisión del material genético de una generación de células a la siguiente es la base de la herencia molecular y el vínculo entre las imágenes clásicas y moleculares de los genes. Los organismos heredan las características de sus progenitores porque las células de la descendencia contienen copias de los genes en las células de sus progenitores. En los organismos que se reproducen asexualmente , la descendencia será una copia genética o clon del organismo progenitor. En los organismos que se reproducen sexualmente , una forma especializada de división celular llamada meiosis produce células llamadas gametos o células germinales que son haploides , o contienen solo una copia de cada gen. [51] : 20.2 Los gametos producidos por las hembras se denominan óvulos , y los producidos por los machos se denominan espermatozoides . Dos gametos se fusionan para formar un óvulo fertilizado diploide , una sola célula que tiene dos conjuntos de genes, con una copia de cada gen de la madre y una del padre. [51] : 20
Durante el proceso de división celular meiótica, a veces puede ocurrir un evento llamado recombinación genética o entrecruzamiento , en el que una longitud de ADN en una cromátida se intercambia con una longitud de ADN en la cromátida homóloga no hermana correspondiente. Esto puede resultar en la reordenación de alelos vinculados de otro modo. [51] : 5.5 El principio mendeliano de la distribución independiente afirma que cada uno de los dos genes de un progenitor para cada rasgo se clasificará independientemente en gametos; el alelo que hereda un organismo para un rasgo no está relacionado con el alelo que hereda para otro rasgo. De hecho, esto solo es cierto para los genes que no residen en el mismo cromosoma o que están ubicados muy lejos uno del otro en el mismo cromosoma. Cuanto más cerca se encuentren dos genes en el mismo cromosoma, más estrechamente estarán asociados en los gametos y más a menudo aparecerán juntos (lo que se conoce como ligamiento genético ). [84] Los genes que están muy cerca esencialmente nunca se separan porque es extremadamente improbable que se produzca un punto de cruce entre ellos. [84]
Evolución molecular
Mutación
La replicación del ADN es en su mayor parte extremadamente precisa, sin embargo, ocurren errores ( mutaciones ). [51] : 7.6 La tasa de error en células eucariotas puede ser tan baja como 10 −8 por nucleótido por replicación, [85] [86] mientras que para algunos virus de ARN puede ser tan alta como 10 −3 . [87] Esto significa que cada generación, cada genoma humano acumula alrededor de 30 nuevas mutaciones. [88] Las mutaciones pequeñas pueden ser causadas por la replicación del ADN y las secuelas del daño del ADN e incluyen mutaciones puntuales en las que se altera una sola base y mutaciones de cambio de marco en las que se inserta o elimina una sola base. Cualquiera de estas mutaciones puede cambiar el gen por falta de sentido (cambiar un codón para codificar un aminoácido diferente) o sin sentido (un codón de parada prematuro ). [89] Las mutaciones más grandes pueden ser causadas por errores en la recombinación para causar anomalías cromosómicas , incluyendo la duplicación , deleción, reordenamiento o inversión de grandes secciones de un cromosoma. Además, los mecanismos de reparación del ADN pueden introducir errores mutacionales al reparar el daño físico a la molécula. La reparación, incluso con mutación, es más importante para la supervivencia que restaurar una copia exacta, por ejemplo, al reparar roturas de doble cadena . [51] : 5.4
Cuando hay múltiples alelos diferentes para un gen en la población de una especie, se dice que es polimórfico . La mayoría de los alelos diferentes son funcionalmente equivalentes, sin embargo, algunos alelos pueden dar lugar a diferentes rasgos fenotípicos . El alelo más común de un gen se llama tipo salvaje y los alelos raros se llaman mutantes . La variación genética en las frecuencias relativas de diferentes alelos en una población se debe tanto a la selección natural como a la deriva genética . [90] El alelo de tipo salvaje no es necesariamente el ancestro de los alelos menos comunes, ni es necesariamente más apto .
La mayoría de las mutaciones dentro de los genes son neutrales y no tienen efecto sobre el fenotipo del organismo ( mutaciones silenciosas ). Algunas mutaciones no cambian la secuencia de aminoácidos porque múltiples codones codifican el mismo aminoácido ( mutaciones sinónimas ). Otras mutaciones pueden ser neutrales si conducen a cambios en la secuencia de aminoácidos, pero la proteína sigue funcionando de manera similar con el nuevo aminoácido (por ejemplo, mutaciones conservadoras ). Sin embargo, muchas mutaciones son perjudiciales o incluso letales y se eliminan de las poblaciones por selección natural. Los trastornos genéticos son el resultado de mutaciones perjudiciales y pueden deberse a una mutación espontánea en el individuo afectado, o pueden heredarse. Finalmente, una pequeña fracción de las mutaciones son beneficiosas , mejoran la aptitud del organismo y son extremadamente importantes para la evolución, ya que su selección direccional conduce a la evolución adaptativa . [51] : 7.6
Homología de secuencia
La relación entre genes se puede medir comparando las secuencias de su ADN. Si el nivel de similitud supera un valor mínimo, se puede concluir que los genes descienden de un ancestro común; son homólogos . [91] [92] Los genes que están relacionados por descendencia directa de un ancestro común son genes ortólogos: suelen encontrarse en el mismo locus en diferentes especies. Los genes que están relacionados como resultado de un evento de duplicación genética son genes parólogos. [93] [94]
A menudo se supone que las funciones de los genes ortólogos son más similares que las de los genes parálogos, aunque la diferencia es mínima. [95] [96]
Orígenes de nuevos genes
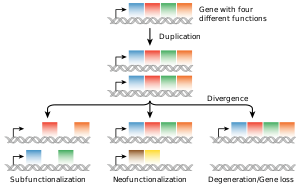
La fuente más común de nuevos genes en los linajes eucariotas es la duplicación genética , que crea una variación en el número de copias de un gen existente en el genoma. [97] [98] Los genes resultantes (parálogos) pueden luego divergir en secuencia y en función. Los conjuntos de genes formados de esta manera componen una familia genética . Las duplicaciones y pérdidas de genes dentro de una familia son comunes y representan una fuente importante de biodiversidad evolutiva . [99] A veces, la duplicación genética puede dar como resultado una copia no funcional de un gen, o una copia funcional puede estar sujeta a mutaciones que resultan en la pérdida de la función; dichos genes no funcionales se denominan pseudogenes . [51] : 7.6
Los genes "huérfanos" , cuya secuencia no muestra similitud con genes existentes, son menos comunes que los duplicados de genes. El genoma humano contiene un estimado de 18 [100] a 60 [101] genes sin homólogos identificables fuera de los humanos. Los genes huérfanos surgen principalmente de la aparición de novo a partir de una secuencia previamente no codificante , o de la duplicación de genes seguida de un cambio de secuencia tan rápido que la relación original se vuelve indetectable. [102] Los genes de novo son típicamente más cortos y de estructura más simple que la mayoría de los genes eucariotas, con pocos intrones o ninguno. [97] Durante largos períodos de tiempo evolutivo, el nacimiento de genes de novo puede ser responsable de una fracción significativa de familias de genes taxonómicamente restringidas. [103]
La transferencia horizontal de genes se refiere a la transferencia de material genético a través de un mecanismo distinto a la reproducción . Este mecanismo es una fuente común de nuevos genes en procariotas , a veces se piensa que contribuye más a la variación genética que la duplicación de genes. [104] Es un medio común de propagación de la resistencia a los antibióticos , la virulencia y las funciones metabólicas adaptativas . [55] [105] Aunque la transferencia horizontal de genes es rara en eucariotas, se han identificado ejemplos probables de genomas de protistas y algas que contienen genes de origen bacteriano. [106] [107]
Genoma
El genoma es el material genético total de un organismo e incluye tanto los genes como las secuencias no codificantes . [108] Los genes eucariotas se pueden anotar utilizando FINDER. [109]
Número de genes

El tamaño del genoma y la cantidad de genes que codifica varían ampliamente entre organismos. Los genomas más pequeños se encuentran en los virus [118] y los viroides (que actúan como un único gen de ARN no codificante). [119] Por el contrario, las plantas pueden tener genomas extremadamente grandes [120] , y el arroz contiene más de 46 000 genes codificadores de proteínas [ 114] . Se estima que la cantidad total de genes codificadores de proteínas (el proteoma de la Tierra ) es de 5 millones de secuencias [121] .
Aunque el número de pares de bases de ADN en el genoma humano se conoce desde la década de 1950, el número estimado de genes ha cambiado con el tiempo a medida que se han refinado las definiciones de genes y los métodos para detectarlos. Las predicciones teóricas iniciales del número de genes humanos en las décadas de 1960 y 1970 se basaron en estimaciones de carga de mutación y el número de ARNm y estas estimaciones tendían a ser de alrededor de 30.000 genes codificadores de proteínas. [122] [123] [124] Durante la década de 1990 hubo estimaciones de hasta 100.000 genes y los primeros datos sobre la detección de ARNm ( etiquetas de secuencia expresada ) sugirieron más que el valor tradicional de 30.000 genes que se había informado en los libros de texto durante la década de 1980. [125]
Las secuencias preliminares del genoma humano confirmaron las predicciones anteriores de unos 30.000 genes codificadores de proteínas; sin embargo, esa estimación ha caído a unos 19.000 con el proyecto de anotación GENCODE en curso. [126] El número de genes no codificantes no se conoce con certeza, pero las últimas estimaciones de Ensembl sugieren 26.000 genes no codificantes. [127]
Genes esenciales
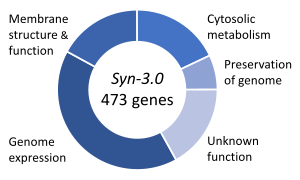
Los genes esenciales son el conjunto de genes que se cree que son críticos para la supervivencia de un organismo. [129] Esta definición supone la abundante disponibilidad de todos los nutrientes relevantes y la ausencia de estrés ambiental. Solo una pequeña parte de los genes de un organismo son esenciales. En las bacterias, se estima que entre 250 y 400 genes son esenciales para Escherichia coli y Bacillus subtilis , lo que supone menos del 10% de sus genes. [130] [131] [132] La mitad de estos genes son ortólogos en ambos organismos y participan en gran medida en la síntesis de proteínas . [132] En la levadura en ciernes Saccharomyces cerevisiae, el número de genes esenciales es ligeramente superior, 1000 genes (~20% de sus genes). [133] Aunque el número es más difícil de medir en eucariotas superiores, se estima que los ratones y los humanos tienen alrededor de 2000 genes esenciales (~10% de sus genes). [134] El organismo sintético, Syn 3 , tiene un genoma mínimo de 473 genes esenciales y genes cuasi-esenciales (necesarios para un crecimiento rápido), aunque 149 tienen una función desconocida. [128]
Los genes esenciales incluyen genes de mantenimiento (cruciales para las funciones celulares básicas) [135] así como genes que se expresan en diferentes momentos en el desarrollo o ciclo de vida de los organismos . [136] Los genes de mantenimiento se utilizan como controles experimentales al analizar la expresión genética , ya que se expresan constitutivamente a un nivel relativamente constante.
Nomenclatura genética y genómica
La nomenclatura genética fue establecida por el Comité de Nomenclatura Genética de HUGO (HGNC), un comité de la Organización del Genoma Humano , para cada gen humano conocido en forma de un nombre de gen aprobado y un símbolo ( abreviatura corta ), al que se puede acceder a través de una base de datos mantenida por el HGNC. Los símbolos se eligen para que sean únicos, y cada gen tiene solo un símbolo (aunque los símbolos aprobados a veces cambian). Los símbolos se mantienen preferiblemente consistentes con otros miembros de una familia de genes y con homólogos en otras especies, particularmente el ratón debido a su papel como organismo modelo común . [137]
Ingeniería genética
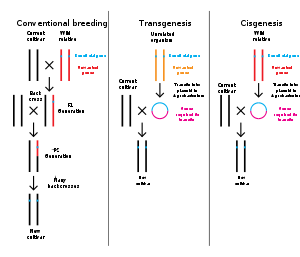
La ingeniería genética es la modificación del genoma de un organismo mediante biotecnología . Desde la década de 1970, se han desarrollado diversas técnicas para agregar, eliminar y editar genes de manera específica en un organismo. [138] Las técnicas de ingeniería genética desarrolladas recientemente utilizan enzimas nucleasas diseñadas para crear una reparación de ADN dirigida en un cromosoma para interrumpir o editar un gen cuando se repara la rotura. [139] [140] [141] [142] El término relacionado biología sintética se utiliza a veces para referirse a la ingeniería genética extensiva de un organismo. [143]
La ingeniería genética es ahora una herramienta de investigación rutinaria con organismos modelo . Por ejemplo, los genes se agregan fácilmente a las bacterias [144] y se utilizan linajes de ratones knockout con la función de un gen específico alterado para investigar la función de ese gen. [145] [146] Muchos organismos han sido modificados genéticamente para aplicaciones en agricultura , biotecnología industrial y medicina .
En el caso de los organismos multicelulares, normalmente se diseña el embrión para que crezca hasta convertirse en un organismo adulto genéticamente modificado . [147] Sin embargo, los genomas de las células de un organismo adulto se pueden editar utilizando técnicas de terapia genética para tratar enfermedades genéticas.
Véase también
- Patente biológica
- Epigenética
- Visión de la evolución centrada en los genes
- Dosis de genes
- Acervo genético
- Redundancia genética
- Silenciamiento genético
- Algoritmo genético
- Haplotipo
- Lista de software de predicción de genes
- Listas de genes humanos
- Medicina predictiva
- Locus de rasgos cuantitativos
- Elemento genético egoísta
- Secuenciación del genoma completo
Referencias
Citas
- ^ abc Orgogozo V, Peluffo AE, Morizot B (2016). "El "gen mendeliano" y el "gen molecular": dos conceptos relevantes de unidades genéticas" (PDF) . Temas actuales en biología del desarrollo . 119 : 1–26. doi :10.1016/bs.ctdb.2016.03.002. PMID 27282022. S2CID 24583286.
- ^ "¿Qué es un gen?: MedlinePlus Genetics". MedlinePlus . 17 de septiembre de 2020 . Consultado el 4 de enero de 2021 .
- ^ Hirsch ED (2002). El nuevo diccionario de alfabetización cultural . Boston: Houghton Mifflin. ISBN 0-618-22647-8.OCLC 50166721 .
- ^ Elston RC, Satagopan JM, Sun S (2012). "Terminología genética". Genética humana estadística . Métodos en biología molecular. Vol. 850. Humana Press. págs. 1–9. doi :10.1007/978-1-61779-555-8_1. ISBN 978-1-61779-554-1. PMC 4450815 . PMID 22307690.
- ^ ab Johannsen W (1909). Elemente der exakten Erblichkeitslehre [ Elementos de la teoría exacta de la herencia ] (en alemán). Jena, Alemania: Gustav Fischer. pag. 124.De la pág. 124: "Dieses" etwas "in den Gameten bezw. in der Zygote, ... – kurz, was wir eben Gene nennen wollen – bedingt sind". (Este "algo" en los gametos o en el cigoto, que tiene una importancia crucial para el carácter del organismo, se suele llamar con el término bastante ambiguo Anlagen [primordio, de la palabra alemana Anlage para "plan, disposición; boceto" ]. Se han sugerido muchos otros términos, la mayoría de ellos, por desgracia, en estrecha relación con ciertas opiniones hipotéticas. La palabra "pangen", que fue introducida por Darwin, es quizás la que se utiliza con más frecuencia en lugar de Anlagen . Sin embargo, la palabra "pangen" no fue bien elegida, ya que es una palabra compuesta que contiene las raíces pan (la forma neutra de Πας todo, cada) y gen (de γί-γ(ε)ν-ομαι, llegar a ser). Solo el significado de esta última [es decir, Aquí entra en consideración el gen ]; sólo debería expresarse la idea básica – [a saber,] que un rasgo en el organismo en desarrollo puede ser determinado o está influido por "algo" en los gametos. No hay hipótesis sobre la naturaleza de este "Algo" debe ser postulado o apoyado por él. Por eso parece más simple utilizar aisladamente la última sílaba gen de la conocida palabra de Darwin, que es la única que nos interesa, para reemplazar con ella la pobre , palabra ambigua Anlage . Por lo tanto, diremos simplemente "gen" y "genes" en lugar de "pangen" y "pangenes". La palabra gen está completamente libre de cualquier hipótesis; expresa solo el hecho establecido de que en cualquier caso muchos rasgos del (Los organismos están determinados por "condiciones", "fundamentos", "planes" específicos, separables y, por tanto, independientes; en una palabra, precisamente lo que queremos llamar genes).
- ^ abcd Kampourakis K (2017). Dando sentido a los genes . Cambridge, Reino Unido: Cambridge University Press.
- ^ Gericke N, Hagberg M (5 de diciembre de 2006). "Definición de modelos históricos de función genética y su relación con la comprensión de la genética por parte de los estudiantes". Ciencia y educación . 16 (7–8): 849–881. Bibcode :2007Sc&Ed..16..849G. doi :10.1007/s11191-006-9064-4. S2CID 144613322.
- ^ Meunier R (2022). «Enciclopedia de filosofía de Stanford: Gene». Enciclopedia de filosofía de Stanford . Consultado el 28 de febrero de 2023 .
- ^ Kellis M, Wold B, Snyder MP, Bernstein BE, Kundaje A, Marinov GK, et al. (abril de 2014). "Definición de elementos funcionales del ADN en el genoma humano". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 111 (17): 6131–8. Bibcode :2014PNAS..111.6131K. doi : 10.1073/pnas.1318948111 . PMC 4035993 . PMID 24753594.
- ^ ab Dawkins R (1976). El gen egoísta . Oxford, Reino Unido: Oxford University Press.
- ^ Stoltz K, Griffiths P (2004). "Genes: análisis filosóficos puestos a prueba". Historia y filosofía de las ciencias de la vida . 26 (1): 5–28. doi :10.1080/03919710412331341621. JSTOR 23333378. PMID 15791804.
- ^ Beadle GW, Tatum EL (noviembre de 1941). "Control genético de las reacciones bioquímicas en Neurospora". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 27 (11): 499–506. Bibcode :1941PNAS...27..499B. doi : 10.1073/pnas.27.11.499 . PMC 1078370 . PMID 16588492.
- ^ Horowitz NH, Berg P, Singer M, Lederberg J, Susman M, Doebley J, Crow JF (enero de 2004). "Un centenario: George W. Beadle, 1903-1989". Genética . 166 (1): 1–10. doi :10.1534/genetics.166.1.1. PMC 1470705 . PMID 15020400.
- ^ Judson HF (1996). El octavo día de la creación (edición ampliada). Plainview, NY (EE. UU.): Cold Spring Harbor Laboratory Press.
- ^ ab Watson JD (1965). Biología molecular del gen . Nueva York, NY, EE. UU.: WA Benjamin, Inc.
- ^ Alberts B, Bray D, Lewis J, Raff M, Roberts K, Watson JD (1994). Biología molecular de la célula: tercera edición . Londres, Reino Unido: Garland Publishing, Inc. ISBN 0-8153-1619-4.
- ^ Moran LA, Horton HR, Scrimgeour KG, Perry MD (2012). Principios de bioquímica: quinta edición . Upper Saddle River, NJ, EE. UU.: Pearson.
- ^ Lewin B (2004). Genes VIII . Upper Saddle River, Nueva Jersey, EE. UU.: Pearson/Prentice Hall.
- ^ Piovesan A, Pelleri MC, Antonaros F, Strippoli P, Caracausi M y Vitale L (2019). "Sobre la longitud, el peso y el contenido de GC del genoma humano". BMC Research Notes . 12 (1): 106–173. doi : 10.1186/s13104-019-4137-z . PMC 6391780 . PMID 30813969.
- ^ Hubé F y Francastel C (2015). "Intrones de mamíferos: cuando la basura genera diversidad molecular". Revista internacional de ciencias moleculares . 16 (3): 4429–4452. doi : 10.3390/ijms16034429 . PMC 4394429 . PMID 25710723.
- ^ Francis WR y Wörheide G (2017). "Relaciones similares de intrones con respecto a secuencias intergénicas en genomas animales". Genome Biology and Evolution . 9 (6): 1582–1598. doi :10.1093/gbe/evx103. PMC 5534336 . PMID 28633296.
- ^ Mortola E, Long M (2021). "Convirtiendo la basura en nosotros: cómo nacen los genes". American Scientist . 109 : 174–182.
- ^ Hopkin K (2009). "La definición cambiante de un gen: con el descubrimiento de que casi todo el genoma se transcribe, la definición de un "gen" necesita otra revisión". BioScience . 59 : 928–931. doi :10.1525/bio.2009.59.11.3. S2CID 88157272.
- ^ Pearson H (2006). "¿Qué es un gen?". Nature . 441 (7092): 399–401. Bibcode :2006Natur.441..398P. doi : 10.1038/441398a . PMID 16724031. S2CID 4420674.
- ^ Pennisi E (2007). "El estudio del ADN obliga a repensar lo que significa ser un gen". Science . 316 (5831): 1556–1557. doi : 10.1126/science.316.5831.1556 . PMID 17569836. S2CID 36463252.
- ^ Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, et al. (noviembre de 2018). Racaniello VR (ed.). "Orígenes y evolución del viroma de ARN global". mBio . 9 (6). Eric Delwart, Luis Enjuanes: e02329–18. doi :10.1128/mBio.02329-18. PMC 6282212 . PMID 30482837.
- ^ ab Pennisi E (junio de 2007). "Genómica. El estudio del ADN obliga a repensar lo que significa ser un gen". Science . 316 (5831): 1556–7. doi : 10.1126/science.316.5831.1556 . PMID 17569836. S2CID 36463252.
- ^ Marande W, Burger G (octubre de 2007). "El ADN mitocondrial como rompecabezas genómico". Science . 318 (5849). AAAS: 415. Bibcode :2007Sci...318..415M. doi :10.1126/science.1148033. PMID 17947575. S2CID 30948765.
- ^ Parra G, Reymond A, Dabbouseh N, Dermitzakis ET, Castelo R, Thomson TM, et al. (enero de 2006). "Quimerismo en tándem como medio para aumentar la complejidad proteica en el genoma humano". Genome Research . 16 (1): 37–44. doi :10.1101/gr.4145906. PMC 1356127 . PMID 16344564.
- ^ abc Gerstein MB, Bruce C, Rozowsky JS, Zheng D, Du J, Korbel JO, et al. (junio de 2007). "¿Qué es un gen, después de ENCODE? Historia y definición actualizada". Genome Research . 17 (6): 669–81. doi : 10.1101/gr.6339607 . PMID 17567988.
- ^ Noble D (septiembre de 2008). "Genes y causalidad". Philosophical Transactions. Series A, Ciencias matemáticas, físicas y de ingeniería . 366 (1878): 3001–15. Bibcode :2008RSPTA.366.3001N. doi : 10.1098/rsta.2008.0086 . PMID 18559318.
- ^ "Mezcla de herencia: una descripción general | Temas de ScienceDirect".
- ^ "génesis" . Oxford English Dictionary (edición en línea). Oxford University Press . (Se requiere suscripción o membresía a una institución participante).
- ^ Magner LN (2002). Una historia de las ciencias de la vida (tercera edición). Marcel Dekker , CRC Press . pág. 371. ISBN. 978-0-203-91100-6.
- ^ Henig RM (2000). El monje en el jardín: el genio perdido y encontrado de Gregor Mendel, el padre de la genética . Boston: Houghton Mifflin. pp. 1–9. ISBN 978-0395-97765-1.
- ^ ab de Vries H (1889). Intracellulare Pangenese [ Pangénesis intracelular ] (en alemán). Traducido por Gager CS . Jena: Verlag von Gustav Fischer.Traducido del alemán al inglés en 1908 por Open Court Publishing Co., Chicago, 1910
- ^ Bateson W (1906). "El progreso de la investigación genética". En Wilks W (ed.). Informe de la Tercera Conferencia Internacional de 1906 sobre Genética . Londres, Inglaterra: Royal Horticultural Society. pp. 90-97.
... la ciencia en sí [es decir, el estudio de la cría e hibridación de plantas] aún no tiene nombre, y sólo podemos describir nuestra búsqueda mediante perífrasis engorrosas y a menudo engañosas. Para resolver esta dificultad, sugiero para la consideración de este Congreso el término
Genética
, que indica suficientemente que nuestros trabajos están dedicados a la elucidación de los fenómenos de la herencia y la variación: en otras palabras, a la fisiología de la Descendencia, con una relación implícita con los problemas teóricos del evolucionista y el sistemático, y la aplicación a los problemas prácticos de los criadores, ya sea de animales o de plantas.
- ^ Avery OT, Macleod CM, McCarty M (febrero de 1944). "Estudios sobre la naturaleza química de la sustancia que induce la transformación de los tipos de neumococo: inducción de la transformación por una fracción de ácido desoxirribonucleico aislada del neumococo tipo III". The Journal of Experimental Medicine . 79 (2): 137–58. doi :10.1084/jem.79.2.137. PMC 2135445 . PMID 19871359. Reimpresión: Avery OT, MacLeod CM, McCarty M (febrero de 1979). "Estudios sobre la naturaleza química de la sustancia que induce la transformación de los tipos de neumococo. Inducciones de transformación por una fracción de ácido desoxirribonucleico aislada del neumococo tipo III". The Journal of Experimental Medicine . 149 (2): 297–326. doi :10.1084/jem.149.2.297. PMC 2184805 . PMID 33226.
- ^ Hershey AD, Chase M (mayo de 1952). "Funciones independientes de la proteína viral y el ácido nucleico en el crecimiento del bacteriófago". The Journal of General Physiology . 36 (1): 39–56. doi :10.1085/jgp.36.1.39. PMC 2147348 . PMID 12981234.
- ^ Judson H (1979). El octavo día de la creación: creadores de la revolución en biología . Cold Spring Harbor Laboratory Press. págs. 51-169. ISBN 978-0-87969-477-7.
- ^ Watson JD, Crick FH (abril de 1953). "Estructura molecular de los ácidos nucleicos: una estructura para el ácido nucleico desoxirribonucleico" (PDF) . Nature . 171 (4356): 737–8. Bibcode :1953Natur.171..737W. doi :10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Benzer S (junio de 1955). "Estructura fina de una región genética en bacteriófagos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 41 (6): 344–54. Bibcode :1955PNAS...41..344B. doi : 10.1073/pnas.41.6.344 . PMC 528093 . PMID 16589677.
- ^ Benzer S (noviembre de 1959). "Sobre la topología de la estructura fina genética". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 45 (11): 1607–20. Bibcode :1959PNAS...45.1607B. doi : 10.1073/pnas.45.11.1607 . PMC 222769 . PMID 16590553.
- ^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (mayo de 1972). "Secuencia de nucleótidos del gen que codifica la proteína de la capa del bacteriófago MS2". Nature . 237 (5350): 82–8. Bibcode :1972Natur.237...82J. doi :10.1038/237082a0. PMID 4555447. S2CID 4153893.
- ^ Sanger F, Nicklen S, Coulson AR (diciembre de 1977). "Secuenciación de ADN con inhibidores de terminación de cadena". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 74 (12): 5463–7. Bibcode :1977PNAS...74.5463S. doi : 10.1073/pnas.74.12.5463 . PMC 431765 . PMID 271968.
- ^ Adams JU (2008). "Tecnologías de secuenciación de ADN". Conocimiento de la educación sobre la naturaleza . SciTable. 1 (1). Nature Publishing Group: 193.
- ^ Huxley J (1942). Evolución: la síntesis moderna . Cambridge, Massachusetts: MIT Press. ISBN 978-0262513661.
- ^ Williams GC (2001). Adaptación y selección natural: una crítica de algunas ideas evolutivas actuales (edición en línea). Princeton: Princeton University Press. ISBN 9781400820108.
- ^ Dawkins R (1989). El fenotipo extendido (edición de bolsillo). Oxford: Oxford University Press. ISBN 978-0-19-286088-0.
- ^ Duret L (2008). "Teoría neutral: la hipótesis nula de la evolución molecular". Nature Education . 1 : 218.
- ^ abcdefghijklmnopqrstu vwxyz aa ab ac ad ae af ag ah ai Alberts B , Johnson A, Lewis J , Raff M , Roberts K, Walter P (2002). Biología molecular de la célula (cuarta edición). Nueva York: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Stryer L, Berg JM, Tymoczko JL (2002). Bioquímica (5ª ed.). San Francisco: WH Freeman. ISBN 978-0-7167-4955-4.
- ^ Bolzer A, Kreth G, Solovei I, Koehler D, Saracoglu K, Fauth C, et al. (mayo de 2005). "Mapas tridimensionales de todos los cromosomas en los núcleos de fibroblastos masculinos humanos y rosetas prometafásicas". PLOS Biology . 3 (5): e157. doi : 10.1371/journal.pbio.0030157 . PMC 1084335 . PMID 15839726.

- ^ Braig M, Schmitt CA (marzo de 2006). "Senescencia inducida por oncogenes: frenando el desarrollo de tumores". Cancer Research . 66 (6): 2881–4. doi : 10.1158/0008-5472.CAN-05-4006 . PMID 16540631.
- ^ ab Bennett PM (marzo de 2008). "Resistencia a antibióticos codificada por plásmidos: adquisición y transferencia de genes de resistencia a antibióticos en bacterias". British Journal of Pharmacology . 153 (Supl 1): S347-57. doi :10.1038/sj.bjp.0707607. PMC 2268074 . PMID 18193080.
- ^ Consorcio Internacional de Secuenciación del Genoma Humano (octubre de 2004). «Terminación de la secuencia eucromática del genoma humano». Nature . 431 (7011): 931–45. Bibcode :2004Natur.431..931H. doi : 10.1038/nature03001 . PMID 15496913.
- ^ ab Shafee, Thomas; Lowe, Rohan (2017). "Estructura de genes eucariotas y procariotas". WikiJournal of Medicine . 4 (1). doi : 10.15347/wjm/2017.002 . ISSN 2002-4436.
- ^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (julio de 2008). "Mapeo y cuantificación de transcriptomas de mamíferos mediante RNA-Seq". Nature Methods . 5 (7): 621–8. doi :10.1038/nmeth.1226. PMID 18516045. S2CID 205418589.
- ^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (abril de 2013). "Potenciadores: cinco preguntas esenciales". Nature Reviews. Genética . 14 (4): 288–95. doi :10.1038/nrg3458. PMC 4445073. PMID 23503198 .
- ^ Maston GA, Evans SK, Green MR (2006). "Elementos reguladores de la transcripción en el genoma humano". Revisión anual de genómica y genética humana . 7 : 29–59. doi : 10.1146/annurev.genom.7.080505.115623 . PMID 16719718.
- ^ Mignone F, Gissi C, Liuni S, Pesole G (28 de febrero de 2002). "Regiones no traducidas de ARNm". Genome Biology . 3 (3): REVIEWS0004. doi : 10.1186/gb-2002-3-3-reviews0004 . PMC 139023 . PMID 11897027.
- ^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (diciembre de 2012). "Intrones en UTR: por qué deberíamos dejar de ignorarlos". BioEssays . 34 (12): 1025–34. doi : 10.1002/bies.201200073 . PMID 23108796. S2CID 5808466.
- ^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (junio de 2000). "Operones en Escherichia coli: análisis genómicos y predicciones". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (12): 6652–7. Bibcode :2000PNAS...97.6652S. doi : 10.1073/pnas.110147297 . PMC 18690 . PMID 10823905.
- ^ Blumenthal T (noviembre de 2004). "Operones en eucariotas". Briefings in Functional Genomics & Proteomics . 3 (3): 199–211. doi : 10.1093/bfgp/3.3.199 . PMID 15642184.
- ^ Jacob F, Monod J (junio de 1961). "Mecanismos reguladores genéticos en la síntesis de proteínas". Journal of Molecular Biology . 3 (3): 318–56. doi :10.1016/S0022-2836(61)80072-7. PMID 13718526. S2CID 19804795.
- ^ Pozzoli U, Menozzi G, Comi GP, Cagliani R, Bresolin N, Sironi M (enero de 2007). "Tamaño de los intrones en los mamíferos: la complejidad se enfrenta a la economía". Tendencias en genética . 23 (1): 20–24. doi :10.1016/j.tig.2006.10.003. PMID 17070957.
- ^ Marais G, Nouvellet P, Keightley PD, Charlesworth B (mayo de 2005). "Tamaño del intrón y evolución del exón en Drosophila". Genética . 170 (1): 481–485. doi :10.1534/genetics.104.037333. PMC 1449718 . PMID 15781704.
- ^ Kumar A (septiembre de 2009). "Una descripción general de los genes anidados en genomas eucariotas". Eukaryotic Cell . 8 (9): 1321–1329. doi :10.1128/EC.00143-09. PMC 2747821 . PMID 19542305. .
- ^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (junio de 2005). "Asociaciones intercromosómicas entre loci expresados alternativamente". Nature . 435 (7042): 637–645. Bibcode :2005Natur.435..637S. doi :10.1038/nature03574. PMID 15880101. S2CID 1755326.
- ^ Williams A, Spilianakis CG, Flavell RA (abril de 2010). "Asociación intercromosómica y regulación génica en trans". Tendencias en genética . 26 (4): 188–197. doi :10.1016/j.tig.2010.01.007. PMC 2865229 . PMID 20236724.
- ^ Lei Q, Li C, Zuo Z, Huang C, Cheng H, Zhou R (marzo de 2016). "Perspectivas evolutivas sobre el transempalme de ARN en vertebrados". Genome Biology and Evolution . 8 (3): 562–577. doi :10.1093/gbe/evw025. PMC 4824033 . PMID 26966239.
- ^ Wright BW, Molloy MP, Jaschke PR (marzo de 2022). "Superposición de genes en genomas naturales y de ingeniería". Nature Reviews. Genética . 23 (3): 154–168. doi :10.1038/s41576-021-00417-w. PMC 8490965 . PMID 34611352.
- ^ ab Eddy SR (diciembre de 2001). "Genes de ARN no codificantes y el mundo moderno del ARN". Nature Reviews. Genetics . 2 (12): 919–29. doi :10.1038/35103511. PMID 11733745. S2CID 18347629.
- ^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (diciembre de 1961). "Naturaleza general del código genético de las proteínas". Nature . 192 (4809): 1227–32. Bibcode :1961Natur.192.1227C. doi :10.1038/1921227a0. PMID 13882203. S2CID 4276146.
- ^ Crick FH (octubre de 1962). "El código genético". Scientific American . 207 (4). WH Freeman and Company: 66–74. Bibcode :1962SciAm.207d..66C. doi :10.1038/scientificamerican1062-66. PMID 13882204.
- ^ Woodson SA (mayo de 1998). "Resolviendo los problemas: empalme y traducción en bacterias". Genes & Development . 12 (9): 1243–7. doi : 10.1101/gad.12.9.1243 . PMID 9573040.
- ^ Jacob F , Monod J (junio de 1961). "Mecanismos reguladores genéticos en la síntesis de proteínas". Journal of Molecular Biology . 3 (3): 318–56. doi :10.1016/S0022-2836(61)80072-7. PMID 13718526. S2CID 19804795.
- ^ Koonin EV, Dolja VV (enero de 1993). "Evolución y taxonomía de virus de ARN de cadena positiva: implicaciones del análisis comparativo de secuencias de aminoácidos". Critical Reviews in Biochemistry and Molecular Biology . 28 (5): 375–430. doi :10.3109/10409239309078440. PMID 8269709.
- ↑ Domingo E (2001). "Genomas del virus ARN". eLS . doi : 10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
- ^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, et al. (junio de 1996). "Conceptos básicos en la evolución de los virus ARN". Revista FASEB . 10 (8): 859–64. doi : 10.1096/fasebj.10.8.8666162 . PMID 8666162. S2CID 20865732.
- ^ Miko I (2008). "Gregor Mendel y los principios de la herencia". Nature Education Knowledge . SciTable. 1 (1). Nature Publishing Group: 134.
- ^ Chial H (2008). "Genética mendeliana: patrones de herencia y trastornos monogenéticos". Nature Education Knowledge . SciTable. 1 (1). Nature Publishing Group: 63.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (octubre de 1976). "Tasas de elongación del ADN y distribuciones de puntos de crecimiento del fago T4 de tipo salvaje y un mutante ámbar con retraso del ADN". Journal of Molecular Biology . 106 (4): 963–81. doi :10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ ab Lobo I, Shaw K (2008). "Descubrimiento y tipos de ligamiento genético". Nature Education Knowledge . SciTable. 1 (1). Nature Publishing Group: 139.
- ^ Nachman MW, Crowell SL (septiembre de 2000). "Estimación de la tasa de mutación por nucleótido en humanos". Genética . 156 (1): 297–304. doi :10.1093/genetics/156.1.297. PMC 1461236 . PMID 10978293.
- ^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, et al. (abril de 2010). "Análisis de la herencia genética en un cuarteto familiar mediante secuenciación del genoma completo". Science . 328 (5978): 636–9. Bibcode :2010Sci...328..636R. doi :10.1126/science.1186802. PMC 3037280 . PMID 20220176.
- ^ Drake JW, Charlesworth B, Charlesworth D, Crow JF (abril de 1998). "Tasas de mutación espontánea". Genética . 148 (4): 1667–86. doi :10.1093/genetics/148.4.1667. PMC 1460098 . PMID 9560386.
- ^ Pyeritz, Reed E., Bruce R. Korf y Wayne W. Grody, eds. Principios y práctica de la genética y genómica médica de Emery y Rimoin: fundamentos. Academic Press, 2018.
- ^ "¿Qué tipos de mutaciones genéticas son posibles?". Genetics Home Reference . Biblioteca Nacional de Medicina de los Estados Unidos. 11 de mayo de 2015. Consultado el 19 de mayo de 2015 .
- ^ Andrews CA (2010). "La selección natural, la deriva genética y el flujo genético no actúan de forma aislada en las poblaciones naturales". Nature Education Knowledge . SciTable. 3 (10). Nature Publishing Group: 5.
- ^ Patterson C (noviembre de 1988). "Homología en biología clásica y molecular". Biología molecular y evolución . 5 (6): 603–25. doi : 10.1093/oxfordjournals.molbev.a040523 . PMID 3065587.
- ^ Graur D (2016). Evolución molecular y genómica . Sunderland, MA (EE. UU.): Sinauer Associates, Inc. ISBN 9781605354699.
- ^ Graur D (2016). Evolución molecular y genómica . Sunderland, MA (EE. UU.): Sinauer Associates, Inc. ISBN 9781605354699.
- ^ Jensen RA (2001). "Ortólogos y parálogos: tenemos que hacerlo bien". Genome Biology . 2 (8): INTERACTIONS1002. doi : 10.1186/gb-2001-2-8-interactions1002 . PMC 138949 . PMID 11532207.
- ^ Studer RA, Robinson-Rechavi M (mayo de 2009). "¿Hasta qué punto podemos estar seguros de que los ortólogos son similares, pero los parálogos difieren?". Trends in Genetics . 25 (5): 210–6. doi :10.1016/j.tig.2009.03.004. PMID 19368988.
- ^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). "Resolución de la conjetura del ortólogo: los ortólogos tienden a ser débilmente, pero significativamente, más similares en función que los parálogos". PLOS Computational Biology . 8 (5): e1002514. Bibcode :2012PLSCB...8E2514A. doi : 10.1371/journal.pcbi.1002514 . PMC 3355068 . PMID 22615551.

- ^ ab Guerzoni D, McLysaght A (noviembre de 2011). "Orígenes de novo de los genes humanos". PLOS Genetics . 7 (11): e1002381. doi : 10.1371/journal.pgen.1002381 . PMC 3213182 . PMID 22102832.

- ^ Reams AB, Roth JR (febrero de 2015). "Mecanismos de duplicación y amplificación de genes". Cold Spring Harbor Perspectives in Biology . 7 (2): a016592. doi :10.1101/cshperspect.a016592. PMC 4315931 . PMID 25646380.
- ^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (diciembre de 2006). "La evolución de las familias de genes de los mamíferos". PLOS ONE . 1 (1): e85. Bibcode :2006PLoSO...1...85D. doi : 10.1371/journal.pone.0000085 . PMC 1762380 . PMID 17183716.

- ^ Knowles DG, McLysaght A (octubre de 2009). "Origen reciente de novo de genes codificadores de proteínas humanas". Genome Research . 19 (10): 1752–9. doi :10.1101/gr.095026.109. PMC 2765279 . PMID 19726446.
- ^ Wu DD, Irwin DM, Zhang YP (noviembre de 2011). "Origen de novo de los genes codificadores de proteínas humanas". PLOS Genetics . 7 (11): e1002379. doi : 10.1371/journal.pgen.1002379 . PMC 3213175 . PMID 22102831.

- ^ McLysaght A, Guerzoni D (septiembre de 2015). "Nuevos genes a partir de secuencias no codificantes: el papel de los genes codificantes de proteínas de novo en la innovación evolutiva eucariota". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 370 (1678): 20140332. doi :10.1098/rstb.2014.0332. PMC 4571571 . PMID 26323763.
- ^ Neme R, Tautz D (febrero de 2013). "Los patrones filogenéticos de la aparición de nuevos genes respaldan un modelo de evolución frecuente de novo". BMC Genomics . 14 (1): 117. doi : 10.1186/1471-2164-14-117 . PMC 3616865 . PMID 23433480.
- ^ Treangen TJ, Rocha EP (enero de 2011). "La transferencia horizontal, no la duplicación, impulsa la expansión de las familias de proteínas en procariotas". PLOS Genetics . 7 (1): e1001284. doi : 10.1371/journal.pgen.1001284 . PMC 3029252 . PMID 21298028.

- ^ Ochman H, Lawrence JG, Groisman EA (mayo de 2000). "Transferencia lateral de genes y la naturaleza de la innovación bacteriana". Nature . 405 (6784): 299–304. Bibcode :2000Natur.405..299O. doi :10.1038/35012500. PMID 10830951. S2CID 85739173.
- ^ Keeling PJ, Palmer JD (agosto de 2008). "Transferencia horizontal de genes en la evolución eucariota". Nature Reviews. Genética . 9 (8): 605–18. doi :10.1038/nrg2386. PMID 18591983. S2CID 213613.
- ^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, et al. (marzo de 2013). "La transferencia de genes de bacterias y arqueas facilitó la evolución de un eucariota extremófilo". Science . 339 (6124): 1207–10. Bibcode :2013Sci...339.1207S. doi :10.1126/science.1231707. PMID 23471408. S2CID 5502148.
- ^ Ridley, M. (2006). Genoma . Nueva York, NY: Harper Perennial. ISBN 0-06-019497-9
- ^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (abril de 2021). "FINDER: un paquete de software automatizado para anotar genes eucariotas a partir de datos de RNA-Seq y secuencias de proteínas asociadas". BMC Bioinformatics . 44 (9): e89. doi : 10.1186/s12859-021-04120-9 . PMC 8056616 . PMID 33879057.
- ^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). "Ch9-10", Biología molecular del gen, 5.ª ed., Peason Benjamin Cummings; CSHL Press.
- ^ "Integr8 – Estadísticas del genoma de A.thaliana".
- ^ "Entender los conceptos básicos". El Proyecto Genoma Humano . Consultado el 26 de abril de 2015 .
- ^ "WS227 Release Letter". WormBase. 10 de agosto de 2011. Archivado desde el original el 28 de noviembre de 2013. Consultado el 19 de noviembre de 2013 .
- ^ ab Yu J, Hu S, Wang J, Wong GK, Li S, Liu B, et al. (abril de 2002). "Un borrador de secuencia del genoma del arroz (Oryza sativa L. ssp. indica)". Science . 296 (5565): 79–92. Bibcode :2002Sci...296...79Y. doi :10.1126/science.1068037. PMID 11935017. S2CID 208529258.
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, et al. (abril de 1981). "Secuencia y organización del genoma mitocondrial humano". Nature . 290 (5806): 457–65. Bibcode :1981Natur.290..457A. doi :10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, et al. (Marzo de 2000). "La secuencia del genoma de Drosophila melanogaster". Ciencia . 287 (5461): 2185–95. Código bibliográfico : 2000Sci...287.2185.. CiteSeerX 10.1.1.549.8639 . doi : 10.1126/ciencia.287.5461.2185. PMID 10731132.
- ^ Pertea M, Salzberg SL (2010). "Entre un pollo y una uva: estimación del número de genes humanos". Genome Biology . 11 (5): 206. doi : 10.1186/gb-2010-11-5-206 . PMC 2898077 . PMID 20441615.
- ^ Belyi VA, Levine AJ, Skalka AM (diciembre de 2010). "Secuencias de virus de ADN monocatenario ancestrales en genomas de vertebrados: los parvoviridae y circoviridae tienen más de 40 a 50 millones de años". Journal of Virology . 84 (23): 12458–62. doi :10.1128/JVI.01789-10. PMC 2976387 . PMID 20861255.
- ^ Flores R, Di Serio F, Hernández C (febrero de 1997). "Viroides: los genomas no codificantes". Seminarios en Virología . 8 (1): 65–73. doi :10.1006/smvy.1997.0107.
- ^ Zonneveld BJ (2010). "Nuevos poseedores de récords de tamaño máximo del genoma en eudicotiledóneas y monocotiledóneas". Journal of Botany . 2010 : 1–4. doi : 10.1155/2010/527357 .
- ^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (diciembre de 2007). "Hacia la finalización del proteoma de la Tierra". EMBO Reports . 8 (12): 1135–41. doi :10.1038/sj.embor.7401117. PMC 2267224 . PMID 18059312.
- ^ Muller HJ (1966). "El material genético como iniciador y base organizadora de la vida". American Naturalist . 100 (915): 493–517. doi :10.1086/282445. JSTOR 2459205. S2CID 84202145.
- ^ Ohno S (1972). "Tanto ADN "basura" en nuestro genoma". Simposios de Brookhaven sobre biología . 23 : 366–370. PMID 5065367.
- ^ Hatje K, Mühlhausen S, Simm D, Killmar M (2019). "El genoma humano codificador de proteínas: anotación de frutos maduros". BioEssays . 41 (11): 1900066. doi : 10.1002/bies.201900066 . PMID 31544971. S2CID 202732556.
- ^ Schuler GD, Boguski MS , Stewart EA, Stein LD, Gyapay G, Rice K, et al. (octubre de 1996). "Un mapa genético del genoma humano". Science . 274 (5287): 540–6. Bibcode :1996Sci...274..540S. doi :10.1126/science.274.5287.540. PMID 8849440. S2CID 22619.
- ^ Chi KR (octubre de 2016). «El lado oscuro del genoma humano». Nature . 538 (7624): 275–277. Bibcode :2016Natur.538..275C. doi : 10.1038/538275a . PMID 27734873.
- ^ "Ensamblaje humano y anotación de genes". Ensembl . 2022 . Consultado el 28 de febrero de 2023 .
- ^ ab Hutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, et al. (marzo de 2016). "Diseño y síntesis de un genoma bacteriano mínimo". Science . 351 (6280): aad6253. Bibcode :2016Sci...351.....H. doi : 10.1126/science.aad6253 . PMID 27013737.
- ^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, et al. (enero de 2006). "Genes esenciales de una bacteria mínima". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (2): 425–30. Bibcode :2006PNAS..103..425G. doi : 10.1073/pnas.0510013103 . PMC 1324956 . PMID 16407165.
- ^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, et al. (octubre de 2003). "Determinación experimental y análisis a nivel de sistema de genes esenciales en Escherichia coli MG1655". Journal of Bacteriology . 185 (19): 5673–84. doi :10.1128/jb.185.19.5673-5684.2003. PMC 193955 . PMID 13129938.
- ^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, et al. (2006). "Construcción de mutantes knockout de un solo gen en el marco de Escherichia coli K-12: la colección Keio". Biología de sistemas moleculares . 2 : 2006.0008. doi :10.1038/msb4100050. PMC 1681482 . PMID 16738554.
- ^ ab Juhas M, Reuß DR, Zhu B, Commichau FM (noviembre de 2014). "Genes esenciales y fábricas celulares mínimas de Bacillus subtilis y Escherichia coli después de una década de ingeniería genómica". Microbiología . 160 (Pt 11): 2341–2351. doi : 10.1099/mic.0.079376-0 . PMID 25092907.
- ^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (febrero de 2006). "Una mayor comprensión de los genes de enfermedades humanas mediante la comparación con los genes de mantenimiento y otros genes". BMC Genomics . 7 : 31. doi : 10.1186/1471-2164-7-31 . PMC 1397819 . PMID 16504025.

- ^ Georgi B, Voight BF, Bućan M (mayo de 2013). "Del ratón al ser humano: análisis genómico evolutivo de ortólogos humanos de genes esenciales". PLOS Genetics . 9 (5): e1003484. doi : 10.1371/journal.pgen.1003484 . PMC 3649967 . PMID 23675308.

- ^ Eisenberg E, Levanon EY (octubre de 2013). "Human housekeeping genes, revisited" (Los genes humanos de mantenimiento, revisados). Trends in Genetics . 29 (10): 569–74. doi :10.1016/j.tig.2013.05.010. PMID 23810203.
- ^ Amsterdam A, Hopkins N (septiembre de 2006). "Estrategias de mutagénesis en pez cebra para identificar genes implicados en el desarrollo y la enfermedad". Tendencias en genética . 22 (9): 473–8. doi :10.1016/j.tig.2006.06.011. PMID 16844256.
- ^ "Acerca del HGNC". Base de datos de nombres de genes humanos del HGNC . Comité de nomenclatura genética de HUGO. Archivado desde el original el 26 de marzo de 2023. Consultado el 14 de mayo de 2015 .
- ^ Cohen SN, Chang AC (mayo de 1973). "Recircularización y replicación autónoma de un segmento de ADN de factor R cortado en transformantes de Escherichia coli". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 70 (5): 1293–7. Bibcode :1973PNAS...70.1293C. doi : 10.1073/pnas.70.5.1293 . PMC 433482 . PMID 4576014.
- ^ Esvelt KM, Wang HH (2013). "Ingeniería a escala genómica para sistemas y biología sintética". Biología de sistemas moleculares . 9 (1): 641. doi :10.1038/msb.2012.66. PMC 3564264 . PMID 23340847.
- ^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). "Edición de precisión de genomas de animales grandes". Advances in Genetics Volumen 80. Vol. 80. págs. 37–97. doi :10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964 . PMID 23084873.
- ^ Puchta H, Fauser F (2013). "Focalización genética en plantas: 25 años después". Revista Internacional de Biología del Desarrollo . 57 (6–8): 629–37. doi : 10.1387/ijdb.130194hp . PMID 24166445.
- ^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (noviembre de 2013). "Ingeniería genómica mediante el sistema CRISPR-Cas9". Nature Protocols . 8 (11): 2281–2308. doi :10.1038/nprot.2013.143. PMC 3969860 . PMID 24157548.
- ^ Kittleson JT, Wu GC, Anderson JC (agosto de 2012). "Éxitos y fracasos en la ingeniería genética modular". Current Opinion in Chemical Biology . 16 (3–4): 329–36. doi :10.1016/j.cbpa.2012.06.009. PMID 22818777.
- ^ Berg P, Mertz JE (enero de 2010). "Reflexiones personales sobre los orígenes y el surgimiento de la tecnología del ADN recombinante". Genética . 184 (1): 9–17. doi :10.1534/genetics.109.112144. PMC 2815933 . PMID 20061565.
- ^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, et al. (septiembre de 2004). "El proyecto del ratón knock-out". Nature Genetics . 36 (9): 921–4. doi :10.1038/ng0904-921. PMC 2716027 . PMID 15340423.
- ^ Guan C, Ye C, Yang X, Gao J (febrero de 2010). "Una revisión de los esfuerzos actuales de eliminación de genes en ratones a gran escala". Genesis . 48 (2): 73–85. doi :10.1002/dvg.20594. PMID 20095055. S2CID 34470273.
- ^ Deng C (octubre de 2007). "En celebración del Premio Nobel del Dr. Mario R. Capecchi". Revista Internacional de Ciencias Biológicas . 3 (7): 417–9. doi :10.7150/ijbs.3.417. PMC 2043165 . PMID 17998949.
Fuentes
- Libro de texto principal
- Alberts B , Johnson A, Lewis J , Raff M , Roberts K, Walter P (2002). Biología molecular de la célula (cuarta edición). Nueva York: Garland Science. ISBN 978-0-8153-3218-3.– Un libro de texto de biología molecular disponible gratis en línea a través de NCBI Bookshelf.

- Glosario
- Cap 1: Células y genomas
- 1.1: Las características universales de las células en la Tierra
- Capítulo 2: Química celular y biosíntesis
- 2.1: Los componentes químicos de una célula
- Cap. 3: Proteínas
- Cap. 4: ADN y cromosomas
- 4.1: La estructura y función del ADN
- 4.2: ADN cromosómico y su empaquetamiento en la fibra de cromatina
- Capítulo 5: Replicación, reparación y recombinación del ADN
- 5.2: Mecanismos de replicación del ADN
- 5.4: Reparación del ADN
- 5.5: Recombinación general
- Capítulo 6: Cómo leen las células el genoma: del ADN a las proteínas
- 6.1: ADN a ARN
- 6.2: ARN a proteína
- Capítulo 7: Control de la expresión genética
- 7.1: Una visión general del control genético
- 7.2: Motivos de unión al ADN en proteínas reguladoras de genes
- 7.3: Cómo funcionan los interruptores genéticos
- 7.5: Controles postranscripcionales
- 7.6: Cómo evolucionan los genomas
- Cap. 14: Conversión de energía: mitocondrias y cloroplastos
- 14.4: Los sistemas genéticos de las mitocondrias y los plástidos
- Capítulo 18: La mecánica de la división celular
- 18.1: Una descripción general de la fase M
- 18.2: Mitosis
- Capítulo 20: Células germinales y fertilización
- 20.2: Meiosis
Lectura adicional
- Watson JD , Baker TA , Bell SP, Gann A, Levine M , Losick R (2013). Biología molecular del gen (7.ª ed.). Benjamin Cummings. ISBN 978-0-321-90537-6.
- Dawkins R (1990). El gen egoísta . Oxford University Press. ISBN 978-0-19-286092-7.
- Ridley M (1999). Genoma: La autobiografía de una especie en 23 capítulos . Fourth Estate. ISBN 978-0-00-763573-3.
- Brown T (2002). Genomas (2.ª ed.). Nueva York: Wiley-Liss. ISBN 978-0-471-25046-3. Número de identificación personal 20821850.
Enlaces externos
- Base de datos de toxicogenómica comparativa
- El ADN desde el principio: una introducción a los genes y al ADN
- Gene – una base de datos de genes con capacidad de búsqueda
- Genes – una revista de acceso abierto
- IDconverter: convierte los identificadores de genes entre bases de datos públicas
- iHOP – Información vinculada a proteínas
- TranscriptomeBrowser: análisis del perfil de expresión genética
- La utilidad de denominación de proteínas, una base de datos para identificar y corregir nombres de genes deficientes
- IMPC (Consorcio Internacional de Fenotipado de Ratones): Enciclopedia de la función genética de los mamíferos
- Global Genes Project: organización líder sin fines de lucro que apoya a personas que viven con enfermedades genéticas
- Explorador de hilos codificados, Naturaleza
- Caracterización de regiones intergénicas y definición de genes, Nature





