Receptor tipo Toll
| Receptor tipo Toll | |
|---|---|
 La región de repetición curva rica en leucina de los receptores tipo Toll, representada aquí por TLR3 | |
| Identificadores | |
| Símbolo | Receptor tipo Toll |
| Membranoma | 7 |
| PIRSF037595 | |
Los receptores tipo Toll ( TLR ) son una clase de proteínas que desempeñan un papel clave en el sistema inmunológico innato . Son receptores de un solo segmento que generalmente se expresan en células centinela, como macrófagos y células dendríticas , que reconocen moléculas estructuralmente conservadas derivadas de microbios . Una vez que estos microbios han alcanzado barreras físicas como la piel o la mucosa del tracto intestinal , son reconocidos por los TLR, que activan las respuestas de las células inmunes . Los TLR incluyen TLR1 , TLR2 , TLR3 , TLR4 , TLR5 , TLR6 , TLR7 , TLR8 , TLR9 , TLR10 , TLR11 , TLR12 y TLR13. Los humanos carecen de genes para TLR11, TLR12 y TLR13 [1] y los ratones carecen de un gen funcional para TLR10. [2] Los receptores TLR1, TLR2, TLR4, TLR5, TLR6 y TLR10 se encuentran en la membrana celular , mientras que TLR3, TLR7, TLR8 y TLR9 se encuentran en vesículas intracelulares (porque son sensores de ácidos nucleicos ). [3]
Los TLR recibieron su nombre por su similitud con la proteína codificada por el gen toll. [4]
Función
La capacidad del sistema inmunológico para reconocer moléculas que son ampliamente compartidas por patógenos se debe, en parte, a la presencia de receptores inmunes llamados receptores tipo Toll (TLR) que se expresan en las membranas de los leucocitos , incluidas las células dendríticas , los macrófagos , las células asesinas naturales , las células de la inmunidad adaptativa ( células T y B ) y las células no inmunes ( células epiteliales y endoteliales y fibroblastos ). [5]
La unión de ligandos (ya sea en forma de adyuvante utilizado en vacunaciones o en forma de fracciones invasivas durante períodos de infección natural) al TLR marca los eventos moleculares clave que finalmente conducen a respuestas inmunes innatas y al desarrollo de inmunidad adquirida específica para el antígeno. [6] [7]
Tras la activación, los TLR reclutan proteínas adaptadoras (proteínas que median otras interacciones proteína-proteína) dentro del citosol de la célula inmunitaria para propagar la vía de transducción de señales inducida por antígenos . Estas proteínas reclutadas son entonces responsables de la activación posterior de otras proteínas posteriores , incluidas las proteína quinasas (IKKi, IRAK1 , IRAK4 y TBK1 ) que amplifican aún más la señal y, en última instancia, conducen a la regulación positiva o la supresión de los genes que orquestan las respuestas inflamatorias y otros eventos transcripcionales . Algunos de estos eventos conducen a la producción, proliferación y supervivencia de citocinas , mientras que otros conducen a una mayor inmunidad adaptativa. [7] Si el ligando es un factor bacteriano, el patógeno podría ser fagocitado y digerido, y sus antígenos presentados a las células T CD4+ . En el caso de un factor viral, la célula infectada puede detener su síntesis de proteínas y puede sufrir muerte celular programada ( apoptosis ). Las células inmunes que han detectado un virus también pueden liberar factores antivirales como los interferones .
También se ha demostrado que los receptores tipo Toll son un vínculo importante entre la inmunidad innata y adaptativa a través de su presencia en las células dendríticas . [8] La flagelina , un ligando de TLR5, induce la secreción de citocinas al interactuar con TLR5 en las células T humanas. [8]
Superfamilia

Los TLR son un tipo de receptor de reconocimiento de patrones (PRR) y reconocen moléculas que son ampliamente compartidas por patógenos pero que se pueden distinguir de las moléculas del huésped, denominadas colectivamente patrones moleculares asociados a patógenos (PAMP). Además del reconocimiento de PAMP exógenos, los TLR también pueden unirse a patrones moleculares asociados a daños (DAMP) endógenos, como proteínas de choque térmico (HSP) o componentes de la membrana plasmática. [9] Los TLR junto con los receptores de interleucina-1 forman una superfamilia de receptores , conocida como la "superfamilia del receptor de interleucina-1 / receptor tipo Toll"; todos los miembros de esta familia tienen en común un dominio denominado TIR (receptor Toll-IL-1).
Existen tres subgrupos de dominios TIR. Las proteínas con dominios TIR del subgrupo 1 son receptores de interleucinas producidas por macrófagos , monocitos y células dendríticas y todas tienen dominios de inmunoglobulina (Ig) extracelulares. Las proteínas con dominios TIR del subgrupo 2 son TLR clásicos y se unen directa o indirectamente a moléculas de origen microbiano. Un tercer subgrupo de proteínas que contienen dominios TIR consiste en proteínas adaptadoras que son exclusivamente citosólicas y median la señalización de las proteínas de los subgrupos 1 y 2.
Familia extendida
En esta sección falta información sobre el TLR de los coanoflagelados (pmid29848444). ( Diciembre de 2021 ) |
Los TLR están presentes tanto en vertebrados como en invertebrados . Los componentes básicos moleculares de los TLR están presentes en bacterias y plantas, y se sabe que los receptores de reconocimiento de patrones de las plantas son necesarios para la defensa del huésped contra las infecciones. Por lo tanto, los TLR parecen ser uno de los componentes más antiguos y conservados del sistema inmunitario .
En los últimos años, los TLR también se han identificado en el sistema nervioso de los mamíferos. Se han detectado miembros de la familia TLR en la glía, las neuronas y las células progenitoras neuronales, en las que regulan la decisión sobre el destino celular. [10]
Se ha estimado que la mayoría de las especies de mamíferos tienen entre diez y quince tipos de receptores tipo Toll. Se han identificado trece TLR (denominados simplemente TLR1 a TLR13) en humanos y ratones juntos, y se han encontrado formas equivalentes de muchos de estos en otras especies de mamíferos. [11] [12] [13] Sin embargo, los equivalentes de ciertos TLR encontrados en humanos no están presentes en todos los mamíferos. Por ejemplo, un gen que codifica una proteína análoga a TLR10 en humanos está presente en ratones , pero parece haber sido dañado en algún momento en el pasado por un retrovirus . Por otro lado, los ratones expresan TLR 11, 12 y 13, ninguno de los cuales está representado en humanos. Otros mamíferos pueden expresar TLR que no se encuentran en humanos. Otras especies no mamíferas pueden tener TLR distintos de los mamíferos, como lo demuestra el TLR14 anti-pared celular, que se encuentra en el pez globo Takifugu . [14] Esto puede complicar el proceso de utilizar animales experimentales como modelos de inmunidad innata humana.
Los TLR de vertebrados se dividen por similitud en las familias TLR 1/2/6/10/14/15, TLR 3, TLR 4, TLR 5, TLR 7/8/9 y TLR 11/12/13/16/21/22/23. [14]
TLR enDrosophilainmunidad

La participación de la señalización toll en la inmunidad se demostró por primera vez en la mosca de la fruta, Drosophila melanogaster . [19] Las moscas de la fruta solo tienen respuestas inmunes innatas, lo que permite que los estudios eviten la interferencia de los mecanismos inmunes adaptativos en la transducción de señales. La respuesta de la mosca a una infección fúngica o bacteriana se produce a través de dos cascadas de señalización distintas, una de las cuales es la vía toll y la otra es la vía de inmunodeficiencia . La vía toll es similar a la señalización TLR de mamíferos, pero a diferencia de los TLR de mamíferos, toll no se activa directamente por patrones moleculares asociados a patógenos ( PAMP ). Su ectodominio receptor reconoce la forma escindida de la citocina spätzle, que se secreta en la hemolinfa como un precursor dimérico inactivo. El receptor toll comparte el dominio TIR citoplasmático con los TLR de mamíferos, pero el ectodominio y la cola intracitoplasmática son diferentes. Esta diferencia podría reflejar una función de estos receptores como receptores de citocinas en lugar de PRR .
La vía toll se activa por diferentes estímulos, como bacterias grampositivas , hongos y factores de virulencia . [17] [20] Primero, la enzima procesadora de Spätzle (SPE) se activa en respuesta a la infección y escinde el spätzle ( spz ). El spätzle escindido luego se une al receptor toll y reticula sus ectodominios. Esto desencadena cambios conformacionales en el receptor que resultan en señalización a través de toll. A partir de este punto, la cascada de señalización es muy similar a la señalización de mamíferos a través de TLR. El complejo de señalización inducida por toll (TICS) está compuesto por MyD88 , Tube y Pelle (el ortólogo de IRAK de mamíferos). La señal de TICS luego se transduce a Cactus (homólogo del IκB de mamífero ), el Cactus fosforilado se poliubiquitina y se degrada, lo que permite la translocación nuclear de DIF (factor de inmunidad relacionado con el dorsal; un homólogo del NF-κB de mamífero ) y la inducción de la transcripción de genes para péptidos antimicrobianos (AMP) como la drosomicina . [21]
La Drosophila tiene un total de 9 genes de la familia Toll y 6 de la familia Spz que interactúan entre sí en diferentes grados. [22]
TLR2
TLR2 también ha sido designado como CD282 (grupo de diferenciación 282).
TLR3
TLR3 no utiliza la vía dependiente de MyD88. Su ligando es el ARN bicatenario retroviral ( dsRNA ), que activa la vía de señalización dependiente de TRIF . Para explorar el papel de esta vía en la reprogramación retroviral, se prepararon técnicas de knock down de TLR3 o TRIF, y los resultados mostraron que solo se requiere la vía TLR3 para la inducción completa de la expresión del gen objetivo por el vector de expresión del retrovirus. Esta expresión retroviral de cuatro factores de transcripción ( Oct4 , Sox2 , Klf4 y c-Myc ; OSKM) induce pluripotencia en células somáticas. Esto está respaldado por un estudio que muestra que la eficiencia y la cantidad de generación de iPSC humanas, utilizando vectores retrovirales, se reduce por el knockdown de la vía con inhibidores de péptidos o el knockdown de shRNA de TLR3 o su proteína adaptadora TRIF. En conjunto, la estimulación de TLR3 provoca grandes cambios en la remodelación de la cromatina y la reprogramación nuclear, y la activación de vías inflamatorias es necesaria para estos cambios, la inducción de genes de pluripotencia y la generación de colonias de células madre pluripotentes inducidas humanas (iPSC). [23]
TLR11
Como se señaló anteriormente, las células humanas no expresan TLR11 , pero las células de ratones sí. El TLR11 específico de ratón reconoce E. coli uropatógeno y el parásito apicomplejo Toxoplasma gondii . Con Toxoplasma su ligando es la proteína profilina y el ligando para E. coli es la flagelina . La flagelina del enteropatógeno Salmonella también es reconocida por TLR11. [24]
Como el TLR11 del ratón es capaz de reconocer eficazmente a la Salmonella , los ratones normales no se infectan con la Salmonella Typhi oral , que causa gastroenteritis transmitida por los alimentos y el agua y fiebre tifoidea en los seres humanos. Los ratones knock out deficientes en TLR11 , por otro lado, se infectan de manera eficiente. Como resultado, este ratón knock out puede actuar como un modelo de enfermedad de la fiebre tifoidea humana. [25]
Resumen de los TLR de mamíferos conocidos
Los receptores tipo Toll se unen y se activan mediante diferentes ligandos, que, a su vez, se encuentran en diferentes tipos de organismos o estructuras. También tienen diferentes adaptadores para responder a la activación y se encuentran a veces en la superficie celular y a veces en compartimentos celulares internos . [26] Además, se expresan en diferentes tipos de leucocitos u otros tipos de células :
| Receptor | Ligando(s) [27] | Ubicación del ligando [27] | Adaptador(es) | Ubicación | Tipos de células [27] |
|---|---|---|---|---|---|
| Límite de refracción 1 | lipopéptidos de triacilo múltiples | Lipoproteína bacteriana | Mi D88 /MAL | superficie celular |
|
| TLR 2 | glicolípidos múltiples | Peptidoglicanos bacterianos | Mi D88/MAL | superficie celular |
|
| lipopéptidos y proteolípidos múltiples | Peptidoglicanos bacterianos | ||||
| ácido lipoteicoico | Bacterias grampositivas | ||||
| HSP70 | Células huésped | ||||
| zimosán ( beta-glucano ) | Hongos | ||||
| Numerosos otros | |||||
| TLR 3 | ARN bicatenario , poli I:C | virus | TRIF | compartimento celular |
|
| TLR 4 | lipopolisacárido | Bacterias gramnegativas | Mi D88/MAL/ TRIF /TRAM | superficie celular |
|
| Varias proteínas de choque térmico | Bacterias y células huésped | ||||
| fibrinógeno | células huésped | ||||
| fragmentos de sulfato de heparán | células huésped | ||||
| fragmentos de ácido hialurónico | células huésped | ||||
| níquel [32] | |||||
| Diversos fármacos opioides | |||||
| TLR 5 | Flagelina bacteriana | Bacteria | MiD88 | superficie celular |
|
| Perfilina [33] | Toxoplasma gondii | ||||
| TLR6 (Respuesta de emergencia 6) | lipopéptidos diacílicos múltiples | Micoplasma | Mi D88/MAL | superficie celular |
|
| TLR7 (Respuesta rápida 7) | imidazoquinolina | pequeños compuestos sintéticos | MiD88 | compartimento celular |
|
| loxoribina (un análogo de la guanosina ) | |||||
| bropirimina | |||||
| Resquimod | |||||
| ARN monocatenario | Virus de ARN | ||||
| TLR8 | Compuestos sintéticos pequeños; ARN viral monocatenario, ARN bacteriano fagocitado (24) | MiD88 | compartimento celular |
| |
| TLR9 (Respuesta rápida 9) | ADN oligodesoxirribonucleico CpG no metilado | Bacterias, virus ADN | MiD88 | compartimento celular |
|
| TLR10 | lipopéptidos triacilados [35] | desconocido | superficie celular |
| |
| TLR11 | Perfilina | Toxoplasma gondii [39] | MiD88 | compartimento celular [40] |
|
| Flagelina | Bacterias ( E. coli , Salmonella ) [24] | ||||
| TLR 12 | Perfilina | Toxoplasma gondii [41] | MiD88 | compartimento celular |
|
| LRT 13 [43] [44] | secuencia de ARN ribosómico bacteriano "CGGAAAGACC" (pero no la versión metilada) [45] | Virus, bacteria | Mi D88, TAK-1 | compartimento celular |
|
Ligandos
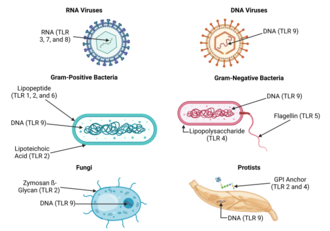
Debido a la especificidad de los receptores tipo toll (y otros receptores inmunes innatos), no se pueden cambiar fácilmente en el curso de la evolución, estos receptores reconocen moléculas que están constantemente asociadas con amenazas (es decir, patógenos o estrés celular) y son altamente específicos para estas amenazas (es decir, no se pueden confundir con moléculas propias que normalmente se expresan en condiciones fisiológicas). Se cree que las moléculas asociadas a patógenos que cumplen con este requisito son fundamentales para la función del patógeno y difíciles de cambiar a través de la mutación; se dice que están conservadas evolutivamente. Las características algo conservadas en los patógenos incluyen lipopolisacáridos de superficie celular bacteriana (LPS), lipoproteínas , lipopéptidos y lipoarabinomanano ; proteínas como la flagelina de los flagelos bacterianos; ARN bicatenario de virus; o las islas CpG no metiladas del ADN bacteriano y viral ; y también de las islas CpG encontradas en los promotores del ADN eucariota; así como ciertas otras moléculas de ARN y ADN. Como los ligandos TLR están presentes en la mayoría de los patógenos, también pueden estar presentes en las vacunas derivadas de patógenos (por ejemplo, vacunas MMR, influenza, polio); la mayoría de las vacunas disponibles comercialmente se han evaluado por la capacidad inherente de sus ligandos TLR para activar distintos subconjuntos de células inmunes. [46] [47] Para la mayoría de los TLR, la especificidad del reconocimiento del ligando ahora se ha establecido mediante la selección de genes (también conocida como "inactivación de genes"): una técnica por la cual se pueden eliminar genes individuales de forma selectiva en ratones. [48] [49] Consulte la tabla anterior para obtener un resumen de los ligandos TLR conocidos.
Ligandos endógenos
La respuesta inflamatoria estereotipada provocada por la activación del receptor tipo Toll ha dado lugar a la especulación de que los activadores endógenos de los receptores tipo Toll podrían participar en las enfermedades autoinmunes. Se ha sospechado que los TLR se unen a las moléculas del huésped, incluido el fibrinógeno (que interviene en la coagulación sanguínea ), las proteínas de choque térmico (HSP), HMGB1 , los componentes de la matriz extracelular y el ADN propio (normalmente se degrada por nucleasas, pero en condiciones inflamatorias y autoinmunes puede formar un complejo con proteínas endógenas, volverse resistente a estas nucleasas y obtener acceso a los TLR endosómicos como TLR7 o TLR9). Estos ligandos endógenos se producen habitualmente como resultado de la muerte celular no fisiológica. [50]
Señalización
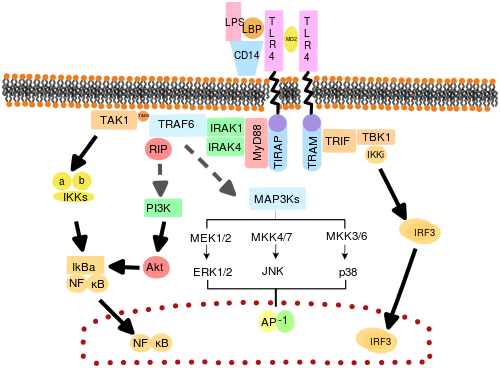
Se cree que los TLR funcionan como dímeros . Aunque la mayoría de los TLR parecen funcionar como homodímeros , TLR2 forma heterodímeros con TLR1 o TLR6, cada dímero tiene una especificidad de ligando diferente. Los TLR también pueden depender de otros correceptores para una sensibilidad completa al ligando, como en el caso del reconocimiento de LPS por parte de TLR4 , que requiere MD-2. Se sabe que CD14 y la proteína de unión a LPS ( LBP ) facilitan la presentación de LPS a MD-2.
Un conjunto de TLR endosómicos que comprende TLR3, TLR7, TLR8 y TLR9 reconocen ácidos nucleicos derivados de virus, así como ácidos nucleicos endógenos en el contexto de eventos patogénicos. La activación de estos receptores conduce a la producción de citocinas inflamatorias , así como de interferones de tipo I ( interferón tipo I ) para ayudar a combatir la infección viral.
También se han identificado las proteínas adaptadoras y las quinasas que median la señalización de TLR. Además, se ha utilizado la mutagénesis de línea germinal aleatoria con ENU para descifrar las vías de señalización de TLR. Cuando se activan, los TLR reclutan moléculas adaptadoras dentro del citoplasma de las células para propagar una señal. Se sabe que cuatro moléculas adaptadoras están involucradas en la señalización. Estas proteínas se conocen como MyD88 , TIRAP (también llamada Mal), TRIF y TRAM (molécula adaptadora relacionada con TRIF). [51] [52] [53]
La señalización de TLR se divide en dos vías de señalización distintas, la vía dependiente de MyD88 y la vía dependiente de TRIF.
Vía dependiente de MyD88
La respuesta dependiente de MyD88 ocurre en la dimerización de los TLR y es utilizada por todos los TLR excepto TLR3. Su efecto principal es la activación de NFκB y la proteína quinasa activada por mitógenos . La unión del ligando y el cambio conformacional que ocurre en el receptor reclutan la proteína adaptadora MyD88, un miembro de la familia TIR . MyD88 luego recluta IRAK4 , IRAK1 e IRAK2 . Las quinasas IRAK luego fosforilan y activan la proteína TRAF6 , que a su vez poliubiquina la proteína TAK1, así como a sí misma para facilitar la unión a IKK-β . Al unirse, TAK1 fosforila IKK-β, que luego fosforila IκB causando su degradación y permitiendo que NFκB se difunda en el núcleo celular y active la transcripción y la consiguiente inducción de citocinas inflamatorias. [50]
Vía dependiente de TRIF
Tanto TLR3 como TLR4 utilizan la vía dependiente de TRIF, que se activa mediante dsRNA y LPS, respectivamente. En el caso de TLR3, dsRNA activa el receptor, reclutando el adaptador TRIF . TRIF activa las quinasas TBK1 y RIPK1 , lo que crea una rama en la vía de señalización. El complejo de señalización TRIF/TBK1 fosforila IRF3 , lo que permite su translocación al núcleo y la producción de interferón tipo I. Mientras tanto, la activación de RIPK1 provoca la poliubiquitinación y la activación de la transcripción de TAK1 y NFκB de la misma manera que la vía dependiente de MyD88. [50]
La señalización de TLR conduce en última instancia a la inducción o supresión de genes que orquestan la respuesta inflamatoria. En total, miles de genes son activados por la señalización de TLR y, en conjunto, los TLR constituyen una de las vías de acceso más pleiotrópicas y, al mismo tiempo, más reguladas para la modulación genética.
TLR4 es el único TLR que utiliza los cuatro adaptadores. El complejo formado por TLR4, MD2 y LPS recluta los adaptadores que contienen el dominio TIR TIRAP y MyD88 y, por lo tanto, inicia la activación de NFκB (fase temprana) y MAPK. El complejo TLR4-MD2-LPS luego experimenta endocitosis y en el endosoma forma un complejo de señalización con los adaptadores TRAM y TRIF. Esta vía dependiente de TRIF nuevamente conduce a la activación de IRF3 y la producción de interferones de tipo I, pero también activa la activación de NFκB en fase tardía. Tanto la activación de fase tardía como la temprana de NFκB son necesarias para la producción de citocinas inflamatorias. [50]
Relevancia médica
El imiquimod (utilizado principalmente en dermatología ) es un agonista de TLR7, y su sucesor, el resiquimod , es un agonista de TLR7 y TLR8. [54] Recientemente, se ha explorado el resiquimod como agente para la inmunoterapia contra el cáncer, [55] actuando a través de la estimulación de los macrófagos asociados a los tumores.
Varios ligandos TLR se encuentran en desarrollo clínico o se están probando en modelos animales como adyuvantes de vacunas , [56] con el primer uso clínico en humanos en una vacuna recombinante contra el herpes zóster en 2017, que contiene un componente de lípido A monofosforilado.
Se han informado los niveles de expresión del ARN mensajero TLR7 en animales lecheros en un brote natural de fiebre aftosa. [57]
Se ha demostrado que el TLR4 es importante para los efectos secundarios a largo plazo de los opioides . Su activación conduce a la liberación descendente de moduladores inflamatorios, incluidos TNF-α e IL-1β , y se cree que la liberación constante de bajo nivel de estos moduladores reduce la eficacia del tratamiento con fármacos opioides con el tiempo, y está involucrada en la tolerancia a los opioides, [58] [59] hiperalgesia y alodinia . [60] [61] La activación de TLR4 inducida por morfina atenúa la supresión del dolor por opioides y mejora el desarrollo de tolerancia y adicción a opioides , abuso de drogas y otros efectos secundarios negativos como depresión respiratoria e hiperalgesia. [62] Se ha demostrado que los medicamentos que bloquean la acción de TNF-α o IL-1β aumentan los efectos analgésicos de los opioides y reducen el desarrollo de tolerancia y otros efectos secundarios, [63] [64] y esto también se ha demostrado con medicamentos que bloquean el propio TLR4.
Los enantiómeros "no naturales" de los fármacos opiáceos, como la (+)-morfina y la (+)-naloxona, carecen de afinidad por los receptores opiáceos, pero aún así producen la misma actividad en TLR4 que sus enantiómeros "normales". [65] [66] Por lo tanto, los enantiómeros "no naturales" de los opiáceos, como la (+)-naloxona, se pueden utilizar para bloquear la actividad TLR4 de los fármacos analgésicos opiáceos sin tener ninguna afinidad por el receptor μ-opioide [67] [66] [68]
Descubrimiento
Cuando se reconoció por primera vez a los microbios como la causa de las enfermedades infecciosas, quedó inmediatamente claro que los organismos multicelulares debían ser capaces de reconocerlos cuando se infectaban y, por lo tanto, capaces de reconocer moléculas exclusivas de los microbios. Una gran cantidad de literatura, que abarca la mayor parte del siglo pasado, da fe de la búsqueda de las moléculas clave y sus receptores. Hace más de 100 años, Richard Pfeiffer , un estudiante de Robert Koch , acuñó el término " endotoxina " para describir una sustancia producida por bacterias gramnegativas que podía provocar fiebre y shock en animales de experimentación . En las décadas siguientes, la endotoxina se caracterizó químicamente y se identificó como un lipopolisacárido (LPS) producido por la mayoría de las bacterias gramnegativas. Este lipopolisacárido es una parte integral de la membrana gramnegativa y se libera tras la destrucción de la bacteria. Se demostró a su vez que otras moléculas ( lipopéptidos bacterianos , flagelina y ADN no metilado ) provocan respuestas del huésped que normalmente son protectoras. Sin embargo, estas respuestas pueden ser perjudiciales si son excesivamente prolongadas o intensas. Lógicamente, se deducía que debían existir receptores para dichas moléculas, capaces de alertar al huésped de la presencia de una infección, pero estos no se encontraron durante muchos años. Los receptores tipo Toll se cuentan ahora entre las moléculas clave que alertan al sistema inmunitario de la presencia de infecciones microbianas.
El miembro prototípico de la familia, el receptor toll ( P08953 ; Tl) en la mosca de la fruta Drosophila melanogaster , fue descubierto en 1985 por los premios Nobel de 1995 Christiane Nüsslein-Volhard y Eric Wieschaus y colegas. Era conocido por su función de desarrollo en la embriogénesis al establecer el eje dorsal - ventral . Recibió su nombre de la exclamación de 1985 de Christiane Nüsslein-Volhard, " Das ist ja toll! " ("¡Eso es increíble!"), en referencia a la porción ventral subdesarrollada de una larva de mosca de la fruta. [4] Fue clonado por el laboratorio de Kathryn Anderson en 1988. [69] En 1996, Jules A. Hoffmann y sus colegas descubrieron que toll tenía un papel esencial en la inmunidad de la mosca a la infección por hongos , que lograba activando la síntesis de péptidos antimicrobianos. [19]
El primer receptor tipo toll humano del que se informó fue descrito por Nomura y colegas en 1994, [70] y mapeado en un cromosoma por Taguchi y colegas en 1996. [71] Debido a que la función inmunológica del toll en Drosophila no se conocía en ese momento, se asumió que el TIL (ahora conocido como TLR1) podría participar en el desarrollo de los mamíferos. Sin embargo, en 1991 (antes del descubrimiento del TIL) se observó que una molécula con un papel claro en la función inmunológica en mamíferos, el receptor de interleucina-1 (IL-1), también tenía homología con el toll de Drosophila; las porciones citoplasmáticas de ambas moléculas eran similares. [72]
En 1997, Charles Janeway y Ruslan Medzhitov demostraron que un receptor tipo Toll, ahora conocido como TLR4, podría, cuando se liga artificialmente utilizando anticuerpos, inducir la activación de ciertos genes necesarios para iniciar una respuesta inmune adaptativa . [7] La función de TLR 4 como receptor sensor de LPS fue descubierta por Bruce A. Beutler y colegas. [73] Estos trabajadores utilizaron la clonación posicional para demostrar que los ratones que no podían responder al LPS tenían mutaciones que abolían la función de TLR4. Esto identificó a TLR4 como uno de los componentes clave del receptor de LPS.

A su vez, los demás genes TLR fueron eliminados en ratones mediante técnicas de selección de genes, en gran medida en el laboratorio de Shizuo Akira y sus colegas. Ahora se cree que cada TLR detecta un conjunto discreto de moléculas (algunas de origen microbiano y otras productos de daño celular) y señala la presencia de infecciones. [74]
Los homólogos vegetales de toll fueron descubiertos por Pamela Ronald en 1995 (arroz XA21) [75] y Thomas Boller en 2000 ( Arabidopsis FLS2). [76]
En 2011, Beutler y Hoffmann recibieron el Premio Nobel de Medicina o Fisiología por su trabajo. [77] Hoffmann y Akira recibieron el Premio Internacional Gairdner de Canadá en 2011. [78]
Notas y referencias
- ^ Mahla RS, Reddy MC, Prasad DV, Kumar H (septiembre de 2013). "Endulzar los PAMP: papel de los PAMP complejos con azúcar en la inmunidad innata y la biología de las vacunas". Frontiers in Immunology . 4 : 248. doi : 10.3389/fimmu.2013.00248 . PMC 3759294 . PMID 24032031.
- ^ Fore, Faith; Indriputri, Cut; Mamutse, Janet; Nugraha, Jusak (2020). "TLR10 y sus propiedades antiinflamatorias únicas y su uso potencial como objetivo en la terapéutica". Red Inmunológica . 20 (3): e21. doi :10.4110/in.2020.20.e21. ISSN 1598-2629. PMC 7327153 . PMID 32655969.
- ^ Kemball CC, Alirezaei M, Whitton JL (2010). "Coxsackievirus tipo B y sus interacciones con los sistemas inmunitarios innato y adaptativo". Future Microbiology . 5 (9): 1329–47. doi :10.2217/fmb.10.101. PMC 3045535 . PMID 20860480.
- ^ ab Hansson GK, Edfeldt K (junio de 2005). "Peaje que se debe pagar en la puerta de entrada a la pared vascular". Arteriosclerosis, trombosis y biología vascular . 25 (6): 1085–7. doi : 10.1161/01.ATV.0000168894.43759.47 . PMID 15923538.
- ^ Delneste Y, Beauvillain C, Jeannin P (enero de 2007). "[Inmunidad innata: estructura y función de los TLR]". Médecine/Sciences . 23 (1): 67–73. doi : 10.1051/medsci/200723167 . PMID 17212934.
- ^ Takeda K, Akira S (enero de 2005). "Receptores tipo Toll en la inmunidad innata". Inmunología internacional . 17 (1): 1–14. doi :10.1093/intimm/dxh186. PMID 15585605.
- ^ abc Medzhitov R, Preston-Hurlburt P, Janeway CA (julio de 1997). "Un homólogo humano de la proteína Toll de Drosophila señala la activación de la inmunidad adaptativa". Nature . 388 (6640): 394–7. Bibcode :1997Natur.388..394M. doi : 10.1038/41131 . PMID 9237759.
- ^ ab Sharma N, Akhade AS, Qadri A (abril de 2013). "La esfingosina-1-fosfato suprime la secreción de CXCL8 inducida por TLR de las células T humanas". Journal of Leukocyte Biology . 93 (4): 521–8. doi :10.1189/jlb.0712328. PMID 23345392.
- ^ Sameer AS, Nissar S (2021). "Receptores tipo Toll (TLR): estructura, funciones, señalización y papel de sus polimorfismos en la susceptibilidad al cáncer colorrectal". Biomed Res Int . 2021 : 1157023. doi : 10.1155/2021/1157023 . PMC 8452412. PMID 34552981 .
- ^ Rolls A, Shechter R, London A, Ziv Y, Ronen A, Levy R, Schwartz M (septiembre de 2007). "Los receptores tipo Toll modulan la neurogénesis del hipocampo adulto". Nature Cell Biology . 9 (9): 1081–8. doi :10.1038/ncb1629. PMID 17704767. S2CID 12517461.
- ^ Du X, Poltorak A, Wei Y, Beutler B (septiembre de 2000). "Tres nuevos receptores tipo toll en mamíferos: estructura, expresión y evolución de los genes". European Cytokine Network . 11 (3): 362–71. PMID 11022119.
- ^ Chuang TH, Ulevitch RJ (septiembre de 2000). "Clonación y caracterización de una subfamilia de receptores humanos tipo Toll: hTLR7, hTLR8 y hTLR9". European Cytokine Network . 11 (3): 372–8. PMID 11022120.
- ^ Tabeta K, Georgel P, Janssen E, Du X, Hoebe K, Crozat K, et al. (marzo de 2004). "Receptores tipo Toll 9 y 3 como componentes esenciales de la defensa inmunitaria innata contra la infección por citomegalovirus en ratones". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 (10): 3516–21. Bibcode :2004PNAS..101.3516T. doi : 10.1073/pnas.0400525101 . PMC 373494 . PMID 14993594.
- ^ ab Roach JC, Glusman G, Rowen L, Kaur A, Purcell MK, Smith KD, et al. (julio de 2005). "La evolución de los receptores tipo Toll en vertebrados". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (27): 9577–82. Bibcode :2005PNAS..102.9577R. doi : 10.1073/pnas.0502272102 . PMC 1172252 . PMID 15976025.
- ^ Lemaitre B, Hoffmann J (2007). "La defensa del huésped de Drosophila melanogaster". Revisión anual de inmunología . 25 : 697–743. doi :10.1146/annurev.immunol.25.022106.141615. PMID 17201680.
- ^ Valanne S, Wang JH, Rämet M (enero de 2011). "La vía de señalización Toll de Drosophila". Journal of Immunology . 186 (2): 649–56. doi : 10.4049/jimmunol.1002302 . PMID 21209287.
- ^ ab Dudzic JP, Hanson MA, Iatsenko I, Kondo S, Lemaitre B (abril de 2019). "Más que negro o blanco: melanización y serina proteasas reguladoras de Toll Share en Drosophila". Cell Reports . 27 (4): 1050–61. doi : 10.1016/j.celrep.2019.03.101 . PMID 31018123.
- ^ Hanson MA, Hamilton PT, Perlman SJ (octubre de 2016). "Genes inmunes y péptidos antimicrobianos divergentes en moscas del subgénero Drosophila". BMC Evolutionary Biology . 16 (1): 228. Bibcode :2016BMCEE..16..228H. doi : 10.1186/s12862-016-0805-y . PMC 5078906 . PMID 27776480.
- ^ ab Lemaitre B, Nicolas E, Michaut L, Reichhart JM, Hoffmann JA (septiembre de 1996). "El casete del gen regulador dorsoventral spätzle/Toll/cactus controla la potente respuesta antifúngica en adultos de Drosophila". Cell . 86 (6): 973–83. doi : 10.1016/s0092-8674(00)80172-5 . PMID 8808632. S2CID 10736743.
- ^ Issa N, Guillaumot N, Lauret E, Matt N, Schaeffer-Reiss C, Van Dorsselaer A, et al. (febrero de 2018). "La proteasa circulante Persephone es un sensor inmunitario para las actividades proteolíticas microbianas aguas arriba de la vía Toll de Drosophila". Molecular Cell . 69 (4): 539–550.e6. doi : 10.1016/j.molcel.2018.01.029 . PMC 5823974 . PMID 29452635.
- ^ Ferrandon D, Imler JL, Hetru C, Hoffmann JA (noviembre de 2007). "La respuesta inmunitaria sistémica de Drosophila: detección y señalización durante infecciones bacterianas y fúngicas". Nature Reviews. Inmunología . 7 (11): 862–74. doi :10.1038/nri2194. PMID 17948019. S2CID 11104900.
- ^ Chowdhury M, Li CF, He Z, Lu Y, Liu XS, Wang YF, et al. (junio de 2019). "Drosophila". Revista de química biológica . 294 (26): 10172–81. doi : 10.1074/jbc.RA118.006804 . PMC 6664172 . PMID 31088910.
- ^ Lee J, Sayed N, Hunter A, Au KF, Wong WH, Mocarski ES, et al. (octubre de 2012). "La activación de la inmunidad innata es necesaria para una reprogramación nuclear eficiente". Cell . 151 (3): 547–58. doi :10.1016/j.cell.2012.09.034. PMC 3506423 . PMID 23101625.
- ^ ab Hatai, Hirotsugu; Lepelley, Alice; Zeng, Wangyong; Hayden, Matthew S.; Ghosh, Sankar (2016). "El receptor tipo Toll 11 (TLR11) interactúa con la flagelina y la profilina a través de mecanismos dispares". PLOS ONE . 11 (2): e0148987. Bibcode :2016PLoSO..1148987H. doi : 10.1371/journal.pone.0148987 . ISSN 1932-6203. PMC 4747465 . PMID 26859749.
- ^ Mathur R, Oh H, Zhang D, Park SG, Seo J, Koblansky A, et al. (octubre de 2012). "Un modelo de ratón de infección por Salmonella typhi". Cell . 151 (3): 590–602. doi :10.1016/j.cell.2012.08.042. PMC 3500584 . PMID 23101627.
- ^ Flajnik, Martin F.; Singh, Nevil J.; Holland, Steven M., eds. (2023). "Cap. 12. Receptores de reconocimiento de patrones y la familia IL-1". Inmunología fundamental de Paul (8.ª ed.). Filadelfia Baltimore Nueva York Londres Buenos Aires Hong Kong Sídney Tokio: Wolters Kluwer/Lippincott Williams & Wilkins. ISBN 978-1-9751-4251-3En términos generales ,
los TLR 1, 2, 4 a 6 y 10 se expresan en la membrana plasmática, y los TLR 3, 7 a 9 y 11 a 13 se confinan a los endosomas.
- ^ abc A menos que se especifique lo contrario en los cuadros, la referencia es: Waltenbaugh C, Doan T, Melvold R, Viselli S (2008). Inmunología . Reseñas ilustradas de Lippincott. Filadelfia: Wolters Kluwer Health/Lippincott Williams & Wilkins. pág. 17. ISBN 978-0-7817-9543-2.
- ^ ab Sabroe I, Dower SK, Whyte MK (noviembre de 2005). "El papel de los receptores tipo Toll en la regulación de la migración, activación y apoptosis de los neutrófilos". Clinical Infectious Diseases . 41 (Supl. 7): S421-6. doi : 10.1086/431992 . PMID 16237641.
- ^ abcd Sallusto F, Lanzavecchia A (2002). "El papel instructivo de las células dendríticas en las respuestas de las células T". Arthritis Research . 4 (Supl 3): S127-32. doi : 10.1186/ar567 . PMC 3240143 . PMID 12110131.
- ^ Gerondakis S, Grumont RJ, Banerjee A (2007). "Regulación de la activación y supervivencia de las células B en respuesta a las señales TLR". Inmunología y biología celular . 85 (6): 471–5. doi : 10.1038/sj.icb.7100097 . PMID 17637697. S2CID 30443009.
- ^ Cario E, Rosenberg IM, Brandwein SL, Beck PL, Reinecker HC, Podolsky DK (enero de 2000). "El lipopolisacárido activa vías de señalización distintas en líneas celulares epiteliales intestinales que expresan receptores tipo Toll". Journal of Immunology . 164 (2): 966–72. doi : 10.4049/jimmunol.164.2.966 . PMID 10623846.
- ^ Peana M, Zdyb K, Medici S, Pelucelli A, Simula G, Gumienna-Kontecka E, Zoroddu MA (diciembre de 2017). "Interacción de Ni(II) con un modelo peptídico del ectodominio TLR4 humano". Journal of Trace Elements in Medicine and Biology . 44 : 151–160. Bibcode :2017JTEMB..44..151P. doi :10.1016/j.jtemb.2017.07.006. PMID 28965571.
- ^ Salazar Gonzalez RM, Shehata H, O'Connell MJ, Yang Y, Moreno-Fernandez ME, Chougnet CA, Aliberti J (2014). "La profilina derivada de Toxoplasma gondii desencadena la producción de citocinas dependiente del receptor tipo Toll 5 humano". Journal of Innate Immunity . 6 (5): 685–94. doi :10.1159/000362367. PMC 4141014 . PMID 24861338.
- ^ Seizer L, Rahimi S, Santos-Sierra S, Drexel M (2022). "Expresión del receptor tipo toll 8 (TLR8) en grupos específicos de interneuronas del hipocampo de ratón". PLOS ONE . 17 (5): e0267860. Bibcode :2022PLoSO..1767860S. doi : 10.1371/journal.pone.0267860 . PMC 9067651 . PMID 35507634.
- ^ Guan Y, Ranoa DR, Jiang S, Mutha SK, Li X, Baudry J, Tapping RI (mayo de 2010). "Los TLR humanos 10 y 1 comparten mecanismos comunes de detección inmunitaria innata pero no de señalización". Journal of Immunology . 184 (9): 5094–103. doi : 10.4049/jimmunol.0901888 . PMID 20348427.
- ^ Chuang T, Ulevitch RJ (marzo de 2001). "Identificación de hTLR10: un nuevo receptor tipo Toll humano expresado preferentemente en células inmunes". Biochimica et Biophysica Acta (BBA) - Estructura y expresión génica . 1518 (1–2): 157–61. doi :10.1016/s0167-4781(00)00289-x. PMID 11267672.
- ^ Hornung V, Rothenfusser S, Britsch S, Krug A, Jahrsdörfer B, Giese T, Endres S, Hartmann G (mayo de 2002). "Expresión cuantitativa del ARNm del receptor tipo Toll 1-10 en subconjuntos celulares de células mononucleares de sangre periférica humana y sensibilidad a oligodesoxinucleótidos CpG". Journal of Immunology . 168 (9): 4531–7. doi : 10.4049/jimmunol.168.9.4531 . PMID 11970999.
- ^ ab Regan T, Nally K, Carmody R, Houston A, Shanahan F, Macsharry J, Brint E (diciembre de 2013). "Identificación de TLR10 como mediador clave de la respuesta inflamatoria a Listeria monocytogenes en células epiteliales intestinales y macrófagos". Journal of Immunology . 191 (12): 6084–92. doi : 10.4049/jimmunol.1203245 . PMID 24198280.
- ^ Yarovinsky F, Zhang D, Andersen JF, Bannenberg GL, Serhan CN, Hayden MS, et al. (junio de 2005). "Activación de células dendríticas por TLR11 mediante una proteína protozoaria similar a la profilina". Science . 308 (5728): 1626–9. Bibcode :2005Sci...308.1626Y. doi : 10.1126/science.1109893 . PMID 15860593. S2CID 34165967.
- ^ Pifer R, Benson A, Sturge CR, Yarovinsky F (febrero de 2011). "UNC93B1 es esencial para la activación de TLR11 y la resistencia del huésped dependiente de IL-12 a Toxoplasma gondii". The Journal of Biological Chemistry . 286 (5): 3307–14. doi : 10.1074/jbc.M110.171025 . PMC 3030336 . PMID 21097503.
- ^ Koblansky AA, Jankovic D, Oh H, Hieny S, Sungnak W, Mathur R, et al. (enero de 2013). "El reconocimiento de la profilina por el receptor tipo Toll 12 es fundamental para la resistencia del huésped a Toxoplasma gondii". Inmunidad . 38 (1): 119–30. doi :10.1016/j.immuni.2012.09.016. PMC 3601573 . PMID 23246311.
- ^ Mishra BB, Gundra UM, Teale JM (diciembre de 2008). "Expresión y distribución de los receptores tipo Toll 11-13 en el cerebro durante la neurocisticercosis murina". Journal of Neuroinflammation . 5 : 53. doi : 10.1186/1742-2094-5-53 . PMC 2631477 . PMID 19077284.
- ^ Shi Z, Cai Z, Sanchez A, Zhang T, Wen S, Wang J, et al. (febrero de 2011). "Un nuevo receptor tipo Toll que reconoce el virus de la estomatitis vesicular". The Journal of Biological Chemistry . 286 (6): 4517–24. doi : 10.1074/jbc.M110.159590 . PMC 3039399 . PMID 21131352.
- ^ Oldenburg M, Krüger A, Ferstl R, Kaufmann A, Nees G, Sigmund A, et al. (agosto de 2012). "TLR13 reconoce el ARNr 23S bacteriano desprovisto de modificación que genera resistencia a la eritromicina". Science . 337 (6098): 1111–5. Bibcode :2012Sci...337.1111O. doi :10.1126/science.1220363. PMID 22821982. S2CID 206540638.
- ^ Hochrein H, Kirschning CJ (marzo de 2013). "Las bacterias evaden el reconocimiento inmunológico a través de TLR13 y la unión de su ARNr 23S por antibióticos MLS mediante los mismos mecanismos". Oncoimmunology . 2 (3): e23141. doi :10.4161/onci.23141. PMC 3661153 . PMID 23802068.
- ^ Schreibelt, Gerty; Benítez-Ribas, Daniel; Schuurhuis, Danita; Lambeck, Annechien JA; van Hout-Kuijer, Maaike; Schaft, Niels; Punt, Cornelis JA; Figdor, Carl G.; Adema, Gosse J.; de Vries, I. Jolanda M. (29 de julio de 2010). "Vacunas profilácticas de uso común como alternativa para ligandos de TLR producidos sintéticamente para madurar células dendríticas derivadas de monocitos". Sangre . 116 (4): 564–574. doi : 10.1182/sangre-2009-11-251884 . hdl : 2066/89493 . ISSN 1528-0020. PMID 20424184.
- ^ Aleynick, Mark; Svensson-Arvelund, Judit; Pantsulaia, Gvantsa; Kim, Kristy; Rose, Samuel A.; Upadhyay, Ranjan; Yellin, Michael; Marsh, Henry; Oreper, Daniel; Jhunjhunwala, Suchit; Moussion, Christine Carine; Merad, Miriam; Brown, Brian D.; Brody, Joshua D. (julio de 2023). "Los agonistas del receptor de reconocimiento de patrones en las vacunas contra patógenos median la preparación cruzada de células T antitumorales". Revista de inmunoterapia del cáncer . 11 (7): e007198. doi :10.1136/jitc-2023-007198. ISSN 2051-1426. PMC 10373699 . PMID 37487664.
- ^ Hoebe K, Du X, Georgel P, Janssen E, Tabeta K, Kim SO, et al. (agosto de 2003). "Identificación de Lps2 como un transductor clave de la señalización TIR independiente de MyD88". Nature . 424 (6950): 743–8. Bibcode :2003Natur.424..743H. doi :10.1038/nature01889. PMID 12872135. S2CID 15608748.
- ^ Hemmi H, Takeuchi O, Kawai T, Kaisho T, Sato S, Sanjo H, et al. (Diciembre de 2000). "Un receptor tipo Toll reconoce el ADN bacteriano". Naturaleza . 408 (6813): 740–5. Código Bib :2000Natur.408..740H. doi :10.1038/35047123. PMID 11130078. S2CID 4405163.
- ^ abcd Kawai T, Akira S (mayo de 2010). "El papel de los receptores de reconocimiento de patrones en la inmunidad innata: actualización sobre los receptores tipo Toll". Nature Immunology . 11 (5): 373–84. doi : 10.1038/ni.1863 . PMID 20404851. S2CID 39414949.
- ^ Shigeoka AA, Holscher TD, King AJ, Hall FW, Kiosses WB, Tobias PS, et al. (mayo de 2007). "TLR2 se expresa de forma constitutiva en el riñón y participa en la lesión renal isquémica a través de vías dependientes e independientes de MyD88". Journal of Immunology . 178 (10): 6252–8. doi : 10.4049/jimmunol.178.10.6252 . PMID 17475853.
- ^ Yamamoto M, Sato S, Hemmi H, Uematsu S, Hoshino K, Kaisho T, et al. (noviembre de 2003). "TRAM está específicamente involucrado en la vía de señalización independiente de MyD88 mediada por el receptor tipo Toll 4". Nature Immunology . 4 (11): 1144–50. doi :10.1038/ni986. PMID 14556004. S2CID 13016860.
- ^ Yamamoto M, Sato S, Hemmi H, Sanjo H, Uematsu S, Kaisho T, et al. (noviembre de 2002). "Función esencial de TIRAP en la activación de la cascada de señalización compartida por TLR2 y TLR4". Nature . 420 (6913): 324–9. Bibcode :2002Natur.420..324Y. doi :10.1038/nature01182. PMID 12447441. S2CID 16163262.
- ^ Peter Fritsch (2004). Dermatología Venerología: Grundlagen. Clínica. Atlas (en alemán). Berlín: Springer. ISBN 3-540-00332-0.
- ^ Rodell CB, Arlauckas SP, Cuccarese MF, Garris CS, Li R, Ahmed MS, et al. (agosto de 2018). "Las nanopartículas cargadas con agonistas TLR7/8 promueven la polarización de los macrófagos asociados a tumores para mejorar la inmunoterapia contra el cáncer". Nature Biomedical Engineering . 2 (8): 578–588. doi :10.1038/s41551-018-0236-8. PMC 6192054 . PMID 31015631.
- ^ Toussi DN, Massari P (abril de 2014). "Efecto adyuvante inmunológico de ligandos de receptores tipo Toll definidos molecularmente". Vacunas . 2 (2): 323–53. doi : 10.3390/vaccines2020323 . PMC 4494261 . PMID 26344622.
- ^ Audarya, SD; Pattnaik, B.; Sanyal, A.; Mohapatra, JK (2017). "Niveles de expresión del ácido ribonucleico mensajero del receptor tipo Toll 7 en animales lecheros en un brote de fiebre aftosa" (PDF) . Buffalo Bulletin . 36 (3): 489–495. Archivado desde el original (PDF) el 28 de abril de 2021.
- ^ Shavit Y, Wolf G, Goshen I, Livshits D, Yirmiya R (mayo de 2005). "La interleucina-1 antagoniza la analgesia por morfina y es la base de la tolerancia a la morfina". Pain . 115 (1–2): 50–9. doi :10.1016/j.pain.2005.02.003. PMID 15836969. S2CID 7286123.
- ^ Mohan S, Davis RL, DeSilva U, Stevens CW (octubre de 2010). "Regulación dual de los receptores opioides mu en células de neuroblastoma SK-N-SH por morfina e interleucina-1β: evidencia de interacción opioide-inmune". Journal of Neuroimmunology . 227 (1–2): 26–34. doi :10.1016/j.jneuroim.2010.06.007. PMC 2942958 . PMID 20615556.
- ^ Komatsu T, Sakurada S, Katsuyama S, Sanai K, Sakurada T (2009). Mecanismo de alodinia provocado por la morfina-3-glucurónido intratecal en ratones . Revista internacional de neurobiología. Vol. 85. págs. 207-19. doi :10.1016/S0074-7742(09)85016-2. ISBN 978-0-12-374893-5. Número de identificación personal 19607972.
- ^ Lewis SS, Hutchinson MR, Rezvani N, Loram LC, Zhang Y, Maier SF, Rice KC, Watkins LR (enero de 2010). "Evidencia de que la morfina-3-glucurónido intratecal puede causar un aumento del dolor a través del receptor tipo toll 4/MD-2 y la interleucina-1 beta". Neurociencia . 165 (2): 569–83. doi :10.1016/j.neuroscience.2009.10.011. PMC 2795035 . PMID 19833175.
- ^ Drahl C (22 de agosto de 2012). "Pequeñas moléculas atacan a receptores tipo Toll". Chemical & Engineering News .
- ^ Shen CH, Tsai RY, Shih MS, Lin SL, Tai YH, Chien CC, Wong CS (febrero de 2011). "Etanercept restaura el efecto antinociceptivo de la morfina y suprime la neuroinflamación espinal en ratas tolerantes a la morfina". Anestesia y analgesia . 112 (2): 454–9. doi : 10.1213/ANE.0b013e3182025b15 . PMID 21081778. S2CID 12295407.
- ^ Hook MA, Washburn SN, Moreno G, Woller SA, Puga D, Lee KH, Grau JW (febrero de 2011). "Un antagonista del receptor de IL-1 bloquea una atenuación inducida por morfina de la recuperación locomotora después de una lesión de la médula espinal". Cerebro, comportamiento e inmunidad . 25 (2): 349–59. doi :10.1016/j.bbi.2010.10.018. PMC 3025088 . PMID 20974246.
- ^ Watkins LR, Hutchinson MR, Rice KC, Maier SF (noviembre de 2009). "El "costo" de la activación glial inducida por opioides: mejorar la eficacia clínica de los opioides al dirigirse a la glía". Tendencias en ciencias farmacológicas . 30 (11): 581–91. doi :10.1016/j.tips.2009.08.002. PMC 2783351 . PMID 19762094.
- ^ ab Hutchinson MR, Zhang Y, Brown K, Coats BD, Shridhar M, Sholar PW, Patel SJ, Crysdale NY, Harrison JA, Maier SF, Rice KC, Watkins LR (julio de 2008). "Reversión no estereoselectiva del dolor neuropático mediante naloxona y naltrexona: participación del receptor tipo Toll 4 (TLR4)". Revista Europea de Neurociencia . 28 (1): 20–9. doi :10.1111/j.1460-9568.2008.06321.x. PMC 2588470 . PMID 18662331.
- ^ Hutchinson MR, Coats BD, Lewis SS, Zhang Y, Sprunger DB, Rezvani N, Baker EM, Jekich BM, Wieseler JL, Somogyi AA, Martin D, Poole S, Judd CM, Maier SF, Watkins LR (noviembre de 2008). "Las citocinas proinflamatorias se oponen a la analgesia aguda y crónica inducida por opioides". Cerebro, comportamiento e inmunidad . 22 (8): 1178–89. doi :10.1016/j.bbi.2008.05.004. PMC 2783238 . PMID 18599265.
- ^ Hutchinson MR, Lewis SS, Coats BD, Rezvani N, Zhang Y, Wieseler JL, Somogyi AA, Yin H, Maier SF, Rice KC, Watkins LR (mayo de 2010). "Posible participación de la actividad del receptor tipo Toll 4/factor de diferenciación mieloide-2 de los isómeros inactivos de opioides causa proinflamación espinal y consecuencias conductuales relacionadas". Neurociencia . 167 (3): 880–93. doi :10.1016/j.neuroscience.2010.02.011. PMC 2854318 . PMID 20178837.
- ^ Hashimoto C, Hudson KL, Anderson KV (enero de 1988). "El gen Toll de Drosophila, necesario para la polaridad embrionaria dorsal-ventral, parece codificar una proteína transmembrana". Cell . 52 (2): 269–79. doi :10.1016/0092-8674(88)90516-8. PMID 2449285. S2CID 19439405.
- ^ Nomura N, Miyajima N, Sazuka T, Tanaka A, Kawarabayasi Y, Sato S, et al. (1994). "Predicción de las secuencias codificantes de genes humanos no identificados. I. Las secuencias codificantes de 40 genes nuevos (KIAA0001-KIAA0040) deducidas mediante el análisis de clones de ADNc muestreados aleatoriamente de la línea celular mieloide humana inmadura KG-1". DNA Research . 1 (1): 27–35. doi : 10.1093/dnares/1.1.27 . PMID 7584026.
- ^ Taguchi T, Mitcham JL, Dower SK, Sims JE, Testa JR (marzo de 1996). "Localización cromosómica de TIL, un gen que codifica una proteína relacionada con el receptor transmembrana Toll de Drosophila, en el cromosoma humano 4p14". Genomics . 32 (3): 486–8. doi :10.1006/geno.1996.0150. PMID 8838819.
- ^ Gay NJ, Keith FJ (mayo de 1991). "Receptor Toll e IL-1 de Drosophila". Nature . 351 (6325): 355–6. Código Bibliográfico :1991Natur.351..355G. doi :10.1038/351355b0. PMID 1851964. S2CID 1700458.
- ^ Poltorak A, He X, Smirnova I, Liu MY, Van Huffel C, Du X, et al. (diciembre de 1998). "Señalización de LPS defectuosa en ratones C3H/HeJ y C57BL/10ScCr: mutaciones en el gen Tlr4". Science . 282 (5396): 2085–8. Bibcode :1998Sci...282.2085P. doi :10.1126/science.282.5396.2085. PMID 9851930.
- ^ Hoshino K, Takeuchi O, Kawai T, Sanjo H, Ogawa T, Takeda Y, et al. (abril de 1999). "Vanguardia: los ratones deficientes en el receptor tipo Toll 4 (TLR4) son hiporresponsivos al lipopolisacárido: evidencia de que el TLR4 es el producto del gen Lps". Journal of Immunology . 162 (7): 3749–52. doi : 10.4049/jimmunol.162.7.3749 . PMID 10201887. S2CID 7419784.
- ^ Song WY, Wang GL, Chen LL, Kim HS, Pi LY, Holsten T, et al. (diciembre de 1995). "Una proteína similar a la quinasa del receptor codificada por el gen de resistencia a enfermedades del arroz, Xa21". Science . 270 (5243): 1804–6. Bibcode :1995Sci...270.1804S. doi :10.1126/science.270.5243.1804. PMID 8525370. S2CID 10548988.
- ^ Gómez-Gómez L, Boller T (junio de 2000). "FLS2: una quinasa similar al receptor LRR implicada en la percepción del elicitor bacteriano flagelina en Arabidopsis". Molecular Cell . 5 (6): 1003–11. doi : 10.1016/S1097-2765(00)80265-8 . PMID 10911994.
- ^ "El Premio Nobel de Fisiología o Medicina 2011". Nobel Media AB . 3 de octubre de 2011.
- ^ Mitchell B (23 de marzo de 2011). "Médico de Columbia Británica gana prestigioso premio médico". The Star .
Véase también
Enlaces externos
- Receptores Toll-Like+ en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Toll+protein,+Drosophila en los Encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- TollML: base de datos de receptores y ligandos tipo Toll de la Universidad de Múnich
- La familia de receptores de tipo Toll de receptores inmunitarios innatos (pdf)
- Vía del receptor tipo Toll
- Animaciones de biociencias
