Operón

En genética , un operón es una unidad funcional de ADN que contiene un grupo de genes bajo el control de un único promotor . [1] Los genes se transcriben juntos en una cadena de ARNm y se traducen juntos en el citoplasma o sufren un empalme para crear ARNm monocistrónicos que se traducen por separado, es decir, varias cadenas de ARNm que codifican cada una un único producto génico. El resultado de esto es que los genes contenidos en el operón se expresan juntos o no se expresan en absoluto. Varios genes deben cotranscribirse para definir un operón. [2]
Originalmente, se pensaba que los operones existían únicamente en procariotas (que incluyen orgánulos como los plástidos que se derivan de bacterias ), pero su descubrimiento en eucariotas se demostró a principios de la década de 1990 y se consideran raros. [3] [4] [5] [6] En general, la expresión de operones procariotas conduce a la generación de ARNm policistrónicos , mientras que los operones eucariotas conducen a ARNm monocistrónicos.
Los operones también se encuentran en virus como los bacteriófagos . [7] [8] Por ejemplo, los fagos T7 tienen dos operones. El primer operón codifica varios productos, incluida una ARN polimerasa T7 especial que puede unirse al segundo operón y transcribirlo. El segundo operón incluye un gen de lisis destinado a hacer que la célula huésped reviente. [9]
Historia
El término "operón" fue propuesto por primera vez en un breve artículo en las Actas de la Academia Francesa de Ciencias en 1960. [10] A partir de este artículo, se desarrolló la llamada teoría general del operón. Esta teoría sugería que en todos los casos, los genes dentro de un operón están controlados negativamente por un represor que actúa en un solo operador ubicado antes del primer gen. Más tarde, se descubrió que los genes podrían ser regulados positivamente y también regulados en los pasos que siguen al inicio de la transcripción. Por lo tanto, no es posible hablar de un mecanismo regulador general, porque diferentes operones tienen diferentes mecanismos. Hoy en día, el operón se define simplemente como un grupo de genes transcritos en una sola molécula de ARNm. Sin embargo, el desarrollo del concepto se considera un evento histórico en la historia de la biología molecular. El primer operón que se describió fue el operón lac en E. coli . [10] El Premio Nobel de Fisiología y Medicina de 1965 fue otorgado a François Jacob , André Michel Lwoff y Jacques Monod por sus descubrimientos relacionados con la síntesis de operones y virus.
Descripción general
Operón policistrónico Estructura de un operón procariota de genes codificadores de proteínas . La secuencia reguladora controla cuándo se produce la expresión de las múltiples regiones codificadoras de proteínas (rojo). Las regiones promotora , operadora y potenciadora (amarillo) regulan la transcripción del gen en un ARNm. Las regiones no traducidas del ARNm (azul) regulan la traducción en los productos proteicos finales. [11] |
Los operones se encuentran principalmente en procariotas , pero también raramente en algunos eucariotas , incluidos nematodos como C. elegans y la mosca de la fruta, Drosophila melanogaster . [3] Los genes de ARNr a menudo existen en operones que se han encontrado en una variedad de eucariotas, incluidos los cordados . Un operón se compone de varios genes estructurales dispuestos bajo un promotor común y regulados por un operador común. Se define como un conjunto de genes estructurales adyacentes, más las señales reguladoras adyacentes que afectan la transcripción de los genes estructurales. 5 [12] Los reguladores de un operón dado, incluidos los represores , correpresores y activadores , no están necesariamente codificados por ese operón. La ubicación y la condición de los reguladores, el promotor, el operador y las secuencias de ADN estructural pueden determinar los efectos de mutaciones comunes.
Los operones están relacionados con los regulones , los estimuladores y los modulones ; mientras que los operones contienen un conjunto de genes regulados por el mismo operador, los regulones contienen un conjunto de genes regulados por una única proteína reguladora, y los estimuladores contienen un conjunto de genes regulados por un único estímulo celular. Según sus autores, el término "operón" se deriva del verbo "operar". [13]
Como unidad de transcripción
Un operón contiene uno o más genes estructurales que generalmente se transcriben en un ARNm policistrónico (una única molécula de ARNm que codifica más de una proteína ). Sin embargo, la definición de un operón no requiere que el ARNm sea policistrónico, aunque en la práctica, generalmente lo es. [6] Corriente arriba de los genes estructurales se encuentra una secuencia promotora que proporciona un sitio para que la ARN polimerasa se una e inicie la transcripción. Cerca del promotor se encuentra una sección de ADN llamada operador .
Operones versus agrupamiento de genes procariotas
Todos los genes estructurales de un operón se activan o desactivan juntos, debido a un único promotor y operador aguas arriba de ellos, pero a veces se necesita un mayor control sobre la expresión génica. Para lograr este aspecto, algunos genes bacterianos se encuentran ubicados cerca unos de otros, pero hay un promotor específico para cada uno de ellos; esto se llama agrupamiento de genes . Por lo general, estos genes codifican proteínas que trabajarán juntas en la misma vía, como una vía metabólica. El agrupamiento de genes ayuda a una célula procariota a producir enzimas metabólicas en un orden correcto. [14] En un estudio, se ha postulado que en Asgard (arqueas) , los genes codificadores de proteínas ribosómicas se presentan en grupos que están menos conservados en su organización que en otras arqueas ; cuanto más cerca está un Asgard (arqueas) de los eucariotas , más dispersa es la disposición de los genes codificadores de proteínas ribosómicas. [15]
Estructura general
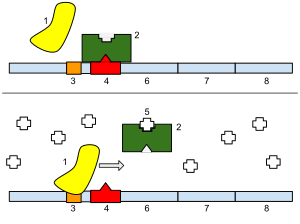
Un operón se compone de 3 componentes básicos de ADN:
- Promotor : secuencia de nucleótidos que permite la transcripción de un gen . La ARN polimerasa reconoce al promotor y, a continuación, inicia la transcripción. En la síntesis de ARN, los promotores indican qué genes se deben utilizar para la creación del ARN mensajero y, por extensión, controlan qué proteínas produce la célula.
- Operador : segmento de ADN al que se une un represor . Se define clásicamente en el operón lac como un segmento entre el promotor y los genes del operón. [16] El operador principal (O1) en el operón lac se encuentra ligeramente aguas abajo del promotor; dos operadores adicionales, O2 y O3, se encuentran en -82 y +412, respectivamente. En el caso de un represor, la proteína represora obstruye físicamente la transcripción de los genes por parte de la ARN polimerasa.
- Genes estructurales : los genes que están co-regulados por el operón.
No siempre está incluido dentro del operón, pero es importante para su función el gen regulador , un gen que se expresa constantemente y que codifica proteínas represoras . El gen regulador no necesita estar dentro, adyacente o incluso cerca del operón para controlarlo. [17]
Un inductor (molécula pequeña) puede desplazar un represor (proteína) del sitio operador (ADN), dando como resultado un operón desinhibido.
Alternativamente, un correpresor puede unirse al represor para permitir su unión al sitio operador. Un buen ejemplo de este tipo de regulación se observa en el operón trp .
Regulación
El control de un operón es un tipo de regulación genética que permite a los organismos regular la expresión de varios genes en función de las condiciones ambientales. La regulación del operón puede ser negativa o positiva por inducción o represión. [16]
El control negativo implica la unión de un represor al operador para evitar la transcripción.
- En los operones inducibles negativos , una proteína represora reguladora normalmente está unida al operador, lo que impide la transcripción de los genes en el operón. Si hay una molécula inductora presente, se une al represor y cambia su conformación de modo que no puede unirse al operador. Esto permite la expresión del operón. El operón lac es un operón inducible controlado negativamente, donde la molécula inductora es alolactosa .
- En los operones reprimibles negativos , la transcripción del operón se lleva a cabo normalmente. Las proteínas represoras son producidas por un gen regulador , pero no pueden unirse al operador en su conformación normal. Sin embargo, ciertas moléculas llamadas correpresores se unen a la proteína represora, lo que provoca un cambio conformacional en el sitio activo. La proteína represora activada se une al operador y evita la transcripción. El operón trp , involucrado en la síntesis de triptófano (que actúa como correpresor), es un operón reprimible controlado negativamente.
Los operones también pueden controlarse positivamente. Con el control positivo, una proteína activadora estimula la transcripción uniéndose al ADN (normalmente en un sitio distinto del operador).
- En los operones inducibles positivos , las proteínas activadoras normalmente no pueden unirse al ADN pertinente. Cuando un inductor se une a la proteína activadora, sufre un cambio en la conformación para poder unirse al ADN y activar la transcripción. Entre los ejemplos de operones inducibles positivos se incluyen la familia MerR de activadores transcripcionales.
- En los operones reprimibles positivos , las proteínas activadoras normalmente están unidas al segmento de ADN pertinente. Sin embargo, cuando un inhibidor se une al activador, se le impide unirse al ADN. Esto detiene la activación y la transcripción del sistema.
Ellacaoperón
El operón lac de la bacteria modelo Escherichia coli fue el primer operón que se descubrió y proporciona un ejemplo típico de la función del operón. Consta de tres genes estructurales adyacentes , un promotor , un terminador y un operador . El operón lac está regulado por varios factores, incluida la disponibilidad de glucosa y lactosa . Puede ser activado por alolactosa . La lactosa se une a la proteína represora y evita que reprima la transcripción génica. Este es un ejemplo del modelo desreprimible (de arriba: inducible negativo). Por lo tanto, es un operón inducible negativo inducido por la presencia de lactosa o alolactosa.
Eltrpoperón
Descubierto en 1953 por Jacques Monod y sus colegas, el operón trp en E. coli fue el primer operón reprimible que se descubrió. Mientras que el operón lac puede ser activado por una sustancia química ( alolactosa ), el operón triptófano (Trp) es inhibido por una sustancia química (triptófano). Este operón contiene cinco genes estructurales: trp E, trp D, trp C, trp B y trp A, que codifica la triptófano sintetasa . También contiene un promotor que se une a la ARN polimerasa y un operador que bloquea la transcripción cuando se une a la proteína sintetizada por el gen represor (trp R) que se une al operador. En el operón lac, la lactosa se une a la proteína represora y evita que reprima la transcripción génica, mientras que en el operón trp, el triptófano se une a la proteína represora y le permite reprimir la transcripción génica. Además, a diferencia del operón lac, el operón trp contiene un péptido líder y una secuencia atenuadora que permite una regulación gradual. [18] Este es un ejemplo del modelo correprimible .
Predicción del número y organización de operones
El número y la organización de los operones se han estudiado de forma más exhaustiva en E. coli . Como resultado, se pueden hacer predicciones basadas en la secuencia genómica de un organismo.
Un método de predicción utiliza la distancia intergénica entre los marcos de lectura como predictor principal del número de operones en el genoma. La separación simplemente cambia el marco y garantiza que la lectura sea eficiente. Existen tramos más largos donde los operones comienzan y terminan, a menudo hasta 40-50 bases. [19]
Un método alternativo para predecir operones se basa en encontrar grupos de genes donde el orden y la orientación de los genes se conservan en dos o más genomas. [20]
La predicción de operones es aún más precisa si se considera la clase funcional de las moléculas. Las bacterias han agrupado sus marcos de lectura en unidades, secuestradas por su participación en complejos proteínicos, vías comunes o sustratos y transportadores compartidos. Por lo tanto, una predicción precisa implicaría todos estos datos, una tarea difícil en verdad.
El laboratorio de Pascale Cossart fue el primero en identificar experimentalmente todos los operones de un microorganismo, Listeria monocytogenes . Los 517 operones policistrónicos se enumeran en un estudio de 2009 que describe los cambios globales en la transcripción que ocurren en L. monocytogenes en diferentes condiciones. [21]
Véase también
- Biología evolutiva del desarrollo
- Código genético
- Red reguladora de genes
- Operón L-arabinosa
- Biosíntesis de proteínas
- Caja TATA
- Prueba de cromoterapia
Referencias
- ^ Sadava DE, Hillis DM, Heller HC, Berenbaum M (2009). La vida: la ciencia de la biología (novena edición). Macmillan. pág. 349. ISBN 978-1-4292-1962-4.
- ^ Lodish H, Zipursky L, Matsudaira P, Baltimore D, Darnel J (2000). "Capítulo 9: Definición molecular de un gen". Biología celular molecular. WH Freeman. ISBN 978-0-7167-3136-8.
- ^ ab Kominek J, Doering DT, Opulente DA, Shen XX, Zhou X, DeVirgilio J, Hulfachor AB, Groenewald M, Mcgee MA, Karlen SD, Kurtzman CP, Rokas A, Hittinger CT (marzo de 2019). "Adquisición eucariota de un operón bacteriano". Cell . 176 (6): 1356–1366.e10. doi :10.1016/j.cell.2019.01.034. PMC 7295392 . PMID 30799038.
- ^ Spieth J, Brooke G, Kuersten S, Lea K, Blumenthal T (mayo de 1993). "Operones en C. elegans: los precursores de ARNm policistrónicos se procesan mediante el empalme trans de SL2 a regiones codificantes posteriores". Cell . 73 (3): 521–32. doi :10.1016/0092-8674(93)90139-H. PMID 8098272. S2CID 26918553.
- ^ Brogna S, Ashburner M (abril de 1997). "El gen relacionado con Adh de Drosophila melanogaster se expresa como un ARN mensajero dicistrónico funcional: transcripción multigénica en organismos superiores". The EMBO Journal . 16 (8): 2023–31. doi :10.1093/emboj/16.8.2023. PMC 1169805 . PMID 9155028.
- ^ ab Blumenthal T (noviembre de 2004). "Operones en eucariotas". Briefings in Functional Genomics & Proteomics . 3 (3): 199–211. doi : 10.1093/bfgp/3.3.199 . PMID 15642184.
- ^ "Definición de operón". Diccionario médico . MedicineNet.com . Consultado el 30 de diciembre de 2012 .
- ^ Liu J, Mushegian A (julio de 2004). "Desplazamientos de genes de proteasas prohead en los operones tardíos de bacteriófagos de ADN bicatenario". Journal of Bacteriology . 186 (13): 4369–75. doi :10.1128/JB.186.13.4369-4375.2004. PMC 421614 . PMID 15205439.
- ^ "Operones de uso de bacteriófagos". Control de genes procariotas . Dartmouth College. Archivado desde el original el 28 de enero de 2013. Consultado el 30 de diciembre de 2012 .
- ^ ab Jacob F, Perrin D, Sanchez C, Monod J (febrero de 1960). «[Operón: un grupo de genes con la expresión coordinada por un operador]» [Operón: un grupo de genes con la expresión coordinada por un operador] (PDF) . Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences (versión facsímil reimpresa en 2005) (en francés). 250 (6): 1727–9. PMID 14406329. Archivado desde el original (PDF) el 2016-03-04 . Consultado el 2015-08-27 .
- ^ Shafee, Thomas; Lowe, Rohan (2017). "Estructura de genes eucariotas y procariotas". WikiJournal of Medicine . 4 (1). doi : 10.15347/wjm/2017.002 . ISSN 2002-4436.
- ^ Miller JH, Suzuki DT, Griffiths AJ, Lewontin RC, Wessler SR, Gelbart WM (2005). Introducción al análisis genético (8.ª ed.). San Francisco: WH Freeman. pág. 740. ISBN 978-0-7167-4939-4.
- ^ Jacob F (mayo de 2011). "El nacimiento del operón". Science . 332 (6031): 767. Bibcode :2011Sci...332..767J. doi : 10.1126/science.1207943 . PMID 21566161.
- ^ Lee JM, Sonnhammer EL (mayo de 2003). "Análisis de agrupamiento de genes genómicos de vías en eucariotas". Genome Research . 13 (5): 875–82. doi :10.1101/gr.737703. PMC 430880 . PMID 12695325.
- ^ Tirumalai MR, Sivaraman RV, Kutty LA, Song EL, Fox GE (septiembre de 2003). "Organización de grupos de proteínas ribosómicas en arqueas de Asgard". Archaea . 2023 : 1–16. doi : 10.1155/2023/5512414 . PMC 10833476 . PMID 38314098.
- ^ ab Lewin B (1990). Genes IV (4.ª ed.). Oxford: Oxford University Press. págs. 243–58. ISBN 978-0-19-854267-4.
- ^ Mayer G. "Bacteriología – Capítulo nueve Mecanismos de regulación genética". Microbiología e inmunología en línea . Facultad de Medicina de la Universidad de Carolina del Sur . Consultado el 30 de diciembre de 2012 .
- ^ Cummings MS, Klug WS (2006). Conceptos de genética (8.ª ed.). Upper Saddle River, NJ: Pearson Education. págs. 394–402. ISBN 978-0-13-191833-7.
- ^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (junio de 2000). "Operones en Escherichia coli: análisis genómicos y predicciones". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (12): 6652–7. Bibcode :2000PNAS...97.6652S. doi : 10.1073/pnas.110147297 . PMC 18690 . PMID 10823905.
- ^ Ermolaeva MD, White O, Salzberg SL (marzo de 2001). "Predicción de operones en genomas microbianos". Nucleic Acids Research . 29 (5): 1216–21. doi :10.1093/nar/29.5.1216. PMC 29727 . PMID 11222772.
- ^ Toledo-Arana A, Dussurget O, Nikitas G, Sesto N, Guet-Revillet H, Balestrino D, Loh E, Gripenland J, Tiensuu T, Vaitkevicius K, Barthelemy M, Vergassola M, Nahori MA, Soubigou G, Régnault B, Coppée JY, Lecuit M, Johansson J, Cossart P (junio de 2009). "El panorama transcripcional de Listeria desde el saprofitismo hasta la virulencia". Naturaleza . 459 (7249): 950–6. Código Bib :2009Natur.459..950T. doi : 10.1038/naturaleza08080. PMID 19448609. S2CID 4341657.
Enlaces externos
- Navegador de correlación de operones H37Rv de Mycobacterium tuberculosis
- OBD - Base de datos de operones (aunque un poco incómoda de usar)


