Haplogrupo R-M269
| Haplogrupo R-M269 | |
|---|---|
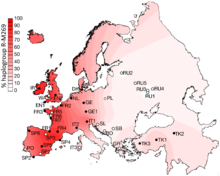 Distribución de frecuencias espaciales proyectadas para el haplogrupo R-M269 en Europa. [1] | |
| Posible época de origen | 4.000–10.000 AP [2] [3] |
| Posible lugar de origen | Europa del Este, asociada a las migraciones indoeuropeas [4] [5] |
| Antepasado | R1b1a1a (R-P297) |
| Descendientes | L23; L51/M412, L151/P310; Z2103 |
| Definición de mutaciones | M269 |
El haplogrupo R-M269 es el subclado del haplogrupo R1b del cromosoma Y humano que se define por el marcador SNP M269 . Según la ISOGG 2020, está clasificado filogenéticamente como R1b1a1b . Fue objeto de una investigación intensiva y anteriormente se clasificó como R1b1a2 (2003 a 2005), R1b1c (2005 a 2008), R1b1b2 (2008 a 2011) y R1b1a1a2 (2011 a 2020). [6]
El R-M269 es de particular interés para la historia genética de Europa occidental , siendo el haplogrupo europeo más común. Aumenta en frecuencia en un gradiente de este a oeste (su prevalencia en Polonia se estima en un 22,7%, en comparación con Gales en un 92,3%). Es portado por aproximadamente 110 millones de hombres europeos (estimación de 2010). [3] La edad de la mutación M269 se estima en 4.000 a 10.000 años atrás. [2] [3]
Origen
Anteriormente se había datado R-M269 en el Paleolítico superior, [7] pero alrededor de 2010 se pensaba que se había formado cerca del comienzo de la Revolución Neolítica, hace unos 10.000 años. [8] [9] [10] Sin embargo, estudios arqueogenéticos más recientes desde 2015 sugieren firmemente un origen entre los cazadores-recolectores del Eneolítico de Europa del Este . [4] [11]
Balaresque et al. (2010) basándose en el patrón de diversidad de Y-STR argumentaron que existía una única fuente en Oriente Próximo y que la introducción en Europa a través de Anatolia en la Revolución Neolítica. En este escenario, los cazadores-recolectores mesolíticos en Europa habrían sido casi reemplazados por los agricultores que llegaron. Por el contrario, Busby et al. (2012) no pudieron confirmar los resultados de Balaresque et al. (2010) y no pudieron hacer estimaciones creíbles de la edad de R-M269 basándose en la diversidad de Y-STR. [3] [12] Además, estudios más recientes han descubierto que el ADN-Y de los primeros agricultores europeos es típicamente el haplogrupo G2a. [13]
Según un estudio de 2015, [4] un cazador-recolector de Samara (fechado entre 5640 y 5555 a. C.) perteneciente al haplogrupo R1b1(*) era ancestro de ambos haplogrupos R-M269 y R-M478. Según los autores, la aparición de formas basales de R1b en cazadores-recolectores de Europa del Este proporciona una "fuente geográficamente plausible" para el haplogrupo R-M269. Se ha descubierto que los subclados de R-M269, como R-Z2103, son frecuentes en el ADN antiguo encontrado en individuos asociados con la cultura Yamnaya y poblaciones relacionadas, [4] [14] y la dispersión de este haplogrupo está asociada con la propagación de la llamada "ascendencia esteparia" y al menos algunas de las lenguas indoeuropeas. [4] [15]
Según Lazaridis et al. (2022), "la hipótesis más probable" es que todo el clado R-M269 se originó "en el Cáucaso Norte y la estepa al norte". [16]
El subclado R-P311 está sustancialmente confinado a Europa Occidental en las poblaciones modernas. R-P311 está ausente del ADN antiguo de la era Neolítica encontrado en Europa Occidental, lo que sugiere fuertemente que su distribución actual se debe a movimientos de población dentro de Europa que tuvieron lugar después del final del Neolítico. Los tres subclados principales de P311 son U106 (S21), L21 (M529, S145) y U152 (S28). Estos muestran una clara articulación dentro de Europa Occidental, con centros en los Países Bajos , las Islas Británicas y los Alpes , respectivamente. [17] Estos linajes están asociados con los grupos no relacionados con la estepa ibérica de la cultura del vaso campaniforme , y demuestran la relación entre la ascendencia relacionada con la estepa y los subclados R1b-M269, [14] que son "el linaje principal asociado con la llegada de la ascendencia esteparia a Europa occidental después de 2500 a. C." [18]
Distribución
El R1b europeo está dominado por el R-M269. Se ha encontrado en frecuencias generalmente bajas en toda Eurasia central , [19] pero con una frecuencia relativamente alta entre los bashkires de la región de Perm (84,0%) y el distrito de Baymaksky (81,0%). [20] Este marcador está presente en China y la India en frecuencias de menos del uno por ciento. La siguiente tabla enumera con más detalle las frecuencias del M269 en regiones de Asia, Europa y África.
La distribución de R-M269 en Europa aumenta en frecuencia de este a oeste. Alcanza su pico a nivel nacional en Gales a una tasa del 92%, del 82% en Irlanda , del 70% en Escocia , del 68% en España , del 60% en Francia (76% en Normandía ), alrededor del 60% en Portugal , [21] el 50% en Alemania , el 50% en los Países Bajos , el 47% en Italia , [22] el 45% en el este de Inglaterra , el 43% en Dinamarca y el 42% en Islandia . Es tan alta como el 95% en partes de Irlanda. También se encuentra en algunas áreas del norte de África , donde su frecuencia alcanza un pico del 10% en algunas partes de Argelia . [23] M269 también se ha observado entre el 8% de los herero en Namibia . [24] El subclado R-M269 se ha encontrado en fósiles guanches (bimbapes) antiguos excavados en Punta Azul, El Hierro , Islas Canarias , que datan del siglo X (~44%). [25] En Asia occidental, se ha informado de R-M269 en el 29,2% de los varones asirios de Irán. [26] El haplogrupo R1b1 y sus subclados en Asia. [27] M269* (xL23) se encuentra con la frecuencia más alta en los Balcanes centrales, especialmente Kosovo con un 7,9%, Macedonia del Norte con un 5,1% y Serbia con un 4,4%. [21] Kosovo es notable por tener un alto porcentaje de descendientes L23* o L23(xM412) en 11.4% a diferencia de la mayoría de las otras áreas con porcentajes significativos de M269* y L23* excepto Polonia con 2.4% y 9.5% y los Bashkirs del sureste de Bashkortostán con 2.4% y 32.2% respectivamente. [21] Cabe destacar que esta población Bashkir también tiene un alto porcentaje de la rama hermana M269 M73 en 23.4%. [21] Cinco individuos de 110 evaluados en el Valle de Ararat, Armenia pertenecían a R1b1a2* y 36 a L23*, sin que ninguno perteneciera a subclados conocidos de L23. [28] Trofimova et al. (2015) encontraron una frecuencia sorprendentemente alta de R1b-L23 (Z2105/2103) entre los pueblos de Idel-Ural . 21 de 58 (36,2%) de los bashkires del distrito de Burzyansky , 11 de 52 (21,2%) de los udmurtos , 4 de 50 (8%) de los komi , 4 de 59 (6,8%) de los mordvinos , 2 de 53 (3,8%) de losBesermyan y 1 de 43 (2,3%) de Chuvash eran R1b-L23 (Z2105/2103), [29] el tipo de R1b encontrado en los restos de Yamna analizados recientemente de Samara Oblast y Orenburg Oblast . [30]
En particular, el R1b de Europa occidental está dominado por subclados específicos de R-M269 (con algunas pequeñas cantidades de otros tipos halladas en áreas como Cerdeña [21] [31] ). Dentro de Europa, el R-M269 está dominado por el R-M412, también conocido como R-L51, que según Myres et al. (2010) está "prácticamente ausente en Oriente Próximo, el Cáucaso y Asia occidental". Esta población de Europa occidental se divide además entre R-P312/S116 y R-U106/S21, que parecen extenderse desde la cuenca occidental y oriental del río Rin respectivamente. Myres et al. señalan además que, en lo que respecta a sus parientes más cercanos, en el R-L23*, es "instructivo" que estos sean a menudo más del 10% de la población en el Cáucaso, Turquía y algunas poblaciones del sudeste europeo y circunurálicas.
En Europa occidental está presente, pero en niveles generalmente mucho más bajos, aparte de "un caso del 27% en el valle superior del Ródano en Suiza ". [21] Además, el mapa de distribución de subclados, Figura 1h titulada "L11(xU106,S116)", en Myres et al. muestra que R-P310/L11* (o subclados aún no definidos de R-P310/L11) ocurre solo en frecuencias mayores del 10% en Inglaterra central, con áreas circundantes de Inglaterra y Gales con frecuencias más bajas. [21] Este R-P310/L11* es casi inexistente en el resto de Eurasia y el norte de África, con la excepción de las tierras costeras que bordean el Báltico occidental y meridional (alcanzando el 10% en el este de Dinamarca y el 6% en el norte de Polonia) y en el este de Suiza y alrededores. [21]
| M269 (R1b1a1a2) [32] |
| ||||||||||||||||||||||||||||||||||||||||||
En 2009, el ADN extraído de los huesos del fémur de 6 esqueletos en un lugar de enterramiento de principios de la Edad Media en Ergolding (Baviera, Alemania) que data de alrededor de c. 670 arrojó los siguientes resultados: se encontró que 4 eran del haplogrupo R1b con las coincidencias más cercanas en las poblaciones modernas de Alemania, Irlanda y los EE. UU., mientras que 2 estaban en el haplogrupo G2a . [33]
Los estudios de población que realizan pruebas para detectar el M269 se han vuelto más comunes en los últimos años, mientras que en estudios anteriores los hombres de este haplogrupo solo son visibles en los datos mediante la extrapolación de lo que es probable. A continuación se ofrece un resumen de la mayoría de los estudios que realizaron pruebas específicas para detectar el M269, mostrando su distribución (como porcentaje de la población total) en Europa, África del Norte , Oriente Medio y Asia Central, hasta China y Nepal .
| País | Muestreo | muestra | R-M269 | Fuente |
|---|---|---|---|---|
| Gales | Nacional | 65 | 92,3% | Balaresque y otros (2009) [3] |
| España | Vascos | 116 | 87,1% | Balaresque y otros (2009) [3] |
| Irlanda | Nacional | 796 | 85,4% | Moore y otros (2006) [34] |
| España | Cataluña | 80 | 81,3% | Balaresque y otros (2009) [3] |
| Italia | Lombardía | 78 | 80,8% | Grugni y otros [22] |
| Francia | Ille y Vilaine | 82 | 80,5% | Balaresque y otros (2009) [3] |
| Francia | Alto Garona | 57 | 78,9% | Balaresque y otros (2009) [3] |
| Inglaterra | Cornualles | 64 | 78,1% | Balaresque y otros (2009) [3] |
| Francia | Loira Atlántico | 48 | 77,1% | Balaresque y otros (2009) [3] |
| Italia | Toscana | 42 | 76,2% | Di Giacomo y otros (2003) [35] |
| Francia | Finisterre | 75 | 76,0% | Balaresque y otros (2009) [3] |
| Francia | Vascos | 61 | 75,4% | Balaresque y otros (2009) [3] |
| Italia | Noreste | 30 | 73,5% | Di Giacomo y otros (2003) [35] |
| España | Andalucía Oriental | 95 | 72,0% | Balaresque y otros (2009) [3] |
| España | Castilla La Mancha | 63 | 72,0% | Balaresque y otros (2009) [3] |
| Francia | Vendée | 50 | 68,0% | Balaresque y otros (2009) [3] |
| República Dominicana | Nacional | 26 | 65,4% | Bryc y otros (2010) [36] |
| Francia | Bahía de Somme | 43 | 62,8% | Balaresque y otros (2009) [3] |
| Inglaterra | Condado de Leicestershire | 43 | 62,0% | Balaresque y otros (2009) [3] |
| Italia | Noreste (Ladino) | 79 | 60,8% | Balaresque y otros (2009) [3] |
| Portugal | Nacional | 657 | 59,9% | Beleza y otros (2006) [37] |
| Italia | Emilia | 29 | 58,5% | Boattini y otros (2013) [38] |
| España | Galicia | 88 | 58,0% | Balaresque y otros (2009) [3] |
| España | Andalucía Occidental | 72 | 55,0% | Balaresque y otros (2009) [3] |
| Portugal | Sur | 78 | 46,2% | Balaresque y otros (2009) [3] |
| Italia | Noroeste | 99 | 45,0% | Balaresque y otros (2009) [3] |
| Dinamarca | Nacional | 56 | 42,9% | Balaresque y otros (2009) [3] |
| Países Bajos | Nacional | 84 | 42,0% | Balaresque y otros (2009) [3] |
| Armenia | Valle de Ararat | 41 | 37,3% | Herrera y col. (2012) [28] |
| Rusia | Bashkires | 471 | 34,40% | Lobov (2009) [20] |
| Italia | Sicilia oriental | 246 | 34,14% | Tofanelli y otros (2015) [39] |
| Italia | Sicilia occidental | 68 | 33,0% | Tofanelli y otros (2015) [39] |
| Alemania | Baviera | 80 | 32,3% | Balaresque y otros (2009) [3] |
| Pavo | Armenios del lago Van | 33 | 32,0% | Herrera y col. (2012) [28] |
| Armenia | Hombre guardián | 30 | 31,3% | Herrera y col. (2012) [28] |
| Irán | Asirios | 48 | 29,2% | Grugni, Viola et al. (2012) [26] |
| Polonia | Nacional | 110 | 22,7% | Myres y otros (2007) [40] |
| Eslovenia | Nacional | 75 | 21,3% | Battaglia y otros (2008) [41] |
| Albaneses de Kosovo | Nacional | 114 | 21,1% | Pericic2005 [42] |
| Eslovenia | Nacional | 70 | 20,6% | Balaresque y otros (2009) [3] |
| Pavo | Central | 152 | 19,1% | Cinnioğlu y otros (2004) [43] |
| Albaneses en Macedonia del Norte | Nacional | 64 | 18,8% | Battaglia y otros (2008) [41] |
| Albaneses | Nacional | 55 | 18,2% | Battaglia y otros (2008) [41] |
| Creta | Nacional | 193 | 17,0% | Rey y otros (2008) [44] |
| Italia | Cerdeña | 930 | 17,0% | Contu y otros (2008) [45] |
| Pavo | Armenios de Sasun | 16 | 15,4% | Herrera y col. (2012) [28] |
| Irán | Norte | 33 | 15,2% | Regueiro y col. (2006) [46] |
| Moldavia | 268 | 14,6% | Varzari (2006) [47] | |
| Grecia | Nacional | 171 | 13,5% | Rey y otros (2008) [44] |
| Pavo | Oeste | 163 | 13,5% | Cinnioğlu y otros (2004) [43] |
| Rumania | Nacional | 54 | 13,0% | Varzari (2006) [47] |
| Croacia | Nacional | 89 | 12,4% | Battaglia y otros (2008) [41] |
| Pavo | Este | 208 | 12,0% | Cinnioğlu y otros (2004) [43] |
| Argelia | Noroeste ( zona de Orán ) | 102 | 11,8% | Robino y otros (2008) [48] |
| Rusia | Roslavl ( óblast de Smolensk ) | 107 | 11,2% | Balanovsky y otros (2008) [49] |
| Irak | Nacional | 139 | 10,8% | Al-Zahery y otros (2003) [50] |
| Nepal | Newar | 66 | 10,6% | Gayden y otros (2007) [51] |
| Bulgaria | Nacional | 808 | 10,5% | Karachanak y otros (2013) [52] |
| Serbia | Nacional | 100 | 10,0% | Belaresque y otros (2009) [3] |
| Líbano | Nacional | 914 | 7,3% | Zalloua y otros (2008) [53] |
| Túnez | Nacional | 601 | 0,3% | Bekada y otros (2013) [54] |
| Túnez | Túnez | 139 | 7,2% | Adams y otros (2008) [55] |
| Marruecos | Nacional | 760 | 3,5% | Bekada y otros (2013) [54] |
| Libia | Nacional | 83 | 0,0% | Bekada y otros (2013) [54] |
| Egipto | Nacional | 360 | 2,9% | Bekada y otros (2013) [54] |
| Argelia | Nacional | 156 | 7.0% | Bekada y otros (2013) [54] |
| Argelia | Argel , Tizi Ouzou | 46 | 6,5% | Adams y otros (2008) [55] |
| Bosnia y Herzegovina | Serbios | 81 | 6,2% | Marjanovic y otros (2005) [56] |
| Irán | Sur | 117 | 6.0% | Regueiro y col. (2006) [46] |
| Rusia | Repyevka ( Óblast de Vorónezh ) | 96 | 5,2% | Balanovsky y otros (2008) [49] |
| Emiratos Árabes Unidos | 164 | 3,7% | Cadenas y col. (2007) [57] | |
| Bosnia y Herzegovina | bosnios | 85 | 3,5% | Marjanovic y otros (2005) [56] |
| Pakistán | 176 | 2,8% | Sengupta y otros (2006) [58] | |
| Rusia | Bélgorod | 143 | 2,8% | Balanovsky y otros (2008) [49] |
| Rusia | Ostrov ( óblast de Pskov ) | 75 | 2,7% | Balanovsky y otros (2008) [49] |
| Rusia | Pristen ( Óblast de Kursk ) | 45 | 2,2% | Balanovsky y otros (2008) [49] |
| Bosnia y Herzegovina | Croatas | 90 | 2,2% | Marjanovic y otros (2005) [56] |
| Katar | 72 | 1,4% | Cadenas y col. (2007) [57] | |
| Porcelana | 128 | 0,8% | Sengupta y otros (2006) [58] | |
| India | varios | 728 | 0,5% | Sengupta y otros (2006) [58] |
| Croacia | Osijek | 29 | 0,0% | Battaglia y otros (2008) [41] |
| Yemen | 62 | 0,0% | Cadenas y col. (2007) [57] | |
| Tíbet | 156 | 0,0% | Gayden y otros (2007) [51] | |
| Nepal | Tamang | 45 | 0,0% | Gayden y otros (2007) [51] |
| Nepal | Katmandú | 77 | 0,0% | Gayden y otros (2007) [51] |
| Japón | 23 | 0,0% | Sengupta y otros (2006) [58] |
Subclados
R1b1a1a2a (R-L23)
En la actualidad, R-L23* (R1b1a1a2a*) se encuentra con mayor frecuencia en Europa, Anatolia y el Cáucaso.
R1b1a1a2a1 (R-L51)
R-L51* (R1b1a1a2a1*) ahora se concentra en un grupo geográfico centrado en el sur de Francia y el norte de Italia .
R1b1a1a2a1a (R-L151)
R-L151 (L151/PF6542, CTS7650/FGC44/PF6544/S1164, L11, L52/PF6541, P310/PF6546/S129, P311/PF6545/S128), también conocido como R1b1a1a2a1, y sus subclados, incluyen la mayoría de los machos con R1b en Europa occidental.
R1b1a1a2a1a1 (R-U106)
Este subclado se define por la presencia del SNP U106, también conocido como S21 y M405. [8] [59] Parece representar más del 25% de R1b en Europa. [8] En términos de porcentaje de la población total, su epicentro es Frisia , donde representa el 44% de la población. [60] En términos de números de población total, su epicentro es Europa Central , donde comprende el 60% de R1 combinado. [60] Véase también el haplogrupo R-Z18
| U106/S21/M405 |
| |||||||||||||||||||||||||||
Si bien este subclado de R1b se analiza con frecuencia entre los genealogistas genéticos , la siguiente tabla representa los hallazgos revisados por pares publicados hasta el momento en los artículos de 2007 de Myres et al. y Sims et al. [40] [59]
| Población | Tamaño de la muestra | R-M269 | R-U106 | R-U106-1 |
|---|---|---|---|---|
| Austria [40] | 22 | 27% | 23% | 0,0% |
| América Central y del Sur [40] | 33 | 0,0% | 0,0% | 0,0% |
| República Checa [40] | 36 | 28% | 14% | 0,0% |
| Dinamarca [40] | 113 | 34% | 17% | 0,9% |
| Europa del Este [40] | 44 | 5% | 0,0% | 0,0% |
| Inglaterra [40] | 138 | 57% | 20% | 1,4% |
| Francia [40] | 56 | 52% | 7% | 0,0% |
| Alemania [40] | 332 | 43% | 19% | 1,8% |
| Irlanda [40] | 102 | 80% | 6% | 0,0% |
| Italia [21] | 34 | 53% | 6% | 0,0% |
| Jordania [40] | 76 | 0,0% | 0,0% | 0,0% |
| Oriente Medio [40] | 43 | 0,0% | 0,0% | 0,0% |
| Países Bajos [40] | 94 | 54% | 35% | 2,1% |
| Oceanía [40] | 43 | 0,0% | 0,0% | 0,0% |
| Omán [40] | 29 | 0,0% | 0,0% | 0,0% |
| Pakistán [40] | 177 | 3% | 0,0% | 0,0% |
| Palestina [40] | 47 | 0,0% | 0,0% | 0,0% |
| Polonia [40] | 110 | 23% | 8% | 0,0% |
| Rusia [40] | 56 | 21% | 5,4% | 1,8% |
| Eslovenia [40] | 105 | 17% | 4% | 0,0% |
| Suiza [40] | 90 | 58% | 13% | 0,0% |
| Turquía [40] | 523 | 14% | 0,4% | 0,0% |
| Ucrania [40] | 32 | 25% | 9% | 0,0% |
| Estados Unidos (Europa) [59] | 125 | 46% | 15% | 0,8% |
| Estados Unidos (afroamericano) [59] | 118 | 14% | 2,5% | 0,8% |
R-P312
R1b1a1a2a1a2, más conocido como R-P312 (o R-S116), es uno de los tipos más comunes de R-M269 en Europa, junto con R-U106. Myres et al. lo describieron como originario y propagado desde el oeste de la cuenca del Rin. [21]
El R-P312 ha sido objeto de importantes estudios en curso sobre su compleja estructura interna.
| P312 | |
R-DF27
R-M153
R-M153 es un subclado de R-DF27 que se ha encontrado principalmente en vascos y gascones , entre los que representa una fracción considerable del acervo de ADN-Y, [55] [61] aunque también se encuentra ocasionalmente entre los ibéricos en general. La primera vez que se localizó (Bosch 2001 [62] ) se describió como H102 e incluía siete vascos y un andaluz .
R-M167 es un subclado de R-DF27 definido por la presencia del marcador M167. El primer autor que realizó pruebas para este marcador (mucho antes de que existiera la nomenclatura actual de haplogrupos) fue Hurles en 1999, quien realizó pruebas a 1158 hombres en varias poblaciones. [63] Lo encontró relativamente común entre los vascos (13/117: 11%) y los catalanes (7/32: 22%). Se encontraron otras apariciones entre otros franceses, británicos, españoles, bearneses y alemanes .
En 2000, Rosser et al., en un estudio que examinó a 3616 hombres en varias poblaciones [64] también probaron ese mismo marcador, nombrando el haplogrupo Hg22, y nuevamente se encontró principalmente entre vascos (19%), en frecuencias más bajas entre franceses (5%), bávaros (3%), españoles (2%), portugueses del sur (2%) y en ocurrencias únicas entre rumanos, eslovenos, holandeses, belgas e ingleses.::En 2001, Bosch describió este marcador como H103, en 5 vascos y 5 catalanes. [62] Estudios regionales posteriores lo han localizado en cantidades significativas en Asturias , Cantabria y Galicia , así como nuevamente entre vascos. [62] Se han reportado casos en las Azores . [ cita requerida ] En 2008, dos artículos de investigación de López-Parra [61] y Adams, [55] respectivamente, confirmaron una fuerte asociación con todos o la mayor parte de los Pirineos y la Iberia Oriental.
En un estudio más amplio realizado en Portugal en 2006, en el que se examinaron a 657 hombres, Beleza et al. confirmaron niveles bajos similares en todas las regiones principales, de 1,5% a 3,5%. [37]
R-L165
Este subclado se define por la presencia del marcador S68, también conocido como L165. Se encuentra en Inglaterra, Escandinavia y Escocia (en este país se encuentra principalmente en las Islas del Norte y las Hébridas Exteriores ). Por lo tanto, se ha sugerido que llegó a las Islas Británicas con los vikingos. [65]
R-U152
R-U152 se define por la presencia del marcador U152, también llamado S28. [8] Su existencia fue confirmada por Sims et al. (2007). [59] Myres et al. informan que este clado "es más frecuente (20-44%) en Suiza, Italia, Francia y Polonia occidental, con casos adicionales que superan el 15% en algunas regiones de Inglaterra y Alemania". [40] De manera similar, Cruciani et al. (2010) [66] informaron picos de frecuencia en el norte y centro de Italia y Francia. De una muestra de 135 hombres en Tirol, Austria, 9 dieron positivo para U152/S28. [67] Muy alejados de esta aparente área central, Myres et al. también mencionan una subpoblación en el norte de Bashkortostán , donde el 71% de los 70 hombres evaluados pertenecen a R-U152. Proponen que esto sea el resultado de un efecto fundador aislado . [21] King et al. (2014) informaron que cuatro descendientes vivos de Henry Somerset, quinto duque de Beaufort en la línea masculina dieron positivo para U-152. [68] Las muestras antiguas de las culturas de los vasos campaniformes , Hallstatt y Túmulus de Europa central pertenecían a este subclado. [14] [69] [70] Los latinos y etruscos de la Edad del Hierro analizados que datan entre 1000 y 100 a. C. pertenecían principalmente al haplogrupo R1b-U152 (incluidos los clados L2, Z56 y Z193). [71] [72]
R-L21
R-L21, también conocido como R-M529 y R-S145, [8] es más común en Irlanda , Escocia y Gales (es decir, al menos entre el 25 y el 50 % de sus poblaciones masculinas). [21] R-L21 tiene dos subclados principales: R-A5846 y R-S552 .
R-DF13
Un subclado primario de R-S552, R-DF13 –también conocido como R-S521, R-Z2542 y R-CTS8221– es uno de los subclados más comunes de R-L21. Al menos un estudio estimó que R-DF13 se encontraría entre más del 50% de los varones irlandeses vivos. Los siguientes son algunos de los subclados más comunes dentro de R-DF13.
- R-DF21 , un subclado primario de R-DF13, definido por la presencia del marcador DF21, también conocido como S192. R-DF21 representa aproximadamente el 10 % de todos los hombres L21 y tiene alrededor de 3000 años de antigüedad. [73]
- R-L159.2 es un subclado de R-DF13, definido por el marcador L159/S169. Se lo conoce como R-L159.2 debido a una mutación paralela no relacionada (L159.1), encontrada dentro del haplogrupo I2a1a1a (también conocido como I-L158 o I-M26). En consecuencia, algunos árboles de ADN-Y excluyen completamente a L159/S169, sobre la base de que puede ser un marcador poco confiable. Por ejemplo, a partir de 2024, Yfull se refiere a un subclado equivalente como R-FGC80001 (es decir, R-L21 > R-S552 > R-DF13 > R-Z255 > R-FGC80001). [74] R-L159.2 parece estar asociado con los Laigin , un grupo etnotribal , en cuyo honor se nombró al Reino de Leinster . Es común en machos de las zonas costeras que rodean el mar de Irlanda , incluido el oeste de Gales , la Isla de Man y las Hébridas ; R-L159.2 también se ha encontrado en niveles significativos en Noruega , el oeste y sur de Escocia continental , partes de Inglaterra , el noroeste de Francia y el norte de Dinamarca . [75]
- R-L193 : este subclado dentro de R-DF13 se define por la presencia del marcador L193. Muchos apellidos con este marcador están asociados geográficamente con la "Región Fronteriza" occidental de Escocia. Algunos otros apellidos tienen una asociación con las Tierras Altas. R-L193 es un subclado relativamente joven, probablemente nacido en los últimos 2000 años.
- El gen R-L226 , bajo el nombre de R-DF13, se define por la presencia del marcador L226, también conocido como S168. Comúnmente denominado tipo III irlandés, se concentra en el centro-oeste de Irlanda y está asociado con la familia Dál gCais . [76]
- R-L371 : un subclado dentro de R-DF13 definido por la presencia del marcador L371, a veces denominado "haplotipo modal galés". Está asociado con antiguos reyes y príncipes galeses. [77] [78] [79]
Véase también
Referencias
- ^ Balaresque et al. (2010), figura 1B: "Distribución geográfica de la frecuencia del haplogrupo hgR1b1b2, mostrada como una superficie de frecuencia espacial interpolada. Los círculos rellenos indican poblaciones para las que se dispone de datos de microsatélites y estimaciones de TMRCA. Los círculos sin relleno indican poblaciones incluidas solo para ilustrar la frecuencia de R1b1b2. Los códigos de población se definen en la Tabla 1".
- ^ ab "Las estimaciones medias para poblaciones individuales varían (Tabla 2), pero el valor más antiguo se encuentra en Turquía central (7.989 años [intervalo de confianza (IC) del 95 %: 5.661–11.014]), y el más reciente en Cornualles (5.460 años [3.764–7.777]). La estimación media para todo el conjunto de datos es de 6.512 años (IC del 95 %: 4.577–9.063 años), con una tasa de crecimiento del 1,95 % (1,02 %–3,30 %). Por lo tanto, vemos evidencia clara de una rápida expansión, que no puede haber comenzado antes del período Neolítico ". Balaresque et al. (2010).
- ^ abcdefghijklmnopqrstu vwxyz aa ab Balaresque P, Bowden GR, Adams SM, Leung HY, King TE, Rosser ZH, et al. (enero de 2010). Penny D (ed.). "Un origen predominantemente neolítico para los linajes paternos europeos". PLOS Biology . 8 (1): e1000285. doi : 10.1371/journal.pbio.1000285 . PMC 2799514 . PMID 20087410.
- ^ abcde Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (junio de 2015). "La migración masiva desde la estepa fue una fuente de lenguas indoeuropeas en Europa". Nature . 522 (7555): 207–11. arXiv : 1502.02783 . Bibcode :2015Natur.522..207H. doi :10.1038/nature14317. PMC 5048219 . PMID 25731166.
- ^ Lazaridis, Iosif; et al. (2022). "La historia genética del Arco Austral: un puente entre Asia Occidental y Europa". Science . 377 (6609): Material complementario, p.332. doi :10.1126/science.abm4247. PMC 10064553 . PMID 36007055.
Dado que dentro de la filogenia de R-M269 (R-PF7562, (se refiere a R-L51, R-Z2103) tanto R-PF7562 como R-Z2103 tienen sus primeros ejemplos en el Cáucaso Norte y la estepa al norte, la hipótesis más probable es que todo el clado R-M269 se originó allí también, con R-L51 representando un linaje que eventualmente tuvo mucho éxito en Europa continental, R-PF7562 un linaje que no alcanzó la prominencia de sus parientes, y R-Z2103 tuvo mucho éxito (brevemente) como parte de la cultura Yamnaya y sus ramificaciones.
- ^ fechas según los árboles ISOGG para cada año respectivo.
- ^ Semino O, Passarino G, Oefner PJ, Lin AA, Arbuzova S, Beckman LE, et al. (Noviembre de 2000). "El legado genético del Homo sapiens sapiens paleolítico en los europeos actuales: una perspectiva del cromosoma Y". Ciencia . 290 (5494): 1155–9. Código Bib : 2000 Ciencia... 290.1155S. doi : 10.1126/ciencia.290.5494.1155. PMID 11073453.
- ^ abcde Sociedad Internacional de Genealogía Genética (ISOGG) – Haplogrupo R del ADN-Y y sus subclados
- ^ Arredi B, Poloni ES, Tyler-Smith C (2007). "El poblamiento de Europa". En Crawford, Michael H. (ed.). Genética antropológica: teoría, métodos y aplicaciones . Cambridge, Reino Unido: Cambridge University Press. p. 394. ISBN 978-0-521-54697-3.
- ^ Cruciani F, Trombetta B, Antonelli C, Pascone R, Valesini G, Scalzi V, et al. (junio de 2011). "Fuerte diferenciación intra e intercontinental revelada por los SNP del cromosoma Y M269, U106 y U152". Forensic Science International. Genética . 5 (3): e49–52. doi :10.1016/j.fsigen.2010.07.006. PMID 20732840.
- ^ Allentoft ME, Sikora M, Sjögren KG, Rasmussen S, Rasmussen M, Stenderup J, et al. (junio de 2015). "Genómica de poblaciones de Eurasia de la Edad del Bronce". Naturaleza . 522 (7555): 167–172. Código Bib :2015Natur.522..167A. doi : 10.1038/naturaleza14507. PMID 26062507. S2CID 4399103.
- ^ Busby GB, Brisighelli F, Sánchez-Diz P, Ramos-Luis E, Martinez-Cadenas C, Thomas MG, et al. (marzo de 2012). "El poblamiento de Europa y la advertencia sobre el linaje del cromosoma Y R-M269". Actas. Ciencias Biológicas . 279 (1730): 884–892. doi :10.1098/rspb.2011.1044. PMC 3259916 . PMID 21865258.
- ^ Mathieson I, Lazaridis I, Rohland N, Mallick S, Patterson N, Roodenberg SA, et al. (diciembre de 2015). "Patrones de selección en todo el genoma en 230 antiguos euroasiáticos". Nature . 528 (7583): 499–503. Bibcode :2015Natur.528..499M. doi :10.1038/nature16152. PMC 4918750 . PMID 26595274.
- ^ abc Olalde I, Brace S, Allentoft ME, Armit I, Kristiansen K, Booth T, et al. (marzo de 2018). "El fenómeno Beaker y la transformación genómica del noroeste de Europa". Nature . 555 (7695): 190–196. Bibcode :2018Natur.555..190O. doi :10.1038/nature25738. PMC 5973796 . PMID 29466337.
- ^ Anthony DW (6 de diciembre de 2019). "ADN antiguo, redes de apareamiento y la división de Anatolia". En Serangeli M, Olander T (eds.). Dispersiones y diversificación . BRILL. págs. 21–53. doi :10.1163/9789004416192_003. ISBN 978-90-04-41619-2.S2CID213909442 .
- ^ Lazaridis, Iosif; et al. (2022). "La historia genética del Arco Austral: un puente entre Asia Occidental y Europa". Science . 377 (6609): Material complementario, p.332. doi :10.1126/science.abm4247. PMC 10064553 . PMID 36007055.
Dado que dentro de la filogenia de R-M269 (R-PF7562, (se refiere a R-L51, R-Z2103) tanto R-PF7562 como R-Z2103 tienen sus primeros ejemplos en el Cáucaso Norte y la estepa al norte, la hipótesis más probable es que todo el clado R-M269 se originó allí también, con R-L51 representando un linaje que eventualmente tuvo mucho éxito en Europa continental, R-PF7562 un linaje que no alcanzó la prominencia de sus parientes, y R-Z2103 tuvo mucho éxito (brevemente) como parte de la cultura Yamnaya y sus ramificaciones.
- ^ Hammer M (2013). Orígenes de la diversidad de R-M269 en Europa . Novena conferencia anual de FamilyTreeDNA.
- ^ Sjögren KG, Olalde I, Carver S, Allentoft ME, Knowles T, Kroonen G, et al. (11 de diciembre de 2019). "Parentesco y organización social en la Europa de la Edad del Cobre. Un análisis interdisciplinario de la arqueología, el ADN, los isótopos y la antropología de dos cementerios de Vaso Campaniforme". PLOS ONE . 15 (11): e0241278. bioRxiv 10.1101/863944 . doi : 10.1371/journal.pone.0241278 . hdl : 10261/236801 . PMC 7668604 . PMID 33196640. S2CID 212833639.
- ^ Underhill PA, Shen P, Lin AA, Jin L, Passarino G, Yang WH, et al. (Noviembre de 2000). "Variación de la secuencia del cromosoma Y y la historia de las poblaciones humanas". Genética de la Naturaleza . 26 (3): 358–61. doi :10.1038/81685. PMID 11062480. S2CID 12893406.
- ^ ab Lobov AS, et al. (2009). "Estructura del acervo genético de las subpoblaciones bashkires" (PDF) (en ruso). Archivado desde el original (PDF) el 16 de agosto de 2011.
- ^ abcdefghijkl Myres NM, Rootsi S, Lin AA, Järve M, King RJ, Kutuev I, et al. (Enero de 2011). "Un importante efecto fundador de la era del Holoceno del haplogrupo R1b del cromosoma Y en Europa central y occidental". Revista europea de genética humana . 19 (1): 95-101. doi :10.1038/ejhg.2010.146. PMC 3039512 . PMID 20736979.
- ^ ab Grugni V, Raveane A, Mattioli F, Battaglia V, Sala C, Toniolo D, et al. (febrero de 2018). "Reconstrucción de la historia genética de los italianos: nuevos conocimientos desde una perspectiva masculina (cromosoma Y)". Anales de biología humana . 45 (1): 44–56. doi :10.1080/03014460.2017.1409801. PMID 29382284. S2CID 43501209.
- ^ Robino C, Crobu F, Di Gaetano C, Bekada A, Benhamamouch S, Cerutti N, et al. (mayo de 2008). "Análisis de haplogrupos de SNP del cromosoma Y y haplotipos de STR en una muestra de población argelina". Revista Internacional de Medicina Legal . 122 (3): 251–255. doi :10.1007/s00414-007-0203-5. PMID 17909833. S2CID 11556974.
- ^ Wood ET, Stover DA, Ehret C, Destro-Bisol G, Spedini G, McLeod H, et al. (julio de 2005). "Patrones contrastantes de variación del cromosoma Y y del ADNmt en África: evidencia de procesos demográficos con sesgo sexual". Revista Europea de Genética Humana . 13 (7): 867–876. doi : 10.1038/sj.ejhg.5201408 . PMID 15856073. S2CID 20279122.
- ^ Ordóñez AC, Fregel R, Trujillo-Mederos A, Hervella M, de-la-Rúa C, Arnay-de-la-Rosa M (2017). "Estudios genéticos sobre la población prehispánica enterrada en la cueva de Punta Azul (El Hierro, Canarias)". Revista de Ciencias Arqueológicas . 78 : 20–28. Código Bib : 2017JArSc..78...20O. doi :10.1016/j.jas.2016.11.004.
- ^ ab Grugni V, Battaglia V, Hooshiar Kashani B, Parolo S, Al-Zahery N, Achilli A, et al. (2012). "Antiguos acontecimientos migratorios en Oriente Medio: nuevas pistas a partir de la variación del cromosoma Y de los iraníes modernos". PLOS ONE . 7 (7): e41252. Bibcode :2012PLoSO...741252G. doi : 10.1371/journal.pone.0041252 . PMC 3399854 . PMID 22815981.
- ^ Actas de la Academia Rusa de Genealogía del ADN , 3, 1676–1695 (en ruso).
- ^ abcde Herrera KJ, Lowery RK, Hadden L, Calderon S, Chiou C, Yepiskoposyan L, Regueiro M, Underhill PA, Herrera RJ (marzo de 2012). "Las señales patrilineales neolíticas indican que la meseta armenia fue repoblada por agricultores". Revista Europea de Genética Humana . 20 (3): 313–20. doi :10.1038/ejhg.2011.192. PMC 3286660 . PMID 22085901.
- ^ Трофимова Натал'я Вадимовна (febrero de 2015), "Изменчивость Митохондриальной ДНК и Y-Хромосомы в Популяциях Волго-Уральског о Региона" Archivado el 2 de abril de 2017 en la Wayback Machine. ("Variación del ADN mitocondrial y del cromosoma Y en la población de la región Volga-Ural"). Автореферат . диссертации на соискание ученой степени кандидата биологических наук. Уфа – 2015.
- ^ Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (junio de 2015). "La migración masiva desde la estepa fue una fuente de lenguas indoeuropeas en Europa". Nature . 522 (7555): 207–11. arXiv : 1502.02783 . Bibcode :2015Natur.522..207H. bioRxiv 10.1101/013433 . doi :10.1038/nature14317. PMC 5048219 . PMID 25731166.
- ^ Morelli L, Contu D, Santoni F, Whalen MB, Francalacci P, Cucca F (abril de 2010). Lalueza-Fox C (ed.). "Una comparación de la variación del cromosoma Y en Cerdeña y Anatolia es más consistente con la difusión cultural que con la difusión démica de la agricultura". MÁS UNO . 5 (4): e10419. Código Bib : 2010PLoSO...510419M. doi : 10.1371/journal.pone.0010419 . PMC 2861676 . PMID 20454687.
- ^ Árbol ISOGG a partir de 2017 (isogg.org)
- ^ Vanek D, Saskova L, Koch H (junio de 2009). "Análisis de parentesco y cromosoma Y de restos humanos del siglo VII: nuevo procedimiento de extracción y tipificación de ADN para material antiguo". Revista Médica Croata . 3. 50 (3): 286–95. doi :10.3325/cmj.2009.50.286. PMC 2702742 . PMID 19480023.
- ^ Moore LT, McEvoy B, Cape E, Simms K, Bradley DG (febrero de 2006). "Una firma del cromosoma Y de hegemonía en la Irlanda gaélica". American Journal of Human Genetics . 78 (2): 334–8. doi :10.1086/500055. PMC 1380239 . PMID 16358217.
- ^ ab Di Giacomo F, Luca F, Anagnou N, Ciavarella G, Corbo RM, Cresta M, et al. (septiembre de 2003). "Los patrones clinales de la diversidad cromosómica humana Y en Italia continental y Grecia están dominados por los efectos de deriva y fundador". Filogenética molecular y evolución . 28 (3): 387–395. Bibcode :2003MolPE..28..387D. CiteSeerX 10.1.1.553.5711 . doi :10.1016/s1055-7903(03)00016-2. PMID 12927125.
- ^ Bryc K, Velez C, Karafet T, Moreno-Estrada A, Reynolds A, Auton A, et al. (mayo de 2010). "Documento de coloquio: patrones de estructura y mezcla de la población en todo el genoma entre poblaciones hispanas/latinas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (Suppl 2): 8954–61. Bibcode :2010PNAS..107.8954B. doi : 10.1073/pnas.0914618107 . PMC 3024022 . PMID 20445096.
- ^ ab Beleza S, Gusmão L, Lopes A, Alves C, Gomes I, Giouzeli M, et al. (marzo de 2006). "Historia microfilogeográfica y demográfica de los linajes masculinos portugueses". Anales de genética humana . 70 (parte 2): 181–94. doi :10.1111/j.1529-8817.2005.00221.x. PMID 16626329. S2CID 4652154.
395/657
- ^ Boattini A, Martinez-Cruz B, Sarno S, Harmant C, Useli A, Sanz P, Yang-Yao D, Manry J, Ciani G, Luiselli D, Quintana-Murci L, Comas D, Pettener D (2013). "Los marcadores uniparentales en Italia revelan una estructura genética sesgada por el sexo y diferentes estratos históricos". MÁS UNO . 8 (5): e65441. Código Bib : 2013PLoSO...865441B. doi : 10.1371/journal.pone.0065441 . PMC 3666984 . PMID 23734255.
- ^ ab Tofanelli S, Brisighelli F, Anagnostou P, Busby GB, Ferri G, Thomas MG, Taglioli L, Rudan I, Zemunik T, Hayward C, Bolnick D, Romano V, Cali F, Luiselli D, Shepherd GB, Tusa S, Facella A, Capelli C (marzo de 2016). "Los griegos en Occidente: firmas genéticas de la colonización helénica en el sur de Italia y Sicilia". Revista Europea de Genética Humana . 24 (3): 429–36. doi :10.1038/ejhg.2015.124. PMC 4757772 . PMID 26173964.
- ^ abcdefghijklmnopqrstu vwxy Myres NM, Ekins JE, Lin AA, Cavalli-Sforza LL, Woodward SR, Underhill PA (agosto de 2007). "Los alelos no consensuados de repetición corta en tándem DYS458.2 del cromosoma Y aparecen de forma independiente en ambos haplogrupos binarios J1-M267 y R1b3-M405". Revista Médica Croata . 48 (4): 450–9. PMC 2080563 . PMID 17696299.
- ^ abcde Battaglia V, Fornarino S, Al-Zahery N, Olivieri A, Pala M, Myres NM, et al. (junio de 2009). "Evidencia cromosómica Y de la difusión cultural de la agricultura en el sudeste de Europa". Revista Europea de Genética Humana . 17 (6): 820–30. doi :10.1038/ejhg.2008.249. PMC 2947100 . PMID 19107149.
- ^ Pericić M, Lauc LB, Klarić IM, Rootsi S, Janićijevic B, Rudan I, et al. (octubre de 2005). "El análisis filogenético de alta resolución del sudeste de Europa rastrea los principales episodios de flujo genético paterno entre las poblaciones eslavas". Biología molecular y evolución . 22 (10): 1964–75. doi : 10.1093/molbev/msi185 . PMID 15944443.
- ^ abc Cinnioğlu C, King R, Kivisild T, Kalfoğlu E, Atasoy S, Cavalleri GL, et al. (enero de 2004). "Excavando los estratos de haplotipos del cromosoma Y en Anatolia" (PDF) . Human Genetics . 114 (2): 127–48. doi :10.1007/s00439-003-1031-4. PMID 14586639. S2CID 10763736. Archivado desde el original (PDF) el 19 de junio de 2006.
- ^ ab King RJ, Ozcan SS, Carter T, Kalfoğlu E, Atasoy S, Triantaphyllidis C, et al. (marzo de 2008). "Influencias anatolias del cromosoma Y diferencial en el Neolítico griego y cretense". Anales de genética humana . 72 (parte 2): 205–14. doi :10.1111/j.1469-1809.2007.00414.x. PMID 18269686. S2CID 22406638.
- ^ Contu D, Morelli L, Santoni F, Foster JW, Francalacci P, Cucca F (enero de 2008). Hawks J (ed.). "Evidencia basada en el cromosoma Y del origen preneolítico de la población sarda, genéticamente homogénea pero diversa: inferencia para los análisis de asociación". PLOS ONE . 3 (1): e1430. Bibcode :2008PLoSO...3.1430C. doi : 10.1371/journal.pone.0001430 . PMC 2174525 . PMID 18183308.
174/930
- ^ ab Regueiro M, Cadenas AM, Gayden T, Underhill PA, Herrera RJ (2006). "Irán: nexo tricontinental para la migración impulsada por el cromosoma Y". Herencia humana . 61 (3): 132–43. doi :10.1159/000093774. PMID 16770078. S2CID 7017701.
- ^ ab Varzari A (2006). Historia de la población de los Dniéster-Cárpatos: evidencia de la inserción de Alu y polimorfismos del cromosoma Y (PDF) (Disertación). Universidad Ludwig-Maximilians de Múnich.
- ^ Robino C, Crobu F, Di Gaetano C, Bekada A, Benhamamouch S, Cerutti N, et al. (mayo de 2008). "Análisis de haplogrupos de SNP del cromosoma Y y haplotipos de STR en una muestra de población argelina". Revista Internacional de Medicina Legal . 122 (3): 251–5. doi :10.1007/s00414-007-0203-5. PMID 17909833. S2CID 11556974.
- ^ abcde Balanovsky O, Rootsi S, Pshenichnov A, Kivisild T, Churnosov M, Evseeva I, et al. (Enero de 2008). "Dos fuentes de la herencia patrilineal rusa en su contexto euroasiático". Revista Estadounidense de Genética Humana . 82 (1): 236–50. doi :10.1016/j.ajhg.2007.09.019. PMC 2253976 . PMID 18179905.
- ^ Al-Zahery N, Semino O, Benuzzi G, Magri C, Passarino G, Torroni A, Santachiara-Benerecetti AS (septiembre de 2003). "Polimorfismos del cromosoma Y y del ADNmt en Irak, una encrucijada de la dispersión humana temprana y de las migraciones post-neolíticas" (PDF) . Filogenética molecular y evolución . 28 (3): 458–72. Código bibliográfico :2003MolPE..28..458A. doi :10.1016/S1055-7903(03)00039-3. PMID 12927131. Archivado desde el original (PDF) el 20 de enero de 2017 . Consultado el 17 de marzo de 2017 .
16/139
- ^ abcd Gayden T, Cadenas AM, Regueiro M, Singh NB, Zhivotovsky LA, Underhill PA, Cavalli-Sforza LL, Herrera RJ (mayo de 2007). "El Himalaya como barrera direccional para el flujo genético". American Journal of Human Genetics . 80 (5): 884–94. doi :10.1086/516757. PMC 1852741 . PMID 17436243.
- ^ Karachanak S, Grugni V, Fornarino S, Nesheva D, Al-Zahery N, Battaglia V, Carossa V, Yordanov Y, Torroni A, Galabov AS, Toncheva D, Semino O (2013). "Diversidad del cromosoma Y en los búlgaros modernos: nuevas pistas sobre su ascendencia". MÁS UNO . 8 (3): e56779. Código Bib : 2013PLoSO...856779K. doi : 10.1371/journal.pone.0056779 . PMC 3590186 . PMID 23483890.
- ^ Zalloua PA, Xue Y, Khalife J, Makhoul N, Debiane L, Platt DE, Royyuru AK, Herrera RJ, Hernanz DF, Blue-Smith J, Wells RS, Comas D, Bertranpetit J, Tyler-Smith C (abril de 2008). "La diversidad del cromosoma Y en el Líbano está estructurada por eventos históricos recientes". American Journal of Human Genetics . 82 (4): 873–82. doi :10.1016/j.ajhg.2008.01.020. PMC 2427286 . PMID 18374297.
- ^ abcde Bekada A, Fregel R, Cabrera VM, Larruga JM, Pestano J, Benhamamouch S, González AM (19 de febrero de 2013). "Introducción de los perfiles argelinos de ADN mitocondrial y cromosoma Y en el paisaje del norte de África". PLOS ONE . 8 (2): e56775. Bibcode :2013PLoSO...856775B. doi : 10.1371/journal.pone.0056775 . PMC 3576335 . PMID 23431392.
- ^ abcd Adams SM, Bosch E, Balaresque PL, Ballereau SJ, Lee AC, Arroyo E, et al. (diciembre de 2008). "El legado genético de la diversidad religiosa y la intolerancia: linajes paternos de cristianos, judíos y musulmanes en la península Ibérica". American Journal of Human Genetics . 83 (6): 725–36. doi :10.1016/j.ajhg.2008.11.007. PMC 2668061 . PMID 19061982.
- ^ abc Marjanovic D, Fornarino S, Montagna S, Primorac D, Hadziselimovic R, Vidovic S, Pojskic N, Battaglia V, Achilli A, Drobnic K, Andjelinovic S, Torroni A, Santachiara-Benerecetti AS, Semino O (noviembre de 2005). "El poblamiento de la Bosnia-Herzegovina moderna: haplogrupos del cromosoma Y en los tres grupos étnicos principales". Anales de genética humana . 69 (parte 6): 757–63. doi :10.1111/j.1529-8817.2005.00190.x. PMID 16266413. S2CID 36632274.
- ^ abc Cadenas AM, Zhivotovsky LA, Cavalli-Sforza LL, Underhill PA, Herrera RJ (marzo de 2008). "La diversidad del cromosoma Y caracteriza al Golfo de Omán". Revista Europea de Genética Humana . 16 (3): 374–86. doi : 10.1038/sj.ejhg.5201934 . PMID 17928816.
- ^ abcd Sengupta S, Zhivotovsky LA, King R, Mehdi SQ, Edmonds CA, Chow CE, et al. (febrero de 2006). "La polaridad y la temporalidad de las distribuciones de alta resolución del cromosoma Y en la India identifican expansiones tanto indígenas como exógenas y revelan una influencia genética menor de los pastores de Asia central". American Journal of Human Genetics . 78 (2): 202–21. doi :10.1086/499411. PMC 1380230 . PMID 16400607.
8/176 R-M73 y 5/176 R-M269 para un total de 13/176 R1b en Pakistán y 4/728 R-M269 en la India
- ^ abcde Sims LM, Garvey D, Ballantyne J (enero de 2007). "Las subpoblaciones dentro de los principales haplogrupos derivados de Europa y África R1b3 y E3a se diferencian por SNP-Y previamente no definidos filogenéticamente". Human Mutation . 28 (1): 97. doi : 10.1002/humu.9469 . PMID 17154278. S2CID 34556775.
- ^ ab "Orígenes de la diversidad de R-M269 en Europa" (PDF) .
- ^ ab López-Parra AM, Gusmão L, Tavares L, Baeza C, Amorim A, Mesa MS, Prata MJ, Arroyo-Pardo E (enero de 2009). "En busca de los sustratos genéticos pre y posneolíticos en Iberia: evidencia del cromosoma Y en poblaciones pirenaicas". Anales de genética humana . 73 (1): 42–53. doi :10.1111/j.1469-1809.2008.00478.x. PMID 18803634. S2CID 43273988.
- ^ abc Bosch E, Calafell F, Comas D, Oefner PJ, Underhill PA, Bertranpetit J (abril de 2001). "El análisis de alta resolución de la variación del cromosoma Y humano muestra una marcada discontinuidad y un flujo genético limitado entre el noroeste de África y la península Ibérica". American Journal of Human Genetics . 68 (4): 1019–29. doi :10.1086/319521. PMC 1275654 . PMID 11254456.
- ^ Hurles ME, Veitia R, Arroyo E, Armenteros M, Bertranpetit J, Pérez-Lezaun A, et al. (noviembre de 1999). "Flujo genético mediado por el sexo masculino reciente a través de una barrera lingüística en Iberia, sugerido por el análisis de un polimorfismo del ADN del cromosoma Y". American Journal of Human Genetics . 65 (5): 1437–48. doi :10.1086/302617. PMC 1288297 . PMID 10521311.
- ^ Rosser ZH, Zerjal T, Hurles ME, Adojaan M, Alavantic D, Amorim A, et al. (diciembre de 2000). "La diversidad del cromosoma Y en Europa es clinal y está influida principalmente por la geografía, más que por el idioma". American Journal of Human Genetics . 67 (6): 1526–43. doi :10.1086/316890. PMC 1287948 . PMID 11078479.
- ^ Moffat A , Wilson JF (2011). Los escoceses: un viaje genético . Birlinn . págs. 181–182, 192. ISBN 978-0-85790-020-3.
- ^ Cruciani F, Trombetta B, Antonelli C, Pascone R, Valesini G, Scalzi V, Vona G, Melegh B, Zagradisnik B, Assum G, Efremov GD, Sellitto D, Scozzari R (junio de 2011). "Fuerte diferenciación intra e intercontinental revelada por los SNP M269, U106 y U152 del cromosoma Y". Internacional de Ciencias Forenses. Genética . 5 (3): e49–52. doi :10.1016/j.fsigen.2010.07.006. PMID 20732840.
- ^ Niederstätter H, Berger B, Erhart D, Parson W (agosto de 2008). "Los SNP-Y introducidos recientemente mejoran la resolución dentro del haplogrupo R1b del cromosoma Y en una muestra de población de Europa central (Tirol, Austria)". Forensic Science International: Genetics Supplement Series . 1 : 226–227. doi : 10.1016/j.fsigss.2007.10.158 .
- ↑ Henry Somerset descendía a su vez por línea patrilineal de Juan de Gante (1340-1399), hijo del rey Eduardo III (1312-1377). En el contexto del análisis de los restos de Ricardo III , que resultaron pertenecer al haplogrupo G2, se discutió la posibilidad de un evento de falsa paternidad , muy probablemente entre Eduardo III y Henry Somerset; posiblemente confirmando rumores en el sentido de que Juan de Gante era ilegítimo (Jonathan Sumption, Divided Houses: The Hundred Years War III , 2009, p. 274). King TE, Fortes GG, Balaresque P, Thomas MG, Balding D, Maisano Delser P, Neumann R, Parson W, Knapp M, Walsh S, Tonasso L, Holt J, Kayser M, Appleby J, Forster P, Ekserdjian D, Hofreiter M, Schürer K (diciembre de 2014). "Identificación de los restos del rey Ricardo III". Nature Communications . 5 (5631): 5631. Bibcode :2014NatCo...5.5631K. doi :10.1038/ncomms6631. PMC 4268703 . PMID 25463651. "Los haplotipos del cromosoma Y de los parientes de línea masculina y los restos no coinciden, lo que podría atribuirse a un evento de falsa paternidad ocurrido en cualquiera de las generaciones intermedias".
- ^ Allentoft ME, Sikora M, Sjögren KG, Rasmussen S, Rasmussen M, Stenderup J, et al. (junio de 2015). "Genómica de poblaciones de Eurasia de la Edad del Bronce". Naturaleza . 522 (7555): 167–72. Código Bib :2015Natur.522..167A. doi : 10.1038/naturaleza14507. PMID 26062507. S2CID 4399103.
- ^ Damgaard PB, Marchi N, Rasmussen S, Peyrot M, Renaud G, Korneliussen T, et al. (mayo de 2018). "137 genomas humanos antiguos de las estepas euroasiáticas". Nature . 557 (7705): 369–374. Bibcode :2018Natur.557..369D. doi :10.1038/s41586-018-0094-2. hdl : 1887/3202709 . PMID 29743675. S2CID 13670282.
- ^ Antonio ML, Gao Z, Moots HM, Lucci M, Candilio F, Sawyer S, et al. (noviembre de 2019). "La antigua Roma: una encrucijada genética de Europa y el Mediterráneo". Science . 366 (6466): 708–714. Bibcode :2019Sci...366..708A. doi :10.1126/science.aay6826. PMC 7093155 . PMID 31699931.
- ^ Posth C, Zaro V, Spyrou MA, Vai S, Gnecchi-Ruscone GA, Modi A, et al. (septiembre de 2021). "El origen y el legado de los etruscos a través de un transecto temporal arqueogenómico de 2000 años". Science Advances . 7 (39). Asociación Estadounidense para el Avance de la Ciencia: eabi7673. Bibcode :2021SciA....7.7673P. doi :10.1126/sciadv.abi7673. PMC 8462907 . PMID 34559560.
- ^ "Proyecto R-DF21 y Subclados".
- ^ Yfull, 2012–24, R-FGC80001(Acceso: 9 de abril de 2024).
- ^ "Objetivos del Proyecto R-L159".
- ^ Wright DM (2009). "Un conjunto de valores distintivos de marcadores define una firma Y-STR para familias gaélicas dalcasianas". Journal of Genetic Genealogy . Archivado desde el original el 24 de agosto de 2012. Consultado el 17 de marzo de 2017 .
- ^ Bevan N (25 de septiembre de 2014). "Las raras raíces genéticas de Dafydd Iwan reveladas en un nuevo proyecto". Gales en línea . Consultado el 5 de abril de 2018 .
- ^ Bodden T (26 de septiembre de 2014). "Dafydd Iwan 'descendiente de reyes galeses' que gobernaron en Inglaterra". northwales . Consultado el 5 de abril de 2018 .
- ^ "Family Tree DNA - Título del sitio web de My FamilyTree DNA Project". www.familytreedna.com . Consultado el 5 de abril de 2018 .
