Historia genética de Europa
La historia genética de Europa incluye información sobre la formación, etnogénesis y otra información específica del ADN sobre las poblaciones indígenas o que viven en Europa .
Los linajes de humanos modernos tempranos europeos (EEMH) entre 40 y 26 ka ( Auriñaciense ) todavía eran parte de una gran "meta-población" de Eurasia occidental, relacionada con las poblaciones de Asia central y occidental. [2] La divergencia en subpoblaciones genéticamente distintas dentro de Eurasia occidental es el resultado de una mayor presión de selección y efectos fundadores durante el Último Máximo Glacial (LGM, Gravetiense ). [3]
A finales del LGM, después de 20 ka, un linaje de Europa occidental, denominado cazador-recolector de Europa occidental (WHG) emergió del refugio solutrense durante el Mesolítico europeo . [4] Estas culturas de cazadores-recolectores mesolíticos son posteriormente reemplazadas en la Revolución Neolítica como resultado de la llegada de linajes de agricultores europeos tempranos (EEF) derivados de poblaciones mesolíticas de Asia occidental ( Anatolia y el Cáucaso ). [5] En la Edad del Bronce europea , hubo nuevamente reemplazos de población sustanciales en partes de Europa por la intrusión de linajes de pastores esteparios occidentales (WSH) de las estepas póntico-caspias , que surgieron de la mezcla entre cazadores-recolectores orientales (EHG) y pueblos relacionados con los habitantes del Cercano Oriente. Estos reemplazos de población de la Edad del Bronce están asociados con las culturas del vaso campaniforme y de la cerámica cordada arqueológicamente y con la expansión indoeuropea lingüísticamente. [6] [7]
Como resultado de los movimientos de población durante el Mesolítico hasta la Edad del Bronce, las poblaciones europeas modernas se distinguen por diferencias en la ascendencia WHG, EEF y Eurasia del Norte Antiguo (ANE). [8] [9] [10] Las tasas de mezcla variaron geográficamente; a finales del Neolítico, la ascendencia WHG en los agricultores de Hungría rondaba el 10%, en Alemania alrededor del 25% y en Iberia hasta el 50%. [11] La contribución de EEF es más significativa en la Europa mediterránea y disminuye hacia el norte y noreste de Europa, donde la ascendencia WHG es más fuerte; los sardos se consideran el grupo europeo más cercano a la población de EEF.
La etnogénesis de los grupos étnicos modernos de Europa en el período histórico está asociada con numerosos eventos de mezcla, principalmente aquellos asociados con el período de migración y la decadencia del Imperio Romano , asociados con las expansiones germánica , nórdica y eslava.
La investigación sobre la historia genética de Europa se hizo posible en la segunda mitad del siglo XX, pero no arrojó resultados de alta resolución hasta la década de 1990. En la década de 1990, se pudieron obtener resultados preliminares, pero se limitaron en su mayoría a estudios de linajes mitocondriales y del cromosoma Y. El ADN autosómico se volvió más accesible en la década de 2000 y, desde mediados de la década de 2010, se han publicado a un ritmo acelerado resultados de una resolución previamente inalcanzable, muchos de ellos basados en análisis del genoma completo del ADN antiguo. [12] [13]
Prehistoria
Debido a la selección natural, el porcentaje de ADN neandertal en los antiguos europeos disminuyó gradualmente con el tiempo. Desde hace 45.000 años hasta hace 7.000 años, el porcentaje se redujo de alrededor del 3-6% al 2%. [13] La eliminación de alelos derivados de neandertales se produjo con mayor frecuencia en torno a los genes que en otras partes del genoma. [13]
Paleolítico
Los neandertales habitaron gran parte de Europa y Asia occidental desde hace 130.000 años. Existieron en Europa hasta hace 30.000 años. Finalmente fueron reemplazados por los humanos anatómicamente modernos (AMH; a veces conocidos como Cro-Magnons ), que comenzaron a aparecer en Europa hace unos 40.000 años. Dado que las dos especies de homínidos probablemente coexistieron en Europa, los antropólogos se han preguntado durante mucho tiempo si las dos interactuaron. [14] La pregunta se resolvió recién en 2010, cuando se estableció que las poblaciones euroasiáticas exhiben una mezcla neandertal, estimada en un 1,5-2,1% en promedio. [15] La pregunta ahora era si esta mezcla había tenido lugar en Europa, o más bien en el Levante, antes de la migración de los AMH a Europa.
También se ha especulado sobre la herencia de genes específicos de los neandertales. Por ejemplo, un locus MAPT 17q 21.3 que se divide en linajes genéticos profundos H1 y H2. Dado que el linaje H2 parece restringido a las poblaciones europeas, varios autores han defendido la herencia de los neandertales a partir de 2005. [16] [17] [18] [19] [20] Sin embargo, los resultados preliminares de la secuenciación del genoma neandertal completo en ese momento (2009), no lograron descubrir evidencia de mestizaje entre neandertales y humanos modernos. [21] [22] En 2010, los hallazgos de Svante Pääbo (Instituto Max Planck de Antropología Evolutiva en Leipzig, Alemania), Richard E. Green (Universidad de California, Santa Cruz) y David Reich (Facultad de Medicina de Harvard), que compararon el material genético de los huesos de tres neandertales con el de cinco humanos modernos, mostraron una relación entre los neandertales y la gente moderna fuera de África.
Paleolítico superior

Se cree que los humanos modernos comenzaron a habitar Europa durante el Paleolítico superior hace unos 40.000 años. Algunas evidencias muestran la expansión de la cultura auriñaciense . [23] : 59
Desde una perspectiva puramente patrilineal, del cromosoma Y , parece que los haplogrupos C1a2 , F y K2a pueden ser los que tienen una presencia más antigua en Europa. Se han encontrado en algunos de los restos humanos más antiguos secuenciados de la era paleolítica . Sin embargo, otros haplogrupos son mucho más comunes entre los varones europeos modernos, debido a cambios demográficos posteriores.
Actualmente, se ha descubierto que la muestra más antigua del haplogrupo I (M170), que ahora es relativamente común y está muy extendido en Europa, es Krems WA3 de la Baja Austria, que data de hace unos 30-31 000 años. [24] Aproximadamente en esta época, también apareció una cultura del Paleolítico Superior, conocida como Gravetiense . [25]
Las investigaciones anteriores sobre el ADN-Y se habían centrado en el haplogrupo R1 (M173): el linaje más numeroso entre los varones europeos vivos; también se creía que el R1 había surgido hace unos 40.000 años en Asia Central . [25] [26] Sin embargo, ahora se estima que el R1 surgió sustancialmente más recientemente: un estudio de 2008 fechó el ancestro común más reciente del haplogrupo IJ en 38.500 años y el haplogrupo R1 en 18.000 años. Esto sugirió que los colonos del haplogrupo IJ formaron la primera ola y el haplogrupo R1 llegó mucho más tarde. [27]
Así, los datos genéticos sugieren que, al menos desde la perspectiva de la ascendencia patrilineal, grupos separados de humanos modernos tomaron dos rutas hacia Europa: desde Medio Oriente a través de los Balcanes y otra desde Asia Central a través de la estepa euroasiática , al norte del Mar Negro .
Martin Richards et al. descubrieron que entre el 15 y el 40 % de los linajes de mtADN existentes se remontan a las migraciones paleolíticas (dependiendo de si se permiten múltiples eventos fundadores). [28] El haplogrupo U5 de mtADN, que data de hace unos 40-50 kYa, llegó durante la primera colonización temprana del Paleolítico superior. Individualmente, representa entre el 5 y el 15 % del total de linajes de mtADN. Los movimientos del Paleolítico superior medio están marcados por los haplogrupos HV, I y U4. HV se dividió en Pre-V (de unos 26 000 años de antigüedad) y la rama más grande H, ambas se extendieron por Europa, posiblemente a través de contactos gravetianos. [25] [29]
El haplogrupo H representa aproximadamente la mitad de las líneas genéticas en Europa, con muchos subgrupos. Los linajes de ADNmt anteriores o sus precursores, es más probable que hayan llegado a Europa a través de Oriente Medio. Esto contrasta con la evidencia del ADN Y , según la cual alrededor del 50% o más de los linajes masculinos se caracterizan por la superfamilia R1, que es de posible origen asiático central. [ cita requerida ] Ornella Semino postula que estas diferencias "pueden deberse en parte a la aparente edad molecular más reciente de los cromosomas Y en relación con otros loci, lo que sugiere un reemplazo más rápido de los cromosomas Y anteriores. Los comportamientos demográficos migratorios diferenciales basados en el género también influirán en los patrones observados de variación del ADNmt y del Y" [ cita requerida ] .
Último máximo glacial
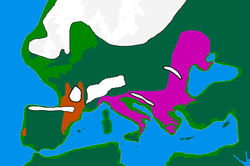
El Último Máximo Glacial ("LGM") comenzó alrededor del año 30.000 a. C., al final del MIS 3 , y provocó una despoblación del norte de Europa. Según el modelo clásico, la gente se refugiaba en santuarios climáticos (o refugios) de la siguiente manera:
- El norte de Iberia y el suroeste de Francia , en conjunto, forman el refugio "Franco-Cantábrico"
- Los Balcanes
- Ucrania y, más en general, la costa norte del Mar Negro [25]
- Italia. [30]
Este evento redujo la diversidad genética general en Europa, un "resultado de la deriva, consistente con un cuello de botella poblacional inferido durante el Último Máximo Glacial". [26] A medida que los glaciares retrocedieron hace unos 16.000 a 13.000 años, Europa comenzó a ser repoblada lentamente por personas de refugios, dejando huellas genéticas. [25]
Algunos clados del haplogrupo I del Y parecen haberse separado de sus haplogrupos parentales en algún momento durante o poco después del LGM. [31]
Cinnioglu ve evidencia de la existencia de un refugio anatolio, que también albergaba Hg R1b1b2. [32] Hoy, R1b domina el paisaje del cromosoma y de Europa occidental, incluidas las Islas Británicas, lo que sugiere que podría haber habido grandes cambios en la composición de la población basados en las migraciones posteriores al LGM.
Semino, Passarino y Pericic sitúan los orígenes del haplogrupo R1a dentro del refugio de la edad de hielo de Ucrania . Su distribución actual en Europa del Este y partes de Escandinavia refleja en parte una repoblación de Europa desde las estepas del sur de Rusia y Ucrania después del Máximo Glacial Tardío . [33] [34] [25]
Desde una perspectiva de ADNmt, Richards et al. descubrieron que la mayor parte de la diversidad de ADNmt en Europa se debe a reexpansiones postglaciales durante el Paleolítico superior tardío/Mesolítico. "Los análisis regionales respaldan en cierta medida la sugerencia de que gran parte de Europa occidental y central fue repoblada principalmente desde el suroeste cuando el clima mejoró. Los linajes involucrados incluyen gran parte del haplogrupo más común, H, así como gran parte de K, T, W y X". El estudio no pudo determinar si hubo nuevas migraciones de linajes de ADNmt desde el Cercano Oriente durante este período; se consideró poco probable que se produjera una aportación significativa. [28]
Bilton et al. analizaron el modelo alternativo de más refugiados [35] .
A partir de un estudio de 51 individuos, los investigadores pudieron identificar cinco grupos genéticos separados de antiguos euroasiáticos durante el LGM: el cúmulo de Věstonice (hace 34.000–26.000 años), asociado con la cultura Gravetiense ; el cúmulo de Mal'ta (hace 24.000–17.000), asociado con la cultura Mal'ta-Buret' , el cúmulo de El Mirón (hace 19.000–14.000 años), asociado con la cultura Magdaleniense ; el cúmulo de Villabruna (hace 14.000–7.000 años) y el cúmulo de Satsurblia (hace 13.000 a 10.000 años). [13]
Hace unos 37.000 años, todos los antiguos europeos comenzaron a compartir cierta ascendencia con los europeos modernos. [13] Esta población fundadora está representada por GoyetQ116-1, un espécimen de 35.000 años de antigüedad de Bélgica. [13] Este linaje desaparece del registro y no se encuentra de nuevo hasta 19.000 BP en España en El Mirón, que muestra fuertes afinidades con GoyetQ116-1. [13] Durante este intervalo, el cúmulo distintivo de Věstonice es predominante en Europa, incluso en Goyet . [13] La reexpansión del cúmulo de El Mirón coincidió con el calentamiento de las temperaturas tras el retroceso de los glaciares durante el Último Máximo Glacial . [13] Desde hace 37.000 a 14.000 años, la población de Europa consistía en una población aislada descendiente de una población fundadora que no se cruzó significativamente con otras poblaciones. [36]
mesolítico
Las poblaciones mesolíticas (post-LGM) habían divergido significativamente debido a su relativo aislamiento durante varios milenios, a las duras presiones de selección durante el LGM y a los efectos fundadores causados por la rápida expansión de los refugios del LGM al comienzo del Mesolítico. [3] Al final del LGM, alrededor de 19 a 11 ka, habían surgido las variedades familiares de fenotipos euroasiáticos. Sin embargo, el linaje de cazadores-recolectores mesolíticos de Europa occidental (WHG) no sobrevive como una contribución mayoritaria en ninguna población moderna. Lo más probable es que tuvieran ojos azules y conservaran la pigmentación oscura de la piel de los EEMH pre-LGM. [37] Las variaciones HERC2 y OCA2 para ojos azules se derivan del linaje WHG y también se encontraron en el pueblo Yamnaya . [37] [ contradictorio ]
Hace unos 14.000 años, el cúmulo de Villabruna se alejó de la afinidad de GoyetQ116-1 y comenzó a mostrar más afinidad con Oriente Próximo, un cambio que coincidió con el calentamiento de las temperaturas del interestadial Bølling-Allerød . [13] Este cambio genético muestra que las poblaciones de Oriente Próximo probablemente ya habían comenzado a trasladarse a Europa durante el final del Paleolítico superior, unos 6.000 años antes de lo que se pensaba anteriormente, antes de la introducción de la agricultura. [36] Algunos especímenes del cúmulo de Villabruna también muestran afinidades genéticas con los asiáticos orientales que se derivan del flujo genético. [13] [36] La variación HERC2 para ojos azules aparece por primera vez hace unos 13.000 a 14.000 años en Italia y el Cáucaso. [13] Se estima que la pigmentación clara de la piel característica de los europeos modernos se ha extendido por Europa en un "barrido selectivo" durante el Mesolítico (19 a 11 ka). Los alelos asociados TYRP1 SLC24A5 y SLC45A2 surgen alrededor de 19 ka, todavía durante el LGM, muy probablemente en el Cáucaso. [3] [4]
Neolítico

_and_modern_Western_Eurasian_populations.jpg/440px-thumbnail.jpg)
En Europa, desde hace mucho tiempo se ha reconocido la existencia de una gran variedad de variaciones genéticas que parecen indicar importantes dispersiones desde Oriente Próximo. Esto se ha relacionado a menudo con la difusión de la tecnología agrícola durante el Neolítico, que se ha considerado uno de los períodos más importantes para determinar la diversidad genética europea moderna.
El Neolítico comenzó con la introducción de la agricultura, que comenzó en el sureste de Europa aproximadamente entre el 10.000 y el 3000 a. C. y se extendió al noroeste de Europa entre el 4500 y el 1700 a. C. Durante esta era, la Revolución Neolítica provocó cambios económicos y socioculturales drásticos en Europa y se cree que esto también tuvo un gran efecto en la diversidad genética de Europa, especialmente en lo que respecta a los linajes genéticos que entraron en Europa desde Oriente Medio hasta los Balcanes. Hubo varias fases de este período:
- En un preludio del Mesolítico europeo tardío al Neolítico, parece que los pueblos del Cercano Oriente de áreas que ya tenían agricultura, y que también tenían tecnología marítima, tuvieron una presencia transitoria en Grecia (por ejemplo en la cueva de Franchthi ). [40] [41]
- Existe consenso en que la tecnología agrícola y las principales razas de animales y plantas que se cultivan ingresaron a Europa desde algún lugar del área de la Media Luna Fértil y específicamente la región del Levante desde el Sinaí hasta el sur de Anatolia . [23] : 1143, 1150 [42] (Con menos certeza, a veces se sostiene que esta revolución agrícola a su vez fue desencadenada en parte por movimientos de personas y tecnología que llegaron a través del Sinaí desde África). Para más información, consulte Media Luna Fértil: difusión cosmopolita .
- Una etapa posterior del Neolítico, el llamado Neolítico de la cerámica , vio una introducción de la cerámica en el Levante, los Balcanes y el sur de Italia (había estado presente en el área del Sudán moderno durante algún tiempo antes de ser encontrada en el Mediterráneo oriental , pero se cree que se desarrolló de forma independiente), y este también puede haber sido un período de transferencia cultural del Levante a los Balcanes.
Una cuestión importante en relación con el impacto genético de las tecnologías neolíticas en Europa es la forma en que se transfirieron a Europa. La agricultura se introdujo mediante una importante migración de agricultores del Cercano Oriente ( modelo de difusión biológica y demográfica de Cavalli-Sforza ) o una " difusión cultural " o una combinación de las dos, y los genetistas de poblaciones han tratado de aclarar si alguna firma genética de origen del Cercano Oriente corresponde a las rutas de expansión postuladas por la evidencia arqueológica. [23] : 146
Martin Richards estimó que sólo el 11% del ADNmt europeo se debe a la inmigración en este período, lo que sugiere que la agricultura se extendió principalmente debido a su adopción por las poblaciones indígenas del Mesolítico, en lugar de debido a la inmigración desde Oriente Próximo. El flujo genético del SE al NO de Europa parece haber continuado en el Neolítico, y el porcentaje disminuyó significativamente hacia las Islas Británicas. La genética clásica también sugirió que la mayor mezcla con el stock europeo del Paleolítico/Mesolítico se debió a la revolución neolítica del séptimo al quinto milenio a. C. [43] Se han identificado tres grupos principales de genes de ADNmt que contribuyeron a la entrada neolítica en Europa: J, T1 y U3 (en ese orden de importancia). Con otros, representan alrededor del 20% del acervo genético . [28]
En 2000, el estudio de Semino sobre el ADN Y reveló la presencia de haplotipos pertenecientes al gran clado E1b1b1 (E-M35). Estos se encontraron predominantemente en los Balcanes meridionales, el sur de Italia y partes de Iberia. Semino relacionó este patrón, junto con los subclados del haplogrupo J, con el componente de ADN-Y de la difusión demica neolítica de Cavalli-Sforza de agricultores del Cercano Oriente. [25] : Aquí, el clado E-M35 se conoce como "Eu 4" . Rosser et al. lo vieron más bien como un "componente norteafricano" (directo) en la genealogía europea, aunque no propusieron un momento y un mecanismo para explicarlo. [44] [45] también describieron a E1b1b como representante de una migración del Pleistoceno tardío de África a Europa sobre la península del Sinaí en Egipto , evidencia de la cual no aparece en el ADN mitocondrial. [46]
En cuanto a la distribución y diversidad de V13, Battaglia [47] propuso un movimiento anterior según el cual el linaje E-M78*, ancestro de todos los hombres E-V13 modernos, se trasladó rápidamente desde su patria en el sur de Egipto y llegó a Europa con solo tecnologías mesolíticas . Luego sugieren que el subclado E-V13 de E-M78 solo se expandió posteriormente cuando los "recolectores-agricultores" nativos de los Balcanes adoptaron tecnologías neolíticas del Cercano Oriente. Proponen que la primera dispersión importante de E-V13 desde los Balcanes puede haber sido en dirección al mar Adriático con la cultura de cerámica impresa neolítica a menudo denominada Impressa o Cardial , [33] más bien proponen que la ruta principal de propagación de E-V13 fue a lo largo del sistema de "autopistas" de los ríos Vardar-Morava-Danubio.
A diferencia de Battaglia, Cruciani [48] sugirió tentativamente (i) un punto diferente en el que la mutación V13 ocurrió en su camino desde Egipto a los Balcanes a través de Oriente Medio, y (ii) un tiempo de dispersión posterior. Los autores propusieron que la mutación V13 apareció por primera vez en Asia occidental, donde se encuentra en frecuencias bajas pero significativas, desde donde entró en los Balcanes en algún momento después de 11 kYa. Más tarde experimentó una rápida dispersión que fechó hace unos 5300 años en Europa, coincidiendo con la Edad de Bronce de los Balcanes. Al igual que Peričic et al., consideran que "la dispersión de los haplogrupos E-V13 y J-M12 parece haber seguido principalmente las vías fluviales que conectan los Balcanes meridionales con el centro-norte de Europa".
Más recientemente, Lacan [49] anunció que un esqueleto de 7000 años de antigüedad en un contexto neolítico en una cueva funeraria española, era un hombre E-V13. (Los otros especímenes analizados del mismo sitio estaban en el haplogrupo G2a , que se ha encontrado en contextos neolíticos en toda Europa). Usando 7 marcadores STR, este espécimen fue identificado como similar a los individuos modernos analizados en Albania , Bosnia , Grecia , Córcega y Provenza . Por lo tanto, los autores propusieron que, independientemente de si la distribución moderna de E-V13 de hoy es el resultado de eventos más recientes o no, E-V13 ya estaba en Europa durante el Neolítico, llevado por los primeros agricultores del Mediterráneo Oriental al Mediterráneo Occidental, mucho antes de la Edad del Bronce. Esto apoya las propuestas de Battaglia et al. en lugar de Cruciani et al. al menos con respecto a las primeras dispersiones europeas, pero E-V13 puede haberse dispersado más de una vez. Incluso más recientemente que la Edad del Bronce, también se ha propuesto que la distribución moderna del E-V13 en Europa se debe, al menos en parte, a los movimientos de personas de la época romana. [50] (Véase más abajo).
La migración de los agricultores neolíticos a Europa trajo consigo varias adaptaciones nuevas. [37] La variación del color de piel claro fue introducida en Europa por los agricultores neolíticos . [37] Después de la llegada de los agricultores neolíticos, se seleccionó una mutación SLC22A4 , una mutación que probablemente surgió para tratar la deficiencia de ergotioneína pero que aumenta el riesgo de colitis ulcerosa , enfermedad celíaca y síndrome del intestino irritable .
Edad del Bronce
La Edad del Bronce fue testigo del desarrollo de redes comerciales de larga distancia , en particular a lo largo de la costa atlántica y en el valle del Danubio. En este período se produjeron migraciones desde Noruega a las islas Orcadas y Shetland (y en menor medida a Escocia continental e Irlanda). También hubo migraciones desde Alemania al este de Inglaterra. Martin Richards estimó que hubo alrededor de un 4 % de inmigración de ADNmt a Europa en la Edad del Bronce.
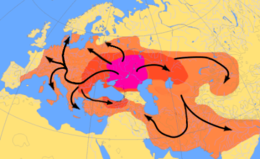
Otra teoría sobre el origen de la lengua indoeuropea se centra en un hipotético pueblo protoindoeuropeo , que, según la hipótesis de los kurganes , se puede rastrear hasta el norte de los mares Negro y Caspio alrededor del 4500 a. C. [51] Domesticaron el caballo y posiblemente inventaron la rueda de disco de madera , y se considera que difundieron su cultura y genes por toda Europa. [52] El haplogrupo Y R1a es un marcador propuesto de estos genes "kurganes", al igual que el haplogrupo Y R1b , aunque estos haplogrupos en su conjunto pueden ser mucho más antiguos que la familia lingüística. [53]
En el extremo norte, los portadores del haplogrupo N llegaron a Europa desde Siberia y finalmente se expandieron hasta Finlandia , aunque el momento específico de su llegada es incierto. Se estima que el subclado más común del norte de Europa, N1c1, tiene alrededor de 8000 años. Hay evidencia de asentamiento humano en Finlandia que data de 8500 a. C., vinculado con la cultura Kunda y su supuesto ancestro, la cultura Swiderian , pero se cree que esta última tiene un origen europeo. La expansión geográfica del haplogrupo N en Europa está bien alineada con la cultura Pit-Comb Ware , cuyo surgimiento se fecha comúnmente alrededor del 4200 a. C., y con la distribución de las lenguas urálicas . Los estudios de ADN mitocondrial del pueblo sami , haplogrupo U5, son consistentes con múltiples migraciones a Escandinavia desde la región del Volga - Ural , que comenzaron entre 6000 y 7000 años antes del presente. [54]
La relación entre los roles de los colonos europeos y asiáticos en la prehistoria de Finlandia es un punto de cierta controversia, y algunos académicos insisten en que los finlandeses son "predominantemente europeos del este y están compuestos por personas que emigraron al norte desde el refugio ucraniano durante la Edad de Hielo". [55] Más al este, el tema es menos polémico. Los portadores del haplogrupo N representan una parte significativa de todos los grupos étnicos no eslavos en el norte de Rusia , incluido el 37% de los carelios , el 35% de los komi (65% según otro estudio [56] ), el 67% de los mari , hasta el 98% de los nenets , el 94% de los nganasanos y entre el 86% y el 94% de los yakutos . [57]
El componente Yamnaya contiene ascendencia parcial de un componente del Antiguo Norte de Eurasia, un linaje siberiano paleolítico pero estrechamente relacionado con los cazadores-recolectores europeos, identificado por primera vez en Mal'ta . [58] [59] Según Iosif Lazaridis, "la ascendencia del Antiguo Norte de Eurasia es proporcionalmente el componente más pequeño en todas partes de Europa, nunca más del 20 por ciento, pero lo encontramos en casi todos los grupos europeos que hemos estudiado". [60] Este componente genético no proviene directamente del linaje Mal'ta en sí, sino de un linaje relacionado que se separó del linaje Mal'ta. [13]
Hasta la mitad del componente Yamnaya puede haber venido de una rama de cazadores-recolectores del Cáucaso . [58] El 16 de noviembre de 2015, en un estudio publicado en la revista Nature Communications , [58] los genetistas anunciaron que habían encontrado una nueva cuarta "tribu" o "rama" ancestral que había contribuido al acervo genético europeo moderno. Analizaron los genomas de dos cazadores-recolectores de Georgia que tenían 13.300 y 9.700 años de antigüedad, y descubrieron que estos cazadores-recolectores del Cáucaso eran probablemente la fuente del ADN de tipo agrícola en los Yamnaya. Según el coautor Dr. Andrea Manica de la Universidad de Cambridge: "La cuestión de dónde vienen los Yamnaya ha sido algo así como un misterio hasta ahora... ahora podemos responder a eso, ya que hemos descubierto que su composición genética es una mezcla de cazadores-recolectores de Europa del Este y una población de este grupo de cazadores-recolectores del Cáucaso que resistieron gran parte de la última Edad de Hielo en aparente aislamiento". [61]
Según Lazaridis et al. (2016), una población relacionada con los pueblos del Calcolítico iraní contribuyó a aproximadamente la mitad de la ascendencia de las poblaciones Yamnaya de la estepa póntico-caspia. Estos pueblos del Calcolítico iraní eran una mezcla de "los pueblos neolíticos del oeste de Irán, el Levante y los cazadores-recolectores del Cáucaso". [62]
Las variaciones genéticas para la persistencia de la lactasa y una mayor altura llegaron con el pueblo Yamnaya. [37] El alelo derivado del gen KITLG (SNP rs12821256) que está asociado con el cabello rubio en los europeos (y probablemente es la causa de este) se encuentra en poblaciones con ascendencia de cazadores-recolectores orientales pero no occidentales , lo que sugiere que su origen está en la población de la antigua euroasiática del norte (ANE) y puede haber sido difundida en Europa por individuos con ascendencia esteparia . En consonancia con esto, el individuo más antiguo conocido con el alelo derivado es un individuo ANE del complejo arqueológico Afontova Gora del Paleolítico Superior Tardío en Siberia central. [63]
Historia reciente

Las expansiones del Imperio Romano no parecen haber dejado firmas genéticas distintivas en Europa. De hecho, se ha descubierto que las poblaciones de habla romance en los Balcanes, como los rumanos , los arrumanos , los moldavos , etc., se parecen genéticamente a los pueblos vecinos de habla griega y eslava meridional en lugar de a los italianos modernos. [65] [66] Steven Bird ha especulado que E1b1b1a se extendió durante la era romana a través de las poblaciones tracias y dacias de los Balcanes al resto de Europa. [50]
En relación con el período romano tardío de " Völkerwanderung " (no solo) germánico , se han hecho algunas sugerencias, al menos para Gran Bretaña, de que el haplogrupo I1a de Y estaría asociado con la inmigración anglosajona en el este de Inglaterra, y el R1a estaría asociado con la inmigración nórdica en el norte de Escocia. [67]
Genética de las poblaciones europeas modernas
Estudios patrilineales
Hay cuatro haplogrupos principales de ADN del cromosoma Y que representan la mayor parte de la descendencia patrilineal de Europa . [25]
- El haplogrupo R1b es común en Europa, particularmente en Europa Occidental , siendo el R1b1a1a2 el más común entre los europeos occidentales. [68] [69] [70] Casi todo este R1b en Europa está en la forma del subclado R1b1a2 (nombre de 2011) (R-M269), específicamente dentro del subclado R-L23, mientras que el R1b que se encuentra en Asia Central , Asia Occidental y África tiende a estar en otros clados. También se ha señalado que los tipos atípicos están presentes en Europa y son particularmente notables en algunas áreas como Cerdeña y Armenia. [71] Las frecuencias del haplogrupo R1b varían desde los máximos en Europa occidental en un clina en constante disminución con la creciente distancia desde el Atlántico: 80-90% ( galeses , vascos , irlandeses , escoceses , bretones ) alrededor del 70-80% en España, Gran Bretaña y Francia y alrededor del 40-60% en partes del este de Alemania y el norte de Italia. Desciende fuera de esta área y es alrededor del 30% o menos en áreas como el sur de Italia, Polonia , los Balcanes y Chipre . R1b sigue siendo el clado más común a medida que uno se mueve al este de Alemania, mientras que más al este, en Polonia, R1a es más común (ver más abajo). [72] En el sudeste de Europa , R1b cae detrás de R1a en el área dentro y alrededor de Hungría y Serbia, pero es más común tanto al sur como al norte de esta región. [33] R1b en Europa occidental está dominado por al menos dos subclados, R-U106, que se distribuye desde el lado este del Rin hasta el norte y centro de Europa (con una fuerte presencia en Inglaterra) y R-P312, que es más común al oeste del Rin, incluidas las Islas Británicas . [69] [70]
- El haplogrupo R1a , casi en su totalidad en el subclado R1a1a, prevalece en gran parte de Europa central y oriental (también en Asia meridional y central ). Por ejemplo, hay un marcado aumento en R1a1 y una disminución en R1b1b2 a medida que uno se dirige hacia el este desde Alemania hasta Polonia. [72] También tiene una presencia sustancial en Escandinavia (particularmente Noruega). [73] [74] En los países bálticos, las frecuencias de R1a disminuyen desde Lituania (45%) hasta Estonia (alrededor del 30%). [75]
- El haplogrupo I se encuentra en forma de varios subclados en toda Europa y se encuentra en las frecuencias más altas en los países nórdicos como I1 ( Noruega , Dinamarca , Suecia , Finlandia ) y en la península de los Balcanes como I2a ( Bosnia y Herzegovina 65%, [56] Croacia y Serbia ). I1 también es frecuente en Alemania , Gran Bretaña y Países Bajos , mientras que I2a es frecuente también en Cerdeña , Rumania / Moldavia , Bulgaria y Ucrania . Este clado se encuentra en su expresión más alta con diferencia en Europa y puede haber estado allí desde antes del LGM . [31]
- El haplogrupo E1b1b (antes conocido como E3b) fue parte de una migración de agricultores neolíticos de Oriente Medio, que portaban E1b1b en una frecuencia baja a media y se introdujo en los habitantes neolíticos de Oriente Medio a lo largo de la deriva genética de una migración de África a Oriente Medio asociada con las lenguas afroasiáticas . Se cree que apareció por primera vez en el noreste de África hace aproximadamente 26.000 años y se dispersó al norte de África y Oriente Próximo durante los períodos Paleolítico tardío y Mesolítico. Los linajes E1b1b están estrechamente vinculados a la difusión de las lenguas afroasiáticas. Aunque está presente en toda Europa, alcanza su punto máximo en la región sur de los Balcanes entre los albaneses y sus vecinos. También es común en Italia y la península Ibérica en una frecuencia más baja. El haplogrupo E1b1b1 , principalmente en forma de su subclado E1b1b1a2 (E-V13), alcanza frecuencias superiores al 47% alrededor del área de Kosovo . [33] Se cree que este clado llegó a Europa desde Asia occidental, ya sea a finales del Mesolítico, [47] o en el Neolítico. [76] El subclado E-M81 del norte de África también está presente en Sicilia y Andalucía.
Dejando de lado los pequeños enclaves, también hay varios haplogrupos aparte de los cuatro mencionados anteriormente que son menos prominentes o más comunes sólo en ciertas áreas de Europa.
- El haplogrupo G , un haplogrupo común entre los agricultores neolíticos europeos, es común en la mayor parte de Europa con una frecuencia baja, alcanzando picos superiores al 70% alrededor de Georgia y entre los madjars (aunque viven en Asia bordean el perímetro oriental de Europa), hasta el 10% en Cerdeña, el 12% en Córcega y Uppsala (Suecia), el 11% en los Balcanes y Portugal, el 10% en España y el 9% en la Rusia europea. Este clado también se encuentra en Oriente Próximo.
- El haplogrupo N , es común sólo en el noreste de Europa y en la forma de su subclado N1c1 alcanza frecuencias de aproximadamente el 60% entre los finlandeses y aproximadamente el 40% entre los estonios, letones y lituanos.
- El haplogrupo J2 , en varios subclados (J2a, J2b), se encuentra en niveles de alrededor del 15-30% en los Balcanes (particularmente Grecia ) e Italia . El haplogrupo J2 es frecuente en Asia occidental y el Mediterráneo oriental . [77]
Estudios matrilineales
Se han realizado numerosos estudios sobre los haplogrupos de ADN mitocondrial (ADNmt) en Europa. A diferencia de los haplogrupos de ADN-Y, los haplogrupos de ADNmt no mostraron tanta estructuración geográfica, pero fueron más uniformemente ubicuos. Aparte de los lejanos saami, todos los europeos se caracterizan por el predominio de los haplogrupos H, U y T. La falta de estructuración geográfica observable del ADNmt puede deberse a factores socioculturales, en concreto a los fenómenos de poligamia y patrilocalidad . [44]
Los estudios genéticos sugieren que hubo algún flujo genético materno hacia Europa del este desde Asia oriental o Siberia meridional hace entre 13.000 y 6.600 años antes del presente . [78] El análisis de esqueletos neolíticos en la Gran Llanura Húngara encontró una alta frecuencia de haplogrupos de ADNmt del este de Asia, algunos de los cuales sobreviven en las poblaciones modernas de Europa del este. [78] El flujo genético materno hacia Europa desde África subsahariana comenzó hace 11.000 años antes del presente, aunque se estima que la mayoría de los linajes, aproximadamente el 65%, llegaron más recientemente, incluso durante el período de romanización, las conquistas árabes del sur de Europa y durante el comercio de esclavos del Atlántico. [79]
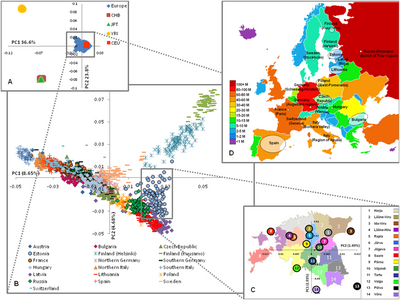
Subestructura de la población europea
Genéticamente, Europa es relativamente homogénea, pero se han encontrado patrones de subpoblación distintos de varios tipos de marcadores genéticos, [80] particularmente a lo largo de una clina sureste-noroeste. [81] Por ejemplo, los análisis de componentes principales de Cavalli-Sforza revelaron cinco patrones clinales principales en toda Europa, y se han seguido encontrando patrones similares en estudios más recientes. [80] : 291–296
- Una clina de genes con frecuencias más altas en Oriente Medio , que se extiende a niveles más bajos en el noroeste. Cavalli-Sforza originalmente describió esto como un fiel reflejo de la expansión de la agricultura en tiempos neolíticos. Esta ha sido la tendencia general en la interpretación de todos los genes con este patrón.
- Una clina de genes con frecuencias más altas entre los finlandeses y los samis en el extremo noreste, y que se extiende a frecuencias más bajas en el suroeste.
- Un clina de genes con frecuencias más altas en el área de los bajos ríos Don y Volga en el sur de Rusia , y extendiéndose a frecuencias más bajas en España, sur de Italia , Grecia y las áreas habitadas por hablantes de saami en el extremo norte de Escandinavia . Cavalli-Sforza asoció esto con la expansión de las lenguas indoeuropeas, que vincula a su vez a una "expansión secundaria" después de la expansión de la agricultura, asociada con el pastoreo de animales.
- Una clina de genes con frecuencias más altas en los Balcanes y el sur de Italia, que se extiende a niveles más bajos en Gran Bretaña y el País Vasco. Cavalli-Sforza asocia esto con "la expansión griega, que alcanzó su punto máximo en tiempos históricos alrededor de 1000 y 500 a. C., pero que ciertamente comenzó antes".
- Una clina de genes con frecuencias más altas en el País Vasco y niveles más bajos más allá del área de Iberia y el sur de Francia . En quizás la conclusión más conocida de Cavalli-Sforza, este patrón, el más débil de los cinco, fue descrito como restos aislados de la población preneolítica de Europa, "que al menos parcialmente resistió la expansión de los cultivadores". Corresponde aproximadamente a la propagación geográfica de los tipos de sangre Rh negativos . En particular, la conclusión de que los vascos son un aislamiento genético ha sido ampliamente discutida, pero también es una conclusión controvertida.
También creó un árbol filogenético para analizar las relaciones internas entre los europeos. Encontró cuatro "excepciones" importantes: vascos , samis , sardos e islandeses ; [82] un resultado que atribuyó a su aislamiento relativo (nota: los islandeses y los sardos hablan lenguas indoeuropeas , mientras que los otros dos grupos no). Los griegos y yugoslavos representaron un segundo grupo de excepciones menos extremas. Las poblaciones restantes se agruparon en varios grupos: " celtas ", " germánicos ", "europeos del sudoeste", " escandinavos " y "europeos del este". [80] : 268
Un estudio realizado en mayo de 2009 [83] que investigó 19 poblaciones de Europa utilizando 270.000 SNP destacó la diversidad genética de las poblaciones europeas correspondientes al gradiente noroeste a sureste y distinguió "cuatro regiones distintas" dentro de Europa:
- Finlandia, que muestra la mayor distancia respecto al resto de europeos.
- la región del Báltico ( Estonia , Letonia y Lituania ), el oeste de Rusia y el este de Polonia .
- Europa central y occidental.
- Italia , debido a que los Alpes actúan como una gran barrera genética.
En este estudio, el análisis de barreras reveló "barreras genéticas" entre Finlandia, Italia y otros países y que también se podían demostrar barreras dentro de Finlandia (entre Helsinki y Kuusamo) e Italia (entre la parte norte y sur, Fst = 0,0050). Se encontró que el Fst ( índice de fijación ) se correlacionaba considerablemente con las distancias geográficas que iban desde ≤0,0010 para las poblaciones vecinas hasta 0,0200-0,0230 para el sur de Italia y Finlandia. Para las comparaciones, los Fst por pares de muestras no europeas fueron los siguientes: europeos - africanos (yoruba) 0,1530; europeos - chinos 0,1100; africanos (yoruba) - chinos 0,1900. [84]
Un estudio realizado por Chao Tian en agosto de 2009 amplió el análisis de la estructura genética de la población europea para incluir grupos adicionales del sur de Europa y poblaciones árabes ( palestinos , drusos ...) del Cercano Oriente. Este estudio determinó el Fst autosómico entre 18 grupos de población y concluyó que, en general, las distancias genéticas correspondían a relaciones geográficas con valores más pequeños entre grupos de población con orígenes en países/regiones vecinos (por ejemplo, griegos / toscanos : Fst=0,0010, griegos / palestinos : Fst=0,0057) en comparación con los de regiones muy diferentes de Europa (por ejemplo, griegos / suecos : Fst=0,0087, griegos / rusos : Fst=0,0108). [85]
ADN autosómico
Seldin (2006) utilizó más de 5000 SNP autosómicos. Mostró "una distinción consistente y reproducible entre los grupos de población europeos 'del norte' y 'del sur'". La mayoría de los participantes individuales con ascendencia europea del sur ( italianos , griegos , portugueses , españoles ) y judíos asquenazíes tienen >85% de pertenencia a la población del sur; y la mayoría de los europeos del norte, oeste, centro y este ( suecos , ingleses , irlandeses , alemanes y ucranianos ) tienen >90% en el grupo de población del norte. Muchos de los participantes en este estudio eran ciudadanos estadounidenses que se autoidentificaron con diferentes etnias europeas basándose en el pedigrí familiar autoinformado. [86]
Un estudio similar realizado en 2007, en el que se utilizaron muestras predominantemente de Europa, concluyó que la diferenciación genética más importante en Europa se produce en una línea que va del norte al sureste ( desde el norte de Europa hasta los Balcanes), con otro eje de diferenciación este-oeste a lo largo de Europa. Sus hallazgos fueron coherentes con resultados anteriores basados en el ADN mitocondrial y el ADN del cromosoma Y que apoyan la teoría de que los ibéricos modernos (españoles y portugueses) poseen la ascendencia genética europea más antigua, además de separar a los vascos y samis de otras poblaciones europeas. [87]
Sugirió que los ingleses e irlandeses se agrupan con otros europeos del norte y del este, como los alemanes y los polacos , mientras que algunos individuos vascos e italianos también se agrupan con los europeos del norte. A pesar de estas estratificaciones, señaló que "existe una baja diversidad aparente en Europa, con muestras de todo el continente apenas más dispersas que muestras de poblaciones individuales en otras partes del mundo". [87]
En 2008, dos equipos de investigación internacionales publicaron análisis de genotipado a gran escala de grandes muestras de europeos, utilizando más de 300.000 SNP autosómicos. Con la excepción de los aislamientos habituales como los vascos , los finlandeses y los sardos , la población europea carecía de discontinuidades marcadas (agrupamiento) como habían encontrado estudios anteriores (véase Seldin et al. 2006 y Bauchet et al. 2007 [87] ), aunque había un gradiente discernible de sur a norte. En general, encontraron solo un bajo nivel de diferenciación genética entre subpoblaciones, y las diferencias que existían se caracterizaban por una fuerte correlación continental entre la distancia geográfica y genética. Además, encontraron que la diversidad era mayor en el sur de Europa debido a un mayor tamaño efectivo de la población y/o expansión de la población del sur al norte de Europa. [81] Los investigadores toman esta observación para implicar que genéticamente, los europeos no están distribuidos en poblaciones discretas. [88] [81]
Dos estudios de genoma completo de las dos poblaciones de Europa del Este en Ucrania ( ucranianos de Ucrania ) y Rusia ( rusos de Rusia ) mostraron diversidad genómica, que no ha sido representada en las encuestas genómicas anteriores, ya que los estudios en Europa están mayoritariamente sesgados hacia las poblaciones en la parte occidental del continente. [89] [90] Dentro de Rusia , el pueblo Komi , que vive en las regiones del noreste y es parte de la familia de lenguas urálicas que también incluye a los finlandeses , forma un polo de diversidad genética que es distinto de otras poblaciones y se caracteriza por una mayor ascendencia de cazadores-recolectores europeos (WHG) y del antiguo norte de Eurasia. [91] [92]
Según el genetista David Reich , basándose en genomas humanos antiguos que su laboratorio secuenció en 2016, los europeos descienden de una mezcla de cuatro componentes ancestrales de Eurasia occidental, a saber, WHG (cazadores-recolectores occidentales), EHG, agricultores neolíticos del Levante/Anatolia, así como de agricultores neolíticos de Irán (a menudo resumidos como "EEF"; primeros agricultores europeos), en diversos grados. [93] [94]
El flujo genético siberiano se encuentra entre varios grupos étnicos europeos de habla urálica. Este componente siberiano es en sí mismo una composición de ascendencia relacionada con el norte de Eurasia antigua y el este de Asia de Siberia oriental, maximizada entre los evenks y los evens o nganasanos . Algunos genetistas vinculan la propagación de esta ascendencia a la dispersión de las lenguas urálicas , otros, sin embargo, sostienen que las lenguas urálicas se propagaron antes de la llegada del flujo genético siberiano, que es una fuente secundaria de diversidad dentro de las poblaciones de habla urálica. [95] [96] Los datos genéticos apuntan a un origen de cazadores-recolectores siberianos occidentales del flujo genético siberiano observado entre los grupos de habla urálica. Los cazadores-recolectores siberianos occidentales se caracterizaron por una alta ascendencia de Eurasia del norte antigua y menores cantidades de mezcla de Siberia oriental. Los datos genéticos sobre los tártaros del Volga o chuvasos , encontrados entre " los hablantes de turco occidental, como los chuvasos y los tártaros del Volga, el componente de Asia oriental se detectó solo en cantidades bajas (~ 5%) ". [97] [98]
La ascendencia del este de Asia se encuentra con baja frecuencia entre algunos europeos, como los británicos (2,5 ± 1%), los orcadianos (3,8 ± 1%), los franceses (0,7 ± 0,8%) y los alemanes (0,7 ± 0,8%). Los finlandeses y los rusos tienen más del 12% de ascendencia del este de Asia, derivada de matrimonios históricos con poblaciones mongolas. [99] Pero un estudio de 2017 no encuentra evidencia de mezcla asiática entre los rusos, a excepción de los residentes de Novosibirsk y los viejos creyentes en Siberia. [100] Los tártaros de Lipka , una minoría turca en Bielorrusia, tienen una ascendencia significativa de Eurasia Oriental, que constituye un tercio de su genoma. [101]
Al igual que otras poblaciones euroasiáticas, las ascendencias mesolíticas, neolíticas o de la Edad del Bronce no se distribuyen de manera homogénea en las poblaciones europeas. Pero las ascendencias relacionadas con la WHG son más abundantes en los individuos actuales de los Estados bálticos, Bielorrusia, Polonia y Rusia, mientras que las ascendencias relacionadas con la EHG son más abundantes en Finlandia y Estonia. Las ascendencias relacionadas con las estepas se encuentran en niveles altos en el norte de Europa, alcanzando su punto máximo en Irlanda, Islandia, Noruega y Suecia, pero disminuyen más al sur, especialmente en el sur de Europa, donde predominan las ascendencias de agricultores relacionadas con el Neolítico de Anatolia. [102]
Distancias genéticas autosómicas (Fst) basadas en SNP (2009)
La distancia genética entre poblaciones se mide a menudo mediante el índice de fijación (Fst), basado en datos de polimorfismos genéticos, como polimorfismos de un solo nucleótido (SNP) o microsatélites . Fst es un caso especial de estadística F , el concepto desarrollado en la década de 1920 por Sewall Wright . Fst es simplemente la correlación de alelos elegidos aleatoriamente dentro de la misma subpoblación en relación con la que se encuentra en toda la población. A menudo se expresa como la proporción de diversidad genética debido a las diferencias de frecuencia de alelos entre poblaciones.
Los valores varían de 0 a 1. Un valor de cero implica que las dos poblaciones son panmícticas, que se están cruzando libremente. Un valor de uno implicaría que las dos poblaciones están completamente separadas. Cuanto mayor sea el valor de Fst, mayor será la distancia genética. Básicamente, estos valores bajos de Fst sugieren que la mayoría de la variación genética se da a nivel de individuos dentro del mismo grupo de población (~ 85%); mientras que pertenecer a un grupo de población diferente dentro de la misma "raza"/continente, e incluso a diferentes grupos raciales/continentales, agregó un grado mucho menor de variación (3-8%; 6-11%, respectivamente).
| Ítaloamericanos | Palestinos | Suecos | Drusos | Españoles | Alemanes | Rusos | irlandés | Griegos americanos | Judíos asquenazíes | Circasianos | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Ítaloamericanos | 0,0064 | 0,0064 | 0,0057 | 0,0010 | 0,0029 | 0,0088 | 0,0048 | 0.0000 | 0,0040 | 0,0067 | |
| Palestinos | 0,0064 | 0,0191 | 0,0064 | 0,0101 | 0,0136 | 0,0202 | 0,0170 | 0,0057 | 0,0093 | 0,0108 | |
| Suecos | 0,0064 | 0,0191 | 0,0167 | 0,0040 | 0,0007 | 0,0030 | 0,0020 | 0,0084 | 0,0120 | 0,0117 | |
| Drusos | 0,0057 | 0,0064 | 0,0167 | 0,0096 | 0,0121 | 0,0194 | 0,0154 | 0,0052 | 0,0088 | 0,0092 | |
| Españoles | 0,0010 | 0,0101 | 0,0040 | 0,0096 | 0,0015 | 0,0070 | 0,0037 | 0,0035 | 0,0056 | 0,0090 | |
| Alemanes | 0,0029 | 0,0136 | 0,0007 | 0,0121 | 0,0015 | 0,0030 | 0,0010 | 0,0039 | 0,0072 | 0,0089 | |
| Rusos | 0,0088 | 0,0202 | 0,0030 | 0,0194 | 0,0070 | 0,0030 | 0,0038 | 0,0108 | 0,0137 | 0,0120 | |
| irlandés | 0,0048 | 0,0170 | 0,0020 | 0,0154 | 0,0037 | 0,0010 | 0,0038 | 0,0067 | 0,0109 | 0,0110 | |
| Griegos americanos | 0.0000 | 0,0057 | 0,0084 | 0,0052 | 0,0035 | 0,0039 | 0,0108 | 0,0067 | 0,0042 | 0,0054 | |
| Judíos asquenazíes | 0,0040 | 0,0093 | 0,0120 | 0,0088 | 0,0056 | 0,0072 | 0,0137 | 0,0109 | 0,0042 | 0,0107 | |
| Circasianos | 0,0067 | 0,0108 | 0,0117 | 0,0092 | 0,0090 | 0,0089 | 0,0120 | 0,0110 | 0,0054 | 0,0107 |
| Austria | Bulgaria | República Checa | Estonia | Finlandia ( Helsinki ) | Finlandia ( Kuusamo ) | Francia | Alemania del Norte | Alemania del Sur | Hungría | Italia del Norte | Italia del Sur | Letonia | Lituania | Polonia | Rusia | España | Suecia | Suiza | CEU | ||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Austria | 1.14 | 1.08 | 1.58 | 2.24 | 3.30 | 1.16 | 1.10 | 1.04 | 1.04 | 1.49 | 1,79 | 1,85 | 1,70 | 1.19 | 1.47 | 1.41 | 1.21 | 1.19 | 1.12 | Austria | |
| Bulgaria | 1.14 | 1.21 | 1,70 | 2.19 | 2.91 | 1.22 | 1.32 | 1.19 | 1.10 | 1.32 | 1.38 | 1.86 | 1,73 | 1.29 | 1.53 | 1.30 | 1.47 | 1.13 | 1.29 | Bulgaria | |
| República Checa | 1.08 | 1.21 | 1.42 | 2.20 | 3.26 | 1.35 | 1.15 | 1.16 | 1.06 | 1.69 | 2.04 | 1.62 | 1.48 | 1.09 | 1.27 | 1.63 | 1.26 | 1.37 | 1.21 | República Checa | |
| Estonia | 1.58 | 1,70 | 1.42 | 1.71 | 2.80 | 2.08 | 1.53 | 1,70 | 1.41 | 2.42 | 2.93 | 1.24 | 1.28 | 1.17 | 1.21 | 2.54 | 1.49 | 2.16 | 1,59 | Estonia | |
| Finlandia (Helsinki) | 2.24 | 2.19 | 2.20 | 1.71 | 1.86 | 2.69 | 2.17 | 2.35 | 1.87 | 2.82 | 3.37 | 2.31 | 2.33 | 1,75 | 2.10 | 3.14 | 1,89 | 2,77 | 1,99 | Finlandia (Helsinki) | |
| Finlandia (Kuusamo) | 3.30 | 2.91 | 3.26 | 2.80 | 1.86 | 3.72 | 3.27 | 3.46 | 2.68 | 3.64 | 4.18 | 3.33 | 3.37 | 2.49 | 3.16 | 4.21 | 2.87 | 3.83 | 2,89 | Finlandia (Kuusamo) | |
| Francia | 1.16 | 1.22 | 1.35 | 2.08 | 2.69 | 3.72 | 1.25 | 1.12 | 1.16 | 1.38 | 1.68 | 2.40 | 2.20 | 1.44 | 1,94 | 1.13 | 1.38 | 1.10 | 1.13 | Francia | |
| Alemania del Norte | 1.10 | 1.32 | 1.15 | 1.53 | 2.17 | 3.27 | 1.25 | 1.08 | 1.11 | 1,72 | 2.14 | 1.84 | 1.66 | 1.18 | 1.49 | 1.62 | 1.12 | 1.36 | 1.06 | Alemania del Norte | |
| Alemania del Sur | 1.04 | 1.19 | 1.16 | 1,70 | 2.35 | 3.46 | 1.12 | 1.08 | 1.08 | 1.53 | 1,85 | 1.20 | 1.84 | 1.23 | 1.58 | 1.40 | 1.21 | 1.17 | 1.07 | Alemania del Sur | |
| Hungría | 1.04 | 1.10 | 1.06 | 1.41 | 1.87 | 2.68 | 1.16 | 1.11 | 1.08 | 1.42 | 1.63 | 1.58 | 1.46 | 1.14 | 1.28 | 1.32 | 1.22 | 1.16 | 1.13 | Hungría | |
| Italia del Norte | 1.49 | 1.32 | 1.69 | 2.42 | 2.82 | 3.64 | 1.38 | 1,72 | 1.53 | 1.42 | 1.54 | 2.64 | 2.48 | 1,75 | 2.24 | 1.42 | 1.86 | 1.36 | 1.56 | Italia del Norte | |
| Italia del Sur | 1,79 | 1.38 | 2.04 | 2.93 | 3.37 | 4.18 | 1.68 | 2.14 | 1,85 | 1.63 | 1.54 | 3.14 | 2,96 | 1,99 | 2.68 | 1.67 | 2.28 | 1.54 | 1.84 | Italia del Sur | |
| Letonia | 1,85 | 1.86 | 1.62 | 1.24 | 2.31 | 3.33 | 2.40 | 1.84 | 1.20 | 1.58 | 2.64 | 3.14 | 1.20 | 1.26 | 1.84 | 2.82 | 1,89 | 2.52 | 1.87 | Letonia | |
| Lituania | 1,70 | 1,73 | 1.48 | 1.28 | 2.33 | 3.37 | 2.20 | 1.66 | 1.84 | 1.46 | 2.48 | 2,96 | 1.20 | 1.20 | 1.26 | 2.62 | 1,74 | 2.29 | 1,74 | Lituania | |
| Polonia | 1.19 | 1.29 | 1.09 | 1.17 | 1,75 | 2.49 | 1.44 | 1.18 | 1.23 | 1.14 | 1,75 | 1,99 | 1.26 | 1.20 | 1.18 | 1.66 | 1.30 | 1.46 | 1.28 | Polonia | |
| Rusia | 1.47 | 1.53 | 1.27 | 1.21 | 2.10 | 3.16 | 1,94 | 1.49 | 1.58 | 1.28 | 2.24 | 2.68 | 1.84 | 1.26 | 1.18 | 2.32 | 1,59 | 1.20 | 1.56 | Rusia | |
| España | 1.41 | 1.30 | 1.63 | 2.54 | 3.14 | 4.21 | 1.13 | 1.62 | 1.40 | 1.32 | 1.42 | 1.67 | 2.82 | 2.62 | 1.66 | 2.32 | 1,73 | 1.16 | 1.34 | España | |
| Suecia | 1.21 | 1.47 | 1.26 | 1.49 | 1,89 | 2.87 | 1.38 | 1.12 | 1.21 | 1.22 | 1.86 | 2.28 | 1,89 | 1,74 | 1.30 | 1,59 | 1,73 | 1,50 | 1.09 | Suecia | |
| Suiza | 1.19 | 1.13 | 1.37 | 2.16 | 2,77 | 3.83 | 1.10 | 1.36 | 1.17 | 1.16 | 1.36 | 1.54 | 2.52 | 2.29 | 1.46 | 1.20 | 1.16 | 1,50 | 1.21 | Suiza | |
| CEU | 1.12 | 1.29 | 1.21 | 1,59 | 1,99 | 2,89 | 1.13 | 1.06 | 1.07 | 1.13 | 1.56 | 1.84 | 1.87 | 1,74 | 1.28 | 1.56 | 1.34 | 1.09 | 1.21 | CEU | |
| Austria | Bulgaria | República Checa | Estonia | Finlandia (Helsinki) | Finlandia (Kuusamo) | Francia | Alemania del Norte | Alemania del Sur | Hungría | Italia del Norte | Italia del Sur | Letonia | Lituania | Polonia | Rusia | España | Suecia | Suiza | CEU |
CEU – Residentes de Utah con ascendencia del norte y oeste de Europa.
Historia de la investigación
Marcadores genéticos clásicos (por poder)
Uno de los primeros investigadores en realizar estudios genéticos fue Luigi Luca Cavalli-Sforza . Utilizó marcadores genéticos clásicos para analizar el ADN por proxy. Este método estudia las diferencias en las frecuencias de rasgos alélicos particulares, a saber, polimorfismos de proteínas que se encuentran en la sangre humana (como los grupos sanguíneos ABO , antígenos sanguíneos Rhesus, loci HLA , inmunoglobulinas , isoenzimas G6PD , entre otros). Posteriormente, su equipo calculó la distancia genética entre poblaciones, basándose en el principio de que dos poblaciones que comparten frecuencias similares de un rasgo están más estrechamente relacionadas que las poblaciones que tienen frecuencias más divergentes del rasgo. [80] : 51
A partir de esto, construyó árboles filogenéticos que mostraban las distancias genéticas en forma de diagrama. Su equipo también realizó análisis de componentes principales , que son buenos para analizar datos multivariados con una pérdida mínima de información. La información que se pierde se puede restaurar parcialmente generando un segundo componente principal, y así sucesivamente. [80] : 39 A su vez, la información de cada componente principal ( CP ) individual se puede presentar gráficamente en mapas sintéticos . Estos mapas muestran picos y valles, que representan poblaciones cuyas frecuencias genéticas toman valores extremos en comparación con otras en el área estudiada. [80] : 51
Los picos y valles que suelen estar conectados por gradientes suaves se denominan clines . Los clines genéticos pueden generarse por adaptación al medio ambiente ( selección natural ), flujo genético continuo entre dos poblaciones inicialmente diferentes o una expansión demográfica en un entorno escasamente poblado, con poca mezcla inicial con poblaciones existentes. [104] : 390 Cavalli-Sforza relacionó estos gradientes con movimientos poblacionales prehistóricos postulados, basados en teorías arqueológicas y lingüísticas. Sin embargo, dado que no se conocen las profundidades temporales de dichos patrones, "asociarlos con eventos demográficos particulares suele ser especulativo". [44]
Análisis directo de ADN
Los estudios que utilizan análisis directo de ADN son ahora abundantes y pueden utilizar ADN mitocondrial (ADNmt) , la porción no recombinante del cromosoma Y (NRY) o incluso ADN autosómico. El ADNmt y el ADN NRY comparten algunas características similares, que los han hecho particularmente útiles en la antropología genética. Estas propiedades incluyen la herencia directa e inalterada del ADNmt y el ADN NRY de madre a descendencia y de padre a hijo, respectivamente, sin los efectos de "alteración" de la recombinación genética . También suponemos que estos loci genéticos no se ven afectados por la selección natural y que el principal proceso responsable de los cambios en los pares de bases ha sido la mutación (que se puede calcular). [23] : 58
El menor tamaño efectivo de la población del NRY y del mtADN aumenta las consecuencias de la deriva y del efecto fundador, en relación con los autosomas, lo que hace que la variación del NRY y del mtADN sea un índice potencialmente sensible de la composición de la población. [44] [28] [25] Estas suposiciones biológicamente plausibles no son concretas; Rosser sugiere que las condiciones climáticas pueden afectar la fertilidad de ciertos linajes. [44]
La tasa de mutación subyacente utilizada por los genetistas es más cuestionable. A menudo utilizan diferentes tasas de mutación y los estudios con frecuencia llegan a conclusiones muy diferentes. [44] El NRY y el ADNmt pueden ser tan susceptibles a la deriva que algunos patrones antiguos pueden haberse oscurecido. Otra suposición es que las genealogías de las poblaciones se aproximan a las genealogías de los alelos . Guido Barbujani señala que esto solo es válido si los grupos de población se desarrollan a partir de un conjunto genéticamente monomórfico de fundadores. Barbujani sostiene que no hay razón para creer que Europa fue colonizada por poblaciones monomórficas. Esto daría lugar a una sobreestimación de la edad del haplogrupo, extendiendo así falsamente la historia demográfica de Europa hasta el Paleolítico Tardío en lugar de la era Neolítica . [105] Se puede obtener una mayor certeza sobre la cronología a partir de estudios del ADN antiguo (véase más adelante), pero hasta ahora estos han sido comparativamente pocos.
Mientras que los haplogrupos de ADN-Y y ADNmt representan sólo un pequeño componente del acervo genético de una persona, el ADN autosómico tiene la ventaja de contener cientos de miles de loci genéticos examinables, lo que proporciona un panorama más completo de la composición genética. Las relaciones de descendencia sólo se pueden determinar sobre una base estadística, porque el ADN autosómico sufre recombinación. Un solo cromosoma puede registrar una historia para cada gen. Los estudios autosómicos son mucho más fiables para mostrar las relaciones entre poblaciones existentes, pero no ofrecen las posibilidades de desentrañar sus historias de la misma manera que prometen los estudios de ADNmt y NRY, a pesar de sus muchas complicaciones.
Los estudios genéticos se basan en numerosos supuestos y sufren limitaciones metodológicas, como el sesgo de selección y fenómenos de confusión como la deriva genética , los efectos de base y de cuello de botella, que causan grandes errores, en particular en los estudios de haplogrupos. No importa cuán precisa sea la metodología, las conclusiones derivadas de tales estudios se compilan sobre la base de cómo el autor prevé que sus datos se ajustan a las teorías arqueológicas o lingüísticas establecidas. [ cita requerida ]
Véase también
- General
- Genética por grupo europeo
- Historia genética de Italia
- Historia genética de las Islas Británicas
- Historia genética de la Península Ibérica
- Estudios genéticos sobre bosnios
- Estudios genéticos sobre búlgaros
- Estudios genéticos sobre croatas
- Estudios genéticos sobre judíos
- Estudios genéticos sobre los rusos
- Estudios genéticos sobre los samis
- Estudios genéticos sobre los serbios
- Estudios genéticos en población turca
Referencias
Citas en línea
- ^ ab Nelis M, Esko T, Mägi R, Zimprich F, Zimprich A, Toncheva D, et al. (2009). "Estructura genética de los europeos: una visión desde el noreste". PLOS ONE . 4 (5): e5472. Bibcode :2009PLoSO...4.5472N. doi : 10.1371/journal.pone.0005472 . PMC 2675054 . PMID 19424496.
- ^ Seguin-Orlando A, Korneliussen TS, Sikora M, Malaspinas AS, Manica A, Moltke I, et al. (noviembre de 2014). "Paleogenómica. Estructura genómica en europeos que data de al menos 36.200 años atrás". Science . 346 (6213): 1113–1118. Bibcode :2014Sci...346.1113S. doi :10.1126/science.aaa0114. PMID 25378462. S2CID 206632421.
- ^ abc Beleza S, Santos AM, McEvoy B, Alves I, Martinho C, Cameron E, et al. (enero de 2013). "El momento del aclaramiento de la pigmentación en los europeos". Biología molecular y evolución . 30 (1): 24–35. doi :10.1093/molbev/mss207. PMC 3525146 . PMID 22923467.
- ^ ab Jones ER, Gonzalez-Fortes G, Connell S, Siska V, Eriksson A, Martiniano R, et al. (noviembre de 2015). "Los genomas del Paleolítico superior revelan raíces profundas de los euroasiáticos modernos". Nature Communications . 6 (1): 8912. Bibcode :2015NatCo...6.8912J. doi :10.1038/ncomms9912. PMC 4660371 . PMID 26567969.
- ^ El reemplazo de la población en el Neolítico, y nuevamente en la Edad del Bronce, fue casi completo en la Gran Bretaña prehistórica , y la población de la Gran Bretaña mesolítica representa aproximadamente el 10% de la ascendencia de la población indígena británica moderna. Olalde I, Brace S, Allentoft ME, Armit I, Kristiansen K, Booth T, et al. (marzo de 2018). "El fenómeno del vaso campaniforme y la transformación genómica del noroeste de Europa". Nature . 555 (7695): 190–196. Bibcode :2018Natur.555..190O. doi :10.1038/nature25738. PMC 5973796 . PMID 29466337.
- ^ Haak, Wolfgang; Lazaridis, Iosif; Patterson, Nick; Rohland, Nadin; Mallick, Swapan; Llamas, Bastien; Brandt, Guido; Nordenfelt, Susanne; Harney, Eadaoin; Stewardson, Kristin; Fu, Qiaomei; Mittnik, Alissa; Bánffy, Eszter; Economou, Christos; Francken, Michael (2015). "La migración masiva desde la estepa fue una fuente de lenguas indoeuropeas en Europa". Nature . 522 (7555): 207–211. arXiv : 1502.02783 . Bibcode :2015Natur.522..207H. doi :10.1038/nature14317. ISSN 1476-4687. PMC 5048219 . Número de modelo: PMID25731166.
- ^ Lazaridis I, Patterson N, Mittnik A, Renaud G, Mallick S, Kirsanow K, et al. (septiembre de 2014). "Los genomas humanos antiguos sugieren tres poblaciones ancestrales para los europeos actuales". Nature . 513 (7518): 409–413. arXiv : 1312.6639 . Bibcode :2014Natur.513..409L. doi :10.1038/nature13673. PMC 4170574 . PMID 25230663.
- ^ Desde Lazaridis et al. (2014), estudios posteriores han refinado el panorama del mestizaje entre EEF y WHG. En un análisis de 2017 de 180 conjuntos de datos de ADN antiguo de los períodos Calcolítico y Neolítico de Hungría, Alemania y España se encontró evidencia de un período prolongado de mestizaje EEF-WHG. La mezcla se produjo a nivel regional, a partir de poblaciones de cazadores-recolectores locales, de modo que las poblaciones de las tres regiones (Alemania, Iberia y Hungría) fueron genéticamente distinguibles en todas las etapas del período Neolítico, con una proporción gradualmente creciente de ascendencia WHG de poblaciones agrícolas a lo largo del tiempo. Esto sugiere que después de la expansión inicial de los primeros agricultores, no hubo más migraciones de largo alcance lo suficientemente sustanciales como para homogeneizar la población agrícola, y que las poblaciones agrícolas y de cazadores-recolectores coexistieron durante muchos siglos, con una mezcla gradual continua a lo largo del quinto al cuarto milenio a. C. (en lugar de un solo evento de mezcla en el contacto inicial). Lipson M, Szécsényi-Nagy A, Mallick S, Pósa A, Stégmár B, Keerl V, et al. (noviembre de 2017). "Los transectos paleogenómicos paralelos revelan una historia genética compleja de los primeros agricultores europeos". Naturaleza . 551 (7680): 368–372. Código Bib :2017Natur.551..368L. doi : 10.1038/naturaleza24476. PMC 5973800 . PMID 29144465.
- ^ "No existe tal cosa como un europeo 'puro', ni ningún otro". Ciencia | AAAS . 15 de mayo de 2017.
- ^ Curry A (2019). «Las pruebas genéticas revelan que Europa es un crisol de razas, formado por inmigrantes». National Geographic . Archivado desde el original el 9 de julio de 2019.
- ^ Lipson y col. (2017), figura 2.
- ^ Dutchen S (23 de noviembre de 2015). "La agricultura está en su ADN". Facultad de Medicina de Harvard . Consultado el 25 de noviembre de 2015 .
- ^ abcdefghijklm Fu Q, Posth C, Hajdinjak M, Petr M, Mallick S, Fernandes D, et al. (junio de 2016). "La historia genética de la Edad de Hielo en Europa". Nature . 534 (7606): 200–205. Bibcode :2016Natur.534..200F. doi :10.1038/nature17993. PMC 4943878 . PMID 27135931.
- ^ Incluso antes de la aparición de los estudios genéticos, algunos antropólogos creían haber descubierto esqueletos que representaban "híbridos" de neandertales y humanos modernos. Estos resultados se consideraron "ambiguos". La evidencia arqueológica apunta a un cambio abrupto de los artefactos neandertales a los relacionados con AMH durante el Paleolítico superior. Klein RG (marzo de 2003). "Paleoantropología. ¿Hacia dónde van los neandertales?". Science . 299 (5612): 1525–1527. doi :10.1126/science.1082025. PMID 12624250. S2CID 161836323.
- ^ Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, et al. (enero de 2014) [En línea 2013]. "La secuencia completa del genoma de un neandertal de las montañas de Altai". Nature . 505 (7481): 43–49. Bibcode :2014Natur.505...43P. doi :10.1038/nature12886. PMC 4031459 . PMID 24352235.
- ^ Hardy J, Pittman A, Myers A, Gwinn-Hardy K, Fung HC, de Silva R, et al. (agosto de 2005). "Evidencia que sugiere que el Homo neanderthalensis contribuyó con el haplotipo H2 MAPT al Homo sapiens". Biochemical Society Transactions . 33 (Pt 4): 582–585. doi :10.1042/bst0330582. PMID 16042549.
Sugerimos que el haplotipo H2 se deriva del Homo neanderthalensis y entró en las poblaciones de H. sapiens durante la coexistencia de estas especies en Europa desde hace aproximadamente 45 000 a 18 000 años y que el haplotipo H2 ha estado bajo presión de selección desde ese momento, posiblemente debido al papel de este haplotipo H1 en la enfermedad neurodegenerativa"...."El locus tau (MAPT ) es muy inusual. En una región de aproximadamente 1,8 Mb, existen dos clados de haplotipos en las poblaciones europeas, H1 y H2 [6,7]. En otras poblaciones, solo se encuentra el H1 y muestra un patrón normal de recombinación.
- ^ Shaw-Smith C, Pittman AM, Willatt L, Martin H, Rickman L, Gribble S, et al. (septiembre de 2006). "La microdeleción que abarca MAPT en el cromosoma 17q21.3 está asociada con retraso del desarrollo y discapacidad de aprendizaje". Nature Genetics . 38 (9): 1032–1037. doi :10.1038/ng1858. PMID 16906163. S2CID 38047848.
- ^ Zody MC, Jiang Z, Fung HC, Antonacci F, Hillier LW, Cardone MF, et al. (septiembre de 2008). "Alternación evolutiva de la región de inversión MAPT 17q21.31". Nature Genetics . 40 (9): 1076–1083. doi :10.1038/ng.193. PMC 2684794 . PMID 19165922.
- ^ Preguntas frecuentes sobre introgresión y microcefalia John Hawks [1]
- ^ Almos PZ, Horváth S, Czibula A, Raskó I, Sipos B, Bihari P, et al. (noviembre de 2008). "Variación genómica relacionada con el haplotipo H1 tau en 17q21.3 como herencia asiática de la población gitana europea". Heredity . 101 (5): 416–419. doi : 10.1038/hdy.2008.70 . PMID 18648385.
En este estudio, examinamos la frecuencia de una inversión de 900 kb en 17q21.3 en las poblaciones gitana y caucásica de Hungría, que puede reflejar el origen asiático de las poblaciones gitanas. De los dos haplotipos (H1 y H2), se cree que H2 es exclusivamente de origen caucásico, y es probable que su aparición en otros grupos raciales refleje una mezcla. En nuestra muestra, el haplotipo H1 fue significativamente más frecuente en la población gitana (89,8 vs 75,5%, P<0,001) y se encontraba en desequilibrio de Hardy-Weinberg (P=0,017). La región 17q21.3 incluye el gen de la proteína tau asociada a microtúbulos, y este resultado podría implicar una mayor sensibilidad a las tauopatías multifactoriales relacionadas con el haplotipo H1 entre los gitanos.
- ^ Wade N (2009-12-02). "Científicos en Alemania redactan el genoma del neandertal". The New York Times . Consultado el 2010-05-03 .
- ^ "Los neandertales son 'diferentes de nosotros'". BBC . 2009-12-02.
- ^ abcd Milisauskas S (2002). Prehistoria europea: un estudio . Birkhauser . ISBN 978-0-306-46793-6.
- ^ Teschler-Nicola, María; Fernández, Daniel; Händel, Marc; Einwögerer, Thomas; Simón, Ulrich; Neugebauer-Maresch, Christine; Tangl, Stefan; Heimel, Patricio; Dobsak, Toni; Retzmann, Anika; Prohaska, Thomas (6 de noviembre de 2020). "El ADN antiguo revela gemelos recién nacidos monocigóticos del Paleolítico superior". Biología de las Comunicaciones . 3 (1): 650.doi : 10.1038 /s42003-020-01372-8 . ISSN 2399-3642. PMC 7648643 . PMID 33159107.
- ^ abcdefghi Semino O, Passarino G, Oefner PJ, Lin AA, Arbuzova S, Beckman LE, et al. (Noviembre de 2000). "El legado genético del Homo sapiens sapiens paleolítico en los europeos actuales: una perspectiva del cromosoma Y". Ciencia . 290 (5494): 1155-1159. Código Bib : 2000 Ciencia... 290.1155S. doi : 10.1126/ciencia.290.5494.1155. PMID 11073453.Nota: Los nombres de los haplogrupos son diferentes en este artículo. Por ejemplo: el haplogrupo I se denomina M170
- ^ ab Wells RS, Yuldasheva N, Ruzibakiev R, Underhill PA, Evseeva I, Blue-Smith J, et al. (agosto de 2001). "El corazón de Eurasia: una perspectiva continental sobre la diversidad del cromosoma Y". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (18): 10244–10249. Bibcode :2001PNAS...9810244W. doi : 10.1073/pnas.171305098 . PMC 56946 . PMID 11526236.
- ^ Karafet TM, Mendez FL, Meilerman MB, Underhill PA, Zegura SL, Hammer MF (mayo de 2008). "Nuevos polimorfismos binarios remodelan y aumentan la resolución del árbol de haplogrupos del cromosoma Y humano". Genome Research . 18 (5): 830–838. doi :10.1101/gr.7172008. PMC 2336805 . PMID 18385274.
- ^ abcd Richards M, Macaulay V, Hickey E, Vega E, Sykes B, Guida V, et al. (noviembre de 2000). "Rastreo de linajes fundadores europeos en el acervo de ADNmt del Cercano Oriente". American Journal of Human Genetics . 67 (5): 1251–1276. doi :10.1016/S0002-9297(07)62954-1. PMC 1288566 . PMID 11032788.
- ^ Torroni A, Bandelt HJ, Macaulay V, Richards M, Cruciani F, Rengo C, et al. (octubre de 2001). "Una señal, del ADNmt humano, de recolonización postglacial en Europa". American Journal of Human Genetics . 69 (4): 844–852. doi :10.1086/323485. PMC 1226069 . PMID 11517423.
- ^ Pala M, Achilli A, Olivieri A, Hooshiar Kashani B, Perego UA, Sanna D, et al. (junio de 2009). "Haplogrupo mitocondrial U5b3: un eco lejano del epipaleolítico en Italia y el legado de los primeros sardos". Revista Estadounidense de Genética Humana . 84 (6): 814–821. doi :10.1016/j.ajhg.2009.05.004. PMC 2694970 . PMID 19500771.
- ^ ab Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, et al. (julio de 2004). "La filogeografía del haplogrupo I del cromosoma Y revela dominios distintos del flujo genético prehistórico en Europa" (PDF) . American Journal of Human Genetics . 75 (1): 128–137. doi :10.1086/422196. PMC 1181996 . PMID 15162323. Archivado desde el original (PDF) el 24 de junio de 2009 . Consultado el 4 de julio de 2009 .
- ^ Cinnioglu et al. Excavación de estratos de haplotipos del cromosoma Y en Anatolia . 2003
- ^ abcd Pericić M, Lauc LB, Klarić IM, Rootsi S, Janićijevic B, Rudan I, et al. (octubre de 2005). "El análisis filogenético de alta resolución del sudeste de Europa rastrea los principales episodios de flujo genético paterno entre las poblaciones eslavas". Biología molecular y evolución . 22 (10): 1964–1975. doi : 10.1093/molbev/msi185 . PMID 15944443.
- ^ Passarino G, Semino O, Magri C, Al-Zahery N, Benuzzi G, Quintana-Murci L, et al. (septiembre de 2001). "El haplotipo 11 de 49a,f es un nuevo marcador del linaje EU19 que rastrea las migraciones desde las regiones septentrionales del Mar Negro". Inmunología humana . 62 (9): 922–932. doi :10.1016/S0198-8859(01)00291-9. PMID 11543894.
- ^ Bilton DT, Mirol PM, Mascheretti S, Fredga K, Zima J, Searle JB (julio de 1998). "La Europa mediterránea como área de endemismo para pequeños mamíferos en lugar de una fuente para la colonización postglacial hacia el norte". Actas. Ciencias Biológicas . 265 (1402): 1219–1226. doi :10.1098/rspb.1998.0423. PMC 1689182. PMID 9699314 .
- ^ abc Dutchen S (2 de mayo de 2016). «Historia sobre hielo». Facultad de Medicina de Harvard . Consultado el 11 de mayo de 2016 .
- ^ abcde Mathieson I, Lazaridis I, Rohland N, Mallick S, Patterson N, Roodenberg SA, et al. (diciembre de 2015). "Patrones de selección en todo el genoma en 230 antiguos euroasiáticos". Nature . 528 (7583): 499–503. Bibcode :2015Natur.528..499M. doi :10.1038/nature16152. PMC 4918750 . PMID 26595274.
- ^ Sikora M, Carpenter ML, Moreno-Estrada A, Henn BM, Underhill PA, Sánchez-Quinto F, et al. (mayo de 2014). "El análisis genómico poblacional de genomas antiguos y modernos proporciona nuevos conocimientos sobre la ascendencia genética del Hombre de los Hielos tirolés y la estructura genética de Europa". PLOS Genetics . 10 (5): e1004353. doi : 10.1371/journal.pgen.1004353 . PMC 4014435 . PMID 24809476.
- ^ Haak W, Balanovsky O, Sanchez JJ, Koshel S, Zaporozhchenko V, Adler CJ, et al. (noviembre de 2010). "El ADN antiguo de los agricultores neolíticos europeos revela sus afinidades con el Cercano Oriente". PLOS Biology . 8 (11): e1000536. doi : 10.1371/journal.pbio.1000536 . PMC 2976717 . PMID 21085689.
- ^ Perlès C, Monthel G (2001) El Neolítico temprano en Grecia: las primeras comunidades agrícolas en Europa. Cambridge University Press, Cambridge.
- ^ Runnels C (2003) Los orígenes del Neolítico griego: una visión personal, en Ammerman y Biagi (eds. 2003).
- ^ Zeder MA (agosto de 2008). "Domesticación y agricultura temprana en la cuenca mediterránea: orígenes, difusión e impacto". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (33): 11597–11604. Bibcode :2008PNAS..10511597Z. doi : 10.1073/pnas.0801317105 . PMC 2575338 . PMID 18697943.
- ^ Piazza A, Cavalli-Sforza LL, Menozzi P (1994). Historia y geografía de los genes humanos . Princeton, NJ: Princeton University Press. ISBN 978-0-691-08750-4.
- ^ abcdef Rosser ZH, Zerjal T, Hurles ME, Adojaan M, Alavantic D, Amorim A, et al. (diciembre de 2000). "La diversidad del cromosoma Y en Europa es clinal y está influida principalmente por la geografía, más que por el idioma". American Journal of Human Genetics . 67 (6): 1526–1543. doi :10.1086/316890. PMC 1287948 . PMID 11078479. Archivado desde el original el 6 de mayo de 2008.
- ^ Underhill PA, Kivisild T (2007). "Uso del cromosoma Y y la estructura poblacional del ADN mitocondrial para rastrear las migraciones humanas". Revisión anual de genética . 41 : 539–564. doi :10.1146/annurev.genet.41.110306.130407. PMID 18076332.
- ^ Los datos del cromosoma Y muestran una señal de una migración separada del Pleistoceno tardío desde África a Europa a través del Sinaí, como lo evidencia la distribución de los linajes del haplogrupo E3b, que no se manifiesta en las distribuciones de haplogrupos de ADNmt.
- ^ ab Battaglia V, Fornarino S, Al-Zahery N, Olivieri A, Pala M, Myres NM, et al. (junio de 2009). "Evidencia cromosómica Y de la difusión cultural de la agricultura en el sudeste de Europa". Revista Europea de Genética Humana . 17 (6): 820–830. doi :10.1038/ejhg.2008.249. PMC 2947100 . PMID 19107149.
- ^ Cruciani F, La Fratta R, Trombetta B, Santolamazza P, Sellitto D, Colomb EB, et al. (junio de 2007). "Rastreando los movimientos humanos masculinos del pasado en el norte y este de África y el oeste de Eurasia: nuevas pistas a partir de los haplogrupos del cromosoma Y E-M78 y J-M12". Biología molecular y evolución . 24 (6): 1300–1311. doi : 10.1093/molbev/msm049 . PMID 17351267. Archivado desde el original el 10 de octubre de 2017 . Consultado el 22 de julio de 2009 .Véase también Datos suplementarios.
- ^ Lacan M, Keyser C, Ricaut FX, Brucato N, Duranthon F, Guilaine J, Crubézy E, Ludes B (junio de 2011). "El ADN antiguo revela la difusión masculina a través de la ruta mediterránea neolítica". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (24): 9788–91. Bibcode :2011PNAS..108.9788L. doi : 10.1073/pnas.1100723108 . PMC 3116412 . PMID 21628562.
- ^ ab Bird S (2007). "El haplogrupo E3b1a2 como posible indicador de asentamiento en la Gran Bretaña romana por soldados de origen balcánico". Journal of Genetic Genealogy . 3 (2). Archivado desde el original el 22 de abril de 2016 . Consultado el 25 de agosto de 2009 .
- ^ Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (junio de 2015). "La migración masiva desde la estepa fue una fuente de lenguas indoeuropeas en Europa". Nature . 522 (7555): 207–211. arXiv : 1502.02783 . Bibcode :2015Natur.522..207H. doi :10.1038/nature14317. PMC 5048219 . PMID 25731166.
- ^ Callaway E (12 de febrero de 2015). «Lenguas europeas vinculadas a la migración desde el este». Nature . doi : 10.1038/nature.2015.16919 . S2CID 184180681.
- ^ Underhill PA, Myres NM, Rootsi S, Metspalu M, Zhivotovsky LA, King RJ, et al. (abril de 2010). "Separación de la coancestría postglacial de los cromosomas Y europeos y asiáticos dentro del haplogrupo R1a". Revista Europea de Genética Humana . 18 (4): 479–484. doi :10.1038/ejhg.2009.194. PMC 2987245 . PMID 19888303.
- ^ Ingman M, Gyllensten U (enero de 2007). "Un vínculo genético reciente entre los sami y la región del Volga-Ural de Rusia". Revista Europea de Genética Humana . 15 (1): 115–120. doi : 10.1038/sj.ejhg.5201712 . PMID 16985502.
- ^ Wiik K. "¿Quiénes son los finlandeses?" (PDF) . En Suominen M, Arppe A, Airola A, Heinämäki O, Miestamo M, Määttä U, Niemi J, Pitkänen KK, Sinnemäki K (eds.). Un hombre de medida Festschrift en honor a Fred Karlsson . págs. 97-108 . Consultado el 16 de marzo de 2016 .
- ^ ab Mirabal S, Varljen T, Gayden T, Regueiro M, Vujovic S, Popovic D, et al. (julio de 2010). "Repeticiones cortas en tándem del cromosoma Y humano: una historia de aculturación y migraciones como mecanismos para la difusión de la agricultura en la península de los Balcanes". American Journal of Physical Anthropology . 142 (3): 380–390. doi :10.1002/ajpa.21235. PMID 20091845.
- ^ Rootsi S, Zhivotovsky LA, Baldovic M, Kayser M, Kutuev IA, Khusainova R, et al. (febrero de 2007). "Una ruta norte en sentido contrario a las agujas del reloj del haplogrupo N del cromosoma Y desde el sudeste asiático hacia Europa". Revista Europea de Genética Humana . 15 (2): 204–211. doi : 10.1038/sj.ejhg.5201748 . PMID 17149388., Tabla suplementaria 1
- ^ abc Jones ER, Gonzalez-Fortes G, Connell S, Siska V, Eriksson A, Martiniano R, et al. (noviembre de 2015). "Los genomas del Paleolítico superior revelan raíces profundas de los euroasiáticos modernos". Nature Communications . 6 (8912): 8912. Bibcode :2015NatCo...6.8912J. doi :10.1038/ncomms9912. PMC 4660371 . PMID 26567969.
- ^ Yang, Melinda A. (6 de enero de 2022). "Una historia genética de la migración, la diversificación y la mezcla en Asia". Genética y genómica de poblaciones humanas . 2 (1): 1–32. doi : 10.47248/hpgg2202010001 . ISSN 2770-5005.
- ^ Dutchen S (17 de septiembre de 2014). «New Branch Added to European Family Tree» (Nueva rama añadida al árbol genealógico europeo). Facultad de Medicina de Harvard . Archivado desde el original el 1 de octubre de 2014. Consultado el 25 de noviembre de 2015 .
- ^ "Revelan una misteriosa 'tribu' ancestral". BBC News . 16 de noviembre de 2015.
- ^ Lazaridis I, Nadel D, Rollefson G, Merrett DC, Rohland N, Mallick S, et al. (2016). "La estructura genética de los primeros agricultores del mundo". bioRxiv 10.1101/059311 .
- ^ Mathieson I, Alpaslan-Roodenberg S, Posth C, Szécsényi-Nagy A, Rohland N, Mallick S, et al. (Marzo de 2018). "La historia genómica del sureste de Europa". Naturaleza . 555 (7695): 197–203. Código Bib :2018Natur.555..197M. doi : 10.1038/naturaleza25778. PMC 6091220 . PMID 29466330.
- ^ Busby GB, Hellenthal G, Montinaro F, Tofanelli S, Bulayeva K, Rudan I, et al. (octubre de 2015). "El papel de la mezcla reciente en la formación del paisaje genómico contemporáneo de Eurasia occidental". Current Biology . 25 (19): 2518–2526. doi :10.1016/j.cub.2015.08.007. PMC 4714572 . PMID 26387712.
- ^ Comas D, Schmid H, Braeuer S, Flaiz C, Busquets A, Calafell F, et al. (Marzo de 2004). "Polimorfismos de inserción de Alu en los Balcanes y los orígenes de los Aromuns". Anales de genética humana . 68 (parte 2): 120-127. doi :10.1046/j.1529-8817.2003.00080.x. PMID 15008791. S2CID 21773796.
- ^ Bosch E, Calafell F, González-Neira A, Flaiz C, Mateu E, Scheil HG, et al. (julio de 2006). "Los linajes paternos y maternos en los Balcanes muestran un paisaje homogéneo a pesar de las barreras lingüísticas, excepto en el caso de los aislados Aromuns". Anales de genética humana . 70 (parte 4): 459–487. doi :10.1111/j.1469-1809.2005.00251.x. PMID 16759179. S2CID 23156886.
- ^ Capelli C, Redhead N, Abernethy JK, Gratrix F, Wilson JF, Moen T, et al. (mayo de 2003). "Censo de cromosomas AY de las Islas Británicas". Current Biology . 13 (11): 979–984. doi : 10.1016/S0960-9822(03)00373-7 . hdl : 20.500.11820/8acb01f3-a7c1-45f5-89de-b796266d651e . PMID 12781138.También en 030705U491 (PDF) , consultado el 1 de junio de 2011
- ^ Balaresque P, Bowden GR, Adams SM, Leung HY, King TE, Rosser ZH, et al. (enero de 2010). "Un origen predominantemente neolítico para los linajes paternos europeos". PLOS Biology . 8 (1): e1000285. doi : 10.1371/journal.pbio.1000285 . PMC 2799514 . PMID 20087410.
- ^ ab Myres NM, Rootsi S, Lin AA, Järve M, King RJ, Kutuev I, et al. (enero de 2011). "Un importante efecto fundador del haplogrupo R1b del cromosoma Y en la era del Holoceno en Europa central y occidental". Revista Europea de Genética Humana . 19 (1): 95–101. doi :10.1038/ejhg.2010.146. PMC 3039512 . PMID 20736979.
- ^ ab Cruciani F, Trombetta B, Antonelli C, Pascone R, Valesini G, Scalzi V, et al. (junio de 2011). "Fuerte diferenciación intra e intercontinental revelada por los SNP del cromosoma Y M269, U106 y U152". Forensic Science International. Genética . 5 (3): e49–e52. doi :10.1016/j.fsigen.2010.07.006. hdl :11573/226727. PMID 20732840.
- ^ Morelli L, Contu D, Santoni F, Whalen MB, Francalacci P, Cucca F (abril de 2010). "Una comparación de la variación del cromosoma Y en Cerdeña y Anatolia es más consistente con la difusión cultural que con la difusión démica de la agricultura". MÁS UNO . 5 (4): e10419. Código Bib : 2010PLoSO...510419M. doi : 10.1371/journal.pone.0010419 . PMC 2861676 . PMID 20454687.
- ^ ab Kayser M, Lao O, Anslinger K, Augustin C, Bargel G, Edelmann J, et al. (septiembre de 2005). "La diferenciación genética significativa entre Polonia y Alemania sigue las fronteras políticas actuales, como lo revela el análisis del cromosoma Y". Human Genetics . 117 (5): 428–443. doi :10.1007/s00439-005-1333-9. PMID 15959808. S2CID 11066186.Se puede encontrar una copia aquí "La diferenciación genética significativa entre Polonia y Alemania sigue las fronteras políticas actuales, según revela el análisis del cromosoma Y" (PDF) . Archivado desde el original (PDF) el 2009-03-04 . Consultado el 2008-11-02 ..
- ^ Bowden GR, Balaresque P, King TE, Hansen Z, Lee AC, Pergl-Wilson G, Hurley E, Roberts SJ, Waite P, Jesch J, Jones AL, Thomas MG, Harding SE, Jobling MA (febrero de 2008). "Excavando estructuras de población pasadas mediante muestreo basado en apellidos: el legado genético de los vikingos en el noroeste de Inglaterra". Biología molecular y evolución . 25 (2): 301–9. doi :10.1093/molbev/msm255. PMC 2628767 . PMID 18032405.
- ^ Lall GM, Larmuseau MH, Wetton JH, Batini C, Hallast P, Huszar TI, et al. (Marzo de 2021). "La subdivisión del haplogrupo R1a1 del cromosoma Y revela linajes de dispersión de los vikingos nórdicos en Gran Bretaña". Revista europea de genética humana . 29 (3): 512–523. doi :10.1038/s41431-020-00747-z. PMC 7940619 . PMID 33139852.
- ^ Kasperaviciūte D, Kucinskas V, Stoneking M (septiembre de 2004). "Variación del cromosoma Y y del ADN mitocondrial en lituanos". Anales de genética humana . 68 (parte 5): 438–52. doi :10.1046/j.1529-8817.2003.00119.x. PMID 15469421. S2CID 26562505.
- ^ Cruciani F, La Fratta R, Santolamazza P, Sellitto D, Pascone R, Moral P, et al. (mayo de 2004). "El análisis filogeográfico de los cromosomas y del haplogrupo E3b (E-M215) revela múltiples eventos migratorios dentro y fuera de África". American Journal of Human Genetics . 74 (5): 1014–1022. doi :10.1086/386294. PMC 1181964 . PMID 15042509.
- ^ Semino O, Magri C, Benuzzi G, Lin AA, Al-Zahery N, Battaglia V, et al. (mayo de 2004). "Origen, difusión y diferenciación de los haplogrupos E y J del cromosoma Y: inferencias sobre la neolitización de Europa y eventos migratorios posteriores en el área mediterránea". American Journal of Human Genetics . 74 (5): 1023–1034. doi :10.1086/386295. PMC 1181965 . PMID 15069642.
- ^ ab Derenko M, Malyarchuk B, Denisova G, Perkova M, Rogalla U, Grzybowski T, et al. (2012). "Análisis completo del ADN mitocondrial de haplogrupos del este de Eurasia que rara vez se encuentran en poblaciones del norte de Asia y Europa del este". PLOS ONE . 7 (2): e32179. Bibcode :2012PLoSO...732179D. doi : 10.1371/journal.pone.0032179 . PMC 3283723 . PMID 22363811.
- ^ Cerezo M, Achilli A, Olivieri A, Perego UA, Gómez-Carballa A, Brisighelli F, et al. (mayo de 2012). "Reconstrucción de antiguos vínculos de ADN mitocondrial entre África y Europa". Investigación del genoma . 22 (5): 821–826. doi :10.1101/gr.134452.111. PMC 3337428 . PMID 22454235.
- ^ abcdef Cavalli-Sforza LL , Menozzi P, Piazza A (1993). La historia y geografía de los genes humanos. Prensa de la Universidad de Princeton . ISBN 978-0-691-08750-4. Consultado el 22 de julio de 2009 .
- ^ abc Lao O, Lu TT, Nothnagel M, Junge O, Freitag-Wolf S, Caliebe A, et al. (agosto de 2008). "Correlación entre la estructura genética y geográfica en Europa". Current Biology . 18 (16): 1241–1248. doi : 10.1016/j.cub.2008.07.049 . PMID 18691889. S2CID 16945780.
- ^ Cavalli-Sforza, Luca; Menozzi, Paolo; Plaza, Alberto (1994). La historia y geografía de los genes humanos . Prensa de la Universidad de Princeton, pág. 272
- ^ Estructura genética de los europeos: una visión desde el noreste, Nelis et al. 2009
- ^ Nelis, Mari; Esko, Tõnu; Mägi, Reedik; Zimprich, Fritz; Zimprich, Alejandro; Toncheva, Draga; Karachanak, Sena; Piskáčková, Teresa; Balaščák, Ivan; Peltonen, Leena; Jakkula, Eveliina; Rehnström, Karola; Lathrop, Marcos; Brezo, Simón; Galán, Pilar (8 de mayo de 2009). "Estructura genética de los europeos: una visión desde el noreste". MÁS UNO . 4 (5): e5472. doi : 10.1371/journal.pone.0005472 . ISSN 1932-6203.
- ^ ab Tian C, Kosoy R, Nassir R, Lee A, Villoslada P, Klareskog L, et al. (2009). "Subestructura genética de la población europea: definición adicional de marcadores informativos de ascendencia para distinguir entre diversos grupos étnicos europeos". Medicina molecular . 15 (11–12): 371–383. doi :10.2119/molmed.2009.00094. PMC 2730349 . PMID 19707526.
- ^ Seldin MF, Shigeta R, Villoslada P, Selmi C, Tuomilehto J, Silva G, et al. (septiembre de 2006). "Subestructura de la población europea: agrupamiento de las poblaciones del norte y del sur". PLOS Genetics . 2 (9): e143. doi : 10.1371/journal.pgen.0020143 . PMC 1564423 . PMID 17044734.
- ^ abc Bauchet M, McEvoy B, Pearson LN, Quillen EE, Sarkisian T, Hovhannesyan K, et al. (mayo de 2007). "Medición de la estratificación de la población europea con datos de genotipos de microarrays". American Journal of Human Genetics . 80 (5): 948–956. doi :10.1086/513477. PMC 1852743 . PMID 17436249.
- ^ Novembre J, Johnson T, Bryc K, Kutalik Z, Boyko AR, Auton A, et al. (noviembre de 2008). "Los genes reflejan la geografía en Europa". Nature . 456 (7218): 98–101. Bibcode :2008Natur.456...98N. doi :10.1038/nature07331. PMC 2735096 . PMID 18758442.
- ^ Oleksyk TK, Wolfsberger WW, Weber AM, Shchubelka K, Oleksyk OT, Levchuk O, et al. (enero de 2021). "Diversidad del genoma en Ucrania". GigaCiencia . 10 (1). doi : 10.1093/gigascience/giaa159 . PMC 7804371 . PMID 33438729.
- ^ Zhernakova DV, Brukhin V, Malov S, Oleksyk TK, Koepfli KP, Zhuk A, et al. (enero de 2020). "Análisis de secuencias de todo el genoma de poblaciones étnicas en Rusia". Genómica . 112 (1): 442–458. doi : 10.1186/s13742-015-0095-0 . PMC 4644275 . PMID 30902755.
- ^ Khrunin AV, Khokhrin DV, Filippova IN, Esko T, Nelis M, Bebyakova NA, et al. (7 de marzo de 2013). "Un análisis de todo el genoma de poblaciones de la Rusia europea revela un nuevo polo de diversidad genética en el norte de Europa". PLOS ONE . 8 (3): e58552. Bibcode :2013PLoSO...858552K. doi : 10.1371/journal.pone.0058552 . PMC 3591355 . PMID 23505534.
- ^ Saag L, Laneman M, Varul L, Malve M, Valk H, Razzak MA, et al. (mayo de 2019). "La llegada de la ascendencia siberiana conectando el Báltico oriental con los hablantes urálicos más al este". Current Biology . 29 (10): 1701–1711.e16. doi :10.1016/j.cub.2019.04.026. PMC 6544527 . PMID 31080083.
- ^ Lazaridis I, Nadel D, Rollefson G, Merrett DC, Rohland N, Mallick S, et al. (agosto de 2016). "Información genómica sobre el origen de la agricultura en el antiguo Cercano Oriente". Nature . 536 (7617): 419–424. Bibcode :2016Natur.536..419L. doi :10.1038/nature19310. PMC 5003663 . PMID 27459054.
abajo a la izquierda: cazadores-recolectores occidentales (WHG), arriba a la izquierda: cazadores-recolectores orientales (EHG), abajo a la derecha: Levante neolítico y natufienses, arriba a la derecha: Irán neolítico. Esto sugiere la hipótesis de que diversos euroasiáticos occidentales antiguos pueden modelarse como mezclas de tan solo cuatro corrientes de ascendencia relacionadas con estas poblaciones.
- ^ Yang MA, Gao X, Theunert C, Tong H, Aximu-Petri A, Nickel B, et al. (octubre de 2017). "Un individuo de 40.000 años de Asia proporciona información sobre la estructura poblacional temprana en Eurasia". Current Biology . 27 (20): 3202–3208.e9. doi :10.1016/j.cub.2017.09.030. PMC 6592271 . PMID 29033327.
- ^ Lamnidis, Thiseas C.; Majander, Kerttu; Jeong, Choongwon; Salmela, Elina; Wesman, Anna; Moiseyev, Vyacheslav; Khartanovich, Valéry; Balanovsky, Oleg; Ongyerth, Matías; Weihmann, Antje; Sajantila, Antti; Kelso, Janet; Pääbo, Svante; Onkamo, Päivi; Haak, Wolfgang (27 de noviembre de 2018). "Los antiguos genomas fennoscandios revelan el origen y la propagación de la ascendencia siberiana en Europa". Comunicaciones de la naturaleza . 9 (1): 5018. Código Bib : 2018NatCo...9.5018L. doi :10.1038/s41467-018-07483-5. ISSN 2041-1723. Número de modelo : PMID 30479341 .
- ^ Santos, Patricia; González-Fortes, Gloria; Trucchi, Emiliano; Ceolín, Andrea; Cordoni, Guido; Guardián, Cristina; Longobardi, Giuseppe; Barbujani, Guido (diciembre de 2020). "Más regla que excepción: evidencia paralela de migraciones antiguas en gramáticas y genomas de hablantes finno-ugrios". Genes . 11 (12): 1491. doi : 10.3390/genes11121491 . ISSN 2073-4425. PMC 7763979 . PMID 33322364.
- ^ Wong, Emily HM (2015). "Reconstrucción de la historia genética de las poblaciones de Siberia y el noreste de Europa". Genome Research . 27 (1): 1–14. doi :10.1101/gr.202945.115. PMC 5204334 . PMID 27965293.
- ^ Triska, Petr; Chekanov, Nikolay; Stepanov, Vadim; Khusnutdinova, Elza K.; Kumar, Ganesh Prasad Arun; Akhmetova, Vita; Babalyan, Konstantin; Boulygina, Eugenia; Jarkov, Vladimir; Gubina, Marina; Khidiyatova, Irina; Khitrinskaya, Irina; Khrameeva, Ekaterina E.; Husainova, Rita; Konovalova, Natalia (28 de diciembre de 2017). "Entre el lago Baikal y el mar Báltico: historia genómica de la puerta de entrada a Europa". Genética BMC . 18 (1): 110. doi : 10.1186/s12863-017-0578-3 . ISSN 1471-2156. PMC 5751809 . PMID 29297395.
El análisis comparativo de los genomas modernos y antiguos sugiere que los siberianos occidentales tienen más ascendencia del norte de Europa antiguo (representada por Mal'ta) que otras poblaciones de la Federación Rusa.
- ^ Qin, Pengfei; Zhou, Ying; Lou, Haiyi; Lu, Dongsheng; Yang, Xiong; Wang, Yuchen; Jin, Li; Chung, Yeun-Jun; Xu, Shuhua (2 de abril de 2015). "Cuantificación y datación del flujo genético reciente entre poblaciones de Europa y Asia oriental". Informes científicos . 5 (1): 9500. Código Bib : 2015NatSR...5E9500Q. doi :10.1038/srep09500. ISSN 2045-2322. PMC 4382708 . PMID 25833680. "El flujo genético desde el EAS a las poblaciones finlandesas y del noreste de Rusia podría remontarse a 64,2 ± 1,1 y 45,2 ± 1,3 generaciones, o 1.862 y 1.311 años, respectivamente".
- ^ Triska, Petr; Chekanov, Nikolay; Stepanov, Vadim; et al. (2017). "Entre el lago Baikal y el mar Báltico: historia genómica de la puerta de entrada a Europa". BMC Genetics . 18 (1). doi : 10.1186/s12863-017-0578-3 . PMID 29297395.
- ^ Pankrátov, Vasili; Litvinov, Sergei; Kassian, Alexei; Shulhin, Dzmitry; Chebotarev, Lieve; Yunusbayev, Bayazit; Möls, Mart; Sahakyan, Hovhannes; Yepiskoposyan, Levón; Rootsi, Siiri; Metspalu, Ene; Golubenko, María; Ekomasova, Natalia; Akhatova, Farida; Khusnutdinova, Elza; Hola, Evelyne; Endicott, Phillip; Derenko, Miroslava; Malyarchuk, Boris; Metspalu, Mait; Davydenko, Oleg; Villems, Richard; Kushniarevich, Alena (25 de julio de 2016). "Ascendencia de Eurasia oriental en el centro de Europa: huellas genéticas de nómadas esteparios en los genomas de los tártaros lipka bielorrusos". Informes científicos . 6 : 30197. Código Bibliográfico : 2016NatSR...630197P. doi :10.1038/srep30197. PMC : 4958967. PMID: 27453128. Este artículo incorpora texto de esta fuente, que está disponible bajo la licencia CC BY 4.0.
- ^ Irving-Pease, Evan K.; Refoyo-Martínez, Alba; Barrie, William; et al. (2024). "El panorama de selección y el legado genético de los antiguos euroasiáticos". Nature . 625 (7994): 312–320. doi :10.1038/s41586-023-06705-1 – vía Nature.com.
- ^ Nelis M, Esko T, Mägi R, Zimprich F, Zimprich A, Toncheva D, et al. (8 de mayo de 2009). "Estructura genética de los europeos: una visión desde el noreste". PLOS ONE . 4 (5): e5472. Bibcode :2009PLoSO...4.5472N. doi : 10.1371/journal.pone.0005472 . PMC 2675054 . PMID 19424496.
- ^ Arredi B, Poloni ES, Tyler-Smith C (2007). "El poblamiento de Europa". Genética antropológica: teoría, métodos y aplicaciones . Cambridge University Press . ISBN 978-0-521-54697-3.
- ^ Barbujani G, Bertorelle G (enero de 2001). "Genética e historia de la población de Europa". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (1): 22–25. Bibcode :2001PNAS...98...22B. doi : 10.1073/pnas.98.1.22 . PMC 33353 . PMID 11136246.
Fuentes citadas
- Adams SM, Bosch E, Balaresque PL, Ballereau SJ, Lee AC, Arroyo E, et al. (diciembre de 2008). "El legado genético de la diversidad religiosa y la intolerancia: linajes paternos de cristianos, judíos y musulmanes en la península Ibérica". American Journal of Human Genetics . 83 (6): 725–736. doi :10.1016/j.ajhg.2008.11.007. PMC 2668061 . PMID 19061982.
- Arredi B, Poloni ES, Paracchini S, Zerjal T, Fathallah DM, Makrelouf M, et al. (Agosto de 2004). "Un origen predominantemente neolítico para la variación del ADN del cromosoma Y en el norte de África". Revista Estadounidense de Genética Humana . 75 (2): 338–345. doi :10.1086/423147. PMC 1216069 . PMID 15202071.
- Auton A, Bryc K, Boyko AR, Lohmueller KE, Novembre J, Reynolds A, et al. (mayo de 2009). "La distribución global de la diversidad genómica subraya la rica y compleja historia de las poblaciones humanas continentales". Genome Research . 19 (5): 795–803. doi :10.1101/gr.088898.108. PMC 2675968 . PMID 19218534.
- Battaglia V, Fornarino S, Al-Zahery N, Olivieri A, Pala M, Myres NM, et al. (junio de 2009). "Evidencia cromosómica Y de la difusión cultural de la agricultura en el sudeste de Europa". Revista Europea de Genética Humana . 17 (6): 820–830. doi :10.1038/ejhg.2008.249. PMC 2947100 . PMID 19107149.
- Bauchet M, McEvoy B, Pearson LN, Quillen EE, Sarkisian T, Hovhannesyan K, et al. (mayo de 2007). "Medición de la estratificación de la población europea con datos de genotipos de microarrays". American Journal of Human Genetics . 80 (5): 948–956. doi :10.1086/513477. PMC 1852743 . PMID 17436249.
- Beleza S, Gusmão L, Lopes A, Alves C, Gomes I, Giouzeli M, et al. (Marzo de 2006). "Historia microfilogeográfica y demográfica de los linajes masculinos portugueses". Anales de genética humana . 70 (Parte 2): 181–194. doi :10.1111/j.1529-8817.2005.00221.x. PMID 16626329. S2CID 4652154.[ enlace muerto ]
- Bosch E, Calafell F, Comas D, Oefner PJ, Underhill PA, Bertranpetit J (abril de 2001). "El análisis de alta resolución de la variación del cromosoma Y humano muestra una marcada discontinuidad y un flujo genético limitado entre el noroeste de África y la península Ibérica". American Journal of Human Genetics . 68 (4): 1019–1029. doi :10.1086/319521. PMC 1275654 . PMID 11254456.
- Brace CL, Seguchi N, Quintyn CB, Fox SC, Nelson AR, Manolis SK, Qifeng P (enero de 2006). "La cuestionable contribución del Neolítico y la Edad del Bronce a la forma craneofacial europea". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (1): 242–247. Bibcode :2006PNAS..103..242B. doi : 10.1073/pnas.0509801102 . PMC 1325007 . PMID 16371462.
- Capelli C, Onofri V, Brisighelli F, Boschi I, Scarnicci F, Masullo M, et al. (junio de 2009). "Moros y sarracenos en Europa: estimación del legado masculino medieval del norte de África en el sur de Europa". Revista europea de genética humana . 17 (6): 848–852. doi :10.1038/ejhg.2008.258. PMC 2947089 . PMID 19156170.
- Cavalli-Sforza LL (julio de 1997). "Genes, peoples, and language" (Genes, pueblos y lenguas). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 94 (15): 7719–7724. Bibcode :1997PNAS...94.7719C. doi : 10.1073/pnas.94.15.7719 . PMC 33682 . PMID 9223254.
- Cruciani F, Santolamazza P, Shen P, Macaulay V, Moral P, Olckers A, et al. (mayo de 2002). "Una migración de regreso desde Asia al África subsahariana está respaldada por un análisis de alta resolución de los haplotipos del cromosoma Y humano". American Journal of Human Genetics . 70 (5): 1197–1214. doi :10.1086/340257. PMC 447595 . PMID 11910562.
- Cruciani F, La Fratta R, Santolamazza P, Sellitto D, Pascone R, Moral P, et al. (mayo de 2004). "El análisis filogeográfico de los cromosomas y del haplogrupo E3b (E-M215) revela múltiples eventos migratorios dentro y fuera de África". American Journal of Human Genetics . 74 (5): 1014–1022. doi :10.1086/386294. PMC 1181964 . PMID 15042509.
- Cruciani F, La Fratta R, Torroni A, Underhill PA, Scozzari R (agosto de 2006). "Disección molecular del haplogrupo E-M78 (E3b1a) del cromosoma Y: evaluación a posteriori de un enfoque basado en redes de microsatélites a través de seis nuevos marcadores bialélicos". Human Mutation . 27 (8): 831–832. doi : 10.1002/humu.9445 . PMID 16835895. S2CID 26886757.
- Di Gaetano C, Cerutti N, Crobu F, Robino C, Inturri S, Gino S, et al. (enero de 2009). "Las migraciones diferenciales griegas y del norte de África a Sicilia están respaldadas por evidencia genética del cromosoma Y". Revista Europea de Genética Humana . 17 (1): 91–99. doi :10.1038/ejhg.2008.120. PMC 2985948 . PMID 18685561.
- Flores C, Maca-Meyer N, González AM, Oefner PJ, Shen P, Pérez JA, et al. (octubre de 2004). "Estructura genética reducida de la península ibérica revelada por análisis del cromosoma Y: implicaciones para la demografía poblacional". Revista Europea de Genética Humana . 12 (10): 855–863. doi : 10.1038/sj.ejhg.5201225 . PMID 15280900. S2CID 16765118.
- Francalacci P, Morelli L, Underhill PA, Lillie AS, Passarino G, Useli A, et al. (julio de 2003). "Población de tres islas mediterráneas (Córcega, Cerdeña y Sicilia) inferida por la variabilidad bialélica del cromosoma Y". American Journal of Physical Anthropology . 121 (3): 270–279. doi :10.1002/ajpa.10265. PMID 12772214.
- Gonçalves R, Freitas A, Branco M, Rosa A, Fernandes AT, Zhivotovsky LA, et al. (Julio de 2005). "Los linajes del cromosoma Y de Portugal, Madeira y Azores registran elementos de ascendencia sefardí y bereber". Anales de genética humana . 69 (parte 4): 443–454. doi :10.1111/j.1529-8817.2005.00161.x. hdl : 10400.13/3018 . PMID 15996172. S2CID 3229760.[ enlace muerto ]
- Halder I, Shriver M, Thomas M, Fernandez JR, Frudakis T (mayo de 2008). "Un panel de marcadores informativos de ascendencia para estimar la ascendencia y la mezcla biogeográficas individuales de cuatro continentes: utilidad y aplicaciones". Human Mutation . 29 (5): 648–658. doi : 10.1002/humu.20695 . PMID 18286470. S2CID 12489449.
- King R, Underhill PA (2002). "Distribución congruente de cerámica pintada y figurillas de cerámica neolíticas con linajes del cromosoma Y". Antiquity . 76 (293): 707–14. doi :10.1017/s0003598x00091158. S2CID 160359661.
- Lao O, Lu TT, Nothnagel M, Junge O, Freitag-Wolf S, Caliebe A, et al. (agosto de 2008). "Correlación entre la estructura genética y geográfica en Europa". Current Biology . 18 (16): 1241–1248. doi : 10.1016/j.cub.2008.07.049 . PMID 18691889. S2CID 16945780.
- Pericić M, Lauc LB, Klarić IM, Rootsi S, Janićijevic B, Rudan I, et al. (octubre de 2005). "El análisis filogenético de alta resolución del sudeste de Europa rastrea los principales episodios de flujo genético paterno entre las poblaciones eslavas". Biología molecular y evolución . 22 (10): 1964–1975. doi : 10.1093/molbev/msi185 . PMID 15944443.[ enlace muerto ]
- Ricaut FX, Waelkens M (octubre de 2008). "Rasgos craneales discretos en una población bizantina y movimientos poblacionales en el Mediterráneo oriental". Human Biology . 80 (5): 535–564. doi :10.3378/1534-6617-80.5.535. PMID 19341322. S2CID 25142338.
- Scozzari R, Cruciani F, Pangrazio A, Santolamazza P, Vona G, Moral P, et al. (Septiembre de 2001). "Variación del cromosoma Y humano en la zona del Mediterráneo occidental: implicaciones para el poblamiento de la región". Inmunología humana . 62 (9): 871–884. CiteSeerX 10.1.1.408.4857 . doi :10.1016/S0198-8859(01)00286-5. PMID 11543889.
- Semino O, Passarino G, Oefner PJ, Lin AA, Arbuzova S, Beckman LE, et al. (noviembre de 2000). "El legado genético del Homo sapiens sapiens paleolítico en los europeos actuales: una perspectiva del cromosoma Y" (PDF) . Science . 290 (5494): 1155–1159. Bibcode :2000Sci...290.1155S. doi :10.1126/science.290.5494.1155. PMID 11073453. Archivado desde el original (PDF) el 2008-03-07 . Consultado el 2009-07-22 ..
- Semino O, Santachiara-Benerecetti AS, Falaschi F, Cavalli-Sforza LL, Underhill PA (enero de 2002). "Los etíopes y los khoisan comparten los clados más profundos de la filogenia del cromosoma Y humano" (PDF) . Revista Estadounidense de Genética Humana . 70 (1): 265–268. doi :10.1086/338306. PMC 384897 . PMID 11719903. Archivado desde el original (PDF) el 15 de marzo de 2006 . Consultado el 22 de julio de 2009 .
- Sykes B (2006). Sangre de las islas: exploración de las raíces genéticas de nuestra historia tribal. Bantam. ISBN 978-0-593-05652-3. Consultado el 22 de julio de 2009 .
- Underhill PA, Shen P, Lin AA, Jin L, Passarino G, Yang WH, et al. (Noviembre de 2000). "Variación de la secuencia del cromosoma Y y la historia de las poblaciones humanas". Genética de la Naturaleza . 26 (3): 358–361. doi :10.1038/81685. PMID 11062480. S2CID 12893406.
- Underhill PA, Passarino G, Lin AA, Shen P, Mirazón Lahr M, Foley RA, et al. (enero de 2001). "La filogeografía de los haplotipos binarios del cromosoma Y y los orígenes de las poblaciones humanas modernas". Anales de genética humana . 65 (parte 1): 43–62. doi : 10.1046/j.1469-1809.2001.6510043.x . PMID 11415522. S2CID 9441236.
- Underhill PA (2002). "Inferencia de historias de población neolíticas utilizando haplotipos del cromosoma Y". En Bellwood y Renfrew (ed.). Examining the farming/language dispersion theory, Instituto McDonald de Investigación Arqueológica . Cambridge: Instituto McDonald de Investigación Arqueológica. ISBN 978-1-902937-20-5.
- Perlès C, Monthel G (2001) El Neolítico temprano en Grecia: las primeras comunidades agrícolas en Europa. Cambridge University Press , Cambridge.
- Runnels C (2003) Los orígenes del Neolítico griego: una visión personal, en Ammerman y Biagi (eds. 2003).
Lectura adicional
- Rodríguez-Varela, Ricardo et al. "La historia genética de Escandinavia desde la Edad del Hierro romana hasta la actualidad". En: Cell . Volumen 186, Número 1, 5 de enero de 2023, Páginas 32–46.e19. doi :10.1016/j.cell.2022.11.024
- Skourtanioti, E., Ringbauer, H., Gnecchi Ruscone, GA et al. "El ADN antiguo revela la historia de la mezcla y la endogamia en el Egeo prehistórico". En: Nature Ecology & Evolution (2023). https://doi.org/10.1038/s41559-022-01952-3
Enlaces externos
- "Un atlas genético de la historia de la mezcla humana". 2014.



