Biosíntesis
Este artículo tiene varios problemas. Ayúdenos a mejorarlo o a discutir estos problemas en la página de discusión . ( Aprenda cómo y cuándo eliminar estos mensajes )
|
La biosíntesis , es decir, la síntesis química que ocurre en contextos biológicos, es un término que se refiere con mayor frecuencia a procesos de múltiples pasos catalizados por enzimas donde las sustancias químicas absorbidas como nutrientes (o previamente convertidas a través de la biosíntesis) sirven como sustratos enzimáticos , y el organismo vivo las convierte en productos más simples o más complejos . Los ejemplos de vías biosintéticas incluyen aquellas para la producción de aminoácidos , componentes de la membrana lipídica y nucleótidos , pero también para la producción de todas las clases de macromoléculas biológicas y de acetil-coenzima A , trifosfato de adenosina , dinucleótido de nicotinamida y adenina y otras moléculas intermedias y transaccionales clave necesarias para el metabolismo . Por lo tanto, en la biosíntesis, cualquiera de una serie de compuestos , desde simples hasta complejos, se convierte en otros compuestos, por lo que incluye tanto el catabolismo como el anabolismo (formación y descomposición) de moléculas complejas (incluidas las macromoléculas ). Los procesos biosintéticos a menudo se representan mediante diagramas de vías metabólicas . Una vía biosintética particular puede estar ubicada dentro de un solo orgánulo celular (por ejemplo, las vías de síntesis de ácidos grasos mitocondriales), mientras que otras involucran enzimas que se encuentran en una variedad de orgánulos y estructuras celulares (por ejemplo, la biosíntesis de proteínas de superficie celular glicosiladas).
Elementos de la biosíntesis
Los elementos de la biosíntesis incluyen: compuestos precursores , energía química (p. ej. , ATP ) y enzimas catalíticas que pueden necesitar coenzimas (p. ej. , NADH , NADPH ). Estos elementos crean monómeros , los bloques de construcción de las macromoléculas. Algunas macromoléculas biológicas importantes incluyen: proteínas , que se componen de monómeros de aminoácidos unidos mediante enlaces peptídicos , y moléculas de ADN , que se componen de nucleótidos unidos mediante enlaces fosfodiéster .
Propiedades de las reacciones químicas
La biosíntesis se produce debido a una serie de reacciones químicas. Para que estas reacciones se lleven a cabo son necesarios los siguientes elementos: [1]
- Compuestos precursores : estos compuestos son las moléculas iniciales o sustratos de una reacción. También pueden considerarse reactivos en un proceso químico determinado.
- Energía química : la energía química se puede encontrar en forma de moléculas de alta energía. Estas moléculas son necesarias para reacciones energéticamente desfavorables. Además, la hidrólisis de estos compuestos impulsa una reacción. Las moléculas de alta energía, como el ATP , tienen tres fosfatos . A menudo, el fosfato terminal se separa durante la hidrólisis y se transfiere a otra molécula.
- Catalizadores : pueden ser por ejemplo iones metálicos o coenzimas y catalizan una reacción aumentando la velocidad de la reacción y disminuyendo la energía de activación .
En el sentido más simple, las reacciones que ocurren en la biosíntesis tienen el siguiente formato: [2]
Algunas variaciones de esta ecuación básica que se discutirán más detalladamente más adelante son: [3]
- Compuestos simples que se convierten en otros compuestos, generalmente como parte de una vía de reacción de varios pasos. Dos ejemplos de este tipo de reacción ocurren durante la formación de ácidos nucleicos y la carga del ARNt antes de la traducción . Para algunos de estos pasos se requiere energía química:
- Compuestos simples que se convierten en otros compuestos con la ayuda de cofactores. Por ejemplo, la síntesis de fosfolípidos requiere acetil CoA, mientras que la síntesis de otro componente de la membrana, los esfingolípidos , requiere NADH y FADH para la formación de la cadena principal de esfingosina . La ecuación general para estos ejemplos es:
- Compuestos simples que se unen para formar una macromolécula. Por ejemplo, los ácidos grasos se unen para formar fosfolípidos. A su vez, los fosfolípidos y el colesterol interactúan de forma no covalente para formar la bicapa lipídica . Esta reacción se puede representar de la siguiente manera:
Lípido

Muchas macromoléculas complejas se sintetizan en un patrón de estructuras simples y repetidas. [4] Por ejemplo, las estructuras más simples de los lípidos son los ácidos grasos . Los ácidos grasos son derivados de hidrocarburos ; contienen una "cabeza" de grupo carboxilo y una "cola" de cadena de hidrocarburos. [4] Estos ácidos grasos crean componentes más grandes, que a su vez incorporan interacciones no covalentes para formar la bicapa lipídica. [4] Las cadenas de ácidos grasos se encuentran en dos componentes principales de los lípidos de membrana: fosfolípidos y esfingolípidos . Un tercer componente principal de la membrana, el colesterol , no contiene estas unidades de ácidos grasos. [5]
Fosfolípidos eucariotas
La base de todas las biomembranas consiste en una estructura de bicapa de fosfolípidos. [6] La molécula de fosfolípido es anfipática ; contiene una cabeza polar hidrófila y una cola no polar hidrófoba . [4] Las cabezas de fosfolípidos interactúan entre sí y con el medio acuoso, mientras que las colas de hidrocarburos se orientan en el centro, lejos del agua. [7] Estas últimas interacciones impulsan la estructura de bicapa que actúa como barrera para iones y moléculas. [8]
Existen varios tipos de fosfolípidos, por lo que sus vías de síntesis difieren. Sin embargo, el primer paso en la síntesis de fosfolípidos implica la formación de fosfatidato o diacilglicerol 3-fosfato en el retículo endoplasmático y la membrana mitocondrial externa . [7] La vía de síntesis se encuentra a continuación:

La vía comienza con el glicerol 3-fosfato, que se convierte en lisofosfatidato mediante la adición de una cadena de ácido graso proporcionada por la acil coenzima A. [ 9] Luego, el lisofosfatidato se convierte en fosfatidato mediante la adición de otra cadena de ácido graso aportada por una segunda acil CoA; todos estos pasos son catalizados por la enzima glicerol fosfato aciltransferasa . [9] La síntesis de fosfolípidos continúa en el retículo endoplásmico y la vía de biosíntesis diverge dependiendo de los componentes del fosfolípido en particular. [9]
Esfingolípidos
Al igual que los fosfolípidos, estos derivados de ácidos grasos tienen una cabeza polar y colas no polares. [5] A diferencia de los fosfolípidos, los esfingolípidos tienen una estructura principal de esfingosina . [10] Los esfingolípidos existen en las células eucariotas y son particularmente abundantes en el sistema nervioso central . [7] Por ejemplo, la esfingomielina es parte de la vaina de mielina de las fibras nerviosas. [11]
Los esfingolípidos se forman a partir de ceramidas que consisten en una cadena de ácidos grasos unida al grupo amino de una cadena principal de esfingosina. Estas ceramidas se sintetizan a partir de la acilación de la esfingosina. [11] La vía biosintética de la esfingosina se encuentra a continuación:

Como lo indica la imagen, durante la síntesis de esfingosina, el palmitoil CoA y la serina experimentan una reacción de condensación que da como resultado la formación de 3-deshidroesfinganina. [7] Este producto luego se reduce para formar dihidroesfingosina, que se convierte en esfingosina a través de la reacción de oxidación por FAD . [7]
Colesterol
Este lípido pertenece a una clase de moléculas llamadas esteroles . [5] Los esteroles tienen cuatro anillos fusionados y un grupo hidroxilo . [5] El colesterol es una molécula particularmente importante. No solo sirve como componente de las membranas lipídicas, sino que también es un precursor de varias hormonas esteroides , entre ellas el cortisol , la testosterona y el estrógeno . [12]
El colesterol se sintetiza a partir de acetil CoA . [12] La vía se muestra a continuación:

De manera más general, esta síntesis ocurre en tres etapas, la primera en el citoplasma y la segunda y tercera en el retículo endoplasmático. [9] Las etapas son las siguientes: [12]
- 1. La síntesis del pirofosfato de isopentenilo , el «bloque constructor» del colesterol
- 2. La formación de escualeno mediante la condensación de seis moléculas de fosfato de isopentenilo.
- 3. La conversión de escualeno en colesterol a través de varias reacciones enzimáticas.
Nucleótidos
La biosíntesis de nucleótidos implica reacciones catalizadas por enzimas que convierten los sustratos en productos más complejos. [1] Los nucleótidos son los componentes básicos del ADN y el ARN . Los nucleótidos están compuestos por un anillo de cinco miembros formado por el azúcar ribosa en el ARN y el azúcar desoxirribosa en el ADN; estos azúcares están unidos a una base de purina o pirimidina con un enlace glucosídico y un grupo fosfato en la posición 5' del azúcar. [13]
Nucleótidos de purina

Los nucleótidos de ADN adenosina y guanosina consisten en una base de purina unida a un azúcar ribosa con un enlace glucosídico. En el caso de los nucleótidos de ARN desoxiadenosina y desoxiguanosina , las bases de purina están unidas a un azúcar desoxirribosa con un enlace glucosídico. Las bases de purina en los nucleótidos de ADN y ARN se sintetizan en un mecanismo de reacción de doce pasos presente en la mayoría de los organismos unicelulares. Los eucariotas superiores emplean un mecanismo de reacción similar en diez pasos de reacción. Las bases de purina se sintetizan convirtiendo el pirofosfato de fosforribosil (PRPP) en monofosfato de inosina (IMP), que es el primer intermediario clave en la biosíntesis de bases de purina. [14] Una modificación enzimática adicional del IMP produce las bases de adenosina y guanosina de los nucleótidos.
- El primer paso en la biosíntesis de purinas es una reacción de condensación , realizada por la glutamina-PRPP amidotransferasa . Esta enzima transfiere el grupo amino de la glutamina a la PRPP, formando 5-fosforribosilamina . El siguiente paso requiere la activación de la glicina mediante la adición de un grupo fosfato del ATP .
- La sintetasa GAR [15] realiza la condensación de la glicina activada sobre PRPP, formando ribonucleótido de glicinamida (GAR).
- La transformilasa GAR agrega un grupo formilo al grupo amino de GAR, formando ribonucleótido de formilglicinamida (FGAR).
- La amidotransferasa FGAR [16] cataliza la adición de un grupo nitrógeno a FGAR, formando ribonucleótido de formilglicinamidina (FGAM).
- La ciclasa FGAM cataliza el cierre del anillo, que implica la eliminación de una molécula de agua, formando el anillo de imidazol de 5 miembros , el ribonucleótido de 5-aminoimidazol (AIR).
- La sintetasa N5-CAIR transfiere un grupo carboxilo , formando el ribonucleótido intermedio N5-carboxiaminoimidazol (N5-CAIR). [17]
- La mutasa N5-CAIR reorganiza el grupo funcional carboxilo y lo transfiere al anillo de imidazol, formando el ribonucleótido carboxiamino-imidazol (CAIR). El mecanismo de dos pasos de la formación de CAIR a partir de AIR se encuentra principalmente en organismos unicelulares. Los eucariotas superiores contienen la enzima AIR carboxilasa, [18] que transfiere un grupo carboxilo directamente al anillo de imidazol de AIR, formando CAIR.
- La sintetasa SAICAR forma un enlace peptídico entre el aspartato y el grupo carboxilo añadido del anillo de imidazol, formando el ribonucleótido N-succinil-5-aminoimidazol-4-carboxamida (SAICAR).
- La liasa SAICAR elimina el esqueleto de carbono del aspartato añadido, dejando el grupo amino y formando ribonucleótido 5-aminoimidazol-4-carboxamida (AICAR).
- La transformilasa AICAR transfiere un grupo carbonilo a AICAR, formando ribonucleótido N-formilaminoimidazol-4-carboxamida (FAICAR).
- El paso final involucra a la enzima IMP sintasa , que realiza el cierre del anillo de purina y forma el intermediario monofosfato de inosina. [5]
Nucleótidos de pirimidina

Otras bases de nucleótidos del ADN y ARN que están unidas al azúcar ribosa a través de un enlace glucosídico son la timina , la citosina y el uracilo (que solo se encuentra en el ARN). La biosíntesis de monofosfato de uridina involucra una enzima que se encuentra en la membrana interna mitocondrial y enzimas multifuncionales que se encuentran en el citosol . [19]
- El primer paso implica que la enzima carbamoil fosfato sintasa combine glutamina con CO2 en una reacción dependiente de ATP para formar carbamoil fosfato .
- La aspartato carbamoiltransferasa condensa el fosfato de carbamoilo con aspartato para formar uridosuccinato.
- La dihidroorotasa realiza el cierre del anillo , una reacción que pierde agua, para formar dihidroorotato .
- La dihidroorotato deshidrogenasa , ubicada dentro de la membrana interna mitocondrial, [19] oxida el dihidroorotato a orotato .
- La orotato fosforribosil hidrolasa (OMP pirofosforilasa) condensa el orotato con PRPP para formar orotidina-5'-fosfato .
- La descarboxilasa OMP cataliza la conversión de orotidina-5'-fosfato a UMP . [20]
Después de sintetizar la base nucleotídica uridina, se sintetizan las otras bases, citosina y timina. La biosíntesis de citosina es una reacción de dos pasos que implica la conversión de UMP a UTP . La adición de fosfato a UMP es catalizada por una enzima quinasa . La enzima CTP sintasa cataliza el siguiente paso de la reacción: la conversión de UTP a CTP mediante la transferencia de un grupo amino de la glutamina a la uridina; esto forma la base citosina de CTP. [21] El mecanismo, que representa la reacción UTP + ATP + glutamina ⇔ CTP + ADP + glutamato, se muestra a continuación:

La citosina es un nucleótido que está presente tanto en el ADN como en el ARN. Sin embargo, el uracilo solo se encuentra en el ARN. Por lo tanto, después de sintetizar el UTP, debe convertirse en una forma desoxi para incorporarse al ADN. Esta conversión involucra a la enzima ribonucleósido trifosfato reductasa . Esta reacción que elimina el 2'-OH del azúcar ribosa para generar desoxirribosa no se ve afectada por las bases unidas al azúcar. Esta falta de especificidad permite que la ribonucleósido trifosfato reductasa convierta todos los nucleótidos trifosfato en desoxirribonucleótidos mediante un mecanismo similar. [21]
A diferencia del uracilo, las bases de timina se encuentran principalmente en el ADN, no en el ARN. Las células normalmente no contienen bases de timina que estén unidas a los azúcares ribosa en el ARN, lo que indica que las células solo sintetizan timina unida a la desoxirribosa. La enzima timidilato sintetasa es responsable de sintetizar residuos de timina de dUMP a dTMP . Esta reacción transfiere un grupo metilo a la base de uracilo de dUMP para generar dTMP. [21] La reacción de la timidilato sintetasa, dUMP + 5,10-metilentetrahidrofolato ⇔ dTMP + dihidrofolato, se muestra a la derecha.
ADN
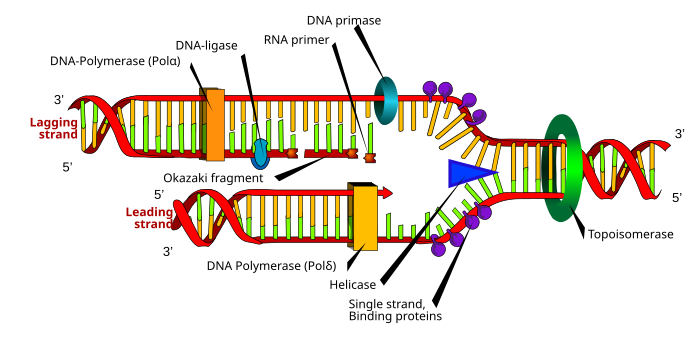
Aunque existen diferencias entre la síntesis de ADN eucariota y procariota , la siguiente sección denota características clave de la replicación del ADN compartidas por ambos organismos.
El ADN está compuesto de nucleótidos que están unidos por enlaces fosfodiéster . [4] La síntesis de ADN , que tiene lugar en el núcleo , es un proceso semiconservativo , lo que significa que la molécula de ADN resultante contiene una cadena original de la estructura parental y una nueva cadena. [22] La síntesis de ADN es catalizada por una familia de ADN polimerasas que requieren cuatro desoxinucleósidos trifosfatos, una cadena molde y un cebador con un 3'OH libre en el que incorporar nucleótidos. [23]
Para que se produzca la replicación del ADN, se crea una horquilla de replicación mediante enzimas llamadas helicasas que desenrollan la hélice del ADN. [23] Las topoisomerasas en la horquilla de replicación eliminan las superenrollaciones causadas por el desenrollado del ADN, y las proteínas de unión al ADN monocatenario mantienen las dos plantillas de ADN monocatenario estabilizadas antes de la replicación. [13]
La síntesis de ADN es iniciada por la ARN polimerasa primasa , que produce un cebador de ARN con un 3'OH libre. [23] Este cebador se une a la plantilla de ADN monocatenario, y la ADN polimerasa alarga la cadena incorporando nucleótidos; la ADN polimerasa también corrige la cadena de ADN recién sintetizada. [23]
Durante la reacción de polimerización catalizada por la ADN polimerasa, se produce un ataque nucleofílico por parte del 3'OH de la cadena en crecimiento sobre el átomo de fósforo más interno de un desoxinucleósido trifosfato; esto produce la formación de un puente fosfodiéster que une un nuevo nucleótido y libera pirofosfato . [9]
Durante la replicación se crean simultáneamente dos tipos de hebras: la hebra líder , que se sintetiza de forma continua y crece hacia la horquilla de replicación, y la hebra rezagada , que se produce de forma discontinua en fragmentos de Okazaki y crece alejándose de la horquilla de replicación. [22] Los fragmentos de Okazaki se unen covalentemente mediante la ADN ligasa para formar una hebra continua. [22] Luego, para completar la replicación del ADN, se eliminan los cebadores de ARN y los huecos resultantes se reemplazan con ADN y se unen mediante la ADN ligasa. [22]
Aminoácidos
Una proteína es un polímero que se compone de aminoácidos que están unidos por enlaces peptídicos . Hay más de 300 aminoácidos que se encuentran en la naturaleza, de los cuales solo veintidós, conocidos como aminoácidos proteinogénicos , son los bloques de construcción de las proteínas. [24] Solo las plantas verdes y la mayoría de los microbios pueden sintetizar los 20 aminoácidos estándar que necesitan todas las especies vivas. Los mamíferos solo pueden sintetizar diez de los veinte aminoácidos estándar. Los otros aminoácidos, valina , metionina , leucina , isoleucina , fenilalanina , lisina , treonina y triptófano para los adultos y la histidina y arginina para los bebés se obtienen a través de la dieta. [25]
Estructura básica de los aminoácidos
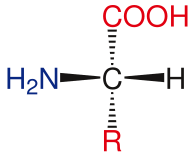
La estructura general de los aminoácidos estándar incluye un grupo amino primario , un grupo carboxilo y el grupo funcional unido al carbono α . Los diferentes aminoácidos se identifican por el grupo funcional. Como resultado de los tres grupos diferentes unidos al carbono α, los aminoácidos son moléculas asimétricas . Para todos los aminoácidos estándar, excepto la glicina , el carbono α es un centro quiral . En el caso de la glicina, el carbono α tiene dos átomos de hidrógeno, lo que añade simetría a esta molécula. Con la excepción de la prolina , todos los aminoácidos que se encuentran en la vida tienen la conformación de isoforma L. La prolina tiene un grupo funcional en el carbono α que forma un anillo con el grupo amino. [24]

Fuente de nitrógeno
Un paso importante en la biosíntesis de aminoácidos implica la incorporación de un grupo de nitrógeno al carbono α. En las células, existen dos vías principales de incorporación de grupos de nitrógeno. Una vía implica la enzima glutamina oxoglutarato aminotransferasa (GOGAT), que elimina el grupo amino amida de la glutamina y lo transfiere al 2-oxoglutarato , produciendo dos moléculas de glutamato . En esta reacción de catálisis, la glutamina sirve como fuente de nitrógeno. A la derecha se encuentra una imagen que ilustra esta reacción.
La otra vía para incorporar nitrógeno al carbono α de los aminoácidos implica la enzima glutamato deshidrogenasa (GDH). La GDH es capaz de transferir amoníaco al 2-oxoglutarato y formar glutamato. Además, la enzima glutamina sintetasa (GS) es capaz de transferir amoníaco al glutamato y sintetizar glutamina, reponiendo la glutamina. [26]
La familia de aminoácidos del glutamato
La familia de aminoácidos glutamato incluye los aminoácidos que se derivan del aminoácido glutamato. Esta familia incluye: glutamato, glutamina , prolina y arginina . Esta familia también incluye el aminoácido lisina , que se deriva del α-cetoglutarato . [27]
La biosíntesis de glutamato y glutamina es un paso clave en la asimilación de nitrógeno que se ha comentado anteriormente. Las enzimas GOGAT y GDH catalizan las reacciones de asimilación de nitrógeno .
En las bacterias, la enzima glutamato 5-quinasa inicia la biosíntesis de prolina transfiriendo un grupo fosfato del ATP al glutamato. La siguiente reacción es catalizada por la enzima pirrolina-5-carboxilato sintasa (P5CS), que cataliza la reducción del grupo ϒ-carboxilo del L-glutamato 5-fosfato. Esto da como resultado la formación de glutamato semialdehído, que se cicla espontáneamente a pirrolina-5-carboxilato. La pirrolina-5-carboxilato se reduce aún más por la enzima pirrolina-5-carboxilato reductasa (P5CR) para producir un aminoácido prolina. [28]
En el primer paso de la biosíntesis de arginina en bacterias, el glutamato se acetila transfiriendo el grupo acetilo del acetil-CoA en la posición N-α; esto evita la ciclización espontánea. La enzima N-acetilglutamato sintasa (glutamato N-acetiltransferasa) es responsable de catalizar el paso de acetilación. Los pasos posteriores son catalizados por las enzimas N-acetilglutamato quinasa , N-acetil-gamma-glutamil-fosfato reductasa y acetilornitina/succinildiamino pimelato aminotransferasa y producen la N-acetil-L-ornitina. El grupo acetilo de la acetilornitina es eliminado por la enzima acetilornitinasa (AO) u ornitina acetiltransferasa (OAT), y esto produce ornitina . Luego, las enzimas citrulina y argininosuccinato convierten la ornitina en arginina. [29]
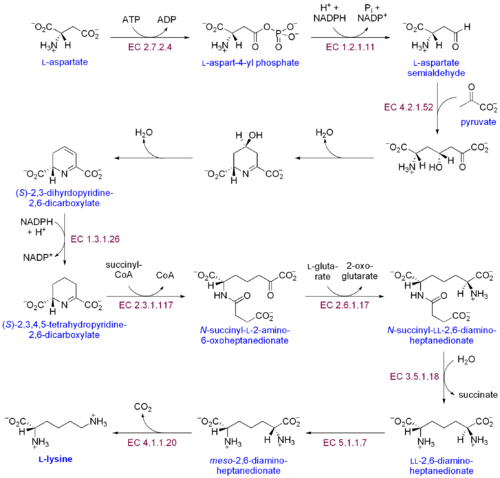
Existen dos vías biosintéticas distintas para la biosíntesis de la lisina: la vía del ácido diaminopimélico y la vía del α-aminoadipato . La más común de las dos vías sintéticas es la vía del ácido diaminopimélico, que consiste en varias reacciones enzimáticas que añaden grupos de carbono al aspartato para producir lisina: [30]
- La aspartato quinasa inicia la vía del ácido diaminopimélico fosforilando aspartato y produciendo fosfato de aspartilo.
- La aspartato semialdehído deshidrogenasa cataliza la reducción dependiente de NADPH del fosfato de aspartilo para producir aspartato semialdehído.
- La 4-hidroxi-tetrahidrodipicolinato sintasa añade un grupo piruvato al β-aspartil-4-semialdehído y elimina una molécula de agua. Esto provoca la ciclización y da lugar al (2S,4S)-4-hidroxi-2,3,4,5-tetrahidrodipicolinato.
- La 4-hidroxi-tetrahidrodipicolinato reductasa cataliza la reducción de (2S,4S)-4-hidroxi-2,3,4,5-tetrahidrodipicolinato por NADPH para producir Δ'-piperideína-2,6-dicarboxilato (2,3,4,5-tetrahidrodipicolinato) y H 2 O.
- La tetrahidrodipicolinato aciltransferasa cataliza la reacción de acetilación que da como resultado la apertura del anillo y produce N-acetil α-amino-ε-cetopimelato.
- La aminotransaminasa N-succinil-α-amino-ε-cetopimelato-glutamato cataliza la reacción de transaminación que elimina el grupo ceto del N-acetil α-amino-ε-cetopimelato y lo reemplaza con un grupo amino para producir N-succinil-L-diaminopimelato. [31]
- La N-acildiaminopimelato desacilasa cataliza la desacilación de N-succinil-L-diaminopimelato para producir L,L-diaminopimelato. [32]
- La epimerasa DAP cataliza la conversión de L,L-diaminopimelato a la forma meso de L,L-diaminopimelato. [33]
- La DAP descarboxilasa cataliza la eliminación del grupo carboxilo, produciendo L-lisina.
La familia de aminoácidos serina
La familia de aminoácidos de la serina incluye: serina, cisteína y glicina . La mayoría de los microorganismos y plantas obtienen el azufre para sintetizar metionina a partir del aminoácido cisteína. Además, la conversión de serina en glicina proporciona los carbonos necesarios para la biosíntesis de la metionina y la histidina . [27]
Durante la biosíntesis de serina, [34] la enzima fosfoglicerato deshidrogenasa cataliza la reacción inicial que oxida el 3-fosfo-D-glicerato para producir 3-fosfonooxipiruvato . [35] La siguiente reacción es catalizada por la enzima fosfoserina aminotransferasa , que transfiere un grupo amino del glutamato al 3-fosfonooxipiruvato para producir L-fosfoserina . [36] El paso final es catalizado por la enzima fosfoserina fosfatasa , que desfosforila la L-fosfoserina para producir L-serina . [37]
Existen dos vías conocidas para la biosíntesis de la glicina. Los organismos que utilizan etanol y acetato como fuente principal de carbono utilizan la vía gluconeogénica para sintetizar glicina . La otra vía de biosíntesis de glicina se conoce como vía glucolítica . Esta vía convierte la serina sintetizada a partir de los intermediarios de la glucólisis en glicina. En la vía glucolítica, la enzima serina hidroximetiltransferasa cataliza la escisión de la serina para producir glicina y transfiere el grupo de carbono escindido de la serina al tetrahidrofolato , formando 5,10-metilen-tetrahidrofolato . [38]
La biosíntesis de cisteína es una reacción de dos pasos que implica la incorporación de azufre inorgánico . En microorganismos y plantas, la enzima serina acetiltransferasa cataliza la transferencia del grupo acetilo de acetil-CoA a L-serina para producir O-acetil-L-serina . [39] El siguiente paso de reacción, catalizado por la enzima O-acetilserina (tiol) liasa , reemplaza el grupo acetilo de O-acetil-L-serina con sulfuro para producir cisteína. [40]
La familia de aminoácidos aspartato
La familia de aminoácidos aspartato incluye: treonina , lisina , metionina , isoleucina y aspartato. La lisina y la isoleucina se consideran parte de la familia aspartato a pesar de que parte de su esqueleto carbonado se deriva del piruvato . En el caso de la metionina, el carbono metilo se deriva de la serina y el grupo azufre, pero en la mayoría de los organismos, se deriva de la cisteína. [27]
La biosíntesis del aspartato es una reacción de un solo paso catalizada por una sola enzima. La enzima aspartato aminotransferasa cataliza la transferencia de un grupo amino del aspartato al α-cetoglutarato para producir glutamato y oxaloacetato . [41] La asparagina se sintetiza mediante la adición dependiente de ATP de un grupo amino al aspartato; la asparagina sintetasa cataliza la adición de nitrógeno de glutamina o amoníaco soluble al aspartato para producir asparagina. [42]

La vía biosintética del ácido diaminopimélico de la lisina pertenece a la familia de aminoácidos del aspartato. Esta vía implica nueve reacciones catalizadas por enzimas que convierten el aspartato en lisina. [43]
- La aspartato quinasa cataliza el paso inicial en la vía del ácido diaminopimélico al transferir un fosforilo del ATP al grupo carboxilato del aspartato, lo que produce aspartil-β-fosfato. [44]
- La aspartato-semialdehído deshidrogenasa cataliza la reacción de reducción por desfosforilación del aspartil-β-fosfato para producir aspartato-β-semialdehído. [45]
- La dihidrodipicolinato sintasa cataliza la reacción de condensación del aspartato-β-semialdehído con piruvato para producir ácido dihidrodipicolínico. [46]
- La 4-hidroxi-tetrahidrodipicolinato reductasa cataliza la reducción del ácido dihidrodipicolínico para producir ácido tetrahidrodipicolínico. [47]
- La tetrahidrodipicolinato N-succiniltransferasa cataliza la transferencia de un grupo succinilo desde succinil-CoA al ácido tetrahidrodipicolínico para producir N-succinil-L-2,6-diaminoheptanodioato. [48]
- La aminotransferasa N-succinildiaminopimelato cataliza la transferencia de un grupo amino del glutamato al N-succinil-L-2,6-diaminoheptanodioato para producir ácido N-succinil-L,L-diaminopimélico. [49]
- La succinil-diaminopimelato desuccinilasa cataliza la eliminación del grupo acilo del ácido N-succinil-L,L-diaminopimélico para producir ácido L,L-diaminopimélico. [50]
- La epimerasa diaminopimelato cataliza la inversión del carbono α del ácido L,L-diaminopimélico para producir ácido meso-diaminopimélico . [51]
- La siaminopimelato descarboxilasa cataliza el paso final en la biosíntesis de lisina que elimina el grupo de dióxido de carbono del ácido meso-diaminopimélico para producir L-lisina. [52]
Proteínas
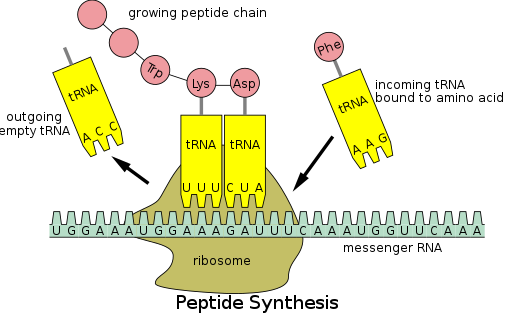
La síntesis de proteínas ocurre a través de un proceso llamado traducción . [53] Durante la traducción, el material genético llamado ARNm es leído por los ribosomas para generar una cadena polipeptídica de proteína. [53] Este proceso requiere ARN de transferencia (ARNt) que sirve como adaptador al unir aminoácidos en un extremo e interactuar con el ARNm en el otro extremo; el último emparejamiento entre el ARNt y el ARNm asegura que se agregue el aminoácido correcto a la cadena. [53] La síntesis de proteínas ocurre en tres fases: iniciación, elongación y terminación. [13] La traducción procariota ( arqueal y bacteriana ) difiere de la traducción eucariota ; sin embargo, esta sección se centrará principalmente en los puntos en común entre los dos organismos.
Antecedentes adicionales
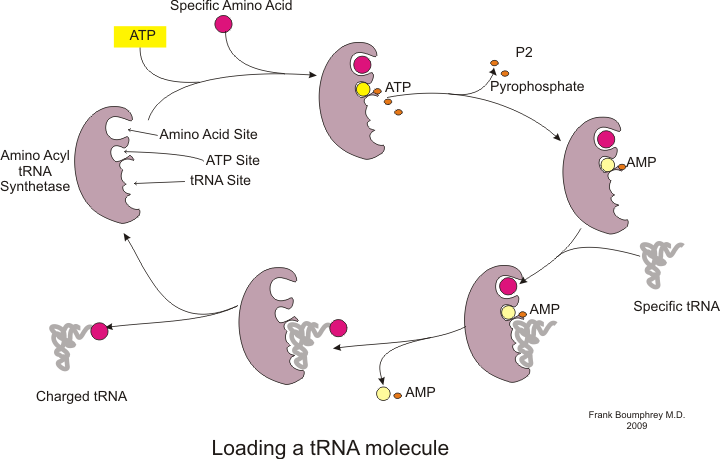
Antes de que pueda comenzar la traducción, debe producirse el proceso de unión de un aminoácido específico a su ARNt correspondiente. Esta reacción, llamada carga del ARNt, es catalizada por la aminoacil-ARNt sintetasa . [54] Una ARNt sintetasa específica es responsable de reconocer y cargar un aminoácido en particular. [54] Además, esta enzima tiene regiones discriminadoras especiales para asegurar la unión correcta entre el ARNt y su aminoácido cognado. [54] El primer paso para unir un aminoácido a su ARNt correspondiente es la formación de aminoacil-AMP: [54]
A continuación se produce la transferencia del grupo aminoacilo del aminoacil-AMP a una molécula de ARNt. La molécula resultante es el aminoacil-ARNt : [54]
La combinación de estos dos pasos, ambos catalizados por la aminoacil ARNt sintetasa, produce un ARNt cargado que está listo para agregar aminoácidos a la cadena polipeptídica en crecimiento.
Además de unirse a un aminoácido, el ARNt tiene una unidad de tres nucleótidos llamada anticodón que se aparea con tripletes de nucleótidos específicos en el ARNm llamados codones ; los codones codifican un aminoácido específico. [55] Esta interacción es posible gracias al ribosoma, que sirve como sitio para la síntesis de proteínas. El ribosoma posee tres sitios de unión del ARNt: el sitio aminoacilo (sitio A), el sitio peptidilo (sitio P) y el sitio de salida (sitio E). [56]
Hay numerosos codones dentro de una transcripción de ARNm, y es muy común que un aminoácido sea especificado por más de un codón; este fenómeno se llama degeneración . [57] En total, hay 64 codones, 61 de cada uno codifican para uno de los 20 aminoácidos, mientras que los codones restantes especifican la terminación de la cadena. [57]
Traducción en pasos
Como se mencionó anteriormente, la traducción ocurre en tres fases: iniciación, elongación y terminación.

Paso 1: Iniciación
La finalización de la fase de iniciación depende de los tres eventos siguientes: [13]
1. El reclutamiento del ribosoma al ARNm
2. La unión de un ARNt iniciador cargado en el sitio P del ribosoma.
3. La alineación adecuada del ribosoma con el codón de inicio del ARNm.
Paso 2: Alargamiento
Tras la iniciación, la cadena polipeptídica se extiende mediante interacciones anticodón:codón, y el ribosoma añade aminoácidos a la cadena polipeptídica de uno en uno. Para garantizar la correcta adición de aminoácidos, deben realizarse los siguientes pasos: [58]
1. La unión del ARNt correcto en el sitio A del ribosoma.
2. La formación de un enlace peptídico entre el ARNt en el sitio A y la cadena polipeptídica unida al ARNt en el sitio P
3. Translocación o avance del complejo ARNt-ARNm por tres nucleótidos
La translocación "activa" el ARNt en el sitio E y lo desplaza del sitio A al sitio P, dejando el sitio A libre para que un ARNt entrante agregue otro aminoácido.
Paso 3: Terminación
La última etapa de la traducción ocurre cuando un codón de parada ingresa al sitio A. [1] Luego, ocurren los siguientes pasos:
1. El reconocimiento de codones por factores de liberación , lo que provoca la hidrólisis de la cadena polipeptídica a partir del ARNt ubicado en el sitio P [1]
2. La liberación de la cadena polipeptídica [57]
3. La disociación y el “reciclaje” del ribosoma para futuros procesos de traducción [57]
A continuación se presenta un cuadro resumen de los actores clave en el ámbito de la traducción:
| Actores clave en la traducción | Etapa de traducción | Objetivo |
|---|---|---|
| ARNt sintetasa | antes de la iniciación | Responsable de la carga del ARNt |
| ARNm | iniciación, elongación, terminación | Plantilla para la síntesis de proteínas; contiene regiones llamadas codones que codifican aminoácidos. |
| ARNt | iniciación, elongación, terminación | Se une a los sitios A, P, E de los ribosomas; los pares de bases del anticodón se combinan con el codón del ARNm para garantizar que se incorpore el aminoácido correcto a la cadena polipeptídica en crecimiento. |
| ribosoma | iniciación, elongación, terminación | Dirige la síntesis de proteínas y cataliza la formación del enlace peptídico. |
Enfermedades asociadas a deficiencia de macromoléculas

Los errores en las vías biosintéticas pueden tener consecuencias perjudiciales, como la malformación de macromoléculas o la producción insuficiente de moléculas funcionales. A continuación se presentan ejemplos que ilustran las alteraciones que se producen debido a estas ineficiencias.
- Hipercolesterolemia familiar : este trastorno se caracteriza por la ausencia de receptores funcionales para LDL . [59] Las deficiencias en la formación de receptores de LDL pueden causar receptores defectuosos que interrumpen la vía endocítica , inhibiendo la entrada de LDL al hígado y otras células. [59] Esto causa una acumulación de LDL en el plasma sanguíneo, lo que resulta en placas ateroscleróticas que estrechan las arterias y aumentan el riesgo de ataques cardíacos. [59]
- Síndrome de Lesch-Nyhan : esta enfermedad genética se caracteriza por automutilación , deficiencia mental y gota . [60] Es causada por la ausencia de hipoxantina-guanina fosforribosiltransferasa , que es una enzima necesaria para la formación de nucleótidos de purina. [60] La falta de enzima reduce el nivel de nucleótidos necesarios y provoca la acumulación de intermediarios de biosíntesis , lo que resulta en el comportamiento inusual antes mencionado. [60]
- Inmunodeficiencia combinada grave (IDCG) : la IDCG se caracteriza por una pérdida de células T. [61] La falta de estos componentes del sistema inmunológico aumenta la susceptibilidad a los agentes infecciosos porque los individuos afectados no pueden desarrollar memoria inmunológica . [61] Este trastorno inmunológico resulta de una deficiencia en la actividad de la adenosina deaminasa , que provoca una acumulación de dATP . Estas moléculas de dATP luego inhiben la ribonucleótido reductasa, que impide la síntesis de ADN. [61]
- Enfermedad de Huntington : esta enfermedad neurológica es causada por errores que ocurren durante la síntesis de ADN. [62] Estos errores o mutaciones conducen a la expresión de una proteína huntingtina mutante, que contiene residuos repetitivos de glutamina que están codificados por repeticiones de trinucleótidos CAG en expansión en el gen. [62] La enfermedad de Huntington se caracteriza por pérdida neuronal y gliosis . Los síntomas de la enfermedad incluyen: trastorno del movimiento, deterioro cognitivo y trastorno del comportamiento. [63]
Véase también
- Lípidos
- Bicapa de fosfolípidos
- Nucleótidos
- ADN
- Replicación del ADN
- Aminoácido proteinogénico
- Tabla de codones
- Prostaglandina
- Porfirinas
- Clorofilas y bacterioclorofilas
- Vitamina B12
Referencias
- ^ abcd Alberts, Bruce (2008). Biología molecular de la célula (5.ª ed.). Nueva York: Garland Science. ISBN 978-0815341055.
- ^ Zumdahl, Steven S. Zumdahl, Susan A. (2008). Química (8.ª ed.). CA: Cengage Learning. ISBN 978-0547125329.
{{cite book}}: CS1 maint: multiple names: authors list (link) - ^ Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2013). Fundamentos de bioquímica: la vida a nivel molecular (4ª ed.). Hoboken, Nueva Jersey: Wiley. ISBN 978-0470547847.
- ^ abcde Lodish, Harvey; et al. (2007). Biología celular molecular (6.ª ed.). Nueva York: WH Freeman. ISBN 978-0716743668.
- ^ abcde Cox, David L. Nelson, Michael M. (2008). Principios de bioquímica de Lehninger (5.ª ed.). Nueva York: WH Freeman. ISBN 9780716771081.
{{cite book}}: CS1 maint: multiple names: authors list (link) - ^ Hanin, Israel (2013). Fosfolípidos: consideraciones bioquímicas, farmacéuticas y analíticas . Springer. ISBN 978-1475713664.
- ^ abcde Vance, Dennis E.; Vance, Jean E. (2008). Bioquímica de lípidos, lipoproteínas y membranas (5.ª ed.). Ámsterdam: Elsevier. ISBN 978-0444532190.
- ^ Katsaras, J.; et al. (2001). Bicapas lipídicas: estructura e interacciones; con 6 tablas . Berlín [ua]: Springer. ISBN 978-3540675556.
- ^ abcde Stryer, Jeremy M. Berg; John L. Tymoczko; Lubert (2007). Bioquímica (6.ª ed., 3.ª ed. impresa). Nueva York: Freeman. ISBN 978-0716787242.
{{cite book}}: CS1 maint: multiple names: authors list (link) - ^ Gault, CR; LM Obeid; YA Hannun (2010). "Una descripción general del metabolismo de los esfingolípidos: desde la síntesis hasta la descomposición". Los esfingolípidos como moléculas señalizadoras y reguladoras . Avances en medicina y biología experimental. Vol. 688. págs. 1–23. doi :10.1007/978-1-4419-6741-1_1. ISBN . 978-1-4419-6740-4. PMC 3069696 . PMID 20919643.
- ^ ab Siegel, George J. (1999). Neuroquímica básica: aspectos moleculares, celulares y médicos (6.ª ed.). Filadelfia, Pensilvania: Lippincott Williams & Wilkins. ISBN 978-0397518203.
- ^ abc Harris, J. Robin (2010). Proteínas transportadoras y de unión al colesterol: estructura y función en la salud y la enfermedad . Dordrecht: Springer. ISBN 978-9048186211.
- ^ abcd Watson, James D.; et al. (2007). Biología molecular del gen (6ª ed.). San Francisco, California: Benjamin Cummings. ISBN 978-0805395921.
- ^ Kappock, TJ; Ealick, SE; Stubbe, J (octubre de 2000). "Evolución modular de la vía biosintética de las purinas". Current Opinion in Chemical Biology . 4 (5): 567–72. doi :10.1016/s1367-5931(00)00133-2. PMID 11006546.
- ^ Sampei, G; Baba, S; Kanagawa, M; Yanai, H; Ishii, T; Kawai, H; Fukai, Y; Ebihara, A; Nakagawa, N; Kawai, G (octubre de 2010). "Estructuras cristalinas de la glicinamida ribonucleótido sintetasa, PurD, de eubacterias termófilas". Journal of Biochemistry . 148 (4): 429–38. doi :10.1093/jb/mvq088. PMID 20716513.
- ^ Hoskins, AA; Anand, R; Ealick, SE; Stubbe, J (17 de agosto de 2004). "El complejo amidotransferasa de ribonucleótido de formilglicinamida de Bacillus subtilis: formación de complejos mediada por metabolitos". Bioquímica . 43 (32): 10314–27. doi :10.1021/bi049127h. PMID 15301530.
- ^ Mueller, EJ; Meyer, E; Rudolph, J; Davisson, VJ; Stubbe, J (1 de marzo de 1994). "N5-carboxiaminoimidazol ribonucleótido: evidencia de un nuevo intermediario y dos nuevas actividades enzimáticas en la vía biosintética de novo de purina de Escherichia coli". Bioquímica . 33 (8): 2269–78. doi :10.1021/bi00174a038. PMID 8117684.
- ^ Firestine, SM; Poon, SW; Mueller, EJ; Stubbe, J; Davisson, VJ (4 de octubre de 1994). "Reacciones catalizadas por carboxilasas de ribonucleótidos de 5-aminoimidazol de Escherichia coli y Gallus gallus: un caso de mecanismos catalíticos divergentes". Bioquímica . 33 (39): 11927–34. doi :10.1021/bi00205a031. PMID 7918411.
- ^ ab Srere, PA (1987). "Complejos de enzimas metabólicas secuenciales". Revista anual de bioquímica . 56 (1): 89–124. doi :10.1146/annurev.bi.56.070187.000513. PMID 2441660.
- ^ Broach, editado por Jeffrey N. Strathern, Elizabeth W. Jones, James R. (1981). Biología molecular de la levadura Saccharomyces . Cold Spring Harbor, NY: Cold Spring Harbor Laboratory. ISBN 978-0879691394.
{{cite book}}:|first=tiene nombre genérico ( ayuda )CS1 maint: multiple names: authors list (link) - ^ abc O'Donovan, GA; Neuhard, J (septiembre de 1970). "Metabolismo de la pirimidina en microorganismos". Bacteriological Reviews . 34 (3): 278–343. doi :10.1128/MMBR.34.3.278-343.1970. PMC 378357 . PMID 4919542.
- ^ abcd Geer, Gerald Karp ; responsable de la revisión del capítulo 15 Peter van der (2004). Biología celular y molecular: conceptos y experimentos (4.ª ed., Wiley International ed.). Nueva York: J. Wiley & Sons. ISBN 978-0471656654.
{{cite book}}: CS1 maint: multiple names: authors list (link) CS1 maint: numeric names: authors list (link) - ^ abcd Griffiths, Anthony JF (1999). Análisis genético moderno (2.ª edición impresa). Nueva York: Freeman. ISBN 978-0716731184.
- ^ ab Wu, G (mayo de 2009). "Aminoácidos: metabolismo, funciones y nutrición". Aminoácidos . 37 (1): 1–17. doi :10.1007/s00726-009-0269-0. PMID 19301095. S2CID 1870305.
- ^ Mousdale, DM; Coggins, JR (1991). "Síntesis de aminoácidos". Sitios objetivo de la acción de los herbicidas . págs. 29-56. doi :10.1007/978-1-4899-2433-9_2. ISBN 978-1-4899-2435-3.
- ^ Miflin, BJ; Lea, PJ (1977). "Metabolismo de aminoácidos". Revista anual de fisiología vegetal . 28 : 299–329. doi :10.1146/annurev.pp.28.060177.001503.
- ^ abc Umbarger, HE (1978). "Biosíntesis de aminoácidos y su regulación". Revista Anual de Bioquímica . 47 (1): 532–606. doi :10.1146/annurev.bi.47.070178.002533. PMID 354503.
- ^ Pérez-Arellano, I; Carmona-Alvarez, F; Martínez, AI; Rodríguez-Díaz, J; Cervera, J (marzo de 2010). "Sintasa de pirrolina-5-carboxilato y biosíntesis de prolina: de la osmotolerancia a una enfermedad metabólica rara". Protein Science . 19 (3): 372–82. doi :10.1002/pro.340. PMC 2866264 . PMID 20091669.
- ^ Xu, Y; Labedan, B; Glansdorff, N (marzo de 2007). "Sorprendente biosíntesis de arginina: una reevaluación de la enzimología y la evolución de la vía en microorganismos". Microbiology and Molecular Biology Reviews . 71 (1): 36–47. doi :10.1128/MMBR.00032-06. PMC 1847373 . PMID 17347518.
- ^ "MetaCyc: Biosíntesis de L-lisina I".
- ^ PETERKOFSKY, B; GILVARG, C (mayo de 1961). "Transaminasa N-succinil-L-diaminopimélico-glutámico". Revista de química biológica . 236 (5): 1432–8. doi : 10.1016/S0021-9258(18)64192-4 . PMID 13734750.
- ^ KINDLER, SH; GILVARG, C (diciembre de 1960). "N-Succinil-L-2,6-diaminopimélico ácido desacilasa". The Journal of Biological Chemistry . 235 : 3532–5. doi : 10.1016/S0021-9258(18)64502-8 . PMID 13756049.
- ^ Born, TL; Blanchard, JS (octubre de 1999). "Estudios de estructura y función de las enzimas en la vía del diaminopimelato de la biosíntesis de la pared celular bacteriana". Current Opinion in Chemical Biology . 3 (5): 607–13. doi :10.1016/s1367-5931(99)00016-2. PMID 10508663.
- ^ "Escherichia coli K-12 substr. MG1655". biosíntesis de serina . SRI International . Consultado el 12 de diciembre de 2013 .
- ^ Bell, JK; Grant, GA; Banaszak, LJ (30 de marzo de 2004). "Estados multiconformacionales en la fosfoglicerato deshidrogenasa". Bioquímica . 43 (12): 3450–8. doi :10.1021/bi035462e. PMID 15035616.
- ^ Dubnovitsky, AP; Kapetaniou, EG; Papageorgiou, AC (enero de 2005). "Adaptación enzimática al pH alcalino: estructura de resolución atómica (1,08 A) de la fosfoserina aminotransferasa de Bacillus alcalophilus". Protein Science . 14 (1): 97–110. doi :10.1110/ps.041029805. PMC 2253317 . PMID 15608117.
- ^ Wang, W; Kim, R; Jancarik, J; Yokota, H; Kim, SH (10 de enero de 2001). "Estructura cristalina de la fosfatasa de fosfoserina de Methanococcus jannaschii, un hipertermófilo, a una resolución de 1,8 A". Structure . 9 (1): 65–71. doi : 10.1016/s0969-2126(00)00558-x . PMID 11342136.
- ^ Monschau, N; Stahmann, KP; Sahm, H; McNeil, JB; Bognar, AL (1 de mayo de 1997). "Identificación de Saccharomyces cerevisiae GLY1 como una treonina aldolasa: una enzima clave en la biosíntesis de glicina". FEMS Microbiology Letters . 150 (1): 55–60. doi : 10.1111/j.1574-6968.1997.tb10349.x . PMID 9163906.
- ^ Pye, VE; Tingey, AP; Robson, RL; Moody, PC (24 de septiembre de 2004). "La estructura y el mecanismo de la serina acetiltransferasa de Escherichia coli". The Journal of Biological Chemistry . 279 (39): 40729–36. doi : 10.1074/jbc.M403751200 . PMID 15231846.
- ^ Huang, B; Vetting, MW; Roderick, SL (mayo de 2005). "El sitio activo de la O-acetilserina sulfhidrilasa es el punto de anclaje para la formación del complejo bienzimático con la serina acetiltransferasa". Journal of Bacteriology . 187 (9): 3201–5. doi :10.1128/JB.187.9.3201-3205.2005. PMC 1082839 . PMID 15838047.
- ^ McPhalen, CA; Vincent, MG; Picot, D; Jansonius, JN ; Lesk, AM; Chothia, C (5 de septiembre de 1992). "Cierre de dominio en la aspartato aminotransferasa mitocondrial". Journal of Molecular Biology . 227 (1): 197–213. doi :10.1016/0022-2836(92)90691-C. PMID 1522585.
- ^ Larsen, TM; Boehlein, SK; Schuster, SM; Richards, NG; Thoden, JB; Holden, HM; Rayment, I (7 de diciembre de 1999). "Estructura tridimensional de la asparagina sintetasa B de Escherichia coli: un breve viaje desde el sustrato hasta el producto". Bioquímica . 38 (49): 16146–57. CiteSeerX 10.1.1.453.5998 . doi :10.1021/bi9915768. PMID 10587437.
- ^ Velasco, AM; Leguina, JI; Lazcano, A (octubre de 2002). "Evolución molecular de las vías biosintéticas de la lisina". Journal of Molecular Evolution . 55 (4): 445–59. Bibcode :2002JMolE..55..445V. doi :10.1007/s00239-002-2340-2. PMID 12355264. S2CID 19460256.
- ^ Kotaka, M; Ren, J; Lockyer, M; Hawkins, AR; Stammers, DK (20 de octubre de 2006). "Estructuras de la aspartoquinasa III de Escherichia coli en estado R y T. Mecanismos de la transición alostérica e inhibición por lisina". The Journal of Biological Chemistry . 281 (42): 31544–52. doi : 10.1074/jbc.M605886200 . PMID 16905770.
- ^ Hadfield, A; Kryger, G; Ouyang, J; Petsko, GA; Ringe, D; Viola, R (18 de junio de 1999). "Estructura de la aspartato-beta-semialdehído deshidrogenasa de Escherichia coli, una enzima clave en la familia aspartato de la biosíntesis de aminoácidos". Journal of Molecular Biology . 289 (4): 991–1002. doi :10.1006/jmbi.1999.2828. PMID 10369777.
- ^ Mirwaldt, C; Korndörfer, I; Huber, R (10 de febrero de 1995). "La estructura cristalina de la dihidrodipicolinato sintasa de Escherichia coli con una resolución de 2,5 A". Journal of Molecular Biology . 246 (1): 227–39. doi :10.1006/jmbi.1994.0078. PMID 7853400.
- ^ Cirilli, M; Zheng, R; Scapin, G; Blanchard, JS (16 de septiembre de 2003). "Las estructuras tridimensionales de los complejos de dihidrodipicolinato reductasa-NADH-2,6-PDC y -NADPH-2,6-PDC de Mycobacterium tuberculosis. Análisis estructural y mutagénico de la especificidad relajada de nucleótidos". Bioquímica . 42 (36): 10644–50. doi :10.1021/bi030044v. PMID 12962488.
- ^ Beaman, TW; Binder, DA; Blanchard, JS; Roderick, SL (21 de enero de 1997). "Estructura tridimensional de la tetrahidrodipicolinato N-succiniltransferasa". Bioquímica . 36 (3): 489–94. doi :10.1021/bi962522q. PMID 9012664.
- ^ Weyand, S; Kefala, G; Weiss, MS (30 de marzo de 2007). "La estructura tridimensional de la N-succinildiaminopimelato aminotransferasa de Mycobacterium tuberculosis". Journal of Molecular Biology . 367 (3): 825–38. doi :10.1016/j.jmb.2007.01.023. PMID 17292400.
- ^ Nocek, BP; Gillner, DM; Fan, Y; Holz, RC; Joachimiak, A (2 de abril de 2010). "Base estructural para la catálisis por las formas mono y dimetaladas de la desuccinilasa del ácido N-succinil-L,L-diaminopimélico codificada por dapE". Journal of Molecular Biology . 397 (3): 617–26. doi :10.1016/j.jmb.2010.01.062. PMC 2885003 . PMID 20138056.
- ^ Pillai, B; Cherney, M; Diaper, CM; Sutherland, A; Blanchard, JS; Vederas, JC; James, MN (23 de noviembre de 2007). "Dinámica de la catálisis revelada a partir de las estructuras cristalinas de mutantes de la diaminopimelato epimerasa". Comunicaciones de investigación bioquímica y biofísica . 363 (3): 547–53. doi :10.1016/j.bbrc.2007.09.012. PMID 17889830.
- ^ Gokulan, K; Rupp, B; Pavelka MS, Jr; Jacobs WR, Jr; Sacchettini, JC (16 de mayo de 2003). "Estructura cristalina de la diaminopimelato descarboxilasa de Mycobacterium tuberculosis, una enzima esencial en la biosíntesis de lisina bacteriana". The Journal of Biological Chemistry . 278 (20): 18588–96. doi : 10.1074/jbc.M301549200 . PMID 12637582.
- ^ abc Weaver, Robert F. (2005). Biología molecular (3.ª ed.). Boston: McGraw-Hill Higher Education. ISBN 978-0-07-284611-9.
- ^ abcde Cooper, Geoffrey M. (2000). La célula: un enfoque molecular (2.ª ed.). Washington (DC): ASM Press. ISBN 978-0878931064.
- ^ Jackson, RJ; et al. (febrero de 2010). "El mecanismo de iniciación de la traducción eucariota y principios de su regulación". Biología celular molecular . 10 (2): 113–127. doi :10.1038/nrm2838. PMC 4461372 . PMID 20094052.
- ^ Green, Rachel; Harry F. Noller; et al. (1997). "Ribosomas y traducción". Annu. Rev. Biochem . 66 : 679–716. doi :10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ abcd Weissbach, Herbert; Pestka, Sidney (1977). Mecanismos moleculares de la biosíntesis de proteínas . Nueva York: Academic Press. ISBN 978-0127442501.
- ^ Frank, J; Haixiao Gao; et al. (septiembre de 2007). "El proceso de translocación ARNm-ARNt". PNAS . 104 (50): 19671–19678. doi : 10.1073/pnas.0708517104 . PMC 2148355 . PMID 18003906.
- ^ abc Bandeali, Salman J.; Daye, Jad; Virani, Salim S. (30 de noviembre de 2013). "Nuevas terapias para el tratamiento de la hipercolesterolemia familiar". Current Atherosclerosis Reports . 16 (1): 382. doi :10.1007/s11883-013-0382-0. PMID 24293346. S2CID 8903481.
- ^ abc Kang, Tae Hyuk; Park, Yongjin; Bader, Joel S.; Friedmann, Theodore; Cooney, Austin John (9 de octubre de 2013). "El gen de mantenimiento hipoxantina guanina fosforibosiltransferasa (HPRT) regula múltiples vías metabólicas y de desarrollo de la diferenciación neuronal de células madre embrionarias murinas". PLOS ONE . 8 (10): e74967. Bibcode :2013PLoSO...874967K. doi : 10.1371/journal.pone.0074967 . PMC 3794013 . PMID 24130677.
- ^ abc Walport, Ken Murphy, Paul Travers, Mark (2011). Inmunobiología de Janeway (8.ª ed.). Oxford: Taylor & Francis. ISBN 978-0815342434.
{{cite book}}: CS1 maint: multiple names: authors list (link) - ^ ab Hughes, editado por Donald C. Lo, Robert E. (2010). Neurobiología de la enfermedad de Huntington: aplicaciones al descubrimiento de fármacos (2.ª ed.). Boca Raton: CRC Press/Taylor & Francis Group. ISBN 978-0849390005.
{{cite book}}:|first=tiene nombre genérico ( ayuda )CS1 maint: multiple names: authors list (link) - ^ Biglan, Kevin M.; Ross, Christopher A.; Langbehn, Douglas R.; Aylward, Elizabeth H.; Stout, Julie C.; Queller, Sarah; Carlozzi, Noelle E.; Duff, Kevin; Beglinger, Leigh J.; Paulsen, Jane S. (26 de junio de 2009). "Anormalidades motoras en personas con enfermedad de Huntington premanifiesta: el estudio PREDICT-HD". Trastornos del movimiento . 24 (12): 1763–1772. doi :10.1002/mds.22601. PMC 3048804 . PMID 19562761.




![{\displaystyle {\ce {Reactivo ->[][enzima] Producto}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d97fbef58f64fb0b7d05d90fa0a73b05f5919fc8)

![{\displaystyle {\ce {{Molécula~precursora}+Cofactor->[][enzima]macromolécula}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b192f25eec9ceebed179bfeb43ae27795f3a91be)




