Secuenciación de ADN
| Parte de una serie sobre |
| Genética |
|---|
 |
La secuenciación del ADN es el proceso de determinar la secuencia de ácidos nucleicos (el orden de los nucleótidos en el ADN) . Incluye cualquier método o tecnología que se utilice para determinar el orden de las cuatro bases: adenina , guanina , citosina y timina . La aparición de métodos rápidos de secuenciación del ADN ha acelerado enormemente la investigación y los descubrimientos biológicos y médicos. [1] [2]
El conocimiento de las secuencias de ADN se ha vuelto indispensable para la investigación biológica básica, los proyectos genográficos de ADN y en numerosos campos aplicados como el diagnóstico médico , la biotecnología , la biología forense , la virología y la sistemática biológica . La comparación de secuencias de ADN sanas y mutadas puede diagnosticar diferentes enfermedades, incluidos varios tipos de cáncer, [3] caracterizar el repertorio de anticuerpos, [4] y puede usarse para guiar el tratamiento del paciente. [5] Tener una forma rápida de secuenciar el ADN permite administrar una atención médica más rápida e individualizada y permitir identificar y catalogar más organismos. [4]
La rápida velocidad de secuenciación lograda con la moderna tecnología de secuenciación de ADN ha sido fundamental en la secuenciación de secuencias de ADN completas, o genomas , de numerosos tipos y especies de vida, incluido el genoma humano y otras secuencias de ADN completas de muchas especies animales, vegetales y microbianas.

Las primeras secuencias de ADN fueron obtenidas a principios de la década de 1970 por investigadores académicos que utilizaban métodos laboriosos basados en cromatografía bidimensional . Tras el desarrollo de métodos de secuenciación basados en fluorescencia con un secuenciador de ADN , [6] la secuenciación de ADN se ha vuelto más sencilla y órdenes de magnitud más rápida. [7] [8]
Aplicaciones
La secuenciación de ADN se puede utilizar para determinar la secuencia de genes individuales , regiones genéticas más grandes (es decir, grupos de genes u operones ), cromosomas completos o genomas enteros de cualquier organismo. La secuenciación de ADN también es la forma más eficiente de secuenciar indirectamente ARN o proteínas (a través de sus marcos de lectura abiertos ). De hecho, la secuenciación de ADN se ha convertido en una tecnología clave en muchas áreas de la biología y otras ciencias como la medicina, la ciencia forense y la antropología .
Biología molecular
La secuenciación se utiliza en biología molecular para estudiar los genomas y las proteínas que codifican. La información obtenida mediante la secuenciación permite a los investigadores identificar cambios en los genes y el ADN no codificante (incluidas las secuencias reguladoras), asociaciones con enfermedades y fenotipos, e identificar posibles dianas farmacológicas.
Biología evolutiva
Dado que el ADN es una macromolécula informativa en términos de transmisión de una generación a otra, la secuenciación del ADN se utiliza en biología evolutiva para estudiar cómo se relacionan los diferentes organismos y cómo evolucionaron. En febrero de 2021, los científicos informaron, por primera vez, de la secuenciación del ADN de restos animales , en este caso un mamut , de más de un millón de años, el ADN más antiguo secuenciado hasta la fecha. [9] [10]
Metagenómica
El campo de la metagenómica implica la identificación de organismos presentes en un cuerpo de agua, aguas residuales , suciedad, desechos filtrados del aire o muestras de organismos extraídas con hisopos. Saber qué organismos están presentes en un entorno particular es fundamental para la investigación en ecología , epidemiología , microbiología y otros campos. La secuenciación permite a los investigadores determinar qué tipos de microbios pueden estar presentes en un microbioma , por ejemplo.
Virología
Como la mayoría de los virus son demasiado pequeños para ser vistos con un microscopio óptico, la secuenciación es una de las principales herramientas en virología para identificar y estudiar el virus. [11] Los genomas virales pueden estar basados en ADN o ARN. Los virus de ARN son más sensibles al tiempo para la secuenciación del genoma, ya que se degradan más rápido en muestras clínicas. [12] La secuenciación tradicional de Sanger y la secuenciación de próxima generación se utilizan para secuenciar virus en investigación básica y clínica, así como para el diagnóstico de infecciones virales emergentes, epidemiología molecular de patógenos virales y pruebas de resistencia a medicamentos. Hay más de 2,3 millones de secuencias virales únicas en GenBank . [11] Recientemente, NGS ha superado a Sanger tradicional como el enfoque más popular para generar genomas virales. [11]
Durante el brote de influenza aviar de 1997 , la secuenciación viral determinó que el subtipo de influenza se originó a través de una recombinación entre codornices y aves de corral. Esto condujo a una legislación en Hong Kong que prohibía la venta de codornices y aves de corral vivas juntas en el mercado. La secuenciación viral también se puede utilizar para estimar cuándo comenzó un brote viral mediante el uso de una técnica de reloj molecular . [12]
Medicamento
Los técnicos médicos pueden secuenciar genes (o, teóricamente, genomas completos) de pacientes para determinar si existe riesgo de enfermedades genéticas. Esta es una forma de prueba genética , aunque algunas pruebas genéticas pueden no implicar la secuenciación de ADN.
A partir de 2013, la secuenciación de ADN se ha utilizado cada vez más para diagnosticar y tratar enfermedades raras. A medida que se identifican más genes que causan enfermedades genéticas raras, los diagnósticos moleculares para los pacientes se vuelven más comunes. La secuenciación de ADN permite a los médicos identificar enfermedades genéticas, mejorar el tratamiento de las enfermedades, brindar asesoramiento reproductivo y terapias más efectivas. [13] Los paneles de secuenciación de genes se utilizan para identificar múltiples causas genéticas potenciales de un trastorno sospechoso. [14]
Además, la secuenciación de ADN puede ser útil para determinar una bacteria específica, lo que permite tratamientos antibióticos más precisos , reduciendo así el riesgo de crear resistencia a los antimicrobianos en las poblaciones de bacterias. [15] [16] [17] [18] [19] [20]
Investigación forense
La secuenciación de ADN puede utilizarse junto con métodos de elaboración de perfiles de ADN para la identificación forense [21] y las pruebas de paternidad . Las pruebas de ADN han evolucionado enormemente en las últimas décadas para vincular en última instancia una huella de ADN con lo que se está investigando. Los patrones de ADN en las huellas dactilares, la saliva, los folículos pilosos, etc., separan de forma única a cada organismo vivo de otro. La prueba de ADN es una técnica que puede detectar genomas específicos en una cadena de ADN para producir un patrón único e individualizado.
La secuenciación de ADN puede utilizarse junto con los métodos de elaboración de perfiles de ADN para la identificación forense y las pruebas de paternidad , ya que ha evolucionado significativamente en las últimas décadas para vincular en última instancia una huella de ADN con lo que se está investigando. Los patrones de ADN en las huellas dactilares, la saliva, los folículos pilosos y otros fluidos corporales separan de forma única a cada organismo vivo de otro, lo que lo convierte en una herramienta invaluable en el campo de la ciencia forense . El proceso de prueba de ADN implica la detección de genomas específicos en una cadena de ADN para producir un patrón único e individualizado, que puede usarse para identificar a las personas o determinar sus relaciones.
Los avances en la tecnología de secuenciación de ADN han hecho posible analizar y comparar grandes cantidades de datos genéticos de forma rápida y precisa, lo que permite a los investigadores reunir pruebas y resolver crímenes de forma más eficiente. Esta tecnología se ha utilizado en diversas aplicaciones, incluida la identificación forense, las pruebas de paternidad y la identificación humana en casos en los que los métodos de identificación tradicionales no están disponibles o no son fiables. El uso de la secuenciación de ADN también ha llevado al desarrollo de nuevas técnicas forenses, como el fenotipado de ADN , que permite a los investigadores predecir las características físicas de un individuo basándose en sus datos genéticos.
Además de sus aplicaciones en la ciencia forense, la secuenciación del ADN también se ha utilizado en la investigación y el diagnóstico médicos. Ha permitido a los científicos identificar mutaciones y variaciones genéticas asociadas con ciertas enfermedades y trastornos, lo que permite diagnósticos más precisos y tratamientos específicos. Además, la secuenciación del ADN también se ha utilizado en la biología de la conservación para estudiar la diversidad genética de especies en peligro de extinción y desarrollar estrategias para su conservación.
Además, el uso de la secuenciación del ADN también ha suscitado importantes consideraciones éticas y jurídicas. Por ejemplo, existen preocupaciones sobre la privacidad y la seguridad de los datos genéticos, así como sobre la posibilidad de un uso indebido o discriminación en función de la información genética. Como resultado, hay debates en curso sobre la necesidad de establecer normas y directrices para garantizar el uso responsable de la tecnología de secuenciación del ADN.
En general, el desarrollo de la tecnología de secuenciación de ADN ha revolucionado el campo de la ciencia forense y tiene implicaciones de largo alcance para nuestra comprensión de la genética, la medicina y la biología de la conservación.
Las cuatro bases canónicas
La estructura canónica del ADN tiene cuatro bases: timina (T), adenina (A), citosina (C) y guanina (G). La secuenciación del ADN es la determinación del orden físico de estas bases en una molécula de ADN. Sin embargo, hay muchas otras bases que pueden estar presentes en una molécula. En algunos virus (específicamente, bacteriófagos ), la citosina puede ser reemplazada por hidroximetil o hidroximetilglucosa citosina. [22] En el ADN de mamíferos, se pueden encontrar bases variantes con grupos metilo o fosfosulfato. [23] [24] Dependiendo de la técnica de secuenciación, una modificación particular, por ejemplo, la 5mC ( 5 metil citosina ) común en humanos, puede o no detectarse. [25]
En casi todos los organismos, el ADN se sintetiza in vivo utilizando sólo las 4 bases canónicas; la modificación que ocurre después de la replicación crea otras bases como 5 metil C. Sin embargo, algunos bacteriófagos pueden incorporar directamente una base no estándar. [26]
Además de las modificaciones, el ADN está bajo el ataque constante de agentes ambientales como los rayos UV y los radicales de oxígeno. En la actualidad, la presencia de estas bases dañadas no se detecta mediante la mayoría de los métodos de secuenciación del ADN, aunque PacBio ha publicado sobre esto. [27]
Historia
Descubrimiento de la estructura y función del ADN
El ácido desoxirribonucleico ( ADN ) fue descubierto y aislado por primera vez por Friedrich Miescher en 1869, pero permaneció poco estudiado durante muchas décadas porque se pensaba que las proteínas, en lugar del ADN, contenían el código genético de la vida. Esta situación cambió después de 1944 como resultado de algunos experimentos de Oswald Avery , Colin MacLeod y Maclyn McCarty que demostraron que el ADN purificado podía transformar una cepa de bacterias en otra. Esta fue la primera vez que se demostró que el ADN era capaz de transformar las propiedades de las células.
En 1953, James Watson y Francis Crick propusieron su modelo de doble hélice del ADN, basado en estructuras de rayos X cristalizadas que estaba estudiando Rosalind Franklin . Según el modelo, el ADN está compuesto por dos cadenas de nucleótidos enrolladas una alrededor de la otra, unidas entre sí por enlaces de hidrógeno y que discurren en direcciones opuestas. Cada cadena está compuesta por cuatro nucleótidos complementarios: adenina (A), citosina (C), guanina (G) y timina (T), con una A en una cadena siempre emparejada con una T en la otra, y una C siempre emparejada con una G. Propusieron que dicha estructura permitía que cada cadena se utilizara para reconstruir la otra, una idea central para la transmisión de información hereditaria entre generaciones. [28]

La base para la secuenciación de proteínas fue establecida por primera vez por el trabajo de Frederick Sanger , quien en 1955 había completado la secuencia de todos los aminoácidos de la insulina , una pequeña proteína secretada por el páncreas. Esto proporcionó la primera evidencia concluyente de que las proteínas eran entidades químicas con un patrón molecular específico en lugar de una mezcla aleatoria de material suspendido en un líquido. El éxito de Sanger en la secuenciación de la insulina estimuló a los cristalógrafos de rayos X, incluidos Watson y Crick, quienes para entonces estaban tratando de entender cómo el ADN dirigía la formación de proteínas dentro de una célula. Poco después de asistir a una serie de conferencias impartidas por Frederick Sanger en octubre de 1954, Crick comenzó a desarrollar una teoría que sostenía que la disposición de los nucleótidos en el ADN determinaba la secuencia de aminoácidos en las proteínas, lo que a su vez ayudaba a determinar la función de una proteína. Publicó esta teoría en 1958. [29]
Secuenciación de ARN
La secuenciación de ARN fue una de las primeras formas de secuenciación de nucleótidos. El hito principal de la secuenciación de ARN es la secuencia del primer gen completo y el genoma completo del bacteriófago MS2 , identificado y publicado por Walter Fiers y sus colaboradores en la Universidad de Ghent ( Ghent , Bélgica ), en 1972 [30] y 1976. [31] Los métodos tradicionales de secuenciación de ARN requieren la creación de una molécula de ADNc que debe secuenciarse. [32]
Métodos tradicionales de secuenciación de ARN
Los métodos tradicionales de secuenciación de ARN implican varios pasos:
1) Transcripción inversa : el primer paso es convertir la molécula de ARN en una molécula de ADN complementario (ADNc) utilizando una enzima llamada transcriptasa inversa .
2) Síntesis de ADNc : La molécula de ADNc se sintetiza luego a través de un proceso llamado PCR ( reacción en cadena de la polimerasa ), que amplifica el ADNc para producir múltiples copias.
3) Secuenciación : A continuación, el ADNc amplificado se secuencia utilizando una técnica como la secuenciación de Sanger o la secuenciación de Maxam-Gilbert .
Desafíos y limitaciones
Los métodos tradicionales de secuenciación de ARN tienen varias limitaciones. Por ejemplo:
Requieren la creación de una molécula de ADNc, lo que puede requerir mucho tiempo y trabajo. Son propensos a errores y sesgos, lo que puede afectar la precisión de los resultados de la secuenciación. Tienen una capacidad limitada para detectar transcripciones raras o de baja abundancia. Avances en la tecnología de secuenciación de ARN
En los últimos años, los avances en la tecnología de secuenciación de ARN han abordado algunas de estas limitaciones. Nuevos métodos como la secuenciación de próxima generación (NGS) y la secuenciación de moléculas individuales en tiempo real (SMRT) han permitido una secuenciación más rápida, más precisa y más rentable de las moléculas de ARN. Estos avances han abierto nuevas posibilidades para estudiar la expresión genética, identificar nuevos genes y comprender la regulación de la expresión genética.
Métodos tempranos de secuenciación de ADN
El primer método para determinar secuencias de ADN implicó una estrategia de extensión de cebador de ubicación específica establecida por Ray Wu en la Universidad de Cornell en 1970. [33] La catálisis de la ADN polimerasa y el etiquetado de nucleótidos específicos, ambos de los cuales figuran prominentemente en los esquemas de secuenciación actuales, se utilizaron para secuenciar los extremos cohesivos del ADN del fago lambda. [34] [35] [36] Entre 1970 y 1973, Wu, R Padmanabhan y colegas demostraron que este método puede emplearse para determinar cualquier secuencia de ADN utilizando cebadores sintéticos de ubicación específica. [37] [38] [8] Frederick Sanger luego adoptó esta estrategia de extensión de cebador para desarrollar métodos de secuenciación de ADN más rápidos en el Centro MRC , Cambridge , Reino Unido y publicó un método para "secuenciación de ADN con inhibidores de terminación de cadena" en 1977. [39] Walter Gilbert y Allan Maxam en Harvard también desarrollaron métodos de secuenciación, incluido uno para "secuenciación de ADN por degradación química". [40] [41] En 1973, Gilbert y Maxam informaron la secuencia de 24 pares de bases utilizando un método conocido como análisis de puntos errantes. [42] Los avances en la secuenciación se vieron facilitados por el desarrollo simultáneo de la tecnología del ADN recombinante , que permitió aislar muestras de ADN de fuentes distintas de los virus.
Secuenciación de genomas completos

El primer genoma de ADN completo que se secuenció fue el del bacteriófago φX174 en 1977. [43] Los científicos del Consejo de Investigación Médica descifraron la secuencia completa de ADN del virus de Epstein-Barr en 1984 y descubrieron que contenía 172.282 nucleótidos. La finalización de la secuencia marcó un punto de inflexión importante en la secuenciación de ADN porque se logró sin conocimiento previo del perfil genético del virus. [44] [8]
A principios de los años 1980, Herbert Pohl y sus colaboradores desarrollaron un método no radiactivo para transferir las moléculas de ADN de las mezclas de reacción de secuenciación a una matriz inmovilizadora durante la electroforesis . [45] [46] A continuación, con la comercialización del secuenciador de ADN "Direct-Blotting-Electrophoresis-System GATC 1500" de GATC Biotech , que se utilizó intensamente en el marco del programa de secuenciación del genoma de la UE, se obtuvo la secuencia completa del ADN del cromosoma II de la levadura Saccharomyces cerevisiae . [47] El laboratorio de Leroy E. Hood en el Instituto de Tecnología de California anunció la primera máquina de secuenciación de ADN semiautomatizada en 1986. [48] A esto le siguió la comercialización por parte de Applied Biosystems de la primera máquina de secuenciación totalmente automatizada, la ABI 370, en 1987 y la Genesis 2000 de Dupont [49] que utilizó una novedosa técnica de etiquetado fluorescente que permitía identificar los cuatro didesoxinucleótidos en un solo carril. En 1990, los Institutos Nacionales de Salud de Estados Unidos (NIH) habían comenzado ensayos de secuenciación a gran escala en Mycoplasma capricolum , Escherichia coli , Caenorhabditis elegans y Saccharomyces cerevisiae a un costo de US$0,75 por base. Mientras tanto, la secuenciación de secuencias de ADNc humano llamadas etiquetas de secuencia expresada comenzó en el laboratorio de Craig Venter , un intento de capturar la fracción codificante del genoma humano . [50] En 1995, Venter, Hamilton Smith y sus colegas del Instituto de Investigación Genómica (TIGR) publicaron el primer genoma completo de un organismo de vida libre, la bacteria Haemophilus influenzae . El cromosoma circular contiene 1.830.137 bases y su publicación en la revista Science [51] marcó el primer uso publicado de la secuenciación shotgun del genoma completo, eliminando la necesidad de esfuerzos iniciales de mapeo.
En 2001, se habían utilizado métodos de secuenciación de escopeta para producir un borrador de la secuencia del genoma humano. [52] [53]
Métodos de secuenciación de alto rendimiento (HTS)

A mediados y finales de la década de 1990 se desarrollaron varios métodos nuevos para la secuenciación de ADN y se implementaron en secuenciadores de ADN comerciales en el año 2000. En conjunto, se los llamó métodos de secuenciación de "próxima generación" o "segunda generación" (NGS), para distinguirlos de los métodos anteriores, incluida la secuenciación de Sanger . A diferencia de la primera generación de secuenciación, la tecnología NGS se caracteriza típicamente por ser altamente escalable, lo que permite secuenciar todo el genoma a la vez. Por lo general, esto se logra fragmentando el genoma en pequeños fragmentos, tomando un fragmento al azar y secuenciándolo utilizando una de varias tecnologías, como las que se describen a continuación. Un genoma completo es posible porque se secuencian múltiples fragmentos a la vez (lo que le da el nombre de secuenciación "masivamente paralela") en un proceso automatizado.
La tecnología NGS ha permitido a los investigadores buscar información sobre la salud, a los antropólogos investigar los orígenes humanos y está catalizando el movimiento de la " medicina personalizada ". Sin embargo, también ha abierto la puerta a un mayor margen de error. Existen muchas herramientas de software para realizar el análisis computacional de los datos de NGS, a menudo compilados en plataformas en línea como CSI NGS Portal, cada una con su propio algoritmo. Incluso los parámetros dentro de un paquete de software pueden cambiar el resultado del análisis. Además, las grandes cantidades de datos producidos por la secuenciación de ADN también han requerido el desarrollo de nuevos métodos y programas para el análisis de secuencias. Se han intentado varios esfuerzos para desarrollar estándares en el campo de NGS para abordar estos desafíos, la mayoría de los cuales han sido esfuerzos a pequeña escala que surgieron de laboratorios individuales. Más recientemente, un esfuerzo grande, organizado y financiado por la FDA ha culminado en el estándar BioCompute .
El 26 de octubre de 1990, Roger Tsien , Pepi Ross, Margaret Fahnestock y Allan J Johnston presentaron una patente que describe la secuenciación por pasos ("base por base") con bloqueadores 3' removibles en matrices de ADN (borrones y moléculas de ADN individuales). [55] En 1996, Pål Nyrén y su estudiante Mostafa Ronaghi en el Instituto Real de Tecnología de Estocolmo publicaron su método de pirosecuenciación . [56]
El 1 de abril de 1997, Pascal Mayer y Laurent Farinelli presentaron patentes a la Organización Mundial de la Propiedad Intelectual que describen la secuenciación de colonias de ADN. [57] Los métodos de preparación de muestras de ADN y de reacción en cadena de la polimerasa (PCR) de superficie aleatoria descritos en esta patente, junto con el método de secuenciación "base por base" de Roger Tsien et al., ahora se implementan en los secuenciadores de genoma Hi-Seq de Illumina .
En 1998, Phil Green y Brent Ewing de la Universidad de Washington describieron su puntuación de calidad phred para el análisis de datos de secuenciadores, [58] una técnica de análisis histórica que ganó una amplia adopción y que sigue siendo la métrica más común para evaluar la precisión de una plataforma de secuenciación. [59]
Lynx Therapeutics publicó y comercializó la secuenciación masiva de firmas paralelas (MPSS) en 2000. Este método incorporó una tecnología de secuenciación en paralelo, mediada por adaptador/ligación y basada en perlas, y sirvió como el primer método de secuenciación de "próxima generación" disponible comercialmente, aunque no se vendieron secuenciadores de ADN a laboratorios independientes. [60]
Métodos básicos
Secuenciación Maxam-Gilbert
Allan Maxam y Walter Gilbert publicaron en 1977 un método de secuenciación de ADN basado en la modificación química del ADN y la posterior escisión en bases específicas. [40] Este método, también conocido como secuenciación química, permitió utilizar muestras purificadas de ADN de doble cadena sin necesidad de clonación adicional. El uso de marcado radiactivo en este método y su complejidad técnica desalentaron su uso extensivo después de que se habían realizado mejoras en los métodos de Sanger.
La secuenciación Maxam-Gilbert requiere el marcaje radiactivo en un extremo 5' del ADN y la purificación del fragmento de ADN que se va a secuenciar. A continuación, el tratamiento químico genera roturas en una pequeña proporción de una o dos de las cuatro bases de nucleótidos en cada una de las cuatro reacciones (G, A+G, C, C+T). La concentración de los productos químicos modificadores se controla para introducir, en promedio, una modificación por molécula de ADN. De este modo, se genera una serie de fragmentos marcados, desde el extremo radiomarcado hasta el primer sitio de "corte" en cada molécula. Los fragmentos de las cuatro reacciones se someten a electroforesis uno al lado del otro en geles de acrilamida desnaturalizantes para separarlos por tamaño. Para visualizar los fragmentos, el gel se expone a una película de rayos X para realizar una autorradiografía, lo que produce una serie de bandas oscuras, cada una de las cuales corresponde a un fragmento de ADN radiomarcado, a partir del cual se puede inferir la secuencia. [40]
Este método está prácticamente obsoleto a partir de 2023. [61]
Métodos de terminación de cadena
El método de terminación de cadena desarrollado por Frederick Sanger y colaboradores en 1977 pronto se convirtió en el método de elección, debido a su relativa facilidad y confiabilidad. [39] [62] Cuando se inventó, el método de terminación de cadena utilizaba menos sustancias químicas tóxicas y menores cantidades de radiactividad que el método de Maxam y Gilbert. Debido a su relativa facilidad, el método de Sanger pronto se automatizó y fue el método utilizado en la primera generación de secuenciadores de ADN .
La secuenciación de Sanger es el método que prevaleció desde la década de 1980 hasta mediados de la década de 2000. Durante ese período, se hicieron grandes avances en la técnica, como el marcaje fluorescente, la electroforesis capilar y la automatización general. Estos avances permitieron una secuenciación mucho más eficiente, lo que condujo a una reducción de costos. El método de Sanger, en forma de producción en masa, es la tecnología que produjo el primer genoma humano en 2001, marcando el comienzo de la era de la genómica . Sin embargo, más tarde en la década, llegaron al mercado enfoques radicalmente diferentes, lo que redujo el costo por genoma de $ 100 millones en 2001 a $ 10,000 en 2011. [63]
Secuenciación por síntesis
El objetivo de la secuenciación secuencial por síntesis (SBS) es determinar la secuenciación de una muestra de ADN detectando la incorporación de un nucleótido por una ADN polimerasa . Se utiliza una polimerasa modificada genéticamente para sintetizar una copia de una sola hebra de ADN y se controla la incorporación de cada nucleótido. El principio de la secuenciación en tiempo real por síntesis se describió por primera vez en 1993 [64] y se publicaron mejoras algunos años después. [65] Las partes clave son muy similares para todas las realizaciones de SBS e incluyen (1) amplificación de ADN (para mejorar la señal posterior) y unir el ADN que se va a secuenciar a un soporte sólido, (2) generación de ADN monocatenario en el soporte sólido, (3) incorporación de nucleótidos utilizando una polimerasa diseñada y (4) detección en tiempo real de la incorporación de nucleótidos. Los pasos 3 y 4 se repiten y la secuencia se ensambla a partir de las señales obtenidas en el paso 4. Este principio de secuenciación en tiempo real por síntesis se ha utilizado para casi todos los instrumentos de secuenciación paralela masiva , incluidos 454 , PacBio , IonTorrent , Illumina y MGI .
Secuenciación a gran escala yDe nuevosecuenciación
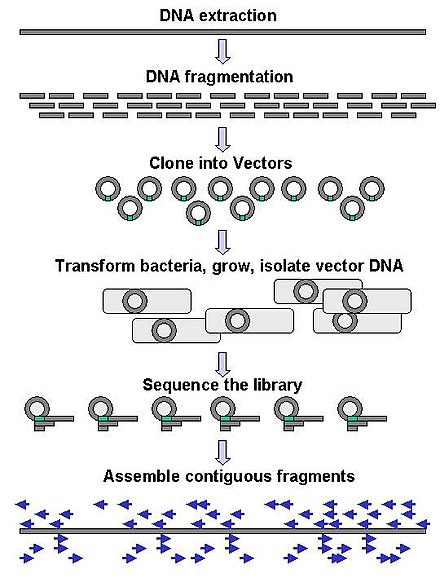
La secuenciación a gran escala a menudo tiene como objetivo secuenciar fragmentos de ADN muy largos, como cromosomas completos , aunque también se puede utilizar para generar cantidades muy grandes de secuencias cortas, como las que se encuentran en la visualización de fagos . Para objetivos más largos, como los cromosomas, los enfoques comunes consisten en cortar (con enzimas de restricción ) o cizallar (con fuerzas mecánicas) fragmentos de ADN grandes en fragmentos de ADN más cortos. Luego, el ADN fragmentado se puede clonar en un vector de ADN y amplificar en un huésped bacteriano como Escherichia coli . Los fragmentos de ADN cortos purificados a partir de colonias bacterianas individuales se secuencian individualmente y se ensamblan electrónicamente en una secuencia larga y contigua. Los estudios han demostrado que agregar un paso de selección de tamaño para recolectar fragmentos de ADN de tamaño uniforme puede mejorar la eficiencia de la secuenciación y la precisión del ensamblaje del genoma. En estos estudios, el dimensionamiento automatizado ha demostrado ser más reproducible y preciso que el dimensionamiento manual en gel. [66] [67] [68]
El término " secuenciación de novo " se refiere específicamente a los métodos utilizados para determinar la secuencia de ADN sin una secuencia conocida previamente. De novo se traduce del latín como "desde el principio". Los huecos en la secuencia ensamblada se pueden llenar mediante el método de primer walking . Las diferentes estrategias tienen diferentes ventajas y desventajas en cuanto a velocidad y precisión; los métodos shotgun se utilizan a menudo para secuenciar genomas grandes, pero su ensamblaje es complejo y difícil, en particular con repeticiones de secuencias que a menudo causan huecos en el ensamblaje del genoma.
La mayoría de los enfoques de secuenciación utilizan un paso de clonación in vitro para amplificar moléculas de ADN individuales, porque sus métodos de detección molecular no son lo suficientemente sensibles para la secuenciación de moléculas individuales. La PCR en emulsión [69] aísla moléculas de ADN individuales junto con perlas recubiertas de cebadores en gotitas acuosas dentro de una fase oleosa. Luego, una reacción en cadena de la polimerasa (PCR) recubre cada perla con copias clonales de la molécula de ADN seguida de inmovilización para una secuenciación posterior. La PCR en emulsión se utiliza en los métodos desarrollados por Marguilis et al. (comercializados por 454 Life Sciences ), Shendure y Porreca et al. (también conocidos como " secuenciación polonía ") y secuenciación SOLiD (desarrollada por Agencourt , más tarde Applied Biosystems , ahora Life Technologies ). [70] [71] [72] La PCR en emulsión también se utiliza en las plataformas GemCode y Chromium desarrolladas por 10x Genomics . [73]
Secuenciación de escopeta
La secuenciación shotgun es un método de secuenciación diseñado para el análisis de secuencias de ADN de más de 1000 pares de bases, incluidos cromosomas enteros. Este método requiere que el ADN objetivo se divida en fragmentos aleatorios. Después de secuenciar fragmentos individuales utilizando el método de terminación de cadena , las secuencias se pueden volver a ensamblar en función de sus regiones superpuestas. [74]
Métodos de alto rendimiento

La secuenciación de alto rendimiento, que incluye métodos de secuenciación de "lectura corta" de próxima generación y de "lectura larga" de tercera generación, [nt 1] se aplica a la secuenciación del exoma , la secuenciación del genoma, la resecuenciación del genoma, el perfil del transcriptoma ( RNA-Seq ), las interacciones ADN-proteína ( secuenciación ChIP ) y la caracterización del epigenoma . [75]
La alta demanda de secuenciación de bajo costo ha impulsado el desarrollo de tecnologías de secuenciación de alto rendimiento que paralelizan el proceso de secuenciación, produciendo miles o millones de secuencias simultáneamente. [76] [77] [78] Las tecnologías de secuenciación de alto rendimiento tienen como objetivo reducir el costo de la secuenciación de ADN más allá de lo que es posible con los métodos estándar de terminación de tinte. [79] En la secuenciación de ultra alto rendimiento, se pueden ejecutar hasta 500.000 operaciones de secuenciación por síntesis en paralelo. [80] [81] [82] Estas tecnologías llevaron a la capacidad de secuenciar un genoma humano completo en tan solo un día. [83] A partir de 2019 [actualizar], los líderes corporativos en el desarrollo de productos de secuenciación de alto rendimiento incluían Illumina , Qiagen y ThermoFisher Scientific . [83]
| Método | Longitud de lectura | Precisión (lectura única, no consenso) | Lecturas por ejecución | Tiempo por carrera | Costo por cada mil millones de bases (en dólares estadounidenses) | Ventajas | Desventajas |
|---|---|---|---|---|---|---|---|
| Secuenciación de moléculas individuales en tiempo real (Pacific Biosciences) | 30.000 pb ( N50 ); longitud máxima de lectura >100.000 bases [86] [87] [88] | 87% de precisión en lecturas sin procesar [89] | 4.000.000 por celda SMRT Sequel 2, 100–200 gigabases [86] [90] [91] | 30 minutos a 20 horas [86] [92] | 7,2-43,3 dólares | Rápido. Detecta 4mC, 5mC, 6mA. [93] | Rendimiento moderado. El equipo puede ser muy costoso. |
| Semiconductor iónico (secuenciación de Ion Torrent) | hasta 600 pb [94] | 99,6% [95] | hasta 80 millones | 2 horas | 66,8-950 dólares | Equipos más económicos. Rápidos. | Errores de homopolímeros. |
| Pirosecuenciación (454) | 700 pb | 99,9% | 1 millón | 24 horas | $10,000 | Lectura larga y rápida. | Las tiradas son caras. Errores de homopolímeros. |
| Secuenciación por síntesis (Illumina) | MiniSeq, NextSeq: 75–300 pb; MiSeq: 50–600 pb; HiSeq 2500: 50–500 pb; HiSeq 3/4000: 50–300 pb; HiSeq X: 300 pb | 99,9 % (Phred30) | MiniSeq/MiSeq: 1–25 millones; SiguienteSeq: 130-00 Millones; HiSeq 2500: 300 millones – 2 mil millones; HiSeq 3/4000 2,5 mil millones; HiSeq X: 3 mil millones | De 1 a 11 días, según el secuenciador y la longitud de lectura especificada [96] | De $5 a $150 | Potencial de alto rendimiento de secuencia, dependiendo del modelo de secuenciador y la aplicación deseada. | El equipo puede ser muy costoso y requiere altas concentraciones de ADN. |
| Síntesis de anclaje de sonda combinatoria (cPAS-BGI/MGI) | BGISEQ-50: 35-50 pb; MGISEQ 200: 50-200 pb; BGISEQ-500, MGISEQ-2000: 50-300 pb [97] | 99,9 % (Phred30) | BGISEQ-50: 160M; MGISEQ 200: 300M; BGISEQ-500: 1300M por celda de flujo; MGISEQ-2000: celda de flujo FCS de 375 M, celda de flujo FCL de 1500 M por celda de flujo. | De 1 a 9 días según el instrumento, la longitud de lectura y la cantidad de celdas de flujo ejecutadas a la vez. | 5-120 dólares | ||
| Secuenciación por ligación (secuenciación SOLiD) | 50+35 o 50+50 pb | 99,9% | 1.2 a 1.4 mil millones | 1 a 2 semanas | 60–130 dólares | Bajo costo por base. | Más lento que otros métodos. Tiene problemas para secuenciar secuencias palindrómicas. [98] |
| Secuenciación de nanoporos | Depende de la preparación de la biblioteca, no del dispositivo, por lo que el usuario elige la longitud de lectura (se han informado hasta 2 272 580 pb [99] ). | ~92–97% lectura única | Depende de la longitud de lectura seleccionada por el usuario | Datos transmitidos en tiempo real. Elija entre 1 minuto y 48 horas. | 7–100 dólares | Lecturas individuales más largas. Comunidad de usuarios accesible. Portátil (tamaño de la palma de la mano). | Rendimiento menor que otras máquinas, precisión de lectura única en 90 segundos. |
| Secuenciación GenapSys | Alrededor de 150 pb de un solo extremo | 99,9 % (Phred30) | 1 a 16 millones | Alrededor de 24 horas | $667 | Bajo costo del instrumento ($10,000) | |
| Terminación de cadena (secuenciación de Sanger) | 400 a 900 pb | 99,9% | N / A | 20 minutos a 3 horas | $2.400.000 | Útil para muchas aplicaciones. | Es más costoso y poco práctico para proyectos de secuenciación más grandes. Este método también requiere el paso de clonación de plásmidos o PCR, que consume mucho tiempo. |
Métodos de secuenciación de lectura larga
Secuenciación de moléculas individuales en tiempo real (SMRT)
La secuenciación SMRT se basa en el enfoque de secuenciación por síntesis. El ADN se sintetiza en guías de onda de modo cero (ZMW, por sus siglas en inglés), pequeños contenedores con forma de pozo con las herramientas de captura ubicadas en el fondo del pozo. La secuenciación se realiza con el uso de polimerasa no modificada (unida al fondo de la ZMW) y nucleótidos marcados con fluorescencia que fluyen libremente en la solución. Los pozos están construidos de manera que solo se detecte la fluorescencia que se produce en el fondo del pozo. La etiqueta fluorescente se desprende del nucleótido al incorporarse a la cadena de ADN, dejando una cadena de ADN sin modificar. Según Pacific Biosciences (PacBio), el desarrollador de la tecnología SMRT, esta metodología permite la detección de modificaciones de nucleótidos (como la metilación de citosina). Esto sucede a través de la observación de la cinética de la polimerasa. Este enfoque permite lecturas de 20.000 nucleótidos o más, con longitudes de lectura promedio de 5 kilobases. [90] [100] En 2015, Pacific Biosciences anunció el lanzamiento de un nuevo instrumento de secuenciación llamado Sequel System, con 1 millón de ZMW en comparación con los 150.000 ZMW del instrumento PacBio RS II. [101] [102] La secuenciación SMRT se conoce como secuenciación de " tercera generación " o de "lectura larga".
Secuenciación de ADN por nanoporos
El ADN que pasa a través del nanoporo cambia su corriente de iones. Este cambio depende de la forma, el tamaño y la longitud de la secuencia de ADN. Cada tipo de nucleótido bloquea el flujo de iones a través del poro durante un período de tiempo diferente. El método no requiere nucleótidos modificados y se realiza en tiempo real. La secuenciación por nanoporo se conoce como secuenciación de " tercera generación " o "de lectura larga", junto con la secuenciación SMRT.
Las primeras investigaciones industriales sobre este método se basaron en una técnica llamada "secuenciación de exonucleasa", donde la lectura de señales eléctricas se producía a medida que los nucleótidos pasaban por los poros de alfa(α)-hemolisina unidos covalentemente con ciclodextrina . [103] Sin embargo, el método comercial posterior, la "secuenciación de hebras", secuenciaba las bases de ADN en una hebra intacta.
Dos áreas principales de la secuenciación de nanoporos en desarrollo son la secuenciación de nanoporos en estado sólido y la secuenciación de nanoporos basada en proteínas. La secuenciación de nanoporos de proteínas utiliza complejos de proteínas de membrana como α-hemolisina, MspA ( Mycobacterium smegmatis Porin A) o CssG, que muestran una gran promesa dada su capacidad para distinguir entre nucleótidos individuales y grupos de nucleótidos. [104] Por el contrario, la secuenciación de nanoporos en estado sólido utiliza materiales sintéticos como nitruro de silicio y óxido de aluminio y se prefiere por su capacidad mecánica superior y su estabilidad térmica y química. [105] El método de fabricación es esencial para este tipo de secuenciación dado que la matriz de nanoporos puede contener cientos de poros con diámetros menores a ocho nanómetros. [104]
El concepto se originó a partir de la idea de que las moléculas de ADN o ARN monocatenario pueden ser impulsadas electroforéticamente en una secuencia lineal estricta a través de un poro biológico que puede tener menos de ocho nanómetros, y pueden detectarse dado que las moléculas liberan una corriente iónica mientras se mueven a través del poro. El poro contiene una región de detección capaz de reconocer diferentes bases, y cada base genera varias señales específicas en el tiempo correspondientes a la secuencia de bases a medida que cruzan el poro, que luego se evalúan. [105] El control preciso sobre el transporte de ADN a través del poro es crucial para el éxito. Se han utilizado varias enzimas, como exonucleasas y polimerasas, para moderar este proceso posicionándolas cerca de la entrada del poro. [106]
Métodos de secuenciación de lectura corta
Secuenciación de firmas masivamente paralela (MPSS)
La primera de las tecnologías de secuenciación de alto rendimiento, la secuenciación masiva de firmas paralelas (o MPSS, también llamada secuenciación de próxima generación), se desarrolló en la década de 1990 en Lynx Therapeutics, una empresa fundada en 1992 por Sydney Brenner y Sam Eletr . La MPSS era un método basado en perlas que utilizaba un enfoque complejo de ligadura de adaptadores seguida de decodificación de adaptadores, leyendo la secuencia en incrementos de cuatro nucleótidos. Este método lo hacía susceptible al sesgo específico de la secuencia o la pérdida de secuencias específicas. Debido a que la tecnología era tan compleja, la MPSS solo se realizaba "internamente" por Lynx Therapeutics y no se vendían máquinas de secuenciación de ADN a laboratorios independientes. Lynx Therapeutics se fusionó con Solexa (posteriormente adquirida por Illumina ) en 2004, lo que llevó al desarrollo de la secuenciación por síntesis, un enfoque más simple adquirido de Manteia Predictive Medicine , que dejó obsoleto a la MPSS. Sin embargo, las propiedades esenciales de los datos de salida del MPSS eran típicas de los tipos de datos de alto rendimiento posteriores, incluidos cientos de miles de secuencias cortas de ADN. En el caso del MPSS, estos se utilizaban normalmente para secuenciar ADNc para mediciones de los niveles de expresión génica . [60]
Secuenciación de polonia
El método de secuenciación polonía , desarrollado en el laboratorio de George M. Church en Harvard, fue uno de los primeros sistemas de secuenciación de alto rendimiento y se utilizó para secuenciar un genoma completo de E. coli en 2005. [107] Combinó una biblioteca de etiquetas pareadas in vitro con PCR en emulsión, un microscopio automatizado y química de secuenciación basada en ligadura para secuenciar un genoma de E. coli con una precisión de >99,9999% y un costo de aproximadamente 1/9 del de la secuenciación de Sanger. [107] La tecnología fue licenciada a Agencourt Biosciences, posteriormente se escindió en Agencourt Personal Genomics y finalmente se incorporó a la plataforma SOLiD de Applied Biosystems . Applied Biosystems fue adquirida más tarde por Life Technologies , ahora parte de Thermo Fisher Scientific .
454 pirosecuenciación
454 Life Sciences , que posteriormente fue adquirida por Roche Diagnostics , desarrolló una versión paralelizada de la pirosecuenciación . El método amplifica el ADN dentro de gotitas de agua en una solución de aceite (PCR en emulsión), y cada gotita contiene una única plantilla de ADN unida a una única perla recubierta de cebador que luego forma una colonia clonal. La máquina de secuenciación contiene muchos pocillos de volumen de picolitros, cada uno de los cuales contiene una única perla y enzimas de secuenciación. La pirosecuenciación utiliza luciferasa para generar luz para la detección de los nucleótidos individuales añadidos al ADN naciente, y los datos combinados se utilizan para generar lecturas de secuencias . [70] Esta tecnología proporciona una longitud de lectura intermedia y un precio por base en comparación con la secuenciación de Sanger en un extremo y Solexa y SOLiD en el otro. [79]
Secuenciación de Illumina (Solexa)
Solexa , ahora parte de Illumina , fue fundada por Shankar Balasubramanian y David Klenerman en 1998, y desarrolló un método de secuenciación basado en tecnología de terminadores de tinte reversibles y polimerasas diseñadas. [108] El concepto de química de terminación reversible fue inventado por Bruno Canard y Simon Sarfati en el Instituto Pasteur de París. [109] [110] Fue desarrollado internamente en Solexa por aquellos nombrados en las patentes relevantes. En 2004, Solexa adquirió la empresa Manteia Predictive Medicine para obtener una tecnología de secuenciación masiva paralela inventada en 1997 por Pascal Mayer y Laurent Farinelli. [57] Se basa en "grupos de ADN" o "colonias de ADN", que implica la amplificación clonal de ADN en una superficie. La tecnología de grupos fue adquirida en conjunto con Lynx Therapeutics de California. Solexa Ltd. luego se fusionó con Lynx para formar Solexa Inc.


En este método, las moléculas de ADN y los cebadores se adhieren primero a un portaobjetos o celda de flujo y se amplifican con polimerasa para que se formen colonias de ADN clonal local, más tarde denominadas "agrupaciones de ADN". Para determinar la secuencia, se añaden cuatro tipos de bases de terminación reversibles (RT-bases) y se eliminan los nucleótidos no incorporados. Una cámara toma imágenes de los nucleótidos marcados con fluorescencia . Luego, el colorante, junto con el bloqueador terminal 3', se elimina químicamente del ADN, lo que permite que comience el siguiente ciclo. A diferencia de la pirosecuenciación, las cadenas de ADN se extienden un nucleótido a la vez y la adquisición de imágenes se puede realizar en un momento diferido, lo que permite capturar conjuntos muy grandes de colonias de ADN mediante imágenes secuenciales tomadas con una sola cámara.

La separación de la reacción enzimática y la captura de imágenes permite un rendimiento óptimo y una capacidad de secuenciación teóricamente ilimitada. Con una configuración óptima, el rendimiento del instrumento que se puede alcanzar en última instancia está determinado únicamente por la tasa de conversión de analógico a digital de la cámara, multiplicada por el número de cámaras y dividida por el número de píxeles por colonia de ADN necesarios para visualizarlas de forma óptima (aproximadamente 10 píxeles/colonia). En 2012, con cámaras que funcionan a tasas de conversión A/D de más de 10 MHz y con la óptica, los fluidos y los enzimas disponibles, el rendimiento puede ser múltiplo de 1 millón de nucleótidos/segundo, lo que corresponde aproximadamente a un genoma humano equivalente a una cobertura de 1x por hora por instrumento, y a un genoma humano resecuenciado (a aproximadamente 30x) por día por instrumento (equipado con una sola cámara). [111]
Síntesis de anclaje de sonda combinatoria (cPAS)
Este método es una modificación mejorada de la tecnología de ligadura de anclaje de sonda combinatoria (cPAL) descrita por Complete Genomics [112], que desde entonces se ha convertido en parte de la empresa de genómica china BGI en 2013. [113] Las dos empresas han perfeccionado la tecnología para permitir longitudes de lectura más largas, reducciones del tiempo de reacción y tiempos más rápidos para obtener resultados. Además, los datos ahora se generan como lecturas completas contiguas en el formato de archivo FASTQ estándar y se pueden usar tal cual en la mayoría de los procesos de análisis bioinformáticos basados en lecturas cortas. [114] [ cita requerida ]
Las dos tecnologías que forman la base de esta tecnología de secuenciación de alto rendimiento son las nanobolas de ADN (DNB) y las matrices con patrones para la fijación de las nanobolas a una superficie sólida. [112] Las nanobolas de ADN se forman simplemente desnaturalizando bibliotecas de doble cadena ligadas con adaptadores y ligando la cadena delantera solo a un oligonucleótido de férula para formar un círculo de ssDNA. Se producen copias fieles de los círculos que contienen el inserto de ADN utilizando la amplificación por círculo rodante que genera aproximadamente entre 300 y 500 copias. La cadena larga de ssDNA se pliega sobre sí misma para producir una estructura de nanobolas tridimensional que tiene aproximadamente 220 nm de diámetro. La fabricación de DNB reemplaza la necesidad de generar copias de PCR de la biblioteca en la celda de flujo y, como tal, puede eliminar grandes proporciones de lecturas duplicadas, ligaduras de adaptador-adaptador y errores inducidos por PCR. [114] [ cita requerida ]

La matriz estampada de puntos cargados positivamente se fabrica mediante técnicas de fotolitografía y grabado seguidas de una modificación química para generar una celda de flujo de secuenciación. Cada punto en la celda de flujo tiene aproximadamente 250 nm de diámetro, están separados por 700 nm (de centro a centro) y permiten una fácil fijación de un solo DNB cargado negativamente a la celda de flujo y, por lo tanto, reducen la sobreagrupación o la subagrupación en la celda de flujo. [112] [ cita requerida ]
Luego se realiza la secuenciación mediante la adición de una sonda de oligonucleótidos que se adhiere en combinación a sitios específicos dentro del DNB. La sonda actúa como un ancla que luego permite que uno de los cuatro nucleótidos individuales inactivados reversiblemente y marcados se una después de fluir a través de la celda de flujo. Los nucleótidos no unidos se eliminan antes de la excitación láser de las etiquetas adheridas, luego emiten fluorescencia y la señal es capturada por cámaras que se convierte en una salida digital para la llamada de bases. La base adherida tiene su terminador y etiqueta escindida químicamente al completarse el ciclo. El ciclo se repite con otro flujo de nucleótidos libres y marcados a través de la celda de flujo para permitir que el siguiente nucleótido se una y tenga su señal capturada. Este proceso se completa varias veces (generalmente de 50 a 300 veces) para determinar la secuencia del fragmento de ADN insertado a una velocidad de aproximadamente 40 millones de nucleótidos por segundo a partir de 2018. [ cita requerida ]
Secuenciación SOLiD


La tecnología SOLiD de Applied Biosystems (ahora una marca de Life Technologies ) emplea la secuenciación por ligadura . Aquí, un grupo de todos los oligonucleótidos posibles de una longitud fija se etiquetan de acuerdo con la posición secuenciada. Los oligonucleótidos se hibridan y se ligan; la ligadura preferencial por la ADN ligasa para secuencias coincidentes da como resultado una señal informativa del nucleótido en esa posición. Cada base en la plantilla se secuencia dos veces y los datos resultantes se decodifican de acuerdo con el esquema de codificación de 2 bases utilizado en este método. Antes de la secuenciación, el ADN se amplifica por PCR en emulsión. Las perlas resultantes, cada una de las cuales contiene copias individuales de la misma molécula de ADN, se depositan en un portaobjetos de vidrio. [115] El resultado son secuencias de cantidades y longitudes comparables a la secuenciación de Illumina. [79] Se ha informado que este método de secuenciación por ligadura tiene algunos problemas para secuenciar secuencias palindrómicas. [98]
Secuenciación de semiconductores mediante Ion Torrent
Ion Torrent Systems Inc. (ahora propiedad de Life Technologies ) desarrolló un sistema basado en el uso de la química de secuenciación estándar, pero con un novedoso sistema de detección basado en semiconductores. Este método de secuenciación se basa en la detección de iones de hidrógeno que se liberan durante la polimerización del ADN , a diferencia de los métodos ópticos utilizados en otros sistemas de secuenciación. Un micropocillo que contiene una cadena de ADN plantilla que se va a secuenciar se inunda con un solo tipo de nucleótido . Si el nucleótido introducido es complementario al nucleótido plantilla principal, se incorpora a la cadena complementaria en crecimiento. Esto provoca la liberación de un ion de hidrógeno que activa un sensor de iones hipersensible, lo que indica que se ha producido una reacción. Si hay repeticiones de homopolímero presentes en la secuencia plantilla, se incorporarán múltiples nucleótidos en un solo ciclo. Esto conduce a un número correspondiente de hidrógenos liberados y una señal electrónica proporcionalmente mayor. [116]

Secuenciación de nanobolas de ADN
La secuenciación de nanobolas de ADN es un tipo de tecnología de secuenciación de alto rendimiento que se utiliza para determinar la secuencia genómica completa de un organismo. La empresa Complete Genomics utiliza esta tecnología para secuenciar muestras enviadas por investigadores independientes. El método utiliza la replicación por círculo rodante para amplificar pequeños fragmentos de ADN genómico en nanobolas de ADN. A continuación, se utiliza la secuenciación desencadenada por ligadura para determinar la secuencia de nucleótidos. [117] Este método de secuenciación de ADN permite secuenciar una gran cantidad de nanobolas de ADN por ejecución y con un bajo coste de reactivos en comparación con otras plataformas de secuenciación de alto rendimiento. [118] Sin embargo, solo se determinan secuencias cortas de ADN a partir de cada nanobola de ADN, lo que dificulta el mapeo de las lecturas cortas a un genoma de referencia . [117]
Secuenciación de moléculas individuales con heliscopio
La secuenciación por heliscopio es un método de secuenciación de moléculas individuales desarrollado por Helicos Biosciences . Utiliza fragmentos de ADN con adaptadores de cola de poli-A añadidos que se adhieren a la superficie de la celda de flujo. Los siguientes pasos implican una secuenciación basada en extensión con lavados cíclicos de la celda de flujo con nucleótidos marcados con fluorescencia (un tipo de nucleótido a la vez, como con el método de Sanger). Las lecturas se realizan mediante el secuenciador Heliscope. [119] [120] Las lecturas son cortas, con un promedio de 35 pb. [121] Lo que hizo que esta tecnología fuera especialmente novedosa fue que fue la primera de su clase en secuenciar ADN no amplificado, evitando así cualquier error de lectura asociado con los pasos de amplificación. [122] En 2009 se secuenció un genoma humano utilizando el Heliscopio, sin embargo, en 2012 la empresa se declaró en quiebra. [123]
Sistemas microfluídicos
Existen dos sistemas microfluídicos principales que se utilizan para secuenciar ADN: los microfluídicos basados en gotas y los microfluídicos digitales . Los dispositivos microfluídicos resuelven muchas de las limitaciones actuales de los sistemas de secuenciación actuales.
Abate et al. estudiaron el uso de dispositivos microfluídicos basados en gotitas para la secuenciación de ADN. [4] Estos dispositivos tienen la capacidad de formar y procesar gotitas del tamaño de picolitros a una velocidad de miles por segundo. Los dispositivos se crearon a partir de polidimetilsiloxano (PDMS) y utilizaron transferencia de energía por resonancia de Forster, ensayos FRET para leer las secuencias de ADN incluidas en las gotitas. Cada posición en la matriz se probó para una secuencia específica de 15 bases. [4]
Fair et al. utilizaron dispositivos microfluídicos digitales para estudiar la pirosecuenciación de ADN . [124] Las ventajas significativas incluyen la portabilidad del dispositivo, el volumen de reactivo, la velocidad de análisis, las capacidades de fabricación en masa y el alto rendimiento. Este estudio proporcionó una prueba de concepto que muestra que los dispositivos digitales se pueden utilizar para la pirosecuenciación; el estudio incluyó el uso de síntesis, que implica la extensión de las enzimas y la adición de nucleótidos marcados. [124]
Boles et al. también estudiaron la pirosecuenciación en dispositivos microfluídicos digitales. [125] Utilizaron un dispositivo de electrohumectación para crear, mezclar y dividir gotas. La secuenciación utiliza un protocolo de tres enzimas y plantillas de ADN ancladas con perlas magnéticas. El dispositivo se probó utilizando dos protocolos y dio como resultado una precisión del 100 % en función de los niveles de pirogramas sin procesar. Las ventajas de estos dispositivos microfluídicos digitales incluyen el tamaño, el costo y los niveles alcanzables de integración funcional. [125]
La investigación de secuenciación de ADN, utilizando microfluidos, también tiene la capacidad de aplicarse a la secuenciación de ARN , utilizando técnicas de microfluidos de gotas similares, como el método inDrops. [126] Esto demuestra que muchas de estas técnicas de secuenciación de ADN podrán aplicarse más y usarse para comprender más sobre los genomas y transcriptomas.
Métodos en desarrollo
Los métodos de secuenciación de ADN actualmente en desarrollo incluyen la lectura de la secuencia a medida que una cadena de ADN transita a través de nanoporos (un método que ahora es comercial pero las generaciones posteriores, como los nanoporos de estado sólido, aún están en desarrollo), [127] [128] y técnicas basadas en microscopía, como la microscopía de fuerza atómica o la microscopía electrónica de transmisión que se utilizan para identificar las posiciones de nucleótidos individuales dentro de fragmentos de ADN largos (> 5000 pb) mediante el etiquetado de nucleótidos con elementos más pesados (por ejemplo, halógenos) para la detección y el registro visual. [129] [130] Las tecnologías de tercera generación tienen como objetivo aumentar el rendimiento y disminuir el tiempo para obtener resultados y el costo eliminando la necesidad de reactivos excesivos y aprovechando la procesividad de la ADN polimerasa. [131]
Secuenciación de ADN mediante corrientes de tunelización
Otro método utiliza mediciones de las corrientes de tunelización eléctrica a través del ADN monocatenario a medida que se mueve a través de un canal. Dependiendo de su estructura electrónica, cada base afecta la corriente de tunelización de manera diferente, [132] lo que permite la diferenciación entre diferentes bases. [133]
El uso de corrientes de tunelización tiene el potencial de secuenciar órdenes de magnitud más rápido que los métodos de corriente iónica y ya se ha logrado la secuenciación de varios oligómeros de ADN y micro-ARN. [134]
Secuenciación por hibridación
La secuenciación por hibridación es un método no enzimático que utiliza una micromatriz de ADN . Un único grupo de ADN cuya secuencia se va a determinar se marca con fluorescencia y se hibrida con una matriz que contiene secuencias conocidas. Las señales de hibridación intensas de un punto determinado de la matriz identifican su secuencia en el ADN que se está secuenciando. [135]
Este método de secuenciación utiliza las características de unión de una biblioteca de moléculas cortas de ADN monocatenario (oligonucleótidos), también llamadas sondas de ADN, para reconstruir una secuencia de ADN objetivo. Los híbridos no específicos se eliminan mediante lavado y el ADN objetivo se eluye. [136] Los híbridos se reorganizan de manera que se pueda reconstruir la secuencia de ADN. El beneficio de este tipo de secuenciación es su capacidad para capturar una gran cantidad de objetivos con una cobertura homogénea. [137] Por lo general, se requiere una gran cantidad de productos químicos y ADN de partida. Sin embargo, con el advenimiento de la hibridación basada en solución, se necesitan muchos menos equipos y productos químicos. [136]
Secuenciación con espectrometría de masas
La espectrometría de masas se puede utilizar para determinar secuencias de ADN. La espectrometría de masas con desorción láser asistida por matriz y tiempo de vuelo, o MALDI-TOF MS , se ha investigado específicamente como un método alternativo a la electroforesis en gel para visualizar fragmentos de ADN. Con este método, los fragmentos de ADN generados por reacciones de secuenciación de terminación de cadena se comparan por masa en lugar de por tamaño. La masa de cada nucleótido es diferente de los demás y esta diferencia es detectable por espectrometría de masas. Las mutaciones de un solo nucleótido en un fragmento se pueden detectar más fácilmente con MS que con electroforesis en gel sola. MALDI-TOF MS puede detectar más fácilmente las diferencias entre los fragmentos de ARN, por lo que los investigadores pueden secuenciar indirectamente el ADN con métodos basados en MS convirtiéndolo primero en ARN. [138]
La mayor resolución de los fragmentos de ADN que permiten los métodos basados en espectrometría de masas es de especial interés para los investigadores en ciencias forenses, ya que pueden desear encontrar polimorfismos de un solo nucleótido en muestras de ADN humano para identificar individuos. Estas muestras pueden estar muy degradadas, por lo que los investigadores forenses a menudo prefieren el ADN mitocondrial por su mayor estabilidad y sus aplicaciones para estudios de linaje. Los métodos de secuenciación basados en espectrometría de masas se han utilizado para comparar las secuencias de ADN mitocondrial humano de muestras de una base de datos de la Oficina Federal de Investigaciones [139] y de huesos encontrados en fosas comunes de soldados de la Primera Guerra Mundial. [140]
Los primeros métodos de terminación de cadena y espectrometría de masas TOF demostraron longitudes de lectura de hasta 100 pares de bases. [141] Los investigadores no han podido superar este tamaño de lectura promedio; al igual que la secuenciación de terminación de cadena por sí sola, la secuenciación de ADN basada en espectrometría de masas puede no ser adecuada para grandes proyectos de secuenciación de novo . Aun así, un estudio reciente utilizó lecturas de secuencia cortas y espectroscopia de masas para comparar polimorfismos de un solo nucleótido en cepas patógenas de Streptococcus . [142]
Secuenciación microfluídica de Sanger
En la secuenciación microfluídica de Sanger, toda la amplificación por termociclado de fragmentos de ADN, así como su separación por electroforesis, se realiza en una única oblea de vidrio (de aproximadamente 10 cm de diámetro), lo que reduce el uso de reactivos y el coste. [143] En algunos casos, los investigadores han demostrado que pueden aumentar el rendimiento de la secuenciación convencional mediante el uso de microchips. [144] Todavía será necesario realizar investigaciones para que este uso de la tecnología sea eficaz.
Técnicas basadas en microscopía
Este enfoque permite visualizar directamente la secuencia de moléculas de ADN mediante microscopía electrónica. Se ha demostrado la primera identificación de pares de bases de ADN dentro de moléculas de ADN intactas mediante la incorporación enzimática de bases modificadas, que contienen átomos de mayor número atómico, la visualización directa y la identificación de bases marcadas individualmente dentro de una molécula de ADN sintética de 3272 pares de bases y un genoma viral de 7249 pares de bases. [145]
Secuenciación de ARN polimerasa
Este método se basa en el uso de la ARN polimerasa (RNAP), que se une a una perla de poliestireno . Un extremo del ADN que se va a secuenciar se une a otra perla, y ambas perlas se colocan en trampas ópticas. El movimiento de la ARN polimerasa durante la transcripción acerca las perlas y su distancia relativa cambia, lo que luego se puede registrar con una resolución de un solo nucleótido. La secuencia se deduce en función de las cuatro lecturas con concentraciones reducidas de cada uno de los cuatro tipos de nucleótidos, de manera similar al método de Sanger. [146] Se realiza una comparación entre regiones y se deduce la información de la secuencia comparando las regiones de secuencia conocidas con las regiones de secuencia desconocidas. [147]
In vitrosecuenciación de alto rendimiento del virus
Se ha desarrollado un método para analizar conjuntos completos de interacciones de proteínas utilizando una combinación de pirosecuenciación 454 y un método de visualización de ARNm de virus in vitro . Específicamente, este método une covalentemente las proteínas de interés a los ARNm que las codifican, luego detecta los fragmentos de ARNm utilizando PCR de transcripción inversa . Luego, el ARNm puede amplificarse y secuenciarse. El método combinado se tituló IVV-HiTSeq y puede realizarse en condiciones libres de células, aunque sus resultados pueden no ser representativos de las condiciones in vivo . [148]
Cuota de mercado
Si bien existen muchas formas diferentes de secuenciar ADN, solo unas pocas dominan el mercado. En 2022, Illumina tenía alrededor del 80 % del mercado; el resto del mercado está en manos de solo unos pocos actores (PacBio, Oxford, 454, MGI) [149]
Preparación de muestras
El éxito de cualquier protocolo de secuenciación de ADN depende de la extracción y preparación de la muestra de ADN o ARN del material biológico de interés.
- Una extracción de ADN exitosa producirá una muestra de ADN con cadenas largas y no degradadas.
- Una extracción de ARN exitosa producirá una muestra de ARN que debe convertirse en ADN complementario (ADNc) utilizando la transcriptasa inversa, una ADN polimerasa que sintetiza un ADN complementario basado en cadenas de ARN existentes de una manera similar a la PCR. [150] El ADN complementario puede luego procesarse de la misma manera que el ADN genómico.
Después de la extracción de ADN o ARN, las muestras pueden requerir una preparación adicional según el método de secuenciación. Para la secuenciación de Sanger, se requieren procedimientos de clonación o PCR antes de la secuenciación. En el caso de los métodos de secuenciación de próxima generación, se requiere la preparación de la biblioteca antes del procesamiento. [151] La evaluación de la calidad y la cantidad de ácidos nucleicos tanto después de la extracción como después de la preparación de la biblioteca identifica muestras degradadas, fragmentadas y de baja pureza y produce datos de secuenciación de alta calidad. [152]
Iniciativas de desarrollo

En octubre de 2006, la Fundación Premio X estableció una iniciativa para promover el desarrollo de tecnologías de secuenciación completa del genoma , llamada Premio Archon X , con la intención de otorgar 10 millones de dólares al "primer equipo que pueda construir un dispositivo y usarlo para secuenciar 100 genomas humanos en 10 días o menos, con una precisión de no más de un error en cada 100.000 bases secuenciadas, con secuencias que cubran con precisión al menos el 98% del genoma, y a un costo recurrente de no más de 10.000 dólares (estadounidenses) por genoma". [153]
Cada año, el Instituto Nacional de Investigación del Genoma Humano , o NHGRI, promueve subvenciones para nuevas investigaciones y desarrollos en genómica . Las subvenciones de 2010 y los candidatos de 2011 incluyen trabajos continuos en metodologías de secuenciación microfluídica, polonía y de bases pesadas. [154]
Desafíos computacionales
Las tecnologías de secuenciación descritas aquí producen datos brutos que deben ensamblarse en secuencias más largas, como genomas completos ( ensamblaje de secuencias ). Existen muchos desafíos computacionales para lograr esto, como la evaluación de los datos de secuencias brutos que se realiza mediante programas y algoritmos como Phred y Phrap . Otros desafíos tienen que ver con secuencias repetitivas que a menudo impiden ensamblajes completos del genoma porque ocurren en muchos lugares del genoma. Como consecuencia, muchas secuencias pueden no asignarse a cromosomas particulares . La producción de datos de secuencias brutos es solo el comienzo de su análisis bioinformático detallado. [155] Sin embargo, se desarrollaron nuevos métodos para secuenciar y corregir errores de secuenciación. [156]
Leer recorte
A veces, las lecturas brutas producidas por el secuenciador son correctas y precisas solo en una fracción de su longitud. El uso de la lectura completa puede introducir artefactos en los análisis posteriores, como el ensamblaje del genoma, la identificación de SNP o la estimación de la expresión génica. Se han introducido dos clases de programas de recorte, basados en las clases de algoritmos basados en ventanas o de suma continua. [157] Esta es una lista parcial de los algoritmos de recorte disponibles actualmente, que especifica la clase de algoritmo a la que pertenecen:
| Nombre del algoritmo | Tipo de algoritmo |
|---|---|
| Adaptación de corte [158] | Suma corriente |
| ConDeTri [159] | Basado en ventana |
| FILTRO ERNE [160] | Suma corriente |
| Recortadora de calidad FASTX | Basado en ventana |
| PRINCIPIO [161] | Basado en ventana |
| Trimmomático [162] | Basado en ventana |
| SolexaQA [163] | Basado en ventana |
| Solexa QA-BWA | Suma corriente |
| Hoz | Basado en ventana |
Cuestiones éticas
![[icono]](http://upload.wikimedia.org/wikipedia/commons/thumb/1/1c/Wiki_letter_w_cropped.svg/20px-Wiki_letter_w_cropped.svg.png) | Esta sección necesita ser ampliada . Puedes ayudar agregándole algo. ( Mayo 2015 ) |
La genética humana se ha incluido en el campo de la bioética desde principios de los años 1970 [164] y el crecimiento en el uso de la secuenciación de ADN (en particular la secuenciación de alto rendimiento) ha introducido una serie de cuestiones éticas. Una cuestión clave es la propiedad del ADN de un individuo y los datos producidos cuando se secuencia ese ADN. [165] En cuanto a la molécula de ADN en sí, el caso legal principal sobre este tema, Moore v. Regents of the University of California (1990) dictaminó que los individuos no tienen derechos de propiedad sobre las células descartadas ni sobre los beneficios obtenidos utilizando estas células (por ejemplo, como una línea celular patentada ). Sin embargo, los individuos tienen derecho al consentimiento informado sobre la extracción y el uso de células. En cuanto a los datos producidos a través de la secuenciación de ADN, Moore no otorga al individuo ningún derecho sobre la información derivada de su ADN. [165]
A medida que la secuenciación de ADN se vuelve más generalizada, el almacenamiento, la seguridad y el intercambio de datos genómicos también se han vuelto más importantes. [165] [166] Por ejemplo, una preocupación es que las aseguradoras pueden usar los datos genómicos de un individuo para modificar su cotización, dependiendo de la salud futura percibida del individuo en función de su ADN. [166] [167] En mayo de 2008, se firmó la Ley de No Discriminación por Información Genética (GINA) en los Estados Unidos, que prohíbe la discriminación sobre la base de la información genética con respecto al seguro de salud y el empleo. [168] [169] En 2012, la Comisión Presidencial de los Estados Unidos para el Estudio de Asuntos Bioéticos informó que la legislación existente sobre privacidad para los datos de secuenciación de ADN, como GINA y la Ley de Portabilidad y Responsabilidad del Seguro Médico, era insuficiente, y señaló que los datos de secuenciación del genoma completo eran particularmente sensibles, ya que podían usarse para identificar no solo al individuo del que se crearon los datos, sino también a sus familiares. [170] [171]
En la mayor parte de los Estados Unidos, el ADN "abandonado", como el que se encuentra en un sello o sobre lamido, una taza de café, un cigarrillo, un chicle, la basura doméstica o el cabello caído en una acera pública, puede ser recolectado y secuenciado legalmente por cualquier persona, incluida la policía, investigadores privados, oponentes políticos o personas involucradas en disputas de paternidad. A partir de 2013, once estados tienen leyes que pueden interpretarse como que prohíben el "robo de ADN". [172]
También se han planteado cuestiones éticas por el uso cada vez mayor de pruebas de detección de variaciones genéticas, tanto en recién nacidos como en adultos, por parte de empresas como 23andMe . [173] [174] Se ha afirmado que las pruebas de detección de variaciones genéticas pueden ser perjudiciales, aumentando la ansiedad en individuos que se ha descubierto que tienen un mayor riesgo de enfermedad. [175] Por ejemplo, en un caso señalado en Time , los médicos que examinaban a un bebé enfermo en busca de variantes genéticas decidieron no informar a los padres de una variante no relacionada vinculada a la demencia debido al daño que causaría a los padres. [176] Sin embargo, un estudio de 2011 en The New England Journal of Medicine ha demostrado que las personas que se sometieron a un perfil de riesgo de enfermedad no mostraron mayores niveles de ansiedad. [175] Además, el desarrollo de tecnologías de secuenciación de próxima generación, como la secuenciación basada en nanoporos, también ha suscitado más preocupaciones éticas. [177]
Véase también
- Bioinformática : análisis computacional de conjuntos grandes y complejos de datos biológicos
- Secuenciación del genoma del cáncer
- Secuenciación de consenso circular
- Computación de ADN : computación con hardware de biología molecular
- Transistor de efecto de campo de ADN : transistor que utiliza el efecto de campo debido a las cargas parciales del ADN.Páginas que muestran descripciones de wikidata como alternativa
- Teoría de la secuenciación del ADN – Teoría biológica
- Secuenciador de ADN : instrumento científico utilizado para automatizar el proceso de secuenciación de ADN.
- Proyecto Genográfico – Proyecto de ciencia ciudadana
- Proyecto Genoma – Iniciativas científicas para determinar la secuencia completa del genoma de un organismo
- Secuenciación del genoma de especies en peligro de extinción : pruebas de ADN para evaluar su peligro de extinción
- Desnatado del genoma : método de secuenciación del genoma
- IsoBase : proteínas relacionadas funcionalmente en redes PPIPáginas que muestran descripciones de wikidata como alternativa
- Secuenciación de lectura enlazada
- Biblioteca de saltos
- Secuencia de ácidos nucleicos – Sucesión de nucleótidos en un ácido nucleico
- Amplificación de sonda dependiente de ligación multiplex
- Medicina personalizada : modelo médico que adapta las prácticas médicas al paciente individual.
- Secuenciación de proteínas : secuenciación de la disposición de aminoácidos en una proteína
- Minería de secuencias
- Herramienta de creación de perfiles de secuencias
- Secuenciación por hibridación : método para determinar los nucleótidos constituyentes de un tamaño fijo en una cadena de ADN.Páginas que muestran descripciones de wikidata como alternativa
- Secuenciación por ligadura : método de secuenciación de ADN que utiliza la enzima ADN ligasa para identificar el nucleótido presente en una posición determinada en una secuencia de ADN.Páginas que muestran descripciones de wikidata como alternativa
- TIARA (base de datos) – Base de datos de información genómica personal
- Secuenciación de ADN mediante microscopía electrónica de transmisión : tecnología de secuenciación de moléculas individuales
Notas
- ^ "La próxima generación" sigue siendo un término de uso generalizado en 2019. Por ejemplo, Straiton J, Free T, Sawyer A, Martin J (febrero de 2019). "From Sanger Sequencing to Genome Databases and Beyond". BioTechniques . 66 (2): 60–63. doi : 10.2144/btn-2019-0011 . PMID 30744413.
Las tecnologías de secuenciación de próxima generación (NGS) han revolucionado la investigación genómica. (frase inicial del artículo)
Referencias
- ^ "Presentamos el 'ADN oscuro': el fenómeno que podría cambiar nuestra forma de pensar sobre la evolución". 24 de agosto de 2017.
- ^ Behjati S, Tarpey PS (diciembre de 2013). "¿Qué es la secuenciación de próxima generación?". Archivos de enfermedades en la infancia: Edición de educación y práctica . 98 (6): 236–8. doi :10.1136/archdischild-2013-304340. PMC 3841808. PMID 23986538 .
- ^ Chmielecki J, Meyerson M (14 de enero de 2014). "Secuenciación de ADN del cáncer: ¿qué hemos aprendido?". Revista Anual de Medicina . 65 (1): 63–79. doi : 10.1146/annurev-med-060712-200152 . PMID 24274178.
- ^ abcd Abate AR, Hung T, Sperling RA, Mary P, Rotem A, Agresti JJ, et al. (diciembre de 2013). "Análisis de secuencia de ADN con microfluídica basada en gotas". Lab on a Chip . 13 (24): 4864–9. doi :10.1039/c3lc50905b. PMC 4090915 . PMID 24185402.
- ^ Pekin D, Skhiri Y, Baret JC, Le Corre D, Mazutis L, Salem CB, et al. (julio de 2011). "Detección cuantitativa y sensible de mutaciones raras mediante microfluídica basada en gotas". Lab on a Chip . 11 (13): 2156–66. doi :10.1039/c1lc20128j. PMID 21594292.
- ^ Olsvik O, Wahlberg J, Petterson B, Uhlén M, Popovic T, Wachsmuth IK, Fields PI (enero de 1993). "Uso de secuenciación automatizada de amplicones generados por reacción en cadena de la polimerasa para identificar tres tipos de subunidad B de la toxina del cólera en cepas O1 de Vibrio cholerae". J. Clin. Microbiol. 31 (1): 22–25. doi :10.1128/JCM.31.1.22-25.1993. PMC 262614 . PMID 7678018.

- ^ Pettersson E, Lundeberg J, Ahmadian A (febrero de 2009). "Generaciones de tecnologías de secuenciación". Genomics . 93 (2): 105–11. doi : 10.1016/j.ygeno.2008.10.003 . PMID 18992322.
- ^ abc Jay E, Bambara R, Padmanabhan R, Wu R (marzo de 1974). "Análisis de secuencia de ADN: un método general, simple y rápido para secuenciar fragmentos grandes de oligodesoxirribonucleótidos mediante mapeo". Nucleic Acids Research . 1 (3): 331–53. doi :10.1093/nar/1.3.331. PMC 344020 . PMID 10793670.
- ^ Hunt, Katie (17 de febrero de 2021). «Secuenciaron el ADN más antiguo del mundo de un mamut que vivió hace más de un millón de años». CNN . Consultado el 17 de febrero de 2021 .
- ^ Callaway, Ewen (17 de febrero de 2021). «Los genomas de mamut de un millón de años rompen el récord del ADN antiguo más antiguo: los dientes preservados en el permafrost, de hasta 1,6 millones de años, identifican un nuevo tipo de mamut en Siberia». Nature . 590 (7847): 537–538. Bibcode :2021Natur.590..537C. doi : 10.1038/d41586-021-00436-x . PMID 33597786.
- ^ abc Castro, Christina; Marine, Rachel; Ramos, Edward; Ng, Terry Fei Fan (2019). "El efecto de la interferencia de variantes en el ensamblaje de novo para la secuenciación profunda viral". BMC Genomics . 21 (1): 421. bioRxiv 10.1101/815480 . doi : 10.1186/s12864-020-06801-w . PMC 7306937 . PMID 32571214.
- ^ ab Wohl, Shirlee; Schaffner, Stephen F.; Sabeti, Pardis C. (2016). "Análisis genómico de brotes virales". Revisión anual de virología . 3 (1): 173–195. doi :10.1146/annurev-virology-110615-035747. PMC 5210220 . PMID 27501264.
- ^ Boycott, Kym M.; Vanstone, Megan R.; Bulman, Dennis E.; MacKenzie, Alex E. (octubre de 2013). "Genética de enfermedades raras en la era de la secuenciación de próxima generación: del descubrimiento a la traducción". Nature Reviews Genetics . 14 (10): 681–691. doi :10.1038/nrg3555. PMID 23999272. S2CID 8496181.
- ^ Bean, Lora; Funke, Birgit; Carlston, Colleen M.; Gannon, Jennifer L.; Kantarci, Sibel; Krock, Bryan L.; Zhang, Shulin; Bayrak-Toydemir, Pinar (marzo de 2020). "Paneles de secuenciación genética diagnóstica: del diseño al informe: un estándar técnico del Colegio Americano de Genética Médica y Genómica (ACMG)". Genética en Medicina . 22 (3): 453–461. doi : 10.1038/s41436-019-0666-z . ISSN 1098-3600. PMID 31732716.
- ^ Schleusener V, Köser CU, Beckert P, Niemann S, Feuerriegel S (2017). "Predicción de la resistencia de Mycobacterium tuberculosis y clasificación de linaje a partir de la secuenciación del genoma: comparación de herramientas de análisis automatizadas". Sci Rep . 7 : 46327. Bibcode :2017NatSR...746327S. doi : 10.1038/srep46327 . PMC 7365310 . PMID 28425484.
- ^ Mahé P, El Azami M, Barlas P, Tournoud M (2019). "Una evaluación a gran escala de TBProfiler y Mykrobe para la predicción de la resistencia a los antibióticos en Mycobacterium tuberculosis". PeerJ . 7 : e6857. doi : 10.7717/peerj.6857 . PMC 6500375 . PMID 31106066.
- ^ Predictor Mykrobe: predicción de la resistencia a los antibióticos para S. aureus y M. tuberculosis a partir de datos de secuenciación del genoma completo
- ^ Bradley, Phelim; Gordon, N. Claire; Walker, Timothy M.; Dunn, Laura; Heys, Simon; Huang, Bill; Earle, Sarah; Pankhurst, Louise J.; Anson, Luke; de Cesare, Mariateresa; Piazza, Paolo; Votintseva, Antonina A.; Golubchik, Tanya; Wilson, Daniel J.; Wyllie, David H.; Diel, Roland; Niemann, Stefan; Feuerriegel, Silke; Kohl, Thomas A.; Ismail, Nazir; Omar, Shaheed V.; Smith, E. Grace; Buck, David; McVean, Gil; Walker, A. Sarah; Peto, Tim EA; Crook, Derrick W.; Iqbal, Zamin (21 de diciembre de 2015). "Predicciones rápidas de resistencia a antibióticos a partir de datos de secuencias genómicas de Staphylococcus aureus y Mycobacterium tuberculosis". Nature Communications . 6 (1): 10063. Código Bibliográfico :2015NatCo...610063B. doi :10.1038/ncomms10063. PMC 4703848 . PMID 26686880.
- ^ "Michael Mosley contra las superbacterias". Archivado desde el original el 24 de noviembre de 2020. Consultado el 21 de octubre de 2019 .
- ^ Mykrobe, Mykrobe-tools, 24 de diciembre de 2022 , consultado el 2 de enero de 2023
- ^ Curtis C, Hereward J (29 de agosto de 2017). "De la escena del crimen a la sala del tribunal: el viaje de una muestra de ADN". The Conversation .
- ^ Moréra S, Larivière L, Kurzeck J, Aschke-Sonnenborn U, Freemont PS, Janin J, Rüger W (agosto de 2001). "Estructuras cristalinas de alta resolución de la beta-glucosiltransferasa del fago T4: ajuste inducido y efecto de la unión al sustrato y al metal". Journal of Molecular Biology . 311 (3): 569–77. doi :10.1006/jmbi.2001.4905. PMID 11493010.
- ^ Ehrlich M, Gama-Sosa MA, Huang LH, Midgett RM, Kuo KC, McCune RA, Gehrke C (abril de 1982). "Cantidad y distribución de 5-metilcitosina en el ADN humano de diferentes tipos de tejidos de células". Nucleic Acids Research . 10 (8): 2709–21. doi :10.1093/nar/10.8.2709. PMC 320645 . PMID 7079182.
- ^ Ehrlich M, Wang RY (junio de 1981). "5-Metilcitosina en el ADN eucariota". Science . 212 (4501): 1350–7. Bibcode :1981Sci...212.1350E. doi :10.1126/science.6262918. PMID 6262918.
- ^ Song CX, Clark TA, Lu XY, Kislyuk A, Dai Q, Turner SW, et al. (noviembre de 2011). "Secuenciación sensible y específica de una sola molécula de 5-hidroximetilcitosina". Nature Methods . 9 (1): 75–7. doi :10.1038/nmeth.1779. PMC 3646335 . PMID 22101853.
- ^ Czernecki, Dariusz; Bonhomme, Frédéric; Kaminski, Pierre-Alexandre; Delarue, Marc (5 de agosto de 2021). "Caracterización de una tríada de genes en el cianófago S-2L suficiente para reemplazar la adenina por 2-aminoadenina en el ADN bacteriano". Nature Communications . 12 (1): 4710. Bibcode :2021NatCo..12.4710C. doi : 10.1038/s41467-021-25064-x . PMC 8342488 . PMID 34354070. S2CID 233745192.
- ^ "Detección directa y secuenciación de bases de ADN dañadas". PacBio . Consultado el 31 de julio de 2024 .
- ^ Watson JD, Crick FH (1953). "La estructura del ADN". Cold Spring Harb. Symp. Quant. Biol . 18 : 123–31. doi :10.1101/SQB.1953.018.01.020. PMID 13168976.
- ^ Marks, L. "El camino hacia la secuenciación del ADN: la vida y la obra de Frederick Sanger". ¿Qué es la biotecnología? . Consultado el 27 de junio de 2023 .
- ^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (mayo de 1972). "Secuencia de nucleótidos del gen que codifica la proteína de la capa del bacteriófago MS2". Nature . 237 (5350): 82–8. Bibcode :1972Natur.237...82J. doi :10.1038/237082a0. PMID 4555447. S2CID 4153893.
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, Min Jou W, Molemans F, Raeymaekers A, Van den Berghe A, Volckaert G, Ysebaert M (abril de 1976). "Secuencia completa de nucleótidos del ARN del bacteriófago MS2: estructura primaria y secundaria del gen de la replicasa". Naturaleza . 260 (5551): 500–7. Código Bib :1976Natur.260..500F. doi :10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Ozsolak F, Milos PM (febrero de 2011). "Secuenciación de ARN: avances, desafíos y oportunidades". Nature Reviews Genetics . 12 (2): 87–98. doi :10.1038/nrg2934. PMC 3031867 . PMID 21191423.
- ^ "Perfil del profesor Ray Wu". Universidad de Cornell. Archivado desde el original el 4 de marzo de 2009.
- ^ Padmanabhan R, Jay E, Wu R (junio de 1974). "Síntesis química de un cebador y su uso en el análisis de secuencia del gen de la lisozima del bacteriófago T4". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 71 (6): 2510–4. Bibcode :1974PNAS...71.2510P. doi : 10.1073/pnas.71.6.2510 . PMC 388489 . PMID 4526223.
- ^ Onaga LA (junio de 2014). "Ray Wu como quinto negocio: demostración de la memoria colectiva en la historia de la secuenciación del ADN". Estudios de historia y filosofía de la ciencia . Parte C. 46 : 1–14. doi :10.1016/j.shpsc.2013.12.006. PMID 24565976.
- ^ Wu R (1972). "Análisis de la secuencia de nucleótidos del ADN". Nature New Biology . 236 (68): 198–200. doi :10.1038/newbio236198a0. PMID 4553110.
- ^ Padmanabhan R, Wu R (1972). "Análisis de secuencia de nucleótidos del ADN. IX. Uso de oligonucleótidos de secuencia definida como iniciadores en el análisis de secuencias de ADN". Biochem. Biophys. Res. Commun . 48 (5): 1295–302. doi :10.1016/0006-291X(72)90852-2. PMID 4560009.
- ^ Wu R, Tu CD, Padmanabhan R (1973). "Análisis de la secuencia de nucleótidos del ADN. XII. La síntesis química y el análisis de la secuencia de un dodecadeoxinucleótido que se une al gen de la endolisina del bacteriófago lambda". Biochem. Biophys. Res. Commun . 55 (4): 1092–99. doi :10.1016/S0006-291X(73)80007-5. PMID 4358929.
- ^ ab Sanger F, Nicklen S, Coulson AR (diciembre de 1977). "Secuenciación de ADN con inhibidores de terminación de cadena". Proc. Natl. Sci. EE. UU . . 74 (12): 5463–77. Bibcode :1977PNAS...74.5463S. doi : 10.1073/pnas.74.12.5463 . PMC 431765 . PMID 271968.
- ^ abc Maxam AM, Gilbert W (febrero de 1977). "Un nuevo método para secuenciar ADN". Proc. Natl. Sci. EE. UU . . 74 (2): 560–64. Bibcode :1977PNAS...74..560M. doi : 10.1073/pnas.74.2.560 . PMC 392330 . PMID 265521.
- ^ Gilbert, W. Secuenciación del ADN y estructura genética. Conferencia Nobel, 8 de diciembre de 1980.
- ^ Gilbert W, Maxam A (diciembre de 1973). "La secuencia de nucleótidos del operador lac". Proc. Natl. Sci. USA . 70 (12): 3581–84. Bibcode :1973PNAS...70.3581G. doi : 10.1073/pnas.70.12.3581 . PMC 427284 . PMID 4587255.
- ^ Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, Hutchison CA, Slocombe PM, Smith M (febrero de 1977). "Secuencia de nucleótidos del ADN del bacteriófago phi X174". Nature . 265 (5596): 687–95. Bibcode :1977Natur.265..687S. doi :10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ Marks, L. "La próxima frontera: los virus humanos". ¿Qué es la biotecnología? . Consultado el 27 de junio de 2023 .
- ^ Beck S, Pohl FM (1984). "Secuenciación de ADN con electroforesis de transferencia directa". EMBO J . 3 (12): 2905–09. doi :10.1002/j.1460-2075.1984.tb02230.x. PMC 557787 . PMID 6396083.
- ^ Patente de los Estados Unidos 4.631.122 (1986)
- ^ Feldmann H, et al. (1994). "Secuencia completa de ADN del cromosoma II de la levadura". EMBO J . 13 (24): 5795–809. doi :10.1002/j.1460-2075.1994.tb06923.x. PMC 395553 . PMID 7813418.
- ^ Smith LM, Sanders JZ, Kaiser RJ, Hughes P, Dodd C, Connell CR, Heiner C, Kent SB, Hood LE (12 de junio de 1986). "Detección de fluorescencia en análisis automatizado de secuencias de ADN". Nature . 321 (6071): 674–79. Bibcode :1986Natur.321..674S. doi :10.1038/321674a0. PMID 3713851. S2CID 27800972.
- ^ Prober JM, Trainor GL, Dam RJ, Hobbs FW, Robertson CW, Zagursky RJ, Cocuzza AJ, Jensen MA, Baumeister K (16 de octubre de 1987). "Un sistema para la secuenciación rápida de ADN con didesoxinucleótidos fluorescentes que terminan la cadena". Science . 238 (4825): 336–41. Bibcode :1987Sci...238..336P. doi :10.1126/science.2443975. PMID 2443975.
- ^ Adams MD, Kelley JM, Gocayne JD, Dubnick M, Polymeropoulos MH, Xiao H, Merril CR, Wu A, Olde B, Moreno RF (junio de 1991). "Secuenciación complementaria de ADN: etiquetas de secuencia expresada y proyecto genoma humano". Science . 252 (5013): 1651–56. Bibcode :1991Sci...252.1651A. doi :10.1126/science.2047873. PMID 2047873. S2CID 13436211.
- ^ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, Bult CJ, Tomb JF, Dougherty BA, Merrick JM (julio de 1995). "Secuenciación aleatoria de todo el genoma y ensamblaje de Haemophilus influenzae Rd ". Science . 269 (5223): 496–512. Bibcode :1995Sci...269..496F. doi :10.1126/science.7542800. PMID 7542800.
- ^ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, et al. (febrero de 2001). "Secuenciación inicial y análisis del genoma humano" (PDF) . Nature . 409 (6822): 860–921. Bibcode :2001Natur.409..860L. doi : 10.1038/35057062 . PMID 11237011.
- ^ Venter JC, Adams MD, et al. (febrero de 2001). "La secuencia del genoma humano". Science . 291 (5507): 1304–51. Bibcode :2001Sci...291.1304V. doi : 10.1126/science.1058040 . PMID 11181995.
- ^ Yang, Aimin; Zhang, Wei; Wang, Jiahao; Yang, Ke; Han, Yang; Zhang, Limin (2020). "Revisión sobre la aplicación de algoritmos de aprendizaje automático en la minería de datos de secuencias de ADN". Frontiers in Bioengineering and Biotechnology . 8 : 1032. doi : 10.3389/fbioe.2020.01032 . PMC 7498545 . PMID 33015010.
- ^ "Espacenet - Datos bibliográficos". worldwide.espacenet.com .
- ^ Ronaghi M, Karamohamed S, Pettersson B, Uhlén M, Nyrén P (1996). "Secuenciación de ADN en tiempo real mediante la detección de la liberación de pirofosfato". Analytical Biochemistry . 242 (1): 84–89. doi :10.1006/abio.1996.0432. PMID 8923969.
- ^ ab Kawashima, Eric H.; Laurent Farinelli; Pascal Mayer (12 de mayo de 2005). «Patente: método de amplificación de ácidos nucleicos». Archivado desde el original el 22 de febrero de 2013. Consultado el 22 de diciembre de 2012 .
- ^ Ewing B, Green P (marzo de 1998). "Llamada de bases de trazas de secuenciadores automatizados utilizando phred. II. Probabilidades de error". Genome Res . 8 (3): 186–94. doi : 10.1101/gr.8.3.186 . PMID 9521922.
- ^ "Puntuaciones de calidad para la secuenciación de próxima generación" (PDF) . Illumina . 31 de octubre de 2011 . Consultado el 8 de mayo de 2018 .
- ^ ab Brenner S, Johnson M, Bridgham J, Golda G, Lloyd DH, Johnson D, Luo S, McCurdy S, Foy M, Ewan M, Roth R, George D, Eletr S, Albrecht G, Vermaas E, Williams SR, Moon K, Burcham T, Pallas M, DuBridge RB, Kirchner J, Fearon K, Mao J, Corcoran K (2000). "Análisis de expresión génica mediante secuenciación masiva de firmas paralelas (MPSS) en matrices de microesferas". Nature Biotechnology . 18 (6): 630–34. doi :10.1038/76469. PMID 10835600. S2CID 13884154.
- ^ "secuenciación de maxam gilbert". PubMed .
- ^ Sanger F, Coulson AR (mayo de 1975). "Un método rápido para determinar secuencias en ADN mediante síntesis cebada con ADN polimerasa". J. Mol. Biol . 94 (3): 441–48. doi :10.1016/0022-2836(75)90213-2. PMID 1100841.
- ^ Wetterstrand, Kris. "Costos de secuenciación de ADN: datos del Programa de secuenciación del genoma del NHGRI (GSP)". Instituto Nacional de Investigación del Genoma Humano . Consultado el 30 de mayo de 2013 .
- ^ Nyren, P.; Pettersson, B.; Uhlen, M. (enero de 1993). "Minisecuenciación de ADN en fase sólida mediante un ensayo enzimático luminométrico de detección de pirofosfato inorgánico". Analytical Biochemistry . 208 (1): 171–175. doi :10.1006/abio.1993.1024. PMID 8382019.
- ^ Ronaghi, Mostafa; Uhlén, Mathías; Nyrén, Pål (17 de julio de 1998). "Un método de secuenciación basado en pirofosfato en tiempo real". Ciencia . 281 (5375): 363–365. doi : 10.1126/ciencia.281.5375.363. PMID 9705713. S2CID 26331871.
- ^ Quail MA, Gu Y, Swerdlow H, Mayho M (2012). "Evaluación y optimización de sistemas de electroforesis semiautomatizada preparativa para la preparación de bibliotecas Illumina". Electroforesis . 33 (23): 3521–28. doi :10.1002/elps.201200128. PMID 23147856. S2CID 39818212.
- ^ Duhaime MB, Deng L, Poulos BT, Sullivan MB (2012). "Hacia la metagenómica cuantitativa de virus salvajes y otras muestras de ADN de concentración ultrabaja: una evaluación rigurosa y optimización del método de amplificación de enlaces". Environ. Microbiol . 14 (9): 2526–37. Bibcode :2012EnvMi..14.2526D. doi :10.1111/j.1462-2920.2012.02791.x. PMC 3466414 . PMID 22713159.
- ^ Peterson BK, Weber JN, Kay EH, Fisher HS, Hoekstra HE (2012). "Doble digestión RADseq: un método económico para el descubrimiento y genotipado de SNP de novo en especies modelo y no modelo". PLOS ONE . 7 (5): e37135. Bibcode :2012PLoSO...737135P. doi : 10.1371/journal.pone.0037135 . PMC 3365034 . PMID 22675423.
- ^ Williams R, Peisajovich SG, Miller OJ, Magdassi S, Tawfik DS, Griffiths AD (2006). "Amplificación de bibliotecas de genes complejos mediante PCR en emulsión". Nature Methods . 3 (7): 545–50. doi :10.1038/nmeth896. PMID 16791213. S2CID 27459628.
- ^ ab Margulies M, Egholm M, et al. (septiembre de 2005). "Secuenciación genómica en reactores de picolitros de alta densidad microfabricados abiertos". Nature . 437 (7057): 376–80. Bibcode :2005Natur.437..376M. doi :10.1038/nature03959. PMC 1464427 . PMID 16056220.
- ^ Shendure J, Porreca GJ, Reppas NB, Lin X, McCutcheon JP, Rosenbaum AM, Wang MD, Zhang K, Mitra RD, Church GM (2005). "Secuenciación de polonía multiplex precisa de un genoma bacteriano evolucionado". Science . 309 (5741): 1728–32. Bibcode :2005Sci...309.1728S. doi : 10.1126/science.1117389 . PMID 16081699. S2CID 11405973.
- ^ "Applied Biosystems – Archivo no encontrado (error 404)". 16 de mayo de 2008. Archivado desde el original el 16 de mayo de 2008.
- ^ Goodwin S, McPherson JD, McCombie WR (mayo de 2016). "Alcanzar la mayoría de edad: diez años de tecnologías de secuenciación de próxima generación". Nature Reviews Genetics . 17 (6): 333–51. doi :10.1038/nrg.2016.49. PMC 10373632 . PMID 27184599. S2CID 8295541.
- ^ Staden R (11 de junio de 1979). "Una estrategia de secuenciación de ADN empleando programas informáticos". Nucleic Acids Research . 6 (7): 2601–10. doi :10.1093/nar/6.7.2601. PMC 327874 . PMID 461197.
- ^ de Magalhães JP, Finch CE, Janssens G (2010). "Secuenciación de próxima generación en la investigación sobre el envejecimiento: aplicaciones emergentes, problemas, dificultades y posibles soluciones". Ageing Research Reviews . 9 (3): 315–23. doi :10.1016/j.arr.2009.10.006. PMC 2878865 . PMID 19900591.
- ^ Grada A (agosto de 2013). "Secuenciación de próxima generación: metodología y aplicación". J Invest Dermatol . 133 (8): 1–4. doi : 10.1038/jid.2013.248 . PMID 23856935.
- ^ Hall N (mayo de 2007). "Tecnologías de secuenciación avanzadas y su impacto más amplio en la microbiología". J. Exp. Biol. 210 (parte 9): 1518–25. doi : 10.1242/jeb.001370 . PMID: 17449817.

- ^ Church GM (enero de 2006). "Genomas para todos". Sci. Am. 294 (1): 46–54. Bibcode :2006SciAm.294a..46C. doi :10.1038/scientificamerican0106-46. PMID 16468433. S2CID 28769137. (se requiere suscripción)
- ^ abc Schuster SC (enero de 2008). "La secuenciación de próxima generación transforma la biología actual". Nat. Methods . 5 (1): 16–18. doi :10.1038/nmeth1156. PMID 18165802. S2CID 1465786.
- ^ Kalb, Gilbert; Moxley, Robert (1992). Computación masivamente paralela, óptica y neuronal en los Estados Unidos . IOS Press . ISBN 978-90-5199-097-3.[ página necesaria ]
- ^ ten Bosch JR, Grody WW (2008). "Mantenerse al día con la próxima generación". Revista de diagnóstico molecular . 10 (6): 484–92. doi :10.2353/jmoldx.2008.080027. PMC 2570630 . PMID 18832462.

- ^ Tucker T, Marra M, Friedman JM (2009). "Secuenciación masivamente paralela: el próximo gran avance en la medicina genética". The American Journal of Human Genetics . 85 (2): 142–54. doi :10.1016/j.ajhg.2009.06.022. PMC 2725244 . PMID 19679224.

- ^ ab Straiton J, Free T, Sawyer A, Martin J (febrero de 2019). "De la secuenciación de Sanger a las bases de datos genómicas y más allá". BioTechniques . 66 (2). Ciencia futura: 60–63. doi : 10.2144/btn-2019-0011 . PMID 30744413.
- ^ Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, Bertoni A, Swerdlow HP, Gu Y (1 de enero de 2012). "Una historia de tres plataformas de secuenciación de próxima generación: comparación de los secuenciadores Ion Torrent, Pacific Biosciences e illumina MiSeq". BMC Genomics . 13 (1): 341. doi : 10.1186/1471-2164-13-341 . PMC 3431227 . PMID 22827831.

- ^ Liu L, Li Y, Li S, Hu N, He Y, Pong R, Lin D, Lu L, Law M (1 de enero de 2012). "Comparación de sistemas de secuenciación de próxima generación". Revista de biomedicina y biotecnología . 2012 : 251364. doi : 10.1155/2012/251364 . PMC 3398667. PMID 22829749 .

- ^ abc "Nuevo software y polimerasa para el sistema Sequel que mejoran el rendimiento y la asequibilidad – PacBio". 7 de marzo de 2018.
- ^ "Tras un año de pruebas, dos de los primeros clientes de PacBio esperan un uso más rutinario del secuenciador RS en 2012". GenomeWeb. 10 de enero de 2012.( se requiere registro )
- ^ "Pacific Biosciences presenta una nueva química con longitudes de lectura más largas para detectar nuevas características en la secuencia de ADN y avanzar en los estudios genómicos de organismos grandes" (Comunicado de prensa). 2013.
- ^ Chin CS, Alexander DH, Marks P, Klammer AA, Drake J, Heiner C, Clum A, Copeland A, Huddleston J, Eichler EE, Turner SW, Korlach J (2013). "Ensamblajes de genomas microbianos terminados y no híbridos a partir de datos de secuenciación SMRT de lectura larga". Nat. Methods . 10 (6): 563–69. doi :10.1038/nmeth.2474. PMID 23644548. S2CID 205421576.
- ^ ab "Ensamblaje de novo del genoma bacteriano: ¿un problema resuelto?". 5 de julio de 2013.
- ^ Rasko DA, Webster DR, Sahl JW, Bashir A, Boisen N, Scheutz F, Paxinos EE, Sebra R, Chin CS, Iliopoulos D, Klammer A, Peluso P, Lee L, Kislyuk AO, Bullard J, Kasarskis A, Wang S, Eid J, Rank D, Redman JC, Steyert SR, Frimodt-Møller J, Struve C, Petersen AM, Krogfelt KA, Nataro JP, Schadt EE, Waldor MK (25 de agosto de 2011). "Orígenes de la cepa que causa un brote de síndrome urémico hemolítico en Alemania". N Inglés J Med . 365 (8): 709–17. doi :10.1056/NEJMoa1106920. PMC 3168948 . Número de modelo: PMID21793740.

- ^ Tran B, Brown AM, Bedard PL, Winquist E, Goss GD, Hotte SJ, Welch SA, Hirte HW, Zhang T, Stein LD , Ferretti V, Watt S, Jiao W, Ng K, Ghai S, Shaw P, Petrocelli T, Hudson TJ , Neel BG, Onetto N, Siu LL, McPherson JD, Kamel-Reid S, Dancey JE (1 de enero de 2012). "Viabilidad de la secuenciación de próxima generación en tiempo real de genes del cáncer vinculados a la respuesta a fármacos: resultados de un ensayo clínico". Int. J. Cancer . 132 (7): 1547–55. doi : 10.1002/ijc.27817 . PMID 22948899. S2CID 72705.(se requiere suscripción)
- ^ Murray IA, Clark TA, Morgan RD, Boitano M, Anton BP, Luong K, Fomenkov A, Turner SW, Korlach J, Roberts RJ (2 de octubre de 2012). "Los metilomas de seis bacterias". Nucleic Acids Research . 40 (22): 11450–62. doi :10.1093/nar/gks891. PMC 3526280 . PMID 23034806.
- ^ "Kit Chef Ion 520 y Ion 530 ExT – Thermo Fisher Scientific". thermofisher.com .
- ^ "Precisión bruta". Archivado desde el original el 30 de marzo de 2018. Consultado el 29 de marzo de 2018 .
- ^ van Vliet AH (1 de enero de 2010). "Secuenciación de próxima generación de transcriptomas microbianos: desafíos y oportunidades". FEMS Microbiology Letters . 302 (1): 1–7. doi : 10.1111/j.1574-6968.2009.01767.x . PMID 19735299.

- ^ "BGI y MGISEQ". en.mgitech.cn . Consultado el 5 de julio de 2018 .
- ^ ab Huang YF, Chen SC, Chiang YS, Chen TH, Chiu KP (2012). "La secuencia palindrómica impide el mecanismo de secuenciación por ligación". BMC Systems Biology . 6 (Supl 2): S10. doi : 10.1186/1752-0509-6-S2-S10 . PMC 3521181 . PMID 23281822.
- ^ Loose, Matthew; Rakyan, Vardhman; Holmes, Nadine; Payne, Alexander (3 de mayo de 2018). "Observación de ballenas con BulkVis: un visualizador gráfico para archivos fast5 masivos de Oxford Nanopore". bioRxiv 10.1101/312256 .
- ^ "Las ventas de PacBio comienzan a repuntar a medida que la empresa mejora sus productos". 12 de febrero de 2013.
- ^ "Bio-IT World". bio-itworld.com . Archivado desde el original el 29 de julio de 2020. Consultado el 16 de noviembre de 2015 .
- ^ "PacBio lanza un sistema de secuenciación de moléculas individuales de mayor rendimiento y menor costo". Octubre de 2015.
- ^ Clarke J, Wu HC, Jayasinghe L, Patel A, Reid S, Bayley H (abril de 2009). "Identificación de bases continua para secuenciación de ADN de nanoporos de moléculas individuales". Nature Nanotechnology . 4 (4): 265–70. Bibcode :2009NatNa...4..265C. doi :10.1038/nnano.2009.12. PMID 19350039.
- ^ ab dela Torre R, Larkin J, Singer A, Meller A (2012). "Fabricación y caracterización de matrices de nanoporos de estado sólido para secuenciación de ADN de alto rendimiento". Nanotecnología . 23 (38): 385308. Bibcode :2012Nanot..23L5308D. doi :10.1088/0957-4484/23/38/385308. PMC 3557807 . PMID 22948520.
- ^ ab Pathak B, Lofas H, Prasongkit J, Grigoriev A, Ahuja R, Scheicher RH (2012). "Electrodos de oro con nanoporos integrados y doblemente funcionalizados para una secuenciación rápida de ADN". Applied Physics Letters . 100 (2): 023701. Bibcode :2012ApPhL.100b3701P. doi :10.1063/1.3673335.
- ^ Korlach J, Marks PJ, Cicero RL, Gray JJ, Murphy DL, Roitman DB, Pham TT, Otto GA, Foquet M, Turner SW (2008). "Pasivación selectiva de aluminio para la inmovilización dirigida de moléculas individuales de ADN polimerasa en nanoestructuras de guía de onda de modo cero". Actas de la Academia Nacional de Ciencias . 105 (4): 1176–81. Bibcode :2008PNAS..105.1176K. doi : 10.1073/pnas.0710982105 . PMC 2234111 . PMID 18216253.
- ^ ab Shendure J, Porreca GJ, Reppas NB, Lin X, McCutcheon JP, Rosenbaum AM, Wang MD, Zhang K, Mitra RD, Church GM (9 de septiembre de 2005). "Secuenciación de polonía multiplex precisa de un genoma bacteriano evolucionado". Science . 309 (5741): 1728–32. Bibcode :2005Sci...309.1728S. doi : 10.1126/science.1117389 . PMID 16081699. S2CID 11405973.
- ^ Bentley DR, Balasubramanian S, et al. (2008). "Secuenciación precisa del genoma humano completo mediante química de terminación reversible". Nature . 456 (7218): 53–59. Bibcode :2008Natur.456...53B. doi :10.1038/nature07517. PMC 2581791 . PMID 18987734.
- ^ Canard B, Sarfati S (13 de octubre de 1994), Nuevos derivados utilizables para la secuenciación de ácidos nucleicos , consultado el 9 de marzo de 2016
- ^ Canard B, Sarfati RS (octubre de 1994). "Sustratos fluorescentes de la ADN polimerasa con etiquetas 3' reversibles". Gene . 148 (1): 1–6. doi :10.1016/0378-1119(94)90226-7. PMID 7523248.
- ^ Mardis ER (2008). "Métodos de secuenciación de ADN de próxima generación". Annu Rev Genom Hum Genet . 9 : 387–402. doi : 10.1146/annurev.genom.9.081307.164359. PMID 18576944.
- ^ abc Drmanac R, Sparks AB, Callow MJ, Halpern AL, Burns NL, Kermani BG, et al. (enero de 2010). "Secuenciación del genoma humano utilizando lecturas de bases no encadenadas en nanoarreglos de ADN autoensamblables". Science . 327 (5961): 78–81. Bibcode :2010Sci...327...78D. doi : 10.1126/science.1181498 . PMID 19892942. S2CID 17309571.
- ^ brandonvd. «Acerca de nosotros – Complete Genomics». Complete Genomics . Consultado el 2 de julio de 2018 .
- ^ ab Huang J, Liang X, Xuan Y, Geng C, Li Y, Lu H, et al. (mayo de 2017). "Un conjunto de datos de referencia del genoma humano del secuenciador BGISEQ-500". GigaScience . 6 (5): 1–9. doi :10.1093/gigascience/gix024. PMC 5467036 . PMID 28379488.
- ^ Valouev A, Ichikawa J, Tonthat T, Stuart J, Ranade S, Peckham H, Zeng K, Malek JA, Costa G, McKernan K, Sidow A, Fire A, Johnson SM (julio de 2008). "Un mapa de posición de nucleosomas de alta resolución de C. elegans revela la falta de un posicionamiento universal dictado por la secuencia". Genome Res . 18 (7): 1051–63. doi :10.1101/gr.076463.108. PMC 2493394 . PMID 18477713.
- ^ Rusk N (2011). "Torrentes de secuencia". Nat Methods . 8 (1): 44. doi : 10.1038/nmeth.f.330 . S2CID 41040192.
- ^ ab Drmanac R, Sparks AB, et al. (2010). "Secuenciación del genoma humano mediante lecturas de bases no encadenadas en nanoarreglos de ADN autoensamblables". Science . 327 (5961): 78–81. Bibcode :2010Sci...327...78D. doi : 10.1126/science.1181498 . PMID 19892942. S2CID 17309571.
- ^ Porreca GJ (2010). "Secuenciación del genoma en nanobolas". Biotecnología de la Naturaleza . 28 (1): 43–44. doi :10.1038/nbt0110-43. PMID 20062041. S2CID 54557996.
- ^ "Sistema de secuenciación genética / analizador genético HeliScope: Helicos BioSciences". 2 de noviembre de 2009. Archivado desde el original el 2 de noviembre de 2009.
- ^ Thompson JF, Steinmann KE (octubre de 2010). "Secuenciación de moléculas individuales con un sistema de análisis genético HeliScope". Protocolos actuales en biología molecular . Capítulo 7: Unidad 7.10. doi :10.1002/0471142727.mb0710s92. PMC 2954431. PMID 20890904 .
- ^ "Explicación técnica de tSMS SeqLL". SeqLL. Archivado desde el original el 8 de agosto de 2014. Consultado el 9 de agosto de 2015 .
- ^ Heather, James M.; Chain, Benjamin (enero de 2016). "La secuencia de secuenciadores: La historia de la secuenciación del ADN". Genómica . 107 (1): 1–8. doi :10.1016/j.ygeno.2015.11.003. PMC 4727787 . PMID 26554401.
- ^ Sara El-Mewally; Osama M. Ouda; Mohamed Helmy (2014). "Nuevos horizontes en la secuenciación de próxima generación". Tecnologías de secuenciación de próxima generación y desafíos en el ensamblaje de secuencias . SpringerBriefs in Systems Biology. Vol. 7. Tecnologías de secuenciación de próxima generación y desafíos en el ensamblaje de secuencias, Springer Briefs in Systems Biology Volumen 7. págs. 51–59. doi :10.1007/978-1-4939-0715-1_6. ISBN 978-1-4939-0714-4.
- ^ ab Fair RB, Khlystov A, Tailor TD, Ivanov V, Evans RD, Srinivasan V, Pamula VK, Pollack MG, Griffin PB, Zhou J (enero de 2007). "Aplicaciones químicas y biológicas de dispositivos microfluídicos digitales". IEEE Design & Test of Computers . 24 (1): 10–24. CiteSeerX 10.1.1.559.1440 . doi :10.1109/MDT.2007.8. hdl :10161/6987. S2CID 10122940.
- ^ ab Boles DJ, Benton JL, Siew GJ, Levy MH, Thwar PK, Sandahl MA, et al. (noviembre de 2011). "Pirosecuenciación basada en gotitas utilizando microfluídica digital". Química analítica . 83 (22): 8439–47. doi :10.1021/ac201416j. PMC 3690483 . PMID 21932784.
- ^ Zilionis R, Nainys J, Veres A, Savova V, Zemmour D, Klein AM, Mazutis L (enero de 2017). "Secuenciación y codificación de códigos de barras de células individuales mediante microfluidos de gotas". Nature Protocols . 12 (1): 44–73. doi :10.1038/nprot.2016.154. PMID 27929523. S2CID 767782.
- ^ "The Harvard Nanopore Group". Mcb.harvard.edu. Archivado desde el original el 21 de febrero de 2002. Consultado el 15 de noviembre de 2009 .
- ^ "La secuenciación de nanoporos podría reducir los costos del análisis de ADN".
- ^ Patente estadounidense 20060029957, ZS Genetics, "Sistemas y métodos de análisis de polímeros de ácidos nucleicos y componentes relacionados", expedida el 14 de julio de 2005
- ^ Xu M, Fujita D, Hanagata N (diciembre de 2009). "Perspectivas y desafíos de las tecnologías emergentes de secuenciación de ADN de moléculas individuales". Small . 5 (23): 2638–49. doi :10.1002/smll.200900976. PMID 19904762.
- ^ Schadt EE, Turner S, Kasarskis A (2010). "Una ventana a la secuenciación de tercera generación". Genética molecular humana . 19 (R2): R227–40. doi : 10.1093/hmg/ddq416 . PMID: 20858600.
- ^ Xu M, Endres RG, Arakawa Y (2007). "Las propiedades electrónicas de las bases del ADN". Small . 3 (9): 1539–43. doi : 10.1002/smll.200600732 . PMID 17786897.
- ^ Di Ventra M (2013). "La secuenciación rápida de ADN por medios eléctricos, cada vez más cerca". Nanotecnología . 24 (34): 342501. Bibcode :2013Nanot..24H2501D. doi :10.1088/0957-4484/24/34/342501. PMID 23899780. S2CID 140101884.
- ^ Ohshiro T, Matsubara K, Tsutsui M, Furuhashi M, Taniguchi M, Kawai T (2012). "Resecuenciación aleatoria eléctrica de una sola molécula de ADN y ARN". Representante de ciencia . 2 : 501. doi : 10.1038/srep00501. PMC 3392642 . PMID 22787559.
- ^ Hanna GJ, Johnson VA, Kuritzkes DR , Richman DD, Martinez-Picado J, Sutton L, Hazelwood JD, D'Aquila RT (1 de julio de 2000). "Comparación de la secuenciación por hibridación y secuenciación cíclica para la genotipificación de la transcriptasa inversa del virus de inmunodeficiencia humana tipo 1". J. Clin. Microbiol . 38 ( 7): 2715–21. doi :10.1128/JCM.38.7.2715-2721.2000. PMC 87006. PMID 10878069.
- ^ ab Morey M, Fernández-Marmiesse A, Castiñeiras D, Fraga JM, Couce ML, Cocho JA (2013). "Un vistazo a la secuenciación de ADN pasada, presente y futura". Genética molecular y metabolismo . 110 (1–2): 3–24. doi :10.1016/j.ymgme.2013.04.024. PMID 23742747.
- ^ Qin Y, Schneider TM, Brenner MP (2012). Gibas C (ed.). "Secuenciación por hibridación de objetivos largos". PLOS ONE . 7 (5): e35819. Bibcode :2012PLoSO...735819Q. doi : 10.1371/journal.pone.0035819 . PMC 3344849 . PMID 22574124.
- ^ Edwards JR, Ruparel H, Ju J (2005). "Secuenciación de ADN por espectrometría de masas". Mutation Research . 573 (1–2): 3–12. Bibcode :2005MRFMM.573....3E. doi :10.1016/j.mrfmmm.2004.07.021. PMID 15829234.
- ^ Hall TA, Budowle B, Jiang Y, Blyn L, Eshoo M, Sannes-Lowery KA, Sampath R, Drader JJ, Hannis JC, Harrell P, Samant V, White N, Ecker DJ, Hofstadler SA (2005). "Análisis de la composición de bases del ADN mitocondrial humano mediante espectrometría de masas de ionización por electrospray: una nueva herramienta para la identificación y diferenciación de humanos". Analytical Biochemistry . 344 (1): 53–69. doi :10.1016/j.ab.2005.05.028. PMID 16054106.
- ^ Howard R, Encheva V, Thomson J, Bache K, Chan YT, Cowen S, Debenham P, Dixon A, Krause JU, Krishan E, Moore D, Moore V, Ojo M, Rodrigues S, Stokes P, Walker J, Zimmermann W, Barallon R (15 de junio de 2011). "Análisis comparativo del ADN mitocondrial humano de muestras óseas de la Primera Guerra Mundial mediante secuenciación de ADN y espectrometría de masas ESI-TOF". Forensic Science International: Genetics . 7 (1): 1–9. doi : 10.1016/j.fsigen.2011.05.009 . PMID 21683667.
- ^ Monforte JA, Becker CH (1 de marzo de 1997). "Análisis de ADN de alto rendimiento mediante espectrometría de masas de tiempo de vuelo". Nature Medicine . 3 (3): 360–62. doi :10.1038/nm0397-360. PMID 9055869. S2CID 28386145.
- ^ Beres SB, Carroll RK, Shea PR, Sitkiewicz I, Martinez-Gutierrez JC, Low DE, McGeer A, Willey BM, Green K, Tyrrell GJ, Goldman TD, Feldgarden M, Birren BW, Fofanov Y, Boos J, Wheaton WD, Honisch C, Musser JM (8 de febrero de 2010). "Complejidad molecular de epidemias bacterianas sucesivas deconvolucionadas por patonomía comparativa". Actas de la Academia Nacional de Ciencias . 107 (9): 4371–76. Bibcode :2010PNAS..107.4371B. doi : 10.1073/pnas.0911295107 . PMC 2840111 . PMID 20142485.
- ^ Kan CW, Fredlake CP, Doherty EA, Barron AE (1 de noviembre de 2004). "Secuenciación de ADN y genotipado en sistemas de electroforesis miniaturizados". Electroforesis . 25 (21–22): 3564–88. doi :10.1002/elps.200406161. PMID 15565709. S2CID 4851728.
- ^ Chen YJ, Roller EE, Huang X (2010). "Secuenciación de ADN por desnaturalización: prueba experimental de concepto con un dispositivo fluídico integrado". Lab on a Chip . 10 (9): 1153–59. doi :10.1039/b921417h. PMC 2881221 . PMID 20390134.
- ^ Bell DC, Thomas WK, Murtagh KM, Dionne CA, Graham AC, Anderson JE, Glover WR (9 de octubre de 2012). "Identificación de bases de ADN mediante microscopía electrónica". Microscopía y microanálisis . 18 (5): 1049–53. Código Bibliográfico :2012MiMic..18.1049B. doi :10.1017/S1431927612012615. PMID 23046798. S2CID 25713635.
- ^ Pareek CS, Smoczynski R, Tretyn A (noviembre de 2011). "Tecnologías de secuenciación y secuenciación del genoma". Journal of Applied Genetics . 52 (4): 413–35. doi :10.1007/s13353-011-0057-x. PMC 3189340 . PMID 21698376.
- ^ Pareek CS, Smoczynski R, Tretyn A (2011). "Tecnologías de secuenciación y secuenciación del genoma". Journal of Applied Genetics . 52 (4): 413–35. doi :10.1007/s13353-011-0057-x. PMC 3189340 . PMID 21698376.
- ^ Fujimori S, Hirai N, Ohashi H, Masuoka K, Nishikimi A, Fukui Y, Washio T, Oshikubo T, Yamashita T, Miyamoto-Sato E (2012). "Secuenciación de próxima generación acoplada con una tecnología de visualización sin células para la producción de alto rendimiento de datos confiables del interactoma". Scientific Reports . 2 : 691. Bibcode :2012NatSR...2..691F. doi :10.1038/srep00691. PMC 3466446 . PMID 23056904.
- ^ "Cuota de mercado de secuenciación en 2022: la misma que siempre (por ahora)". 25 de junio de 2023.
- ^ Harbers M (2008). "El estado actual de la clonación de ADNc". Genomics . 91 (3): 232–42. doi : 10.1016/j.ygeno.2007.11.004 . PMID 18222633.
- ^ Alberti A, Belser C, Engelen S, Bertrand L, Orvain C, Brinas L, Cruaud C, et al. (2014). "La comparación de los métodos de preparación de bibliotecas revela su impacto en la interpretación de los datos metatranscriptómicos". BMC Genomics . 15 (1): 912–12. doi : 10.1186/1471-2164-15-912 . PMC 4213505 . PMID 25331572.
- ^ "Evaluaciones escalables de la calidad de los ácidos nucleicos para la preparación de la biblioteca de secuenciación de próxima generación de Illumina" (PDF) . Consultado el 27 de diciembre de 2017 .
- ^ "Archon Genomics XPRIZE". Archon Genomics XPRIZE . Archivado desde el original el 17 de junio de 2013. Consultado el 9 de agosto de 2007 .
- ^ "Información sobre la subvención". Instituto Nacional de Investigación del Genoma Humano (NHGRI) .
- ^ Severin J, Lizio M, Harshbarger J, Kawaji H, Daub CO, Hayashizaki Y, Bertin N, Forrest AR (2014). "Visualización interactiva y análisis de conjuntos de datos de secuenciación a gran escala utilizando ZENBU". Nat. Biotechnol . 32 (3): 217–19. doi :10.1038/nbt.2840. PMID 24727769. S2CID 26575621.
- ^ Shmilovici A, Ben-Gal I (2007). "Uso de un modelo VOM para reconstruir regiones codificantes potenciales en secuencias EST" (PDF) . Computational Statistics . 22 (1): 49–69. doi :10.1007/s00180-007-0021-8. S2CID 2737235. Archivado desde el original (PDF) el 31 de mayo de 2020 . Consultado el 10 de enero de 2014 .
- ^ Del Fabbro C, Scalabrin S, Morgante M, Giorgi FM (2013). "Una evaluación exhaustiva de los efectos de recorte de lectura en el análisis de datos de NGS de Illumina". PLOS ONE . 8 (12): e85024. Bibcode :2013PLoSO...885024D. doi : 10.1371/journal.pone.0085024 . PMC 3871669 . PMID 24376861.
- ^ Martin, Marcel (2 de mayo de 2011). "Cutadapt elimina secuencias adaptadoras de lecturas de secuenciación de alto rendimiento". EMBnet.journal . 17 (1): 10. doi : 10.14806/ej.17.1.200 .
- ^ Smeds L, Künstner A (19 de octubre de 2011). "ConDeTri: un recortador de lectura dependiente del contenido para datos de Illumina". PLOS ONE . 6 (10): e26314. Bibcode :2011PLoSO...626314S. doi : 10.1371/journal.pone.0026314 . PMC 3198461 . PMID 22039460.
- ^ Prezza N, Del Fabbro C, Vezzi F, De Paoli E, Policriti A (2012). "Erne-Bs5". Actas de la Conferencia ACM sobre Bioinformática, Biología Computacional y Biomedicina . vol. 12. págs. 12-19. doi :10.1145/2382936.2382938. ISBN 9781450316705.S2CID5673753 .
- ^ Schmieder R, Edwards R (marzo de 2011). "Control de calidad y preprocesamiento de conjuntos de datos metagenómicos". Bioinformática . 27 (6): 863–4. doi :10.1093/bioinformatics/btr026. PMC 3051327 . PMID 21278185.
- ^ Bolger AM, Lohse M, Usadel B (agosto de 2014). "Trimmomatic: un recortador flexible para datos de secuencias de Illumina". Bioinformática . 30 (15): 2114–20. doi :10.1093/bioinformatics/btu170. PMC 4103590 . PMID 24695404.
- ^ Cox MP, Peterson DA, Biggs PJ (septiembre de 2010). "SolexaQA: evaluación de calidad de un vistazo de los datos de secuenciación de segunda generación de Illumina". BMC Bioinformatics . 11 (1): 485. doi : 10.1186/1471-2105-11-485 . PMC 2956736 . PMID 20875133.
- ^ Murray TH (enero de 1991). "Cuestiones éticas en la investigación del genoma humano". FASEB Journal . 5 (1): 55–60. doi : 10.1096/fasebj.5.1.1825074 . PMID 1825074. S2CID 20009748.
- ^ abc Robertson JA (agosto de 2003). "El genoma de los 1000 dólares: cuestiones éticas y legales en la secuenciación completa del genoma de individuos". The American Journal of Bioethics . 3 (3): W–IF1. doi :10.1162/152651603322874762. PMID 14735880. S2CID 15357657.
- ^ ab Henderson, Mark (9 de septiembre de 2013). «Secuenciación del genoma humano: los verdaderos dilemas éticos». The Guardian . Consultado el 20 de mayo de 2015 .
- ^ Harmon, Amy (24 de febrero de 2008). "El temor a las aseguradoras hace que muchos eviten las pruebas de ADN". The New York Times . Consultado el 20 de mayo de 2015 .
- ^ Declaración de política de la Administración, Oficina Ejecutiva del Presidente, Oficina de Administración y Presupuesto, 27 de abril de 2007
- ^ Instituto Nacional de Investigación del Genoma Humano (21 de mayo de 2008). "President Bush Signs the Genetic Information Nondiscrimination Act of 2008" (El presidente Bush firma la Ley de no discriminación por información genética de 2008) . Consultado el 17 de febrero de 2014 .
- ^ Baker, Monya (11 de octubre de 2012). "Informe del comité de ética de Estados Unidos sobre la secuenciación del ADN y la privacidad". Nature News Blog .
- ^ "Privacidad y progreso en la secuenciación del genoma completo" (PDF) . Comisión Presidencial para el Estudio de Asuntos Bioéticos. Archivado desde el original (PDF) el 12 de junio de 2015 . Consultado el 20 de mayo de 2015 .
- ^ Hartnett, Kevin (12 de mayo de 2013). "El ADN de la basura: en juego" . The Boston Globe . Consultado el 2 de enero de 2023 .
- ^ Goldenberg AJ, Sharp RR (febrero de 2012). "Los riesgos éticos y los desafíos programáticos del cribado genómico neonatal". JAMA . 307 (5): 461–2. doi :10.1001/jama.2012.68. PMC 3868436 . PMID 22298675.
- ^ Hughes, Virginia (7 de enero de 2013). "Es hora de dejar de obsesionarse con los peligros de la información genética". Revista Slate . Consultado el 22 de mayo de 2015 .
- ^ ab Bloss CS, Schork NJ, Topol EJ (febrero de 2011). "Efecto de la elaboración de perfiles genómicos directos al consumidor para evaluar el riesgo de enfermedad". The New England Journal of Medicine . 364 (6): 524–34. doi :10.1056/NEJMoa1011893. PMC 3786730 . PMID 21226570.
- ^ Rochman, Bonnie (25 de octubre de 2012). "Lo que su médico no le dice sobre su ADN". Time.com . Consultado el 22 de mayo de 2015 .
- ^ Sajeer P, Muhammad (4 de mayo de 2023). "Tecnología disruptiva: exploración de las implicaciones éticas, legales, políticas y sociales de la tecnología de secuenciación de nanoporos". EMBO Reports . 24 (5): e56619. doi :10.15252/embr.202256619. PMC 10157308 . PMID 36988424. S2CID 257803254.
Enlaces externos
- Un wikilibro sobre secuenciación de próxima generación.




