Secuenciación del exoma

La secuenciación del exoma , también conocida como secuenciación del exoma completo ( WES ), es una técnica genómica para secuenciar todas las regiones codificantes de proteínas de los genes en un genoma (conocido como exoma ). [1] Consta de dos pasos: el primer paso es seleccionar solo el subconjunto de ADN que codifica proteínas . Estas regiones se conocen como exones : los humanos tienen alrededor de 180.000 exones, que constituyen aproximadamente el 1% del genoma humano , o aproximadamente 30 millones de pares de bases . El segundo paso es secuenciar el ADN exónico utilizando cualquier tecnología de secuenciación de ADN de alto rendimiento . [2]
El objetivo de este enfoque es identificar variantes genéticas que alteran las secuencias de proteínas, y hacerlo a un costo mucho menor que la secuenciación del genoma completo . Dado que estas variantes pueden ser responsables tanto de enfermedades mendelianas como de enfermedades poligénicas comunes , como la enfermedad de Alzheimer , la secuenciación del exoma completo se ha aplicado tanto en la investigación académica como en el diagnóstico clínico.

Motivación y comparación con otros enfoques
La secuenciación del exoma es especialmente eficaz en el estudio de enfermedades mendelianas raras, porque es una forma eficiente de identificar las variantes genéticas en todos los genes de un individuo. Estas enfermedades suelen estar causadas por variantes genéticas muy raras que solo están presentes en un pequeño número de individuos; [3] por el contrario, técnicas como las matrices de SNP solo pueden detectar variantes genéticas compartidas que son comunes a muchos individuos en la población en general. [4] Además, debido a que las variantes causantes de enfermedades graves tienen muchas más probabilidades (pero de ninguna manera exclusivamente) de estar en la secuencia codificante de la proteína, [5] [6] centrarse en este 1% cuesta mucho menos que la secuenciación del genoma completo pero aún así detecta un alto rendimiento de variantes relevantes.
En el pasado, las pruebas genéticas clínicas se elegían en función de la presentación clínica del paciente (es decir, se centraban en un gen o en un pequeño número conocido por estar asociado con un síndrome particular), o examinaban solo ciertos tipos de variación (por ejemplo, hibridación genómica comparativa ), pero proporcionaban diagnósticos genéticos definitivos en menos de la mitad de todos los pacientes. [7] La secuenciación del exoma se utiliza cada vez más para complementar estas otras pruebas: tanto para encontrar mutaciones en genes que ya se sabe que causan enfermedades como para identificar genes nuevos comparando exomas de pacientes con características similares. [ cita requerida ]
Metodología técnica
Paso 1: Estrategias de enriquecimiento de objetivos
Los métodos de enriquecimiento de objetivos permiten capturar selectivamente regiones genómicas de interés de una muestra de ADN antes de la secuenciación. Se han desarrollado varias estrategias de enriquecimiento de objetivos desde la descripción original del método de selección genómica directa (DGS) en 2005. [8]
Aunque se han descrito muchas técnicas para la captura dirigida, solo unas pocas de ellas se han ampliado para capturar exomas completos. [9] La primera estrategia de enriquecimiento de objetivos que se aplicó a la secuenciación de exomas completos fue el método de captura híbrida basada en matrices en 2007, pero la captura en solución ha ganado popularidad en los últimos años.
Captura basada en matrices
Los microarrays contienen oligonucleótidos monocatenarios con secuencias del genoma humano para cubrir la región de interés fijada a la superficie. El ADN genómico se corta para formar fragmentos bicatenarios. Los fragmentos se someten a una reparación de extremos para producir extremos romos y se añaden adaptadores con secuencias de cebado universales. Estos fragmentos se hibridan con oligos en el microarray. Los fragmentos no hibridados se eliminan por lavado y se eluyen los fragmentos deseados. A continuación, los fragmentos se amplifican mediante PCR . [10] [11]
Roche NimbleGen fue el primero en adoptar la tecnología DGS original [8] y adaptarla para la secuenciación de próxima generación. Desarrollaron el Sequence Capture Human Exome 2.1M Array para capturar aproximadamente 180 000 exones codificantes. [12] Este método ahorra tiempo y es rentable en comparación con los métodos basados en PCR. El Agilent Capture Array y el array de hibridación genómica comparativa son otros métodos que se pueden utilizar para la captura híbrida de secuencias objetivo. Las limitaciones de esta técnica incluyen la necesidad de hardware costoso, así como una cantidad relativamente grande de ADN. [13]
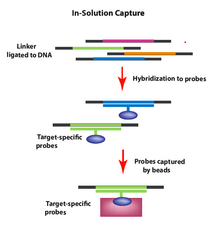
Captura en solución
Para capturar regiones genómicas de interés mediante captura en solución, se sintetiza un conjunto de oligonucleótidos personalizados (sondas) y se hibridan en solución con una muestra de ADN genómico fragmentado. Las sondas (marcadas con perlas) se hibridan selectivamente con las regiones genómicas de interés, después de lo cual las perlas (que ahora incluyen los fragmentos de ADN de interés) se pueden retirar y lavar para eliminar el exceso de material. Luego se retiran las perlas y se pueden secuenciar los fragmentos genómicos, lo que permite la secuenciación selectiva del ADN de las regiones genómicas (por ejemplo, los exones) de interés.
Este método se desarrolló para mejorar el método de enriquecimiento de dianas mediante captura por hibridación. En la captura en solución (a diferencia de la captura híbrida) hay un exceso de sondas para las regiones de interés diana sobre la cantidad de plantilla requerida. [13] El tamaño óptimo de la diana es de aproximadamente 3,5 megabases y produce una excelente cobertura de secuencia de las regiones diana. El método preferido depende de varios factores, entre ellos: número de pares de bases en la región de interés, demandas de lecturas en la diana, equipo interno, etc. [14]
Paso 2: secuenciación
Existen muchas plataformas de secuenciación de próxima generación disponibles, posteriores a las metodologías clásicas de secuenciación de Sanger. Otras plataformas incluyen el secuenciador Roche 454 y los sistemas SOLiD de Life Technologies , el Ion Torrent de Life Technologies y el Illumina Genome Analyzer II (descontinuado) de Illumina y los instrumentos posteriores de las series MiSeq, HiSeq y NovaSeq de Illumina, todos los cuales pueden usarse para la secuenciación masiva paralela del exoma. Estos sistemas NGS de "lectura corta" son particularmente adecuados para analizar muchos tramos relativamente cortos de secuencia de ADN, como los que se encuentran en los exones humanos.
Comparación con otras tecnologías
Existen múltiples tecnologías disponibles para identificar variantes genéticas. Cada tecnología tiene ventajas y desventajas en términos de factores técnicos y financieros. Dos de estas tecnologías son los microarrays y la secuenciación del genoma completo .
Genotipado basado en microarrays
Los microarrays utilizan sondas de hibridación para probar la prevalencia de secuencias de ADN conocidas, por lo que no se pueden utilizar para identificar cambios genéticos inesperados. [13] Por el contrario, las tecnologías de secuenciación de alto rendimiento utilizadas en la secuenciación del exoma proporcionan directamente las secuencias de nucleótidos del ADN en los miles de loci exónicos analizados. [15] Por lo tanto, WES aborda algunas de las limitaciones actuales de los microarrays de genotipado por hibridación .
Aunque la secuenciación del exoma es más costosa que las tecnologías basadas en hibridación en términos de cada muestra, su costo ha ido disminuyendo debido a la caída de los costos y al aumento del rendimiento de la secuenciación del genoma completo . [ cita requerida ]
Secuenciación del genoma completo
La secuenciación del exoma sólo es capaz de identificar aquellas variantes que se encuentran en la región codificante de los genes que afectan la función de las proteínas. No es capaz de identificar las variantes estructurales y no codificantes asociadas con la enfermedad, que se pueden encontrar utilizando otros métodos como la secuenciación del genoma completo . [2] Queda un 99% del genoma humano que no está cubierto mediante la secuenciación del exoma, y la secuenciación del exoma permite la secuenciación de porciones del genoma en al menos 20 veces más muestras en comparación con la secuenciación del genoma completo. [2] Para la traducción de variantes raras identificadas a la clínica, el tamaño de la muestra y la capacidad de interpretar los resultados para proporcionar un diagnóstico clínico indican que con el conocimiento actual en genética, hay informes de que la secuenciación del exoma se utiliza para ayudar al diagnóstico. [12] El costo de la secuenciación del exoma es típicamente menor que la secuenciación del genoma completo. [16]
Análisis de datos
El análisis estadístico de la gran cantidad de datos generados a partir de métodos de secuenciación es un desafío. Incluso al secuenciar únicamente los exomas de individuos, se genera una gran cantidad de datos e información de secuencias que requiere una cantidad significativa de análisis de datos. Los desafíos asociados con el análisis de estos datos incluyen cambios en los programas utilizados para alinear y ensamblar lecturas de secuencias. [13] Varias tecnologías de secuenciación también tienen diferentes tasas de error y generan varias longitudes de lectura que pueden plantear desafíos a la hora de comparar resultados de diferentes plataformas de secuenciación.
Los resultados falsos positivos y falsos negativos están asociados con los métodos de resecuenciación genómica y son cuestiones críticas. Se han desarrollado algunas estrategias para mejorar la calidad de los datos del exoma, como:
- Comparación de las variantes genéticas identificadas entre la secuenciación y la genotipificación basada en matrices [2]
- Comparación de los SNP codificantes con un genoma completo secuenciado de un individuo con el trastorno [2]
- Comparación de los SNP codificantes con la secuenciación de Sanger de individuos HapMap [2]
Es posible que los trastornos recesivos raros no tengan polimorfismos de un solo nucleótido (SNP) en bases de datos públicas como dbSNP . Los fenotipos recesivos más comunes tendrían más probabilidades de tener variantes causantes de enfermedades informadas en dbSNP. Por ejemplo, la variante de fibrosis quística más común tiene una frecuencia de alelos de aproximadamente el 3% en la mayoría de las poblaciones. La detección de dichas variantes podría excluir erróneamente la consideración de dichos genes. Los genes de los trastornos recesivos suelen ser más fáciles de identificar que los trastornos dominantes porque es menos probable que los genes tengan más de una variante no sinónima rara. [2] El sistema que examina las variantes genéticas comunes se basa en dbSNP, que puede no tener información precisa sobre la variación de los alelos. El uso de listas de variación común de un exoma de estudio o de un individuo secuenciado a nivel de genoma sería más confiable. Un desafío en este enfoque es que a medida que aumenta el número de exomas secuenciados, dbSNP también aumentará en el número de variantes poco comunes. Será necesario desarrollar umbrales para definir las variantes comunes que es poco probable que estén asociadas con un fenotipo de enfermedad. [15]
La heterogeneidad genética y la etnicidad de la población también son limitaciones importantes, ya que pueden aumentar el número de resultados falsos positivos y falsos negativos, lo que dificultará la identificación de genes candidatos. Por supuesto, es posible reducir la rigurosidad de los umbrales en presencia de heterogeneidad y etnicidad, pero esto también reducirá la capacidad para detectar variantes. El uso de un enfoque que priorice el genotipo para identificar genes candidatos también podría ofrecer una solución para superar estas limitaciones.
A diferencia del análisis de variantes comunes, el análisis de variantes raras en estudios de secuenciación del exoma completo evalúa conjuntos de variantes en lugar de variantes individuales. [17] [18] Las anotaciones funcionales predicen el efecto o la función de las variantes raras y ayudan a priorizar las variantes funcionales raras. La incorporación de estas anotaciones puede aumentar de manera efectiva el poder del análisis de asociación genética de variantes raras en estudios de secuenciación del genoma completo. [19] Se han desarrollado algunos métodos y herramientas para realizar análisis de asociación de variantes raras con información funcional mediante la incorporación de anotaciones funcionales para potenciar el análisis en estudios de secuenciación del exoma completo. [20] [21]
Implicaciones éticas
Las nuevas tecnologías en genómica han cambiado la forma en que los investigadores abordan la investigación básica y la translacional. Con enfoques como la secuenciación del exoma, es posible mejorar significativamente los datos generados a partir de genomas individuales, lo que ha planteado una serie de preguntas sobre cómo manejar la enorme cantidad de información. ¿Se debe permitir que los individuos que participan en estos estudios tengan acceso a su información de secuenciación? ¿Se debe compartir esta información con las compañías de seguros? Estos datos pueden conducir a hallazgos inesperados y complicar la utilidad clínica y el beneficio para el paciente. Esta área de la genómica sigue siendo un desafío y los investigadores están estudiando cómo abordar estas preguntas. [15]
Aplicaciones de la secuenciación del exoma
Mediante la secuenciación del exoma, los estudios de costo fijo pueden secuenciar muestras con una profundidad mucho mayor que la que se podría lograr con la secuenciación del genoma completo. Esta profundidad adicional hace que la secuenciación del exoma sea adecuada para varias aplicaciones que necesitan determinaciones de variantes confiables.
Mapeo de variantes raras en trastornos complejos
Los estudios de asociación actuales se han centrado en la variación común en todo el genoma, ya que son los más fáciles de identificar con nuestros ensayos actuales. Sin embargo, se ha descubierto que las variantes causantes de enfermedades de gran efecto se encuentran dentro de los exomas en estudios de genes candidatos y, debido a la selección negativa , se encuentran en frecuencias de alelos mucho más bajas y pueden permanecer sin tipificar en los ensayos de genotipado estándar actuales. La secuenciación del genoma completo es un método potencial para analizar nuevas variantes en todo el genoma. Sin embargo, en trastornos complejos (como el autismo), se cree que una gran cantidad de genes están asociados con el riesgo de enfermedad. [1] [22] Esta heterogeneidad del riesgo subyacente significa que se requieren tamaños de muestra muy grandes para el descubrimiento de genes y, por lo tanto, la secuenciación del genoma completo no es particularmente rentable. Este problema del tamaño de la muestra se alivia con el desarrollo de nuevos métodos analíticos avanzados, que mapean eficazmente los genes de la enfermedad a pesar de que las mutaciones genéticas son raras a nivel de variante. [22] Además, las variantes en las regiones codificantes se han estudiado mucho más ampliamente y sus implicaciones funcionales son mucho más fáciles de derivar, lo que hace que las aplicaciones prácticas de las variantes dentro de la región del exoma en cuestión sean más inmediatamente accesibles.
La secuenciación del exoma en el descubrimiento de variantes genéticas raras sigue siendo un área de investigación muy activa y en curso, y hay cada vez más evidencia de que se observa una carga significativa de riesgo en los distintos conjuntos de genes. Se ha informado de variantes raras del gen KRT82 en la alopecia areata, un trastorno autoinmune. [1]
Descubrimiento de los trastornos mendelianos
En los trastornos mendelianos de gran importancia, los hallazgos hasta el momento sugieren que una o una cantidad muy pequeña de variantes dentro de los genes codificantes subyacen a toda la afección. Debido a la gravedad de estos trastornos, se presume que las pocas variantes causales son extremadamente raras o nuevas en la población, y que cualquier ensayo de genotipificación estándar no las detectaría. La secuenciación del exoma proporciona una alta cobertura de las variantes en las regiones codificantes, que son necesarias para separar las variantes verdaderas del ruido. Un modelo exitoso de descubrimiento de genes mendelianos implica el descubrimiento de variantes de novo mediante la secuenciación de tríos, donde se genotipifican los progenitores y el probando.
Estudios de caso
En un estudio publicado en septiembre de 2009 se analizó un experimento de prueba de concepto para determinar si era posible identificar variantes genéticas causales mediante la secuenciación del exoma. Secuenciaron cuatro individuos con síndrome de Freeman-Sheldon (FSS) (OMIM 193700), un trastorno autosómico dominante poco común que se sabe que es causado por una mutación en el gen MYH3 . [2] También se secuenciaron ocho individuos de HapMap para eliminar las variantes comunes con el fin de identificar el gen causal del FSS. Después de la exclusión de las variantes comunes, los autores pudieron identificar MYH3 , lo que confirma que la secuenciación del exoma se puede utilizar para identificar variantes causales de trastornos raros. [2] Este fue el primer estudio informado que utilizó la secuenciación del exoma como un enfoque para identificar un gen causal desconocido para un trastorno mendeliano raro.
Posteriormente, otro grupo informó de un diagnóstico clínico exitoso de un paciente de origen turco con sospecha de síndrome de Bartter . [12] El síndrome de Bartter es una enfermedad renal con pérdida de sal. La secuenciación del exoma reveló una mutación recesiva inesperada y bien conservada en un gen llamado SLC26A3 que está asociado con la diarrea congénita por cloruro (EHC). Este diagnóstico molecular de EHC fue confirmado por el médico que lo refirió. Este ejemplo proporcionó una prueba de concepto del uso de la secuenciación completa del exoma como herramienta clínica en la evaluación de pacientes con enfermedades genéticas no diagnosticadas. Este informe se considera la primera aplicación de la tecnología de secuenciación de próxima generación para el diagnóstico molecular de un paciente.
Se realizó un segundo informe sobre la secuenciación del exoma de individuos con un trastorno mendeliano conocido como síndrome de Miller (MIM#263750), un trastorno poco común de herencia autosómica recesiva . Se estudiaron dos hermanos y dos individuos no relacionados con el síndrome de Miller. Se analizaron variantes que tienen el potencial de ser patógenas, como mutaciones no sinónimas, sitios donantes y aceptores de empalme e inserciones o deleciones de codificación cortas. [3] Dado que el síndrome de Miller es un trastorno poco común, se espera que la variante causal no haya sido identificada previamente. Se utilizaron estudios previos de secuenciación del exoma de polimorfismos de un solo nucleótido (SNP) comunes en bases de datos de SNP públicas para excluir aún más genes candidatos. Después de la exclusión de estos genes, los autores encontraron mutaciones en DHODH que eran compartidas entre individuos con síndrome de Miller. Cada individuo con síndrome de Miller era un heterocigoto compuesto para las mutaciones de DHODH que se heredaban, ya que se descubrió que cada padre de un individuo afectado era portador. [3]
Esta fue la primera vez que se demostró que la secuenciación del exoma identifica un gen nuevo responsable de una enfermedad mendeliana rara. Este emocionante hallazgo demuestra que la secuenciación del exoma tiene el potencial de localizar genes causantes de enfermedades complejas, algo que antes no había sido posible debido a las limitaciones de los métodos tradicionales. La captura dirigida y la secuenciación masiva en paralelo representan una estrategia rentable, reproducible y robusta con alta sensibilidad y especificidad para detectar variantes que causan cambios en la codificación de proteínas en genomas humanos individuales.
Diagnóstico clínico
La secuenciación del exoma se puede utilizar para diagnosticar la causa genética de una enfermedad en un paciente. La identificación de las mutaciones genéticas subyacentes a la enfermedad puede tener implicaciones importantes para los enfoques diagnósticos y terapéuticos, puede orientar la predicción de la historia natural de la enfermedad y permite realizar pruebas a los miembros de la familia en riesgo. [2] [3] [12] [23] [24] [25] Hay muchos factores que hacen que la secuenciación del exoma sea superior al análisis de un solo gen, incluida la capacidad de identificar mutaciones en genes que no se analizaron debido a una presentación clínica atípica [25] o la capacidad de identificar casos clínicos en los que las mutaciones de diferentes genes contribuyen a los diferentes fenotipos en el mismo paciente. [3]
Una vez diagnosticada la causa genética de una enfermedad, esta información puede orientar la selección del tratamiento adecuado. La primera vez que se aplicó esta estrategia con éxito en la clínica fue en el tratamiento de un lactante con enfermedad inflamatoria intestinal. [24] [26] Se habían utilizado anteriormente varios diagnósticos convencionales, pero los resultados no pudieron explicar los síntomas del lactante. El análisis de los datos de secuenciación del exoma identificó una mutación en el gen XIAP . El conocimiento de la función de este gen orientó el tratamiento del lactante, lo que condujo a un trasplante de médula ósea que curó al niño de la enfermedad. [24]
Los investigadores han utilizado la secuenciación del exoma para identificar la mutación subyacente en un paciente con síndrome de Bartter y diarrea congénita por cloruro. [12] El grupo de Bilgular también utilizó la secuenciación del exoma e identificó la mutación subyacente en un paciente con malformaciones cerebrales graves, y afirmó que "[Estos hallazgos] destacan el uso de la secuenciación completa del exoma para identificar los loci de la enfermedad en entornos en los que los métodos tradicionales han demostrado ser un desafío... Nuestros resultados demuestran que esta tecnología será particularmente valiosa para el descubrimiento de genes en aquellas condiciones en las que el mapeo se ha visto confundido por la heterogeneidad de los locus y la incertidumbre sobre los límites de la clasificación diagnóstica, lo que apunta a un futuro brillante para su amplia aplicación en la medicina". [23]
Investigadores de la Universidad de Ciudad del Cabo, Sudáfrica, utilizaron la secuenciación del exoma para descubrir la mutación genética de CDH2 como la causa subyacente de un trastorno genético conocido como miocardiopatía arritmogénica del ventrículo derecho (ARVC), que aumenta el riesgo de enfermedad cardíaca y paro cardíaco. [1]
Costos comerciales
Varias empresas han ofrecido la secuenciación del exoma a los consumidores. Knome fue la primera empresa en ofrecer servicios de secuenciación del exoma a los consumidores [ ¿cuándo? ] , a un coste de varios miles de dólares. [27] Más tarde, 23andMe puso en marcha un programa piloto de secuenciación del exoma que se anunció en septiembre de 2011 y se interrumpió en 2012. Los consumidores podían obtener datos del exoma a un coste de 999 dólares. La empresa proporcionaba datos sin procesar y no ofrecía análisis. [27] [28] [29]
En noviembre de 2012, DNADTC, una división de Gene by Gene, comenzó a ofrecer exomas con una cobertura de 80X y un precio de lanzamiento de $695. [30] Este precio por sitio web de DNADTC es actualmente de $895. En octubre de 2013, BGI anunció una promoción para la secuenciación personal de exomas completos con una cobertura de 50X por $499. [31] En junio de 2016, Genos pudo lograr un precio aún más bajo de $399 con un exoma de consumidor con certificación CLIA de 75X secuenciado a partir de saliva. [32] [33] [34]
Una revisión de 36 estudios realizada en 2018 determinó que el costo de la secuenciación del exoma oscilaba entre 555 USD y 5169 USD, con un rendimiento diagnóstico que oscilaba entre el 3 % y el 79 % según los grupos de pacientes. [16]
Véase también
- Perfil de ADN
- Asesoramiento genético
- Medicina personalizada
- Transcriptómica
- Secuenciación del genoma completo
Referencias
- ^ abc Erjavec SO, Gelfman S, Abdelaziz AR, Lee EY, Monga I, Alkelai A, Ionita-Laza I, Petukhova L, Christiano AM (febrero de 2022). "La secuenciación completa del exoma en Alopecia Areata identifica variantes raras en KRT82". Comuna Nacional . 13 (1): 800. Código Bib : 2022NatCo..13..800E. doi :10.1038/s41467-022-28343-3. PMC 8831607 . PMID 35145093.
- ^ abcdefghij Ng SB, Turner EH, Robertson PD, Flygare SD, Bigham AW, Lee C, Shaffer T, Wong M, Bhattacharjee A, Eichler EE, Bamshad M, Nickerson DA, Shendure J (10 de septiembre de 2009). "Captura dirigida y secuenciación masiva paralela de 12 exomas humanos". Naturaleza . 461 (7261): 272–276. Código Bib :2009Natur.461..272N. doi : 10.1038/naturaleza08250. PMC 2844771 . PMID 19684571.
- ^ abcde Sarah B Ng; Kati J Buckingham; Choli Lee; Abigail W Bigham; Holly K Tabor; Karin M Dent; Chad D Huff; Paul T Shannon; Ethylin Wang Jabs; Deborah A Nickerson; Jay Shendure; Michael J Bamshad (2010). "La secuenciación del exoma identifica la causa de un trastorno mendeliano". Nature Genetics . 42 (1): 30–35. doi :10.1038/ng.499. PMC 2847889 . PMID 19915526.
- ^ Wang, DG; Fan, JB; Siao, CJ; Berno, A.; Young, P.; Sapolsky, R.; Ghandour, G.; Perkins, N.; Winchester, E. (15 de mayo de 1998). "Identificación, mapeo y genotipado a gran escala de polimorfismos de un solo nucleótido en el genoma humano". Science . 280 (5366): 1077–1082. Bibcode :1998Sci...280.1077W. doi :10.1126/science.280.5366.1077. ISSN 0036-8075. PMID 9582121.
- ^ Petersen, Britt-Sabina; Fredrich, Broder; Hoeppner, Marc P.; Ellinghaus, David; Franke, Andre (14 de febrero de 2017). "Oportunidades y desafíos de la secuenciación del genoma completo y del exoma". BMC Genetics . 18 (1): 14. doi : 10.1186/s12863-017-0479-5 . ISSN 1471-2156. PMC 5307692 . PMID 28193154.
- ^ Botstein, David; Risch, Neil (marzo de 2003). "Descubrimiento de genotipos subyacentes a los fenotipos humanos: éxitos pasados en enfermedades mendelianas, enfoques futuros para enfermedades complejas". Nature Genetics . 33 (3): 228–237. doi :10.1038/ng1090. ISSN 1546-1718. PMID 12610532. S2CID 10599219.
- ^ Rauch, A; Hoyer, J; Guth, S; Zweier, C; Kraus, C; Becker, C; Zenker, M; Hüffmeier, U; Thiel, C; Rüschendorf, F; Nürnberg, P; Reis, A; Trautmann, U (1 de octubre de 2006). "Rendimiento diagnóstico de varios enfoques genéticos en pacientes con retraso mental o retraso del desarrollo inexplicado". American Journal of Medical Genetics Part A. 140 ( 19): 2063–74. doi : 10.1002/ajmg.a.31416 . PMID: 16917849. S2CID : 24570999.
- ^ por Stavros Basiardes; Rose Veile; Cindy Helms; Elaine R. Mardis; Anne M. Bowcock; Michael Lovett (2005). "Selección genómica directa". Nature Methods . 1 (2): 63–69. doi :10.1038/nmeth0105-63. PMID 16152676. S2CID 667227.
- ^ Teer, JK; Mullikin, JC (12 de agosto de 2010). "Secuenciación del exoma: el punto óptimo antes de los genomas completos". Genética molecular humana . 19 (R2): R145–R151. doi :10.1093/hmg/ddq333. PMC 2953745 . PMID 20705737.
- ^ Emily H. Turner; Sarah B. Ng; Deborah A. Nickerson; Jay Shendure (2009). "Métodos para la partición genómica". Annu Rev Genom Hum Genet . 10 (1): 30–35. doi :10.1146/annurev-genom-082908-150112. PMID 19630561.
- ^ Mertes F, Elsharawy A, Sauer S, van Helvoort JM, van der Zaag PJ, Franke A, Nilsson M, Lehrach H, Brookes AJ (2011). "Enriquecimiento dirigido de regiones de ADN genómico para secuenciación de próxima generación". Brief. Funct. Genomics . 10 (6): 374–386. doi :10.1093/bfgp/elr033. PMC 3245553. PMID 22121152. Archivado desde el original (PDF) el 2017-09-21 . Consultado el 2018-10-24 .
- ^ abcde Choi M, Scholl UI, Ji W, Liu T, Tikhonova IR, Zumbo P, Nayir A, Bakkaloğlu A, Ozen S, Sanjad S, Nelson-Williams C, Farhi A, Mane S, Lifton RP (10 de noviembre de 2009) . "Diagnóstico genético mediante captura del exoma completo y secuenciación masiva de ADN en paralelo". Proc Natl Acad Sci Estados Unidos . 106 (45): 19096–19101. Código bibliográfico : 2009PNAS..10619096C. doi : 10.1073/pnas.0910672106 . PMC 2768590 . PMID 19861545.
- ^ abcd Kahvejian A, Quackenbush J, Thompson JF (2008). "¿Qué harías si pudieras secuenciarlo todo?". Nature Biotechnology . 26 (10): 1125–1133. doi :10.1038/nbt1494. PMC 4153598 . PMID 18846086.
- ^ Lira Mamanova; Coffey, Alison J; Scott, Carol E; Kozarewa, Iwanka; Turner, Emily H; Kumar, Akash; Howard, Eleanor; Shendure, Jay; Turner, Daniel J; et al. (febrero de 2010). "Estrategias de enriquecimiento de objetivos para la secuenciación de próxima generación". Nature Methods . 7 (2): 111–118. doi :10.1038/nmeth.1419. PMID 20111037. S2CID 21410733.
- ^ abc Biesecker LG (enero de 2010). "La secuenciación del exoma hace que la genómica médica sea una realidad". Nat. Genet . 42 (1): 13–14. doi : 10.1038/ng0110-13 . PMID 20037612.
- ^ ab Schwarze, K; Buchanan, J; Taylor, JC; Wordsworth, S (mayo de 2018). "¿Son rentables los enfoques de secuenciación del exoma y el genoma completos? Una revisión sistemática de la literatura". Value in Health . 21 : S100. doi : 10.1016/j.jval.2018.04.677 . ISSN 1098-3015.
- ^ Lee, Seunggeung; Abecasis, Goncalo R.; Boehnke, Michael; Lin, Xihong (julio de 2014). "Análisis de asociación de variantes raras: diseños de estudios y pruebas estadísticas". The American Journal of Human Genetics . 95 (1): 5–23. doi :10.1016/j.ajhg.2014.06.009. PMC 4085641 . PMID 24995866.
- ^ Wang, Quanli; Dhindsa, Ryan S.; Carss, Keren; Harper, Andrew R.; et al. (23 de septiembre de 2021). "Contribución de variantes raras a enfermedades humanas en 281.104 exomas del biobanco del Reino Unido". Naturaleza . 597 (7877): 527–532. Código Bib :2021Natur.597..527W. doi :10.1038/s41586-021-03855-y. PMC 8458098 . PMID 34375979.
- ^ Li, Xihao; Li, Zilin; Zhou, Hufeng; Gaynor, Sheila M.; et al. (septiembre de 2020). "La incorporación dinámica de múltiples anotaciones funcionales in silico potencia el análisis de asociación de variantes raras en estudios de secuenciación de todo el genoma a gran escala". Nature Genetics . 52 (9): 969–983. doi :10.1038/s41588-020-0676-4. PMC 7483769 . PMID 32839606.
- ^ "STAARpipeline: una herramienta todo en uno para detectar variantes raras y obtener datos de secuenciación del genoma completo a escala de biobanco". Nature Methods . 19 (12): 1532–1533. Diciembre de 2022. doi :10.1038/s41592-022-01641-w. PMID 36316564. S2CID 253246835.
- ^ Li, Xihao; Rápido, Corbin; Zhou, Hufeng; Gaynor, Sheila M.; Liu, Yaowu; Chen, Han; Selvaraj, Margaret Sunitha; Sol, Ryan; Dey, Rounak; Arnett, Donna K.; Bielak, Lawrence F.; Bis, Josué C.; Blangero, Juan; Boerwinkle, Eric; Bowden, Donald W.; Brody, Jennifer A.; Cade, Brian E.; Correa, Adolfo; Cupples, L. Adrienne; Curran, Joanne E.; de Vries, Paul S.; Duggirala, Ravindranath; Freedman, Barry I.; Göring, Harald HH; Guo, Xiuqing; Haessler, Jeffrey; Kalyani, Rita R.; Kooperberg, Charles; Kral, Brian G.; Lange, Leslie A.; Manichaikul, Ani; Martín, Lisa W.; McGarvey, Stephen T.; Mitchell, Braxton D.; Montasser, May E.; Morrison, Alanna C.; Naseri, Take; O'Connell, Jeffrey R.; Palmer, Nicholette D.; Peyser, Patricia A.; Psaty, Bruce M.; Raffield, Laura M.; Redline, Susan; Reiner, Alexander P.; Reupena, Muagututi'a Sefuiva; Rice, Kenneth M.; Rich, Stephen S.; Sitlani, Colleen M.; Smith, Jennifer A.; Taylor, Kent D. ; Vasan, Ramachandran S.; Willer, Cristen J.; Wilson, James G.; Yanek, Lisa R.; Zhao, Wei; Consorcio NHLBI Trans-Omics for Precision Medicine (TOPMed); Grupo de trabajo de lípidos TOPMed; Rotter, Jerome I .; Natarajan, Pradeep; Peloso, Gina M.; Li, Zilin; Lin, Xihong (enero de 2023). "Metaanálisis potente, escalable y eficiente en términos de recursos de asociaciones de variantes raras en estudios de secuenciación del genoma completo a gran escala". Nature Genetics . 55 (1): 154–164. doi :10.1038/s41588-022-01225-6. PMC 10084891 . Número de modelo: PMID 36564505. Número de modelo: S2CID 255084231.
- ^ ab Weijun Luo; Chaolin Zhang; Yong-hui Jiang; Cory R. Brouwer (2018). "Reconstrucción sistemática de la biología del autismo a partir de perfiles de mutación genética masiva". Science Advances . 4 (4): e1701799. Bibcode :2018SciA....4.1799L. doi :10.1126/sciadv.1701799. PMC 5895441 . PMID 29651456.
- ^ ab Bilgüvar K, Oztürk AK, Louvi A, Kwan KY, Choi M, Tatli B, Yalnizoğlu D, Tüysüz B, Cağlayan AO, Gökben S, Kaymakçalan H, Barak T, Bakircioğlu M, Yasuno K, Ho W, Sanders S, Zhu Y, Yilmaz S, Dinçer A, Johnson MH, Bronen RA, Koçer N, Per H, Mane S, Pamir MN, Yalçinkaya C, Kumandaş S, Topçu M, Ozmen M, Sestan N, Lifton RP, State MW, Günel M (9 de septiembre de 2010). "La secuenciación del exoma completo identifica mutaciones recesivas de WDR62 en malformaciones cerebrales graves". Naturaleza . 467 (7312): 207–210. Código Bibliográfico : 2010Natur.467..207B. doi :10.1038/nature09327. PMC 3129007. PMID 20729831 .
- ^ abc Worthey EA, Mayer AN, Syverson GD, Helbling D, Bonacci BB, Decker B, Serpe JM, Dasu T, Tschannen MR, Veith RL, Basehore MJ, Broeckel U, Tomita-Mitchell A, Arca MJ, Casper JT, Margolis DA, Bick DP, Hessner MJ, Routes JM, Verbsky JW, Jacob HJ, Dimmock DP (marzo de 2011). "Cómo hacer un diagnóstico definitivo: aplicación clínica exitosa de la secuenciación del exoma completo en un niño con enfermedad inflamatoria intestinal intratable". Genet. Med . 13 (3): 255–262. doi : 10.1097/GIM.0b013e3182088158 . PMID 21173700.
- ^ ab Raffan E, Hurst LA, Turki SA, Carpenter G, Scott C, Daly A, Coffey A, Bhaskar S, Howard E, Khan N, Kingston H, Palotie A, Savage DB, O'Driscoll M, Smith C, O'Rahilly S, Barroso I, Semple RK (2011). "Diagnóstico temprano del síndrome de Werner mediante secuenciación de todo el exoma en un único paciente atípico". Front Endocrinol (Lausana) . 2 (8): 8. doi : 10.3389/fendo.2011.00008 . PMC 3356119. PMID 22654791 .
- ^ Warr, A.; Robert, C.; Hume, D.; Archibald, A.; Deeb, N.; Watson, M. (2 de julio de 2015). "Secuenciación del exoma: perspectivas actuales y futuras". G3 . 5 (8): 1543–1550. doi :10.1534/g3.115.018564. PMC 4528311 . PMID 26139844.
- ^ ab Herper, Matthew (27 de septiembre de 2011). "El futuro es ahora: 23andMe ahora ofrece todos tus genes por 999 dólares". Forbes . Consultado el 11 de diciembre de 2011 .
- ^ "23andMe lanza un programa piloto para la secuenciación directa del exoma al consumidor". GenomeWeb . GenomeWeb LLC. 28 de septiembre de 2011 . Consultado el 11 de diciembre de 2011 .
- ^ "Secuenciación del genoma personal en individuos aparentemente sanos y el consorcio PeopleSeq" (PDF) . Genomes2People. 14 de junio de 2016.
- ^ Vorhaus, Dan (29 de noviembre de 2012). "DNA DTC: The Return of Direct to Consumer Whole Genome Sequencing". Informe sobre la ley de genómica . Consultado el 30 de mayo de 2013 .
- ^ "Promoción definitiva del exoma". BGI Americas. 18 de octubre de 2013. Archivado desde el original el 10 de noviembre de 2013. Consultado el 17 de noviembre de 2013 .
- ^ "Ser dueño de sus datos: el modelo Genos". Bio-IT World. 6 de julio de 2016.
- ^ "La startup de genómica de consumo Genos Research planea permitir que los clientes exploren y compartan sus datos". GenomeWeb LLC. 13 de junio de 2016.
- ^ "Genos - Aprópiese de su ADN, aprenda sobre usted mismo, impulse la investigación". www.genosresearch.com . Archivado desde el original el 25 de agosto de 2016 . Consultado el 18 de octubre de 2016 .
Enlaces externos
- Noticias sobre la secuenciación del exoma [ enlace muerto permanente ]
- Guía para pacientes y familiares
