V-ATPasa
| V-ATPasa | |
|---|---|
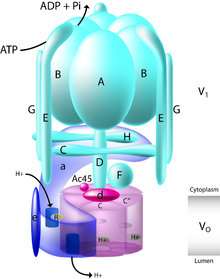 Esquema de la V-ATPasa | |
| Identificadores | |
| Símbolo | V-ATPasa |
| Base de datos de datos termodinámica | 3.A.2 |
| Superfamilia OPM | 5 |
| Proteína OPM | 2bl2 |
| Membranoma | 226 |
| V-ATPasa, subunidad c (Vo) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
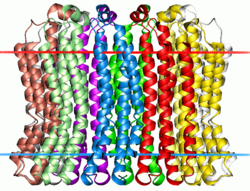 Región que atraviesa la membrana de la ATPasa de sodio de tipo V de Enterococcus hirae . Los límites de hidrocarburos calculados de la bicapa lipídica se muestran mediante puntos rojos y azules. | |||||||||
| Identificadores | |||||||||
| Símbolo | ATP-sint_C | ||||||||
| Pfam | PF00137 | ||||||||
| Interprofesional | IPR002379 | ||||||||
| PROSITIO | PDOC00526 | ||||||||
| SCOP2 | 1aty / ALCANCE / SUPFAM | ||||||||
| |||||||||
| V-ATPasa, subunidad C (V1) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura cristalina de la subunidad C (vma5p) de la v-atpasa de la levadura | |||||||||
| Identificadores | |||||||||
| Símbolo | V-ATPasa_C | ||||||||
| Pfam | PF03223 | ||||||||
| Interprofesional | IPR004907 | ||||||||
| SCOP2 | 1u7l / ALCANCE / SUPFAM | ||||||||
| |||||||||
| V-ATPasa, subunidad I/a | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | V_ATPasa_I | ||||||||
| Pfam | PF01496 | ||||||||
| Interprofesional | IPR002490 | ||||||||
| SCOP2 | 3rrk / ALCANCE / SUPFAM | ||||||||
| Base de datos de datos termodinámica | 3.A.2 | ||||||||
| |||||||||
| V-ATPasa, subunidad E | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | vATP-sint_E | ||||||||
| Pfam | PF01991 | ||||||||
| Clan Pfam | CL0255 | ||||||||
| Interprofesional | IPR002842 | ||||||||
| |||||||||
| V-ATPasa, subunidad d/d2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura cristalina de la subunidad C (subunidad D de levadura) de la v-atpasa | |||||||||
| Identificadores | |||||||||
| Símbolo | vATP-sint_AC39 | ||||||||
| Pfam | PF01992 | ||||||||
| Interprofesional | IPR002843 | ||||||||
| SCOP2 | 1r5z / ALCANCE / SUPFAM | ||||||||
| |||||||||
| V-ATPasa, subunidad H, N-terminal | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura cristalina de la subunidad reguladora H de la atpasa de tipo V de Saccharomyces cerevisiae | |||||||||
| Identificadores | |||||||||
| Símbolo | V-ATPasa_H_N | ||||||||
| Pfam | PF03224 | ||||||||
| Clan Pfam | CL0020 | ||||||||
| Interprofesional | IPR004908 | ||||||||
| SCOP2 | 1ho8 / ALCANCE / SUPFAM | ||||||||
| |||||||||
| V-ATPasa, subunidad G | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | V-ATPasa_G | ||||||||
| Pfam | PF03179 | ||||||||
| Clan Pfam | CL0255 | ||||||||
| Interprofesional | IPR005124 | ||||||||
| |||||||||
La ATPasa de tipo vacuolar ( V-ATPasa ) es una enzima muy conservada desde el punto de vista evolutivo y con funciones muy diversas en los organismos eucariotas . [1] Las V-ATPasas acidifican una amplia variedad de orgánulos intracelulares y bombean protones a través de las membranas plasmáticas de numerosos tipos de células. Las V-ATPasas acoplan la energía de la hidrólisis del ATP al transporte de protones a través de las membranas intracelulares y plasmáticas de las células eucariotas. Generalmente se la considera el polo opuesto de la ATP sintasa porque la ATP sintasa es un canal de protones que utiliza la energía de un gradiente de protones para producir ATP. Sin embargo, la V-ATPasa es una bomba de protones que utiliza la energía de la hidrólisis del ATP para producir un gradiente de protones.
La ATPasa de tipo Archaea ( A-ATPasa ) es un grupo relacionado de ATPasas que se encuentran en las arqueas y que a menudo funcionan como una ATP sintasa . Forma un clado V/A-ATPasa con V-ATPasa. La mayoría de los miembros de cualquiera de los dos grupos transportan protones ( H+
), pero algunos miembros han evolucionado para utilizar iones de sodio ( Na+
) en cambio.
Papeles desempeñados por las V-ATPasas
Las V-ATPasas se encuentran dentro de las membranas de muchos orgánulos, como endosomas , lisosomas y vesículas secretoras, donde desempeñan una variedad de funciones cruciales para el funcionamiento de estos orgánulos. Por ejemplo, el gradiente de protones a través de la membrana vacuolar de la levadura generado por las V-ATPasas impulsa la absorción de calcio en la vacuola a través de una enzima H+
/California2+
sistema antiportador. [2] En la transmisión sináptica en células neuronales, la V-ATPasa acidifica las vesículas sinápticas. [3] La noradrenalina ingresa a las vesículas por la V-ATPasa [ cita requerida ] .
Las V-ATPasas también se encuentran en las membranas plasmáticas de una amplia variedad de células, como las células intercaladas del riñón , los osteoclastos (células que reabsorben los huesos), los macrófagos , los neutrófilos , los espermatozoides , las células del intestino medio de los insectos y ciertas células tumorales . [4] Las V-ATPasas de la membrana plasmática están involucradas en procesos como la homeostasis del pH , el transporte acoplado y la metástasis tumoral . Las V-ATPasas en la membrana acrosómica del esperma acidifican el acrosoma . Esta acidificación activa las proteasas necesarias para perforar la membrana plasmática del óvulo . Las V-ATPasas en la membrana plasmática de los osteoclastos bombean protones a la superficie del hueso, lo que es necesario para la resorción ósea. En las células intercaladas del riñón, las V-ATPasas bombean protones a la orina , lo que permite la reabsorción de bicarbonato en la sangre. Además, otra variedad de procesos biológicos, como la administración de toxinas, la entrada viral, la orientación a la membrana, la apoptosis, la regulación del pH citoplasmático, el proceso proteolítico y la acidificación de los sistemas intracelulares, son funciones importantes de las V-ATPasas. [5]
Las V-ATPasas también desempeñan un papel importante en el desarrollo de la morfogénesis celular. La alteración del gen vma-1, que codifica la subunidad catalítica (A) de la enzima, afecta gravemente la tasa de crecimiento, la diferenciación y la capacidad de producir esporas viables en el hongo Neurospora crassa. [6]
Estructura
La V-ATPasa de levadura es la mejor caracterizada. Se han identificado al menos trece subunidades que forman un complejo de V-ATPasa funcional, que consta de dos dominios. Las subunidades pertenecen al dominio V o (subunidades asociadas a la membrana, letras minúsculas en la figura) o al dominio V 1 (subunidades asociadas periféricamente, letras mayúsculas en la figura).
El dominio V 1 incluye ocho subunidades, AH, con tres copias de las subunidades catalíticas A y B, tres copias de las subunidades del estator E y G, y una copia de las subunidades reguladoras C y H. Además, el dominio V 1 también contiene las subunidades D y F, que forman un eje central del rotor. [7] El dominio V 1 contiene isoformas de subunidades específicas de tejido que incluyen B, C, E y G. Las mutaciones en la isoforma B1 dan como resultado la enfermedad humana acidosis tubular renal distal y sordera neurosensorial.
El dominio V o contiene seis subunidades diferentes, a, d, c, c', c", y e, y la estequiometría del anillo c sigue siendo un tema de debate, ya que se postula un decámero para la V-ATPasa del gusano cuerno del tabaco ( Manduca sexta ). El dominio V o de los mamíferos contiene isoformas específicas de tejido para las subunidades a y d, mientras que la V-ATPasa de levadura contiene dos isoformas de subunidades específicas de orgánulos de a, Vph1p y Stv1p. Las mutaciones en la isoforma a3 dan lugar a la enfermedad humana osteopetrosis maligna infantil , y las mutaciones en la isoforma a4 dan lugar a acidosis tubular renal distal, en algunos casos con sordera neurosensorial.
El dominio V 1 es responsable de la hidrólisis de ATP, mientras que el dominio V o es responsable de la translocación de protones. La hidrólisis de ATP en los sitios de unión de nucleótidos catalíticos en la subunidad A impulsa la rotación de un tallo central compuesto por las subunidades D y F, que a su vez impulsa la rotación de un barril de subunidades c en relación con la subunidad a. La estructura compleja de la V-ATPasa se ha revelado a través de la estructura de los complejos M. Sexta y Yeast que se resolvieron mediante crio-EM de partícula única y tinción negativa, respectivamente. [8] [9] [10] Estas estructuras han revelado que la V-ATPasa tiene una red de 3 estatores, unidos por un collar de densidad formado por las subunidades C, H y a, que, aunque dividen los dominios V 1 y V o , no interactúan con el eje del rotor central formado por las subunidades F, D y d. La rotación de este eje central del rotor, causada por la hidrólisis del ATP dentro de los dominios catalíticos AB, da como resultado el movimiento del barril de subunidades c más allá de la subunidad a, lo que impulsa el transporte de protones a través de la membrana. Johnson propuso una estequiometría de dos protones translocados por cada ATP hidrolizado. [11]
Además de las subunidades estructurales de la V-ATPasa de levadura, se han identificado proteínas asociadas que son necesarias para el ensamblaje. Estas proteínas asociadas son esenciales para el ensamblaje del dominio V o y se denominan Vma12p, Vma21p y Vma22p. [12] [13] [14] [15] Dos de las tres proteínas, Vma12p y Vma22p, forman un complejo que se une transitoriamente a Vph1p (subunidad a) para ayudar a su ensamblaje y maduración. [14] [16] [17] [18] Vma21p coordina el ensamblaje de las subunidades V o y escolta el dominio V o hacia las vesículas para su transporte al Golgi . [19]
V1
El dominio V 1 de la V-ATPasa es el sitio de hidrólisis de ATP. A diferencia de V o , el dominio V 1 es hidrófilo. [5] Este dominio soluble consta de un hexámero de subunidades A y B alternadas, un rotor central D, estatores periféricos G y E, y subunidades reguladoras C y H. La hidrólisis de ATP impulsa un cambio conformacional en las seis interfaces A|B y con ello la rotación del rotor central D. A diferencia de la ATP sintasa, el dominio V 1 no es una ATPasa activa cuando se disocia.
| Subunidad | Gen humano | Nota |
|---|---|---|
| A, B | ATP6V1A , ATP6V1B1 , ATP6V1B2 | Hexámero catalítico. |
| do | ATP6V1C1 , ATP6V1C2 | |
| D | ATP6V1D | Tallo del rotor central, responsable de la especificidad iónica. |
| P.EJ | ATP6V1E1 , ATP6V1E2 , ATP6V1G1 , ATP6V1G2 , ATP6V1G3 | |
| F | ATP6V1F | |
| yo | ATP6V1H |
Subunidad C
La V-ATPasa (Vacuolar-ATPasa) C representa la subunidad C terminal que forma parte del complejo V1 y está localizada en la interfaz entre los complejos V1 y Vo. [21]
Función de la subunidad C
La subunidad C desempeña un papel esencial en el control del ensamblaje de la V-ATPasa, actuando como un estator flexible que mantiene unidos los sectores catalítico (V1) y de membrana (VO) de la enzima. [22] La liberación de la subunidad C del complejo ATPasa da como resultado la disociación de los subcomplejos V1 y Vo, que es un mecanismo importante para controlar la actividad de la V-ATPasa en las células . Esencialmente, al crear un alto gradiente electroquímico y un pH bajo, esto impulsa a la enzima a crear más ATP.
Subunidades E, G
Estas subunidades relacionadas forman el tallo o tallos de la A/V-ATPasa. Son importantes para el ensamblaje y pueden funcionar como varillas de empuje en la actividad. E tiene una tapa para conectarse a A/B, mientras que G no la tiene. [20] Es probable que hayan evolucionado a partir de una sola proteína por duplicación genética . [23]
Subunidad H
La subunidad H sólo participa en la actividad y no en el ensamblaje. Esta subunidad también actúa como inhibidor de las subunidades V1 libres; detiene la hidrólisis de ATP cuando V1 y Vo se disocian. [24]
Vo
El dominio V o es responsable de la translocación de protones. A diferencia de la ATP sintasa de tipo F , el dominio V o generalmente transporta protones en contra de su propio gradiente de concentración. La rotación del dominio V o transporta los protones en movimiento coordinado con el dominio V 1 , que es responsable de la hidrólisis del ATP. El dominio V o es hidrófobo y está compuesto por varias subunidades disociables. [5] Estas subunidades están presentes en el dominio V o para convertirlo en una translocasa de protones funcional; se describen a continuación.
| Subunidad | Gen humano | Nota |
|---|---|---|
| ai | ATP6V0A1 , ATP6V0A2 , ATP6V0A4 | |
| do | ATP6V0B , ATP6V0C | Anillo de tamaño variado. |
| corriente continua | ATP6V0D1 , ATP6V0D2 | |
| mi | ATP6V0E1 , ATP6V0E2 | Proteína de ensamblaje hidrofóbica de 9 kDa. |
| AC45/S1 | ATP6AP1 | Subunidad accesoria |
| S2 | ATP6AP2 | Subunidad accesoria |
Subunidad a/I
La subunidad de 116 kDa (o subunidad a) y la subunidad I se encuentran en el complejo Vo o Ao de las ATPasas V o A, respectivamente. La subunidad de 116 kDa es una glicoproteína transmembrana necesaria para el ensamblaje y la actividad de transporte de protones del complejo ATPasa. Existen varias isoformas de la subunidad de 116 kDa, que brindan un papel potencial en la orientación y regulación diferencial de la ATPasa V para orgánulos específicos.
La función de la subunidad de 116 kDa no está definida, pero su estructura prevista consta de 6 a 8 sectores transmembranosos, lo que sugiere que puede funcionar de manera similar a la subunidad a de FO.
Subunidad d/C
La subunidad d de las V-ATPasas, llamada subunidad C de las A-ATPasas, es parte del complejo Vo. Se coloca en el medio del anillo c, por lo que se cree que funciona como un rotor. Existen dos versiones de esta subunidad en eucariotas, d/d1 y d2. [25]
En los mamíferos, d1 ( ATP6V0D1 ) es la versión expresada de forma ubicua y d2 ( ATP6V0D2 ) se expresa solo en tipos de células específicos. [25]
Subunidad c
De manera similar a la ATP sintasa de tipo F, la región transmembrana de la V-ATPasa incluye un anillo de subunidades que atraviesan la membrana y que son las principales responsables de la translocación de protones. Sin embargo, a diferencia de la ATP sintasa de tipo F, la V-ATPasa tiene múltiples subunidades relacionadas en el anillo C; en hongos como la levadura hay tres subunidades relacionadas (de estequiometría variada) y en la mayoría de los demás eucariotas hay dos.
Ensamblaje de V-ATPasa
Las V-ATPasas de levadura no se ensamblan cuando se elimina alguno de los genes que codifican las subunidades, excepto las subunidades H y c". [26] [27] [28] Sin la subunidad H, la V-ATPasa ensamblada no está activa, [13] [29] y la pérdida de la subunidad c" da como resultado el desacoplamiento de la actividad enzimática. [27]
Los mecanismos precisos por los cuales se ensamblan las V-ATPasas aún son controvertidos, y la evidencia sugiere dos posibilidades diferentes. El análisis mutacional y los ensayos in vitro han demostrado que los dominios V o y V 1 preensamblados pueden combinarse para formar un complejo en un proceso llamado ensamblaje independiente. El respaldo del ensamblaje independiente incluye los hallazgos de que el dominio V o ensamblado se puede encontrar en la vacuola en ausencia del dominio V 1 , mientras que los dominios V 1 libres se pueden encontrar en el citoplasma y no en la vacuola . [30] [31] En contraste, los experimentos de pulso-persecución in vivo han revelado interacciones tempranas entre las subunidades V o y V 1 , para ser específicos, las subunidades a y B, lo que sugiere que las subunidades se agregan de manera escalonada para formar un solo complejo en un proceso de ensamblaje concertado. [32]
Evolución de la V-ATPasa
Una técnica relativamente nueva llamada resurrección de genes ancestrales ha arrojado nueva luz sobre la historia evolutiva de la V-ATPasa. Se ha demostrado cómo la estructura de la V-ATPasa de la forma ancestral que consiste en dos proteínas diferentes evoluciona a la versión de hongos con tres proteínas diferentes. [33] [34] [35] La ATPasa de tipo V es similar a la ATP sintasa arqueal (llamada) de tipo A, un hecho que apoya un origen arqueológico de los eucariotas (como la hipótesis del eocitos , ver también Lokiarchaeota ). La ocurrencia excepcional de algunos linajes de arqueas con tipo F y de algunos linajes de bacterias con ATPasa de tipo A respectivamente se considera como resultado de la transferencia horizontal de genes . [36]
Regulación de la actividad de la V-ATPasa
Se sabe que las V-ATPasas son inhibidas específicamente por antibióticos macrólidos, como la concanamicina (CCA) y la balifomicina A 1 . [37] La regulación in vivo de la actividad de la V-ATPasa se logra mediante la disociación reversible del dominio V 1 del dominio V o . Después del ensamblaje inicial, tanto las V-ATPasas de Manduca sexta de insecto como las de levadura pueden desensamblarse reversiblemente en los dominios V o y V 1 libres después de una privación de glucosa de 2 a 5 minutos. [30] El desensamblaje reversible puede ser un mecanismo general de regulación de la actividad de la V-ATPasa, ya que existe en levaduras e insectos. Se propone que el reensamblaje sea ayudado por un complejo denominado RAVE (regulador de H+
-ATPasa de membranas vacuolares y endosómicas). [38] El desmontaje y reensamblaje de las V-ATPasas no requiere la síntesis de nuevas proteínas, pero sí necesita una red microtubular intacta. [39]
Enfermedades humanas
Osteopetrosis
La osteopetrosis es un nombre genérico que representa un grupo de enfermedades hereditarias en las que hay un defecto en la resorción ósea osteoclástica . Tanto la osteopetrosis dominante como la recesiva se producen en humanos. [40] [41] La osteopetrosis autosómica dominante muestra síntomas leves en adultos que experimentan fracturas óseas frecuentes debido a huesos frágiles. [40] Una forma más grave de osteopetrosis se denomina osteopetrosis maligna infantil autosómica recesiva. [41] [42] [43] Se han identificado tres genes que son responsables de la osteopetrosis recesiva en humanos. Todos ellos están directamente involucrados en las vías de generación y secreción de protones que son esenciales para la resorción ósea. Un gen es la anhidrasa carbónica II (CAII), que, cuando muta, causa osteopetrosis con acidosis tubular renal (tipo 3). [44] Las mutaciones en el gen del canal de cloruro ClC7 también conducen a osteopetrosis dominante y recesiva. [40] Aproximadamente el 50% de los pacientes con osteopetrosis maligna infantil recesiva tienen mutaciones en la isoforma a3 de la subunidad V-ATPasa. [42] [45] [46] En humanos, se han identificado 26 mutaciones en la isoforma a3 de la subunidad V-ATPasa, que se encuentra en los osteoclastos, que dan lugar a la enfermedad ósea autosómica recesiva osteopetrosis. [42] [41] [45] [47]
Acidosis tubular renal distal (ATRd)
La importancia de la actividad de la V-ATPasa en la secreción renal de protones se destaca por la enfermedad hereditaria acidosis tubular renal distal . En todos los casos, la acidosis tubular renal resulta de una falla de los mecanismos renales normales que regulan el pH sistémico. Hay cuatro tipos de acidosis tubular renal. El tipo 1 es la acidosis tubular renal distal y resulta de una falla del conducto colector cortical para acidificar la orina por debajo de un pH de 5. [48] Algunos pacientes con dRTA autosómica recesiva también tienen pérdida auditiva neurosensorial . [49] La herencia de este tipo de RTA resulta de mutaciones en la subunidad V-ATPasa isoforma B1 o isoforma a4 o mutaciones de la banda 3 (también llamada AE1), un intercambiador Cl-/HCO3-. [49] [50] [51] Doce mutaciones diferentes en la isoforma B1 de la V-ATPasa [52] y veinticuatro mutaciones diferentes en a4 conducen a dRTA. [52] [49] Los estudios de reacción en cadena de la polimerasa con transcripción inversa han demostrado la expresión de la subunidad a4 en la célula intercalada del riñón y en la cóclea . [52] La dRTA causada por mutaciones en el gen de la subunidad a4 en algunos casos puede estar asociada con sordera debido a una falla en la acidificación normal de la endolinfa del oído interno . [51]
Miopatía ligada al cromosoma X con autofagia excesiva (XMEA)
La miopatía ligada al cromosoma X con autofagia excesiva es una enfermedad genética rara que resulta de mutaciones en el gen VMA21. [53] La enfermedad comienza en la infancia y provoca una debilidad muscular de progresión lenta, que suele comenzar en las piernas, y algunos pacientes pueden llegar a necesitar asistencia en silla de ruedas con la edad avanzada. La proteína Vma21 ayuda al ensamblaje de la V-ATPasa, y las mutaciones asociadas a XMEA provocan una disminución de la actividad de la V-ATPasa y un aumento del pH lisosomal . [53]
Nomenclatura
El término V o tiene una letra minúscula "o" (no el número "cero") en subíndice. La "o" representa oligomicina , que se une a la región homóloga de la F-ATPasa . Vale la pena señalar que las notaciones de genes humanos en NCBI lo designan como "cero" en lugar de la letra "o". Por ejemplo, el gen para la subunidad c humana de Vo aparece en la base de datos de genes de NCBI como "ATP6V0C" (con un cero), en lugar de "ATP6VOC" (con una "o"). Muchas publicaciones también cometen este error.
Véase también
Referencias
- ^ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H (enero de 2000). "La biología celular de la generación de fuerza protónica por V-ATPases". The Journal of Experimental Biology . 203 (Pt 1): 89–95. doi :10.1242/jeb.203.1.89. PMID 10600677.
- ^ Ohya Y, Umemoto N, Tanida I, Ohta A, Iida H, Anraku Y (julio de 1991). "Los mutantes cls sensibles al calcio de Saccharomyces cerevisiae que muestran un fenotipo Pet- son atribuibles a defectos en la actividad de la H(+)-ATPasa de la membrana vacuolar". The Journal of Biological Chemistry . 266 (21): 13971–7. doi : 10.1016/S0021-9258(18)92798-5 . PMID 1830311.
- ^ Wienisch M, Klingauf J (agosto de 2006). "Las proteínas vesiculares exocitosis y posteriormente recuperadas por endocitosis compensatoria no son idénticas". Nature Neuroscience . 9 (8): 1019–27. doi :10.1038/nn1739. hdl : 11858/00-001M-0000-0012-E436-F . PMID 16845386. S2CID 12808314.
- ^ Izumi H, Torigoe T, Ishiguchi H, Uramoto H, Yoshida Y, Tanabe M, Ise T, Murakami T, Yoshida T, Nomoto M, Kohno K (diciembre de 2003). "Reguladores de pH celular: objetivos moleculares potencialmente prometedores para la quimioterapia contra el cáncer". Cancer Treatment Reviews . 29 (6): 541–9. doi :10.1016/S0305-7372(03)00106-3. PMID 14585264.
- ^ abc Emma B, Forest O, Barry B (junio de 1997). "Las mutaciones de pma-1, el gen que codifica la H+ATPasa de la membrana plasmática de Neurospora crassa, suprimen la inhibición del crecimiento por concanamicina A, un inhibidor específico de las ATPasas vacuolares". The Journal of Biological Chemistry . 272 (23): 14776–14786. doi : 10.1074/jbc.272.23.14776 . PMID 9169444. S2CID 29865381.
- ^ Bowman, EJ y Bowman, BJ (2000). Función celular de la V-ATPasa en Neurospora crassa: análisis de mutantes resistentes a la concanamicina o que carecen de la subunidad catalítica A. The Journal of experimental biology, 203(Pt 1), 97–106.
- ^ Kitagawa N, Mazon H, Heck AJ, Wilkens S (febrero de 2008). "Estequiometría de las subunidades periféricas E y G del tallo de la V1-ATPasa de levadura determinada por espectrometría de masas". The Journal of Biological Chemistry . 283 (6): 3329–37. doi : 10.1074/jbc.M707924200 . PMID 18055462. S2CID 27627066.
- ^ Muench SP, Huss M, Song CF, Phillips C, Wieczorek H, Trinick J, Harrison MA (marzo de 2009). "La criomicroscopía electrónica del motor de la ATPasa vacuolar revela su complejidad mecánica y regulatoria". Journal of Molecular Biology . 386 (4): 989–99. doi :10.1016/j.jmb.2009.01.014. PMID 19244615.
- ^ Diepholz M, Börsch M, Böttcher B (octubre de 2008). "Organización estructural de la V-ATPasa y sus implicaciones para el ensamblaje y desensamblaje regulatorios". Biochemical Society Transactions . 36 (Pt 5): 1027–31. doi :10.1042/BST0361027. PMID 18793183. S2CID 23852611.
- ^ Zhang Z, Zheng Y, Mazon H, Milgrom E, Kitagawa N, Kish-Trier E, Heck AJ, Kane PM, Wilkens S (diciembre de 2008). "Estructura de la ATPasa vacuolar de la levadura". The Journal of Biological Chemistry . 283 (51): 35983–95. doi : 10.1074/jbc.M805345200 . PMC 2602884 . PMID 18955482.
- ^ Johnson RG, Beers MF, Scarpa A (septiembre de 1982). "H+ ATPasa de gránulos cromafines. Cinética, regulación y estequiometría". The Journal of Biological Chemistry . 257 (18): 10701–7. doi : 10.1016/S0021-9258(18)33879-1 . PMID 6213624.
- ^ Hirata R, Umemoto N, Ho MN, Ohya Y, Stevens TH, Anraku Y (enero de 1993). "VMA12 es esencial para el ensamblaje de las subunidades de la H(+)-ATPasa vacuolar en la membrana vacuolar en Saccharomyces cerevisiae". The Journal of Biological Chemistry . 268 (2): 961–7. doi : 10.1016/S0021-9258(18)54027-8 . PMID 8419376.
- ^ ab Ho MN, Hirata R, Umemoto N, Ohya Y, Takatsuki A, Stevens TH, Anraku Y (agosto de 1993). "VMA13 codifica una subunidad vacuolar de H(+)-ATPasa de 54 kDa necesaria para la actividad, pero no para el ensamblaje, del complejo enzimático en Saccharomyces cerevisiae". The Journal of Biological Chemistry . 268 (24): 18286–92. doi : 10.1016/S0021-9258(17)46842-6 . PMID 8349704.
- ^ ab Hill KJ, Stevens TH (septiembre de 1994). "Vma21p es una proteína de membrana de levadura retenida en el retículo endoplásmico por un motivo de di-lisina y es necesaria para el ensamblaje del complejo vacuolar H(+)-ATPasa". Biología molecular de la célula . 5 (9): 1039–50. doi :10.1091/mbc.5.9.1039. PMC 301125 . PMID 7841520.
- ^ Jackson DD, Stevens TH (octubre de 1997). "VMA12 codifica una proteína del retículo endoplasmático de levadura necesaria para el ensamblaje de la H+-ATPasa vacuolar". The Journal of Biological Chemistry . 272 (41): 25928–34. doi : 10.1074/jbc.272.41.25928 . PMID 9325326. S2CID 38400074.
- ^ Hill KJ, Stevens TH (septiembre de 1995). "Vma22p es una nueva proteína asociada al retículo endoplasmático necesaria para el ensamblaje del complejo H(+)-ATPasa vacuolar de levadura". The Journal of Biological Chemistry . 270 (38): 22329–36. doi : 10.1074/jbc.270.38.22329 . PMID 7673216. S2CID 34639779.
- ^ Graham LA, Hill KJ, Stevens TH (julio de 1998). "El ensamblaje de la H+-ATPasa vacuolar de levadura ocurre en el retículo endoplasmático y requiere un complejo de ensamblaje Vma12p/Vma22p". The Journal of Cell Biology . 142 (1): 39–49. doi :10.1083/jcb.142.1.39. PMC 2133036 . PMID 9660861.
- ^ Graham LA, Flannery AR, Stevens TH (agosto de 2003). "Estructura y ensamblaje de la V-ATPasa de levadura". Journal of Bioenergetics and Biomembranes . 35 (4): 301–12. doi :10.1023/A:1025772730586. PMID 14635776. S2CID 37806912.
- ^ Malkus P, Graham LA, Stevens TH, Schekman R (noviembre de 2004). "Función de Vma21p en el ensamblaje y transporte de la ATPasa vacuolar de la levadura". Biología molecular de la célula . 15 (11): 5075–91. doi :10.1091/mbc.E04-06-0514. PMC 524777 . PMID 15356264.
- ^ abc Stewart AG, Laming EM, Sobti M, Stock D (abril de 2014). "ATPases rotatorias: máquinas moleculares dinámicas". Current Opinion in Structural Biology . 25 : 40–8. doi : 10.1016/j.sbi.2013.11.013 . PMID 24878343.
- ^ Inoue T, Forgac M (julio de 2005). "La reticulación mediada por cisteína indica que la subunidad C de la V-ATPasa está muy próxima a las subunidades E y G del dominio V1 y a la subunidad a del dominio V0". The Journal of Biological Chemistry . 280 (30): 27896–903. doi : 10.1074/jbc.M504890200 . PMID 15951435. S2CID 23648833.
- ^ Drory O, Frolow F, Nelson N (diciembre de 2004). "La estructura cristalina de la subunidad C de la V-ATPasa de levadura revela su función de estator". EMBO Reports . 5 (12): 1148–52. doi :10.1038/sj.embor.7400294. PMC 1299189 . PMID 15540116.
- ^ Imada K, Minamino T, Uchida Y, Kinoshita M, Namba K (marzo de 2016). "Información sobre la exportación de flagelos tipo III revelada por la compleja estructura de la ATPasa tipo III y su regulador". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (13): 3633–8. Bibcode :2016PNAS..113.3633I. doi : 10.1073/pnas.1524025113 . PMC 4822572 . PMID 26984495.
- ^ Jefferies KC, Forgac M (febrero de 2008). "La subunidad H de la ATPasa vacuolar (H+) inhibe la hidrólisis de ATP por el dominio V1 libre mediante interacción con la subunidad rotatoria F". The Journal of Biological Chemistry . 283 (8): 4512–9. doi : 10.1074/jbc.M707144200 . PMC 2408380 . PMID 18156183.
- ^ ab Toei M, Saum R, Forgac M (junio de 2010). "Regulación y función de isoformas de las V-ATPases". Bioquímica . 49 (23): 4715–23. doi :10.1021/bi100397s. PMC 2907102 . PMID 20450191.
- ^ Forgac M (enero de 1999). "La H+-ATPasa vacuolar de vesículas revestidas de clatrina es inhibida reversiblemente por S-nitrosoglutatión". The Journal of Biological Chemistry . 274 (3): 1301–5. doi : 10.1074/jbc.274.3.1301 . PMID 9880499. S2CID 21784089.
- ^ ab Whyteside G, Gibson L, Scott M, Finbow ME (junio de 2005). "El ensamblaje de la H+-ATPasa vacuolar de la levadura y la hidrólisis de ATP ocurren en ausencia de la subunidad c"". FEBS Letters . 579 (14): 2981–5. doi : 10.1016/j.febslet.2005.04.049 . PMID 15907326. S2CID 32086585.
- ^ Stevens TH, Forgac M (1997). "Estructura, función y regulación de la (H+)-ATPasa vacuolar". Revisión anual de biología celular y del desarrollo . 13 : 779–808. doi :10.1146/annurev.cellbio.13.1.779. PMID 9442887.
- ^ Parra KJ, Keenan KL, Kane PM (julio de 2000). "La subunidad H (Vma13p) de la V-ATPasa de levadura inhibe la actividad ATPasa de los complejos V1 citosólicos". The Journal of Biological Chemistry . 275 (28): 21761–7. doi : 10.1074/jbc.M002305200 . PMID 10781598. S2CID 46127337.
- ^ ab Kane PM (julio de 1995). "Desensamblaje y reensamblaje de la H(+)-ATPasa vacuolar de levadura in vivo". The Journal of Biological Chemistry . 270 (28): 17025–32. doi : 10.1016/S0021-9258(17)46944-4 . PMID 7622524.
- ^ Sumner JP, Dow JA, Earley FG, Klein U, Jäger D, Wieczorek H (marzo de 1995). "Regulación de la actividad de la V-ATPasa de la membrana plasmática mediante disociación de subunidades periféricas". The Journal of Biological Chemistry . 270 (10): 5649–53. doi : 10.1074/jbc.270.10.5649 . PMID 7890686. S2CID 38963775.
- ^ Kane PM, Tarsio M, Liu J (junio de 1999). "Primeros pasos en el ensamblaje de la H+-ATPasa vacuolar de levadura". The Journal of Biological Chemistry . 274 (24): 17275–83. doi : 10.1074/jbc.274.24.17275 . PMID 10358087. S2CID 42610386.
- ^ Pearson H (9 de enero de 2012). "Resucitar proteínas extintas muestra cómo evoluciona una máquina". Blog de noticias de Nature.com .
- ^ Finnigan GC, Hanson-Smith V, Stevens TH, Thornton JW (enero de 2012). "Evolución de una mayor complejidad en una máquina molecular". Nature . 481 (7381): 360–4. Bibcode :2012Natur.481..360F. doi :10.1038/nature10724. PMC 3979732 . PMID 22230956.
- ^ Vista instantánea de la máquina molecular de la V-ATPasa: animales vs. hongos Archivado el 28 de abril de 2012 en Wayback Machine , Universidad de Oregón (consultado el 11 de enero de 2012)
- ^ Hilario E, Gogarten JP (1993). "Transferencia horizontal de genes de ATPasa: el árbol de la vida se convierte en una red de vida" (PDF) . Bio Systems . 31 (2–3): 111–9. doi :10.1016/0303-2647(93)90038-E. PMID 8155843.
- ^ Bowman EJ, O'Neill FJ, Bowman BJ (junio de 1997). "Las mutaciones de pma-1, el gen que codifica la H+-ATPasa de la membrana plasmática de Neurospora crassa, suprimen la inhibición del crecimiento por concanamicina A, un inhibidor específico de las ATPasas vacuolares". The Journal of Biological Chemistry . 272 (23): 14776–86. doi : 10.1074/jbc.272.23.14776 . PMID 9169444. S2CID 29865381.
- ^ Kane PM, Smardon AM (agosto de 2003). "Ensamblaje y regulación de la H+-ATPasa vacuolar de la levadura". Journal of Bioenergetics and Biomembranes . 35 (4): 313–21. doi :10.1023/A:1025724814656. PMID 14635777. S2CID 7535580.
- ^ Holliday LS, Lu M, Lee BS, Nelson RD, Solivan S, Zhang L, Gluck SL (octubre de 2000). "El dominio amino-terminal de la subunidad B de la H+-ATPasa vacuolar contiene un sitio de unión a actina filamentosa". The Journal of Biological Chemistry . 275 (41): 32331–7. doi : 10.1074/jbc.M004795200 . PMID 10915794. S2CID 2601649.
- ^ abc Michigami T, Kageyama T, Satomura K, Shima M, Yamaoka K, Nakayama M, Ozono K (febrero de 2002). "Nuevas mutaciones en la subunidad a3 de la H(+)-adenosina trifosfatasa vacuolar en un paciente japonés con osteopetrosis maligna infantil". Bone . 30 (2): 436–9. doi :10.1016/S8756-3282(01)00684-6. PMID 11856654.
- ^ abc Frattini A, Orchard PJ, Sobacchi C, Giliani S, Abinun M, Mattsson JP, Keeling DJ, Andersson AK, Wallbrandt P, Zecca L, Notarangelo LD, Vezzoni P, Villa A (julio de 2000). "Los defectos en la subunidad TCIRG1 de la bomba de protones vacuolar son responsables de un subconjunto de osteopetrosis autosómica recesiva humana". Nature Genetics . 25 (3): 343–6. doi :10.1038/77131. PMID 10888887. S2CID 21316081.
- ^ abc Sobacchi C, Frattini A, Orchard P, Porras O, Tezcan I, Andolina M, et al. (agosto de 2001). "El espectro mutacional de la osteopetrosis autosómica recesiva maligna humana". Genética molecular humana . 10 (17): 1767–73. doi : 10.1093/hmg/10.17.1767 . PMID 11532986.
- ^ Fasth A, Porras O (1999). "Osteopetrosis maligna humana: fisiopatología, tratamiento y función del trasplante de médula ósea". Pediatric Transplantation . 3 (Supl 1): 102–7. doi :10.1034/j.1399-3046.1999.00063.x. PMID 10587979. S2CID 31745272.
- ^ Sly WS, Hewett-Emmett D, Whyte MP, Yu YS, Tashian RE (mayo de 1983). "Deficiencia de anhidrasa carbónica II identificada como el defecto primario en el síndrome autosómico recesivo de osteopetrosis con acidosis tubular renal y calcificación cerebral". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 80 (9): 2752–6. Bibcode :1983PNAS...80.2752S. doi : 10.1073/pnas.80.9.2752 . PMC 393906 . PMID 6405388.
- ^ ab Kornak U, Schulz A, Friedrich W, Uhlhaas S, Kremens B, Voit T, Hasan C, Bode U, Jentsch TJ, Kubisch C (agosto de 2000). "Las mutaciones en la subunidad a3 de la H (+) -ATPasa vacuolar provocan osteopetrosis maligna infantil". Genética Molecular Humana . 9 (13): 2059–63. doi : 10.1093/hmg/9.13.2059 . PMID 10942435.
- ^ Frattini A, Pangrazio A, Susani L, Sobacchi C, Mirolo M, Abinun M, Andolina M, Flanagan A, Horwitz EM, Mihci E, Notarangelo LD, Ramenghi U, Teti A, Van Hove J, Vujic D, Young T, Albertini A, Orchard PJ, Vezzoni P, Villa A (octubre de 2003). "Las mutaciones del canal de cloruro ClCN7 son responsables de la osteopetrosis recesiva, dominante e intermedia grave". Revista de investigación de huesos y minerales . 18 (10): 1740–7. doi : 10.1359/jbmr.2003.18.10.1740 . PMID 14584882. S2CID 20966489.
- ^ Susani L, Pangrazio A, Sobacchi C, Taranta A, Mortier G, Savarirayan R, Villa A, Orchard P, Vezzoni P, Albertini A, Frattini A, Pagani F (septiembre de 2004). "Ostepetrosis recesiva dependiente de TCIRG1: análisis de mutaciones, identificación funcional de los defectos de empalme y rescate in vitro mediante snRNA U1". Mutación humana . 24 (3): 225–35. doi : 10.1002/humu.20076 . PMID 15300850. S2CID 31788054.
- ^ Alper SL (2002). "Enfermedades genéticas de los transportadores ácido-base". Revisión anual de fisiología . 64 : 899–923. doi :10.1146/annurev.physiol.64.092801.141759. PMID 11826292.
- ^ abc Karet FE, Finberg KE, Nelson RD, Nayir A, Mocan H, Sanjad SA, et al. (enero de 1999). "Las mutaciones en el gen que codifica la subunidad B1 de la H+-ATPasa causan acidosis tubular renal con sordera neurosensorial". Nature Genetics . 21 (1): 84–90. doi :10.1038/5022. PMID 9916796. S2CID 34262548.
- ^ Karet FE, Gainza FJ, Györy AZ, Unwin RJ, Wrong O, Tanner MJ, et al. (mayo de 1998). "Las mutaciones en el gen intercambiador de cloruro-bicarbonato AE1 causan acidosis tubular renal distal autosómica dominante pero no autosómica recesiva". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 95 (11): 6337–42. Bibcode :1998PNAS...95.6337K. doi : 10.1073/pnas.95.11.6337 . PMC 27686 . PMID 9600966.
- ^ ab Stehberger PA, Schulz N, Finberg KE, Karet FE, Giebisch G, Lifton RP, Geibel JP, Wagner CA (diciembre de 2003). "Localización y regulación de la subunidad H+-ATPasa vacuolar ATP6V0A4 (a4) defectuosa en una forma hereditaria de acidosis tubular renal distal". Revista de la Sociedad Americana de Nefrología . 14 (12): 3027–38. doi : 10.1097/01.ASN.0000099375.74789.AB . PMID 14638902.
- ^ abc Stover EH, Borthwick KJ, Bavalia C, Eady N, Fritz DM, Rungroj N, et al. (noviembre de 2002). "Nuevas mutaciones ATP6V1B1 y ATP6V0A4 en la acidosis tubular renal distal autosómica recesiva con nueva evidencia de pérdida auditiva". Journal of Medical Genetics . 39 (11): 796–803. doi :10.1136/jmg.39.11.796. PMC 1735017 . PMID 12414817.
- ^ ab Ramachandran N, Munteanu I, Wang P, Ruggieri A, Rilstone JJ, Israelian N, et al. (marzo de 2013). "La deficiencia de VMA21 impide el ensamblaje de la ATPasa vacuolar y causa miopatía vacuolar autofágica". Acta Neuropathologica . 125 (3): 439–57. doi :10.1007/s00401-012-1073-6. PMID 23315026. S2CID 20528180.
Enlaces externos
- V-Type+ATPasa en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.