Diferenciación celular


La diferenciación celular es el proceso en el cual una célula madre cambia de un tipo a uno diferenciado. [2] [3] Por lo general, la célula cambia a un tipo más especializado. La diferenciación ocurre varias veces durante el desarrollo de un organismo multicelular a medida que cambia de un simple cigoto a un sistema complejo de tejidos y tipos de células. La diferenciación continúa en la edad adulta a medida que las células madre adultas se dividen y crean células hijas completamente diferenciadas durante la reparación de tejidos y durante la renovación celular normal. Parte de la diferenciación ocurre en respuesta a la exposición a antígenos . La diferenciación cambia drásticamente el tamaño, la forma, el potencial de membrana , la actividad metabólica y la capacidad de respuesta a las señales de una célula . Estos cambios se deben en gran medida a modificaciones altamente controladas en la expresión genética y son el estudio de la epigenética . Con algunas excepciones, la diferenciación celular casi nunca implica un cambio en la secuencia de ADN en sí. Sin embargo, la composición metabólica se altera drásticamente [4] donde las células madre se caracterizan por abundantes metabolitos con estructuras altamente insaturadas cuyos niveles disminuyen con la diferenciación. Por lo tanto, diferentes células pueden tener características físicas muy diferentes a pesar de tener el mismo genoma .
Un tipo especializado de diferenciación, conocido como diferenciación terminal , es importante en algunos tejidos, incluidos el sistema nervioso de los vertebrados , el músculo estriado , la epidermis y el intestino. Durante la diferenciación terminal, una célula precursora que antes era capaz de dividirse celular abandona permanentemente el ciclo celular, desmantela la maquinaria del ciclo celular y, a menudo, expresa una serie de genes característicos de la función final de la célula (por ejemplo, miosina y actina para una célula muscular). La diferenciación puede continuar ocurriendo después de la diferenciación terminal si la capacidad y las funciones de la célula sufren más cambios.
Entre las células en división, existen múltiples niveles de potencia celular , que es la capacidad de la célula para diferenciarse en otros tipos de células. Una mayor potencia indica un mayor número de tipos de células que pueden derivarse. Una célula que puede diferenciarse en todos los tipos de células, incluido el tejido placentario, se conoce como totipotente . En los mamíferos, solo el cigoto y los blastómeros posteriores son totipotentes, mientras que en las plantas, muchas células diferenciadas pueden volverse totipotentes con técnicas de laboratorio simples. Una célula que puede diferenciarse en todos los tipos de células del organismo adulto se conoce como pluripotente . Dichas células se denominan células meristemáticas en plantas superiores y células madre embrionarias en animales, aunque algunos grupos informan la presencia de células pluripotentes adultas. La expresión inducida por virus de cuatro factores de transcripción Oct4 , Sox2 , c-Myc y Klf4 ( factores de Yamanaka ) es suficiente para crear células pluripotentes (iPS) a partir de fibroblastos adultos . [5] Una célula multipotente es aquella que puede diferenciarse en múltiples tipos de células diferentes, pero estrechamente relacionadas. [6] Las células oligopotentes son más restringidas que las multipotentes, pero aún pueden diferenciarse en unos pocos tipos de células estrechamente relacionadas. [6] Finalmente, las células unipotentes pueden diferenciarse en un solo tipo de célula, pero son capaces de auto-renovarse . [6] En citopatología , el nivel de diferenciación celular se utiliza como una medida de la progresión del cáncer . El " grado " es un marcador de qué tan diferenciada está una célula en un tumor. [7]
Tipos de células de mamíferos
Tres categorías básicas de células componen el cuerpo de los mamíferos: células germinales , células somáticas y células madre . Cada una de las aproximadamente 37,2 billones (3,72x10 13 ) de células de un ser humano adulto tiene su propia copia o copias del genoma , excepto ciertos tipos de células , como los glóbulos rojos , que carecen de núcleo en su estado completamente diferenciado. La mayoría de las células son diploides ; tienen dos copias de cada cromosoma . Estas células, llamadas células somáticas, componen la mayor parte del cuerpo humano, como las células de la piel y los músculos. Las células se diferencian para especializarse en diferentes funciones. [8]
Las células de la línea germinal son cualquier línea de células que dan origen a los gametos (óvulos y espermatozoides) y, por lo tanto, son continuas a lo largo de las generaciones. Las células madre, por otro lado, tienen la capacidad de dividirse durante períodos indefinidos y dar lugar a células especializadas. Se describen mejor en el contexto del desarrollo humano normal. [ cita requerida ]
El desarrollo comienza cuando un espermatozoide fecunda un óvulo y crea una sola célula que tiene el potencial de formar un organismo completo. En las primeras horas después de la fecundación, esta célula se divide en células idénticas. En los humanos, aproximadamente cuatro días después de la fecundación y después de varios ciclos de división celular, estas células comienzan a especializarse, formando una esfera hueca de células, llamada blastocisto . [9] El blastocisto tiene una capa externa de células, y dentro de esta esfera hueca, hay un grupo de células llamado masa celular interna . Las células de la masa celular interna pasan a formar prácticamente todos los tejidos del cuerpo humano. Aunque las células de la masa celular interna pueden formar prácticamente todos los tipos de células que se encuentran en el cuerpo humano, no pueden formar un organismo. Estas células se denominan pluripotentes . [10]
Las células madre pluripotentes experimentan una mayor especialización y se convierten en células progenitoras multipotentes que luego dan lugar a células funcionales. Entre los ejemplos de células madre y progenitoras se incluyen: [ cita requerida ]
- Células gliales radiales (células madre neurales embrionarias) que dan lugar a neuronas excitatorias en el cerebro fetal a través del proceso de neurogénesis . [11] [12] [13]
- Células madre hematopoyéticas (células madre adultas) de la médula ósea que dan lugar a glóbulos rojos, glóbulos blancos y plaquetas .
- Células madre mesenquimales (células madre adultas) de la médula ósea que dan lugar a células del estroma, células grasas y tipos de células óseas.
- Células madre epiteliales (células progenitoras) que dan origen a los distintos tipos de células de la piel.
- Células satélite musculares (células progenitoras) que contribuyen al tejido muscular diferenciado .
Una vía guiada por las moléculas de adhesión celular que constan de cuatro aminoácidos, arginina , glicina , asparagina y serina , se crea a medida que el blastómero celular se diferencia de la blástula de una sola capa a las tres capas primarias de las células germinales en los mamíferos, a saber, el ectodermo , el mesodermo y el endodermo (enumerados desde la más distal (exterior) a la proximal (interior)). El ectodermo termina formando la piel y el sistema nervioso, el mesodermo forma los huesos y el tejido muscular, y el endodermo forma los tejidos de los órganos internos.
Desdiferenciación

La desdiferenciación , o integración, es un proceso celular que se observa en las formas de vida más basales de los animales, como los gusanos y los anfibios , donde una célula diferenciada vuelve a una etapa de desarrollo anterior, generalmente como parte de un proceso regenerativo . [14] [15] La desdiferenciación también ocurre en las células vegetales. [16] Y, en el cultivo celular en el laboratorio, las células pueden cambiar de forma o pueden perder propiedades específicas como la expresión de proteínas, procesos que también se denominan desdiferenciación. [17]
Algunos plantean la hipótesis de que la desdiferenciación es una aberración que probablemente produce cánceres , [18] pero otros la explican como una parte natural de la respuesta inmune que los humanos perdieron en algún momento de la evolución.
Se ha demostrado que una molécula recién descubierta, llamada reversina , un análogo de la purina , induce la desdiferenciación en los miotubos . Estas células manifiestamente desdiferenciadas (que ahora funcionan esencialmente como células madre) podrían luego rediferenciarse en osteoblastos y adipocitos . [19]

Mecanismos

Cada tipo de célula especializada en un organismo expresa un subconjunto de todos los genes que constituyen el genoma de esa especie . Cada tipo de célula se define por su patrón particular de expresión génica regulada . La diferenciación celular es, por tanto, una transición de una célula de un tipo celular a otro e implica un cambio de un patrón de expresión génica a otro. La diferenciación celular durante el desarrollo puede entenderse como el resultado de una red reguladora de genes . Un gen regulador y sus módulos cis-reguladores son nodos en una red reguladora de genes; reciben información y crean una salida en otra parte de la red. [20] El enfoque de la biología de sistemas para la biología del desarrollo enfatiza la importancia de investigar cómo interactúan los mecanismos de desarrollo para producir patrones predecibles ( morfogénesis ). Sin embargo, recientemente se ha propuesto una visión alternativa [ ¿cuándo? ] [¿ por quién? ] . Basada en la expresión génica estocástica , la diferenciación celular es el resultado de un proceso selectivo darwiniano que ocurre entre las células. En este marco, las redes de proteínas y genes son el resultado de los procesos celulares y no su causa. [ cita requerida ]
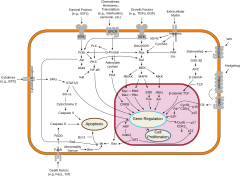
Si bien los procesos moleculares conservados evolutivamente están involucrados en los mecanismos celulares subyacentes a estos cambios, en las especies animales estos son muy diferentes de los mecanismos reguladores genéticos bien caracterizados de las bacterias , e incluso de los de los parientes unicelulares más cercanos de los animales . [21] Específicamente, la diferenciación celular en animales depende en gran medida de condensados biomoleculares de proteínas reguladoras y secuencias potenciadoras de ADN.
La diferenciación celular suele estar controlada por la señalización celular . Muchas de las moléculas de señal que transmiten información de una célula a otra durante el control de la diferenciación celular se denominan factores de crecimiento . Aunque los detalles de las vías de transducción de señales específicas varían, estas vías suelen compartir los siguientes pasos generales. Un ligando producido por una célula se une a un receptor en la región extracelular de otra célula, lo que induce un cambio conformacional en el receptor. La forma del dominio citoplasmático del receptor cambia y el receptor adquiere actividad enzimática. A continuación, el receptor cataliza reacciones que fosforilan otras proteínas y las activan. Una cascada de reacciones de fosforilación acaba activando un factor de transcripción latente o una proteína del citoesqueleto, lo que contribuye al proceso de diferenciación en la célula diana. [22] Las células y los tejidos pueden variar en competencia, su capacidad para responder a señales externas. [23]
La inducción de señales se refiere a cascadas de eventos de señalización, durante los cuales una célula o tejido envía señales a otra célula o tejido para influir en su destino de desarrollo. [23] Yamamoto y Jeffery [24] investigaron el papel del cristalino en la formación del ojo en peces que habitan en cuevas y en la superficie, un ejemplo sorprendente de inducción. [23] A través de trasplantes recíprocos, Yamamoto y Jeffery [24] descubrieron que la vesícula del cristalino de los peces de la superficie puede inducir el desarrollo de otras partes del ojo en los peces que habitan en cuevas y en la superficie, mientras que la vesícula del cristalino de los peces que habitan en cuevas no puede. [23]
Otros mecanismos importantes caen bajo la categoría de divisiones celulares asimétricas , divisiones que dan lugar a células hijas con destinos de desarrollo distintos. Las divisiones celulares asimétricas pueden ocurrir debido a determinantes citoplasmáticos maternos expresados asimétricamente o debido a la señalización. [23] En el primer mecanismo, se crean células hijas distintas durante la citocinesis debido a una distribución desigual de moléculas reguladoras en la célula madre; el citoplasma distinto que hereda cada célula hija da como resultado un patrón distinto de diferenciación para cada célula hija. Un ejemplo bien estudiado de formación de patrones por divisiones asimétricas es el patrón del eje corporal en Drosophila . Las moléculas de ARN son un tipo importante de señal de control de diferenciación intracelular. La base molecular y genética de las divisiones celulares asimétricas también se ha estudiado en algas verdes del género Volvox , un sistema modelo para estudiar cómo los organismos unicelulares pueden evolucionar a organismos multicelulares. [23] En Volvox carteri , las 16 células del hemisferio anterior de un embrión de 32 células se dividen de forma asimétrica, y cada una de ellas produce una célula hija grande y una pequeña. El tamaño de la célula al final de todas las divisiones celulares determina si se convierte en una célula germinal o somática especializada. [23] [25]
Control epigenético
Dado que cada célula, independientemente del tipo celular, posee el mismo genoma, la determinación del tipo celular debe ocurrir a nivel de expresión génica. Si bien la regulación de la expresión génica puede ocurrir a través de elementos reguladores cis y trans, incluidos el promotor y los potenciadores de un gen , surge el problema de cómo se mantiene este patrón de expresión a lo largo de numerosas generaciones de división celular . [26] Como resultado, los procesos epigenéticos desempeñan un papel crucial en la regulación de la decisión de adoptar un destino de célula madre, progenitora o madura. Esta sección se centrará principalmente en las células madre de mamíferos .
En biología de sistemas y modelado matemático de redes reguladoras de genes, se predice que la determinación del destino celular exhibirá cierta dinámica, como la convergencia del atractor (el atractor puede ser un punto de equilibrio, un ciclo límite o un atractor extraño ) u oscilatoria. [27]
Importancia del control epigenético
La primera pregunta que se puede plantear es la extensión y la complejidad del papel de los procesos epigenéticos en la determinación del destino celular. Una respuesta clara a esta pregunta se puede ver en el artículo de 2011 de Lister R, et al. [28] sobre la programación epigenómica aberrante en células madre pluripotentes inducidas humanas . Como se cree que las células madre pluripotentes inducidas (iPSC) imitan a las células madre embrionarias en sus propiedades pluripotentes, deberían existir pocas diferencias epigenéticas entre ellas. Para probar esta predicción, los autores realizaron un perfil de todo el genoma de los patrones de metilación del ADN en varias líneas de células madre embrionarias (ESC), iPSC y células progenitoras humanas.
Las células adiposas femeninas , los fibroblastos pulmonares y los fibroblastos del prepucio se reprogramaron en un estado pluripotente inducido con los genes OCT4 , SOX2 , KLF4 y MYC . Se compararon los patrones de metilación del ADN en las células madre embrionarias, las células iPSC y las células somáticas. Lister R, et al. observaron una semejanza significativa en los niveles de metilación entre las células pluripotentes embrionarias e inducidas. Alrededor del 80% de los dinucleótidos CG en las células madre embrionarias y las iPSC estaban metilados, lo mismo ocurrió con solo el 60% de los dinucleótidos CG en las células somáticas. Además, las células somáticas poseían niveles mínimos de metilación de citosina en dinucleótidos no CG, mientras que las células pluripotentes inducidas poseían niveles similares de metilación que las células madre embrionarias, entre el 0,5 y el 1,5%. Por lo tanto, en consonancia con sus respectivas actividades transcripcionales, [28] los patrones de metilación del ADN, al menos a nivel genómico, son similares entre las ESC y las iPSC.
Sin embargo, al examinar los patrones de metilación más de cerca, los autores descubrieron 1175 regiones de metilación diferencial del dinucleótido CG entre al menos una línea de células ES o iPS. Al comparar estas regiones de metilación diferencial con las regiones de metilación de citosina en las células somáticas originales, el 44-49% de las regiones metiladas diferencialmente reflejaban patrones de metilación de las respectivas células somáticas progenitoras, mientras que el 51-56% de estas regiones eran diferentes a las líneas celulares progenitoras y embrionarias. La diferenciación inducida in vitro de las líneas de iPSC vio la transmisión del 88% y el 46% de las regiones metiladas diferencialmente hiper e hipometiladas, respectivamente.
Dos conclusiones son claramente evidentes a partir de este estudio. En primer lugar, los procesos epigenéticos están muy involucrados en la determinación del destino celular , como se ve a partir de los niveles similares de metilación de citosina entre las células madre pluripotentes inducidas y las embrionarias, en consonancia con sus respectivos patrones de transcripción . En segundo lugar, los mecanismos de reprogramación (y por extensión, de diferenciación) son muy complejos y no se pueden duplicar fácilmente, como se ve por el número significativo de regiones metiladas diferencialmente entre las líneas de células ES e iPS. Ahora que estos dos puntos han sido establecidos, podemos examinar algunos de los mecanismos epigenéticos que se cree que regulan la diferenciación celular.
Mecanismos de regulación epigenética
Factores pioneros (Oct4, Sox2, Nanog)
Tres factores de transcripción, OCT4, SOX2 y NANOG (los dos primeros de los cuales se utilizan en la reprogramación de células madre pluripotentes inducidas, junto con Klf4 y c-Myc ) se expresan en gran medida en células madre embrionarias indiferenciadas y son necesarios para el mantenimiento de su pluripotencia . [29] Se cree que logran esto a través de alteraciones en la estructura de la cromatina , como la modificación de histonas y la metilación del ADN, para restringir o permitir la transcripción de genes diana. Aunque se expresan en gran medida, sus niveles requieren un equilibrio preciso para mantener la pluripotencia, cuya perturbación promoverá la diferenciación hacia diferentes linajes en función de cómo cambien los niveles de expresión genética. Se ha demostrado que la regulación diferencial de los niveles de Oct-4 y SOX2 precede a la selección del destino de la capa germinal. [30] Los niveles aumentados de Oct4 y los niveles disminuidos de Sox2 promueven un destino mesendodérmico , y Oct4 suprime activamente los genes asociados con un destino ectodérmico neuronal . De manera similar, el aumento de los niveles de Sox2 y la disminución de los niveles de Oct4 promueven la diferenciación hacia un destino ectodérmico neuronal, mientras que Sox2 inhibe la diferenciación hacia un destino mesendodérmico. Independientemente del linaje en el que las células se diferencien, se ha identificado la supresión de NANOG como un prerrequisito necesario para la diferenciación. [30]
Complejo represivo Polycomb (PRC2)
En el ámbito del silenciamiento génico , el complejo represor Polycomb 2 , una de las dos clases de la familia de proteínas del grupo Polycomb (PcG), cataliza la di- y tri-metilación de la lisina 27 de la histona H3 (H3K27me2/me3). [29] [31] [32] Al unirse al nucleosoma marcado con H3K27me2/3, PRC1 (también un complejo de proteínas de la familia PcG) cataliza la mono-ubiquitinilación de la histona H2A en la lisina 119 (H2AK119Ub1), bloqueando la actividad de la ARN polimerasa II y dando como resultado la supresión transcripcional. [29] Las células madre embrionarias sin PcG no se diferencian de manera eficiente en las tres capas germinales, y la eliminación de los genes PRC1 y PRC2 conduce a una mayor expresión de genes afiliados al linaje y una diferenciación no programada. [29] Presumiblemente, los complejos PcG son responsables de reprimir transcripcionalmente los genes que promueven la diferenciación y el desarrollo.
Proteínas del grupo Tritórax (TrxG)
Alternativamente, al recibir señales de diferenciación, las proteínas PcG son reclutadas a los promotores de factores de transcripción de pluripotencia. Las células madre embrionarias deficientes en PcG pueden comenzar la diferenciación pero no pueden mantener el fenotipo diferenciado. [29] Simultáneamente, los genes promotores de la diferenciación y el desarrollo son activados por los reguladores de la cromatina del grupo Trithorax (TrxG) y pierden su represión. [29] [32] Las proteínas TrxG son reclutadas en regiones de alta actividad transcripcional, donde catalizan la trimetilación de la histona H3 lisina 4 ( H3K4me3 ) y promueven la activación génica a través de la acetilación de histonas. [32] Los complejos PcG y TrxG participan en competencia directa y se cree que son funcionalmente antagónicos, creando en los loci promotores de la diferenciación y el desarrollo lo que se denomina un "dominio bivalente" y volviendo a estos genes sensibles a la inducción o represión rápida. [33]
Metilación del ADN
La regulación de la expresión génica se logra además a través de la metilación del ADN, en la que la metilación de los residuos de citosina en los dinucleótidos CpG mediada por la ADN metiltransferasa mantiene la represión hereditaria al controlar la accesibilidad del ADN. [33] La mayoría de los sitios CpG en las células madre embrionarias no están metilados y parecen estar asociados con nucleosomas portadores de H3K4me3. [29] Tras la diferenciación, una pequeña cantidad de genes, incluidos OCT4 y NANOG, [33] se metilan y sus promotores se reprimen para evitar su expresión posterior. De manera consistente, las células madre embrionarias deficientes en metilación del ADN entran rápidamente en apoptosis tras la diferenciación in vitro. [29]
Posicionamiento de los nucleosomas
Si bien la secuencia de ADN de la mayoría de las células de un organismo es la misma, los patrones de unión de los factores de transcripción y los patrones de expresión génica correspondientes son diferentes. En gran medida, las diferencias en la unión de los factores de transcripción están determinadas por la accesibilidad de la cromatina a sus sitios de unión a través de la modificación de histonas y/o factores pioneros . En particular, es importante saber si un nucleosoma cubre un sitio de unión genómico determinado o no. Esto se puede determinar utilizando un ensayo de inmunoprecipitación de cromatina . [34]
Acetilación y metilación de histonas
Las interacciones entre el ADN y los nucleosomas se caracterizan por dos estados: o bien fuertemente unidos por los nucleosomas y transcripcionalmente inactivos, llamados heterocromatina , o débilmente unidos y habitualmente, pero no siempre, transcripcionalmente activos, llamados eucromatina . Los procesos epigenéticos de metilación y acetilación de histonas, y sus inversos desmetilación y desacetilación, son los principales responsables de estos cambios. Los efectos de la acetilación y desacetilación son más predecibles. Un grupo acetilo se añade o se elimina de los residuos de lisina con carga positiva en las histonas mediante enzimas llamadas histona acetiltransferasas o histona desacetilasas, respectivamente. El grupo acetilo impide la asociación de la lisina con la cadena principal del ADN con carga negativa. La metilación no es tan sencilla, ya que ni la metilación ni la desmetilación se correlacionan consistentemente con la activación o represión de genes. Sin embargo, se ha demostrado repetidamente que ciertas metilaciones activan o reprimen genes. La trimetilación de la lisina 4 en la histona 3 (H3K4Me3) está asociada con la activación genética, mientras que la trimetilación de la lisina 27 en la histona 3 reprime genes [35] [36] [37]
En células madre
Durante la diferenciación, las células madre cambian sus perfiles de expresión genética. Estudios recientes han implicado un papel para el posicionamiento de los nucleosomas y las modificaciones de las histonas durante este proceso. [38] Hay dos componentes de este proceso: la desactivación de la expresión de los genes de las células madre embrionarias (ESC) y la activación de los genes del destino celular. Se cree que la desmetilasa específica de lisina 1 ( KDM1A ) previene el uso de regiones potenciadoras de los genes de pluripotencia, inhibiendo así su transcripción. [39] Interactúa con el complejo Mi-2/NuRD (remodelación de nucleosomas y desacetilasa de histonas), [39] dando un ejemplo en el que la metilación y la acetilación no son procesos discretos y mutuamente excluyentes, sino entrelazados.
Papel de la señalización en el control epigenético
Una última pregunta que se debe hacer se refiere al papel de la señalización celular en la influencia de los procesos epigenéticos que rigen la diferenciación. Tal papel debería existir, ya que sería razonable pensar que la señalización extrínseca puede conducir a la remodelación epigenética, así como puede conducir a cambios en la expresión génica a través de la activación o represión de diferentes factores de transcripción. Hay pocos datos directos disponibles sobre las señales específicas que influyen en el epigenoma , y la mayoría del conocimiento actual sobre el tema consiste en especulaciones sobre posibles candidatos reguladores de la remodelación epigenética. [40] Primero discutiremos varios candidatos principales que se cree que están involucrados en la inducción y el mantenimiento de las células madre embrionarias y su progenie diferenciada, y luego nos centraremos en un ejemplo de vías de señalización específicas en las que existe evidencia más directa de su papel en el cambio epigenético.
El primer candidato importante es la vía de señalización de Wnt . La vía de Wnt está involucrada en todas las etapas de la diferenciación, y el ligando Wnt3a puede sustituir la sobreexpresión de c-Myc en la generación de células madre pluripotentes inducidas. [40] Por otro lado, la interrupción de la β-catenina , un componente de la vía de señalización de Wnt, conduce a una disminución de la proliferación de progenitores neuronales.
Los factores de crecimiento constituyen el segundo grupo principal de candidatos a reguladores epigenéticos de la diferenciación celular. Estos morfógenos son cruciales para el desarrollo e incluyen proteínas morfogenéticas óseas , factores de crecimiento transformantes (TGF) y factores de crecimiento de fibroblastos (FGF). Se ha demostrado que los TGF y los FGF sostienen la expresión de OCT4, SOX2 y NANOG mediante la señalización descendente a las proteínas Smad . [40] La disminución de los factores de crecimiento promueve la diferenciación de las células madre embrionarias, mientras que los genes con cromatina bivalente pueden volverse más restrictivos o permisivos en su transcripción. [40]
Varias otras vías de señalización también se consideran candidatas primarias. Los factores inhibidores de la leucemia por citocinas están asociados con el mantenimiento de las células madre embrionarias de ratón en un estado indiferenciado. Esto se logra a través de su activación de la vía Jak-STAT3, que ha demostrado ser necesaria y suficiente para mantener la pluripotencia de las células madre de ratón. [41] El ácido retinoico puede inducir la diferenciación de las células madre humanas y de ratón, [40] y la señalización Notch está involucrada en la proliferación y autorrenovación de las células madre. Finalmente, Sonic hedgehog , además de su papel como morfógeno, promueve la diferenciación de células madre embrionarias y la autorrenovación de células madre somáticas. [40]
El problema, por supuesto, es que la candidatura de estas vías de señalización se dedujo principalmente sobre la base de su papel en el desarrollo y la diferenciación celular. Si bien la regulación epigenética es necesaria para impulsar la diferenciación celular, ciertamente no es suficiente para este proceso. La modulación directa de la expresión génica a través de la modificación de los factores de transcripción desempeña un papel clave que debe distinguirse de los cambios epigenéticos hereditarios que pueden persistir incluso en ausencia de las señales ambientales originales. Actualmente solo existen unos pocos ejemplos de vías de señalización que conducen a cambios epigenéticos que alteran el destino celular, y nos centraremos en uno de ellos.
La expresión de Shh (Sonic hedgehog) regula positivamente la producción de BMI1 , un componente del complejo PcG que reconoce H3K27me3 . Esto ocurre de manera dependiente de Gli, ya que Gli1 y Gli2 son efectores descendentes de la vía de señalización Hedgehog . En cultivo, Bmi1 media la capacidad de la vía Hedgehog para promover la autorrenovación de células madre mamarias humanas. [42] Tanto en humanos como en ratones, los investigadores demostraron que Bmi1 se expresa en gran medida en precursores de células granulares cerebelosas inmaduras en proliferación. Cuando se eliminó Bmi1 en ratones, se produjo un desarrollo cerebeloso deficiente, lo que llevó a reducciones significativas en la masa cerebral posnatal junto con anomalías en el control motor y el comportamiento. [43] Un estudio separado mostró una disminución significativa en la proliferación de células madre neuronales junto con una mayor proliferación de astrocitos en ratones sin Bmi. [44]
Un modelo alternativo de diferenciación celular durante la embriogénesis es que la información posicional se basa en la señalización mecánica del citoesqueleto mediante ondas de diferenciación embrionaria . La señal mecánica se transduce epigenéticamente a través de sistemas de transducción de señales (de los cuales forman parte moléculas específicas como Wnt) para dar lugar a una expresión genética diferencial.
En resumen, el papel de la señalización en el control epigenético del destino celular en los mamíferos es en gran medida desconocido, pero existen ejemplos concretos que indican la probable existencia de otros mecanismos de este tipo.
Efecto de la elasticidad de la matriz
Para cumplir con el propósito de regenerar una variedad de tejidos, se sabe que las células madre adultas migran desde sus nichos, se adhieren a nuevas matrices extracelulares (ECM) y se diferencian. La ductilidad de estos microambientes es exclusiva de los diferentes tipos de tejidos. La ECM que rodea los tejidos cerebrales, musculares y óseos varía de blanda a rígida. La transducción de las células madre en estos tipos de células no está dirigida únicamente por señales de quimiocinas y señalización de célula a célula. La elasticidad del microambiente también puede afectar la diferenciación de las células madre mesenquimales (MSC que se originan en la médula ósea). Cuando las MSC se colocan en sustratos de la misma rigidez que la ECM cerebral, muscular y ósea, las MSC adquieren propiedades de esos respectivos tipos de células. [45] La detección de la matriz requiere que la célula tire contra la matriz en las adherencias focales, lo que activa un mecanotransductor celular para generar una señal para informar qué fuerza se necesita para deformar la matriz. Para determinar los actores clave en la especificación del linaje impulsada por la elasticidad de la matriz en las MSC, se imitaron diferentes microambientes de la matriz. A partir de estos experimentos, se concluyó que las adherencias focales de las MSC eran el mecanotransductor celular que detectaba las diferencias de la elasticidad de la matriz. Las isoformas de miosina no muscular IIa-c generan las fuerzas en la célula que conducen a la señalización de marcadores de compromiso temprano. La miosina no muscular IIa genera la menor fuerza que aumenta a la miosina no muscular IIc. También hay factores en la célula que inhiben la miosina no muscular II, como la blebbistatina . Esto hace que la célula sea efectivamente ciega a la matriz circundante. [45] Los investigadores han logrado cierto éxito en la inducción de propiedades similares a las de las células madre en células HEK 239 al proporcionar una matriz blanda sin el uso de factores de difusión. [46] Las propiedades de las células madre parecen estar vinculadas a la tensión en la red de actina de las células. Un mecanismo identificado para la diferenciación inducida por la matriz son las proteínas inducidas por tensión, que remodelan la cromatina en respuesta al estiramiento mecánico. [47] La vía RhoA también está implicada en este proceso. [ cita requerida ]
Historia evolutiva
Un protista holozoario de mil millones de años , Bicellum brasieri , con dos tipos de células, muestra que la evolución de la multicelularidad diferenciada , posiblemente pero no necesariamente de linajes animales, ocurrió hace al menos mil millones de años y posiblemente principalmente en lagos de agua dulce en lugar de en el océano. [48] [49] [50] [ aclaración necesaria ]
Véase también
- Fuerzas entre capas en la fusión de membranas
- Mecanismo de fusión
- Fusión de bicapa lipídica
- Fusógenos célula-célula
- CAF-1
- Lista de tipos de células humanas derivadas de las capas germinales
Referencias
- ^ Kryven, I.; Röblitz, S.; Schütte, Ch. (2015). "Solución de la ecuación maestra química mediante aproximación de funciones de base radial con seguimiento de interfaz". BMC Systems Biology . 9 (1): 67. doi : 10.1186/s12918-015-0210-y . PMC 4599742 . PMID 26449665.

- ^ Slack, JMW (2013) Biología esencial del desarrollo. Wiley-Blackwell, Oxford.
- ^ Slack, JMW (2007). "Metaplasia y transdiferenciación: de la biología pura a la clínica". Nature Reviews Molecular Cell Biology . 8 (5): 369–378. doi :10.1038/nrm2146. PMID 17377526. S2CID 3353748.
- ^ Yanes, Oscar; Clark, Julie; Wong, Diana M.; Patti, Gary J.; Sánchez-Ruiz, Antonio; Benton, H. Paul; Trauger, Sunia A.; Desponts, Caroline; Ding, Sheng; Siuzdak, Gary (junio de 2010). "La oxidación metabólica regula la diferenciación de células madre embrionarias". Nature Chemical Biology . 6 (6): 411–417. doi :10.1038/nchembio.364. ISSN 1552-4469. PMC 2873061 . PMID 20436487.
- ^ Takahashi, K; Yamanaka, S (2006). "Inducción de células madre pluripotentes a partir de cultivos de fibroblastos embrionarios y adultos de ratón mediante factores definidos". Cell . 126 (4): 663–76. doi :10.1016/j.cell.2006.07.024. hdl : 2433/159777 . PMID 16904174. S2CID 1565219.
- ^ abc Schöler, Hans R. (2007). "El potencial de las células madre: un inventario". En Nikolaus Knoepffler; Dagmar Schipanski; Stefan Lorenz Sorgner (eds.). La biotecnología humana como desafío social . Ashgate Publishing. p. 28. ISBN 978-0-7546-5755-2.
- ^ "NCI Dictionary of Cancer Terms" (Diccionario de términos oncológicos del NCI). Instituto Nacional del Cáncer . Consultado el 1 de noviembre de 2013 .
- ^ Lodish, Harvey (2000). Biología celular molecular (4.ª ed.). Nueva York: WH Freeman. Sección 14.2. ISBN 978-0-7167-3136-8.
- ^ Kumar, Rani (2008). Libro de texto de embriología humana. Editorial IK International. pág. 22. ISBN 9788190675710.
- ^ D. Carpeta, Marc; Hirokawa, Nobutaka; Windhorst, Uwe (2009). Enciclopedia de Neurociencia . Saltador. ISBN 978-3540237358.
- ^ Rakic, P (octubre de 2009). "Evolución del neocórtex: una perspectiva desde la biología del desarrollo". Nature Reviews. Neuroscience . 10 (10): 724–35. doi :10.1038/nrn2719. PMC 2913577 . PMID 19763105.
- ^ Lui, JH; Hansen, DV; Kriegstein, AR (8 de julio de 2011). "Desarrollo y evolución del neocórtex humano". Cell . 146 (1): 18–36. doi :10.1016/j.cell.2011.06.030. PMC 3610574 . PMID 21729779.
- ^ Rash, BG; Ackman, JB; Rakic, P (febrero de 2016). "La actividad radial bidireccional del Ca(2+) regula la neurogénesis y la migración durante la formación temprana de la columna cortical". Science Advances . 2 (2): e1501733. Bibcode :2016SciA....2E1733R. doi :10.1126/sciadv.1501733. PMC 4771444 . PMID 26933693.
- ^ Stocum DL (2004). "Regeneración de anfibios y células madre". Regeneración: células madre y más allá . Temas actuales en microbiología e inmunología. Vol. 280. págs. 1–70. doi :10.1007/978-3-642-18846-6_1. ISBN 978-3-540-02238-1. Número de identificación personal 14594207.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Casimir CM, Gates PB, Patient RK, Brockes JP (1988-12-01). "Evidencia de desdiferenciación y metaplasia en la regeneración de miembros de anfibios a partir de la herencia de la metilación del ADN". Desarrollo . 104 (4): 657–668. doi :10.1242/dev.104.4.657. PMID 3268408.
- ^ Giles KL (1971). "Desdiferenciación y regeneración en briofitas: una revisión selectiva". New Zealand Journal of Botany . 9 (4): 689–94. Bibcode :1971NZJB....9..689G. doi :10.1080/0028825x.1971.10430231. Archivado desde el original el 2008-12-04 . Consultado el 2008-01-01 .
- ^ Schnabel M, Marlovits S, Eckhoff G, et al. (enero de 2002). "Cambios asociados a la desdiferenciación en la morfología y la expresión génica en condrocitos articulares humanos primarios en cultivo celular". Osteoarthr. Cartil . 10 (1): 62–70. doi : 10.1053/joca.2001.0482 . PMID 11795984.
- ^ Sell S (diciembre de 1993). "Origen celular del cáncer: ¿desdiferenciación o detención de la maduración de las células madre?". Environ. Health Perspect . 101 (Supl. 5): 15–26. doi :10.2307/3431838. JSTOR 3431838. PMC 1519468 . PMID 7516873.
- ^ Tsonis PA (abril de 2004). «Células madre a partir de células diferenciadas». Mol. Interv . 4 (2): 81–3. doi :10.1124/mi.4.2.4. PMID 15087480. Archivado desde el original el 23 de mayo de 2016. Consultado el 26 de diciembre de 2010 .
- ^ Ben-Tabou de-Leon S, Davidson EH (2007). "Regulación genética: red de control genético en el desarrollo" (PDF) . Annu Rev Biophys Biomol Struct . 36 (191): 191–212. doi :10.1146/annurev.biophys.35.040405.102002. PMID 17291181.
- ^ Newman, Stuart A. (2020). «Diferenciación celular: ¿qué hemos aprendido en 50 años?». Journal of Theoretical Biology . 485 : 110031. arXiv : 1907.09551 . Bibcode :2020JThBi.48510031N. doi : 10.1016/j.jtbi.2019.110031 . PMID: 31568790.
- ^ Knisely, Karen; Gilbert, Scott F. (2009). Biología del desarrollo (8.ª ed.). Sunderland, Mass.: Sinauer Associates. pág. 147. ISBN 978-0-87893-371-6.
- ^ abcdefg Rudel y Sommer; La evolución de los mecanismos de desarrollo. Biología del desarrollo 264, 15-37, 2003 Rudel, D.; Sommer, RJ (2003). "La evolución de los mecanismos de desarrollo". Biología del desarrollo . 264 (1): 15–37. doi : 10.1016/S0012-1606(03)00353-1 . PMID 14623229.
- ^ ab Yamamoto Y y WR Jeffery; Función central del cristalino en la degeneración del ojo de los peces de las cavernas. Science 289 (5479), 631-633, 2000 Yamamoto, Y.; Jeffery, WR (2000). "Función central del cristalino en la degeneración del ojo de los peces de las cavernas". Science . 289 (5479): 631–633. Bibcode :2000Sci...289..631Y. doi :10.1126/science.289.5479.631. PMID 10915628.
- ^ Kirk MM, A Ransick, SE Mcrae, DL Kirk; La relación entre el tamaño celular y el destino celular en Volvox carteri . Revista de biología celular 123, 191-208, 1993 Kirk, MM; Ransick, A.; McRae, SE; Kirk, DL (1993). "La relación entre el tamaño celular y el destino celular en Volvox carteri". Revista de biología celular . 123 (1): 191–208. doi :10.1083/jcb.123.1.191. PMC 2119814 . PMID 8408198.
- ^ Madrigal P, Deng S, Feng Y, Militi S, Goh KJ, Nibhani R, Grandy R, Osnato A, Ortmann D, Brown S, Pauklin S (25 de enero de 2023). "Las regulaciones epigenéticas y transcripcionales condicionan el destino celular antes de la división durante la diferenciación de células madre pluripotentes humanas" (PDF) . Nature Communications . 14 (405): 405. Bibcode :2023NatCo..14..405M. doi :10.1038/s41467-023-36116-9. PMC 9876972 . PMID 36697417.
- ^ Rabajante JF, Babierra AL (30 de enero de 2015). "Ramificación y oscilaciones en el panorama epigenético de la determinación del destino celular" (PDF) . Progreso en biofísica y biología molecular . 117 (2–3): 240–9. doi :10.1016/j.pbiomolbio.2015.01.006. PMID 25641423. S2CID 2579314.
- ^ ab Lister R; et al. (2011). "Puntos calientes de reprogramación epigenómica aberrante en células madre pluripotentes inducidas humanas". Nature . 471 (7336): 68–73. Bibcode :2011Natur.471...68L. doi :10.1038/nature09798. PMC 3100360 . PMID 21289626.
- ^ abcdefgh Christophersen NS, Helin K (2010). "Control epigenético del destino de las células madre embrionarias". J Exp Med . 207 (11): 2287–95. doi :10.1084/jem.20101438. PMC 2964577 . PMID 20975044.
- ^ ab Thomson, M; Liu, SJ; Zou, LN; Smith, Z; Meissner, A; Ramanathan, S (2011). "Los factores de pluripotencia en células madre embrionarias regulan la diferenciación en capas germinales". Cell . 145 (6): 875–89. doi :10.1016/j.cell.2011.05.017. PMC 5603300 . PMID 21663792.
- ^ Zhu, J.; et al. (2013). "Transiciones del estado de la cromatina en todo el genoma asociadas con señales ambientales y de desarrollo". Cell . 152 (3): 642–654. doi :10.1016/j.cell.2012.12.033. PMC 3563935 . PMID 23333102.
- ^ abc Guenther MG, Young RA (2010). "Transcripción represiva" (PDF) . Science . 329 (5988): 150–1. Bibcode :2010Sci...329..150G. doi :10.1126/science.1193995. PMC 3006433 . PMID 20616255.
- ^ abc Meissner A (2010). "Modificaciones epigenéticas en células pluripotentes y diferenciadas". Nat Biotechnol . 28 (10): 1079–88. doi :10.1038/nbt.1684. PMID 20944600. S2CID 205274850.
- ^ "Inmuprecipitación de cromatina". www.bio.brandeis.edu . Archivado desde el original el 25 de noviembre de 2017. Consultado el 26 de diciembre de 2016 .
- ^ Krogan NJ, Dover J, Wood A, Schneider J, Heidt J, Boateng MA, Dean K, Ryan OW, Golshani A, Johnston M, Greenblatt JF, Shilatifard A (marzo de 2003). "El complejo Paf1 es necesario para la metilación de la histona H3 por COMPASS y Dot1p: vinculando la elongación transcripcional a la metilación de la histona". Molecular Cell . 11 (3): 721–9. doi : 10.1016/S1097-2765(03)00091-1 . PMID 12667454.
- ^ Ng HH, Robert F, Young RA, Struhl K (marzo de 2003). "El reclutamiento dirigido de la metilasa de histona Set1 mediante la elongación de Pol II proporciona una marca localizada y una memoria de la actividad transcripcional reciente". Molecular Cell . 11 (3): 709–19. doi : 10.1016/S1097-2765(03)00092-3 . PMID 12667453.
- ^ Bernstein BE, Kamal M, Lindblad-Toh K, Bekiranov S, Bailey DK, Huebert DJ, McMahon S, Karlsson EK, Kulbokas EJ, Gingeras TR, Schreiber SL, Lander ES (enero de 2005). "Mapas genómicos y análisis comparativo de modificaciones de histonas en humanos y ratones". Cell . 120 (2): 169–81. doi : 10.1016/j.cell.2005.01.001 . PMID 15680324. S2CID 7193829.
- ^ Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K (2012). "Posicionamiento de nucleosomas en todo el genoma durante el desarrollo de células madre embrionarias". Nat Struct Mol Biol . 19 (11): 1185–92. doi :10.1038/nsmb.2419. PMID 23085715. S2CID 34509771.
- ^ ab Whyte, WA; Bilodeau, S; Orlando, DA; Hoke, HA; Frampton, GM; Foster, CT; Cowley, SM; Young, RA (2012). "Desmantelamiento del potenciador por LSD1 durante la diferenciación de células madre embrionarias". Nature . 482 (7384): 221–5. Bibcode :2012Natur.482..221W. doi :10.1038/nature10805. PMC 4144424 . PMID 22297846.
- ^ abcdef Mohammad HP, Baylin SB (2010). "Vinculación de la señalización celular y la maquinaria epigenética". Nat Biotechnol . 28 (10): 1033–8. doi :10.1038/nbt1010-1033. PMID 20944593. S2CID 6911946.
- ^ Niwa H, Burdon T, Chambers I, Smith A (1998). "La autorrenovación de células madre embrionarias pluripotentes está mediada por la activación de STAT3". Genes Dev . 12 (13): 2048–60. doi :10.1101/gad.12.13.2048. PMC 316954 . PMID 9649508.
- ^ Liu S; et al. (2006). "La señalización Hedgehog y Bmi-1 regulan la autorrenovación de células madre mamarias humanas normales y malignas". Cancer Res . 66 (12): 6063–71. doi :10.1158/0008-5472.CAN-06-0054. PMC 4386278 . PMID 16778178.
- ^ Leung C; et al. (2004). "Bmi1 es esencial para el desarrollo cerebeloso y se sobreexpresa en meduloblastomas humanos". Nature . 428 (6980): 337–41. Bibcode :2004Natur.428..337L. doi :10.1038/nature02385. PMID 15029199. S2CID 29965488.
- ^ Zencak D; et al. (2005). "La pérdida de Bmi1 produce un aumento de las células astrogliales y una disminución de la población y proliferación de células madre neurales". J Neurosci . 25 (24): 5774–83. doi :10.1523/JNEUROSCI.3452-04.2005. PMC 6724881 . PMID 15958744.
- ^ ab Engler, AJ; Sen, S; Sweeney, HL; Discher, DE (agosto de 2006). "La elasticidad de la matriz dirige la especificación del linaje de células madre". Cell . 126 (4): 677–689. doi : 10.1016/j.cell.2006.06.044 . PMID 16923388. S2CID 16109483.
- ^ Guo, Jun; Wang, Yuexiu; Sachs, Frederick ; Meng, Fanjie (9 de diciembre de 2014). "El estrés de la actina en la reprogramación celular". Actas de la Academia Nacional de Ciencias . 111 (49): E5252–E5261. Bibcode :2014PNAS..111E5252G. doi : 10.1073/pnas.1411683111 . ISSN 0027-8424. PMC 4267376 . PMID 25422450.
- ^ Guilak, Farshid; Cohen, Daniel M.; Estes, Bradley T.; Gimble, Jeffrey M.; Liedtke, Wolfgang; Chen, Christopher S. (2 de julio de 2009). "Control del destino de las células madre mediante interacciones físicas con la matriz extracelular". Cell Stem Cell . 5 (1): 17–26. doi :10.1016/j.stem.2009.06.016. PMC 2768283 . PMID 19570510.
- ^ "Un fósil de mil millones de años revela un eslabón perdido en la evolución de los animales". phys.org . Consultado el 9 de mayo de 2021 .
- ^ "Se encuentra un fósil de mil millones de años preservado en las rocas de Torridon". BBC News . 2021-04-29 . Consultado el 22 de mayo de 2021 .
- ^ Strother, Paul K.; Brasier, Martin D.; Wacey, David; Timpe, Leslie; Saunders, Martin; Wellman, Charles H. (13 de abril de 2021). "Un posible holozoo de mil millones de años con multicelularidad diferenciada". Current Biology . 31 (12): 2658–2665.e2. Código Bibliográfico :2021CBio...31E2658S. doi : 10.1016/j.cub.2021.03.051 . ISSN 0960-9822. PMID 33852871.
 Disponible bajo CC BY 4.0.
Disponible bajo CC BY 4.0.



