ARN mensajero
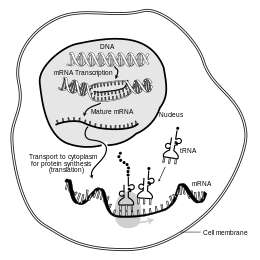
En biología molecular , el ácido ribonucleico mensajero ( ARNm ) es una molécula monocatenaria de ARN que corresponde a la secuencia genética de un gen , y es leída por un ribosoma en el proceso de síntesis de una proteína .
El ARNm se crea durante el proceso de transcripción , donde una enzima ( ARN polimerasa ) convierte el gen en ARNm de transcripción primaria (también conocido como pre-ARNm ). Este pre-ARNm generalmente todavía contiene intrones , regiones que no codificarán la secuencia final de aminoácidos . Estos se eliminan en el proceso de empalme de ARN , dejando solo exones , regiones que codificarán la proteína. Esta secuencia de exones constituye el ARNm maduro . Luego, el ribosoma lee el ARNm maduro y el ribosoma crea la proteína utilizando aminoácidos transportados por el ARN de transferencia (ARNt). Este proceso se conoce como traducción . Todos estos procesos forman parte del dogma central de la biología molecular , que describe el flujo de información genética en un sistema biológico.
Al igual que en el ADN , la información genética del ARNm está contenida en la secuencia de nucleótidos , que están dispuestos en codones que constan de tres ribonucleótidos cada uno. Cada codón codifica un aminoácido específico , excepto los codones de terminación , que terminan la síntesis de proteínas. La traducción de los codones en aminoácidos requiere otros dos tipos de ARN: el ARN de transferencia, que reconoce el codón y proporciona el aminoácido correspondiente, y el ARN ribosómico (ARNr), el componente central de la maquinaria de fabricación de proteínas del ribosoma.
El concepto de ARN mensajero fue desarrollado por Sydney Brenner y Francis Crick en 1960 durante una conversación con François Jacob . En 1961, el ARN mensajero fue identificado y descrito de forma independiente por un equipo formado por Brenner, Jacob y Matthew Meselson y otro equipo dirigido por James Watson . Mientras analizaban los datos para su publicación, Jacob y Jacques Monod acuñaron el nombre de "ARN mensajero".
Síntesis
La breve existencia de una molécula de ARNm comienza con la transcripción y finalmente termina con la degradación. Durante su vida, una molécula de ARNm también puede ser procesada, editada y transportada antes de la traducción. Las moléculas de ARNm eucariotas a menudo requieren un procesamiento y transporte extensos, mientras que las moléculas de ARNm procariotas no. Una molécula de ARNm eucariota y las proteínas que la rodean se denominan juntas RNP mensajero . [ cita requerida ]
Transcripción
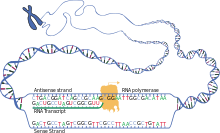
La transcripción es cuando el ARN se copia del ADN. Durante la transcripción, la ARN polimerasa hace una copia de un gen del ADN al ARNm según sea necesario. Este proceso difiere ligeramente en eucariotas y procariotas. Una diferencia notable es que la ARN polimerasa procariota se asocia con enzimas de procesamiento de ADN durante la transcripción para que el procesamiento pueda continuar durante la transcripción. Por lo tanto, esto hace que la nueva cadena de ARNm se vuelva bicatenaria al producir una cadena complementaria conocida como cadena de ARNt, que cuando se combinan no pueden formar estructuras a partir del apareamiento de bases. Además, la plantilla para el ARNm es la cadena complementaria del ARNt, que es idéntica en secuencia a la secuencia del anticodón a la que se une el ADN. El producto de corta duración, no procesado o parcialmente procesado se denomina ARNm precursor o pre-ARNm ; una vez procesado por completo, se denomina ARNm maduro . [ cita requerida ]
Sustitución de timina por uracilo
El ARNm utiliza uracilo (U) en lugar de timina (T) en el ADN. El uracilo (U) es la base complementaria de la adenina (A) durante la transcripción en lugar de la timina (T). Por lo tanto, cuando se utiliza una cadena de ADN molde para construir ARN, la timina se reemplaza por uracilo. Esta sustitución permite que el ARNm lleve la información genética adecuada del ADN al ribosoma para su traducción. En cuanto a la historia natural, el uracilo apareció primero y luego la timina; la evidencia sugiere que el ARN apareció antes que el ADN en la evolución. [1] La hipótesis del mundo del ARN propone que la vida comenzó con moléculas de ARN, antes de la aparición de los genomas de ADN y las proteínas codificadas. En el ADN, la sustitución evolutiva de timina por uracilo puede haber aumentado la estabilidad del ADN y mejorado la eficiencia de la replicación del ADN. [2] [3]
Procesamiento del pre-ARNm eucariota

El procesamiento del ARNm difiere mucho entre eucariotas , bacterias y arqueas . El ARNm no eucariota es, en esencia, maduro tras la transcripción y no requiere procesamiento, excepto en casos raros. [4] Sin embargo, el pre-ARNm eucariota requiere varios pasos de procesamiento antes de su transporte al citoplasma y su traducción por el ribosoma.
Empalme
El procesamiento extenso del pre-ARNm eucariota que conduce al ARNm maduro es el empalme del ARN , un mecanismo por el cual se eliminan los intrones o los outrones (regiones no codificantes) y se unen los exones (regiones codificantes). [ cita requerida ]
Adición de tapa de 5'
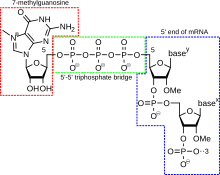
Una tapa 5' (también llamada tapa de ARN, tapa de ARN 7-metilguanosina o tapa de ARN m 7 G) es un nucleótido de guanina modificado que se ha añadido al "frente" o extremo 5' de un ARN mensajero eucariota poco después del inicio de la transcripción. La tapa 5' consiste en un residuo terminal de 7-metilguanosina que está unido a través de un enlace 5'-5'-trifosfato al primer nucleótido transcrito. Su presencia es fundamental para el reconocimiento por parte del ribosoma y la protección contra las ARNasas . [ cita requerida ]
La adición de la tapa está asociada a la transcripción y se produce de manera cotranscripcional, de modo que cada una influye en la otra. Poco después del inicio de la transcripción, el extremo 5' del ARNm que se está sintetizando se une mediante un complejo sintetizador de tapa asociado con la ARN polimerasa . Este complejo enzimático cataliza las reacciones químicas necesarias para la colocación de la tapa en el ARNm. La síntesis se lleva a cabo como una reacción bioquímica de varios pasos. [ cita requerida ]
Edición
En algunos casos, se edita un ARNm , lo que cambia la composición de nucleótidos de ese ARNm. Un ejemplo en humanos es el ARNm de la apolipoproteína B , que se edita en algunos tejidos, pero no en otros. La edición crea un codón de terminación temprano que, tras la traducción, produce una proteína más corta.
Poliadenilación

La poliadenilación es la unión covalente de una porción poliadenililo a una molécula de ARN mensajero. En los organismos eucariotas, la mayoría de las moléculas de ARN mensajero (ARNm) están poliadeniladas en el extremo 3', pero estudios recientes han demostrado que también son comunes los tramos cortos de uridina (oligouridilación). [5] La cola de poli(A) y la proteína unida a ella ayudan a proteger el ARNm de la degradación por exonucleasas. La poliadenilación también es importante para la terminación de la transcripción, la exportación del ARNm desde el núcleo y la traducción. El ARNm también puede estar poliadenilado en organismos procariotas, donde las colas de poli(A) actúan para facilitar, en lugar de impedir, la degradación exonucleolítica. [ cita requerida ]
La poliadenilación ocurre durante y/o inmediatamente después de la transcripción del ADN en ARN. Una vez finalizada la transcripción, la cadena de ARNm se escinde mediante la acción de un complejo de endonucleasas asociado con la ARN polimerasa. Después de que el ARNm se ha escindido, se añaden alrededor de 250 residuos de adenosina al extremo 3' libre en el sitio de escisión. Esta reacción es catalizada por la poliadenilato polimerasa . Al igual que en el splicing alternativo , puede haber más de una variante de poliadenilación de un ARNm.
También se producen mutaciones en el sitio de poliadenilación. El transcrito primario de ARN de un gen se corta en el sitio de adición de poli-A y se añaden entre 100 y 200 A al extremo 3' del ARN. Si se altera este sitio, se formará una construcción de ARNm anormalmente larga e inestable.
Transporte
Otra diferencia entre eucariotas y procariotas es el transporte de ARNm. Debido a que la transcripción y la traducción eucariotas están separadas por compartimentos, los ARNm eucariotas deben exportarse desde el núcleo al citoplasma , un proceso que puede estar regulado por diferentes vías de señalización. [6] Los ARNm maduros son reconocidos por sus modificaciones procesadas y luego exportados a través del poro nuclear mediante la unión a las proteínas de unión a la tapa CBP20 y CBP80, [7] así como al complejo de transcripción/exportación (TREX). [8] [9] Se han identificado múltiples vías de exportación de ARNm en eucariotas. [10]
En células espacialmente complejas, algunos ARNm se transportan a destinos subcelulares particulares. En neuronas maduras , ciertos ARNm se transportan desde el soma hasta las dendritas . Un sitio de traducción del ARNm se encuentra en polirribosomas localizados selectivamente debajo de las sinapsis. [11] El ARNm para Arc/Arg3.1 es inducido por la actividad sináptica y se localiza selectivamente cerca de sinapsis activas en función de las señales generadas por los receptores NMDA . [12] Otros ARNm también se mueven hacia las dendritas en respuesta a estímulos externos, como el ARNm de β-actina . [13] Para la exportación desde el núcleo, el ARNm de actina se asocia con ZBP1 [14] y más tarde con la subunidad 40S . El complejo está unido por una proteína motora y se transporta a la ubicación objetivo ( extensión de neurita ) a lo largo del citoesqueleto . Finalmente, ZBP1 es fosforilado por Src para que se inicie la traducción. [15] En las neuronas en desarrollo, los ARNm también se transportan a los axones en crecimiento y, especialmente, a los conos de crecimiento. Muchos ARNm están marcados con los llamados "códigos postales", que dirigen su transporte a una ubicación específica. [16] [17] Los ARNm también pueden transferirse entre células de mamíferos a través de estructuras llamadas nanotubos de tunelización . [18] [19]
Traducción
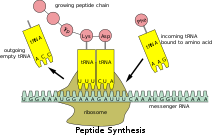
Debido a que el ARNm procariota no necesita ser procesado ni transportado, la traducción por parte del ribosoma puede comenzar inmediatamente después del final de la transcripción. Por lo tanto, se puede decir que la traducción procariota está acoplada a la transcripción y ocurre de manera cotranscripcional . [ cita requerida ]
El ARNm eucariota que ha sido procesado y transportado al citoplasma (es decir, el ARNm maduro) puede ser traducido por el ribosoma. La traducción puede ocurrir en los ribosomas que flotan libremente en el citoplasma o ser dirigida al retículo endoplásmico por la partícula de reconocimiento de señales . Por lo tanto, a diferencia de lo que ocurre en los procariotas, la traducción eucariota no está directamente acoplada a la transcripción. Incluso es posible en algunos contextos que los niveles reducidos de ARNm estén acompañados de niveles aumentados de proteína, como se ha observado para los niveles de ARNm/proteína de EEF1A1 en el cáncer de mama . [20] [ fuente no primaria necesaria ]
Estructura

Regiones codificantes
Las regiones codificantes se componen de codones , que son decodificados y traducidos en proteínas por el ribosoma; en eucariotas generalmente en uno y en procariotas generalmente en varios. Las regiones codificantes comienzan con el codón de inicio y terminan con un codón de terminación . En general, el codón de inicio es un triplete AUG y el codón de terminación es UAG ("ámbar"), UAA ("ocre") o UGA ("ópalo"). Las regiones codificantes tienden a ser estabilizadas por pares de bases internos; esto impide la degradación. [21] [22] Además de codificar proteínas, partes de las regiones codificantes pueden servir como secuencias reguladoras en el pre-ARNm como potenciadores del empalme exónico o silenciadores del empalme exónico .
Regiones no traducidas

Las regiones no traducidas (UTR) son secciones del ARNm antes del codón de inicio y después del codón de terminación que no se traducen, denominadas región no traducida de cinco puntos (UTR 5') y región no traducida de tres puntos (UTR 3'), respectivamente. Estas regiones se transcriben con la región codificante y, por lo tanto, son exónicas , ya que están presentes en el ARNm maduro. Se han atribuido varias funciones en la expresión génica a las regiones no traducidas, incluida la estabilidad del ARNm, la localización del ARNm y la eficiencia de la traducción . La capacidad de una UTR para realizar estas funciones depende de la secuencia de la UTR y puede diferir entre ARNm. Las variantes genéticas en la UTR 3' también se han implicado en la susceptibilidad a las enfermedades debido al cambio en la estructura del ARN y la traducción de proteínas. [23]
La estabilidad de los ARNm puede ser controlada por el UTR 5' y/o el UTR 3' debido a la afinidad variable por las enzimas que degradan el ARN llamadas ribonucleasas y por las proteínas auxiliares que pueden promover o inhibir la degradación del ARN. (Véase también, elemento de estabilidad rico en C ).
La eficiencia de la traducción, que a veces incluye la inhibición completa de la traducción, puede ser controlada por los UTR. Las proteínas que se unen a los UTR 3' o 5' pueden afectar la traducción al influir en la capacidad del ribosoma de unirse al ARNm. Los microARN unidos al UTR 3' también pueden afectar la eficiencia de la traducción o la estabilidad del ARNm.
Se cree que la localización citoplasmática del ARNm es una función del UTR 3'. Las proteínas que se necesitan en una región particular de la célula también pueden traducirse allí; en tal caso, el UTR 3' puede contener secuencias que permitan localizar la transcripción en esta región para su traducción.
Algunos de los elementos contenidos en las regiones no traducidas forman una estructura secundaria característica cuando se transcriben en ARN. Estos elementos estructurales del ARNm participan en la regulación del ARNm. Algunos, como el elemento SECIS , son objetivos a los que se unen las proteínas. Una clase de elemento del ARNm, los riboswitches , se unen directamente a moléculas pequeñas, cambiando su plegamiento para modificar los niveles de transcripción o traducción. En estos casos, el ARNm se regula a sí mismo.
Cola de poli(A)
La cola poli(A) 3' es una secuencia larga de nucleótidos de adenina (a menudo varios cientos) que se agrega al extremo 3' del pre-ARNm. Esta cola promueve la exportación desde el núcleo y la traducción, y protege al ARNm de la degradación.
ARNm monocistrónico versus policistrónico
Se dice que una molécula de ARNm es monocistrónica cuando contiene la información genética para traducir solo una única cadena de proteína (polipéptido). Este es el caso de la mayoría de los ARNm eucariotas . [24] [25] Por otro lado, el ARNm policistrónico lleva varios marcos de lectura abiertos (ORF), cada uno de los cuales se traduce en un polipéptido. Estos polipéptidos suelen tener una función relacionada (a menudo son las subunidades que componen una proteína compleja final) y su secuencia codificante se agrupa y regula en una región reguladora, que contiene un promotor y un operador . La mayoría del ARNm que se encuentra en bacterias y arqueas es policistrónico, [24] al igual que el genoma mitocondrial humano. [26] El ARNm dicistrónico o bicistrónico codifica solo dos proteínas .
Circularización del ARNm

En los eucariotas, las moléculas de ARNm forman estructuras circulares debido a una interacción entre eIF4E y la proteína de unión a poli(A) , que se unen a eIF4G y forman un puente ARNm-proteína-ARNm. [27] Se cree que la circularización promueve el ciclo de los ribosomas en el ARNm, lo que conduce a una traducción eficiente en términos de tiempo, y también puede funcionar para garantizar que solo se traduzca el ARNm intacto (el ARNm parcialmente degradado no suele tener una tapa m7G ni una cola de poli-A). [28]
Existen otros mecanismos de circularización, en particular en el ARNm del virus. El ARNm del virus de la poliomielitis utiliza una sección en forma de hoja de trébol hacia su extremo 5' para unirse a PCBP2, que se une a la proteína de unión a poli(A) , formando el conocido círculo ARNm-proteína-ARNm. El virus del enanismo amarillo de la cebada tiene unión entre segmentos de ARNm en su extremo 5' y 3' (llamados bucles de tallos besándose), lo que hace circular el ARNm sin que intervenga ninguna proteína.
Los genomas de virus de ARN (cuyas hebras + se traducen como ARNm) también suelen circularizarse. [29] Durante la replicación del genoma, la circularización actúa para mejorar las velocidades de replicación del genoma, haciendo que la ARN polimerasa dependiente del ARN viral se ciclice de la misma manera que se supone que lo hace el ribosoma.
Degradación
Los diferentes ARNm dentro de la misma célula tienen tiempos de vida distintos (estabilidades). En las células bacterianas, los ARNm individuales pueden sobrevivir desde segundos hasta más de una hora. Sin embargo, el tiempo de vida promedio es de entre 1 y 3 minutos, lo que hace que el ARNm bacteriano sea mucho menos estable que el ARNm eucariota. [30] En las células de mamíferos, los tiempos de vida del ARNm varían desde varios minutos hasta días. [31] Cuanto mayor sea la estabilidad de un ARNm, más proteínas se pueden producir a partir de ese ARNm. El tiempo de vida limitado del ARNm permite que una célula altere la síntesis de proteínas rápidamente en respuesta a sus necesidades cambiantes. Hay muchos mecanismos que conducen a la destrucción de un ARNm, algunos de los cuales se describen a continuación.
Degradación del ARNm procariota

En general, en los procariotas la vida del ARNm es mucho más corta que en los eucariotas. Los procariotas degradan los mensajes mediante una combinación de ribonucleasas, incluidas las endonucleasas , las exonucleasas 3' y las exonucleasas 5'. En algunos casos, las pequeñas moléculas de ARN (sRNA) de decenas a cientos de nucleótidos de longitud pueden estimular la degradación de ARNm específicos mediante el apareamiento de bases con secuencias complementarias y facilitando la escisión de la ribonucleasa por la ARNasa III . Recientemente se ha demostrado que las bacterias también tienen una especie de tapa 5' que consiste en un trifosfato en el extremo 5' . [32] La eliminación de dos de los fosfatos deja un monofosfato 5', lo que hace que el mensaje sea destruido por la exonucleasa ARNasa J, que degrada 5' a 3'.
Recambio de ARNm eucariota
Dentro de las células eucariotas, existe un equilibrio entre los procesos de traducción y descomposición del ARNm. Los mensajes que se traducen activamente están unidos por los ribosomas , los factores de iniciación eucariotas eIF-4E y eIF-4G y la proteína de unión a poli(A) . eIF-4E y eIF-4G bloquean la enzima de decapitación ( DCP2 ), y la proteína de unión a poli(A) bloquea el complejo de exosomas , protegiendo los extremos del mensaje. El equilibrio entre la traducción y la descomposición se refleja en el tamaño y la abundancia de estructuras citoplasmáticas conocidas como cuerpos P. [33] La cola de poli(A) del ARNm se acorta mediante exonucleasas especializadas que se dirigen a ARN mensajeros específicos mediante una combinación de secuencias reguladoras cis en el ARN y proteínas de unión al ARN que actúan en trans. Se cree que la eliminación de la cola de poli(A) altera la estructura circular del mensaje y desestabiliza el complejo de unión de la tapa . El mensaje se degrada entonces por el complejo de exosomas o por el complejo de descapsulación . De esta manera, los mensajes inactivos desde el punto de vista traduccional pueden destruirse rápidamente, mientras que los mensajes activos permanecen intactos. El mecanismo por el cual se detiene la traducción y el mensaje se transfiere a los complejos de desintegración no se comprende en detalle. Se creía que la mayor parte de la desintegración del ARNm se producía en el citoplasma; sin embargo, recientemente se describió una nueva vía de desintegración del ARNm que comienza en el núcleo. [34]
Desintegración de elementos ricos en AU
La presencia de elementos ricos en AU en algunos ARNm de mamíferos tiende a desestabilizar esas transcripciones a través de la acción de proteínas celulares que se unen a estas secuencias y estimulan la eliminación de la cola de poli(A) . Se cree que la pérdida de la cola de poli(A) promueve la degradación del ARNm al facilitar el ataque tanto del complejo de exosomas [35] como del complejo de descapuchado . [36] La degradación rápida del ARNm a través de elementos ricos en AU es un mecanismo crítico para prevenir la sobreproducción de citocinas potentes como el factor de necrosis tumoral (TNF) y el factor estimulante de colonias de granulocitos y macrófagos (GM-CSF). [37] Los elementos ricos en AU también regulan la biosíntesis de factores de transcripción protooncogénicos como c-Jun y c-Fos . [38]
Decadencia mediada por tonterías
Los mensajes eucariotas están sujetos a vigilancia mediante la desintegración mediada por sinsentidos (NMD), que comprueba la presencia de codones de terminación prematuros (codones sin sentido) en el mensaje. Estos pueden surgir a través de un empalme incompleto, recombinación V(D)J en el sistema inmunológico adaptativo , mutaciones en el ADN, errores de transcripción, escaneo con fugas por parte del ribosoma que causa un cambio de marco y otras causas. La detección de un codón de terminación prematuro desencadena la degradación del ARNm mediante la desprotección de 5', la eliminación de la cola de poli(A) de 3' o la escisión endonucleolítica . [39]
ARN interferente pequeño (siRNA)
En los metazoos , los ARN interferentes pequeños (siRNA) procesados por Dicer se incorporan a un complejo conocido como complejo de silenciamiento inducido por ARN o RISC. Este complejo contiene una endonucleasa que corta los mensajes perfectamente complementarios a los que se une el siRNA. Los fragmentos de ARNm resultantes son destruidos por exonucleasas . El siRNA se usa comúnmente en los laboratorios para bloquear la función de los genes en cultivos celulares. Se cree que es parte del sistema inmunológico innato como defensa contra los virus de ARN bicatenario. [40]
MicroARN (miARN)
Los microARN (miARN) son ARN pequeños que normalmente son parcialmente complementarios a las secuencias de los ARN mensajeros de los metazoos. [41] [42] La unión de un miARN a un mensaje puede reprimir la traducción de ese mensaje y acelerar la eliminación de la cola de poli(A), acelerando así la degradación del ARNm. El mecanismo de acción de los miARN es objeto de una investigación activa. [43] [44]
Otros mecanismos de desintegración
Hay otras formas mediante las cuales se pueden degradar los mensajes, incluida la descomposición continua y el silenciamiento mediante ARN que interactúa con Piwi (piRNA), entre otros.
Aplicaciones
La administración de una secuencia de ARN mensajero modificada con nucleósidos puede hacer que una célula produzca una proteína, que a su vez podría tratar directamente una enfermedad o podría funcionar como una vacuna ; de manera más indirecta, la proteína podría impulsar a una célula madre endógena a diferenciarse de una manera deseada. [45] [46]
Los principales desafíos de la terapia de ARN se centran en hacer llegar el ARN a las células adecuadas. [47] Los desafíos incluyen el hecho de que las secuencias de ARN desnudas se degradan naturalmente después de la preparación; pueden provocar que el sistema inmunológico del cuerpo las ataque como invasoras; y son impermeables a la membrana celular . [46] Una vez dentro de la célula, deben abandonar el mecanismo de transporte de la célula para actuar dentro del citoplasma , que alberga los ribosomas necesarios . [45]
Para superar estos desafíos, el ARNm como terapia se propuso por primera vez en 1989 "después del desarrollo de una técnica de transfección in vitro de amplia aplicación". [48] En la década de 1990, se desarrollaron vacunas de ARNm personalizadas para el cáncer, que se basaban en ARNm modificado sin nucleósidos. Las terapias basadas en ARNm continúan siendo investigadas como un método de tratamiento o terapia tanto para el cáncer como para las enfermedades inflamatorias autoinmunes, metabólicas y respiratorias. Las terapias de edición genética como CRISPR también pueden beneficiarse del uso de ARNm para inducir a las células a producir la proteína Cas deseada . [49]
Desde la década de 2010, las vacunas de ARN y otras terapias de ARN se consideran "una nueva clase de medicamentos". [50] Las primeras vacunas basadas en ARNm recibieron una autorización restringida y se implementaron en todo el mundo durante la pandemia de COVID-19 , por ejemplo , Pfizer -BioNTech COVID-19 vaccine y Moderna . [51] El Premio Nobel de Fisiología o Medicina de 2023 fue otorgado a Katalin Karikó y Drew Weissman por el desarrollo de vacunas de ARNm efectivas contra COVID-19. [52] [53] [54]
Historia
Varios estudios de biología molecular durante la década de 1950 indicaron que el ARN desempeñaba algún tipo de papel en la síntesis de proteínas, pero ese papel no se entendía claramente. Por ejemplo, en uno de los primeros informes, Jacques Monod y su equipo demostraron que la síntesis de ARN era necesaria para la síntesis de proteínas, específicamente durante la producción de la enzima β-galactosidasa en la bacteria E. coli . [55] Arthur Pardee también encontró una acumulación de ARN similar en 1954. [ 56] En 1953, Alfred Hershey , June Dixon y Martha Chase describieron un cierto ADN que contenía citosina (lo que indicaba que era ARN) que desapareció rápidamente después de su síntesis en E. coli . [57] En retrospectiva, esta puede haber sido una de las primeras observaciones de la existencia de ARNm, pero no se reconoció en ese momento como tal. [58]
La idea del ARN mensajero fue concebida por primera vez por Sydney Brenner y Francis Crick el 15 de abril de 1960 en el King's College de Cambridge , mientras François Jacob les contaba sobre un experimento reciente realizado por Arthur Pardee y Monod (el llamado experimento PaJaMo, que no demostró la existencia del ARN mensajero, pero sugirió la posibilidad de su existencia). Con el apoyo de Crick, Brenner y Jacob se propusieron inmediatamente probar esta nueva hipótesis y se pusieron en contacto con Matthew Meselson del Instituto de Tecnología de California para solicitar ayuda. Durante el verano de 1960, Brenner, Jacob y Meselson llevaron a cabo un experimento en el laboratorio de Meselson en Caltech que fue el primero en demostrar la existencia del ARN mensajero. Ese otoño, Jacob y Monod acuñaron el nombre de "ARN mensajero" y desarrollaron el primer marco teórico para explicar su función. [58]
En febrero de 1961, James Watson reveló que su grupo de investigación con sede en Harvard los había estado apoyando con una serie de experimentos cuyos resultados apuntaban aproximadamente en la misma dirección. Brenner y los demás aceptaron la solicitud de Watson de retrasar la publicación de los hallazgos de su investigación. Como resultado, los artículos de Brenner y Watson se publicaron simultáneamente en el mismo número de Nature en mayo de 1961, mientras que ese mismo mes, Jacob y Monod publicaron su marco teórico para el ARNm en el Journal of Molecular Biology . [58]
Véase también
- Prueba de extensión poli(A)
- GeneCalling , una tecnología de creación de perfiles de ARNm
- ARNm sin sentido
- visualización de ARNm
- Vigilancia del ARNm
- Degradación del ARNm procariota
- Transcriptoma , la suma de todo el ARN en una célula.
Referencias
- ^ "La información del ADN se decodifica mediante transcripción | Aprende ciencias en Scitable". www.nature.com . Consultado el 3 de mayo de 2024 .
- ^ "Mundo ARN (artículo) | Selección natural". Khan Academy . Consultado el 3 de mayo de 2024 .
- ^ "La presencia de timina en lugar de uracilo también confiere estabilidad adicional al ADN. ¿Cómo?". Toppr Ask . Consultado el 4 de mayo de 2024 .
- ^ Watson JD (22 de febrero de 2013). Biología molecular del gen, 7.ª edición . Pearson Higher Ed USA. ISBN 9780321851499.
- ^ Choi YS, Patena W, Leavitt AD, McManus MT (marzo de 2012). "Oliguridilación generalizada del extremo 3' del ARN en mamíferos". ARN . 18 (3): 394–401. doi :10.1261/rna.029306.111. PMC 3285928 . PMID 22291204.
- ^ Quaresma AJ, Sievert R, Nickerson JA (abril de 2013). "Regulación de la exportación de ARNm por la vía de transducción de señales PI3 quinasa/AKT". Biología molecular de la célula . 24 (8): 1208–1221. doi :10.1091/mbc.E12-06-0450. PMC 3623641 . PMID 23427269.
- ^ Kierzkowski D, Kmieciak M, Piontek P, Wojtaszek P, Szweykowska-Kulinska Z, Jarmolowski A (septiembre de 2009). "La CBP20 de Arabidopsis dirige el complejo de unión de la tapa al núcleo y es estabilizada por CBP80". The Plant Journal . 59 (5): 814–825. doi : 10.1111/j.1365-313X.2009.03915.x . PMID 19453442.
- ^ Strässer K, Masuda S, Mason P, Pfannstiel J, Oppizzi M, Rodriguez-Navarro S, Rondón AG, Aguilera A, Struhl K, Reed R, Hurt E (mayo de 2002). "TREX es un complejo conservado que acopla la transcripción con la exportación de ARN mensajero". Nature . 417 (6886): 304–308. Bibcode :2002Natur.417..304S. doi :10.1038/nature746. PMID 11979277. S2CID 1112194.
- ^ Katahira J, Yoneda Y (27 de octubre de 2014). "Funciones del complejo TREX en la exportación nuclear de ARNm". RNA Biology . 6 (2): 149–152. doi : 10.4161/rna.6.2.8046 . PMID 19229134.
- ^ Cenik C, Chua HN, Zhang H, Tarnawsky SP, Akef A, Derti A, Tasan M, Moore MJ, Palazzo AF, Roth FP (abril de 2011). "El análisis del genoma revela la interacción entre los intrones 5'UTR y la exportación de ARNm nuclear para genes secretores y mitocondriales". PLOS Genetics . 7 (4): e1001366. doi : 10.1371/journal.pgen.1001366 . PMC 3077370 . PMID 21533221.
- ^ Steward O, Levy WB (marzo de 1982). "Localización preferencial de polirribosomas bajo la base de las espinas dendríticas en células granulares del giro dentado". The Journal of Neuroscience . 2 (3): 284–291. doi :10.1523/JNEUROSCI.02-03-00284.1982. PMC 6564334 . PMID 7062109.
- ^ Steward O, Worley PF (abril de 2001). "La orientación selectiva del ARNm de Arc recién sintetizado hacia sinapsis activas requiere la activación del receptor NMDA". Neuron . 30 (1): 227–240. doi : 10.1016/s0896-6273(01)00275-6 . PMID 11343657. S2CID 13395819.
- ^ Job C, Eberwine J (diciembre de 2001). "Localización y traducción del ARNm en dendritas y axones". Nature Reviews. Neuroscience . 2 (12): 889–898. doi :10.1038/35104069. PMID 11733796. S2CID 5275219.
- ^ Oleynikov Y, Singer RH (febrero de 2003). "Visualización en tiempo real de la asociación de ZBP1 con el ARNm de beta-actina durante la transcripción y localización". Current Biology . 13 (3): 199–207. Bibcode :2003CBio...13..199O. doi :10.1016/s0960-9822(03)00044-7. PMC 4765734 . PMID 12573215.
- ^ Hüttelmaier S, Zenklusen D, Lederer M, Dictenberg J, Lorenz M, Meng X, et al. (noviembre de 2005). "Regulación espacial de la traducción de beta-actina mediante fosforilación de ZBP1 dependiente de Src". Nature . 438 (7067): 512–515. Bibcode :2005Natur.438..512H. doi :10.1038/nature04115. PMID 16306994. S2CID 2453397.
- ^ Oleynikov Y, Singer RH (octubre de 1998). "Localización del ARN: diferentes códigos postales, ¿el mismo cartero?". Tendencias en biología celular . 8 (10): 381–383. doi :10.1016/s0962-8924(98)01348-8. PMC 2136761. PMID 9789325 .
- ^ Ainger K, Avossa D, Diana AS, Barry C, Barbarese E, Carson JH (septiembre de 1997). "Elementos de transporte y localización en el ARNm de la proteína básica de la mielina". The Journal of Cell Biology . 138 (5): 1077–1087. doi :10.1083/jcb.138.5.1077. PMC 2136761 . PMID 9281585.
- ^ Haimovich G, Ecker CM, Dunagin MC, Eggan E, Raj A, Gerst JE, Singer RH (noviembre de 2017). "Tráfico intercelular de ARNm a través de extensiones similares a nanotubos de membrana en células de mamíferos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 114 (46): E9873–E9882. Bibcode :2017PNAS..114E9873H. doi : 10.1073/pnas.1706365114 . PMC 5699038 . PMID 29078295.
- ^ Haimovich G, Dasgupta S, Gerst JE (febrero de 2021). "Transferencia de ARN a través de nanotubos de tunelización". Biochemical Society Transactions . 49 (1): 145–160. doi :10.1042/BST20200113. PMID 33367488. S2CID 229689880.
- ^ Lin CY, Beattie A, Baradaran B, Dray E, Duijf PH (septiembre de 2018). "Expresión contradictoria de ARNm y proteína de EEF1A1 en carcinoma ductal de mama debido a la regulación del ciclo celular y estrés celular". Scientific Reports . 8 (1): 13904. Bibcode :2018NatSR...813904L. doi :10.1038/s41598-018-32272-x. PMC 6141510 . PMID 30224719.
- ^ Shabalina SA, Ogurtsov AY, Spiridonov NA (2006). "Un patrón periódico de la estructura secundaria del ARNm creado por el código genético". Investigación de ácidos nucleicos . 34 (8): 2428–2437. doi :10.1093/nar/gkl287. PMC 1458515 . PMID 16682450.
- ^ Katz L, Burge CB (septiembre de 2003). "Selección generalizada de la estructura secundaria del ARN local en regiones codificantes de genes bacterianos". Genome Research . 13 (9): 2042–2051. doi :10.1101/gr.1257503. PMC 403678 . PMID 12952875.
- ^ Lu YF, Mauger DM, Goldstein DB, Urban TJ, Weeks KM, Bradrick SS (noviembre de 2015). "La estructura del ARNm de IFNL3 se remodela mediante un polimorfismo no codificante funcional asociado con la eliminación del virus de la hepatitis C". Scientific Reports . 5 : 16037. Bibcode :2015NatSR...516037L. doi :10.1038/srep16037. PMC 4631997 . PMID 26531896.
- ^ ab Kozak M (marzo de 1983). "Comparación de la iniciación de la síntesis de proteínas en procariotas, eucariotas y orgánulos". Microbiological Reviews . 47 (1): 1–45. doi :10.1128/MMBR.47.1.1-45.1983. PMC 281560 . PMID 6343825.
- ^ Niehrs C, Pollet N (diciembre de 1999). "Grupos de sinexpresión en eucariotas". Nature . 402 (6761): 483–487. Bibcode :1999Natur.402..483N. doi :10.1038/990025. PMID 10591207. S2CID 4349134.
- ^ Mercer TR, Neph S, Dinger ME, Crawford J, Smith MA, Shearwood AM, Haugen E, Bracken CP, Rackham O, Stamatoyannopoulos JA , Filipovska A, Mattick JS (agosto de 2011). "El transcriptoma mitocondrial humano". Cell . 146 (4): 645–658. doi :10.1016/j.cell.2011.06.051. PMC 3160626. PMID 21854988 .
- ^ Wells SE, Hillner PE, Vale RD, Sachs AB (julio de 1998). "Circularización del ARNm por factores de iniciación de la traducción eucariota". Molecular Cell . 2 (1): 135–140. CiteSeerX 10.1.1.320.5704 . doi :10.1016/S1097-2765(00)80122-7. PMID 9702200.
- ^ López-Lastra M, Rivas A, Barría MI (2005). "Síntesis de proteínas en eucariotas: la creciente relevancia biológica de la iniciación de la traducción independiente del cap". Biological Research . 38 (2–3): 121–146. doi : 10.4067/S0716-97602005000200003 . PMID 16238092.
- ^ Zhang X, Liang Z, Wang C, Shen Z, Sun S, Gong C, Hu X (2022). "ARN circulares virales y sus posibles funciones en la interacción virus-huésped". Frontiers in Immunology . 13 : 939768. doi : 10.3389/fimmu.2022.939768 . PMC 9247149 . PMID 35784275.
- ^ Lewin B , Krebs JE, Kilpatrick ST, Goldstein ES, eds. (2011). Genes X de Lewin (10.ª ed.). Sudbury, Mass.: Jones y Bartlett. ISBN 9780763766320.OCLC 456641931 .
- ^ Yu J, Russell JE (septiembre de 2001). "Análisis estructural y funcional de un complejo de ARNm que media la alta estabilidad del ARNm de beta-globina humana". Biología molecular y celular . 21 (17): 5879–5888. doi :10.1128/mcb.21.17.5879-5888.2001. PMC 87307 . PMID 11486027.
- ^ Deana A, Celesnik H, Belasco JG (enero de 2008). "La enzima bacteriana RppH desencadena la degradación del ARN mensajero mediante la eliminación del pirofosfato 5'". Nature . 451 (7176): 355–358. Bibcode :2008Natur.451..355D. doi :10.1038/nature06475. PMID 18202662. S2CID 4321451.
- ^ Parker R, Sheth U (marzo de 2007). "Cuerpos P y el control de la traducción y degradación del ARNm". Molecular Cell . 25 (5): 635–646. doi : 10.1016/j.molcel.2007.02.011 . PMID 17349952.
- ^ Chattopadhyay S, Garcia Martinez J, Haimovich G, Fischer J, Khwaja A, Barkai O, Chuartzman SG, Schuldiner M, Elran R, Rosenberg M, Urim S, Deshmukh S, Bohnsack K, Bohnsack M, Perez Ortin J, Choder M (noviembre de 2022). "El transporte nucleocitoplasmático controlado por ARN de los factores de desintegración del ARNm regula la síntesis del ARNm y una nueva vía de desintegración del ARNm". Nature Communications . 13(1): 7184 (1): 7184. Bibcode :2022NatCo..13.7184C. doi : 10.1038/s41467-022-34417-z . PMC 9684461 . PMID 36418294.
- ^ Chen CY, Gherzi R, Ong SE, Chan EL, Raijmakers R, Pruijn GJ, Stoecklin G, Moroni C, Mann M, Karin M (noviembre de 2001). "Las proteínas de unión a AU reclutan el exosoma para degradar los ARNm que contienen ARE". Cell . 107 (4): 451–464. doi : 10.1016/S0092-8674(01)00578-5 . PMID 11719186. S2CID 14817671.
- ^ Fenger-Grøn M, Fillman C, Norrild B, Lykke-Andersen J (diciembre de 2005). "Múltiples factores de procesamiento corporal y la proteína de unión a ARE TTP activan el descapuchado del ARNm". Molecular Cell . 20 (6): 905–915. doi : 10.1016/j.molcel.2005.10.031 . PMID 16364915.
- ^ Shaw G, Kamen R (agosto de 1986). "Una secuencia AU conservada de la región 3' no traducida del ARNm de GM-CSF media la degradación selectiva del ARNm". Cell . 46 (5): 659–667. doi :10.1016/0092-8674(86)90341-7. PMID 3488815. S2CID 40332253.
- ^ Chen CY, Shyu AB (noviembre de 1995). "Elementos ricos en AU: caracterización e importancia en la degradación del ARNm". Tendencias en ciencias bioquímicas . 20 (11): 465–470. doi :10.1016/S0968-0004(00)89102-1. PMID 8578590.
- ^ Isken O, Maquat LE (agosto de 2007). "Control de calidad del ARNm eucariota: salvaguardar las células de la función anormal del ARNm". Genes & Development . 21 (15): 1833–1856. doi : 10.1101/gad.1566807 . PMID 17671086.
- ^ Obbard DJ, Gordon KH, Buck AH, Jiggins FM (enero de 2009). "La evolución del ARNi como defensa contra virus y elementos transponibles". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 364 (1513): 99–115. doi :10.1098/rstb.2008.0168. PMC 2592633 . PMID 18926973.
- ^ Robert E. Farrell, Jr. Metodologías del ARN, quinta edición. Academic Press, 2017
- ^ Brennecke J, Stark A, Russell RB, Cohen SM (marzo de 2005). "Principios del reconocimiento de dianas de microARN". PLOS Biology . 3 (3): e85. doi : 10.1371/journal.pbio.0030085 . PMC 1043860 . PMID 15723116.
- ^ Tasuku Honjo, Michael Reth, Andreas Radbruch, Frederick Alt. Molecular Biology of B Cells, 2nd Edition. Academic Press, 2014 (incluida la "investigación actualizada sobre microARN")
- ^ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (enero de 2009). "La deadenilación es un efecto generalizado de la regulación de los miARN". ARN . 15 (1): 21–32. doi :10.1261/rna.1399509. PMC 2612776 . PMID 19029310.
- ^ ab Hajj KA, Whitehead KA (12 de septiembre de 2017). "Herramientas para la traducción: materiales no virales para la administración terapéutica de ARNm". Nature Reviews Materials . 2 (10): 17056. Bibcode :2017NatRM...217056H. doi : 10.1038/natrevmats.2017.56 .
- ^ ab Gousseinov E, Kozlov M, Scanlan C (15 de septiembre de 2015). "Vacunas y terapias basadas en ARN". Noticias de ingeniería genética .
- ^ Kaczmarek JC, Kowalski PS, Anderson DG (junio de 2017). "Avances en la administración de terapias de ARN: del concepto a la realidad clínica". Genome Medicine . 9 (1): 60. doi : 10.1186/s13073-017-0450-0 . PMC 5485616 . PMID 28655327.
- ^ Schlake T, Thess A, Fotin-Mleczek M, Kallen KJ (noviembre de 2012). "Desarrollo de tecnologías de vacunas de ARNm". RNA Biology . 9 (11): 1319–30. doi :10.4161/rna.22269. PMC 3597572 . PMID 23064118.
- ^ Haridi R (23 de abril de 2021). "La revolución del ARNm: cómo la COVID-19 aceleró el avance de una tecnología experimental". New Atlas . Consultado el 26 de abril de 2021 .
- ^ Kowalska J, Wypijewska del Nogal A, Darzynkiewicz ZM, Buck J, Nicola C, Kuhn AN, Lukaszewicz M, Zuberek J, Strenkowska M, Ziemniak M, Maciejczyk M, Bojarska E, Rhoads RE, Darzynkiewicz E, Sahin U, Jemielity J (2014), "terapias basadas en ARNm: desarrollo de una nueva clase de fármacos". Nature Reviews Drug Discovery , vol. 13, núm. 10, págs. 759–780, doi : 10.1093/nar/gku757 , PMC 4176373 , PMID 25150148
- ^ Barbier AJ, Jiang AY, Zhang P, Wooster R, Anderson DG (junio de 2022). "El progreso clínico de las vacunas de ARNm y las inmunoterapias". Nature Biotechnology . 40 (6): 840–854. doi : 10.1038/s41587-022-01294-2 . PMID 35534554. S2CID 248667843.
- ^ "El Premio Nobel de Fisiología o Medicina 2023". NobelPrize.org . Consultado el 3 de octubre de 2023 .
- ^ "Científicos húngaros y estadounidenses ganan el Nobel por descubrimientos de vacunas contra el COVID-19". Reuters . 2023-10-02 . Consultado el 2023-10-03 .
- ^ "El Premio Nobel de Fisiología o Medicina 2023". NobelPrize.org . Consultado el 3 de octubre de 2023 .
- ^ Monod J, Pappenheimer AM, Cohen-Bazire G (1952). "La cinemática de la biosíntesis de la β-galactosidasa de E. coli se considera como función del croissance". Biochimica et Biophysica Acta (en francés). 9 (6): 648–660. doi :10.1016/0006-3002(52)90227-8. PMID 13032175.
- ^ Pardee AB (mayo de 1954). "Precursores de ácidos nucleicos y síntesis de proteínas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 40 (5): 263–270. Bibcode :1954PNAS...40..263P. doi : 10.1073/pnas.40.5.263 . PMC 534118 . PMID 16589470.
- ^ Hershey AD, Dixon J, Chase M (julio de 1953). "Economía de ácidos nucleicos en bacterias infectadas con el bacteriófago T2. I. Composición de purinas y pirimidinas". The Journal of General Physiology . 36 (6): 777–789. doi :10.1085/jgp.36.6.777. PMC 2147416 . PMID 13069681.
- ^ abc Cobb M (29 de junio de 2015). "¿Quién descubrió el ARN mensajero?". Current Biology . 25 (13): R526–R532. Bibcode :2015CBio...25.R526C. doi : 10.1016/j.cub.2015.05.032 . PMID 26126273.
Lectura adicional
- Alsaweed M, Lai CT, Hartmann PE, Geddes DT, Kakulas F (febrero de 2016). "Los miRNA de la leche humana se originan principalmente en la glándula mamaria, lo que da lugar a perfiles de miRNA únicos de la leche fraccionada". Scientific Reports . 6 (1): 20680. Bibcode :2016NatSR...620680A. doi :10.1038/srep20680. PMC 4745068 . PMID 26854194.
- Lillycrop KA , Burdge GC (octubre de 2012). "Mecanismos epigenéticos que vinculan la nutrición temprana con la salud a largo plazo". Mejores prácticas e investigación. Endocrinología clínica y metabolismo . 26 (5): 667–676. doi :10.1016/j.beem.2012.03.009. PMID 22980048.
- Melnik BC, Kakulas F, Geddes DT, Hartmann PE, John SM, Carrera-Bastos P, Cordain L, Schmitz G (21 de junio de 2016). "MiRNA de la leche: ¿simples nutrientes o reguladores funcionales sistémicos?". Nutrition & Metabolism . 13 (1): 42. doi : 10.1186/s12986-016-0101-2 . PMC 4915038 . PMID 27330539.
- Vickers MH (junio de 2014). "Nutrición en la primera infancia, epigenética y programación de enfermedades en etapas posteriores de la vida". Nutrients . 6 (6): 2165–2178. doi : 10.3390/nu6062165 . PMC 4073141 . PMID 24892374.
- Zhou Q, Li M, Wang X, Li Q, Wang T, Zhu Q, Zhou X, Wang X, Gao X, Li X (2012). "Los microARN relacionados con el sistema inmunitario son abundantes en los exosomas de la leche materna". Revista Internacional de Ciencias Biológicas . 8 (1): 118–123. doi :10.7150/ijbs.8.118. PMC 3248653 . PMID 22211110.
- Krause W (2023). "ARNm: del tratamiento de la COVID-19 a la inmunoterapia contra el cáncer". Biomedicinas . 11 (2): 308. doi : 10.3390/biomedicines11020308 . PMC 9953480 . PMID 36830845.
Enlaces externos
- Atlas de RNAi: una base de datos de bibliotecas de RNAi y los resultados de sus análisis de objetivos
- miRSearch Archivado el 4 de diciembre de 2012 en Wayback Machine : Herramienta para encontrar microARN que se dirigen al ARNm
- ¿Cómo se codifica el ARNm?: Vídeo de YouTube
- ¿Qué es el ARNm?: theconversation.com

