visualización de ARNm
La visualización de ARNm es una técnica de visualización utilizada para la evolución de proteínas y/o péptidos in vitro para crear moléculas que puedan unirse a un objetivo deseado. El proceso da como resultado péptidos o proteínas traducidos que se asocian con su progenitor de ARNm a través de un enlace de puromicina . Luego, el complejo se une a un objetivo inmovilizado en un paso de selección ( cromatografía de afinidad ). Las fusiones de ARNm-proteína que se unen bien se transcriben luego de manera inversa a ADNc y su secuencia se amplifica a través de una reacción en cadena de la polimerasa . El resultado es una secuencia de nucleótidos que codifica un péptido con alta afinidad por la molécula de interés.
La puromicina es un análogo del extremo 3' de un tirosil-ARNt con una parte de su estructura que imita una molécula de adenosina y la otra parte imita una molécula de tirosina . En comparación con el enlace éster escindible en un tirosil-ARNt, la puromicina tiene un enlace amida no hidrolizable. Como resultado, la puromicina interfiere con la traducción y causa la liberación prematura de los productos de traducción.

Todas las plantillas de ARNm utilizadas para la tecnología de visualización de ARNm tienen puromicina en su extremo 3'. A medida que avanza la traducción, el ribosoma se mueve a lo largo de la plantilla de ARNm y, una vez que alcanza el extremo 3' de la plantilla, la puromicina fusionada ingresará al sitio A del ribosoma y se incorporará al péptido naciente. Luego, la fusión ARNm-polipéptido se libera del ribosoma (Figura 1).
Para sintetizar una fusión ARNm-polipéptido, la puromicina fusionada no es la única modificación de la plantilla de ARNm. [1] Es necesario reclutar oligonucleótidos y otros espaciadores junto con la puromicina para proporcionar flexibilidad y longitud adecuada para que la puromicina entre en el sitio A. Idealmente, el enlace entre el extremo 3' de un ARNm y la puromicina tiene que ser flexible y lo suficientemente largo para permitir que la puromicina entre en el sitio A tras la traducción del último codón. Esto permite la producción eficiente de una fusión ARNm-polipéptido de longitud completa y alta calidad. Rihe Liu et al. optimizaron el espaciador de oligonucleótidos de 3'-puromicina. Informaron que dA25 en combinación con un espaciador 9 (Glen Research) y dAdCdCP en el extremo 5' funcionaron mejor para la reacción de fusión. Encontraron que los enlaces más largos de 40 nucleótidos y más cortos de 16 nucleótidos mostraron una eficiencia de formación de fusión muy reducida. Además, cuando la secuencia rUrUP se presentó adyacente a la puromicina, la fusión no se formó de manera eficiente. [2]
Además de proporcionar flexibilidad y longitud, la porción poli dA del enlazador también permite una mayor purificación de la fusión ARNm-polipéptido debido a su alta afinidad por la resina de celulosa dT. [3] Las fusiones ARNm-polipéptido se pueden seleccionar sobre objetivos de selección inmovilizados durante varias rondas con una rigurosidad creciente. Después de cada ronda de selección, los miembros de la biblioteca que permanecen unidos al objetivo inmovilizado se amplifican por PCR y los que no se unen se eliminan por lavado.
Método

La síntesis de una biblioteca de visualización de ARNm comienza con la síntesis de una biblioteca de ADN. Una biblioteca de ADN para cualquier proteína o péptido pequeño de interés se puede sintetizar mediante síntesis en fase sólida seguida de amplificación por PCR. Por lo general, cada miembro de esta biblioteca de ADN tiene un sitio de transcripción de la ARN polimerasa T7 y un sitio de unión ribosomal en el extremo 5'. La región promotora T7 permite la transcripción in vitro a gran escala de T7 para transcribir la biblioteca de ADN en una biblioteca de ARNm, que proporciona plantillas para la reacción de traducción in vitro posterior. El sitio de unión ribosomal en la región 5' no traducida (5' UTR) se diseña de acuerdo con el sistema de traducción in vitro que se utilizará. Hay dos sistemas de traducción in vitro populares disponibles comercialmente . Uno es E. coli S30 Extract System (Promega) que requiere una secuencia Shine-Dalgarno en la 5' UTR como sitio de unión ribosomal; [4] el otro es Red Nova Lysate (Novagen), que necesita un sitio de unión ribosomal ΔTMV.
Una vez que se genera la biblioteca de ARNm, se purifica mediante electroforesis en gel de urea y se liga utilizando la ADN ligasa T4 al espaciador de ADN que contiene puromicina en el extremo 3'. En este paso de ligación, se liga un fragmento de ARNm con un ADN monocatenario con la ayuda de la ADN ligasa T4. Esta no es una reacción de ligación estándar de la ADN ligasa T4, en la que se ligan dos fragmentos de ADN bicatenario. Para aumentar el rendimiento de esta ligación especial, se puede utilizar una férula de ADN monocatenario para ayudar a la reacción de ligación. El extremo 5' de la férula está diseñado para ser complementario al extremo 3' del ARNm, y el extremo 3' de la férula está diseñado para ser complementario al extremo 5' del espaciador de ADN, que normalmente consta de nucleótidos de poli dA (Figura 2).
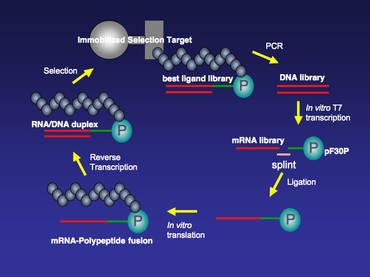
La biblioteca de ARNm-ADN-puromicina ligada se traduce en el lisado Red Nova (Novagen) o en el sistema de extracto de E. coli S30 (Promega), lo que da como resultado polipéptidos unidos covalentemente en cis al ARNm codificante. La traducción in vitro también se puede realizar en un sistema PURE (síntesis de proteínas utilizando elementos recombinantes). El sistema PURE es un sistema de traducción sin células de E. coli en el que solo están presentes los componentes de traducción esenciales. Algunos componentes, como los aminoácidos y las aminoacil-ARNt sintasas (AARS), se pueden omitir del sistema. En su lugar, se puede agregar ARNt acilado químicamente al sistema PURE. Se ha demostrado que algunos aminoácidos no naturales, como el ARNt acilado con N-metilaminoácido, se pueden incorporar a péptidos o fusiones de ARNm-polipéptido en un sistema PURE. [5]
Después de la traducción, las porciones de ARNm monocatenario de las fusiones se convertirán en heterodúplex de ARN/ADN mediante la transcriptasa inversa para eliminar cualquier estructura secundaria de ARN no deseada y hacer que la porción de ácido nucleico de la fusión sea más estable. Este paso es una reacción de transcripción inversa estándar. Por ejemplo, se puede realizar utilizando Superscript II (GIBCO-BRL) siguiendo el protocolo del fabricante.
Las fusiones ARNm/ADN-polipéptido pueden seleccionarse sobre dianas de selección inmovilizadas durante varias rondas (Figura 3). Puede haber un fondo relativamente alto durante las primeras rondas de selección, y esto puede minimizarse aumentando la rigurosidad de la selección, como ajustando la concentración de sal, la cantidad de detergente y/o la temperatura durante el período de unión diana/fusión. Después de la selección de unión, los miembros de la biblioteca que permanecen unidos a la diana inmovilizada se amplifican por PCR. El paso de amplificación por PCR enriquecerá la población de la biblioteca de visualización de ARNm que tenga mayor afinidad por la diana inmovilizada. La PCR propensa a errores también se puede realizar entre cada ronda de selección para aumentar aún más la diversidad de la biblioteca de visualización de ARNm y reducir el fondo en la selección. [6]
Recientemente se publicó un protocolo que requiere menos tiempo para la visualización del ARNm. [7]
Ventajas
Aunque existen muchas otras tecnologías de visualización molecular, como la visualización en fagos , la visualización en bacterias , la visualización en levaduras y la visualización en ribosomas , la tecnología de visualización de ARNm tiene muchas ventajas sobre las demás. [8] Las tres primeras bibliotecas de visualización biológica enumeradas tienen polipéptidos o proteínas expresados en la superficie del microorganismo respectivo y la información de codificación que acompaña a cada polipéptido o proteína se puede recuperar del genoma del microorganismo. Sin embargo, el tamaño de la biblioteca para estos tres sistemas de visualización in vivo está limitado por la eficiencia de transformación de cada organismo. Por ejemplo, el tamaño de la biblioteca para la visualización en fagos y bacterias está limitado a 1-10 × 10^9 miembros diferentes. El tamaño de la biblioteca para la visualización en levaduras es incluso menor. Además, estos sistemas de visualización basados en células solo permiten la selección y el enriquecimiento de péptidos/proteínas que contienen aminoácidos naturales. Por el contrario, la visualización de ARNm y la visualización en ribosomas son métodos de selección in vitro . Permiten un tamaño de biblioteca de hasta 10^15 miembros diferentes. El gran tamaño de la biblioteca aumenta la probabilidad de seleccionar secuencias muy raras y también mejora la diversidad de las secuencias seleccionadas. Además, los métodos de selección in vitro eliminan la presión de selección no deseada, como la expresión deficiente de proteínas y la degradación rápida de proteínas, que pueden reducir la diversidad de las secuencias seleccionadas. Finalmente, los métodos de selección in vitro permiten la aplicación de técnicas de mutagénesis in vitro [9] y recombinación a lo largo del proceso de selección.
Aunque tanto la visualización de ribosomas como la visualización de ARNm son métodos de selección in vitro , la visualización de ARNm tiene algunas ventajas sobre la tecnología de visualización de ribosomas. [10] La visualización de ARNm utiliza complejos covalentes de ARNm-polipéptido unidos a través de puromicina; mientras que la visualización de ribosomas utiliza complejos de ribosoma-ARNm-polipéptido no covalentes estancados. [11] Para la visualización de ribosomas, la rigurosidad de la selección se limita a mantener el ribosoma-ARNm-polipéptido en un complejo debido a los complejos no covalentes de ribosoma-ARNm-polipéptido. Esto puede causar dificultades para reducir la unión de fondo durante el ciclo de selección. Además, los polipéptidos bajo selección en un sistema de visualización de ribosomas están unidos a un enorme complejo de ARNr-proteína, un ribosoma, que tiene un peso molecular de más de 2.000.000 Da. Puede haber alguna interacción impredecible entre el objetivo de selección y el ribosoma, y esto puede conducir a una pérdida de aglutinantes potenciales durante el ciclo de selección. Por el contrario, el espaciador de ADN de puromicina utilizado en la tecnología de visualización de ARNm es mucho más pequeño en comparación con un ribosoma. Este espaciador puede tener menos posibilidades de interactuar con un objetivo de selección inmovilizado. Por lo tanto, es más probable que la tecnología de visualización de ARNm proporcione resultados menos sesgados.
Solicitud
En 1997, Roberts y Szostak demostraron que las fusiones entre un ARNm sintético y su epítopo myc codificado podían enriquecerse a partir de un conjunto de fusiones de ARNm-polipéptido de secuencia aleatoria mediante inmunoprecipitación. [6]
Nueve años después, Fukuda y sus colegas eligieron el método de visualización de ARNm para la evolución in vitro de fragmentos de anticuerpos Fv de cadena sencilla (scFv). [12] Seleccionaron seis mutantes scFv diferentes con cinco mutaciones de consenso. Sin embargo, el análisis cinético de estos mutantes mostró que su especificidad antigénica seguía siendo similar a la del tipo salvaje. Sin embargo, han demostrado que dos de las cinco mutaciones de consenso estaban dentro de las regiones determinantes de complementariedad (CDR). Y concluyeron que la visualización de ARNm tiene el potencial de una rápida evolución artificial de anticuerpos diagnósticos y terapéuticos de alta afinidad mediante la optimización de sus CDR.
Roberts y colaboradores han demostrado que los oligómeros peptídicos no naturales que consisten en un aminoácido N-sustituido pueden sintetizarse como fusiones de ARNm y polipéptidos. [13] Los péptidos que contienen aminoácidos N-sustituidos se han asociado con una buena estabilidad proteolítica y propiedades farmacocinéticas mejoradas. Este trabajo indica que la tecnología de visualización de ARNm tiene el potencial de seleccionar péptidos similares a fármacos para uso terapéutico resistentes a la proteólisis. [14]
Véase también
Referencias
- ^ Roberts RW, Szostak JW (1997). "Fusiones ARN-péptido para la selección in vitro de péptidos y proteínas". PNAS . 94 (23): 12297–12302. Bibcode :1997PNAS...9412297R. doi : 10.1073/pnas.94.23.12297 . PMC 24913 . PMID 9356443.
- ^ Liu R, Barrick JE, Szostak JW, Roberts RW (2000). "Síntesis optimizada de fusiones de ARN-proteína para la selección de proteínas in vitro". Interacciones ARN-ligando Parte B. Métodos en enzimología . Vol. 318. págs. 268–93. doi :10.1016/S0076-6879(00)18058-9. ISBN. 9780121822194. Número de identificación personal 10889994.
- ^ Kurz M, Gu K, Lohse PA (2000). "Conjugados de ARNm-puromicina fotoentrecruzados con psoraleno: una nueva plantilla para la preparación rápida y sencilla de fusiones de ARNm-proteína". Nucleic Acids Research . 28 (18): 83e–83. doi :10.1093/nar/28.18.e83. PMC 110755 . PMID 10982894.
- ^ Mattheakis LC, Bhatt RR, Dower WJ (1994). "Un sistema de visualización de polisomas in vitro para identificar ligandos de bibliotecas de péptidos muy grandes". Proc Natl Acad Sci USA . 91 (19): 9022–6. Bibcode :1994PNAS...91.9022M. doi : 10.1073/pnas.91.19.9022 . PMC 44739 . PMID 7522328.
- ^ Kawakami T, Murakami H, Suga H (enero de 2008). "Incorporación programada por ARN mensajero de múltiples N-metilaminoácidos en péptidos lineales y cíclicos". Química y biología . 15 (1): 32–42. doi :10.1016/j.chembiol.2007.12.008. PMID 18215771.
- ^ ab Roberts RW, Szostak JW (1997). "Fusiones ARN-péptido para la selección in vitro de péptidos y proteínas". Proc Natl Acad Sci USA . 94 (23): 12297–302. Bibcode :1997PNAS...9412297R. doi : 10.1073/pnas.94.23.12297 . PMC 24913 . PMID 9356443.
- ^ Barendt PA, Ng DT, McQuade CN, Sarkar CA (2013). "Protocolo optimizado para la visualización de ARNm". ACS Combinatorial Science . 15 (2): 77–81. doi :10.1021/co300135r. PMC 3666848 . PMID 23305392.
- ^ Roberts RW (junio de 1999). "Selección de proteínas totalmente in vitro mediante fusiones de ARNm-proteína y visualización de ribosomas". Current Opinion in Chemical Biology . 3 (3): 268–73. doi :10.1016/S1367-5931(99)80042-8. PMID 10359713.
- ^ Jing D, Li F, Jiang M, Cai J, Wu Y, Xie K, Wu X, Tang C, Liu J, Guo W, Shen G, Luo E (noviembre de 2013). "Los campos electromagnéticos pulsados mejoran la microestructura y la resistencia ósea en ratas ovariectomizadas". PLoS ONE . 8 (11): e79377. Bibcode :2013PLoSO...879377J. doi : 10.1371/journal.pone.0079377 . PMC 3828367 . PMID 24244491.
- ^ Gold L (abril de 2001). "Presentación del ARNm: la diversidad importa durante la selección in vitro". Proc Natl Acad Sci USA . 98 (9): 4825–6. Bibcode :2001PNAS...98.4825G. doi : 10.1073/pnas.091101698 . PMC 33119 . PMID 11320229.
- ^ Andrew Buchanan; Lutz Jermutus. "Método de visualización de ribosomas o visualización de ARNm con selección para aumentar la estabilidad de la proteína". Google Patents . Consultado el 9 de junio de 2014 .
- ^ Fukuda I, Kojoh K, Tabata N, et al. (2006). "Evolución in vitro de anticuerpos monocatenarios mediante visualización de ARNm". Investigación de ácidos nucleicos . 34 (19): e127. doi :10.1093/nar/gkl618. PMC 1636464 . PMID 17012279.
- ^ Frankel A, Millward SW, Roberts RW (noviembre de 2003). "Encodificadores: oligómeros peptídicos no naturales codificados en ARN" (PDF) . Química y biología . 10 (11): 1043–50. doi : 10.1016/j.chembiol.2003.11.004 . PMID 14652071.
- ^ White, E. Railey; Sun, Luxin; Ma, Zhong; Beckta, Jason M.; Danzig, Brittany A.; Hacker, David E.; Huie, Melissa; Williams, David C.; Edwards, Ross A. (15 de mayo de 2015). "Enfoque de biblioteca de péptidos para descubrir inhibidores fosfomiméticos del dominio C-terminal de BRCA1". ACS Chemical Biology . 10 (5): 1198–1208. doi :10.1021/cb500757u. PMC 4433557 . PMID 25654734.