ADN mitocondrial


El ADN mitocondrial ( ADNmt y ADNm ) es el ADN que se encuentra en los orgánulos mitocondriales de una célula eucariota y que convierte la energía química de los alimentos en trifosfato de adenosina (ATP). El ADN mitocondrial es una pequeña porción del ADN contenido en una célula eucariota; la mayor parte del ADN se encuentra en el núcleo celular y, en las plantas y las algas, el ADN también se encuentra en los plástidos , como los cloroplastos . [3]
El ADN mitocondrial humano fue la primera parte significativa del genoma humano que se secuenció. [4] Esta secuenciación reveló que el ADNmt humano tiene 16.569 pares de bases y codifica 13 proteínas . Al igual que en otros vertebrados, el código genético mitocondrial humano difiere ligeramente del ADN nuclear. [5]
Dado que el ADNmt animal evoluciona más rápido que los marcadores genéticos nucleares , [6] [7] [8] representa un pilar de la filogenética y la biología evolutiva . También permite rastrear las relaciones entre poblaciones, por lo que ha adquirido importancia en la antropología y la biogeografía .
Origen
Se cree que el ADN nuclear y mitocondrial tienen orígenes evolutivos separados , y que el ADNmt deriva de los genomas circulares de bacterias engullidas por los ancestros de las células eucariotas modernas. Esta teoría se denomina teoría endosimbiótica . En las células de los organismos actuales, la gran mayoría de las proteínas de las mitocondrias (que suman aproximadamente 1500 tipos diferentes en los mamíferos ) están codificadas por el ADN nuclear , pero se cree que los genes de algunas de ellas, si no la mayoría, son de origen bacteriano, habiendo sido transferidos al núcleo eucariota durante la evolución . [9]
Las razones por las que las mitocondrias han retenido algunos genes son motivo de debate. La existencia en algunas especies de orgánulos derivados de mitocondrias que carecen de genoma [10] sugiere que es posible la pérdida completa de genes, y la transferencia de genes mitocondriales al núcleo tiene varias ventajas [11] . La dificultad de dirigir productos proteicos hidrófobos producidos de forma remota a la mitocondria es una hipótesis de por qué algunos genes se retienen en el ADNmt; [12] la colocalización para la regulación redox es otra, citando la conveniencia de un control localizado sobre la maquinaria mitocondrial [13] . Un análisis reciente de una amplia gama de genomas de ADNmt sugiere que ambas características pueden determinar la retención de genes mitocondriales [9] .
Estructura y diversidad del genoma
En todos los organismos, hay seis tipos principales de genomas mitocondriales, clasificados por estructura (es decir, circular versus lineal), tamaño, presencia de intrones o estructuras similares a plásmidos y si el material genético es una molécula singular o una colección de moléculas homogéneas o heterogéneas . [14]
En muchos organismos unicelulares (p. ej., el ciliado Tetrahymena y el alga verde Chlamydomonas reinhardtii ), y en casos raros también en organismos multicelulares (p. ej. en algunas especies de Cnidaria ), el ADNmt es ADN lineal . La mayoría de estos ADNmt lineales poseen telómeros independientes de la telomerasa (es decir, los extremos del ADN lineal ) con diferentes modos de replicación, lo que los ha convertido en objetos de investigación interesantes porque muchos de estos organismos unicelulares con ADNmt lineal son patógenos conocidos . [15]
Animales
La mayoría de los animales ( bilaterales ) tienen un genoma mitocondrial circular. Sin embargo, los clados Medusozoa y Calcarea incluyen especies con cromosomas mitocondriales lineales. [16] Con algunas excepciones, los animales tienen 37 genes en su ADN mitocondrial: 13 para proteínas , 22 para ARNt y 2 para ARNr . [17]
Los genomas mitocondriales de los animales tienen una longitud media de unos 16.000 pares de bases. [17] La anémona Isarachnanthus nocturnus tiene el genoma mitocondrial más grande de todos los animales, con 80.923 pb. [18] El genoma mitocondrial más pequeño conocido en animales pertenece a la medusa peine Vallicula multiformis , que consta de 9.961 pb. [19]
En febrero de 2020, se descubrió un parásito relacionado con las medusas, Henneguya salminicola , que carece de genoma mitocondrial pero conserva estructuras consideradas orgánulos relacionados con las mitocondrias. Además, los genes del ADN nuclear involucrados en la respiración aeróbica y en la replicación y transcripción del ADN mitocondrial estaban ausentes o presentes solo como pseudogenes . Este es el primer organismo multicelular conocido que tiene esta ausencia de respiración aeróbica y vive completamente libre de dependencia del oxígeno. [20] [21]
Plantas y hongos
Existen tres tipos diferentes de genomas mitocondriales en plantas y hongos. El primer tipo es un genoma circular que tiene intrones (tipo 2) y puede tener una longitud de entre 19 y 1000 kbp. El segundo tipo de genoma es un genoma circular (de unos 20 a 1000 kbp) que también tiene una estructura similar a la de un plásmido (1 kb) (tipo 3). El último tipo de genoma que se encuentra en plantas y hongos es un genoma lineal formado por moléculas de ADN homogéneas (tipo 5). [22] [23] [24]
Existe una gran variación en el contenido y tamaño de los genes del ADNmt entre hongos y plantas, aunque parece haber un subconjunto central de genes presente en todos los eucariotas (excepto en los pocos que no tienen mitocondrias en absoluto). [9] En los hongos, sin embargo, no hay un único gen compartido entre todos los mitogenomas. [25] Algunas especies de plantas tienen genomas mitocondriales enormes, y el ADNmt de Silene conica contiene hasta 11.300.000 pares de bases. [26] Sorprendentemente, incluso esos enormes ADNmt contienen la misma cantidad y tipos de genes que las plantas relacionadas con ADNmt mucho más pequeños. [27] El genoma de la mitocondria del pepino ( Cucumis sativus ) consta de tres cromosomas circulares (longitudes de 1556, 84 y 45 kilobases), que son total o en gran medida autónomos con respecto a su replicación . [28]
Protistas
Los protistas contienen los genomas mitocondriales más diversos, con cinco tipos diferentes encontrados en este reino. Los tipos 2, 3 y 5 de los genomas de plantas y hongos también existen en algunos protistas, al igual que dos tipos únicos de genoma. Uno de estos tipos únicos es una colección heterogénea de moléculas de ADN circulares (tipo 4), mientras que el otro es una colección heterogénea de moléculas lineales (tipo 6). Los tipos de genoma 4 y 6 varían cada uno de 1 a 200 kbp en tamaño. [ cita requerida ]
El genoma mitocondrial más pequeño secuenciado hasta la fecha es el ADNmt de 5.967 pb del parásito Plasmodium falciparum . [29] [30]
La transferencia genética endosimbiótica , el proceso por el cual los genes que fueron codificados en el genoma mitocondrial se transfieren al genoma principal de la célula, probablemente explica por qué organismos más complejos como los humanos tienen genomas mitocondriales más pequeños que organismos más simples como los protistas.
| Tipo de genoma [14] | Reino | Intrones | Tamaño | Forma | Descripción |
|---|---|---|---|---|---|
| 1 | Animal | No | 11–28 kbps | Circular | Molécula única |
| 2 | Hongos, Plantas, Protistas | Sí | 19–1000 kbps | Circular | Molécula única |
| 3 | Hongos, Plantas, Protistas | No | 20–1000 kbps | Circular | Estructuras similares a moléculas grandes y plásmidos pequeños |
| 4 | Protista | No | 1–200 kbps | Circular | Grupo heterogéneo de moléculas |
| 5 | Hongos, Plantas, Protistas | No | 1–200 kbps | Lineal | Grupo homogéneo de moléculas |
| 6 | Protista | No | 1–200 kbps | Lineal | Grupo heterogéneo de moléculas |
Replicación
El ADN mitocondrial es replicado por el complejo de la ADN polimerasa gamma, que está compuesto por una ADN polimerasa catalítica de 140 kDa codificada por el gen POLG y dos subunidades accesorias de 55 kDa codificadas por el gen POLG2 . [31] La maquinaria del replisoma está formada por la ADN polimerasa, TWINKLE y las proteínas SSB mitocondriales . TWINKLE es una helicasa , que desenrolla tramos cortos de dsADN en la dirección 5' a 3'. [32] Todos estos polipéptidos están codificados en el genoma nuclear.
Durante la embriogénesis , la replicación del ADNmt está estrictamente regulada a la baja desde el ovocito fertilizado hasta el embrión preimplantacional. [33] La reducción resultante en el número de copias por célula del ADNmt desempeña un papel en el cuello de botella mitocondrial, explotando la variabilidad de célula a célula para mejorar la herencia de mutaciones dañinas. [34] Según Justin St. John y colegas, "En la etapa de blastocisto , el inicio de la replicación del ADNmt es específico de las células del trofectodermo . [33] Por el contrario, las células de la masa celular interna restringen la replicación del ADNmt hasta que reciben las señales para diferenciarse en tipos celulares específicos". [33]

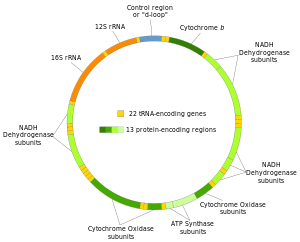
Genes del mtADN humano y su transcripción

Las dos hebras del ADN mitocondrial humano se distinguen como la hebra pesada y la hebra ligera. La hebra pesada es rica en guanina y codifica 12 subunidades del sistema de fosforilación oxidativa , dos ARN ribosómicos (12S y 16S) y 14 ARN de transferencia (ARNt). La hebra ligera codifica una subunidad y 8 ARNt. Por lo tanto, en total, el ADNmt codifica dos ARNr, 22 ARNt y 13 subunidades proteicas , todas ellas implicadas en el proceso de fosforilación oxidativa. [37] [38]
- La secuencia completa del ADN mitocondrial humano en forma gráfica
| Gene | Tipo | Producto | Posiciones en el mitogenoma | Hebra |
|---|---|---|---|---|
| MT-ATP8 | codificación de proteínas | ATP sintasa , subunidad Fo 8 (complejo V) | 08.366–08.572 (superposición con MT-ATP6) | yo |
| MT-ATP6 | codificación de proteínas | ATP sintasa , subunidad Fo 6 (complejo V) | 08.527–09.207 (superposición con MT-ATP8) | yo |
| CO1 MT | codificación de proteínas | Citocromo c oxidasa , subunidad 1 (complejo IV) | 05.904–07.445 | yo |
| CO2 Mt | codificación de proteínas | Citocromo c oxidasa , subunidad 2 (complejo IV) | 07.586–08.269 | yo |
| CO3-MT | codificación de proteínas | Citocromo c oxidasa , subunidad 3 (complejo IV) | 09.207–09.990 | yo |
| MT-CyB | codificación de proteínas | Citocromo b (complejo III) | 14.747–15.887 | yo |
| MT-ND1 | codificación de proteínas | NADH deshidrogenasa , subunidad 1 (complejo I) | 03.307–04.262 | yo |
| MT-ND2 | codificación de proteínas | NADH deshidrogenasa , subunidad 2 (complejo I) | 04.470–05.511 | yo |
| MT-ND3 | codificación de proteínas | NADH deshidrogenasa , subunidad 3 (complejo I) | 10.059–10.404 | yo |
| MT-ND4L | codificación de proteínas | NADH deshidrogenasa , subunidad 4L (complejo I) | 10 470–10 766 (superposición con MT-ND4) | yo |
| MT-ND4 | codificación de proteínas | NADH deshidrogenasa , subunidad 4 (complejo I) | 10 760–12 137 (superposición con MT-ND4L) | yo |
| MT-ND5 | codificación de proteínas | NADH deshidrogenasa , subunidad 5 (complejo I) | 12.337–14.148 | yo |
| MT-ND6 | codificación de proteínas | NADH deshidrogenasa , subunidad 6 (complejo I) | 14.149–14.673 | yo |
| MT-RNR2 | codificación de proteínas | Humanina | — | — |
| MT-TA | ARN de transferencia | ARNt- Alanina (Ala o A) | 05.587–05.655 | yo |
| MT-TR | ARN de transferencia | ARNt- Arginina (Arg o R) | 10.405–10.469 | yo |
| MT-TN | ARN de transferencia | ARNt- Asparagina (Asn o N) | 05.657–05.729 | yo |
| MT-TD | ARN de transferencia | ARNt- Ácido aspártico (Asp o D) | 07.518–07.585 | yo |
| MT-TC | ARN de transferencia | ARNt- Cisteína (Cys o C) | 05.761–05.826 | yo |
| MT-TE | ARN de transferencia | ARNt- Ácido glutámico (Glu o E) | 14.674–14.742 | yo |
| MT-TQ | ARN de transferencia | ARNt- Glutamina (Gln o Q) | 04.329–04.400 | yo |
| MT-TG | ARN de transferencia | ARNt- Glicina (Gly o G) | 09.991–10.058 | yo |
| De lunes a jueves | ARN de transferencia | ARNt- Histidina (His o H) | 12.138–12.206 | yo |
| MT-TI | ARN de transferencia | ARNt- Isoleucina (Ile o I) | 04.263–04.331 | yo |
| MT-TL1 | ARN de transferencia | ARNt- Leucina (Leu-UUR o L) | 03.230–03.304 | yo |
| MT-TL2 | ARN de transferencia | ARNt- Leucina (Leu-CUN o L) | 12.266–12.336 | yo |
| MT-TK | ARN de transferencia | ARNt- Lisina (Lys o K) | 08.295–08.364 | yo |
| MT-TM | ARN de transferencia | ARNt- Metionina (Met o M) | 04.402–04.469 | yo |
| MT-TF | ARN de transferencia | ARNt- Fenilalanina (Phe o F) | 00,577–00,647 | yo |
| MT-TP | ARN de transferencia | ARNt- Prolina (Pro o P) | 15.956–16.023 | yo |
| MT-TS1 | ARN de transferencia | ARNt- Serina (Ser-UCN o S) | 07.446–07.514 | yo |
| MT-TS2 | ARN de transferencia | ARNt- Serina (Ser-AGY o S) | 12.207–12.265 | yo |
| MT-TT | ARN de transferencia | ARNt- Treonina (Thr o T) | 15.888–15.953 | yo |
| MT-TW | ARN de transferencia | ARNt- Triptófano (Trp o W) | 05.512–05.579 | yo |
| MT-TY | ARN de transferencia | ARNt- Tirosina (Tyr o Y) | 05.826–05.891 | yo |
| Televisión MT | ARN de transferencia | ARNt- Valina (Val o V) | 01.602–01.670 | yo |
| MT-RNR1 | ARN ribosómico | Subunidad pequeña: SSU (12S) | 00,648–01,601 | yo |
| MT-RNR2 | ARN ribosómico | Subunidad grande: LSU (16S) | 01.671–03.229 | yo |
Entre la mayoría de las regiones codificantes de proteínas (pero no todas) se encuentran ARNt (véase el mapa del genoma mitocondrial humano). Durante la transcripción, los ARNt adquieren su forma característica de L, que es reconocida y escindida por enzimas específicas. Con el procesamiento del ARN mitocondrial, se liberan secuencias individuales de ARNm, ARNr y ARNt de la transcripción primaria. [40] Por lo tanto, los ARNt plegados actúan como puntuaciones de estructura secundaria. [41]
Regulación de la transcripción
Los promotores para la iniciación de la transcripción de las cadenas pesadas y ligeras se encuentran en la principal región no codificante del ADNmt llamada bucle de desplazamiento, el D-loop . [37] Existe evidencia de que la transcripción de los ARNr mitocondriales está regulada por el promotor de la cadena pesada 1 (HSP1), y la transcripción de las transcripciones policistrónicas que codifican las subunidades de la proteína está regulada por HSP2. [37]
La medición de los niveles de ARN codificados por ADNmt en tejidos bovinos ha demostrado que existen diferencias importantes en la expresión de los ARN mitocondriales en relación con el ARN tisular total. [42] Entre los 12 tejidos examinados, el nivel más alto de expresión se observó en el corazón, seguido del cerebro y las muestras de tejido esteroidogénico. [42]
Como lo demuestra el efecto de la hormona trófica ACTH sobre las células de la corteza suprarrenal, la expresión de los genes mitocondriales puede estar fuertemente regulada por factores externos, aparentemente para mejorar la síntesis de proteínas mitocondriales necesarias para la producción de energía. [42] Curiosamente, mientras que la expresión de genes codificadores de proteínas fue estimulada por ACTH, los niveles del ARNr 16S mitocondrial no mostraron cambios significativos. [42]
Herencia mitocondrial
En la mayoría de los organismos multicelulares , el ADNmt se hereda de la madre (herencia materna). Los mecanismos para esto incluyen la dilución simple (un óvulo contiene en promedio 200.000 moléculas de ADNmt, mientras que se ha informado que un espermatozoide humano sano contiene en promedio 5 moléculas), [43] [44] la degradación del ADNmt del esperma en el tracto genital masculino y en el óvulo fertilizado; y, al menos en algunos organismos, el fracaso del ADNmt del esperma para entrar en el óvulo. Cualquiera que sea el mecanismo, este patrón de herencia monoparental ( herencia uniparental ) de ADNmt se encuentra en la mayoría de los animales, la mayoría de las plantas y también en los hongos. [45]
En un estudio publicado en 2018, se informó que los bebés humanos heredan el ADNmt tanto de sus padres como de sus madres, lo que resulta en heteroplasmia del ADNmt , [46] un hallazgo que ha sido rechazado por otros científicos. [47] [48] [49]
Herencia femenina
En la reproducción sexual , las mitocondrias normalmente se heredan exclusivamente de la madre; las mitocondrias en los espermatozoides de los mamíferos suelen ser destruidas por el óvulo después de la fertilización. Además, las mitocondrias solo se encuentran en la cola del espermatozoide, que se utiliza para propulsar las células espermáticas y, a veces, la cola se pierde durante la fertilización. En 1999 se informó que las mitocondrias del espermatozoide paterno (que contienen ADNmt) están marcadas con ubiquitina para seleccionarlas para su posterior destrucción dentro del embrión . [50] Algunas técnicas de fertilización in vitro , en particular la inyección de un espermatozoide en un ovocito , pueden interferir con esto.
El hecho de que el ADN mitocondrial se herede principalmente por vía materna permite a los investigadores genealógicos rastrear el linaje materno muy atrás en el tiempo. ( El ADN del cromosoma Y , heredado por vía paterna, se utiliza de forma análoga para determinar la historia patrilineal ). Esto generalmente se logra en el ADN mitocondrial humano mediante la secuenciación de las regiones de control hipervariables (HVR1 o HVR2), y a veces la molécula completa del ADN mitocondrial, como una prueba de ADN genealógico . [51] HVR1, por ejemplo, consta de unos 440 pares de bases. Estos 440 pares de bases se comparan con las mismas regiones de otros individuos (ya sea personas específicas o sujetos en una base de datos) para determinar el linaje materno. La mayoría de las veces, la comparación se realiza con la secuencia de referencia de Cambridge revisada . Vilà et al. han publicado estudios que rastrean la descendencia matrilineal de los perros domésticos a partir de los lobos. [52] El concepto de Eva Mitocondrial se basa en el mismo tipo de análisis, intentando descubrir el origen de la humanidad rastreando el linaje en el tiempo.
El cuello de botella mitocondrial
Se puede esperar que las entidades sujetas a herencia uniparental y con poca o ninguna recombinación estén sujetas al trinquete de Muller , la acumulación de mutaciones deletéreas hasta que se pierde la funcionalidad. Las poblaciones animales de mitocondrias evitan esto a través de un proceso de desarrollo conocido como el cuello de botella del ADNmt . El cuello de botella explota procesos aleatorios en la célula para aumentar la variabilidad de célula a célula en la carga mutante a medida que un organismo se desarrolla: un solo óvulo con cierta proporción de ADNmt mutante produce un embrión en el que diferentes células tienen diferentes cargas mutantes. La selección a nivel celular puede entonces actuar para eliminar aquellas células con más ADNmt mutante, lo que lleva a una estabilización o reducción en la carga mutante entre generaciones. El mecanismo subyacente al cuello de botella es debatido, [53] [54] [55] [56] con un metaestudio matemático y experimental reciente que proporciona evidencia de una combinación de la partición aleatoria de ADNmt en las divisiones celulares y la renovación aleatoria de moléculas de ADNmt dentro de la célula. [34]
Herencia masculina
Se ha descubierto la herencia del ADN mitocondrial masculino en los pollos Plymouth Rock . [57] La evidencia también respalda casos raros de herencia mitocondrial masculina en algunos mamíferos. Específicamente, existen casos documentados en ratones, [58] [59] donde las mitocondrias heredadas del macho fueron posteriormente rechazadas. También se ha encontrado en ovejas, [60] y en ganado clonado. [61] Se han documentado casos raros de herencia mitocondrial masculina en humanos. [62] [63] [64] [46] Aunque muchos de estos casos involucran embriones clonados o rechazo posterior de las mitocondrias paternas, otros documentan herencia in vivo y persistencia en condiciones de laboratorio.
En los moluscos bivalvos se observa una herencia uniparental doble del ADNmt. En esas especies, las hembras tienen solo un tipo de ADNmt (F), mientras que los machos tienen ADNmt de tipo F en sus células somáticas, pero ADNmt de tipo M (que puede ser hasta un 30% divergente) en las células de la línea germinal . [65] También se han descrito mitocondrias heredadas por vía paterna en algunos insectos como las moscas de la fruta , [66] [67] las abejas melíferas , [68] y las cigarras periódicas . [69]
Donación de mitocondrias
Una técnica de FIV conocida como donación mitocondrial o terapia de reemplazo mitocondrial (TRM) da como resultado una descendencia que contiene ADNmt de una donante femenina y ADN nuclear de la madre y el padre. En el procedimiento de transferencia de huso, el núcleo de un óvulo se inserta en el citoplasma de un óvulo de una donante femenina al que se le ha extraído el núcleo, pero que aún contiene el ADNmt de la donante femenina. Luego, el óvulo compuesto se fertiliza con el esperma del macho. El procedimiento se utiliza cuando una mujer con mitocondrias genéticamente defectuosas desea procrear y producir descendencia con mitocondrias sanas. [70] El primer niño conocido que nació como resultado de la donación mitocondrial fue un niño nacido de una pareja jordana en México el 6 de abril de 2016. [71]
Mutaciones y enfermedades
Susceptibilidad
El concepto de que el ADNmt es particularmente susceptible a las especies reactivas de oxígeno generadas por la cadena respiratoria debido a su proximidad sigue siendo controvertido. [72] El ADNmt no acumula más daño oxidativo de bases que el ADN nuclear. [73] Se ha informado de que al menos algunos tipos de daño oxidativo del ADN se reparan de forma más eficiente en las mitocondrias que en el núcleo. [74] El ADNmt está empaquetado con proteínas que parecen ser tan protectoras como las proteínas de la cromatina nuclear. [75] Además, las mitocondrias desarrollaron un mecanismo único que mantiene la integridad del ADNmt a través de la degradación de genomas excesivamente dañados seguida de la replicación del ADNmt intacto/reparado. Este mecanismo no está presente en el núcleo y es posible gracias a las múltiples copias de ADNmt presentes en las mitocondrias. [76] El resultado de la mutación en el ADNmt puede ser una alteración en las instrucciones de codificación de algunas proteínas, [77] lo que puede tener un efecto en el metabolismo y/o la aptitud del organismo.
Enfermedad genética
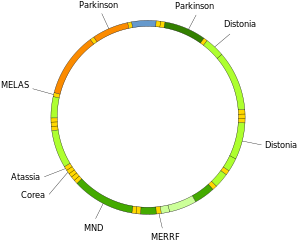
Las mutaciones del ADN mitocondrial pueden provocar una serie de enfermedades, entre ellas la intolerancia al ejercicio y el síndrome de Kearns-Sayre (KSS), que hace que una persona pierda la función completa del corazón, los ojos y los movimientos musculares. Algunas pruebas sugieren que podrían ser importantes contribuyentes al proceso de envejecimiento y las patologías asociadas a la edad . [78] Particularmente en el contexto de la enfermedad, la proporción de moléculas de ADNmt mutantes en una célula se denomina heteroplasmia . Las distribuciones intracelulares e intercelulares de la heteroplasmia dictan el inicio y la gravedad de la enfermedad [79] y están influenciadas por procesos estocásticos complicados dentro de la célula y durante el desarrollo. [34] [80]
Las mutaciones en los ARNt mitocondriales pueden ser responsables de enfermedades graves como los síndromes MELAS y MERRF . [81]
Las mutaciones en los genes nucleares que codifican proteínas que utilizan las mitocondrias también pueden contribuir a las enfermedades mitocondriales. Estas enfermedades no siguen patrones de herencia mitocondrial, sino patrones de herencia mendeliana. [82]
Uso en el diagnóstico de enfermedades
Recientemente, se ha utilizado una mutación en el ADNmt para ayudar a diagnosticar el cáncer de próstata en pacientes con biopsia de próstata negativa . [83] [84] Se pueden detectar alteraciones del ADNmt en los biofluidos de pacientes con cáncer. [85] El ADNmt se caracteriza por la alta tasa de polimorfismos y mutaciones. Algunos de los cuales se reconocen cada vez más como una causa importante de patología humana, como los trastornos de la fosforilación oxidativa (OXPHOS), la diabetes y sordera hereditarias maternas (MIDD), la diabetes mellitus tipo 2, las enfermedades neurodegenerativas , la insuficiencia cardíaca y el cáncer.
Relación con el envejecimiento
Aunque la idea es controvertida, algunas evidencias sugieren un vínculo entre el envejecimiento y la disfunción del genoma mitocondrial. [86] En esencia, las mutaciones en el ADNmt alteran un equilibrio cuidadoso de la producción de especies reactivas de oxígeno (ROS) y la eliminación enzimática de ROS (por enzimas como la superóxido dismutasa , la catalasa , la glutatión peroxidasa y otras). Sin embargo, algunas mutaciones que aumentan la producción de ROS (por ejemplo, al reducir las defensas antioxidantes) en los gusanos aumentan, en lugar de disminuir, su longevidad. [72] Además, las ratas topo desnudas , roedores del tamaño de los ratones , viven aproximadamente ocho veces más que los ratones a pesar de haber reducido, en comparación con los ratones, las defensas antioxidantes y aumentado el daño oxidativo a las biomoléculas. [87] Una vez, se pensó que había un bucle de retroalimentación positiva en funcionamiento (un "círculo vicioso"); a medida que el ADN mitocondrial acumula daño genético causado por radicales libres, las mitocondrias pierden su función y filtran radicales libres al citosol . Una disminución en la función mitocondrial reduce la eficiencia metabólica general. [88] Sin embargo, este concepto fue refutado concluyentemente cuando se demostró que los ratones, que fueron alterados genéticamente para acumular mutaciones de mtADN a un ritmo acelerado, envejecen prematuramente, pero sus tejidos no producen más ROS como predijo la hipótesis del "círculo vicioso". [89] Apoyando un vínculo entre la longevidad y el ADN mitocondrial, algunos estudios han encontrado correlaciones entre las propiedades bioquímicas del ADN mitocondrial y la longevidad de las especies. [90] La aplicación de un eliminador de ROS específico mitocondrial, que condujo a una longevidad significativa de los ratones estudiados, [91] sugiere que las mitocondrias aún pueden estar bien implicadas en el envejecimiento. Se están realizando investigaciones exhaustivas para investigar más a fondo este vínculo y los métodos para combatir el envejecimiento. Actualmente, la terapia génica y la suplementación nutracéutica son áreas populares de investigación en curso. [92] [93] Bjelakovic et al. analizaron los resultados de 78 estudios realizados entre 1977 y 2012, con un total de 296.707 participantes, y concluyeron que los suplementos antioxidantes no reducen la mortalidad por todas las causas ni prolongan la esperanza de vida, mientras que algunos de ellos, como el betacaroteno, la vitamina E y dosis más altas de vitamina A, pueden en realidad aumentar la mortalidad. [94]En un estudio reciente se demostró que la restricción dietética puede revertir las alteraciones del envejecimiento al afectar la acumulación de daño en el mtADN en varios órganos de ratas. Por ejemplo, la restricción dietética impidió la acumulación de daño en el mtADN relacionada con la edad en la corteza y la disminuyó en los pulmones y los testículos. [95]
Enfermedades neurodegenerativas
El aumento del daño del ADNmt es una característica de varias enfermedades neurodegenerativas .
Los cerebros de las personas con enfermedad de Alzheimer presentan niveles elevados de daño oxidativo del ADN tanto en el ADN nuclear como en el ADNmt, pero el ADNmt tiene niveles aproximadamente 10 veces más altos que el ADN nuclear. [96] Se ha propuesto que las mitocondrias envejecidas son el factor crítico en el origen de la neurodegeneración en la enfermedad de Alzheimer. [97] El análisis de los cerebros de pacientes con EA sugirió una función deteriorada de la vía de reparación del ADN , lo que causaría una reducción de la calidad general del ADNmt. [98]
En la enfermedad de Huntington , la proteína huntingtina mutante causa disfunción mitocondrial que implica la inhibición del transporte de electrones mitocondriales , niveles más altos de especies reactivas de oxígeno y un mayor estrés oxidativo . [99] La proteína huntingtina mutante promueve el daño oxidativo al ADNmt, así como al ADN nuclear, que puede contribuir a la patología de la enfermedad de Huntington . [100]
El producto de oxidación del ADN, 8-oxoguanina (8-oxoG), es un marcador bien establecido de daño oxidativo del ADN. En personas con esclerosis lateral amiotrófica (ELA), las enzimas que normalmente reparan los daños del ADN causados por 8-oxoG en el ADNmt de las neuronas motoras espinales están alteradas. [101] Por lo tanto, el daño oxidativo al ADNmt de las neuronas motoras puede ser un factor significativo en la etiología de la ELA.
Correlación de la composición de bases del ADNmt con la longevidad de los animales
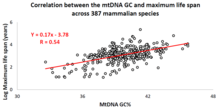
Durante la última década, un grupo de investigación israelí dirigido por el profesor Vadim Fraifeld ha demostrado que existen correlaciones fuertes y significativas entre la composición de la base del ADNmt y las esperanzas de vida máximas específicas de las especies animales. [102] [103] [104] Como se demostró en su trabajo, un mayor contenido de guanina + citosina ( GC% ) del ADNmt se asocia fuertemente con esperanzas de vida máximas más largas en las especies animales. Una observación adicional es que la correlación del GC% del ADNmt con las esperanzas de vida máximas es independiente de la correlación bien conocida entre la tasa metabólica de las especies animales y las esperanzas de vida máximas. El GC% del ADNmt y la tasa metabólica en reposo explican las diferencias en las esperanzas de vida máximas de las especies animales de manera multiplicativa (es decir, esperanza de vida máxima de las especies = su GC% de ADNmt * tasa metabólica). [103] Para apoyar a la comunidad científica en la realización de análisis comparativos entre las características del ADNmt y la longevidad en los animales, se creó una base de datos dedicada llamada MitoAge. [105]
El espectro mutacional del ADNmt es sensible a los rasgos del ciclo de vida específicos de cada especie
Las mutaciones de novo surgen debido a errores durante la replicación del ADN o debido a daños no reparados causados a su vez por mutágenos endógenos y exógenos. Durante mucho tiempo se ha creído que el ADNmt puede ser particularmente sensible al daño causado por especies reactivas de oxígeno (ROS), sin embargo, las sustituciones G>T, el sello distintivo del daño oxidativo en el genoma nuclear, son muy raras en el ADNmt y no aumentan con la edad. Al comparar los espectros mutacionales del ADNmt de cientos de especies de mamíferos, se ha demostrado recientemente que las especies con vidas prolongadas tienen una mayor tasa de sustituciones A>G en la cadena pesada monocatenaria. [106] Este descubrimiento condujo a la hipótesis de que A>G es un marcador específico de las mitocondrias del daño oxidativo asociado con la edad. Este hallazgo proporciona una explicación mutacional (contraria a la selectiva) para la observación de que las especies de larga vida tienen ADNmt rico en GC: las especies de larga vida se vuelven ricas en GC simplemente debido a su proceso sesgado de mutagénesis. Una asociación entre el espectro mutacional del ADNmt y los rasgos del ciclo de vida específicos de las especies de mamíferos abre la posibilidad de vincular estos factores y descubrir nuevos mutágenos específicos del ciclo de vida en diferentes grupos de organismos.
Relación con estructuras de ADN no B (no canónicas)
Los puntos de corte por deleción se producen frecuentemente dentro o cerca de regiones que muestran conformaciones no canónicas (no B), a saber, horquillas, cruciformes y elementos similares a hojas de trébol. [107] Además, hay datos que respaldan la participación de regiones intrínsecamente curvadas que distorsionan la hélice y tétradas G largas en la generación de eventos de inestabilidad. Además, se observaron consistentemente mayores densidades de puntos de corte dentro de regiones sesgadas por GC y en las proximidades del motivo de secuencia degenerada YMMYMNNMMHM. [108]
Uso en la ciencia forense
A diferencia del ADN nuclear, que se hereda de ambos padres y en el que los genes se reorganizan en el proceso de recombinación , normalmente no hay cambios en el ADNmt de los padres a los hijos. Aunque el ADNmt también se recombina, lo hace con copias de sí mismo dentro de la misma mitocondria. Debido a esto y a que la tasa de mutación del ADNmt animal es mayor que la del ADN nuclear, [109] el ADNmt es una herramienta poderosa para rastrear la ascendencia a través de las hembras ( matrilinaje ) y se ha utilizado en esta función para rastrear la ascendencia de muchas especies a lo largo de cientos de generaciones.
Los científicos forenses pueden utilizar la prueba de ADNmt en casos en los que el ADN nuclear está gravemente degradado. Las células autosómicas solo tienen dos copias de ADN nuclear, pero pueden tener cientos de copias de ADNmt debido a las múltiples mitocondrias presentes en cada célula. Esto significa que la evidencia altamente degradada que no sería beneficiosa para el análisis de STR podría usarse en el análisis de ADNmt. El ADNmt puede estar presente en huesos, dientes o cabello, que podrían ser los únicos restos que queden en el caso de una degradación grave. A diferencia del análisis de STR, la secuenciación de ADNmt utiliza la secuenciación de Sanger . La secuencia conocida y la secuencia cuestionada se comparan con la secuencia de referencia revisada de Cambridge para generar sus respectivos haplotipos. Si la secuencia de muestra conocida y la secuencia cuestionada se originaron a partir de la misma matrilina, uno esperaría ver secuencias idénticas y diferencias idénticas del rCRS. [110] Surgen casos en los que no hay muestras conocidas para recolectar y la secuencia desconocida se puede buscar en una base de datos como EMPOP. El Grupo de Trabajo Científico sobre Métodos de Análisis de ADN recomienda tres conclusiones para describir las diferencias entre una secuencia de ADNmt conocida y una secuencia de ADNmt cuestionada: exclusión para dos o más diferencias entre las secuencias, no concluyente si hay una diferencia de nucleótidos, o no se puede excluir si no hay diferencias de nucleótidos entre las dos secuencias. [111]
La rápida tasa de mutación (en animales) hace que el ADNmt sea útil para evaluar las relaciones genéticas de individuos o grupos dentro de una especie y también para identificar y cuantificar la filogenia (relaciones evolutivas; ver filogenética ) entre diferentes especies. Para ello, los biólogos determinan y luego comparan las secuencias de ADNmt de diferentes individuos o especies. Los datos de las comparaciones se utilizan para construir una red de relaciones entre las secuencias, que proporciona una estimación de las relaciones entre los individuos o especies de los que se tomaron los ADNmt. El ADNmt se puede utilizar para estimar la relación entre especies estrechamente relacionadas y distantemente relacionadas. Debido a la alta tasa de mutación del ADNmt en animales, las 3.ª posiciones de los codones cambian con relativa rapidez y, por lo tanto, proporcionan información sobre las distancias genéticas entre individuos o especies estrechamente relacionados. Por otra parte, la tasa de sustitución de las proteínas mt es muy baja, por lo que los cambios de aminoácidos se acumulan lentamente (con los correspondientes cambios lentos en las posiciones del 1.º y 2.º codón) y, por lo tanto, proporcionan información sobre las distancias genéticas de las especies distantes. Los modelos estadísticos que tratan las tasas de sustitución entre las posiciones de los codones por separado pueden utilizarse para estimar simultáneamente filogenias que contienen especies cercanas y distantes [81].
El ADN mitocondrial fue admitido como prueba por primera vez en un tribunal de los Estados Unidos en 1996 durante el caso Estado de Tennessee contra Paul Ware . [112]
En el caso de 1998 de la Commonwealth of Pennsylvania v. Patricia Lynne Rorrer en los Estados Unidos, [113] el ADN mitocondrial fue admitido como prueba en el estado de Pensilvania por primera vez. [114] [115] El caso apareció en el episodio 55 de la quinta temporada de la serie dramática sobre crímenes reales Forensic Files (temporada 5) . [116]
El ADN mitocondrial fue admitido por primera vez como prueba en California , Estados Unidos, en el exitoso procesamiento de David Westerfield por el secuestro y asesinato en 2002 de Danielle van Dam , de 7 años, en San Diego : se utilizó para la identificación tanto de humanos como de perros. [117] Este fue el primer juicio en los EE. UU. en admitir ADN canino. [118]
Los restos del rey Ricardo III , que murió en 1485, fueron identificados comparando su ADNmt con el de dos descendientes matrilineales de su hermana que estaban vivos en 2013, 527 años después de su muerte. [119]
Uso en biología evolutiva y biología sistemática.
El ADNmt se conserva en todos los organismos eucariotas debido al papel fundamental de las mitocondrias en la respiración celular . Sin embargo, debido a que la reparación del ADN es menos eficiente (en comparación con el ADN nuclear), tiene una tasa de mutación relativamente alta (pero lenta en comparación con otras regiones del ADN, como los microsatélites ), lo que lo hace útil para estudiar las relaciones evolutivas ( filogenia ) de los organismos. Los biólogos pueden determinar y luego comparar secuencias de ADNmt entre diferentes especies y usar las comparaciones para construir un árbol evolutivo para las especies examinadas.
Por ejemplo, mientras que la mayoría de los genes nucleares son casi idénticos entre los humanos y los chimpancés , sus genomas mitocondriales son un 9,8% diferentes. Los genomas mitocondriales de los humanos y los gorilas son un 11,8% diferentes, lo que sugiere que los humanos pueden estar más estrechamente relacionados con los chimpancés que con los gorilas. [120]
ADNmt en el ADN nuclear
Las secuencias completas del genoma de más de 66.000 personas revelaron que la mayoría de ellas tenían ADN mitocondrial insertado en sus genomas nucleares . Más del 90% de estos segmentos mitocondriales nucleares ( NUMT ) se insertaron después de que los humanos divergieran de los otros simios . Los resultados indican que dichas transferencias ocurren actualmente con una frecuencia de una vez cada aproximadamente 4.000 nacimientos humanos. [121]
Parece que el ADN organelar se transfiere al ADN nuclear con mucha más frecuencia de lo que se creía anteriormente. Esta observación también respalda la idea de la teoría endosimbionte de que los eucariotas han evolucionado a partir de endosimbiontes que se convirtieron en organelos mientras transferían la mayor parte de su ADN al núcleo, de modo que el genoma organelar se encogió en el proceso. [122]
Historia
El ADN mitocondrial fue descubierto en la década de 1960 por Margit MK Nass y Sylvan Nass mediante microscopía electrónica como hilos sensibles a la DNasa dentro de las mitocondrias, [123] y por Ellen Haslbrunner, Hans Tuppy y Gottfried Schatz mediante ensayos bioquímicos en fracciones mitocondriales altamente purificadas. [124]
Bases de datos de secuencias mitocondriales
Se han creado varias bases de datos especializadas para recopilar secuencias del genoma mitocondrial y otra información. Aunque la mayoría de ellas se centran en datos de secuencias, algunas de ellas incluyen información filogenética o funcional.
- AmtDB: una base de datos de genomas mitocondriales humanos antiguos. [125]
- InterMitoBase : una base de datos anotada y una plataforma de análisis de interacciones proteína-proteína para mitocondrias humanas. [126] (aparentemente, última actualización en 2010, pero aún disponible)
- MitoBreak : la base de datos de puntos de ruptura del ADN mitocondrial. [127]
- MitoFish y MitoAnnotator : una base de datos del genoma mitocondrial de peces. [128] Véase también Cawthorn et al. [129]
- Mitome: una base de datos para la genómica mitocondrial comparativa en animales metazoos [130] (ya no está disponible)
- MitoRes: un recurso de genes mitocondriales codificados en el núcleo y sus productos en metazoos [131] (aparentemente ya no se actualiza)
- MitoSatPlant : base de datos de microsatélites mitocondriales de viridiplantae. [132]
- MitoZoa 2.0: una base de datos para análisis comparativos y evolutivos de genomas mitocondriales en metazoos. [133] (ya no está disponible)
Bases de datos de asociación entre ADNmt y fenotipo
Los estudios de asociación de todo el genoma pueden revelar asociaciones de genes de ADNmt y sus mutaciones con fenotipos que incluyen la esperanza de vida y los riesgos de enfermedades. En 2021, el estudio de asociación de todo el genoma del ADN mitocondrial más grande, basado en el Biobanco del Reino Unido , reveló 260 nuevas asociaciones con fenotipos que incluyen la esperanza de vida y los riesgos de enfermedades, por ejemplo, la diabetes tipo 2. [134] [135]
Bases de datos de mutaciones mitocondriales
Existen varias bases de datos especializadas que reportan polimorfismos y mutaciones en el ADN mitocondrial humano, junto con la evaluación de su patogenicidad.
- MitImpact : una colección de predicciones de patogenicidad precalculadas para todos los cambios de nucleótidos que causan sustituciones no sinónimas en genes codificadores de proteínas mitocondriales humanas MitImpact 3D - Laboratorio de bioinformática IRCCS-CSS.
- MITOMAP : Un compendio de polimorfismos y mutaciones en el ADN mitocondrial humano WebHome < MITOMAP < Foswiki.
Véase también
Referencias
- ^ Siekevitz P (1957). "La central eléctrica de la célula". Scientific American . 197 (1): 131–40. Código Bibliográfico :1957SciAm.197a.131S. doi :10.1038/scientificamerican0757-131.
- ^ Iborra FJ, Kimura H, Cook PR (mayo de 2004). "La organización funcional de los genomas mitocondriales en células humanas". BMC Biology . 2 : 9. doi : 10.1186/1741-7007-2-9 . PMC 425603 . PMID 15157274.
- ^ Sykes B (10 de septiembre de 2003). «ADN mitocondrial e historia humana». El genoma humano . Wellcome Trust . Archivado desde el original el 7 de septiembre de 2015. Consultado el 5 de febrero de 2012 .
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, et al. (abril de 1981). "Secuencia y organización del genoma mitocondrial humano". Nature . 290 (5806): 457–465. Bibcode :1981Natur.290..457A. doi :10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ Chinnery, Patrick Francis; Hudson, Gavin (junio de 2013). "Genética mitocondrial". British Medical Bulletin . 106 (1): 135–159. doi :10.1093/bmb/ldt017. ISSN 0007-1420. PMC 3675899 . PMID 23704099.
- ^ Boursot P, Bonhomme F (1 de enero de 1986). "[No disponible]". Génétique, Selección, Évolución . 18 (1): 73–98. doi : 10.1186/1297-9686-18-1-73 . PMC 2713894 . PMID 22879234.
- ^ Delsuc F, Stanhope MJ, Douzery EJ (agosto de 2003). "Sistemática molecular de armadillos (Xenarthra, Dasypodidae): contribución de los análisis de máxima verosimilitud y bayesianos de genes mitocondriales y nucleares". Filogenética molecular y evolución . 28 (2): 261–275. Bibcode :2003MolPE..28..261D. doi :10.1016/s1055-7903(03)00111-8. PMID 12878463. Archivado desde el original el 4 de noviembre de 2018. Consultado el 4 de noviembre de 2018 .
- ^ Hassanin A, An J, Ropiquet A, Nguyen TT, Couloux A (marzo de 2013). "Combinación de múltiples intrones autosómicos para estudiar la filogenia y taxonomía superficial de los mamíferos laurasiaterios: aplicación a la tribu Bovini (Cetartiodactyla, Bovidae)". Filogenética molecular y evolución . 66 (3): 766–775. Bibcode :2013MolPE..66..766H. doi :10.1016/j.ympev.2012.11.003. PMID 23159894.
- ^ abc Johnston IG, Williams BP (febrero de 2016). "La inferencia evolutiva entre eucariotas identifica presiones específicas que favorecen la retención de genes mitocondriales" (PDF) . Cell Systems . 2 (2): 101–111. doi : 10.1016/j.cels.2016.01.013 . PMID 27135164.
- ^ van der Giezen M, Tovar J, Clark CG (2005). "Organelos derivados de mitocondrias en protistas y hongos". Un estudio de biología celular . Revista internacional de citología. Vol. 244. págs. 175–225. doi :10.1016/S0074-7696(05)44005-X. ISBN 978-0-12-364648-4. Número de identificación personal 16157181.
- ^ Adams KL, Palmer JD (diciembre de 2003). "Evolución del contenido genético mitocondrial: pérdida de genes y transferencia al núcleo". Filogenética molecular y evolución . 29 (3): 380–395. Bibcode :2003MolPE..29..380A. doi :10.1016/S1055-7903(03)00194-5. PMID 14615181.
- ^ Björkholm P, Harish A, Hagström E, Ernst AM, Andersson SG (agosto de 2015). "Los genomas mitocondriales se conservan mediante restricciones selectivas en la selección de proteínas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (33): 10154–10161. Bibcode :2015PNAS..11210154B. doi : 10.1073/pnas.1421372112 . PMC 4547212 . PMID 26195779.
- ^ Allen JF (agosto de 2015). "Por qué los cloroplastos y las mitocondrias conservan sus propios genomas y sistemas genéticos: coubicación para la regulación redox de la expresión génica". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (33): 10231–10238. Bibcode :2015PNAS..11210231A. doi : 10.1073/pnas.1500012112 . PMC 4547249 . PMID 26286985.
- ^ ab Kolesnikov AA, Gerasimov ES (diciembre de 2012). "Diversidad de la organización del genoma mitocondrial". Bioquímica. Biokhimiia . 77 (13): 1424–1435. doi :10.1134/S0006297912130020. PMID 23379519. S2CID 14441187.
- ^ Nosek J, Tomáska L, Fukuhara H, Suyama Y, Kovác L (mayo de 1998). "Genomas mitocondriales lineales: 30 años después". Tendencias en genética . 14 (5): 184–188. doi :10.1016/S0168-9525(98)01443-7. PMID 9613202.
- ^ Lavrov DV, Pett W (septiembre de 2016). "ADN mitocondrial animal como no lo conocemos: organización y evolución del genoma mitocondrial en linajes no bilaterales". Genome Biology and Evolution . 8 (9): 2896–2913. doi :10.1093/gbe/evw195. PMC 5633667 . PMID 27557826.
- ^ ab Boore, JL (1 de abril de 1999). "Animal mitochondrial genomes" (Genomas mitocondriales animales). Nucleic Acids Research (Investigación de ácidos nucleicos ) . 27 (8): 1767–1780. doi :10.1093/nar/27.8.1767. PMC 148383. PMID 10101183 .
- ^ Stampar SN, Broe MB, Macrander J, Reitzel AM, Brugler MR, Daly M (abril de 2019). "Genoma mitocondrial lineal en Anthozoa (Cnidaria): un estudio de caso en Ceriantharia". Scientific Reports . 9 (1): 6094. Bibcode :2019NatSR...9.6094S. doi :10.1038/s41598-019-42621-z. PMC 6465557 . PMID 30988357.
- ^ Polimorfismo en el genoma mitocondrial del ctenóforo Pleurobrachia bachei y su rápida evolución continua - bioRxiv
- ^ Yahalomi D, Atkinson SD, Neuhof M, Chang ES, Philippe H, Cartwright P, et al. (marzo de 2020). "Un parásito cnidario del salmón (Myxozoa: Henneguya) carece de un genoma mitocondrial". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 117 (10): 5358–5363. Bibcode :2020PNAS..117.5358Y. doi : 10.1073/pnas.1909907117 . PMC 7071853 . PMID 32094163.
- ^ Starr M (25 de febrero de 2020). «Los científicos encuentran el primer animal que no necesita oxígeno para sobrevivir». ScienceAlert . Archivado desde el original el 25 de febrero de 2020. Consultado el 25 de febrero de 2020 .
- ^ de Queiroz, Casley Borges; Santana, Mateus Ferreira; Pereira Vidigal, Pedro M.; de Queiroz, Marisa Vieira (15 de marzo de 2018). "Análisis comparativo del genoma mitocondrial del hongo Colletotrichum lindemuthianum, agente causal de la antracnosis en el frijol". Microbiología y Biotecnología Aplicadas . 102 (6): 2763–2778. doi :10.1007/s00253-018-8812-0. PMID 29453633. S2CID 3341230.
- ^ Masutani, Bansho; Arimura, Shin-ichi; Morishita, Shinichi (12 de enero de 2021). "Investigación del panorama genómico mitocondrial de Arabidopsis thaliana mediante secuenciación de lectura larga". PLOS Computational Biology . 17 (1): e1008597. Bibcode :2021PLSCB..17E8597M. doi : 10.1371/journal.pcbi.1008597 . PMC 7833223 . PMID 33434206.
- ^ Kozik, Alexander; Rowan, Beth A.; Lavelle, Dean; Berke, Lidija; Schranz, M. Eric; Michelmore, Richard W.; Christensen, Alan C. (30 de agosto de 2019). "La realidad alternativa del ADN mitocondrial de las plantas: un anillo no las gobierna a todas". PLOS Genetics . 15 (8): e1008373. doi : 10.1371/journal.pgen.1008373 . PMC 6742443 . PMID 31469821.
- ^ Fonseca PL, De-Paula RB, Araújo DS, Tomé LM, Mendes-Pereira T, Rodrigues WF, et al. (2021). "Caracterización global de mitogenomas fúngicos: nuevos conocimientos sobre la diversidad genómica y el dinamismo de los genes codificantes y elementos accesorios". Frontiers in Microbiology . 12 : 787283. doi : 10.3389/fmicb.2021.787283 . PMC 8672057 . PMID 34925295.
- ^ Sloan DB, Alverson AJ, Chuckalovcak JP, Wu M, McCauley DE, Palmer JD, Taylor DR (enero de 2012). "Evolución rápida de genomas enormes y multicromosómicos en mitocondrias de plantas con flores con tasas de mutación excepcionalmente altas". PLOS Biology . 10 (1): e1001241. doi : 10.1371/journal.pbio.1001241 . PMC 3260318 . PMID 22272183.
- ^ Ward BL, Anderson RS, Bendich AJ (septiembre de 1981). "El genoma mitocondrial es grande y variable en una familia de plantas (cucurbitáceas)". Cell . 25 (3): 793–803. doi :10.1016/0092-8674(81)90187-2. PMID 6269758. S2CID 45090891.
- ^ Alverson AJ, Rice DW, Dickinson S, Barry K, Palmer JD (julio de 2011). "Orígenes y recombinación del genoma mitocondrial multicromosómico de tamaño bacteriano del pepino". The Plant Cell . 23 (7): 2499–2513. doi : 10.1105 /tpc.111.087189. JSTOR 41433488. PMC 3226218. PMID 21742987.
- ^ "ADN mitocondrial (ADNmt)" (PDF) . Integrated DNA Technologies. Archivado desde el original (PDF) el 29 de julio de 2016 . Consultado el 25 de febrero de 2016 .
- ^ Tyagi S, Pande V, Das A (febrero de 2014). "Secuencia completa del genoma mitocondrial de un aislado de campo de Plasmodium falciparum de la India". The Korean Journal of Parasitology . 52 (1): 99–103. doi :10.3347/kjp.2014.52.1.99. PMC 3949004 . PMID 24623891.
- ^ Yakubovskaya E, Chen Z, Carrodeguas JA, Kisker C, Bogenhagen DF (enero de 2006). "La ADN polimerasa mitocondrial humana funcional gamma forma un heterotrímero". The Journal of Biological Chemistry . 281 (1): 374–382. doi : 10.1074/jbc.M509730200 . PMID 16263719.
- ^ Jemt E, Farge G, Bäckström S, Holmlund T, Gustafsson CM, Falkenberg M (noviembre de 2011). "La helicasa de ADN mitocondrial TWINKLE puede ensamblarse en una plantilla circular cerrada y apoyar la iniciación de la síntesis de ADN". Nucleic Acids Research . 39 (21): 9238–9249. doi :10.1093/nar/gkr653. PMC 3241658 . PMID 21840902.
- ^ abc St John JC, Facucho-Oliveira J, Jiang Y, Kelly R, Salah R (2010). "Transmisión, replicación y herencia del ADN mitocondrial: un viaje desde el gameto a través del embrión y hacia la descendencia y las células madre embrionarias". Actualización sobre reproducción humana . 16 (5): 488–509. doi : 10.1093/humupd/dmq002 . PMID 20231166.
- ^ abc Johnston IG, Burgstaller JP, Havlicek V, Kolbe T, Rülicke T, Brem G, et al. (junio de 2015). "Modelado estocástico, inferencia bayesiana y nuevas mediciones in vivo aclaran el debatido mecanismo de cuello de botella del ADNmt". eLife . 4 : e07464. arXiv : 1512.02988 . doi : 10.7554/eLife.07464 . PMC 4486817 . PMID 26035426.
- ^ Shuster RC, Rubenstein AJ, Wallace DC (septiembre de 1988). "ADN mitocondrial en células sanguíneas humanas anucleadas". Comunicaciones de investigación bioquímica y biofísica . 155 (3): 1360–1365. doi :10.1016/s0006-291x(88)81291-9. PMID 3178814.
- ^ Zhang D, Keilty D, Zhang ZF, Chian RC (marzo de 2017). "Mitocondrias en el envejecimiento de los ovocitos: conocimiento actual". Facts, Views & Vision in ObGyn . 9 (1): 29–38. PMC 5506767 . PMID 28721182.
- ^ abc Barshad G, Marom S, Cohen T, Mishmar D (septiembre de 2018). "Transcripción del ADN mitocondrial y su regulación: una perspectiva evolutiva". Tendencias en genética . 34 (9): 682–692. doi :10.1016/j.tig.2018.05.009. PMID 29945721. S2CID 49430452.
- ^ Barchiesi A, Vascotto C (mayo de 2019). "Transcripción, procesamiento y desintegración del ARN mitocondrial en la salud y la enfermedad". Revista internacional de ciencias moleculares . 20 (9): 2221. doi : 10.3390/ijms20092221 . PMC 6540609 . PMID 31064115.
- ^ Mitocondria del Homo sapiens , genoma completo. «Secuencia de referencia de Cambridge revisada (rCRS): acceso NC_012920» Archivado el 6 de abril de 2020 en Wayback Machine . Recuperado el 20 de febrero de 2017.
- ^ Falkenberg M, Larsson NG, Gustafsson CM (19 de junio de 2007). "Replicación y transcripción del ADN en mitocondrias de mamíferos". Revista Anual de Bioquímica . 76 (1): 679–699. doi :10.1146/annurev.biochem.76.060305.152028. PMID 17408359.
- ^ Ojala D, Montoya J, Attardi G (abril de 1981). "Modelo de puntuación de ARNt del procesamiento del ARN en mitocondrias humanas". Nature . 290 (5806): 470–474. Bibcode :1981Natur.290..470O. doi :10.1038/290470a0. PMID 7219536. S2CID 4323371.
- ^ abcd Raikhinstein M, Hanukoglu I (noviembre de 1993). "ARN codificados por el genoma mitocondrial: regulación diferencial por corticotropina en células adrenocorticales bovinas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 90 (22): 10509–10513. Bibcode :1993PNAS...9010509R. doi : 10.1073/pnas.90.22.10509 . PMC 47806 . PMID 7504267.
- ^ Wolff JN, Gemmell NJ (noviembre de 2008). "Perdido en el cigoto: la dilución del ADNmt paterno tras la fertilización". Heredity . 101 (5): 429–434. doi : 10.1038/hdy.2008.74 . PMID 18685570. S2CID 5065133.
- ^ Gabriel MS, Chan SW, Alhathal N, Chen JZ, Zini A (agosto de 2012). "Influencia de la varicocelectomía microquirúrgica en el número de copias del ADN mitocondrial del esperma humano: un estudio piloto". Revista de reproducción asistida y genética . 29 (8): 759–764. doi :10.1007/s10815-012-9785-z. PMC 3430774 . PMID 22562241.
- ^ Mendoza H, Perlin MH, Schirawski J (mayo de 2020). "Herencia mitocondrial en hongos fitopatógenos: ¿todo se sabe o no?". Int J Mol Sci . 21 (11): 3883. doi : 10.3390/ijms21113883 . PMC 7312866 . PMID 32485941.
- ^ ab Luo S, Valencia CA, Zhang J, Lee NC, Slone J, Gui B, et al. (diciembre de 2018). "Herencia biparental del ADN mitocondrial en humanos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 115 (51): 13039–13044. Bibcode :2018PNAS..11513039L. doi : 10.1073/pnas.1810946115 . PMC 6304937 . PMID 30478036.
- ^ Pagnamenta AT, Wei W, Rahman S, Chinnery PF (agosto de 2021). "Revisión de la herencia biparental del ADN mitocondrial". Reseñas de la naturaleza. Genética . 22 (8): 477–478. doi :10.1038/s41576-021-00380-6. PMID 34031572. S2CID 235199218.
- ^ Salas A, Schönherr S, Bandelt HJ, Gómez-Carballa A, Weissensteiner H (julio de 2020). "Las afirmaciones extraordinarias requieren evidencia extraordinaria en la herencia biparental del ADNmt". Forensic Science International. Genética . 47 : 102274. doi :10.1016/j.fsigen.2020.102274. PMID 32330850. S2CID 216131636.
- ^ Wei W, Pagnamenta AT, Gleadall N, Sanchis-Juan A, Stephens J, Broxholme J, Tuna S, Odhams CA, Fratter C, Turro E, Caulfield MJ, Taylor JC, Rahman S, Chinnery PF (abril de 2020). "Los segmentos de ADN mitocondrial nuclear se parecen al ADN mitocondrial heredado por vía paterna en humanos". Nature Communications . 11 (1): 1740. Bibcode :2020NatCo..11.1740W. doi :10.1038/s41467-020-15336-3. PMC 7142097 . PMID 32269217.
- ^ Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G (noviembre de 1999). "Etiqueta de ubiquitina para mitocondrias de espermatozoides". Nature . 402 (6760): 371–372. Bibcode :1999Natur.402..371S. doi :10.1038/46466. PMID 10586873. S2CID 205054671.Discutido en: Travis J (2000). "Los óvulos de mamá ejecutan las mitocondrias de papá". Science News . 157 (1): 5. doi :10.2307/4012086. JSTOR 4012086. Archivado desde el original el 19 de diciembre de 2007.
- ^ "Contratación de una empresa de pruebas de ADN Genealogía". Family Search . La Iglesia de Jesucristo de los Santos de los Últimos Días. 2016. Archivado desde el original el 3 de octubre de 2016 . Consultado el 2 de octubre de 2016 .
- ^ Vilà C, Savolainen P, Maldonado JE, Amorim IR, Rice JE, Honeycutt RL, et al. (junio de 1997). «Múltiples y antiguos orígenes del perro doméstico» (PDF) . Science . 276 (5319): 1687–1689. doi :10.1126/science.276.5319.1687. PMID 9180076. Archivado (PDF) desde el original el 9 de octubre de 2022.
- ^ Wolff JN, White DJ, Woodhams M, White HE, Gemmell NJ (2011). "La fuerza y el momento del cuello de botella mitocondrial en el salmón sugieren un mecanismo conservado en los vertebrados". PLOS ONE . 6 (5): e20522. Bibcode :2011PLoSO...620522W. doi : 10.1371/journal.pone.0020522 . PMC 3105079 . PMID 21655224.
- ^ Cree LM, Samuels DC, de Sousa Lopes SC, Rajasimha HK, Wonnapinij P, Mann JR, et al. (febrero de 2008). "Una reducción de las moléculas de ADN mitocondrial durante la embriogénesis explica la rápida segregación de genotipos". Genética de la Naturaleza . 40 (2): 249–254. doi :10.1038/ng.2007.63. PMID 18223651. S2CID 205344980.
- ^ Cao L, Shitara H, Horii T, Nagao Y, Imai H, Abe K, et al. (Marzo de 2007). "El cuello de botella mitocondrial se produce sin reducción del contenido de ADNmt en las células germinales de ratón hembra". Genética de la Naturaleza . 39 (3): 386–390. doi :10.1038/ng1970. PMID 17293866. S2CID 10686347.
- ^ Wai T, Teoli D, Shoubridge EA (diciembre de 2008). "El cuello de botella genético del ADN mitocondrial resulta de la replicación de una subpoblación de genomas". Nature Genetics . 40 (12): 1484–1488. doi :10.1038/ng.258. PMID 19029901. S2CID 225349.
- ^ Alexander M, Ho SY, Molak M, Barnett R, Carlborg Ö, Dorshorst B, et al. (octubre de 2015). "El análisis mitogenómico de un pedigrí de pollos de 50 generaciones revela una tasa rápida de evolución mitocondrial y evidencia de herencia paterna del ADNmt". Biology Letters . 11 (10): 20150561. doi :10.1098/rsbl.2015.0561. PMC 4650172 . PMID 26510672.
- ^ Gyllensten U, Wharton D, Josefsson A, Wilson AC (julio de 1991). "Herencia paterna del ADN mitocondrial en ratones". Nature . 352 (6332): 255–257. Bibcode :1991Natur.352..255G. doi :10.1038/352255a0. PMID 1857422. S2CID 4278149.
- ^ Shitara H, Hayashi JI, Takahama S, Kaneda H, Yonekawa H (febrero de 1998). "Herencia materna del ADNmt de ratón en híbridos interespecíficos: segregación del ADNmt paterno filtrado seguida de la prevención de la fuga paterna posterior". Genética . 148 (2): 851–857. doi :10.1093/genetics/148.2.851. PMC 1459812 . PMID 9504930.
- ^ Zhao X, Li N, Guo W, Hu X, Liu Z, Gong G, et al. (octubre de 2004). "Más pruebas de la herencia paterna del ADN mitocondrial en las ovejas (Ovis aries)". Heredity . 93 (4): 399–403. doi : 10.1038/sj.hdy.6800516 . PMID 15266295.
- ^ Steinborn R, Zakhartchenko V, Jelyazkov J, Klein D, Wolf E, Müller M, Brem G (abril de 1998). "Composición del ADN mitocondrial parental en embriones bovinos clonados". FEBS Letters . 426 (3): 352–356. Bibcode :1998FEBSL.426..352S. doi : 10.1016/S0014-5793(98)00350-0 . PMID 9600265.
- ^ Singh AN (abril de 2012). "Familia A105 decodificada: descubrimiento de huellas dactilares de todo el genoma para la medicina genómica personalizada". ScienceMED . 3 (2): 115–126.
- ^ Singh AN (mayo de 2018). "Informática biomédica personalizada". Análisis de big data . 3 . doi : 10.1186/s41044-018-0030-3 .
- ^ Schwartz M, Vissing J (agosto de 2002). "Herencia paterna del ADN mitocondrial". The New England Journal of Medicine . 347 (8): 576–580. doi : 10.1056/NEJMoa020350 . PMID 12192017.
- ^ Passamonti M, Ghiselli F (febrero de 2009). "Herencia doblemente uniparental: dos genomas mitocondriales, un modelo valioso para la herencia y evolución del ADN de orgánulos". ADN y biología celular . 28 (2): 79–89. doi :10.1089/dna.2008.0807. PMID 19196051.
- ^ Kondo R, Matsuura ET, Chigusa SI (abril de 1992). "Observación adicional de la transmisión paterna del ADN mitocondrial de Drosophila mediante el método de amplificación selectiva por PCR". Investigación genética . 59 (2): 81–84. doi : 10.1017/S0016672300030287 . PMID 1628820.
- ^ Wolff JN, Nafisinia M, Sutovsky P, Ballard JW (enero de 2013). "Transmisión paterna del ADN mitocondrial como parte integral de la herencia mitocondrial en metapoblaciones de Drosophila simulans". Heredity . 110 (1): 57–62. doi :10.1038/hdy.2012.60. PMC 3522233 . PMID 23010820.
- ^ Meusel MS, Moritz RF (diciembre de 1993). "Transferencia de ADN mitocondrial paterno durante la fertilización de huevos de abejas melíferas (Apis mellifera L.)". Genética actual . 24 (6): 539–543. doi :10.1007/BF00351719. PMID 8299176. S2CID 15621371.
- ^ Fontaine KM, Cooley JR, Simon C (septiembre de 2007). "Evidencia de fuga paterna en cigarras periódicas híbridas (Hemiptera: Magicicada spp.)". PLOS ONE . 2 (9): e892. Bibcode :2007PLoSO...2..892F. doi : 10.1371/journal.pone.0000892 . PMC 1963320 . PMID 17849021.
- ^ Frith M (14 de octubre de 2003). "Prohibición a los científicos que intenten crear bebés con tres padres". The Independent .[ enlace muerto permanente ]
- ^ Roberts M (27 de septiembre de 2016). «Nace el primer 'bebé de tres personas' gracias al nuevo método». BBC News . Archivado desde el original el 30 de mayo de 2019. Consultado el 28 de septiembre de 2016 .
- ^ ab Alexeyev MF (octubre de 2009). "¿Hay algo más en el envejecimiento que el ADN mitocondrial y las especies reactivas de oxígeno?". The FEBS Journal . 276 (20): 5768–5787. doi :10.1111/j.1742-4658.2009.07269.x. PMC 3097520 . PMID 19796285.
- ^ Anson RM, Hudson E, Bohr VA (febrero de 2000). "Se ha sobreestimado el daño oxidativo endógeno mitocondrial". FASEB Journal . 14 (2): 355–360. doi : 10.1096/fasebj.14.2.355 . PMID 10657991. S2CID 19771110.
- ^ Thorslund T, Sunesen M, Bohr VA, Stevnsner T (abril de 2002). "La reparación de 8-oxoG es más lenta en genes nucleares endógenos que en ADN mitocondrial y no presenta sesgo de hebra". Reparación de ADN . 1 (4): 261–273. doi :10.1016/S1568-7864(02)00003-4. PMID 12509245. Archivado desde el original el 31 de diciembre de 2019 . Consultado el 30 de junio de 2019 .
- ^ Guliaeva NA, Kuznetsova EA, Gaziev AI (2006). "[Las proteínas asociadas con el ADN mitocondrial lo protegen contra la acción de los rayos X y el peróxido de hidrógeno]" [Las proteínas asociadas con el ADN mitocondrial lo protegen contra la acción de los rayos X y el peróxido de hidrógeno]. Biofizika (en ruso). 51 (4): 692–697. PMID 16909848.
- ^ Alexeyev M, Shokolenko I, Wilson G, LeDoux S (mayo de 2013). "El mantenimiento de la integridad del ADN mitocondrial: análisis crítico y actualización". Cold Spring Harbor Perspectives in Biology . 5 (5): a012641. doi :10.1101/cshperspect.a012641. PMC 3632056 . PMID 23637283.
- ^ Hogan CM (2010). "Mutación". En Monosson E, Cleveland CJ (eds.). Enciclopedia de la Tierra . Washington DC: Consejo Nacional para la Ciencia y el Medio Ambiente. Archivado desde el original el 30 de abril de 2011. Consultado el 18 de abril de 2011 .
- ^ Alexeyev MF, Ledoux SP, Wilson GL (octubre de 2004). "ADN mitocondrial y envejecimiento". Clinical Science . 107 (4): 355–364. doi :10.1042/CS20040148. PMID 15279618. S2CID 5747202.
- ^ Burgstaller JP, Johnston IG, Poulton J (enero de 2015). "Enfermedad del ADN mitocondrial e implicaciones del desarrollo para las estrategias reproductivas". Reproducción humana molecular . 21 (1): 11–22. doi :10.1093/molehr/gau090. PMC 4275042 . PMID 25425607.
- ^ Burgstaller JP, Johnston IG, Jones NS, Albrechtová J, Kolbe T, Vogl C, et al. (junio de 2014). "La segregación de ADNmt en tejidos heteroplásmicos es común in vivo y modulada por diferencias de haplotipos y estadio de desarrollo". Cell Reports . 7 (6): 2031–2041. doi :10.1016/j.celrep.2014.05.020. PMC 4570183 . PMID 24910436.
- ^ ab Taylor RW, Turnbull DM (mayo de 2005). "Mutaciones del ADN mitocondrial en enfermedades humanas". Nature Reviews. Genetics . 6 (5): 389–402. doi :10.1038/nrg1606. PMC 1762815 . PMID 15861210.
- ^ Angelini C, Bello L, Spinazzi M, Ferrati C (julio de 2009). "Trastornos mitocondriales del genoma nuclear". Acta Myologica . 28 (1): 16–23. PMC 2859630 . PMID 19772191.
- ^ Reguly B, Jakupciak JP, Parr RL (octubre de 2010). "La deleción del genoma mitocondrial de 3,4 kb sirve como biomarcador predictivo sustituto del cáncer de próstata en muestras de biopsia histopatológicamente benignas". Revista de la Asociación Urológica Canadiense . 4 (5): E118–E122. doi :10.5489/cuaj.932. PMC 2950771 . PMID 20944788.
- ^ Robinson K, Creed J, Reguly B, Powell C, Wittock R, Klein D, et al. (junio de 2010). "Predicción precisa de los resultados de la biopsia de próstata repetida mediante un ensayo de deleción de ADN mitocondrial". Cáncer de próstata y enfermedades prostáticas . 13 (2): 126–131. doi :10.1038/pcan.2009.64. PMID 20084081. S2CID 25050759.
- ^ Mair R, Mouliere F, Smith CG, Chandrananda D, Gale D, Marass F, et al. (enero de 2019). "La medición del ADN tumoral mitocondrial libre de células plasmáticas mejora la detección del glioblastoma en modelos de xenoinjerto ortotópico derivados del paciente". Cancer Research . 79 (1): 220–230. doi :10.1158/0008-5472.CAN-18-0074. PMC 6753020 . PMID 30389699. Archivado desde el original el 24 de septiembre de 2019 . Consultado el 24 de septiembre de 2019 .
- ^ de Grey A (1999). La teoría de los radicales libres mitocondriales del envejecimiento (PDF) . Landes Bioscience. ISBN 978-1-57059-564-6. Archivado (PDF) del original el 3 de junio de 2016 . Consultado el 1 de mayo de 2016 .[ página necesaria ]
- ^ Lewis KN, Andziak B, Yang T, Buffenstein R (octubre de 2013). "La respuesta de la rata topo desnuda al estrés oxidativo: simplemente hay que afrontarlo". Antioxidantes y señalización redox . 19 (12): 1388–1399. doi :10.1089/ars.2012.4911. PMC 3791056 . PMID 23025341.
- ^ Shigenaga MK, Hagen TM, Ames BN (noviembre de 1994). "Daño oxidativo y decaimiento mitocondrial en el envejecimiento". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (23): 10771–10778. Bibcode :1994PNAS...9110771S. doi : 10.1073/pnas.91.23.10771 . JSTOR 2365473. PMC 45108 . PMID 7971961.
- ^ Trifunovic A, Hansson A, Wredenberg A, Rovio AT, Dufour E, Khvorostov I, et al. (diciembre de 2005). "Las mutaciones somáticas del mtADN causan fenotipos de envejecimiento sin afectar la producción de especies reactivas de oxígeno". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (50): 17993–17998. Bibcode :2005PNAS..10217993T. doi : 10.1073/pnas.0508886102 . JSTOR 4152716. PMC 1312403 . PMID 16332961.
- ^ Aledo JC, Li Y, de Magalhães JP, Ruíz-Camacho M, Pérez-Claros JA (abril de 2011). "La metionina codificada mitocondrialmente está inversamente relacionada con la longevidad en mamíferos". Aging Cell . 10 (2): 198–207. doi : 10.1111/j.1474-9726.2010.00657.x . PMID 21108730.
- ^ Shabalina IG, Vyssokikh MY, Gibanova N, Csikasz RI, Edgar D, Hallden-Waldemarson A, et al. (febrero de 2017). "Mejora de la salud y la esperanza de vida en ratones mutantes de mtADN tratados con el antioxidante dirigido a las mitocondrias SkQ1". Envejecimiento . 9 (2): 315–339. doi :10.18632/aging.101174. PMC 5361666 . PMID 28209927.
- ^ Ferrari CK (2004). "Alimentos funcionales, hierbas y nutracéuticos: hacia los mecanismos bioquímicos del envejecimiento saludable". Biogerontología . 5 (5): 275–289. doi :10.1007/s10522-004-2566-z. PMID 15547316. S2CID 11568208.
- ^ Taylor RW (febrero de 2005). "Terapia génica para el tratamiento de trastornos del ADN mitocondrial". Opinión de expertos sobre terapia biológica . 5 (2): 183–194. doi :10.1517/14712598.5.2.183. PMID 15757380. S2CID 35276183.
- ^ Bjelakovic G, Nikolova D, Gluud C (septiembre de 2013). "Suplementos antioxidantes para prevenir la mortalidad". JAMA . 310 (11): 1178–1179. doi :10.1001/jama.2013.277028. PMID 24045742.
- ^ Gureev AP, Andrianova NV, Pevzner IB, Zorova LD, Chernyshova EV, Sadovnikova IS, et al. (septiembre de 2022). "La restricción dietética modula el daño del ADN mitocondrial y el perfil de oxilipinas en ratas envejecidas". The FEBS Journal . 289 (18): 5697–5713. doi :10.1111/febs.16451. PMID 35373508. S2CID 247938550. Archivado desde el original el 4 de mayo de 2022. Consultado el 11 de mayo de 2022 .
- ^ Wang J, Xiong S, Xie C, Markesbery WR, Lovell MA (mayo de 2005). "Aumento del daño oxidativo en el ADN nuclear y mitocondrial en la enfermedad de Alzheimer". Journal of Neurochemistry . 93 (4): 953–962. doi : 10.1111/j.1471-4159.2005.03053.x . PMID 15857398.
- ^ Bonda DJ, Wang X, Lee HG, Smith MA, Perry G, Zhu X (abril de 2014). "Falla neuronal en la enfermedad de Alzheimer: una mirada a través del espejo del estrés oxidativo". Boletín de neurociencia . 30 (2): 243–252. doi :10.1007/s12264-013-1424-x. PMC 4097013 . PMID 24733654.
- ^ Canugovi C, Shamanna RA, Croteau DL, Bohr VA (junio de 2014). "Niveles de reparación del ADN por escisión de bases en lisados mitocondriales de la enfermedad de Alzheimer". Neurobiología del envejecimiento . 35 (6): 1293–1300. doi :10.1016/j.neurobiolaging.2014.01.004. PMC 5576885 . PMID 24485507.
- ^ Liu Z, Zhou T, Ziegler AC, Dimitrion P, Zuo L (2017). "Estrés oxidativo en enfermedades neurodegenerativas: de mecanismos moleculares a aplicaciones clínicas". Medicina oxidativa y longevidad celular . 2017 : 2525967. doi : 10.1155/2017/2525967 . PMC 5529664. PMID 28785371 .
- ^ Ayala-Peña S (septiembre de 2013). "El papel del daño oxidativo del ADN en la disfunción mitocondrial y la patogénesis de la enfermedad de Huntington". Free Radical Biology & Medicine . 62 : 102–110. doi :10.1016/j.freeradbiomed.2013.04.017. PMC 3722255 . PMID 23602907.
- ^ Kikuchi H, Furuta A, Nishioka K, Suzuki SO, Nakabeppu Y, Iwaki T (abril de 2002). "Deterioro de las enzimas reparadoras del ADN mitocondrial contra la acumulación de 8-oxoguanina en las neuronas motoras espinales de la esclerosis lateral amiotrófica". Acta Neuropatológica . 103 (4): 408–414. doi :10.1007/s00401-001-0480-x. PMID 11904761. S2CID 2102463.
- ^ Lehmann G, Budovsky A, Muradian KK, Fraifeld VE (2006). "Anatomía del genoma mitocondrial y esperanza de vida específica de la especie". Rejuvenation Research . 9 (2): 223–226. doi :10.1089/rej.2006.9.223. PMID 16706648.
- ^ ab Lehmann G, Segal E, Muradian KK, Fraifeld VE (abril de 2008). "¿Se complementan el ADN mitocondrial y la tasa metabólica en la determinación de la longevidad máxima de los mamíferos?". Rejuvenation Research . 11 (2): 409–417. doi :10.1089/rej.2008.0676. PMID 18442324.
- ^ Lehmann G, Muradian KK, Fraifeld VE (2013). "¿La longitud de los telómeros y los determinantes independientes de la temperatura corporal de la longevidad de los mamíferos?". Frontiers in Genetics . 4 (111): 111. doi : 10.3389/fgene.2013.00111 . PMC 3680702 . PMID 23781235.
- ^ Toren D, Barzilay T, Tacutu R, Lehmann G, Muradian KK, Fraifeld VE (enero de 2016). "MitoAge: una base de datos para el análisis comparativo del ADN mitocondrial, con especial atención a la longevidad animal". Nucleic Acids Research . 44 (D1): D1262–D1265. doi :10.1093/nar/gkv1187. PMC 4702847 . PMID 26590258.
- ^ Mikhailova AG, Mikhailova AA, Ushakova K, Tretiakov EO, Iliushchenko D, Shamansky V, et al. (octubre de 2022). "Una firma mutacional específica de la mitocondria del envejecimiento: aumento de la tasa de sustituciones A > G en la cadena pesada". Nucleic Acids Research . 50 (18): 10264–10277. doi :10.1093/nar/gkac779. PMC 9561281 . PMID 36130228.
- ^ Damas J, Carneiro J, Gonçalves J, Stewart JB, Samuels DC, Amorim A, Pereira F (septiembre de 2012). "Las eliminaciones del ADN mitocondrial están asociadas con conformaciones de ADN no B". Investigación de ácidos nucleicos . 40 (16): 7606–7621. doi :10.1093/nar/gks500. PMC 3439893 . PMID 22661583.
- ^ Oliveira PH, da Silva CL, Cabral JM (2013). "Una evaluación de la inestabilidad del ADN mitocondrial humano: nuevos conocimientos sobre el papel de las estructuras de ADN no canónicas y los motivos de secuencia". PLOS ONE . 8 (3): e59907. Bibcode :2013PLoSO...859907O. doi : 10.1371/journal.pone.0059907 . PMC 3612095 . PMID 23555828.
- ^ Brown WM, George M, Wilson AC (abril de 1979). "Evolución rápida del ADN mitocondrial animal". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 76 (4): 1967–1971. Bibcode :1979PNAS...76.1967B. doi : 10.1073/pnas.76.4.1967 . JSTOR 69636. PMC 383514 . PMID 109836.
- ^ Butler JM (2005). Tipificación forense de ADN: biología, tecnología y genética de los marcadores STR (2.ª ed.). Ámsterdam: Elsevier Academic Press. pp. 241–242, 246, 258–259. ISBN 0-12-147952-8.
- ^ Syndercombe Court D (septiembre de 2021). "ADN mitocondrial en uso forense". Temas emergentes en ciencias de la vida . 5 (3): 415–426. doi :10.1042/ETLS20210204. PMC 8457767 . PMID 34374411.
- ^ Davis CL (1998). «ADN mitocondrial: Estado de Tennessee contra Paul Ware» (PDF) . Perfiles en ADN . 1 (3): 6–7. Archivado (PDF) desde el original el 21 de agosto de 2016. Consultado el 21 de octubre de 2013 .
- ^ [1] Archivado el 21 de agosto de 2016 en Wayback Machine . Nombre del caso judicial que figura en la apelación. [ cita completa necesaria ] Consultado el 17 de abril de 2015.
- ^ [2] Archivado el 17 de mayo de 2015 en Wayback Machine [ ¿fuente autopublicada? ] Abogado defensor. Consultado el 17 de abril de 2015.
- ^ Garlicki D (11 de marzo de 1998). "Las pruebas de ADN consiguieron una condena de por vida en prisión". The Morning Call .
- ^ Pellegrino P, Thomas P (2000), "Archivos forenses. Una mujer despreciada Episodio 55 Episodio 55", Courtroom Television Network , Medstar Television, OCLC 456260156
- ^ "Juez permite ADN en caso de Samantha Runnion". Associated Press. 18 de febrero de 2005. Consultado el 4 de abril de 2007 .
- ^ "Se admite ADN canino en caso de asesinato en California". Pit Bulletin Legal News . 5 de diciembre de 2013. Archivado desde el original el 2 de febrero de 2014. Consultado el 21 de enero de 2014 .
- ^ Kennedy M (4 de febrero de 2013). «Ricardo III: el ADN confirma que los huesos retorcidos pertenecen al rey». The Guardian . Archivado desde el original el 8 de enero de 2019. Consultado el 7 de diciembre de 2014 .
- ^ Xu X, Arnason U (mayo de 1996). "Una secuencia completa del genoma mitocondrial del gorila occidental de las tierras bajas". Biología molecular y evolución . 13 (5): 691–698. doi : 10.1093/oxfordjournals.molbev.a025630 . PMID 8676744. Archivado desde el original el 4 de agosto de 2020. Consultado el 3 de febrero de 2020 .
- ^ Wei W, Schon KR, Elgar G, Orioli A, Tanguy M, Giess A, et al. (noviembre de 2022). "Secuencias de ADN mitocondrial con inclusión nuclear en 66.083 genomas humanos". Naturaleza . 611 (7934): 105–114. Código Bib :2022Natur.611..105W. doi :10.1038/s41586-022-05288-7. PMC 9630118 . PMID 36198798.
- ^ Wei W, Schon KR, Elgar G, Orioli A, Tanguy M, Giess A, et al. (noviembre de 2022). "Secuencias de ADN mitocondrial con inclusión nuclear en 66.083 genomas humanos". Naturaleza . 611 (7934): 105–114. Código Bib :2022Natur.611..105W. doi : 10.1038/s41586-022-05288-7 . PMC 9630118 . PMID 36198798.
- ^ Nass MM, Nass S (diciembre de 1963). "Fibras intramitocondriales con características de ADN". Revista de biología celular . 19 (3): 593–611. doi :10.1083/jcb.19.3.593. PMC 2106331 . PMID 14086138.
- ^ Schatz G, Haslbrunner E, Tuppy H (marzo de 1964). "Ácido desoxirribonucleico asociado con mitocondrias de levadura". Comunicaciones de investigación bioquímica y biofísica . 15 (2): 127–132. doi :10.1016/0006-291X(64)90311-0. PMID 26410904.
- ^ Ehler E, Novotný J, Juras A, Chylenski M, Moravcík O, Paces J (enero de 2019). "AmtDB: una base de datos de genomas mitocondriales humanos antiguos". Investigación de ácidos nucleicos . 47 (D1): D29-D32. doi : 10.1093/nar/gky843. PMC 6324066 . PMID 30247677.
- ^ Gu Z, Li J, Gao S, Gong M, Wang J, Xu H, et al. (junio de 2011). "InterMitoBase: una base de datos anotada y una plataforma de análisis de interacciones proteína-proteína para mitocondrias humanas". BMC Genomics . 12 : 335. doi : 10.1186/1471-2164-12-335 . PMC 3142533 . PMID 21718467.
- ^ Damas J, Carneiro J, Amorim A, Pereira F (enero de 2014). "MitoBreak: la base de datos de puntos de interrupción del ADN mitocondrial". Investigación de ácidos nucleicos . 42 (Problema de la base de datos): D1261 – D1268. doi : 10.1093/nar/gkt982. PMC 3965124 . PMID 24170808.
- ^ Iwasaki W, Fukunaga T, Isagozawa R, Yamada K, Maeda Y, Satoh TP, et al. (noviembre de 2013). "MitoFish y MitoAnnotator: una base de datos del genoma mitocondrial de peces con un proceso de anotación preciso y automático". Biología molecular y evolución . 30 (11): 2531–2540. doi :10.1093/molbev/mst141. PMC 3808866 . PMID 23955518.
- ^ Cawthorn DM, Steinman HA, Witthuhn RC (noviembre de 2011). "Establecimiento de una base de datos de secuencias de ADN mitocondrial para la identificación de especies de peces disponibles comercialmente en Sudáfrica". Recursos de ecología molecular . 11 (6): 979–991. doi :10.1111/j.1755-0998.2011.03039.x. PMID 21689383. S2CID 205971257.
- ^ Lee YS, Oh J, Kim YU, Kim N, Yang S, Hwang UW (enero de 2008). "Mitome: base de datos dinámica e interactiva para la genómica mitocondrial comparativa en animales metazoarios". Nucleic Acids Research . 36 (número de la base de datos): D938–D942. doi :10.1093/nar/gkm763. PMC 2238945 . PMID 17940090.
- ^ Catalano D, Licciulli F, Turi A, Grillo G, Saccone C, D'Elia D (enero de 2006). "MitoRes: un recurso de genes mitocondriales codificados nuclearmente y sus productos en Metazoa". Bioinformática BMC . 7 : 36. doi : 10.1186/1471-2105-7-36 . PMC 1395343 . PMID 16433928.
- ^ Kumar M, Kapil A, Shanker A (noviembre de 2014). "MitoSatPlant: base de datos de microsatélites mitocondriales de viridiplantae". Mitochondrion . 19 (Pt B): 334–337. doi :10.1016/j.mito.2014.02.002. PMID 24561221.
- ^ D'Onorio de Meo P, D'Antonio M, Griggio F, Lupi R, Borsani M, Pavesi G, et al. (enero de 2012). "MitoZoa 2.0: un recurso de base de datos y herramientas de búsqueda para análisis comparativos y evolutivos de genomas mitocondriales en metazoos". Nucleic Acids Research . 40 (número de base de datos): D1168–D1172. doi :10.1093/nar/gkr1144. PMC 3245153 . PMID 22123747.
- ^ "Las madres pueden influir en la altura, la esperanza de vida y el riesgo de enfermedades de sus hijos a través de las mitocondrias". phys.org . Archivado desde el original el 14 de junio de 2021 . Consultado el 14 de junio de 2021 .
- ^ Yonova-Doing E, Calabrese C, Gomez-Duran A, Schon K, Wei W, Karthikeyan S, et al. (julio de 2021). "Atlas de asociaciones genotipo-fenotipo del ADN mitocondrial en el Biobanco del Reino Unido". Nature Genetics . 53 (7): 982–993. doi :10.1038/s41588-021-00868-1. PMC 7611844 . PMID 34002094. S2CID 234768578.


