Cromosoma X
| Cromosoma X humano | |
|---|---|
 Cromosoma X humano (después de la formación de bandas G ) | |
 Cromosoma X en el cariograma masculino humano | |
| Características | |
| Longitud ( pb ) | 154.259.566 pb (CHM13) |
| Número de genes | 804 ( Código de distribución de contenidos ) |
| Tipo | Alosoma |
| Posición del centrómero | Submetacéntrico [1] (61,0 Mbp) [2] |
| Listas completas de genes | |
| CCDS | Lista de genes |
| HGNC | Lista de genes |
| Protección unificada | Lista de genes |
| Instituto Nacional de Biología | Lista de genes |
| Visores de mapas externos | |
| Conjunto | Cromosoma X |
| Entre | Cromosoma X |
| Instituto Nacional de Biología | Cromosoma X |
| Universidad del Sur de California | Cromosoma X |
| Secuencias completas de ADN | |
| Secuencia de referencia | NC_000023 ( FASTA ) |
| Banco Genético | CM000685 ( FASTA ) |
El cromosoma X es uno de los dos cromosomas sexuales de muchos organismos, incluidos los mamíferos, y se encuentra tanto en machos como en hembras. Forma parte del sistema de determinación sexual XY y del sistema de determinación sexual XO . Los primeros investigadores le dieron el nombre de cromosoma X por sus propiedades únicas, lo que dio lugar a que su homólogo, el cromosoma Y , se denominara así por la siguiente letra del alfabeto, tras su posterior descubrimiento. [3]
Descubrimiento
En 1890, Hermann Henking, en Leipzig, fue el primero en advertir que el cromosoma X era especial . Henking estaba estudiando los testículos de Pyrrhocoris y se dio cuenta de que un cromosoma no participaba en la meiosis . Los cromosomas se llaman así por su capacidad de absorber coloración ( croma en griego significa color ). Aunque el cromosoma X podía teñirse tan bien como los demás, Henking no estaba seguro de si se trataba de una clase diferente del objeto y, en consecuencia, lo denominó elemento X , [4] que más tarde se convirtió en cromosoma X después de que se estableciera que era, de hecho, un cromosoma. [5]
La idea de que el cromosoma X recibió su nombre por su similitud con la letra "X" es errónea. Todos los cromosomas aparecen normalmente como una masa amorfa bajo el microscopio y adquieren una forma bien definida solo durante la mitosis. Esta forma tiene vagamente la forma de una X para todos los cromosomas. Es pura coincidencia que el cromosoma Y , durante la mitosis , tenga dos ramas muy cortas que pueden verse fusionadas bajo el microscopio y aparecer como el descendiente de una Y. [6]
En 1901, Clarence Erwin McClung fue el primero en sugerir que el cromosoma X estaba involucrado en la determinación del sexo. Después de comparar su trabajo sobre las langostas con el de Henking y otros, McClung notó que solo la mitad de los espermatozoides recibían un cromosoma X. Llamó a este cromosoma un cromosoma accesorio e insistió (correctamente) en que era un cromosoma apropiado y teorizó (incorrectamente) que era el cromosoma determinante masculino. [4]
Patrón de herencia
This section relies largely or entirely upon a single source. (August 2021) |

Luke Hutchison notó que un número de posibles ancestros en la línea de herencia del cromosoma X en una generación ancestral dada sigue la secuencia de Fibonacci . [7] Un individuo masculino tiene un cromosoma X, que recibió de su madre, y un cromosoma Y , que recibió de su padre. El hombre cuenta como el "origen" de su propio cromosoma X ( ), y en la generación de sus padres, su cromosoma X provenía de un solo progenitor ( ). La madre del hombre recibió un cromosoma X de su madre (la abuela materna del hijo) y uno de su padre (el abuelo materno del hijo), por lo que dos abuelos contribuyeron al cromosoma X del descendiente masculino ( ). El abuelo materno recibió su cromosoma X de su madre, y la abuela materna recibió cromosomas X de ambos padres, por lo que tres bisabuelos contribuyeron al cromosoma X del descendiente masculino ( ). Cinco tatarabuelos contribuyeron al cromosoma X del descendiente masculino ( ), etc. (Tenga en cuenta que esto supone que todos los antepasados de un descendiente dado son independientes, pero si se rastrea alguna genealogía lo suficientemente atrás en el tiempo, los antepasados comienzan a aparecer en múltiples líneas de la genealogía, hasta que, finalmente, un fundador de la población aparece en todas las líneas de la genealogía).
Humanos
Función

El cromosoma X en los humanos abarca más de 153 millones de pares de bases (el material de construcción del ADN ). Representa alrededor de 800 genes codificadores de proteínas en comparación con el cromosoma Y que contiene alrededor de 70 genes, de un total de 20.000 a 25.000 genes en el genoma humano. Cada persona suele tener un par de cromosomas sexuales en cada célula. Las mujeres suelen tener dos cromosomas X, mientras que los hombres suelen tener un cromosoma X y uno Y. Tanto los hombres como las mujeres conservan uno de los cromosomas X de su madre, y las mujeres conservan su segundo cromosoma X de su padre. Dado que el padre conserva su cromosoma X de su madre, una mujer humana tiene un cromosoma X de su abuela paterna (por el lado del padre) y un cromosoma X de su madre. Este patrón de herencia sigue los números de Fibonacci a una profundidad ancestral dada. [ cita requerida ]
Los trastornos genéticos que se deben a mutaciones en los genes del cromosoma X se describen como ligados al cromosoma X. Si el cromosoma X tiene un gen de enfermedad genética, siempre causa la enfermedad en pacientes varones, ya que los hombres solo tienen un cromosoma X y, por lo tanto, solo una copia de cada gen. Las mujeres, en cambio, necesitan ambos cromosomas X para tener la enfermedad y, como resultado, potencialmente solo podrían ser portadoras de la enfermedad genética, ya que su segundo cromosoma X anula al primero. Por ejemplo, la hemofilia A y B y el daltonismo congénito rojo-verde se transmiten en familias de esta manera.
El cromosoma X contiene cientos de genes, pero pocos de ellos, si es que hay alguno, tienen algo que ver directamente con la determinación del sexo. En las primeras fases del desarrollo embrionario de las hembras, uno de los dos cromosomas X se inactiva de forma permanente en casi todas las células somáticas (células distintas de los óvulos y los espermatozoides ). Este fenómeno se denomina inactivación del cromosoma X o lionización y crea un corpúsculo de Barr . Si la inactivación del cromosoma X en la célula somática significara una desfuncionalización completa de uno de los cromosomas X, garantizaría que las hembras, al igual que los machos, tuvieran solo una copia funcional del cromosoma X en cada célula somática. Anteriormente se suponía que esto era así. Sin embargo, investigaciones recientes sugieren que el corpúsculo de Barr puede ser biológicamente más activo de lo que se suponía anteriormente. [8]
La inactivación parcial del cromosoma X se debe a la heterocromatina represiva que compacta el ADN e impide la expresión de la mayoría de los genes. La compactación de la heterocromatina está regulada por el complejo represivo Polycomb 2 ( PRC2 ). [9]
Genes
Número de genes
A continuación se presentan algunas de las estimaciones del recuento de genes del cromosoma X humano. Debido a que los investigadores utilizan diferentes enfoques para la anotación del genoma, sus predicciones del número de genes en cada cromosoma varían (para obtener detalles técnicos, consulte predicción de genes ). Entre los diversos proyectos, el proyecto de secuencia de codificación de consenso colaborativo ( CCDS ) adopta una estrategia extremadamente conservadora. Por lo tanto, la predicción del número de genes del CCDS representa un límite inferior en el número total de genes codificadores de proteínas humanas. [10]
| Estimado por | Genes codificadores de proteínas | Genes de ARN no codificantes | Pseudogenes | Fuente | Fecha de lanzamiento |
|---|---|---|---|---|---|
| CCDS | 804 | — | — | [11] | 08-09-2016 |
| HGNC | 825 | 260 | 606 | [12] | 12 de mayo de 2017 |
| Conjunto | 841 | 639 | 871 | [13] | 29-03-2017 |
| Protección unificada | 839 | — | — | [14] | 28-02-2018 |
| Instituto Nacional de Biología | 874 | 494 | 879 | [15] [16] [17] | 19 de mayo de 2017 |
Lista de genes
La siguiente es una lista parcial de genes del cromosoma X humano. Para ver la lista completa, consulte el enlace en el cuadro de información de la derecha.
- AD16 : proteína que codifica la enfermedad de Alzheimer 16
- AIC : proteína codificante AIC
- APOO : proteína codificante Apolipoproteína O
- ARMCX6 : proteína codificante que contiene la repetición Armadillo 6 ligada al cromosoma X
- BEX1 : proteína codificante Proteína 1 ligada al cromosoma X expresada en el cerebro
- BEX2 : proteína codificante Proteína 2 ligada al cromosoma X expresada en el cerebro
- BEX4 : proteína codificante expresada en el cerebro, ligada al cromosoma X 4
- CCDC120 : proteína codificante Dominio de bobina enrollada que contiene la proteína 120
- CCDC22 : proteína codificante de dominio en espiral que contiene 22
- CD99L2 : proteína similar al antígeno CD99 2
- CDR1-AS : ARN antisentido que codifica la proteína CDR1
- CFAP47 : proteína codificante de la proteína asociada a cilios y flagelos 47
- CHRDL1 : proteína codificante de tipo Cordina 1
- Proteína codificante CMTX2 Neuropatía de Charcot-Marie-Tooth, ligada al cromosoma X 2 (recesiva)
- Proteína codificante CMTX3 de la neuropatía de Charcot-Marie-Tooth, ligada al cromosoma X 3 (dominante)
- CT45A5 : proteína codificante de la familia 45 de antígenos de cáncer/testículo, miembro A5
- CT55 : proteína codificante del antígeno 55 del cáncer/testículo
- CXorf36 : proteína codificante de la proteína hipotética LOC79742
- CXorf57 : proteína codificante del marco de lectura abierto del cromosoma X 57
- CXorf40A : Marco de lectura abierto 40 del cromosoma X
- CXorf49 : marco de lectura abierto 49 del cromosoma X. Proteína codificante
- CXorf66 : proteína codificante del marco de lectura abierto del cromosoma X 66
- CXorf67 : proteína codificante Proteína no caracterizada CXorf67
- DACH2 : proteína codificante del homólogo 2 del Dachshund
- EFHC2 : proteína codificante del dominio EF-hand (C-terminal) que contiene 2
- Proteína codificante ERCC6L, reparación por escisión ERCC similar a 6, punto de control de ensamblaje del huso helicasa
- FAAH2 : Amida hidrolasa de ácidos grasos 2
- F8A1 : proteína del intrón 22 del factor VIII
- FAM104B : proteína codificante de la familia con similitud de secuencia de 104 miembros B
- FAM120C : familia de proteínas codificantes con similitud de secuencia 120C
- FAM122B : Familia con similitud de secuencia 122 miembro B
- FAM122C : familia de proteínas codificantes con similitud de secuencia 122C
- FAM127A : proteína de la caja CAAX 1
- FAM155B : familia de proteínas codificantes con similitud de secuencia 155 miembros B
- FAM50A : Familia con similitud de secuencia de 50 miembros A
- FATE1 : proteína de transcripción expresada en testículos fetales y adultos
- FMR1-AS1 : codifica un ARN largo no codificante ARN antisentido FMR1 1
- FRMPD3 : proteína codificante del dominio FERM y PDZ que contiene 3
- FRMPD4 : proteína codificante del dominio FERM y PDZ que contiene 4
- FUNDC1 : proteína codificante del dominio FUN14 que contiene 1
- FUNDC2 : proteína 2 que contiene el dominio FUN14
- GAGE12F : proteína codificante del antígeno G 12F
- GAGE2A : proteína codificante del antígeno G 2A
- GATA1 : codifica el factor de transcripción GATA1
- Proteína codificante GNL3L, proteína G, nucleolar 3 similar
- GPRASP2 : proteína de clasificación asociada al receptor acoplado a proteína G 2
- GRIPAP1 : proteína codificante de la proteína 1 asociada a GRIP1
- GRDX : proteína codificante de la enfermedad de Graves, susceptibilidad a la misma, ligada al cromosoma X
- HDHD1A : enzima codificante de la proteína 1A que contiene el dominio hidrolasa similar a la deshalogenasa de haloácido
- HS6ST2 : proteína codificante de la heparán sulfato 6-O-sulfotransferasa 2
- ITM2A : proteína codificante Proteína integral de membrana 2A
- LAS1L : proteína codificante de proteína similar a LAS1
- LOC101059915 : codifica la proteína *LOC101059915
- MAGEA2 : proteína codificante del antígeno 2 asociado al melanoma
- MAGEA5 : proteína codificante de la familia A del antígeno del melanoma, 5
- MAGEA8 : proteína codificante de la familia A del antígeno del melanoma, 8
- MAGED4B : proteína codificante del antígeno D4 asociado al melanoma
- MAGT1 : proteína codificante Proteína transportadora de magnesio 1
- MAGED4 : proteína codificante del miembro D4 de la familia MAGE
- MAO-A : Monoaminooxidasa A
- MAO-B : Monoaminooxidasa B
- MAP3K15 : proteína codificante de la proteína quinasa activada por mitógeno quinasa 15
- MBNL3 : proteína codificante de la proteína 3 similar a Muscleblind
- MBTPS2 : enzima codificante del sitio 2 del factor de transcripción unido a la membrana proteasa
- MCT-1 : proteína codificante MCTS1, factor de reiniciación y liberación
- MIR106A : microARN codificante MicroARN 106
- MIR222 : microARN codificante MicroARN 222
- MIR223 : proteína codificante MicroRNA 223
- MIR361 : microARN codificante MicroARN 361
- MIR503 : microARN codificante MicroARN 503
- MIR6087 : microARN codificante MicroARN 6087
- MIR660 : microARN codificante MicroARN 660
- MIRLET7F2 : proteína codificante del microARN let-7f-2
- MORF4L2 : proteína codificante de la proteína 2 similar al factor de mortalidad 4
- MOSPD1 : proteína codificante del dominio de espermatozoides móviles que contiene 1
- MOSPD2 : proteína codificante del dominio de esperma móvil que contiene 2
- NAP1L3 : proteína codificante Proteína de ensamblaje de nucleosomas 1 como 3
- NBDY : proteína codificante Regulador negativo de la asociación del cuerpo P
- NKRF : proteína codificante del factor represor NF-kappa-B
- NRK : enzima codificante de la proteína quinasa relacionada con Nik
- OPN1LW : opsina sensible a ondas largas (cono rojo)
- OPN1MW : opsina sensible a ondas medias (cono verde)
- OTUD5 : proteína codificante OTU deubiquitinasa 5
- PASD1 : proteína codificante que contiene el dominio PAS de la proteína 1
- PAGE1 : proteína codificante miembro 1 de la familia PAGE
- PAGE2B : codifica la proteína 2B del miembro de la familia PAGE
- PBDC1 : codifica una proteína de función no establecida
- PCYT1B : enzima codificante de la colina-fosfato citidiltransferasa B
- PIN4 : enzima codificante de la peptidil-prolil cis-trans isomerasa que interactúa con NIMA 4
- PLAC1 : proteína codificante Proteína 1 específica de la placenta
- PLP2 : proteína codificante Proteína proteolipídica 2
- PRR32 : proteína codificante PRR32
- RPA4 : proteína codificante de la subunidad A de la proteína de replicación de 30 kDa
- RPS6KA6 : proteína codificante de la proteína quinasa ribosomal S6, 90 kDa, polipéptido 6
- RRAGB : proteína codificante de la proteína B de unión a GTP relacionada con Ras
- RTL3 : proteína codificante del retrotransposón Gag like 3
- SFRS17A : factor de empalme de proteínas codificante, 17A rico en arginina/serina
- SLC38A5 : proteína codificante de la familia de transportadores de solutos 38 miembro 5
- SLITRK2 : proteína codificante de SLIT y proteína similar a NTRK 2
- SMARCA1 : proteína codificante del probable activador global de la transcripción SNF2L1
- SMS : enzima codificante de la espermina sintetasa
- SPANXN1 : proteína codificante del miembro N1 de la familia SPANX
- SPANXN5 : proteína codificante del miembro N5 de la familia SPANX
- SPG16 : proteína codificante de la paraplejía espástica 16 (complicada, recesiva ligada al cromosoma X)
- SSR4 : proteína codificante de la subunidad delta de la proteína asociada al translocón
- TAF7L : proteína codificante del factor 7 asociado a la proteína de unión a la caja TATA
- TCEAL1 : proteína codificante Factor de elongación de transcripción A proteína-similar 1
- TCEAL4 : proteína codificante Factor de elongación de transcripción A proteína-similar 4
- TENT5D : proteína codificante de la nucleotidiltransferasa terminal 5D
- TEX11 : proteína codificante expresada en testículos 11
- THOC2 : proteína codificante de la subunidad 2 del complejo THO
- TMEM29 : proteína codificante Proteína FAM156A
- TMEM47 : proteína codificante Proteína transmembrana 47
- TMLHE : enzima codificante trimetillisina dioxigenasa, mitocondrial
- Proteína codificante TNMD Tenomodulina (también conocida como tendina, miodulina, Tnmd y TeM)
- Subunidad 2 del complejo de partículas proteicas que codifica TRAPPC2P1
- TREX2 : enzima codificante de la exonucleasa 2 de reparación primaria tres
- TRO : proteína codificante trofinina
- TSPYL2 : proteína codificante Proteína 2 similar a la codificada por el gen Y específica de los testículos
- TTC3P1 : proteína codificante del pseudogen 1 del dominio 3 de repetición tetratricopeptídica
- USP51 : enzima codificante de la hidrolasa carboxilo-terminal de ubiquitina 51
- VSIG1 : proteína codificante del conjunto V y dominio de inmunoglobulina que contiene 1
- YIPF6 : proteína codificante Proteína YIPF6
- ZC3H12B : proteína codificante ZC3H12B
- ZC4H2 : deficiencia de la proteína codificante ZC4H2
- ZCCHC18 : proteína codificante de tipo CCHC con dedo de zinc que contiene 18
- ZFP92 : proteína codificante de la proteína de dedo de zinc ZFP92
- ZMYM3 : proteína codificante de la proteína 3 de tipo MYM con dedo de zinc
- ZNF157 : proteína codificante Proteína de dedo de zinc 157
- Proteína codificante ZNF182 Proteína de dedo de zinc 182
- ZNF275 : proteína codificante Proteína de dedo de zinc 275
- ZNF674 : proteína codificante Proteína de dedo de zinc 674
Estructura
Ross et al. (2005) y Ohno (1967) teorizan que el cromosoma X se deriva al menos parcialmente del genoma autosómico (no relacionado con el sexo) de otros mamíferos, como lo demuestran los alineamientos de secuencias genómicas entre especies.
El cromosoma X es notablemente más grande y tiene una región de eucromatina más activa que su homólogo, el cromosoma Y. Una comparación más detallada de los cromosomas X e Y revela regiones de homología entre ambos. Sin embargo, la región correspondiente en el cromosoma Y parece mucho más corta y carece de regiones que se conservan en el cromosoma X en todas las especies de primates, lo que implica una degeneración genética del cromosoma Y en esa región. Debido a que los machos tienen solo un cromosoma X, es más probable que tengan una enfermedad relacionada con el cromosoma X.
Se estima que alrededor del 10% de los genes codificados por el cromosoma X están asociados a una familia de genes "CT", llamados así porque codifican marcadores que se encuentran tanto en las células tumorales (en pacientes con cáncer) como en los testículos humanos (en pacientes sanos). [18]
Papel en la enfermedad
Anormalidades numéricas
- El síndrome de Klinefelter es causado por la presencia de una o más copias adicionales del cromosoma X en las células de un hombre.
- Los varones con síndrome de Klinefelter suelen tener una copia extra del cromosoma X en cada célula, lo que da un total de dos cromosomas X y un cromosoma Y (47,XXY). Es menos común que los varones afectados tengan dos o tres cromosomas X adicionales (48,XXXY o 49,XXXXY) o copias extra de los cromosomas X e Y (48,XXYY) en cada célula. El material genético adicional puede provocar estatura alta, problemas de aprendizaje y de lectura y otros problemas médicos. Cada cromosoma X adicional reduce el cociente intelectual del niño en unos 15 puntos, [19] [20] lo que significa que el cociente intelectual promedio en el síndrome de Klinefelter está en general dentro del rango normal, aunque por debajo del promedio. Cuando hay cromosomas X y/o Y adicionales en 48,XXXY, 48,XXYY o 49,XXXXY, los retrasos del desarrollo y las dificultades cognitivas pueden ser más graves y puede presentarse una discapacidad intelectual leve.
- El síndrome de Klinefelter también puede ser consecuencia de la presencia de un cromosoma X adicional en solo algunas células del organismo. Estos casos se denominan mosaico 46,XY/47,XXY.
- Este síndrome es el resultado de una copia adicional del cromosoma X en cada una de las células de la mujer. Las mujeres con trisomía X tienen tres cromosomas X, lo que hace un total de 47 cromosomas por célula. El coeficiente intelectual promedio de las mujeres con este síndrome es de 90, mientras que el coeficiente intelectual promedio de los hermanos no afectados es de 100. [21] Su estatura, en promedio, es más alta que la de las mujeres normales. Son fértiles y sus hijos no heredan la afección. [22]
- Se han identificado mujeres con más de una copia extra del cromosoma X (48, tetrasomía X o 49, pentasomía X ), pero estas afecciones son raras.
- Esto se produce cuando cada una de las células de una mujer tiene un cromosoma X normal y el otro cromosoma sexual falta o está alterado. El material genético faltante afecta el desarrollo y causa las características de la afección, incluida la baja estatura y la infertilidad.
- Aproximadamente la mitad de las personas con síndrome de Turner tienen monosomía X (45,X), lo que significa que cada célula del cuerpo de una mujer tiene solo una copia del cromosoma X en lugar de las dos copias habituales. El síndrome de Turner también puede ocurrir si uno de los cromosomas sexuales falta parcialmente o está reorganizado en lugar de faltar por completo. Algunas mujeres con síndrome de Turner tienen un cambio cromosómico solo en algunas de sus células. Estos casos se denominan mosaicos del síndrome de Turner (45,X/46,XX).
Trastornos recesivos ligados al cromosoma X
El ligamiento sexual se descubrió por primera vez en insectos, por ejemplo, en 1910, cuando TH Morgan descubrió el patrón de herencia de la mutación de ojos blancos en Drosophila melanogaster . [23] Dichos descubrimientos ayudaron a explicar los trastornos ligados al cromosoma X en humanos, por ejemplo, la hemofilia A y B, la adrenoleucodistrofia y el daltonismo rojo-verde .
Otros trastornos
El síndrome del varón XX es un trastorno poco frecuente en el que la región SRY del cromosoma Y se ha recombinado para ubicarse en uno de los cromosomas X. Como resultado, la combinación XX después de la fecundación tiene el mismo efecto que una combinación XY, lo que da como resultado un varón. Sin embargo, los otros genes del cromosoma X también causan feminización.
La distrofia corneal endotelial ligada al cromosoma X es una enfermedad extremadamente rara de la córnea asociada con la región Xq25. La distrofia corneal epitelial de Lisch está asociada con Xp22.3.
La megalocórnea 1 está asociada con Xq21.3-q22 [ cita médica necesaria ]
Adrenoleucodistrofia , un trastorno poco frecuente y mortal que se transmite por la madre a través de la célula X. Afecta solo a los niños de entre 5 y 10 años y destruye la mielina , la célula protectora que rodea los nervios del cerebro. La mujer portadora apenas presenta síntomas porque las mujeres tienen una copia de la célula X. Este trastorno hace que un niño que antes estaba sano pierda todas las capacidades de caminar, hablar, ver, oír e incluso tragar. En los 2 años posteriores al diagnóstico, la mayoría de los niños con adrenoleucodistrofia mueren.
Banda citogenética

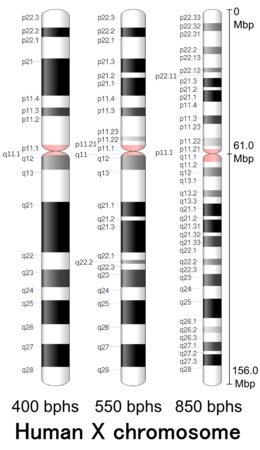
| Cristo. | Brazo [28] | Banda [29] | Inicio ISCN [30] | Parada ISCN [30] | Inicio del par base | Stop de par base | Mancha [31] | Densidad |
|---|---|---|---|---|---|---|---|---|
| incógnita | pag | 22.33 | 0 | 323 | 1 | 4.400.000 | negrito | |
| incógnita | pag | 22.32 | 323 | 504 | 4.400.001 | 6.100.000 | puntos de observación de GPS | 50 |
| incógnita | pag | 22.31 | 504 | 866 | 6.100.001 | 9.600.000 | negrito | |
| incógnita | pag | 22.2 | 866 | 1034 | 9.600.001 | 17.400.000 | puntos de observación de GPS | 50 |
| incógnita | pag | 22.13 | 1034 | 1345 | 17.400.001 | 19.200.000 | negrito | |
| incógnita | pag | 22.12 | 1345 | 1448 | 19.200.001 | 21.900.000 | puntos de observación de GPS | 50 |
| incógnita | pag | 22.11 | 1448 | 1577 | 21.900.001 | 24.900.000 | negrito | |
| incógnita | pag | 21.3 | 1577 | 1784 | 24.900.001 | 29.300.000 | puntos de observación de GPS | 100 |
| incógnita | pag | 21.2 | 1784 | 1862 | 29.300.001 | 31.500.000 | negrito | |
| incógnita | pag | 21.1 | 1862 | 2120 | 31.500.001 | 37.800.000 | puntos de observación de GPS | 100 |
| incógnita | pag | 11.4 | 2120 | 2430 | 37.800.001 | 42.500.000 | negrito | |
| incógnita | pag | 11.3 | 2430 | 2624 | 42.500.001 | 47.600.000 | puntos de observación de GPS | 75 |
| incógnita | pag | 11.23 | 2624 | 2948 | 47.600.001 | 50.100.000 | negrito | |
| incógnita | pag | 11.22 | 2948 | 3129 | 50.100.001 | 54.800.000 | puntos de observación de GPS | 25 |
| incógnita | pag | 11.21 | 3129 | 3206 | 54.800.001 | 58.100.000 | negrito | |
| incógnita | pag | 11.1 | 3206 | 3297 | 58.100.001 | 61.000.000 | Acenar | |
| incógnita | q | 11.1 | 3297 | 3491 | 61.000.001 | 63.800.000 | Acenar | |
| incógnita | q | 11.2 | 3491 | 3620 | 63.800.001 | 65.400.000 | negrito | |
| incógnita | q | 12 | 3620 | 3827 | 65.400.001 | 68.500.000 | puntos de observación de GPS | 50 |
| incógnita | q | 13.1 | 3827 | 4137 | 68.500.001 | 73.000.000 | negrito | |
| incógnita | q | 13.2 | 4137 | 4292 | 73.000.001 | 74.700.000 | puntos de observación de GPS | 50 |
| incógnita | q | 13.3 | 4292 | 4447 | 74.700.001 | 76.800.000 | negrito | |
| incógnita | q | 21.1 | 4447 | 4732 | 76.800.001 | 85.400.000 | puntos de observación de GPS | 100 |
| incógnita | q | 21.2 | 4732 | 4809 | 85.400.001 | 87.000.000 | negrito | |
| incógnita | q | 21.31 | 4809 | 5107 | 87.000.001 | 92.700.000 | puntos de observación de GPS | 100 |
| incógnita | q | 21.32 | 5107 | 5184 | 92.700.001 | 94.300.000 | negrito | |
| incógnita | q | 21.33 | 5184 | 5430 | 94.300.001 | 99.100.000 | puntos de observación de GPS | 75 |
| incógnita | q | 22.1 | 5430 | 5701 | 99.100.001 | 103.300.000 | negrito | |
| incógnita | q | 22.2 | 5701 | 5843 | 103.300.001 | 104.500.000 | puntos de observación de GPS | 50 |
| incógnita | q | 22.3 | 5843 | 6050 | 104.500.001 | 109.400.000 | negrito | |
| incógnita | q | 23 | 6050 | 6322 | 109.400.001 | 117.400.000 | puntos de observación de GPS | 75 |
| incógnita | q | 24 | 6322 | 6619 | 117.400.001 | 121.800.000 | negrito | |
| incógnita | q | 25 | 6619 | 7059 | 121.800.001 | 129.500.000 | puntos de observación de GPS | 100 |
| incógnita | q | 26.1 | 7059 | 7253 | 129.500.001 | 131.300.000 | negrito | |
| incógnita | q | 26.2 | 7253 | 7395 | 131.300.001 | 134.500.000 | puntos de observación de GPS | 25 |
| incógnita | q | 26.3 | 7395 | 7602 | 134.500.001 | 138.900.000 | negrito | |
| incógnita | q | 27.1 | 7602 | 7808 | 138.900.001 | 141.200.000 | puntos de observación de GPS | 75 |
| incógnita | q | 27.2 | 7808 | 7886 | 141.200.001 | 143.000.000 | negrito | |
| incógnita | q | 27.3 | 7886 | 8145 | 143.000.001 | 148.000.000 | puntos de observación de GPS | 100 |
| incógnita | q | 28 | 8145 | 8610 | 148.000.001 | 156.040.895 | negrito |
Investigación
En julio de 2020, los científicos informaron sobre el primer ensamblaje completo y sin espacios de un cromosoma X humano . [32] [33]
Véase también
- Lista de marcadores X-STR
- Vinculación sexual
- Inactivación del cromosoma X
- Región pseudoautosómica
- Cromosoma Y
Referencias
- Las versiones anteriores de este artículo contienen material de la Biblioteca Nacional de Medicina, parte de los Institutos Nacionales de Salud (EE. UU.), que, como publicación del gobierno de EE. UU., es de dominio público.
- ^ Tom Strachan; Andrew Read (2 de abril de 2010). Genética molecular humana. Garland Science. pág. 45. ISBN 978-1-136-84407-2.
- ^ Página de decoración del genoma de abc, NCBI. Datos de ideogramas de Homo sapience (850 bphs, ensamblaje GRCh38.p3). Última actualización: 2014-06-03. Consultado el 2017-04-26.
- ^ Angier, Natalie (1 de mayo de 2007). "Para el cromosoma X materno, el género es solo el comienzo". The New York Times . Consultado el 1 de mayo de 2007 .
- ^ de James Schwartz, En busca del gen: de Darwin al ADN , páginas 155-158, Harvard University Press, 2009 ISBN 0674034910
- ^ David Bainbridge, 'El X en el sexo: cómo el cromosoma X controla nuestras vidas' , páginas 3-5, Harvard University Press, 2003 ISBN 0674016211 .
- ^ Bainbridge, páginas 65-66
- ^ ab Hutchison, Luke (septiembre de 2004). "Growing the Family Tree: The Power of DNA in Reconstructing Family Relationships" (PDF) . Actas del Primer Simposio sobre Bioinformática y Biotecnología (BIOT-04) . Consultado el 3 de septiembre de 2016 .
- ^ Carrel L, Willard H (2005). "El perfil de inactivación del cromosoma X revela una amplia variabilidad en la expresión génica ligada al cromosoma X en mujeres". Nature . 434 (7031): 400–4. Bibcode :2005Natur.434..400C. doi :10.1038/nature03479. PMID 15772666. S2CID 4358447.
- ^ Veneti Z, Gkouskou KK, Eliopoulos AG (julio de 2017). "Complejo represor Polycomb 2 en la inestabilidad genómica y el cáncer". Int J Mol Sci . 18 (8): 1657. doi : 10.3390/ijms18081657 . PMC 5578047 . PMID 28758948.
- ^ Pertea M, Salzberg SL (2010). "Entre un pollo y una uva: estimación del número de genes humanos". Genome Biol . 11 (5): 206. doi : 10.1186/gb-2010-11-5-206 . PMC 2898077. PMID 20441615 .
- ^ "Resultados de la búsqueda - X[CHR] AND "Homo sapiens"[Organismo] AND ("tiene ccds"[Propiedades] AND vivo[prop]) - Gen". NCBI . Versión 20 de CCDS para Homo sapiens . 2016-09-08 . Consultado el 2017-05-28 .
- ^ "Estadísticas y descargas para el cromosoma X". Comité de Nomenclatura Genética de HUGO . 2017-05-12. Archivado desde el original el 2017-06-29 . Consultado el 2017-05-19 .
- ^ "Cromosoma X: Resumen cromosómico - Homo sapiens". Ensembl Release 88 . 2017-03-29 . Consultado el 2017-05-19 .
- ^ "Cromosoma X humano: entradas, nombres de genes y referencias cruzadas a MIM". UniProt . 2018-02-28 . Consultado el 2018-03-16 .
- ^ "Resultados de la búsqueda - X[CHR] AND "Homo sapiens"[Organismo] AND ("genetype protein coding"[Properties] AND alive[prop]) - Gene". NCBI . 2017-05-19 . Consultado el 2017-05-20 .
- ^ "Resultados de la búsqueda - X[CHR] AND "Homo sapiens"[Organismo] AND ( ("genetype miscrna"[Propiedades] OR "genetype ncrna"[Propiedades] OR "genetype rrna"[Propiedades] OR "genetype trna"[Propiedades] OR "genetype scrna"[Propiedades] OR "genetype snrna"[Propiedades] OR "genetype snorna"[Propiedades]) NOT "genetype protein coding"[Propiedades] AND alive[prop]) - Gene". NCBI . 2017-05-19 . Consultado el 2017-05-20 .
- ^ "Resultados de la búsqueda - X[CHR] AND "Homo sapiens"[Organismo] AND ("genetype pseudo"[Propiedades] AND alive[prop]) - Gene". NCBI . 2017-05-19 . Consultado el 2017-05-20 .
- ^ Ross M, et al. (2005). "La secuencia de ADN del cromosoma X humano". Nature . 434 (7031): 325–37. Bibcode :2005Natur.434..325R. doi :10.1038/nature03440. PMC 2665286 . PMID 15772651.
- ^ Harold Chen; Ian Krantz; Mary L Windle; Margaret M McGovern; Paul D Petry; Bruce Buehler (22 de febrero de 2013). "Fisiopatología del síndrome de Klinefelter". Medscape . Consultado el 18 de julio de 2014 .
- ^ Visootsak J, Graham JM (2006). "Síndrome de Klinefelter y otras aneuploidías cromosómicas sexuales". Orphanet J Rare Dis . 1 : 42. doi : 10.1186/1750-1172-1-42 . PMC 1634840 . PMID 17062147.
- ^ Bender B, Puck M, Salbenblatt J, Robinson A (1986). Smith S (ed.). Desarrollo cognitivo de niños con anomalías en los cromosomas sexuales . San Diego: College Hill Press. págs. 175–201.
- ^ "Síndrome del triple X". Genetics Home Reference . 2014-07-14 . Consultado el 2014-07-18 .
- ^ Morgan, TH (1910). "Herencia limitada por sexo en Drosophila". Science . 32 (812): 120–122. Bibcode :1910Sci....32..120M. doi :10.1126/science.32.812.120. PMID 17759620.
- ^ Página de decoración del genoma, NCBI. Datos de ideogramas para Homo sapience (400 bphs, ensamblaje GRCh38.p3). Última actualización: 4 de marzo de 2014. Consultado el 26 de abril de 2017.
- ^ Página de decoración del genoma, NCBI. Datos de ideogramas de Homo sapience (550 bphs, ensamblaje GRCh38.p3). Última actualización: 2015-08-11. Consultado el 2017-04-26.
- ^ Comité Permanente Internacional de Nomenclatura Citogenética Humana (2013). ISCN 2013: Un sistema internacional para la nomenclatura citogenética humana (2013). Karger Medical and Scientific Publishers. ISBN 978-3-318-02253-7.
- ^ Sethakulvichai, W.; Manitpornsut, S.; Wiboonrat, M.; Lilakiatsakun, W.; Assawamakin, A.; Tongsima, S. (2012). "Estimación de resoluciones a nivel de banda de imágenes de cromosomas humanos". Novena Conferencia Internacional sobre Ciencias de la Computación e Ingeniería de Software (JCSSE) de 2012. págs. 276–282. doi :10.1109/JCSSE.2012.6261965. ISBN 978-1-4673-1921-8.S2CID16666470 .
- ^ " p ": Brazo corto; " q ": Brazo largo.
- ^ Para la nomenclatura de las bandas citogenéticas, consulte el artículo locus .
- ^ ab Estos valores (inicio/fin del ISCN) se basan en la longitud de las bandas/ideogramas del libro ISCN, An International System for Human Cytogenetic Nomenclature (2013). Unidad arbitraria .
- ^ gpos : Región que se tiñe positivamente mediante bandas G , generalmente rica en AT y pobre en genes; gneg : Región que se tiñe negativamente mediante bandas G, generalmente rica en CG y rica en genes; acen Centrómero . var : Región variable; stalk : Tallo.
- ^ "Los científicos logran el primer ensamblaje completo del cromosoma X humano". phys.org . Consultado el 16 de agosto de 2020 .
- ^ Miga, Karen H .; Koren, Sergey; Rhie, Arang; Vollger, Mitchell R.; Gershman, Ariel; Bzikadze, Andrey; Brooks, Shelise; Howe, Edmundo; Porubsky, David; Logsdon, Glennis A.; Schneider, Valerie A.; Potapova, Tamara; Madera, Jonathan; Chow, William; Armstrong, Joel; Fredrickson, Jeanne; Pak, Evgenia; Tigyi, Kristof; Kremitzki, Milinn; Markovic, Christopher; Maduro, Valeria; Dutra, Amalia; Bouffard, Gerard G.; Chang, Alejandro M.; Hansen, Nancy F.; Wilfert, Amy B.; Thibaud-Nissen, Françoise; Schmitt, Antonio D.; Belton, Jon-Matthew; Selvaraj, Siddarth; Dennis, Megan Y.; Soto, Daniela C.; Sahasrabudhe, Ruta; Kaya, Gulhan; Quick, Josh; Loman, Nicholas J.; Holmes, Nadine; Loose, Matthew; Surti, Urvashi; Risques, Rosa ana; Lindsay, Tina A. Graves; Fulton, Robert; Hall, Ira; Paten, Benedict; Howe, Kerstin; Timp, Winston; Young, Alice; Mullikin, James C.; Pevzner, Pavel A.; Gerton, Jennifer L.; Sullivan, Beth A.; Eichler, Evan E.; Phillippy, Adam M . (14 de julio de 2020). "Ensamblaje telómero a telómero de un cromosoma X humano completo". Nature . 585 (7823): 79–84. Bibcode :2020Natur.585...79M. doi : 10.1038/s41586-020 -2547-7 . Revista de Biología Molecular y Genética .
Enlaces externos
- Institutos Nacionales de Salud. «Cromosoma X». Genetics Home Reference . Archivado desde el original el 2007-07-08 . Consultado el 2017-05-06 .
- «Cromosoma X». Archivo de información del Proyecto Genoma Humano 1990–2003 . Consultado el 6 de mayo de 2017 .







