Inactivación del cromosoma X

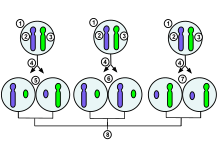
1. Célula embrionaria en etapa temprana de una mujer humana
2. Cromosoma X materno
3. Cromosoma X paterno
4. Mitosis y evento de inactivación aleatoria del cromosoma X
5. El cromosoma paterno se inactiva aleatoriamente en una célula hija, el cromosoma materno se inactiva en la otra
6. El cromosoma paterno se inactiva aleatoriamente en ambas células hijas
7. El cromosoma materno se inactiva aleatoriamente en ambas células hijas
8. Tres posibles resultados de combinación aleatoria


Izquierda: Núcleo teñido con ADN (DAPI). La flecha indica la ubicación del cuerpo de Barr (Xi). Derecha: Proteína histona asociada al ADN detectada

La inactivación del cromosoma X (también llamada Lyonización , en honor a la genetista inglesa Mary Lyon ) es un proceso por el cual una de las copias del cromosoma X se inactiva en los mamíferos hembras de la familia Therian . El cromosoma X inactivo se silencia al ser empaquetado en una estructura transcripcionalmente inactiva llamada heterocromatina . Como casi todos los mamíferos hembras tienen dos cromosomas X, la inactivación del cromosoma X evita que tengan el doble de productos génicos del cromosoma X que los machos, que solo poseen una única copia del cromosoma X (ver compensación de dosis ).
La elección del cromosoma X que se inactivará en una célula embrionaria particular es aleatoria en mamíferos placentarios como los humanos, pero una vez que un cromosoma X se inactiva, permanecerá inactivo durante toda la vida de la célula y sus descendientes en el organismo (su línea celular). El resultado es que la elección del cromosoma X inactivado en todas las células del organismo es una distribución aleatoria, con frecuencia con aproximadamente la mitad de las células que tienen el cromosoma X paterno inactivado y la otra mitad con un cromosoma X materno inactivado; pero comúnmente, la inactivación del cromosoma X se distribuye de manera desigual a través de las líneas celulares dentro de un organismo ( inactivación del cromosoma X sesgada ).
A diferencia de la inactivación aleatoria del cromosoma X en los mamíferos placentarios, la inactivación en los marsupiales se aplica exclusivamente al cromosoma X derivado del padre.
Mecanismo
Ciclo de activación del cromosoma X en roedores
Los párrafos siguientes se refieren únicamente a roedores y no reflejan la presencia de XI en la mayoría de los mamíferos. La inactivación del cromosoma X forma parte del ciclo de activación del cromosoma X a lo largo de la vida de la hembra. El óvulo y el cigoto fertilizado utilizan inicialmente las transcripciones maternas, y todo el genoma embrionario queda silenciado hasta la activación del genoma cigótico . A partir de entonces, todas las células del ratón experimentan una inactivación temprana e impronta del cromosoma X de origen paterno en los embriones en estadio de 4 a 8 células . [3] [4] [5] [6] Los tejidos extraembrionarios (que dan lugar a la placenta y otros tejidos que sostienen al embrión) conservan esta inactivación temprana impronta y, por tanto, solo el cromosoma X materno está activo en estos tejidos.
En el blastocisto temprano , esta inactivación inicial del cromosoma X se revierte en las células de la masa celular interna (que dan lugar al embrión), y en estas células ambos cromosomas X se vuelven a activar. Cada una de estas células inactiva entonces de forma independiente y aleatoria una copia del cromosoma X. [5] Este evento de inactivación es irreversible durante la vida del individuo, con la excepción de la línea germinal. En la línea germinal femenina antes de la entrada meiótica, la inactivación del cromosoma X se revierte, de modo que después de la meiosis todos los ovocitos haploides contienen un solo cromosoma X activo.
Descripción general
Xi marca el cromosoma X inactivo, Xa el cromosoma X activo. X P denota el cromosoma X paterno y X M to denota el cromosoma X materno. Cuando el óvulo (que lleva X M ) es fertilizado por un espermatozoide (que lleva un Y o un X P ) se forma un cigoto diploide. Desde el cigoto, pasando por la etapa adulta, hasta la siguiente generación de óvulos, el cromosoma X sufre los siguientes cambios:
- Xi P Xi M cigoto → experimentando una activación del genoma cigótico , lo que conduce a:
- Xa P Xa M → sufre inactivación del cromosoma X (paternal) impresa , lo que conduce a:
- Xi P Xa M → experimentando activación X en la etapa temprana de blastocisto , lo que conduce a:
- Xa P Xa M → sufre una inactivación aleatoria de X en el linaje embrionario (masa celular interna) en la etapa de blastocisto, lo que conduce a:
- Xi P Xa M O Xa P Xi M → experimentando una reactivación X en las células germinales primordiales antes de la meiosis , lo que conduce a:
- Células germinales diploides Xa M Xa P en detención meiótica. Como la meiosis I sólo se completa con la ovulación , las células germinales humanas existen en esta etapa desde las primeras semanas de desarrollo hasta la pubertad. La finalización de la meiosis conduce a:
- Células germinales haploides Xa M y Xa P (óvulos).
El ciclo de activación del cromosoma X se ha estudiado mejor en ratones, pero existen múltiples estudios en humanos. Como la mayoría de las pruebas provienen de ratones, el esquema anterior representa los eventos en ratones. La finalización de la meiosis se simplifica aquí para mayor claridad. Los pasos 1 a 4 se pueden estudiar en embriones fertilizados in vitro y en células madre en diferenciación; la reactivación del cromosoma X ocurre en el embrión en desarrollo y los pasos subsiguientes (6 a 7) dentro del cuerpo femenino, por lo que es mucho más difícil de estudiar.
Momento
El momento en que se produce cada proceso depende de la especie y, en muchos casos, el momento preciso es motivo de intenso debate. [Toda la parte de esta tabla que habla del momento en que se produce la inactivación del cromosoma X en humanos es muy cuestionable y debería eliminarse hasta que se confirme adecuadamente con datos empíricos].
| Proceso | Ratón | Humano | |
| 1 | Activación del genoma cigótico | Etapa de 2 a 4 células [7] | Etapa de 2 a 8 células [7] |
| 2 | Inactivación del cromosoma X impreso (paterno) | Etapa de 4 a 8 células [6] [8] | No está claro si ocurre en humanos [9] |
| 3 | Activación X | Etapa temprana de blastocisto | Etapa temprana de blastocisto |
| 4 | Inactivación aleatoria del cromosoma X en el linaje embrionario (masa celular interna) | Etapa tardía de blastocisto | Estadio tardío de blastocisto, después de la implantación [9] |
| 5 | Reactivación del cromosoma X en células germinales primordiales antes de la meiosis | Desde antes de la semana 4 de desarrollo hasta la semana 14 [10] [11] |
Herencia del estado de inactivación a lo largo de generaciones celulares
Los descendientes de cada célula que inactiva un cromosoma X en particular también inactivarán ese mismo cromosoma. Este fenómeno, que se puede observar en la coloración de los gatos carey cuando las hembras son heterocigotas para el gen del pigmento ligado al cromosoma X , no debe confundirse con el mosaicismo , que es un término que se refiere específicamente a las diferencias en el genotipo de varias poblaciones celulares en el mismo individuo; la inactivación del cromosoma X, que es un cambio epigenético que da como resultado un fenotipo diferente, no es un cambio a nivel genotípico . Para una célula o linaje individual, la inactivación está, por lo tanto , sesgada o " no aleatoria ", y esto puede dar lugar a síntomas leves en las hembras "portadoras" de trastornos genéticos ligados al cromosoma X. [12]
Selección de un cromosoma X activo
Las mujeres poseen dos cromosomas X y, en cualquier célula, un cromosoma estará activo (denominado Xa) y el otro inactivo (Xi). Sin embargo, los estudios de individuos con copias adicionales del cromosoma X muestran que en las células con más de dos cromosomas X, todavía hay un solo cromosoma Xa y todos los cromosomas X restantes están inactivados. Esto indica que el estado predeterminado del cromosoma X en las mujeres es la inactivación, pero siempre se selecciona un cromosoma X para que permanezca activo.
Se entiende que la inactivación del cromosoma X es un proceso aleatorio que ocurre aproximadamente en el momento de la gastrulación en el epiblasto (células que darán origen al embrión). Los cromosomas X maternos y paternos tienen la misma probabilidad de inactivación. Esto sugeriría que se esperaría que las mujeres sufrieran trastornos ligados al cromosoma X aproximadamente un 50% más a menudo que los hombres (porque las mujeres tienen dos cromosomas X, mientras que los hombres tienen solo uno); sin embargo, en realidad, la incidencia de estos trastornos en las mujeres es mucho menor que eso. Una explicación para esta disparidad es que entre el 12 y el 20% [13] de los genes en el cromosoma X inactivado permanecen expresados, lo que proporciona a las mujeres una protección adicional contra los genes defectuosos codificados por el cromosoma X. Algunos [¿ quiénes? ] sugieren que esta disparidad debe ser evidencia de una inactivación preferencial (no aleatoria). La inactivación preferencial del cromosoma X paterno ocurre tanto en marsupiales como en linajes celulares que forman las membranas que rodean al embrión, [14] mientras que en los mamíferos placentarios el cromosoma X derivado de la madre o del padre puede inactivarse en diferentes líneas celulares. [15]
El período de tiempo para la inactivación del cromosoma X explica esta disparidad. La inactivación ocurre en el epiblasto durante la gastrulación, que da lugar al embrión. [16] La inactivación ocurre a nivel celular, lo que resulta en una expresión en mosaico, en la que parches de células tienen un cromosoma X materno inactivo, mientras que otros parches tienen un cromosoma X paterno inactivo. Por ejemplo, una mujer heterocigota para hemofilia (una enfermedad ligada al cromosoma X) tendría aproximadamente la mitad de sus células hepáticas funcionando correctamente, lo que suele ser suficiente para garantizar una coagulación sanguínea normal. [17] [18] El azar podría dar lugar a significativamente más células disfuncionales; sin embargo, tales extremos estadísticos son poco probables. Las diferencias genéticas en el cromosoma también pueden hacer que un cromosoma X sea más propenso a sufrir inactivación. Además, si un cromosoma X tiene una mutación que impide su crecimiento o lo vuelve no viable, las células que inactivaron aleatoriamente ese X tendrán una ventaja selectiva sobre las células que inactivaron aleatoriamente el alelo normal. Por lo tanto, aunque la inactivación es inicialmente aleatoria, las células que inactivan un alelo normal (dejando activo el alelo mutado) eventualmente serán superadas y reemplazadas por células funcionalmente normales en las que casi todas tienen el mismo cromosoma X activado. [17]
Se ha planteado la hipótesis de que existe un «factor de bloqueo» codificado autosómicamente que se une al cromosoma X y evita su inactivación. [19] El modelo postula que existe un factor de bloqueo limitante, por lo que una vez que la molécula de factor de bloqueo disponible se une a un cromosoma X, el resto de los cromosomas X no están protegidos de la inactivación. Este modelo está respaldado por la existencia de un único cromosoma Xa en células con muchos cromosomas X y por la existencia de dos cromosomas X activos en líneas celulares con el doble del número normal de autosomas. [20]
Las secuencias en el centro de inactivación X ( XIC ), presente en el cromosoma X, controlan el silenciamiento del cromosoma X. Se predice que el factor de bloqueo hipotético se une a secuencias dentro del XIC.
Expresión de trastornos ligados al cromosoma X en mujeres heterocigotas
El efecto de la heterocigosidad del cromosoma X de la hembra es evidente en algunos rasgos localizados, como el patrón único del pelaje de un gato atigrado . Sin embargo, puede resultar más difícil comprender por completo la expresión de rasgos no localizados en estas hembras, como la expresión de enfermedades.
Dado que los hombres sólo tienen una copia del cromosoma X, todos los genes del cromosoma X expresados (o alelos , en el caso de múltiples formas variantes para un gen dado en la población) se encuentran en esa copia del cromosoma. Las mujeres, sin embargo, expresarán principalmente los genes o alelos ubicados en la copia del cromosoma X que permanece activa. Considerando la situación de un gen o múltiples genes que causan diferencias individuales en un fenotipo particular (es decir, causan variación observada en la población para ese fenotipo), en las mujeres homocigotas no importa particularmente qué copia del cromosoma está inactivada, ya que los alelos en ambas copias son los mismos. Sin embargo, en las mujeres que son heterocigotas en los genes causales, la inactivación de una copia del cromosoma sobre la otra puede tener un impacto directo en su valor fenotípico. Debido a este fenómeno, hay un aumento observado en la variación fenotípica en las mujeres que son heterocigotas en el gen o genes involucrados que en las mujeres que son homocigotas en ese gen o esos genes. [21] La variación fenotípica puede manifestarse de muchas formas diferentes. En muchos casos, las mujeres heterocigotas pueden ser asintomáticas o presentar solo síntomas menores de un trastorno determinado, como la adrenoleucodistrofia ligada al cromosoma X. [22]
La diferenciación del fenotipo en hembras heterocigotas se ve favorecida por la presencia de un sesgo de inactivación del cromosoma X. Normalmente, cada cromosoma X está silenciado en la mitad de las células, pero este proceso se ve sesgado cuando se produce una inactivación preferencial de un cromosoma. Se cree que el sesgo se produce por casualidad o por una característica física de un cromosoma que puede hacer que se silencie con mayor o menor frecuencia, como una mutación desfavorable. [23] [24]
En promedio, cada cromosoma X está inactivado en la mitad de las células, aunque entre el 5 y el 20 % de las mujeres presentan una desviación de la inactivación del cromosoma X. [23] En los casos en los que hay desviación, puede darse una amplia gama de expresión de síntomas, lo que da como resultado una expresión que varía de leve a grave según la proporción de desviación. Se observó un caso extremo de esto en el que dos gemelas monocigóticas tenían una variación extrema en la expresión de la enfermedad de Menkes (un trastorno ligado al cromosoma X), lo que dio como resultado la muerte de una gemela mientras que la otra permaneció asintomática. [25]
Se cree que la desviación de la inactivación del cromosoma X podría deberse a problemas en el mecanismo que provoca la inactivación o a problemas en el propio cromosoma. [23] [24] Sin embargo, el vínculo entre el fenotipo y la desviación aún se cuestiona y debe examinarse caso por caso. Un estudio que examinó mujeres sintomáticas y asintomáticas que eran heterocigotas para las distrofias musculares de Duchenne y Becker (DMD) no encontró un vínculo aparente entre la expresión de la transcripción y la desviación de la inactivación del cromosoma X. El estudio sugiere que ambos mecanismos se regulan de forma independiente y que hay otros factores desconocidos en juego. [26]
Componente cromosómico
El centro de inactivación del cromosoma X (o simplemente XIC) es necesario y suficiente para provocar la inactivación del cromosoma X. Las translocaciones cromosómicas que colocan el XIC en un autosoma provocan la inactivación del autosoma, y los cromosomas X que carecen del XIC no se inactivan. [27] [28]
El XIC contiene cuatro genes de ARN no traducidos , Xist , Tsix , Jpx y Ftx , que participan en la inactivación del cromosoma X. El XIC también contiene sitios de unión para proteínas reguladoras conocidas y desconocidas . [29]
ARN Xist y Tsix
El gen de transcripción específica X-inactiva ( Xist ) codifica un ARN no codificante de gran tamaño que es responsable de mediar el silenciamiento específico del cromosoma X del que se transcribe. [30] El cromosoma X inactivo está recubierto por el ARN Xist, [31] mientras que el Xa no lo está (véase la figura de la derecha). Los cromosomas X que carecen del gen Xist no pueden inactivarse. [32] La colocación y expresión artificial del gen Xist en otro cromosoma conduce al silenciamiento de ese cromosoma. [33] [27]
Antes de la inactivación, ambos cromosomas X expresan débilmente el ARN Xist del gen Xist. Durante el proceso de inactivación, el futuro Xa deja de expresar Xist, mientras que el futuro Xi aumenta drásticamente la producción de ARN Xist. En el futuro Xi, el ARN Xist recubre progresivamente el cromosoma, extendiéndose desde el XIC; [33] el ARN Xist no se localiza en el Xa. El silenciamiento de los genes a lo largo del Xi ocurre poco después del recubrimiento por el ARN Xist.
Al igual que Xist, el gen Tsix codifica un ARN grande que no se cree que codifique una proteína. El ARN Tsix se transcribe en sentido antisentido con respecto a Xist, lo que significa que el gen Tsix se superpone al gen Xist y se transcribe en la cadena de ADN opuesta a la del gen Xist. [28] Tsix es un regulador negativo de Xist; los cromosomas X que carecen de la expresión de Tsix (y, por lo tanto, tienen altos niveles de transcripción de Xist) se inactivan con mucha más frecuencia que los cromosomas normales.
Al igual que Xist, antes de la inactivación, ambos cromosomas X expresan débilmente el ARN Tsix del gen Tsix. Al iniciarse la inactivación de X, el futuro Xi deja de expresar el ARN Tsix (y aumenta la expresión de Xist), mientras que Xa continúa expresando Tsix durante varios días.
Rep A es un ARN largo no codificante que trabaja con otro ARN largo no codificante, Xist, para la inactivación de X. Rep A inhibe la función de Tsix, el antisentido de Xist, junto con la eliminación de la expresión de Xite. Promueve la metilación de la región Tsix atrayendo a PRC2 y, por lo tanto, inactivando uno de los cromosomas X. [29]
Silenciamiento
El cromosoma X inactivo no expresa la mayoría de sus genes, a diferencia del cromosoma X activo. Esto se debe al silenciamiento del Xi por la heterocromatina represiva , que compacta el ADN Xi e impide la expresión de la mayoría de los genes.
En comparación con Xa, Xi tiene altos niveles de metilación de ADN , bajos niveles de acetilación de histonas , bajos niveles de metilación de lisina-4 de histona H3 y altos niveles de metilación de lisina-9 de histona H3 y marca de metilación de lisina-27 de H3 que es colocada por el complejo PRC2 reclutado por Xist , todos los cuales están asociados con el silenciamiento de genes. [34] PRC2 regula la compactación de la cromatina y la remodelación de la cromatina en varios procesos, incluida la respuesta al daño del ADN . [35] Además, una variante de histona llamada macroH2A ( H2AFY ) se encuentra exclusivamente en los nucleosomas a lo largo de Xi. [36] [37]
Cuerpos de Barr
El ADN empaquetado en heterocromatina, como Xi, está más condensado que el ADN empaquetado en eucromatina , como Xa. La X inactiva forma un cuerpo discreto dentro del núcleo llamado cuerpo de Barr . [38] El cuerpo de Barr generalmente se encuentra en la periferia del núcleo , se replica tardíamente dentro del ciclo celular y, como contiene Xi, contiene modificaciones de heterocromatina y el ARN Xist.
Genes expresados en el cromosoma X inactivo
Una fracción de los genes a lo largo del cromosoma X escapan a la inactivación en el Xi. El gen Xist se expresa en niveles altos en el Xi y no se expresa en el Xa. [39] Muchos otros genes escapan a la inactivación; algunos se expresan por igual en el Xa y el Xi, y otros, aunque se expresan en ambos cromosomas, siguen expresándose predominantemente en el Xa. [40] [41] [42] Hasta una cuarta parte de los genes en el Xi humano son capaces de escapar. [40] Los estudios en el ratón sugieren que en cualquier tipo de célula dado, entre el 3% y el 15% de los genes escapan a la inactivación, y que la identidad del gen que escapa varía entre los tejidos. [41] [42]
Muchos de los genes que escapan a la inactivación están presentes en regiones del cromosoma X que, a diferencia de la mayoría del cromosoma X, contienen genes también presentes en el cromosoma Y. Estas regiones se denominan regiones pseudoautosómicas , ya que los individuos de ambos sexos recibirán dos copias de cada gen en estas regiones (como un autosoma), a diferencia de la mayoría de los genes a lo largo de los cromosomas sexuales. Dado que los individuos de ambos sexos recibirán dos copias de cada gen en una región pseudoautosómica , no se necesita compensación de dosis para las mujeres, por lo que se postula que estas regiones de ADN han desarrollado mecanismos para escapar de la inactivación del cromosoma X. Los genes de las regiones pseudoautosómicas del Xi no tienen las modificaciones típicas del Xi y tienen poco ARN Xist unido.
La existencia de genes a lo largo del cromosoma X inactivo que no están silenciados explica los defectos en humanos con números anormales del cromosoma X, como el síndrome de Turner (X0, causado por el gen SHOX [43] ) o el síndrome de Klinefelter (XXY). Teóricamente, la inactivación del cromosoma X debería eliminar las diferencias en la dosis génica entre los individuos afectados y los individuos con un complemento cromosómico normal. Sin embargo, en los individuos afectados, la inactivación del cromosoma X es incompleta y la dosis de estos genes no silenciados diferirá a medida que escapen a la inactivación del cromosoma X, de manera similar a una aneuploidía autosómica .
No se conocen los mecanismos precisos que controlan el escape de la inactivación del cromosoma X, pero se ha demostrado que las regiones silenciadas y de escape tienen marcas de cromatina distintas. [41] [44] Se ha sugerido que el escape de la inactivación del cromosoma X podría estar mediado por la expresión de ARN largo no codificante (lncRNA) dentro de los dominios cromosómicos de escape. [2]
Usos en biología experimental
Stanley Michael Gartler utilizó la inactivación del cromosoma X para demostrar el origen clonal de los cánceres. El examen de tejidos normales y tumores de mujeres heterocigotas para las isoenzimas del gen G6PD ligado al sexo demostró que las células tumorales de dichos individuos expresan sólo una forma de G6PD, mientras que los tejidos normales están compuestos por una mezcla casi igual de células que expresan los dos fenotipos diferentes. Este patrón sugiere que una sola célula, y no una población, se convierte en un cáncer. [45] Sin embargo, se ha demostrado que este patrón es erróneo para muchos tipos de cáncer, lo que sugiere que algunos cánceres pueden ser de origen policlonal. [46]
Además, la medición del estado de metilación (inactivación) del receptor de andrógenos humano polimórfico (HUMARA) ubicado en el cromosoma X se considera el método más preciso para evaluar la clonalidad en biopsias de cáncer femenino. [47] Se evaluó una gran variedad de tumores mediante este método, algunos, como el carcinoma de células renales, [48] se encontraron monoclonales, mientras que otros (por ejemplo, el mesotelioma [49] ) se informaron como policlonales.
Los investigadores también han investigado el uso de la inactivación del cromosoma X para silenciar la actividad de los cromosomas autosómicos. Por ejemplo, Jiang et al. insertaron una copia del gen Xist en una copia del cromosoma 21 en células madre derivadas de un individuo con trisomía 21 ( síndrome de Down ). [50] El gen Xist insertado induce la formación de cuerpos de Barr, desencadena modificaciones estables de la heterocromatina y silencia la mayoría de los genes en la copia adicional del cromosoma 21. En estas células madre modificadas, el silenciamiento del gen mediado por Xist parece revertir algunos de los defectos asociados con el síndrome de Down.
Historia
En 1959 Susumu Ohno demostró que los dos cromosomas X de los mamíferos eran diferentes: uno parecía similar a los autosomas ; el otro era condensado y heterocromático. [51] Este hallazgo sugirió, independientemente a dos grupos de investigadores, que uno de los cromosomas X sufrió inactivación.
En 1961, Mary Lyon propuso la inactivación aleatoria de un cromosoma X femenino para explicar el fenotipo moteado de ratones hembra heterocigotos para genes de color de pelaje . [52] La hipótesis de Lyon también explicó los hallazgos de que una copia del cromosoma X en células femeninas estaba altamente condensada, y que los ratones con solo una copia del cromosoma X se desarrollaban como hembras infértiles. Esto sugirió [53] a Ernest Beutler , que estudiaba hembras heterocigotas para la deficiencia de glucosa-6-fosfato deshidrogenasa (G6PD), que había dos poblaciones de glóbulos rojos de eritrocitos en tales heterocigotos: células deficientes y células normales, [54] dependiendo de si el cromosoma X inactivado (en el núcleo de la célula precursora del glóbulo rojo) contiene el alelo G6PD normal o defectuoso.
Véase también
- Sistema de determinación del sexo
- Compensación de dosis
- Cuerpo de barra
- Heterocromatina
- Epigenética
- Inactivación sesgada del cromosoma X
- Trastornos del desarrollo que se cree que están relacionados con la inactivación del cromosoma X:
Referencias
- ^ Gartler SM, Varadarajan KR, Luo P, Canfield TK, Traynor J, Francke U, Hansen RS (septiembre de 2004). "Modificaciones normales de histonas en el cromosoma X inactivo en células con síndrome de Rett e ICF: implicaciones para las proteínas de unión a metil-CpG". BMC Biology . 2 : 21. doi : 10.1186/1741-7007-2-21 . PMC 521681 . PMID 15377381.
- ^ ab Reinius B, Shi C, Hengshuo L, Sandhu KS, Radomska KJ, Rosen GD, Lu L, Kullander K, Williams RW, Jazin E (noviembre de 2010). "Expresión sesgada hacia las hembras de ARN largos no codificantes en dominios que escapan a la inactivación del cromosoma X en ratones". BMC Genomics . 11 : 614. doi : 10.1186/1471-2164-11-614 . PMC 3091755 . PMID 21047393.
- ^ Takagi N, Sasaki M (agosto de 1975). "Inactivación preferencial del cromosoma X de origen paterno en las membranas extraembrionarias del ratón". Nature . 256 (5519): 640–2. Bibcode :1975Natur.256..640T. doi :10.1038/256640a0. PMID 1152998. S2CID 4190616.
- ^ Cheng MK, Disteche CM (agosto de 2004). "El silencio de los padres: inactivación temprana del cromosoma X". BioEssays . 26 (8): 821–4. doi :10.1002/bies.20082. PMID 15273983.[ enlace muerto ]
- ^ ab Okamoto I, Otte AP, Allis CD, Reinberg D, Heard E (enero de 2004). "Dinámica epigenética de la inactivación del cromosoma X impreso durante el desarrollo temprano del ratón". Science . 303 (5658): 644–9. Bibcode :2004Sci...303..644O. doi :10.1126/science.1092727. PMID 14671313. S2CID 26326026.
- ^ ab Deng Q, Ramsköld D, Reinius B, Sandberg R (enero de 2014). "La secuenciación de ARN de una sola célula revela una expresión génica monoalélica aleatoria y dinámica en células de mamíferos". Science . 343 (6167): 193–6. Bibcode :2014Sci...343..193D. doi :10.1126/science.1245316. PMID 24408435. S2CID 206552108.
- ^ ab Xue Z, Huang K, Cai C, Cai L, Jiang CY, Feng Y, Liu Z, Zeng Q, Cheng L, Sun YE, Liu JY, Horvath S, Fan G (agosto de 2013). "Programas genéticos en embriones humanos y de ratón tempranos revelados por secuenciación de ARN unicelular". Nature . 500 (7464): 593–7. Bibcode :2013Natur.500..593X. doi :10.1038/nature12364. PMC 4950944 . PMID 23892778.
- ^ Borensztein M, Syx L, Ancelin K, Diabangouaya P, Picard C, Liu T, Liang JB, Vassilev I, Galupa R, Servant N, Barillot E, Surani A, Chen CJ, Heard E (marzo de 2017). "Inactivación del cromosoma X impreso dependiente de Xist y las consecuencias tempranas de su falla en el desarrollo". Nature Structural & Molecular Biology . 24 (3): 226–233. doi :10.1038/nsmb.3365. PMC 5337400 . PMID 28134930.
- ^ ab Deng X, Berletch JB, Nguyen DK, Disteche CM (junio de 2014). "Regulación del cromosoma X: diversos patrones en el desarrollo, los tejidos y la enfermedad". Nature Reviews. Genetics . 15 (6): 367–78. doi :10.1038/nrg3687. PMC 4117651 . PMID 24733023.
- ^ Vértesy Á, Arindrarto W, Roost MS, Reinius B, Torrens-Juaneda V, Bialecka M, et al. (mayo de 2018). "La transcriptómica unicelular específica del haplotipo parental revela una reprogramación epigenética incompleta en células germinales femeninas humanas". Nature Communications . 9 (1): 1873. Bibcode :2018NatCo...9.1873V. doi :10.1038/s41467-018-04215-7. PMC 5951918 . PMID 29760424.
- ^ Guo F, Yan L, Guo H, Li L, Hu B, Zhao Y, et al. (junio de 2015). "Los paisajes del transcriptoma y del metiloma del ADN de las células germinales primordiales humanas". Cell . 161 (6): 1437–52. doi : 10.1016/j.cell.2015.05.015 . PMID 26046443.
- ^ Puck JM, Willard HF (enero de 1998). "Inactivación del cromosoma X en mujeres con enfermedad ligada al cromosoma X". The New England Journal of Medicine . 338 (5): 325–8. doi :10.1056/NEJM199801293380611. PMID 9445416.
- ^ Balaton BP, Cotton AM, Brown CJ (30 de diciembre de 2015). "Derivación del estado de inactivación de consenso para genes ligados al cromosoma X a partir de estudios de todo el genoma". Biology of Sex Differences . 6 (35): 35. doi : 10.1186/s13293-015-0053-7 . PMC 4696107 . PMID 26719789.
- ^ Graves JA (1996). "Mamíferos que rompen las reglas: genética de marsupiales y monotremas". Revisión anual de genética . 30 : 233–60. doi :10.1146/annurev.genet.30.1.233. PMID 8982455.
- ^ Lyon MF (enero de 1972). "Inactivación del cromosoma X y patrones de desarrollo en mamíferos". Biological Reviews of the Cambridge Philosophical Society . 47 (1): 1–35. doi :10.1111/j.1469-185X.1972.tb00969.x. PMID 4554151. S2CID 39402646.
- ^ Migeon, B (2010). "Inactivación del cromosoma X en células humanas". The Biomedical & Life Sciences Collection . Henry Stewart Talks, Ltd: 1–54 . Consultado el 15 de diciembre de 2013 .
- ^ ab Gartler SM, Goldman MA (2001). "Inactivación del cromosoma X" (PDF) . Enciclopedia de ciencias de la vida . Nature Publishing Group: 1–2.
- ^ Connallon T, Clark AG (abril de 2013). "Selección diferencial de sexos y evolución de las estrategias de inactivación del cromosoma X". PLOS Genetics . 9 (4): e1003440. doi : 10.1371/journal.pgen.1003440 . PMC 3630082 . PMID 23637618.
- ^ Avner, Philip; Heard, Edith (enero de 2001). "Inactivación del cromosoma X: recuento, elección e iniciación". Nature Reviews Genetics . 2 (1): 59–67. doi :10.1038/35047580. ISSN 1471-0064. PMID 11253071. S2CID 5234164.
- ^ Barakat TS, Gribnau J (2010). "Inactivación del cromosoma X y células madre embrionarias". En Meshorer E, Plath K (eds.). La biología celular de las células madre . Landes Bioscience y Springer Science+Business Media.
- ^ Ma L, Hoffman G, Keinan A (marzo de 2015). "La inactivación del cromosoma X informa las pruebas basadas en la varianza para la asociación ligada al cromosoma X de un rasgo cuantitativo". BMC Genomics . 16 (1): 241. doi : 10.1186/s12864-015-1463-y . PMC 4381508 . PMID 25880738.
- ^ Habekost CT, Pereira FS, Vargas CR, Coelho DM, Torrez V, Oses JP, Portela LV, Schestatsky P, Felix VT, Matte U, Torman VL, Jardim LB (octubre de 2015). "Tasa de progresión de la mielopatía en heterocigotos con adrenoleucodistrofia ligada al cromosoma X". Metabolic Brain Disease . 30 (5): 1279–84. doi :10.1007/s11011-015-9672-2. PMID 25920484. S2CID 11375978.
- ^ abc Belmont JW (junio de 1996). "Control genético de la inactivación del cromosoma X y procesos que conducen a un sesgo en la inactivación del cromosoma X". American Journal of Human Genetics . 58 (6): 1101–8. PMC 1915050 . PMID 8651285.
- ^ ab Holle JR, Marsh RA, Holdcroft AM, Davies SM, Wang L, Zhang K, Jordan MB (julio de 2015). "Linfohistiocitosis hemofagocítica en una paciente femenina debido a una mutación XIAP heterocigótica y una inactivación sesgada del cromosoma X". Pediatric Blood & Cancer . 62 (7): 1288–90. doi :10.1002/pbc.25483. PMID 25801017. S2CID 5516967.
- ^ Burgemeister AL, Zirn B, Oeffner F, Kaler SG, Lemm G, Rossier E, Büttel HM (noviembre de 2015). "Enfermedad de Menkes con fenotipo discordante en gemelas monocigóticas". Revista Estadounidense de Genética Médica. Parte A. 167A (11): 2826–9. doi :10.1002/ajmg.a.37276. PMC 6475897 . PMID 26239182.
- ^ Brioschi S, Gualandi F, Scotton C, Armaroli A, Bovolenta M, Falzarano MS, Sabatelli P, Selvatici R, D'Amico A, Pane M, Ricci G, Siciliano G, Tedeschi S, Pini A, Vercelli L, De Grandis D, Mercuri E, Bertini E, Merlini L, Mongini T, Ferlini A (agosto de 2012). "Caracterización genética en mujeres portadoras sintomáticas de DMD: falta de relación entre inactivación de X, equilibrio de alelos transcripcionales de DMD y fenotipo". BMC Genética Médica . 13 : 73. doi : 10.1186/1471-2350-13-73 . PMC 3459813 . PMID 22894145.
- ^ ab Lee JT, Jaenisch R (marzo de 1997). "Efectos cis de largo alcance de los centros de inactivación ectópicos del cromosoma X en un autosoma de ratón". Nature . 386 (6622): 275–9. Bibcode :1997Natur.386..275L. doi :10.1038/386275a0. PMID 9069285. S2CID 10899129.
- ^ ab Lee JT, Davidow LS, Warshawsky D (abril de 1999). "Tsix, un gen antisentido de Xist en el centro de inactivación de X". Nature Genetics . 21 (4): 400–4. doi :10.1038/7734. PMID 10192391. S2CID 30636065.
- ^ ab Mercer, TR, Dinger, ME, Mattick, JS, (2009). ARN largos no codificantes: conocimiento de sus funciones. Nature Reviews Genetics. (10) 155–159.
- ^ Hoki Y, Kimura N, Kanbayashi M, Amakawa Y, Ohhata T, Sasaki H, Sado T (enero de 2009). "Una repetición conservada proximal en el gen Xist es esencial como elemento genómico para la inactivación del cromosoma X en ratones". Desarrollo . 136 (1): 139–46. doi : 10.1242/dev.026427 . PMID 19036803.
- ^ Ng K, Pullirsch D, Leeb M, Wutz A (enero de 2007). "Xist y el orden del silenciamiento" (artículo de revisión) . EMBO Reports . 8 (1): 34–9. doi :10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100. Figura
1 El ARN Xist abarca el cromosoma X a partir del cual se transcribe.
{{cite journal}}: Enlace externo en|quote= - ^ Penny GD, Kay GF, Sheardown SA, Rastan S, Brockdorff N (1996). "Requisitos de Xist en la inactivación del cromosoma X". Nature . 379 (6561): 131–7. Bibcode :1996Natur.379..131P. doi :10.1038/379131a0. PMID 8538762. S2CID 4329368.
- ^ ab Herzing LB, Romer JT, Horn JM, Ashworth A (marzo de 1997). "Xist tiene propiedades del centro de inactivación del cromosoma X". Nature . 386 (6622): 272–5. Bibcode :1997Natur.386..272H. doi :10.1038/386272a0. PMID 9069284. S2CID 4371247.
- ^ Ng K, Pullirsch D, Leeb M, Wutz A (enero de 2007). "Xist y el orden del silenciamiento" (artículo de revisión) . EMBO Reports . 8 (1): 34–9. doi :10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100. Tabla
1 Características del territorio X inactivo
{{cite journal}}: Enlace externo en|quote=
Lucchesi JC, Kelly WG, Panning B (2005). "Remodelación de la cromatina en la compensación de dosis". Revisión anual de genética . 39 : 615–51. CiteSeerX 10.1.1.328.2992 . doi :10.1146/annurev.genet.39.073003.094210. PMID 16285873. - ^ Veneti Z, Gkouskou KK, Eliopoulos AG (julio de 2017). "Complejo represor Polycomb 2 en la inestabilidad genómica y el cáncer". Int J Mol Sci . 18 (8): 1657. doi : 10.3390/ijms18081657 . PMC 5578047 . PMID 28758948.
- ^ Costanzi C, Pehrson JR (junio de 1998). "La histona macroH2A1 se concentra en el cromosoma X inactivo de los mamíferos hembra". Nature . 393 (6685): 599–601. Bibcode :1998Natur.393..599C. doi :10.1038/31275. PMID 9634239. S2CID 205001095.
- ^ Costanzi C, Stein P, Worrad DM, Schultz RM, Pehrson JR (junio de 2000). "La histona macroH2A1 se concentra en el cromosoma X inactivo de embriones de ratón preimplantacionales femeninos" (PDF) . Desarrollo . 127 (11): 2283–9. doi :10.1242/dev.127.11.2283. PMID 10804171.
- ^ Barr ML, Bertram EG (abril de 1949). "Una distinción morfológica entre las neuronas masculinas y femeninas, y el comportamiento del satélite nucleolar durante la síntesis acelerada de nucleoproteínas". Nature . 163 (4148): 676–677. Bibcode :1949Natur.163..676B. doi :10.1038/163676a0. PMID 18120749. S2CID 4093883.
- ^ Plath K, Mlynarczyk-Evans S, Nusinow DA, Panning B (2002). "Xist RNA y el mecanismo de inactivación del cromosoma X". Revisión anual de genética . 36 : 233–78. doi :10.1146/annurev.genet.36.042902.092433. PMID 12429693.
- ^ ab Carrel L, Willard HF (marzo de 2005). "El perfil de inactivación del cromosoma X revela una amplia variabilidad en la expresión génica ligada al cromosoma X en mujeres". Nature . 434 (7031): 400–4. Bibcode :2005Natur.434..400C. doi :10.1038/nature03479. PMID 15772666. S2CID 4358447.
- ^ abc Calabrese JM, Sun W, Song L, Mugford JW, Williams L, Yee D, Starmer J, Mieczkowski P, Crawford GE, Magnuson T (noviembre de 2012). "Silenciamiento específico del sitio de elementos reguladores como mecanismo de inactivación de X". Cell . 151 (5): 951–63. doi :10.1016/j.cell.2012.10.037. PMC 3511858 . PMID 23178118.
- ^ ab Yang F, Babak T, Shendure J, Disteche CM (mayo de 2010). "Estudio global sobre el escape de la inactivación del cromosoma X mediante secuenciación de ARN en ratones". Genome Research . 20 (5): 614–22. doi :10.1101/gr.103200.109. PMC 2860163 . PMID 20363980.
- ^ "Síndrome de Turner: MedlinePlus Genetics". medlineplus.gov . Consultado el 10 de febrero de 2023 .
- ^ Berletch JB, Yang F, Disteche CM (junio de 2010). "Escape de la inactivación del cromosoma X en ratones y humanos". Genome Biology . 11 (6): 213. doi : 10.1186/gb-2010-11-6-213 . PMC 2911101 . PMID 20573260.
- ^ Linder D, Gartler SM (octubre de 1965). "Mosaicismo de la glucosa-6-fosfato deshidrogenasa: utilización como marcador celular en el estudio de los leiomiomas". Science . 150 (3692): 67–9. Bibcode :1965Sci...150...67L. doi :10.1126/science.150.3692.67. PMID 5833538. S2CID 33941451.
- ^ Parsons BL (2008). "Muchos tipos de tumores diferentes tienen un origen tumoral policlonal: evidencia e implicaciones". Mutation Research . 659 (3): 232–47. doi :10.1016/j.mrrev.2008.05.004. PMID 18614394.
- ^ Chen GL, Prchal JT (septiembre de 2007). "Pruebas de clonalidad ligadas al cromosoma X: interpretación y limitaciones". Blood . 110 (5): 1411–9. doi :10.1182/blood-2006-09-018655. PMC 1975831 . PMID 17435115.
- ^ Petersson F, Branzovsky J, Martinek P, Korabecna M, Kruslin B, Hora M, et al. (julio de 2014). "El estroma leiomiomatoso en carcinomas de células renales es policlonal y no forma parte del proceso neoplásico". Virchows Archiv . 465 (1): 89–96. doi :10.1007/s00428-014-1591-9. PMID 24838683. S2CID 24870232.
- ^ Comertpay S, Pastorino S, Tanji M, Mezzapelle R, Strianese O, Napolitano A, Baumann F, Weigel T, Friedberg J, Sugarbaker P, Krausz T, Wang E, Powers A, Gaudino G, Kanodia S, Pass HI, Parsons BL, Yang H, Carbone M (diciembre de 2014). "Evaluación del origen clonal del mesotelioma maligno". Revista de medicina traslacional . 12 : 301. doi : 10.1186/s12967-014-0301-3 . PMC 4255423 . PMID 25471750.
- ^ Jiang J, Jing Y, Cost GJ, Chiang JC, Kolpa HJ, Cotton AM, et al. (agosto de 2013). "Traducción de la compensación de dosis a la trisomía 21". Nature . 500 (7462): 296–300. Bibcode :2013Natur.500..296J. doi :10.1038/nature12394. PMC 3848249 . PMID 23863942.
- ^ Ohno S, Kaplan WD, Kinosita R (octubre de 1959). "Formación de la cromatina sexual por un solo cromosoma X en células hepáticas de Rattus norvegicus". Experimental Cell Research . 18 (2): 415–8. doi :10.1016/0014-4827(59)90031-X. PMID 14428474.
- ^ Lyon MF (abril de 1961). "Acción génica en el cromosoma X del ratón (Mus musculus L.)". Nature . 190 (4773): 372–3. Bibcode :1961Natur.190..372L. doi :10.1038/190372a0. PMID 13764598. S2CID 4146768.
- ^ Beutler E (enero de 2008). "Deficiencia de glucosa-6-fosfato deshidrogenasa: una perspectiva histórica". Blood . 111 (1): 16–24. doi : 10.1182/blood-2007-04-077412 . PMID 18156501.
- ^ Beutler E, Yeh M, Fairbanks VF (enero de 1962). "La hembra humana normal como un mosaico de actividad del cromosoma X: estudios que utilizan el gen de la deficiencia de C-6-PD como marcador". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 48 (1): 9–16. Bibcode :1962PNAS...48....9B. doi : 10.1073/pnas.48.1.9 . PMC 285481 . PMID 13868717.
Lectura adicional
- Huynh KD, Lee JT (mayo de 2005). "Inactivación del cromosoma X: una hipótesis que vincula la ontogenia y la filogenia". Nature Reviews. Genetics . 6 (5): 410–8. doi :10.1038/nrg1604. PMID 15818384. S2CID 19245310.
- Goto T, Monk M (junio de 1998). "Regulación de la inactivación del cromosoma X en el desarrollo de ratones y humanos" (artículo de revisión) . Microbiology and Molecular Biology Reviews . 62 (2): 362–78. doi :10.1128 / MMBR.62.2.362-378.1998. PMC 98919. PMID 9618446.
- Lyon MF (diciembre de 2003). "La hipótesis de Lyon y LINE". Seminars in Cell & Developmental Biology (artículo de revisión). 14 (6): 313–8. doi :10.1016/j.semcdb.2003.09.015. PMID 15015738.
- Ng K, Pullirsch D, Leeb M, Wutz A (enero de 2007). "Xist y el orden del silenciamiento" (artículo de revisión) . EMBO Reports . 8 (1): 34–9. doi :10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100 .
- Cerase A, Pintacuda G, Tattermusch A, Avner P (agosto de 2015). "Localización y función de Xist: nuevos conocimientos desde múltiples niveles". Genome Biology . 16 (1): 166. doi : 10.1186/s13059-015-0733-y . PMC 4539689 . PMID 26282267.
