Ciclo celular


El ciclo celular , o ciclo de división celular , es la serie secuencial de eventos que tienen lugar en una célula y que provocan su división en dos células hijas. Estos eventos incluyen el crecimiento de la célula, la duplicación de su ADN ( replicación del ADN ) y algunos de sus orgánulos , y posteriormente la partición de su citoplasma, cromosomas y otros componentes en dos células hijas en un proceso llamado división celular .
En las células eucariotas (que tienen un núcleo celular ), incluidas las células animales , vegetales , fúngicas y protistas , el ciclo celular se divide en dos etapas principales: la interfase y la fase M , que incluye la mitosis y la citocinesis. [1] Durante la interfase, la célula crece, acumula los nutrientes necesarios para la mitosis y replica su ADN y algunos de sus orgánulos. Durante la fase M, los cromosomas , orgánulos y citoplasma replicados se separan en dos nuevas células hijas. Para garantizar la replicación adecuada de los componentes celulares y la división, existen mecanismos de control conocidos como puntos de control del ciclo celular después de cada uno de los pasos clave del ciclo que determinan si la célula puede progresar a la siguiente fase.
En las células sin núcleo, como las procariotas , las bacterias y las arqueas , el ciclo celular se divide en los períodos B, C y D. El período B se extiende desde el final de la división celular hasta el comienzo de la replicación del ADN. La replicación del ADN ocurre durante el período C. El período D se refiere a la etapa entre el final de la replicación del ADN y la división de la célula bacteriana en dos células hijas. [2]
En los organismos unicelulares, un solo ciclo de división celular es la forma en que el organismo se reproduce para asegurar su supervivencia. En los organismos multicelulares, como las plantas y los animales, una serie de ciclos de división celular es la forma en que el organismo se desarrolla a partir de un óvulo fertilizado unicelular hasta convertirse en un organismo maduro, y también es el proceso por el cual el cabello , la piel , las células sanguíneas y algunos órganos internos se regeneran y curan (con la posible excepción de los nervios ; ver daño a los nervios ). Después de la división celular, cada una de las células hijas comienza la interfase de un nuevo ciclo celular. Aunque las diversas etapas de la interfase no suelen ser morfológicamente distinguibles, cada fase del ciclo celular tiene un conjunto distinto de procesos bioquímicos especializados que preparan a la célula para el inicio de la división celular.
Fases
El ciclo celular eucariota consta de cuatro fases distintas: fase G 1 , fase S (síntesis), fase G 2 (conocidas colectivamente como interfase ) y fase M (mitosis y citocinesis). La fase M se compone a su vez de dos procesos estrechamente acoplados: la mitosis, en la que se divide el núcleo de la célula, y la citocinesis , en la que el citoplasma y la membrana celular de la célula se dividen formando dos células hijas. La activación de cada fase depende de la progresión y finalización adecuadas de la anterior. Se dice que las células que han dejado de dividirse temporal o reversiblemente han entrado en un estado de inactividad llamado fase G 0 o fase de reposo.
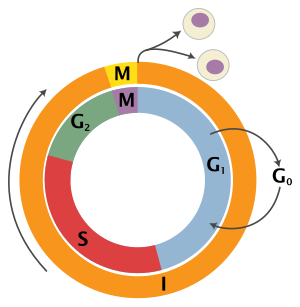
| Estado | Fase | Abreviatura | Descripción |
|---|---|---|---|
| Descansando | Brecha 0 | G 0 | Una fase en la que la célula ha abandonado el ciclo y ha dejado de dividirse. |
| Interfase | Brecha 1 | G 1 | Crecimiento celular. El punto de control G1 garantiza que todo esté listo para la síntesis de ADN . |
| Síntesis | S | Replicación del ADN . | |
| Brecha 2 | G2 | Crecimiento y preparación para la mitosis. El punto de control G2 garantiza que todo esté listo para entrar en la fase M (mitosis) y dividirse. | |
| División celular | Mitosis | METRO | Se produce la división celular. El punto de control de la metafase garantiza que la célula esté lista para completar la división celular. |
GRAMO0fase (quiescencia)

G 0 es una fase de reposo en la que la célula ha abandonado el ciclo y ha dejado de dividirse. El ciclo celular comienza con esta fase. Las células no proliferativas (que no se dividen) en eucariotas multicelulares generalmente entran en el estado G 0 quiescente desde G 1 y pueden permanecer quiescentes durante largos períodos de tiempo, posiblemente indefinidamente (como suele ser el caso de las neuronas ). Esto es muy común para las células que están completamente diferenciadas . Algunas células entran en la fase G 0 de forma semipermanente y se consideran posmitóticas, por ejemplo, algunas células del hígado, los riñones y el estómago. Muchas células no entran en G 0 y continúan dividiéndose a lo largo de la vida de un organismo, por ejemplo, las células epiteliales.
La palabra "postmitótica" se utiliza a veces para referirse tanto a células quiescentes como senescentes . La senescencia celular se produce en respuesta al daño del ADN y al estrés externo y, por lo general, constituye una detención de G 1 . La senescencia celular puede hacer que la progenie de una célula no sea viable; a menudo es una alternativa bioquímica a la autodestrucción de una célula dañada por apoptosis .
Interfase
La interfase representa la fase entre dos fases M sucesivas. La interfase es una serie de cambios que tienen lugar en una célula recién formada y su núcleo antes de que pueda volver a dividirse. También se denomina fase preparatoria o intermitosis. Normalmente, la interfase dura al menos el 91 % del tiempo total necesario para el ciclo celular.
La interfase transcurre en tres etapas, G 1 , S y G 2 , seguidas por el ciclo de mitosis y citocinesis. El contenido de ADN nuclear de la célula se duplica durante la fase S.
GRAMO1Fase (primera fase de crecimiento o fase de brecha post mitótica)

La primera fase dentro de la interfase, desde el final de la fase M anterior hasta el comienzo de la síntesis de ADN, se llama G 1 (G indica gap ). También se llama fase de crecimiento. Durante esta fase, las actividades biosintéticas de la célula, que se ralentizan considerablemente durante la fase M, se reanudan a un ritmo elevado. La duración de G 1 es muy variable, incluso entre diferentes células de la misma especie. [4] En esta fase, la célula aumenta su suministro de proteínas, aumenta el número de orgánulos (como mitocondrias, ribosomas) y crece en tamaño. En la fase G 1 , una célula tiene tres opciones.
- Para continuar el ciclo celular y entrar en la fase S
- Detener el ciclo celular y entrar en la fase G0 para realizar la diferenciación .
- Queda detenido en la fase G 1 , por lo que puede entrar en la fase G 0 o reingresar al ciclo celular.
El punto de decisión se denomina punto de control ( punto de restricción ). Este punto de control se denomina punto de restricción o START y está regulado por las ciclinas G 1 /S, que provocan la transición de la fase G 1 a la fase S. El paso por el punto de control G 1 compromete a la célula a la división.
Fase S (replicación del ADN)
La fase S que sigue comienza cuando comienza la síntesis de ADN ; cuando se completa, todos los cromosomas se han replicado, es decir, cada cromosoma consta de dos cromátidas hermanas . Por lo tanto, durante esta fase, la cantidad de ADN en la célula se ha duplicado, aunque la ploidía y el número de cromosomas no han cambiado. Las tasas de transcripción de ARN y síntesis de proteínas son muy bajas durante esta fase. Una excepción a esto es la producción de histonas , la mayor parte de la cual ocurre durante la fase S. [5] [6] [7]
GRAMO2fase (crecimiento)
La fase G2 ocurre después de la replicación del ADN y es un período de síntesis de proteínas y crecimiento celular rápido para preparar la célula para la mitosis. Durante esta fase, los microtúbulos comienzan a reorganizarse para formar un huso (preprofase). Antes de pasar a la fase mitótica , las células deben revisarse en el punto de control G2 para detectar cualquier daño en el ADN dentro de los cromosomas. El punto de control G2 está regulado principalmente por la proteína tumoral p53 . Si el ADN está dañado, p53 reparará el ADN o desencadenará la apoptosis de la célula. Si p53 es disfuncional o mutado, las células con ADN dañado pueden continuar a través del ciclo celular, lo que lleva al desarrollo del cáncer.
Fase mitótica (separación de cromosomas)
La fase M, relativamente breve, consiste en la división nuclear ( cariocinesis ) y la división del citoplasma ( citocinesis ). Es un período relativamente corto del ciclo celular. La fase M es compleja y está muy regulada. La secuencia de eventos se divide en fases, que corresponden a la finalización de un conjunto de actividades y al inicio del siguiente. Estas fases se conocen secuencialmente como:

La mitosis es el proceso mediante el cual una célula eucariota separa los cromosomas de su núcleo celular en dos conjuntos idénticos en dos núcleos. [8] Durante el proceso de mitosis, los pares de cromosomas se condensan y se adhieren a microtúbulos que atraen a las cromátidas hermanas hacia lados opuestos de la célula. [9]
La mitosis se produce exclusivamente en células eucariotas , pero se produce de forma diferente en distintas especies. Por ejemplo, las células animales experimentan una mitosis "abierta", en la que la envoltura nuclear se rompe antes de que los cromosomas se separen, mientras que los hongos como Aspergillus nidulans y Saccharomyces cerevisiae ( levadura ) experimentan una mitosis "cerrada", en la que los cromosomas se dividen dentro de un núcleo celular intacto . [10]
Fase de citocinesis (separación de todos los componentes celulares)
La mitosis es seguida inmediatamente por la citocinesis , que divide los núcleos, el citoplasma , los orgánulos y la membrana celular en dos células que contienen proporciones aproximadamente iguales de estos componentes celulares. La citocinesis ocurre de manera diferente en las células vegetales y animales. Mientras que la membrana celular forma un surco que se profundiza gradualmente para separar el citoplasma en las células animales, se forma una placa celular para separarlo en las células vegetales. La posición de la placa celular está determinada por la posición de una banda preprofásica de microtúbulos y filamentos de actina . La mitosis y la citocinesis juntas definen la división de la célula madre en dos células hijas, genéticamente idénticas entre sí y a su célula madre. Esto representa aproximadamente el 10% del ciclo celular.
Debido a que la citocinesis suele ocurrir junto con la mitosis, "mitosis" se usa a menudo indistintamente con "fase M". Sin embargo, hay muchas células en las que la mitosis y la citocinesis ocurren por separado, formando células individuales con múltiples núcleos en un proceso llamado endorreplicación . Esto ocurre más notablemente entre los hongos y los mohos mucilaginosos , pero se encuentra en varios grupos. Incluso en animales, la citocinesis y la mitosis pueden ocurrir de forma independiente, por ejemplo durante ciertas etapas del desarrollo embrionario de la mosca de la fruta . [11] Los errores en la mitosis pueden provocar la muerte celular por apoptosis o causar mutaciones que pueden conducir al cáncer .
Regulación del ciclo celular eucariota

La regulación del ciclo celular implica procesos cruciales para la supervivencia de una célula, entre los que se incluyen la detección y reparación de daños genéticos, así como la prevención de la división celular descontrolada. Los eventos moleculares que controlan el ciclo celular son ordenados y direccionales; es decir, cada proceso ocurre de manera secuencial y es imposible "revertir" el ciclo.
Papel de las ciclinas y las CDK
 Premio Nobel Paul Nurse |  Premio Nobel Tim Hunt |
Dos clases clave de moléculas reguladoras, las ciclinas y las quinasas dependientes de ciclina (CDK), determinan el progreso de una célula a través del ciclo celular. [12] Leland H. Hartwell , R. Timothy Hunt y Paul M. Nurse ganaron el Premio Nobel de Fisiología o Medicina de 2001 por su descubrimiento de estas moléculas centrales. [13] Muchos de los genes que codifican ciclinas y CDK se conservan entre todos los eucariotas, pero en general, los organismos más complejos tienen sistemas de control del ciclo celular más elaborados que incorporan más componentes individuales. Muchos de los genes relevantes se identificaron por primera vez estudiando la levadura, especialmente Saccharomyces cerevisiae ; [14] la nomenclatura genética en levaduras denomina a muchos de estos genes cdc (para "ciclo de división celular") seguido de un número de identificación, p. ej. cdc25 o cdc20 .
Las ciclinas forman las subunidades reguladoras y las CDK las subunidades catalíticas de un heterodímero activado ; las ciclinas no tienen actividad catalítica y las CDK son inactivas en ausencia de una ciclina asociada. Cuando se activan por una ciclina unida, las CDK realizan una reacción bioquímica común llamada fosforilación que activa o inactiva las proteínas objetivo para orquestar la entrada coordinada a la siguiente fase del ciclo celular. Diferentes combinaciones de ciclina-CDK determinan las proteínas diana posteriores. Las CDK se expresan constitutivamente en las células, mientras que las ciclinas se sintetizan en etapas específicas del ciclo celular, en respuesta a varias señales moleculares. [15]
Mecanismo general de interacción ciclina-CDK
Al recibir una señal extracelular pro-mitótica, los complejos G 1 ciclina-CDK se activan para preparar la célula para la fase S, promoviendo la expresión de factores de transcripción que a su vez promueven la expresión de ciclinas S y de enzimas requeridas para la replicación del ADN . Los complejos G 1 ciclina-CDK también promueven la degradación de moléculas que funcionan como inhibidores de la fase S al dirigirlas a la ubiquitinación . Una vez que una proteína ha sido ubiquitinada, es dirigida a la degradación proteolítica por el proteasoma . Sin embargo, los resultados de un estudio reciente de la dinámica transcripcional de E2F a nivel de célula única sostienen que el papel de las actividades G1 ciclina-CDK, en particular ciclina D-CDK4/6, es ajustar el tiempo en lugar del compromiso de la entrada al ciclo celular. [16]
Los complejos de S-ciclina-CDK activos fosforilan las proteínas que forman los complejos de prerreplicación ensamblados durante la fase G 1 en los orígenes de replicación del ADN . La fosforilación tiene dos propósitos: activar cada complejo de prerreplicación ya ensamblado y evitar que se formen nuevos complejos. Esto garantiza que cada porción del genoma de la célula se replicará una vez y solo una vez. La razón para la prevención de brechas en la replicación es bastante clara, porque las células hijas a las que les faltan todos o parte de los genes cruciales morirán. Sin embargo, por razones relacionadas con los efectos del número de copias de genes , la posesión de copias adicionales de ciertos genes también es perjudicial para las células hijas.
Los complejos ciclina-CDK mitóticos, que se sintetizan pero se inactivan durante las fases S y G2 , promueven el inicio de la mitosis al estimular las proteínas posteriores involucradas en la condensación de los cromosomas y el ensamblaje del huso mitótico . Un complejo crítico activado durante este proceso es una ligasa de ubiquitina conocida como complejo promotor de anafase (APC), que promueve la degradación de las proteínas estructurales asociadas con el cinetocoro cromosómico . El APC también se dirige a las ciclinas mitóticas para su degradación, lo que garantiza que la telofase y la citocinesis puedan continuar. [17]
Acción específica de los complejos ciclina-CDK
La ciclina D es la primera ciclina producida en las células que entran en el ciclo celular, en respuesta a señales extracelulares (p. ej. factores de crecimiento ). Los niveles de ciclina D se mantienen bajos en células en reposo que no están proliferando. Además, CDK4/6 y CDK2 también están inactivos porque CDK4/6 está unido por miembros de la familia INK4 (p. ej., p16), lo que limita la actividad de la quinasa. Mientras tanto, los complejos CDK2 son inhibidos por las proteínas CIP/KIP como p21 y p27, [18] Cuando llega el momento de que una célula entre en el ciclo celular, lo que se desencadena por un estímulo mitogénico, los niveles de ciclina D aumentan. En respuesta a este desencadenante, la ciclina D se une al CDK4 /6 existente, formando el complejo ciclina D-CDK4/6 activo. Los complejos ciclina D-CDK4/6 a su vez monofosforilan la proteína de susceptibilidad al retinoblastoma ( Rb ) a pRb. El supresor tumoral Rb no fosforilado actúa induciendo la salida del ciclo celular y manteniendo el arresto de G0 (senescencia). [19]
En las últimas décadas, se ha aceptado ampliamente un modelo según el cual las proteínas pRB se inactivan mediante la fosforilación mediada por ciclina D-Cdk4/6. Rb tiene más de 14 sitios de fosforilación potenciales. La ciclina D-Cdk 4/6 fosforila progresivamente a Rb hasta el estado hiperfosforilado, lo que desencadena la disociación de los complejos pRB- E2F , induciendo así la expresión génica del ciclo celular G1/S y la progresión a la fase S. [20]
Sin embargo, las observaciones científicas de un estudio reciente muestran que Rb está presente en tres tipos de isoformas: (1) Rb no fosforilado en estado G0; (2) Rb monofosforilado, también denominado Rb "hipofosforilado" o "parcialmente" fosforilado en el estado G1 temprano; y (3) Rb hiperfosforilado inactivo en el estado G1 tardío. [21] [22] [23] En las células G1 tempranas, el Rb monofosforilado existe como 14 isoformas diferentes, una de cada una tiene una afinidad de unión a E2F distinta . [23] Se ha descubierto que el Rb se asocia con cientos de proteínas diferentes [24] y la idea de que diferentes isoformas de Rb monofosforiladas tienen diferentes parejas proteicas era muy atractiva. [25] Un informe reciente confirmó que la monofosforilación controla la asociación de Rb con otras proteínas y genera formas funcionales distintas de Rb. [26] Todas las diferentes isoformas de Rb monofosforiladas inhiben el programa transcripcional de E2F y pueden detener las células en la fase G1. Es importante destacar que las diferentes formas monofosforiladas de Rb tienen resultados transcripcionales distintos que son extendido más allá de la regulación E2F. [26]
En general, la unión de pRb a E2F inhibe la expresión del gen diana E2F de ciertos genes de transición G1/S y S, incluidas las ciclinas de tipo E. La fosforilación parcial de Rb desreprime la supresión mediada por Rb de la expresión del gen diana E2F, comienza la expresión de ciclina E. El mecanismo molecular que hace que la célula cambie a la activación de ciclina E actualmente no se conoce, pero a medida que aumentan los niveles de ciclina E, se forma el complejo ciclina E-CDK2 activo, lo que hace que Rb se inactive por hiperfosforilación. [23] El Rb hiperfosforilado se disocia completamente de E2F, lo que permite una mayor expresión de una amplia gama de genes diana E2F que son necesarios para impulsar a las células a pasar a la fase S [1]. Recientemente, se ha identificado que la ciclina D-Cdk4/6 se une a una región de hélice alfa C-terminal de Rb que solo se distingue de la ciclina D en lugar de otras ciclinas, ciclina E , A y B. [27] Esta observación basada en el análisis estructural de la fosforilación de Rb respalda que Rb se fosforila en un nivel diferente a través de múltiples complejos de ciclina-Cdk. Esto también hace factible el modelo actual de una inactivación simultánea de tipo interruptor de todas las isoformas monofosforiladas de Rb a través de un tipo de mecanismo de hiperfosforilación de Rb. Además, el análisis mutacional de la hélice C-terminal de Rb específica de ciclina D-Cdk 4/6 muestra que las interrupciones de la unión de ciclina D-Cdk 4/6 a Rb previenen la fosforilación de Rb, detienen las células en G1 y refuerzan las funciones de Rb en la supresión tumoral. [27] Este mecanismo de transición del ciclo celular impulsado por la ciclina-Cdk gobierna una célula comprometida con el ciclo celular que permite la proliferación celular. El crecimiento de una célula cancerosa suele ir acompañado de una desregulación de la actividad de la ciclina D-Cdk 4/6.
El Rb hiperfosforilado se disocia del complejo E2F/DP1/Rb (que estaba unido a los genes sensibles a E2F , "bloqueándolos" eficazmente de la transcripción), activando E2F. La activación de E2F da como resultado la transcripción de varios genes como ciclina E , ciclina A , ADN polimerasa , timidina quinasa , etc. La ciclina E así producida se une a CDK2 , formando el complejo ciclina E-CDK2, que empuja a la célula de la fase G 1 a la S (G 1 /S, que inicia la transición G 2 /M). [28] La activación del complejo ciclina B -cdk1 causa la ruptura de la envoltura nuclear y el inicio de la profase , y posteriormente, su desactivación hace que la célula salga de la mitosis. [15] Un estudio cuantitativo de la dinámica transcripcional de E2F a nivel de célula única mediante el uso de células reporteras fluorescentes diseñadas proporcionó un marco cuantitativo para comprender la lógica de control de la entrada al ciclo celular, desafiando el modelo canónico de los libros de texto. Los genes que regulan la amplitud de la acumulación de E2F, como Myc, determinan el compromiso en el ciclo celular y la entrada en la fase S. Las actividades de la ciclina G1-CDK no son el impulsor de la entrada en el ciclo celular. En cambio, principalmente ajustan el momento del aumento de E2F, modulando así el ritmo de la progresión del ciclo celular. [16]
Inhibidores
Endógeno

Dos familias de genes, la familia cip/kip ( proteína que interactúa con CDK/proteína inhibidora de quinasas ) y la familia INK4a/ARF ( inhibidor de la quinasa 4/ marco de lectura alternativo ) , impiden la progresión del ciclo celular. Debido a que estos genes son fundamentales para la prevención de la formación de tumores , se los conoce como supresores tumorales .
La familia cip/kip incluye los genes p21 , p27 y p57 . Estos genes detienen el ciclo celular en la fase G1 uniéndose a los complejos ciclina-CDK y desactivándolos. p21 es activado por p53 (que, a su vez, es activado por daño del ADN, por ejemplo, debido a la radiación). p27 es activado por el factor de crecimiento transformante β ( TGF β ), un inhibidor del crecimiento.
La familia INK4a/ARF incluye p16 INK4a , que se une a CDK4 y detiene el ciclo celular en la fase G 1 , y p14 ARF , que previene la degradación de p53.
Sintético
Los inhibidores sintéticos de Cdc25 también podrían ser útiles para detener el ciclo celular y, por lo tanto, ser útiles como agentes antineoplásicos y anticancerígenos. [29]
Muchos cánceres humanos poseen actividades hiperactivadas de Cdk 4/6. [30] Dadas las observaciones de las funciones de ciclina D-Cdk 4/6, la inhibición de Cdk 4/6 debería resultar en la prevención de la proliferación de un tumor maligno. En consecuencia, los científicos han tratado de inventar el inhibidor sintético de Cdk4/6, ya que Cdk4/6 se ha caracterizado por ser un objetivo terapéutico para la eficacia antitumoral. Tres inhibidores de Cdk4/6 ( palbociclib , ribociclib y abemaciclib ) recibieron actualmente la aprobación de la FDA para uso clínico para tratar el cáncer de mama en etapa avanzada o metastásico , positivo para el receptor de hormonas (HR-positivo, HR+), negativo para HER2 (HER2-). [31] [32] Por ejemplo, palbociclib es un inhibidor de CDK4/6 activo por vía oral que ha demostrado mejores resultados para el cáncer de mama avanzado ER-positivo/HER2-negativo. El principal efecto secundario es la neutropenia , que puede controlarse mediante la reducción de la dosis. [33]
La terapia dirigida a Cdk4/6 solo tratará los tipos de cáncer en los que se expresa Rb. Las células cancerosas con pérdida de Rb tienen resistencia primaria a los inhibidores de Cdk4/6.
Red reguladora de la transcripción
La evidencia actual sugiere que una red transcripcional semiautónoma actúa en conjunto con la maquinaria de ciclinas CDK para regular el ciclo celular. Varios estudios de expresión génica en Saccharomyces cerevisiae han identificado entre 800 y 1200 genes que cambian su expresión a lo largo del ciclo celular. [14] [34] [35] Se transcriben en niveles altos en puntos específicos del ciclo celular y permanecen en niveles más bajos durante el resto del ciclo. Si bien el conjunto de genes identificados difiere entre los estudios debido a los métodos computacionales y los criterios utilizados para identificarlos, cada estudio indica que una gran parte de los genes de la levadura están regulados temporalmente. [36]
Muchos genes expresados periódicamente son impulsados por factores de transcripción que también se expresan periódicamente. Un análisis de genes knockouts identificó 48 factores de transcripción (alrededor del 20% de todos los factores de transcripción no esenciales) que muestran defectos en la progresión del ciclo celular. [37] Estudios de todo el genoma que utilizan tecnologías de alto rendimiento han identificado los factores de transcripción que se unen a los promotores de los genes de levadura, y la correlación de estos hallazgos con patrones de expresión temporal ha permitido la identificación de factores de transcripción que impulsan la expresión génica específica de fase. [34] [38] Los perfiles de expresión de estos factores de transcripción son impulsados por los factores de transcripción que alcanzan su pico en la fase anterior, y los modelos computacionales han demostrado que una red autónoma de CDK de estos factores de transcripción es suficiente para producir oscilaciones de estado estable en la expresión génica). [35] [39]
La evidencia experimental también sugiere que la expresión génica puede oscilar con el período observado en la división de células de tipo salvaje independientemente de la maquinaria CDK. Orlando et al. utilizaron microarrays para medir la expresión de un conjunto de 1.271 genes que identificaron como periódicos tanto en células de tipo salvaje como en células que carecían de todas las ciclinas de fase S y mitóticas ( clb1,2,3,4,5,6 ). De los 1.271 genes ensayados, 882 continuaron expresándose en las células deficientes en ciclina al mismo tiempo que en las células de tipo salvaje, a pesar del hecho de que las células deficientes en ciclina se detienen en el límite entre la fase G 1 y S . Sin embargo, 833 de los genes ensayados cambiaron de comportamiento entre las células de tipo salvaje y mutantes, lo que indica que es probable que estos genes estén regulados directa o indirectamente por la maquinaria CDK-ciclina. Algunos genes que continuaron expresándose a tiempo en las células mutantes también se expresaron en diferentes niveles en las células mutantes y de tipo salvaje. Estos hallazgos sugieren que si bien la red transcripcional puede oscilar independientemente del oscilador CDK-ciclina, están acoplados de una manera que requiere de ambos para asegurar la sincronización adecuada de los eventos del ciclo celular. [35] Otros trabajos indican que la fosforilación , una modificación postraduccional, de los factores de transcripción del ciclo celular por Cdk1 puede alterar la localización o actividad de los factores de transcripción para controlar estrictamente la sincronización de los genes objetivo. [37] [40] [41]
Si bien la transcripción oscilatoria desempeña un papel clave en la progresión del ciclo celular de la levadura, la maquinaria de ciclinas CDK opera de manera independiente en el ciclo celular embrionario temprano. Antes de la transición a la blástula media , no se produce la transcripción cigótica y todas las proteínas necesarias, como las ciclinas de tipo B, se traducen a partir del ARNm cargado por la madre . [42]
Actividad de replicación del ADN y origen de la replicación del ADN
Los análisis de cultivos sincronizados de Saccharomyces cerevisiae en condiciones que impiden el inicio de la replicación del ADN sin retrasar la progresión del ciclo celular mostraron que la concesión de licencias de origen disminuye la expresión de genes con orígenes cerca de sus extremos 3', lo que revela que los orígenes descendentes pueden regular la expresión de genes ascendentes. [43] Esto confirma predicciones previas a partir del modelado matemático de una coordinación causal global entre la actividad del origen de replicación del ADN y la expresión del ARNm, [44] [45] [46] y muestra que el modelado matemático de datos de microarrays de ADN se puede utilizar para predecir correctamente modos biológicos de regulación previamente desconocidos.
Puestos de control
Los puntos de control del ciclo celular son utilizados por la célula para monitorear y regular el progreso del ciclo celular. [47] Los puntos de control previenen la progresión del ciclo celular en puntos específicos, lo que permite la verificación de los procesos de fase necesarios y la reparación del daño del ADN . La célula no puede avanzar a la siguiente fase hasta que se hayan cumplido los requisitos del punto de control. Los puntos de control generalmente consisten en una red de proteínas reguladoras que monitorean y dictan la progresión de la célula a través de las diferentes etapas del ciclo celular.
Se estima que en las células humanas normales aproximadamente el 1% de los daños en el ADN monocatenario se convierten en aproximadamente 50 roturas de doble cadena de ADN endógeno por célula por ciclo celular. [48] Aunque dichas roturas de doble cadena suelen repararse con alta fidelidad, se considera que los errores en su reparación contribuyen significativamente a la tasa de cáncer en humanos. [48]
Existen varios puntos de control para garantizar que el ADN dañado o incompleto no pase a las células hijas. Existen tres puntos de control principales: el punto de control G 1 /S, el punto de control G 2 /M y el punto de control de la metafase (mitótica). Otro punto de control es el punto de control Go, en el que se comprueba la madurez de las células. Si las células no pasan este punto de control porque aún no están preparadas, se descartarán de la división.
La transición G 1 /S es un paso limitante de la velocidad en el ciclo celular y también se conoce como punto de restricción . [15] Aquí es donde la célula verifica si tiene suficientes materias primas para replicar completamente su ADN (bases de nucleótidos, ADN sintasa, cromatina, etc.). Una célula enferma o desnutrida se quedará atascada en este punto de control.
El punto de control G2 / M es donde la célula se asegura de tener suficiente citoplasma y fosfolípidos para dos células hijas. Pero a veces, lo que es más importante, verifica si es el momento adecuado para replicarse. Hay algunas situaciones en las que muchas células necesitan replicarse simultáneamente (por ejemplo, un embrión en crecimiento debe tener una distribución celular simétrica hasta que alcanza la transición a mitad de la blástula). Esto se hace controlando el punto de control G2 / M.
El punto de control de la metafase es un punto de control bastante menor, ya que una vez que una célula está en metafase, se ha comprometido a realizar la mitosis. Sin embargo, eso no quiere decir que no sea importante. En este punto de control, la célula verifica que se haya formado el huso y que todos los cromosomas estén alineados en el ecuador del huso antes de que comience la anafase. [49]
Aunque estos son los tres puntos de control "principales", no todas las células tienen que pasar por cada uno de ellos en este orden para replicarse. Muchos tipos de cáncer son causados por mutaciones que permiten a las células pasar rápidamente por los distintos puntos de control o incluso saltárselos por completo, pasando de la fase S a la M y de nuevo a la S casi consecutivamente. Como estas células han perdido sus puntos de control, cualquier mutación del ADN que pueda haberse producido se ignora y se transmite a las células hijas. Esta es una de las razones por las que las células cancerosas tienen tendencia a adquirir mutaciones de forma exponencial. Aparte de las células cancerosas, muchos tipos de células completamente diferenciadas ya no se replican, por lo que abandonan el ciclo celular y permanecen en G 0 hasta su muerte, eliminando así la necesidad de los puntos de control celulares. También se ha propuesto un modelo alternativo de la respuesta del ciclo celular al daño del ADN, conocido como punto de control de posreplicación .
La regulación de los puntos de control desempeña un papel importante en el desarrollo de un organismo. En la reproducción sexual, cuando se produce la fecundación del óvulo, cuando el espermatozoide se une al óvulo, libera factores de señalización que notifican al óvulo que ha sido fecundado. Entre otras cosas, esto induce al ovocito ahora fecundado a regresar de su estado previamente latente, G 0 , al ciclo celular y a la replicación y división mitótica.
El p53 desempeña un papel importante en la activación de los mecanismos de control en los puntos de control G 1 /S y G 2 /M. Además del p53, se están investigando intensamente los reguladores de los puntos de control por su papel en el crecimiento y la proliferación del cáncer.
Imágenes de fluorescencia del ciclo celular

El trabajo pionero de Atsushi Miyawaki y sus colaboradores desarrolló el indicador de ciclo celular basado en ubiquitinación fluorescente (FUCCI), que permite la obtención de imágenes de fluorescencia del ciclo celular. Originalmente, una proteína fluorescente verde , mAG, se fusionó a hGem(1/110) y una proteína fluorescente naranja (mKO 2 ) se fusionó a hCdt1(30/120). Tenga en cuenta que estas fusiones son fragmentos que contienen una señal de localización nuclear y sitios de ubiquitinación para la degradación , pero no son proteínas funcionales. La proteína fluorescente verde se produce durante la fase S, G 2 o M y se degrada durante la fase G 0 o G 1 , mientras que la proteína fluorescente naranja se produce durante la fase G 0 o G 1 y se destruye durante la fase S, G 2 o M. [50] Se desarrolló un FUCCI de infrarrojo cercano y rojo lejano utilizando una proteína fluorescente derivada de cianobacterias ( smURFP ) y una proteína fluorescente derivada de bacteriofitocromo (película disponible en este enlace). [51] Se han realizado varias modificaciones al sistema FUCCI original para mejorar su utilidad en varios sistemas in vitro y organismos modelo. Estos avances han aumentado la sensibilidad y precisión de la detección de la fase del ciclo celular, lo que permite evaluaciones más precisas de la proliferación celular. [52]
Papel en la formación de tumores
Una desregulación de los componentes del ciclo celular puede conducir a la formación de tumores . [53] Como se mencionó anteriormente, cuando algunos genes como los inhibidores del ciclo celular, RB , p53 , etc. mutan, pueden hacer que la célula se multiplique sin control, formando un tumor. Aunque la duración del ciclo celular en las células tumorales es igual o mayor que la del ciclo celular normal, la proporción de células que están en división celular activa (en comparación con las células quiescentes en fase G 0 ) en los tumores es mucho mayor que en el tejido normal. [54] Por lo tanto, hay un aumento neto en el número de células, ya que el número de células que mueren por apoptosis o senescencia permanece igual.
Las células que se encuentran en pleno proceso de división celular son el objetivo de la terapia contra el cáncer, ya que el ADN está relativamente expuesto durante la división celular y, por lo tanto, es susceptible a daños por fármacos o radiación . Este hecho se aprovecha en el tratamiento del cáncer; mediante un proceso conocido como desbulking , se elimina una masa significativa del tumor, lo que empuja a una cantidad significativa de las células tumorales restantes de la fase G 0 a la G 1 (debido a una mayor disponibilidad de nutrientes, oxígeno, factores de crecimiento, etc.). La radiación o quimioterapia posteriores al procedimiento de desbulking matan estas células que han entrado recientemente en el ciclo celular. [15]
Las células de mamíferos con ciclos más rápidos en cultivo, las células de las criptas del epitelio intestinal, tienen un ciclo de tan solo 9 a 10 horas. Las células madre de la piel de ratón en reposo pueden tener un ciclo de más de 200 horas. La mayor parte de esta diferencia se debe a la duración variable de G 1 , la fase más variable del ciclo. M y S no varían mucho.
En general, las células son más radiosensibles en las últimas fases M y G2 y más resistentes en la última fase S. En el caso de las células con un ciclo celular más largo y una fase G1 significativamente larga , hay un segundo pico de resistencia en la última fase G1 . El patrón de resistencia y sensibilidad se correlaciona con el nivel de compuestos sulfhidrilos en la célula. Los sulfhidrilos son sustancias naturales que protegen a las células del daño por radiación y tienden a alcanzar sus niveles más altos en la fase S y los más bajos cerca de la mitosis.
La recombinación homóloga (HR) es un proceso preciso para reparar roturas de doble cadena de ADN. La HR está casi ausente en la fase G1 , es más activa en la fase S y disminuye en G 2 /M. [55] La unión de extremos no homólogos , un proceso menos preciso y más mutagénico para reparar roturas de doble cadena, está activa durante todo el ciclo celular.
Evolución del ciclo celular
Evolución del genoma
El ciclo celular debe duplicar todos los constituyentes celulares y repartirlos equitativamente en dos células hijas. Muchos constituyentes, como las proteínas y los ribosomas , se producen continuamente durante todo el ciclo celular (excepto durante la fase M ). Sin embargo, los cromosomas y otros elementos asociados, como los MTOC , se duplican solo una vez durante el ciclo celular. Un componente central del ciclo celular es su capacidad para coordinar las duplicaciones continuas y periódicas de diferentes elementos celulares, que evolucionaron con la formación del genoma.
El entorno precelular contenía ARN funcionales y autorreplicantes . [56] Todas las concentraciones de ARN dependían de las concentraciones de otros ARN que podrían estar ayudando o dificultando la recolección de recursos. En este entorno, el crecimiento era simplemente la producción continua de ARN. Estas estructuras precelulares habrían tenido que lidiar con ARN parásitos, problemas de herencia y control del número de copias de ARN específicos. [56] [57]
La separación del ARN "genómico" del ARN "funcional" ayudó a resolver estos problemas. [58] La fusión de múltiples ARN en un genoma proporcionó una plantilla a partir de la cual se escindieron los ARN funcionales. Ahora, los ARN parásitos tendrían que incorporarse al genoma, una barrera mucho mayor, para sobrevivir. El control del número de copias del ARN genómico también permitió que la concentración de ARN se determinara a través de las tasas de síntesis y las vidas medias del ARN, en lugar de la competencia. [56] Separar la duplicación de ARN genómicos de la generación de ARN funcionales permitió una fidelidad de duplicación mucho mayor de los ARN genómicos sin comprometer la producción de ARN funcionales. Finalmente, la sustitución del ARN genómico por ADN , que es una molécula más estable, permitió genomas más grandes. La transición de la síntesis enzimática por autocatálisis a la síntesis enzimática dirigida por el genoma fue un paso crítico en la evolución celular y tuvo implicaciones duraderas en el ciclo celular, que debe regular la síntesis funcional y la duplicación genómica de maneras muy diferentes. [56]
Quinasa dependiente de ciclina y evolución de la ciclina
La progresión del ciclo celular está controlada por las concentraciones oscilantes de diferentes ciclinas y las interacciones moleculares resultantes de las diversas quinasas dependientes de ciclina (CDK). En la levadura, solo una CDK (Cdc28 en S. cerevisiae y Cdc2 en S. pombe ) controla el ciclo celular. [59] Sin embargo, en animales, han evolucionado familias completas de CDK. [60] [61] Cdk1 controla la entrada a la mitosis y Cdk2, Cdk4 y Cdk6 regulan la entrada a la fase S. A pesar de la evolución de la familia CDK en animales, estas proteínas tienen funciones relacionadas o redundantes. [62] [63] [64] Por ejemplo, las células de ratones con triple knock out de cdk2, cdk4 y cdk6 aún pueden progresar a través del ciclo celular básico. [65] Los knockouts de cdk1 son letales, lo que sugiere una quinasa ancestral de tipo CDK1 que en última instancia controla el ciclo celular. [65]
Arabidopsis thaliana tiene un homólogo de Cdk1 llamado CDKA;1, sin embargo, los mutantes cdka;1 de A. thaliana aún son viables, [66] lo que va en contra del patrón opistoconto de las quinasas de tipo CDK1 como reguladores esenciales que controlan el ciclo celular. [67] Las plantas también tienen un grupo único de CDK de tipo B, cuyas funciones pueden variar desde funciones específicas del desarrollo hasta actores principales en la regulación mitótica. [68] [69]
Evolución del punto de control G1/S

El punto de control G1/S es el punto en el que la célula se compromete a dividirse a través del ciclo celular. Redes reguladoras complejas conducen a la decisión de transición G1/S. En los opistocontos, existen secuencias de proteínas altamente divergentes, así como topologías de red sorprendentemente similares. [67] [70]
La entrada en la fase S tanto en levaduras como en animales está controlada por los niveles de dos reguladores opuestos. [67] Las redes que regulan estos factores de transcripción son bucles de retroalimentación doblemente negativos y bucles de retroalimentación positiva tanto en levaduras como en animales. [67] [70] [71] La regulación adicional de la red reguladora para el punto de control G1/S en levaduras y animales incluye la fosforilación /desfosforilación de complejos CDK-ciclina. La suma de estas redes reguladoras crea un esquema histérico y biestable, a pesar de que las proteínas específicas están altamente divergentes. [72] [73] Para levaduras, Whi5 debe ser suprimido por la fosforilación de Cln3 para que se exprese SBF, [74] mientras que en animales Rb debe ser suprimido por el complejo Cdk4/6-ciclina D para que se exprese E2F . [75] Tanto Rb como Whi5 inhiben la transcripción a través del reclutamiento de proteínas histona desacetilasas a los promotores. [76] [77] Ambas proteínas tienen además múltiples sitios de fosforilación de CDK a través de los cuales son inhibidas. [78] [75] Sin embargo, estas proteínas no comparten ninguna similitud de secuencia.
Los estudios en A. thaliana amplían nuestro conocimiento de la transición G1/S en los eucariotas en su conjunto. Las plantas también comparten una serie de características de red conservadas con los opistocontos, y muchos reguladores vegetales tienen homólogos animales directos. [79] Por ejemplo, las plantas también necesitan suprimir Rb para la traducción de E2F en la red. [80] Estos elementos conservados de los ciclos celulares de plantas y animales pueden ser ancestrales en los eucariotas. Si bien la levadura comparte una topología de red conservada con las plantas y los animales, la naturaleza altamente divergente de los reguladores de la levadura sugiere una posible evolución rápida a lo largo del linaje de la levadura. [67]
Véase también
- Modelo celular
- Replicación del ADN eucariota
- Catástrofe mitótica
- Complejo de reconocimiento de origen
- Proteína del retinoblastoma
- Cultivo sincrónico : sincronización de cultivos celulares
- Nosotros 1
Referencias
- ^ Alberts B, Hopkin K, Johnson A, Morgan D, Raff M, Roberts K, Walter P (2019). Biología celular esencial (quinta edición). Nueva York Londres: WW Norton & Company. págs. 624–625. ISBN 9780393680393.
- ^ Wang JD, Levin PA (noviembre de 2009). "Metabolismo, crecimiento celular y ciclo celular bacteriano". Nature Reviews. Microbiology . 7 (11): 822–827. doi :10.1038/nrmicro2202. PMC 2887316 . PMID 19806155.
- ^ Cooper GM (2000). "Capítulo 14: El ciclo celular eucariota". La célula: un enfoque molecular (2.ª ed.). Washington, DC: ASM Press. ISBN 978-0-87893-106-4.
- ^ Smith JA, Martin L (abril de 1973). "¿Las células realizan ciclos?". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 70 (4): 1263–1267. Bibcode :1973PNAS...70.1263S. doi : 10.1073/pnas.70.4.1263 . PMC 433472 . PMID 4515625.
- ^ Wu RS, Bonner WM (diciembre de 1981). "Separación de la síntesis de histonas basales de la síntesis de histonas en fase S en células en división". Cell . 27 (2 Pt 1): 321–330. doi :10.1016/0092-8674(81)90415-3. PMID 7199388. S2CID 12215040.
- ^ Nelson DM, Ye X, Hall C, Santos H, Ma T, Kao GD, et al. (noviembre de 2002). "Acoplamiento de la síntesis de ADN y la síntesis de histonas en la fase S independiente de la actividad de ciclina/cdk2". Biología molecular y celular . 22 (21): 7459–7472. doi :10.1128/MCB.22.21.7459-7472.2002. PMC 135676 . PMID 12370293.
- ^ Cameron IL, Greulich RC (julio de 1963). "Evidencia de una duración esencialmente constante de la síntesis de ADN en la renovación de los epitelios del ratón adulto". The Journal of Cell Biology . 18 (1): 31–40. doi :10.1083/jcb.18.1.31. PMC 2106275 . PMID 14018040.
- ^ Rubenstein I, Wick SM (2008). «Célula». Centro de referencia en línea del libro mundial . Archivado desde el original el 30 de mayo de 2011. Consultado el 10 de julio de 2009 .
- ^ Maton A, Lahart D, Hopkins J, Warner MQ, Johnson S, Wright JD (1997). Células: elementos básicos de la vida. Nueva Jersey: Prentice Hall. pp. 70–4. ISBN 978-0-13-423476-2.
- ^ De Souza CP, Osmani SA (septiembre de 2007). "Mitosis, no sólo abierta o cerrada". Eukaryotic Cell . 6 (9): 1521–1527. doi :10.1128/EC.00178-07. PMC 2043359 . PMID 17660363.
- ^ Lilly MA, Duronio RJ (abril de 2005). "Nuevos conocimientos sobre el control del ciclo celular a partir del endociclo de Drosophila". Oncogene . 24 (17): 2765–2775. doi :10.1038/sj.onc.1208610. PMID 15838513. S2CID 25473573.
- ^ Nigg EA (junio de 1995). "Proteínas quinasas dependientes de ciclina: reguladores clave del ciclo celular eucariota". BioEssays . 17 (6): 471–480. doi :10.1002/bies.950170603. PMID 7575488. S2CID 44307473.
- ^ "El Premio Nobel de Fisiología o Medicina 2001 – Nota de prensa". Nobelprize.org.
- ^ ab Spellman PT, Sherlock G, Zhang MQ, Iyer VR, Anders K, Eisen MB, et al. (diciembre de 1998). "Identificación completa de genes regulados por el ciclo celular de la levadura Saccharomyces cerevisiae mediante hibridación de microarrays". Biología molecular de la célula . 9 (12): 3273–3297. doi :10.1091/mbc.9.12.3273. PMC 25624 . PMID 9843569.
- ^ abcd Robbins SL, Cotran RS (2004). Kumar V, Abbas AK, Fausto N (eds.). Bases patológicas de la enfermedad . Elsevier . ISBN 978-81-8147-528-2.
- ^ ab Dong P, Maddali MV, Srimani JK, Thélot F, Nevins JR, Mathey-Prevot B, You L (septiembre de 2014). "División del trabajo entre las ciclinas Myc y G1 en el compromiso del ciclo celular y el control del ritmo". Nature Communications . 5 : 4750. Bibcode :2014NatCo...5.4750D. doi :10.1038/ncomms5750. PMC 4164785 . PMID 25175461.
- ^ Mahmoudi M, Azadmanesh K, Shokrgozar MA, Journeay WS, Laurent S (mayo de 2011). "Efecto de las nanopartículas en el ciclo de vida celular". Chemical Reviews . 111 (5): 3407–3432. doi :10.1021/cr1003166. PMID 21401073.
- ^ Goel S, DeCristo MJ, McAllister SS, Zhao JJ (noviembre de 2018). "Inhibición de CDK4/6 en el cáncer: más allá de la detención del ciclo celular". Tendencias en biología celular . 28 (11): 911–925. doi :10.1016/j.tcb.2018.07.002. PMC 6689321 . PMID 30061045.
- ^ Burkhart DL, Sage J (septiembre de 2008). "Mecanismos celulares de supresión tumoral por el gen del retinoblastoma". Nature Reviews. Cancer . 8 (9): 671–682. doi :10.1038/nrc2399. PMC 6996492 . PMID 18650841.
- ^ Morgan DO (2007). El ciclo celular: principios de control . Londres: New Science Press. ISBN 978-0-19-920610-0.OCLC 70173205 .
- ^ Paternot S, Bockstaele L, Bisteau X, Kooken H, Coulonval K, Roger PP (febrero de 2010). "Inactivación de Rb en el ciclo celular y el cáncer: el rompecabezas de la fosforilación activadora altamente regulada de CDK4 frente a la quinasa activadora de CDK constitutivamente activa". Ciclo celular . 9 (4): 689–699. doi : 10.4161/cc.9.4.10611 . PMID 20107323.
- ^ Henley SA, Dick FA (marzo de 2012). "La familia de proteínas del retinoblastoma y sus funciones reguladoras en el ciclo de división celular de los mamíferos". División celular . 7 (1): 10. doi : 10.1186/1747-1028-7-10 . PMC 3325851 . PMID 22417103.
- ^ abc Narasimha AM, Kaulich M, Shapiro GS, Choi YJ, Sicinski P, Dowdy SF (junio de 2014). "La ciclina D activa el supresor tumoral Rb mediante monofosforilación". eLife . 3 : e02872. doi : 10.7554/eLife.02872 . PMC 4076869 . PMID 24876129.
- ^ Morris EJ, Dyson NJ (1 de enero de 2001). Socios proteicos del retinoblastoma. Avances en la investigación del cáncer. Vol. 82. Academic Press. págs. 1–54. doi :10.1016/s0065-230x(01)82001-7. ISBN 9780120066827. Número de identificación personal 11447760.
- ^ Dyson NJ (julio de 2016). "RB1: un prototipo de supresor tumoral y un enigma". Genes & Development . 30 (13): 1492–1502. doi :10.1101/gad.282145.116. PMC 4949322 . PMID 27401552.
- ^ ab Sanidas I, Morris R, Fella KA, Rumde PH, Boukhali M, Tai EC, et al. (Marzo de 2019). "Un código de monofosforilación modula la función de RB". Célula molecular . 73 (5): 985–1000.e6. doi :10.1016/j.molcel.2019.01.004. PMC 6424368 . PMID 30711375.
- ^ ab Topacio BR, Zatulovskiy E, Cristea S, Xie S, Tambo CS, Rubin SM, et al. (mayo de 2019). "La ciclina D-Cdk4,6 impulsa la progresión del ciclo celular a través de la hélice C-terminal de la proteína del retinoblastoma". Molecular Cell . 74 (4): 758–770.e4. doi :10.1016/j.molcel.2019.03.020. PMC 6800134 . PMID 30982746.
- ^ Norbury C (1995). "Cdk2 protein kinase (vertebrates)". En Hardie DG, Hanks S (eds.). Datos sobre la proteína quinasa . Boston: Academic Press. pp. 184. ISBN 978-0-12-324719-3.
- ^ "Presentación sobre las FOSFATASAS CDC25: un objetivo potencial para nuevos agentes anticancerígenos". Archivado desde el original el 3 de marzo de 2016 . Consultado el 11 de marzo de 2010 .
- ^ Sherr CJ, Beach D, Shapiro GI (abril de 2016). "Dirigir CDK4 y CDK6: del descubrimiento a la terapia". Cancer Discovery . 6 (4): 353–367. doi :10.1158/2159-8290.cd-15-0894. PMC 4821753 . PMID 26658964.
- ^ O'Leary B, Finn RS, Turner NC (julio de 2016). "Tratamiento del cáncer con inhibidores selectivos de CDK4/6". Nature Reviews. Oncología clínica . 13 (7): 417–430. doi :10.1038/nrclinonc.2016.26. PMID 27030077. S2CID 23646632.
- ^ Bilgin B, Sendur MA, Şener Dede D, Akıncı MB, Yalçın B (septiembre de 2017). "Una revisión actual y completa de los inhibidores de la quinasa dependiente de ciclina para el tratamiento del cáncer de mama metastásico". Investigación médica actual y opinión . 33 (9): 1559–1569. doi :10.1080/03007995.2017.1348344. PMID 28657360. S2CID 205542255.
- ^ Schmidt M, Sebastian M (agosto de 2018). "Palbociclib: el primero de una nueva clase de inhibidores del ciclo celular". Small Molecules in Oncology . Recent Results in Cancer Research. Vol. 211. págs. 153–175. doi :10.1007/978-3-319-91442-8_11. ISBN 978-3-319-91441-1. Número PMID 30069766.
- ^ ab Pramila T, Wu W, Miles S, Noble WS, Breeden LL (agosto de 2006). "El factor de transcripción Forkhead Hcm1 regula los genes de segregación cromosómica y llena el vacío de la fase S en el circuito transcripcional del ciclo celular". Genes & Development . 20 (16): 2266–2278. doi :10.1101/gad.1450606. PMC 1553209 . PMID 16912276.
- ^ abc Orlando DA, Lin CY, Bernard A, Wang JY, Socolar JE, Iversen ES, et al. (junio de 2008). "Control global de la transcripción del ciclo celular mediante osciladores de red y CDK acoplados". Nature . 453 (7197): 944–947. Bibcode :2008Natur.453..944O. doi :10.1038/nature06955. PMC 2736871 . PMID 18463633.
- ^ de Lichtenberg U, Jensen LJ, Fausbøll A, Jensen TS, Bork P, Brunak S (abril de 2005). "Comparación de métodos computacionales para la identificación de genes regulados por el ciclo celular". Bioinformática . 21 (7): 1164–1171. doi : 10.1093/bioinformatics/bti093 . PMID 15513999.
- ^ ab White MA, Riles L, Cohen BA (febrero de 2009). "Un análisis sistemático de los reguladores transcripcionales del ciclo celular de la levadura". Genética . 181 (2): 435–446. doi :10.1534/genetics.108.098145. PMC 2644938 . PMID 19033152.
- ^ Lee TI, Rinaldi NJ, Robert F, Odom DT, Bar-Joseph Z, Gerber GK, et al. (octubre de 2002). "Redes reguladoras de la transcripción en Saccharomyces cerevisiae". Science . 298 (5594): 799–804. Bibcode :2002Sci...298..799L. doi :10.1126/science.1075090. PMID 12399584. S2CID 4841222.
- ^ Simon I, Barnett J, Hannett N, Harbison CT, Rinaldi NJ, Volkert TL, et al. (septiembre de 2001). "Regulación serial de reguladores transcripcionales en el ciclo celular de la levadura". Cell . 106 (6): 697–708. doi : 10.1016/S0092-8674(01)00494-9 . PMID 11572776. S2CID 9308235.
- ^ Sidorova JM, Mikesell GE, Breeden LL (diciembre de 1995). "La fosforilación regulada por el ciclo celular de Swi6 controla su localización nuclear". Biología molecular de la célula . 6 (12): 1641–1658. doi :10.1091/mbc.6.12.1641. PMC 301322 . PMID 8590795.
- ^ Ubersax JA, Woodbury EL, Quang PN, Paraz M, Blethrow JD, Shah K, et al. (octubre de 2003). "Objetivos de la quinasa dependiente de ciclina Cdk1". Nature . 425 (6960): 859–864. Bibcode :2003Natur.425..859U. doi :10.1038/nature02062. PMID 14574415. S2CID 4391711.
- ^ Morgan DO (2007). "2–3". El ciclo celular: principios de control . Londres: New Science Press. pág. 18. ISBN 978-0-9539181-2-6.
- ^ Omberg L, Meyerson JR, Kobayashi K, Drury LS, Diffley JF, Alter O (octubre de 2009). "Efectos globales de la replicación del ADN y la actividad del origen de la replicación del ADN en la expresión génica eucariota". Biología de sistemas moleculares . 5 : 312. doi :10.1038/msb.2009.70. PMC 2779084 . PMID 19888207.
- ^ Alter O, Golub GH, Brown PO, Botstein D (febrero de 2004). Deutscher MP, Black S, Boehmer PE, D'Urso G, Fletcher TM, Huijing F, et al. (eds.). Novel Genome-Scale Correlation between DNA Replication and RNA Transcription During the Cell Cycle in Yeast is Predicted by Data-Driven Models (PDF) (La correlación a escala genómica novedosa entre la replicación del ADN y la transcripción del ARN durante el ciclo celular en levaduras se predice mediante modelos basados en datos) . Simposio de invierno de biotecnología de Miami Nature. Ciclo celular, cromosomas y cáncer. Vol. 15. Miami Beach, FL: Facultad de Medicina de la Universidad de Miami. Archivado desde el original (PDF) el 9 de septiembre de 2014 . Consultado el 7 de febrero de 2014 .
- ^ Alter O, Golub GH (noviembre de 2004). "El análisis integrativo de datos a escala del genoma mediante proyección pseudoinversa predice una nueva correlación entre la replicación del ADN y la transcripción del ARN". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 (47): 16577–16582. Bibcode :2004PNAS..10116577A. doi : 10.1073/pnas.0406767101 . PMC 534520 . PMID 15545604.
- ^ Omberg L, Golub GH, Alter O (noviembre de 2007). "Una descomposición en valores singulares de orden superior de tensor para el análisis integrador de datos de microarrays de ADN de diferentes estudios". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (47): 18371–18376. Bibcode :2007PNAS..10418371O. doi : 10.1073/pnas.0709146104 . PMC 2147680 . PMID 18003902.
- ^ Elledge SJ (diciembre de 1996). "Puntos de control del ciclo celular: prevención de una crisis de identidad". Science . 274 (5293): 1664–1672. Bibcode :1996Sci...274.1664E. doi :10.1126/science.274.5293.1664. PMID 8939848. S2CID 39235426.
- ^ ab Vilenchik MM, Knudson AG (octubre de 2003). "Roturas endógenas de doble cadena de ADN: producción, fidelidad de reparación e inducción de cáncer". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (22): 12871–12876. Bibcode :2003PNAS..10012871V. doi : 10.1073/pnas.2135498100 . PMC 240711 . PMID 14566050.
- ^ LeMaire-Adkins R, Radke K, Hunt PA (diciembre de 1997). "Falta de control de puntos de control en la transición metafase/anafase: un mecanismo de no disyunción meiótica en hembras de mamíferos". The Journal of Cell Biology . 139 (7): 1611–1619. doi :10.1083/jcb.139.7.1611. PMC 2132649 . PMID 9412457.
- ^ Sakaue-Sawano A, Kurokawa H, Morimura T, Hanyu A, Hama H, Osawa H, et al. (febrero de 2008). "Visualización de la dinámica espaciotemporal de la progresión del ciclo celular multicelular". Celúla . 132 (3): 487–498. doi : 10.1016/j.cell.2007.12.033 . PMID 18267078. S2CID 15704902.
- ^ Rodríguez EA, Tran GN, Gross LA, Crisp JL, Shu X, Lin JY, Tsien RY (septiembre de 2016). "Una proteína fluorescente de color rojo lejano evolucionó a partir de una ficobiliproteína cianobacteriana". Nature Methods . 13 (9): 763–769. doi :10.1038/nmeth.3935. PMC 5007177 . PMID 27479328.
- ^ Salmenov R, Mummery C, ter Huurne M (octubre de 2024). "Herramientas de visualización del ciclo celular para estudiar la proliferación de cardiomiocitos en tiempo real". Open Biology . 14 (10): 240167. doi :10.1098/rsob.240167. PMID 39378987.
- ^ Champeris Tsaniras S, Kanellakis N, Symeonidou IE, Nikolopoulou P, Lygerou Z, Taraviras S (junio de 2014). "Licencias para la replicación del ADN, el cáncer, la pluripotencia y la diferenciación: ¿un mundo interconectado?". Seminarios en biología celular y del desarrollo . 30 : 174–180. doi : 10.1016/j.semcdb.2014.03.013 . PMID 24641889.
- ^ Baserga R (junio de 1965). "La relación del ciclo celular con el crecimiento tumoral y el control de la división celular: una revisión". Investigación sobre el cáncer . 25 (5): 581–595. PMID 14347544.
- ^ Mao Z, Bozzella M, Seluanov A, Gorbunova V (septiembre de 2008). "Reparación del ADN mediante unión de extremos no homólogos y recombinación homóloga durante el ciclo celular en células humanas". Cell Cycle . 7 (18): 2902–2906. doi :10.4161/cc.7.18.6679. PMC 2754209 . PMID 18769152.
- ^ abcd Nasmyth K (septiembre de 1995). "Evolución del ciclo celular". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 349 (1329): 271–281. doi :10.1098/rstb.1995.0113. PMID 8577838.
- ^ Cavalier-Smith T (julio de 1987). "El origen de las células eucariotas y arqueobacterianas". Anales de la Academia de Ciencias de Nueva York . 503 (1): 17–54. Bibcode :1987NYASA.503...17C. doi :10.1111/j.1749-6632.1987.tb40596.x. PMID 3113314. S2CID 38405158.
- ^ Maizels N, Weiner AM (julio de 1994). "Filogenia a partir de la función: evidencia del registro fósil molecular de que el ARNt se originó en la replicación, no en la traducción". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (15): 6729–6734. Bibcode :1994PNAS...91.6729M. doi : 10.1073/pnas.91.15.6729 . PMC 44276 . PMID 8041690.
- ^ Morgan DO (noviembre de 1997). "Quinasas dependientes de ciclina: motores, relojes y microprocesadores". Revisión anual de biología celular y del desarrollo . 13 (1): 261–291. doi :10.1146/annurev.cellbio.13.1.261. PMID 9442875.
- ^ Malumbres M, Harlow E, Hunt T, Hunter T, Lahti JM, Manning G, et al. (noviembre de 2009). "Quinasas dependientes de ciclina: un retrato de familia". Nature Cell Biology . 11 (11): 1275–1276. doi :10.1038/ncb1109-1275. PMC 2914104 . PMID 19884882.
- ^ Satyanarayana A, Kaldis P (agosto de 2009). "Regulación del ciclo celular en mamíferos: varias Cdk, numerosas ciclinas y diversos mecanismos compensatorios". Oncogene . 28 (33): 2925–2939. doi :10.1038/onc.2009.170. PMID 19561645. S2CID 3096776.
- ^ Barrière C, Santamaría D, Cerqueira A, Galán J, Martín A, Ortega S, et al. (junio de 2007). "Los ratones prosperan sin Cdk4 y Cdk2". Oncología Molecular . 1 (1): 72–83. doi :10.1016/j.molonc.2007.03.001. PMC 5543859 . PMID 19383288.
- ^ Ortega S, Prieto I, Odajima J, Martín A, Dubus P, Sotillo R, et al. (Septiembre de 2003). "La quinasa 2 dependiente de ciclina es esencial para la meiosis pero no para la división celular mitótica en ratones". Genética de la Naturaleza . 35 (1): 25–31. doi :10.1038/ng1232. PMID 12923533. S2CID 19522248.
- ^ Aleem E, Kiyokawa H, Kaldis P (agosto de 2005). "Los complejos Cdc2-ciclina E regulan la transición de fase G1/S". Biología celular de la naturaleza . 7 (8): 831–836. doi :10.1038/ncb1284. PMID 16007079. S2CID 10842071.
- ^ ab Santamaría D, Barrière C, Cerqueira A, Hunt S, Tardy C, Newton K, et al. (agosto de 2007). "Cdk1 es suficiente para impulsar el ciclo celular de los mamíferos". Nature . 448 (7155): 811–815. Bibcode :2007Natur.448..811S. doi :10.1038/nature06046. PMID 17700700. S2CID 4412772.
- ^ Nowack MK, Harashima H, Dissmeyer N, Zhao X, Bouyer D, Weimer AK y col. (mayo de 2012). "Marco genético de la función quinasa dependiente de ciclina en Arabidopsis". Célula del desarrollo . 22 (5): 1030-1040. doi : 10.1016/j.devcel.2012.02.015 . PMID 22595674.
- ^ abcdef Harashima H, Dissmeyer N, Schnittger A (julio de 2013). "Control del ciclo celular en el reino eucariota". Tendencias en biología celular . 23 (7): 345–356. doi :10.1016/j.tcb.2013.03.002. PMID 23566594.
- ^ Boudolf V, Barrôco R, Engler J, Verkest A, Beeckman T, Naudts M, et al. (abril de 2004). "Las quinasas dependientes de ciclina de tipo B1 son esenciales para la formación de complejos estomáticos en Arabidopsis thaliana". The Plant Cell . 16 (4): 945–955. doi : 10.1105/tpc.021774 . PMC 412868 . PMID 15031414.
- ^ Andersen SU, Buechel S, Zhao Z, Ljung K, Novák O, Busch W, et al. (enero de 2008). "Requisitos de quinasas dependientes de ciclina de tipo B2 para la integridad del meristemo en Arabidopsis thaliana". The Plant Cell . 20 (1): 88–100. doi :10.1105/tpc.107.054676. PMC 2254925 . PMID 18223038.
- ^ ab Cross FR, Buchler NE, Skotheim JM (diciembre de 2011). "Evolución de redes y secuencias en el control del ciclo celular eucariota". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 366 (1584): 3532–3544. doi :10.1098/rstb.2011.0078. PMC 3203458 . PMID 22084380.
- ^ Skotheim JM, Di Talia S, Siggia ED, Cross FR (julio de 2008). "La retroalimentación positiva de las ciclinas G1 asegura una entrada coherente al ciclo celular". Nature . 454 (7202): 291–296. Bibcode :2008Natur.454..291S. doi :10.1038/nature07118. PMC 2606905 . PMID 18633409.
- ^ Ferrell JE (abril de 2002). "Estados autoperpetuantes en la transducción de señales: retroalimentación positiva, retroalimentación doblemente negativa y biestabilidad". Current Opinion in Cell Biology . 14 (2): 140–148. doi :10.1016/S0955-0674(02)00314-9. PMID 11891111.
- ^ Venta R, Valk E, Kõivomägi M, Loog M (2012). "La retroalimentación doblemente negativa entre la ciclina-CDK de la fase S y la CKI genera brusquedad en el cambio G1/S". Frontiers in Physiology . 3 : 459. doi : 10.3389/fphys.2012.00459 . PMC 3515773 . PMID 23230424.
- ^ Eser U, Falleur-Fettig M, Johnson A, Skotheim JM (agosto de 2011). "El compromiso con una transición celular precede al cambio transcripcional en todo el genoma". Molecular Cell . 43 (4): 515–527. doi :10.1016/j.molcel.2011.06.024. PMC 3160620 . PMID 21855792.
- ^ ab Narasimha AM, Kaulich M, Shapiro GS, Choi YJ, Sicinski P, Dowdy SF (junio de 2014). Davis R (ed.). "La ciclina D activa el supresor tumoral Rb mediante monofosforilación". eLife . 3 : e02872. doi : 10.7554/eLife.02872 . PMC 4076869 . PMID 24876129.
- ^ Harbour JW, Luo RX, Dei Santi A, Postigo AA, Dean DC (septiembre de 1999). "La fosforilación de CDK desencadena interacciones intramoleculares secuenciales que bloquean progresivamente las funciones de Rb a medida que las células pasan por G1". Cell . 98 (6): 859–869. doi : 10.1016/s0092-8674(00)81519-6 . PMID 10499802.
- ^ Takahata S, Yu Y, Stillman DJ (noviembre de 2009). "El análogo funcional E2F SBF recluta el Rpd3 (L) HDAC, a través de Whi5 y Stb1, y el reorganizador de cromatina FACT, para los promotores de ciclina G1 de levadura". La Revista EMBO . 28 (21): 3378–3389. doi :10.1038/emboj.2009.270. PMC 2776103 . PMID 19745812.
- ^ de Bruin RA, McDonald WH, Kalashnikova TI, Yates J, Wittenberg C (junio de 2004). "Cln3 activa la transcripción específica de G1 a través de la fosforilación del represor Whi5 unido a SBF". Cell . 117 (7): 887–898. doi : 10.1016/j.cell.2004.05.025 . PMID 15210110.
- ^ Zhao X, Harashima H, Dissmeyer N, Pusch S, Weimer AK, Bramsiepe J, et al. (2 de agosto de 2012). Palanivelu R (ed.). "Un módulo general de control del ciclo celular en fase G1/S en la planta con flores Arabidopsis thaliana". PLOS Genetics . 8 (8): e1002847. doi : 10.1371/journal.pgen.1002847 . PMC 3410867 . PMID 22879821.
- ^ Weimer AK, Nowack MK, Bouyer D, Zhao X, Harashima H, Naseer S, et al. (octubre de 2012). "El retinoblastoma relacionado1 regula las divisiones celulares asimétricas en Arabidopsis". La célula vegetal . 24 (10): 4083–4095. doi :10.1105/tpc.112.104620. PMC 3517237 . PMID 23104828.
Lectura adicional
- Morgan DO (2007). El ciclo celular: principios de control . Londres: Publicado por New Science Press en asociación con Oxford University Press. ISBN 978-0-87893-508-6.
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). "Capítulo 17". Biología molecular de la célula (5.ª ed.). Nueva York: Garland Science. ISBN 978-0-8153-4111-6.
- Krieger M, Scott MP, Matsudaira PT, Lodish HF, Darnell JE, Zipursky L, Kaiser C, Berk A (2004). Biología celular molecular. Nueva York: WH Freeman and CO. ISBN 978-0-7167-4366-8.
- Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). "Capítulo 7". Biología molecular del gen (5.ª ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-4642-8.
Enlaces externos
 Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.
Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.- Seminario de David Morgan: Control del ciclo celular
- El ciclo celular y la muerte celular Archivado el 30 de octubre de 2018 en Wayback Machine.
- Programa transcripcional del ciclo celular: cronometraje de alta resolución
- Transcripción regulada por el ciclo celular y el ciclo metabólico en levaduras
- Animación del ciclo celular 1Lec.com
- Ciclo celular
- Fucci: Utilizando GFP para visualizar el ciclo celular
- Descripción general del ciclo celular de Science Creative Quarterly
- KEGG – Ciclo celular humano Archivado el 3 de noviembre de 2008 en Wayback Machine



