Repetición de Ankyrin
This article possibly contains original research. (July 2015) |
| Dominio de repetición de anquirina | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
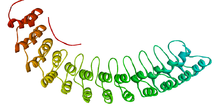 Diagrama de cinta de un fragmento del dominio de unión a la membrana de la anquirina R. [ 1] | |||||||||||
| Identificadores | |||||||||||
| Símbolo | Ank | ||||||||||
| Pfam | PF00023 | ||||||||||
| Interprofesional | IPR002110 | ||||||||||
| ELEGANTE | SM00248 | ||||||||||
| PROSITIO | PDOC50088 | ||||||||||
| SCOP2 | 1awc / ALCANCE / SUPFAM | ||||||||||
| |||||||||||
La repetición de anquirina es un motivo de 33 residuos en proteínas que consiste en dos hélices alfa separadas por bucles , descubierto por primera vez en proteínas de señalización en levadura Cdc10 y Drosophila Notch . Los dominios que consisten en repeticiones en tándem de anquirina median las interacciones proteína-proteína y se encuentran entre los motivos estructurales más comunes en las proteínas conocidas. Aparecen en proteínas bacterianas , arqueales y eucariotas , pero son mucho más comunes en eucariotas. Las proteínas de repetición de anquirina, aunque ausentes en la mayoría de los virus, son comunes entre los poxvirus . La mayoría de las proteínas que contienen el motivo tienen de cuatro a seis repeticiones, aunque su homónimo anquirina contiene 24, y el mayor número conocido de repeticiones es 34, predicho en una proteína expresada por Giardia lamblia . [2]
Las repeticiones de anquirina generalmente se pliegan juntas para formar una única estructura de solenoide lineal llamada dominios de repetición de anquirina . Estos dominios son una de las plataformas de interacción proteína-proteína más comunes en la naturaleza. Se encuentran en una gran cantidad de proteínas funcionalmente diversas, principalmente de eucariotas . Los pocos ejemplos conocidos de procariotas y virus pueden ser el resultado de transferencias horizontales de genes. [3] La repetición se ha encontrado en proteínas de diversas funciones, como iniciadores de la transcripción, reguladores del ciclo celular , citoesqueleto , transportadores de iones y transductores de señales . El pliegue de anquirina parece definirse por su estructura en lugar de su función, ya que no hay una secuencia o estructura específica que sea universalmente reconocida por él.
Teniendo en cuenta las estructuras atómicas de las repeticiones de anquirina individuales, el bucle es a menudo un bucle de protuberancia beta tipo 1 , mientras que ambas hélices alfa comúnmente tienen un bucle de Schellman en su extremo N.
Papel en el plegamiento de proteínas
El motivo de secuencia de repetición de anquirina se ha estudiado utilizando alineamiento de secuencias múltiples para determinar residuos de aminoácidos conservados críticos para el plegamiento y la estabilidad. Los residuos en la amplia superficie lateral de las estructuras de repetición de anquirina son variables, a menudo hidrófobos e involucrados principalmente en la mediación de interacciones proteína-proteína. Se ha sintetizado un diseño de proteína artificial basado en una secuencia de consenso derivada del alineamiento de secuencias y se ha descubierto que se pliega de manera estable, lo que representa la primera proteína diseñada con múltiples repeticiones. [4] Estrategias de diseño más extensas han utilizado secuencias combinatorias para "evolucionar" repeticiones de anquirina que reconocen dianas proteicas particulares, una técnica que se ha presentado como una alternativa al diseño de anticuerpos para aplicaciones que requieren una unión de alta afinidad. [5] Un estudio basado en la estructura que involucra una variedad de proteínas anquirina de estructuras conocidas, muestra que las proteínas anquirina basadas en el consenso son muy estables ya que maximizan la brecha energética entre las estructuras de plegamiento y desplegado, codificando una red densamente conectada de interacciones favorables entre motivos de secuencia conservados, como el motivo TPLX. [6] El mismo estudio muestra que las inserciones en el marco canónico de las repeticiones de anquirina se enriquecen con interacciones conflictivas, que están relacionadas con la función. Lo mismo se aplica a las interacciones que rodean los puntos calientes de deleción. Estas podrían estar relacionadas con transiciones complejas de plegamiento/desplegamiento que son importantes para el reconocimiento y la interacción de los socios.
Las proteínas con repeticiones de anquirina presentan un problema inusual en el estudio del plegamiento de proteínas , que se ha centrado en gran medida en las proteínas globulares que forman una estructura terciaria bien definida estabilizada por contactos residuo-residuo no locales de largo alcance . Las repeticiones de anquirina, por el contrario, contienen muy pocos de estos contactos (es decir, tienen un orden de contacto bajo ). La mayoría de los estudios han encontrado que las repeticiones de anquirina se pliegan en un mecanismo de plegamiento de dos estados, lo que sugiere un alto grado de cooperatividad de plegamiento a pesar de los contactos locales entre residuos y la evidente necesidad de un plegamiento exitoso con números variables de repeticiones. Algunas evidencias, basadas en la síntesis de versiones truncadas de proteínas de repetición naturales, [7] y en el examen de los valores phi , [8] sugieren que el extremo C forma el sitio de nucleación de plegamiento.
Importancia clínica
Las proteínas de repetición de anquirina se han asociado con una serie de enfermedades humanas . Estas proteínas incluyen el inhibidor del ciclo celular p16 , que está asociado con el cáncer , y la proteína Notch (un componente clave de las vías de señalización celular) que puede causar el trastorno neurológico CADASIL cuando el dominio de repetición se ve alterado por mutaciones. [2]
Una familia especializada de proteínas anquirina, conocidas como proteínas de repetición de anquirina muscular (MARP), participan en la reparación y regeneración del tejido muscular después de daños causados por lesiones y estrés. [9]
A una variación natural entre la glutamina y la lisina en la posición 703 en la undécima repetición de anquirina de ANKK1 , conocida como el alelo TaqI A1, [10] se le ha atribuido el fomento de conductas adictivas como la obesidad, el alcoholismo, la dependencia de la nicotina y el estilo de amor Eros [ cita requerida ] al tiempo que desalienta la delincuencia juvenil y el neuroticismo-ansiedad. [11] [ verificación fallida ] La variación puede afectar la especificidad de las interacciones proteicas realizadas por la proteína quinasa ANKK1 a través de esta repetición [ cita requerida ] .
Proteínas humanas que contienen esta repetición
ABTB1 ; ABTB2; ACBD6; ACTBL1; ANK1 ; ANK2 ; ANK3 ; ANKAR; ANKDD1A; ANKEF1; ANKFY1 ; ANKHD1 ; ANKIB1; ANKK1 ; ANKMY1 ; ANKMY2; ANKRA2; ANKRD1 ; ANKRD10; ANKRD11 ; ANKRD12; ANKRD13; ANKRD13A; ANKRD13B; ANKRD13C ; ANKRD13D; ANKRD15 ; ANKRD16; ANKRD17 ; ANKRD18A; ANKRD18B; ANKRD19; ANKRD2 ; ANKRD20A1; ANKRD20A2; ANKRD20A3; ANKRD20A4; ANKRD21; ANKRD22; ANKRD23 ; ANKRD24; ANKRD25; ANKRD26 ; ANKRD27 ; ANKRD28 ; ANKRD30A; ANKRD30B; ANKRD30BL; ANKRD32; ANKRD33; ANKRD35; ANKRD36; ANKRD36B ; ANKRD37 ; ANKRD38; ANKRD39; ANKRD40; ANKRD41; ANKRD42; ANKRD43; ANKRD44; ANKRD45; ANKRD46; ANKRD47; ANKRD49 ; ANKRD50; ANKRD52; ANKRD53; ANKRD54; ANKRD55; ANKRD56; ANKRD57; ANKRD58; ANKRD60; ANKRD6; ANKRD7; ANKRD9; ANKS1A ; ASB1; ASB2; ASB3 ; ASB4 ; ASB5 ; ASB6 ; ASB7 ; ASB8 ; ASB9; ASZ1 ; BARD1 ; BAT4 ; BAT8 ; BCL3 ; BCOR ; BCORL1; BTBD11; CAMTA1 ; CAMTA2; CASKIN1; CASKIN2; CCM1 ; CDKN2A ; CDKN2B ; CDKN2C ; CDKN2D ; CENTB1 ; CENTB2 ; CENTB5; CENTG1 ; CENTG2 ; CENTG3 ; CLIP3; CLIP4 ; CLPB ; CTGLF1; CTGLF2; CTGLF3; CTGLF4; CTGLF5; CTTNBP2 ; DAPK1 ; DDEF1 ; DDEF2 ; DDEFL1; DGKI; DGKZ ; DP58; DYSFIP1; DZANK; EHMT1 ; EHMT2 ; ESPN ; FANK1; FEM1A ; FEM1B; GABPB2 ; GIT1 ; GIT2 ; GLS; GLS2 ; HACE1 ; HECTD1; IBTK; ILK ; INVS ; NIÑOS220 ; KRIT1 ; LRRK1 ; CORREO; MIB1 ; MIB2 ; MPHOSPH8 ; MTPN ; MYO16; NFKB1 ; NFKB2 ; NFKBIA ; NFKBIB ; NFKBIE ; NFKBIL1 ; NFKBIL2; MUESCA1 ; MUESCA2 ; MUESCA3 ; MUESCA4 ; NRARP ; NUDT12 ; OSBPL1A ; OSTF1 ; PLA2G6 ; POTE14; POTE15; POTE8; PPP1R12A ; PPP1R12B ; PPP1R12C ; PPP1R13B ; PPP1R13L ; PPP1R16A; PPP1R16B; PSMD10 ; RAI14 ; RFXANK ; RIPK4 ; RNASEL ; SHANK1 ; SHANK2 ; SHANK3 ; SNCAIP ; TA-NFKBH; TEX14 ; TNKS ; TNKS2 ; TNNI3K ; TP53BP2 ; TRP7; TRPA1 ; TRPC3 ; TRPC4 ; TRPC5 ; TRPC6 ; TRPC7 ; TRPV1 ; TRPV2 ; TRPV3 ; TRPV4 ; TRPV5 ; TRPV6 ; UACA ; USH1G ; ZDHHC13; ZDHHC17 ;
Véase también
- DARPin (proteína de repetición de anquirina diseñada), un anticuerpo mimético diseñado basado en la estructura de las repeticiones de anquirina
Referencias
- ^ PDB : 1N11 ; Michaely P, Tomchick DR, Machius M, Anderson RG (diciembre de 2002). "Estructura cristalina de una pila de 12 repeticiones ANK de ANK1 humano". EMBO J . 21 (23): 6387–96. doi :10.1093/emboj/cdf651. PMC 136955 . PMID 12456646.
- ^ ab Mosavi L, Cammett T, Desrosiers D, Peng Z (2004). "La repetición de anquirina como arquitectura molecular para el reconocimiento de proteínas". Protein Sci . 13 (6): 1435–48. doi :10.1110/ps.03554604. PMC 2279977 . PMID 15152081. Archivado desde el original el 7 de septiembre de 2004.
- ^ Bork P (diciembre de 1993). "Cientos de repeticiones similares a anquirina en proteínas funcionalmente diversas: ¿módulos móviles que cruzan filos horizontalmente?". Proteins . 17 (4): 363–74. doi :10.1002/prot.340170405. PMID 8108379. S2CID 35224626.
- ^ Mosavi LK, Minor DL, Peng ZY (diciembre de 2002). "Determinantes estructurales derivados del consenso del motivo de repetición de anquirina". Proc Natl Acad Sci USA . 99 (25): 16029–34. Bibcode :2002PNAS...9916029M. doi : 10.1073/pnas.252537899 . PMC 138559 . PMID 12461176.
- ^ Binz HK, Amstutz P, Kohl A, et al. (mayo de 2004). "Aglutinantes de alta afinidad seleccionados a partir de bibliotecas de proteínas con repetición de anquirina diseñadas". Nat. Biotechnol . 22 (5): 575–82. doi :10.1038/nbt962. PMID 15097997. S2CID 1191035.
- ^ Parra RG, Espada R, Verstraete N, Ferreiro DU, et al. (diciembre de 2015). "Caracterización estructural y energética de la familia de proteínas de repetición de anquirina". PLOS Comput. Biol . 12 (11): 575–82. Bibcode :2015PLSCB..11E4659P. doi : 10.1371/journal.pcbi.1004659 . PMC 4687027. PMID 26691182 .
- ^ Zhang B, Peng Z (junio de 2000). "Una unidad de plegamiento mínima en la proteína de repetición de anquirina p16(INK4)". J Mol Biol . 299 (4): 1121–32. doi :10.1006/jmbi.2000.3803. PMID 10843863.
- ^ Tang KS, Fersht AR, Itzhaki LS (enero de 2003). "Despliegue secuencial de repeticiones de anquirina en el supresor tumoral p16". Structure . 11 (1): 67–73. doi : 10.1016/S0969-2126(02)00929-2 . PMID 12517341.
- ^ Miller MK, Bang ML, Witt CC, et al. (noviembre de 2003). "Las proteínas de repetición de anquirina muscular: CARP, ankrd2/Arpp y DARP como una familia de moléculas de respuesta al estrés basadas en filamentos de titina". J Mol Biol . 333 (5): 951–64. doi :10.1016/j.jmb.2003.09.012. PMID 14583192.
- ^ Neville MJ, Johnstone EC, Walton RT (junio de 2004). "Identificación y caracterización de ANKK1: un nuevo gen de quinasa estrechamente vinculado a DRD2 en la banda cromosómica 11q23.1". Hum. Mutat . 23 (6): 540–5. doi : 10.1002/humu.20039 . PMID: 15146457. S2CID : 22242611.
- ^ "Resumen genético del NCBI para DRD2".(referencia provisional)
Enlaces externos
- Recurso de motivo lineal eucariota de clase LIG_TNKBM_1
- Ankyrin+repeat en los encabezamientos de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
