Señalización paracrina
En biología celular , la señalización paracrina es una forma de señalización celular , un tipo de comunicación celular en la que una célula produce una señal para inducir cambios en las células cercanas, alterando el comportamiento de esas células. Las moléculas de señalización conocidas como factores paracrinos se difunden en una distancia relativamente corta (acción local), a diferencia de la señalización celular por factores endocrinos , hormonas que viajan distancias considerablemente más largas a través del sistema circulatorio ; interacciones yuxtacrinas ; y señalización autocrina . Las células que producen factores paracrinos los secretan en el entorno extracelular inmediato . Luego, los factores viajan a las células cercanas en las que el gradiente de factor recibido determina el resultado. Sin embargo, la distancia exacta que pueden viajar los factores paracrinos no es segura.
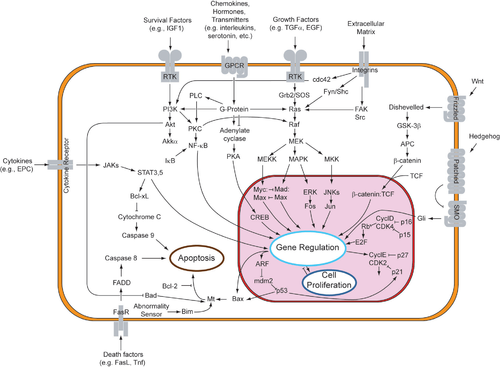
Aunque la señalización paracrina provoca una variedad de respuestas en las células inducidas, la mayoría de los factores paracrinos utilizan un conjunto relativamente simplificado de receptores y vías. De hecho, se sabe que diferentes órganos del cuerpo, incluso entre diferentes especies, utilizan conjuntos similares de factores paracrinos en el desarrollo diferencial. [1] Los receptores y vías altamente conservados se pueden organizar en cuatro familias principales basadas en estructuras similares: familia del factor de crecimiento de fibroblastos (FGF), familia Hedgehog , familia Wnt y superfamilia TGF-β . La unión de un factor paracrino a su respectivo receptor inicia cascadas de transducción de señales , provocando diferentes respuestas.
Los factores paracrinos inducen respuestas competentes
Para que los factores paracrinos induzcan con éxito una respuesta en la célula receptora, esa célula debe tener los receptores adecuados disponibles en la membrana celular para recibir las señales, lo que también se conoce como ser competente . Además, la célula que responde también debe tener la capacidad de ser inducida mecánicamente.
Familia del factor de crecimiento de fibroblastos (FGF)
Aunque la familia FGF de factores paracrinos tiene una amplia gama de funciones, los hallazgos principales respaldan la idea de que estimulan principalmente la proliferación y la diferenciación. [2] [3] Para cumplir muchas funciones diversas, los FGF pueden empalmarse alternativamente o incluso tener diferentes codones de iniciación para crear cientos de isoformas de FGF diferentes . [4]
Una de las funciones más importantes de los receptores de FGF (FGFR) es el desarrollo de las extremidades. Esta señalización implica nueve isoformas diferentes del receptor empalmadas alternativamente . [5] Fgf 8 y Fgf 10 son dos de los actores críticos en el desarrollo de las extremidades. En la iniciación de las extremidades anteriores y el crecimiento de las extremidades en ratones, las señales axiales (longitudinales) del mesodermo intermedio producen Tbx 5, que posteriormente envía señales al mismo mesodermo para que produzca Fgf 10. Fgf 10 luego envía señales al ectodermo para que comience la producción de Fgf 8, que también estimula la producción de Fgf 10. La eliminación de Fgf 10 da como resultado ratones sin extremidades. [6]
Además, la señalización paracrina de Fgf es esencial en el desarrollo del ojo de los polluelos. El ARNm de fgf 8 se localiza en lo que se diferencia en la retina neural de la copa óptica . Estas células están en contacto con las células del ectodermo externo, que finalmente se convertirán en el cristalino. [4]
Fenotipo y supervivencia de ratones tras la eliminación de algunos genes FGFR: [5]
| Gen knock-out del FGFR | Supervivencia | Fenotipo |
|---|---|---|
| Fgf1 | Viable | No claro |
| FGF3 | Viable | Diferenciación esquelética (cola) del oído interno |
| 4 de febrero | Letal | Proliferación de la masa celular interna |
| 8 de febrero | Letal | Defecto de gastrulación , desarrollo del sistema nervioso central, desarrollo de las extremidades |
| FGF10 | Letal | Desarrollo de múltiples órganos (incluidas las extremidades, el timo y la hipófisis) |
| Fgf17 | Viable | Desarrollo cerebeloso |
Vía de la tirosina quinasa del receptor (RTK)
La señalización paracrina a través de los factores de crecimiento de fibroblastos y sus respectivos receptores utiliza la vía de la tirosina del receptor . Esta vía de señalización ha sido ampliamente estudiada, utilizando ojos de Drosophila y cánceres humanos. [7]
La unión de FGF a FGFR fosforila la quinasa inactiva y activa la vía RTK. Esta vía comienza en la superficie de la membrana celular, donde un ligando se une a su receptor específico. Los ligandos que se unen a RTK incluyen factores de crecimiento de fibroblastos , factores de crecimiento epidérmico, factores de crecimiento derivados de plaquetas y factor de células madre . [7] Esto dimeriza el receptor transmembrana a otro receptor RTK, lo que causa la autofosforilación y el cambio conformacional posterior del receptor homodimerizado . Este cambio conformacional activa la quinasa inactiva de cada RTK en el residuo de tirosina. Debido al hecho de que el receptor se extiende a través de la membrana desde el entorno extracelular, a través de la bicapa lipídica y hacia el citoplasma , la unión del receptor al ligando también causa la fosforilación trans del dominio citoplasmático del receptor. [8]
Una proteína adaptadora (como SOS) reconoce la tirosina fosforilada en el receptor. Esta proteína funciona como un puente que conecta la RTK con una proteína intermedia (como GNRP), iniciando la cascada de señalización intracelular. A su vez, la proteína intermedia estimula la unión de Ras a GDP hacia la unión de Ras a GTP activada. GAP finalmente devuelve a Ras a su estado inactivo. La activación de Ras tiene el potencial de iniciar tres vías de señalización posteriores a Ras: la vía de la quinasa Ras→Raf→MAP, la vía de la quinasa PI3 y la vía Ral. Cada vía conduce a la activación de factores de transcripción que ingresan al núcleo para alterar la expresión génica. [9]

Receptor RTK y cáncer
Se ha demostrado que la señalización paracrina de factores de crecimiento entre células cercanas exacerba la carcinogénesis . De hecho, las formas mutantes de un único RTK pueden desempeñar un papel causal en tipos muy diferentes de cáncer. El protooncogén Kit codifica un receptor de tirosina quinasa cuyo ligando es una proteína paracrina llamada factor de células madre (SCF), que es importante en la hematopoyesis (formación de células en la sangre). [10] El receptor Kit y los receptores de tirosina quinasa relacionados en realidad son inhibidores y suprimen eficazmente la activación del receptor. Las formas mutantes del receptor Kit, que se activan constitutivamente de manera independiente del ligando, se encuentran en una amplia gama de neoplasias malignas cancerosas. [11]
Vía RTK y cáncer
Las investigaciones sobre el cáncer de tiroides han esclarecido la teoría de que la señalización paracrina puede ayudar a crear microambientes tumorales. La transcripción de quimiocinas aumenta cuando Ras está en estado unido a GTP. Las quimiocinas se liberan de la célula, libres para unirse a otra célula cercana. La señalización paracrina entre células vecinas crea este ciclo de retroalimentación positiva. Por lo tanto, la transcripción constitutiva de proteínas reguladas al alza forma entornos ideales para que surjan tumores. [12] Efectivamente, las uniones múltiples de ligandos a los receptores RTK sobreestimulan la vía Ras-Raf-MAPK, que sobreexpresa la capacidad mitogénica e invasiva de las células. [13]
Ruta JAK-STAT
Además de la vía RTK, los factores de crecimiento de fibroblastos también pueden activar la vía de señalización JAK-STAT . En lugar de llevar dominios de tirosina quinasa asociados covalentemente, los receptores Jak-STAT forman complejos no covalentes con tirosina quinasas de la clase Jak ( Janus quinasa ). Estos receptores se unen a la eritropoyetina (importante para la eritropoyesis ), la trombopoyetina (importante para la formación de plaquetas ) y el interferón (importante para mediar la función de las células inmunitarias). [14]
Después de la dimerización de los receptores de citocinas tras la unión del ligando, las JAK se transfosforilan entre sí. Las fosfotirosinas resultantes atraen a las proteínas STAT. Las proteínas STAT se dimerizan y entran en el núcleo para actuar como factores de transcripción que alteran la expresión génica. [14] En particular, las STAT transcriben genes que ayudan a la proliferación y supervivencia celular, como myc. [15]
Fenotipo y supervivencia de ratones tras la eliminación de algunos genes JAK o STAT: [16]
| Gen knockout | Supervivencia | Fenotipo |
|---|---|---|
| Jak1 | Letal | Déficits neurológicos |
| Jak2 | Letal | Fallo en la eritropoyesis |
| Estadística 1 | Viable | Síndromes de enanismo y craneosinostosis humanos |
| Estadística3 | Letal | Fenotipos específicos de tejido |
| Estadísticas 4 | Viable | Diferenciación defectuosa de Th1 impulsada por IL-12, mayor susceptibilidad a patógenos intracelulares |
Vía JAK-STAT aberrante y mutaciones óseas
La vía de señalización JAK-STAT es fundamental en el desarrollo de las extremidades, específicamente en su capacidad para regular el crecimiento óseo a través de la señalización paracrina de citocinas. Sin embargo, las mutaciones en esta vía se han relacionado con formas graves de enanismo: displasia tanatofórica (letal) y enanismo acondroplásico (viable). [17] Esto se debe a una mutación en un gen Fgf , que provoca una activación prematura y constitutiva del factor de transcripción Stat1 . La división celular de los condrocitos se termina prematuramente, lo que da lugar a un enanismo letal. Las células de la placa de crecimiento de los huesos de las costillas y de las extremidades no se transcriben. Por tanto, la incapacidad de la caja torácica para expandirse impide la respiración del recién nacido. [18]
La vía JAK-STAT y el cáncer
La investigación sobre la señalización paracrina a través de la vía JAK-STAT reveló su potencial para activar el comportamiento invasivo de las células epiteliales ováricas . Esta transición de epitelio a mesenquimal es muy evidente en la metástasis . [19] La señalización paracrina a través de la vía JAK-STAT es necesaria en la transición de células epiteliales estacionarias a células mesenquimales móviles, que son capaces de invadir el tejido circundante. Se ha descubierto que solo la vía JAK-STAT induce células migratorias. [20]
Familia de erizos
La familia de proteínas Hedgehog está involucrada en la inducción de tipos celulares y la creación de límites y patrones de tejido y se encuentran en todos los organismos bilaterales. Las proteínas Hedgehog fueron descubiertas y estudiadas por primera vez en Drosophila . Las proteínas Hedgehog producen señales clave para el establecimiento del plan corporal y de las extremidades de las moscas de la fruta, así como la homeostasis de los tejidos adultos, involucradas en la embriogénesis tardía y la metamorfosis . Se han encontrado al menos tres homólogos de hedgehog "Drosophila" en vertebrados: sonic hedgehog, desert hedgehog y Indian hedgehog. Sonic hedgehog ( SHH ) tiene varias funciones en el desarrollo de las vértebras, mediando la señalización y regulando la organización del sistema nervioso central, las extremidades y la polaridad de los somitas . Desert hedgehog ( DHH ) se expresa en las células de Sertoli involucradas en la espermatogénesis . Indian hedgehog ( IHH ) se expresa en el intestino y el cartílago, importante en el crecimiento óseo posnatal. [21] [22] [23]
Vía de señalización del erizo


Los miembros de la familia de proteínas Hedgehog actúan uniéndose a un receptor transmembrana " Patched ", que está unido a la proteína " Smoothened ", por el cual se puede transducir la señal Hedgehog . En ausencia de Hedgehog, el receptor Patched inhibe la acción de Smoothened. La inhibición de Smoothened hace que el complejo proteico Cubitus interruptus (Ci), Fused y Cos unido a los microtúbulos permanezca intacto. En esta conformación, la proteína Ci se escinde de modo que una porción de la proteína puede entrar en el núcleo y actuar como un represor transcripcional . En presencia de Hedgehog, Patched ya no inhibe a Smoothened. Entonces, la proteína Smoothened activa es capaz de inhibir PKA y Slimb, de modo que la proteína Ci no se escinde. Esta proteína Ci intacta puede entrar en el núcleo, asociarse con la proteína CPB y actuar como un activador transcripcional , induciendo la expresión de genes de respuesta a Hedgehog. [23] [24] [25]
La vía de señalización del erizo y el cáncer
La vía de señalización Hedgehog es fundamental para la correcta formación y orientación de los tejidos durante el desarrollo normal de la mayoría de los animales. Las proteínas Hedgehog inducen la proliferación celular en ciertas células y la diferenciación en otras. La activación aberrante de la vía Hedgehog se ha relacionado con varios tipos de cáncer , en particular el carcinoma basocelular . Esta activación descontrolada de las proteínas Hedgehog puede deberse a mutaciones en la vía de señalización, que serían independientes del ligando , o a una mutación que provoque la sobreexpresión de la proteína Hedgehog, que sería dependiente del ligando. Además, se ha demostrado que la activación de la vía Hedgehog inducida por terapia es necesaria para la progresión de los tumores de cáncer de próstata después de la terapia de privación de andrógenos . [26] Esta conexión entre la vía de señalización Hedgehog y los cánceres humanos puede permitir la posibilidad de una intervención terapéutica como tratamiento para dichos cánceres. La vía de señalización Hedgehog también participa en la regulación normal de las poblaciones de células madre y es necesaria para el crecimiento normal y la regeneración de los órganos dañados. Esto puede proporcionar otra ruta posible para la tumorigénesis a través de la vía Hedgehog. [27] [28] [29]
Familia Wnt

La familia de proteínas Wnt incluye una gran cantidad de glicoproteínas ricas en cisteína . Las proteínas Wnt activan cascadas de transducción de señales a través de tres vías diferentes: la vía Wnt canónica , la vía de polaridad celular planar (PCP) no canónica y la vía Wnt/Ca 2+ no canónica . Las proteínas Wnt parecen controlar una amplia gama de procesos de desarrollo y se las ha considerado necesarias para el control de la orientación del huso , la polaridad celular, la adhesión mediada por cadherina y el desarrollo temprano de embriones en muchos organismos diferentes. Las investigaciones actuales han indicado que la desregulación de la señalización Wnt desempeña un papel en la formación de tumores, porque a nivel celular, las proteínas Wnt a menudo regulan la proliferación celular , la morfología celular, la motilidad celular y el destino celular. [30]
La vía de señalización canónica de Wnt

En la vía canónica , las proteínas Wnt se unen a su receptor transmembrana de la familia de proteínas Frizzled . La unión de Wnt a una proteína Frizzled activa la proteína Dishevelled . En su estado activo, la proteína Dishevelled inhibe la actividad de la enzima glucógeno sintasa quinasa 3 ( GSK3 ). Normalmente, la GSK3 activa evita la disociación de la β-catenina a la proteína APC , lo que da como resultado la degradación de la β-catenina . Por lo tanto, la GSK3 inhibida permite que la β-catenina se disocie de la APC, se acumule y viaje al núcleo. En el núcleo, la β-catenina se asocia con el factor de transcripción Lef/Tcf , que ya está trabajando en el ADN como represor, inhibiendo la transcripción de los genes a los que se une. La unión de la β-catenina a Lef/Tcf funciona como un activador de la transcripción, activando la transcripción de los genes que responden a Wnt. [31] [32] [33]
Las vías de señalización de Wnt no canónicas
Las vías no canónicas de Wnt proporcionan una vía de transducción de señales para Wnt que no involucra a la β-catenina . En las vías no canónicas, Wnt afecta la actina y el citoesqueleto microtubular , así como la transcripción génica .
La vía de polaridad celular planar (PCP) no canónica

La vía no canónica de PCP regula la morfología , la división y el movimiento celular . Una vez más, las proteínas Wnt se unen a Frizzled y lo activan, de modo que Frizzled activa una proteína Dishevelled que está unida a la membrana plasmática a través de una proteína Prickle y una proteína transmembrana Stbm. La Dishevelled activa activa la GTPasa RhoA a través del activador asociado a Dishevelled de la morfogénesis 1 (Daam1) y la proteína Rac . La RhoA activa puede inducir cambios en el citoesqueleto activando la quinasa asociada a Roh (ROCK) y afectar directamente la transcripción génica. La Rac activa puede inducir directamente cambios en el citoesqueleto y afectar la transcripción génica a través de la activación de JNK. [31] [32] [33]
El Wnt/Ca no canónico2+camino

La vía no canónica Wnt/Ca 2+ regula los niveles intracelulares de calcio . Nuevamente, Wnt se une a Frizzled y se activa. En este caso, sin embargo, Frizzled activado hace que una proteína G acoplada active una fosfolipasa (PLC), que interactúa con PIP 2 y la divide en DAG e IP 3 . El IP 3 puede entonces unirse a un receptor en el retículo endoplásmico para liberar las reservas intracelulares de calcio, para inducir la expresión génica dependiente del calcio. [31] [32] [33]
Vías de señalización de Wnt y cáncer
Las vías de señalización de Wnt son fundamentales en la señalización de célula a célula durante el desarrollo normal y la embriogénesis y son necesarias para el mantenimiento del tejido adulto, por lo tanto, no es difícil entender por qué la alteración de las vías de señalización de Wnt puede promover enfermedades degenerativas y cáncer en humanos .
Las vías de señalización de Wnt son complejas, involucran muchos elementos diferentes y, por lo tanto, tienen muchos objetivos para la regulación incorrecta. Las mutaciones que causan la activación constitutiva de la vía de señalización de Wnt conducen a la formación de tumores y cáncer. La activación aberrante de la vía de Wnt puede conducir a un aumento de la proliferación celular. La investigación actual se centra en la acción de la vía de señalización de Wnt en la regulación de la elección de las células madre para proliferar y autorenovarse. Esta acción de la señalización de Wnt en el posible control y mantenimiento de las células madre, puede proporcionar un posible tratamiento en cánceres que exhiben una señalización Wnt aberrante. [34] [35] [36]
Superfamilia TGF-β
" TGF " (factor de crecimiento transformante) es una familia de proteínas que incluye 33 miembros que codifican polipéptidos diméricos secretados que regulan el desarrollo. [37] Muchos procesos de desarrollo están bajo su control, incluida la gastrulación, la simetría axial del cuerpo, la morfogénesis de órganos y la homeostasis tisular en adultos. [38] Todos los ligandos TGF-β se unen a receptores de tipo I o tipo II para crear complejos heterotetrámicos. [39]
Vía del TGF-β
La vía TGF-β regula muchos procesos celulares en el desarrollo de embriones y organismos adultos, incluyendo el crecimiento celular , la diferenciación , la apoptosis y la homeostasis . Hay cinco tipos de receptores tipo II y siete tipos de receptores tipo I en humanos y otros mamíferos. Estos receptores se conocen como "quinasas de doble especificidad" porque su dominio quinasa citoplasmático tiene una actividad débil de tirosina quinasa pero una fuerte actividad de serina / treonina quinasa. [40] Cuando un ligando de la superfamilia TGF-β se une al receptor tipo II, recluta un receptor tipo I y lo activa fosforilando los residuos de serina o treonina de su caja "GS". [41] Esto forma un complejo de activación que luego puede fosforilar proteínas SMAD.

Vía SMAD
Hay tres clases de SMAD:
Ejemplos de SMAD en cada clase: [42] [43] [44]
| Clase | SMAD |
|---|---|
| R-SMAD | SMAD1 , SMAD2 , SMAD3 , SMAD5 y SMAD8/9 |
| Co-SMAD | SMAD4 |
| I-SMAD | SMAD6 y SMAD7 |
La superfamilia TGF-β activa a los miembros de la familia SMAD , que funcionan como factores de transcripción. En concreto, el receptor de tipo I, activado por el receptor de tipo II, fosforila los R-SMAD que luego se unen al co-SMAD, SMAD4 . El R-SMAD/Co-SMAD forma un complejo con la importina y entra en el núcleo, donde actúan como factores de transcripción y regulan al alza o a la baja la expresión de un gen diana.
Los ligandos específicos de TGF-β darán como resultado la activación de los R-SMAD SMAD2/3 o SMAD1/5 . Por ejemplo, cuando la activina , el ligando Nodal o TGF-β se unen a los receptores, el complejo receptor fosforilado puede activar SMAD2 y SMAD3 a través de la fosforilación. Sin embargo, cuando un ligando BMP se une a los receptores, el complejo receptor fosforilado activa SMAD1 y SMAD5 . Luego, los complejos Smad2/3 o Smad1/5 forman un complejo dímero con SMAD4 y se convierten en factores de transcripción . Aunque hay muchos R-SMAD involucrados en la vía, solo hay un co-SMAD, SMAD4 . [45]
Vía no SMAD
Las proteínas de señalización no Smad contribuyen a las respuestas de la vía TGF-β de tres maneras. En primer lugar, las vías de señalización no Smad fosforilan las Smad. En segundo lugar, las Smad envían señales directamente a otras vías comunicándose directamente con otras proteínas de señalización, como las quinasas. Por último, los receptores TGF-β fosforilan directamente las proteínas no Smad. [46]
Miembros de la superfamilia TGF-β
1. Familia TGF-β
Esta familia incluye TGF-β1 , TGF-β2 , TGF-β3 y TGF-β5. Están involucrados en la regulación positiva y negativa de la división celular , la formación de la matriz extracelular entre las células, la apoptosis y la embriogénesis . Se unen al receptor de tipo II de TGF-β (TGFBRII).
El TGF-β1 estimula la síntesis de colágeno y fibronectina e inhibe la degradación de la matriz extracelular . En última instancia, aumenta la producción de matriz extracelular por parte de las células epiteliales . [39] Las proteínas TGF-β regulan los epitelios controlando dónde y cuándo se ramifican para formar conductos de riñones, pulmones y glándulas salivales. [39]
2. Familia de proteínas morfogenéticas óseas (BMP)
Se descubrió originalmente que los miembros de la familia BMP inducen la formación ósea , como sugiere su nombre. Sin embargo, las BMP son muy multifuncionales y también pueden regular la apoptosis , la migración celular , la división celular y la diferenciación . También especifican el eje anterior/posterior, inducen el crecimiento y regulan la homeostasis . [37]
Las BMP se unen al receptor de proteína morfogenética ósea tipo II (BMPR2). Algunas de las proteínas de la familia BMP son BMP4 y BMP7 . BMP4 promueve la formación ósea, causa la muerte celular o señala la formación de la epidermis , dependiendo del tejido sobre el que actúa. BMP7 es crucial para el desarrollo del riñón, la síntesis de esperma y la polarización del tubo neural. Tanto BMP4 como BMP7 regulan la estabilidad y el procesamiento de los ligandos maduros, incluida la degradación de ligandos en los lisosomas. [37] Las BMP actúan difundiéndose desde las células que las crean. [47]
Otros miembros de la superfamilia TGF-β
- Familia Vg1
- Familia Activin
- Participa en la embriogénesis y osteogénesis.
- Regular la insulina y las hormonas pituitarias , gonadales e hipotalámicas.
- Factores de supervivencia de las células nerviosas
- 3 Activinas: Activina A , Activina B y Activina AB .
- Factor neurotrófico derivado de la glía (GDNF)
- Necesario para la diferenciación de neuronas entéricas y renales.
- Factor inhibidor de Müller
- Interviene en la determinación del sexo en los mamíferos.
- Nodal
- Se une al receptor de activina A tipo 2B
- Forma un complejo receptor con el receptor de activina A tipo 1B o con el receptor de activina A tipo 1C . [48]
- Factores de crecimiento y diferenciación (FDC)
Tabla resumen de la vía de señalización del TGF-β
| Ligando de la superfamilia TGF Beta | Receptor tipo II | Receptor tipo I | R-SMAD | Co-SMAD | Inhibidores de ligandos |
|---|---|---|---|---|---|
| Activina A | ACVR2A | ACVR1B (ALK4) | SMAD2 , SMAD3 | SMAD4 | Folistatina |
| GDF1 | ACVR2A | ACVR1B (ALK4) | SMAD2 , SMAD3 | SMAD4 | |
| GDF11 | ACVR2B | ACVR1B (ALK4), TGFβRI (ALK5) | SMAD2 , SMAD3 | SMAD4 | |
| Proteínas morfogenéticas óseas | BMPR2 | BMPR1A (ALK3), BMPR1B (ALK6) | SMAD1 SMAD5 , SMAD8 | SMAD4 | Noggin , acordeón , DAN |
| Nodal | ACVR2B | ACVR1B (ALK4), ACVR1C (ALK7) | SMAD2 , SMAD3 | SMAD4 | Zurdo |
| TGFβ | TGFβRII | TGFβRI (ALK5) | SMAD2 , SMAD3 | SMAD4 | LTBP1 , THBS1 , Decorina |
Ejemplos
Los factores de crecimiento y de coagulación son agentes de señalización paracrinos. La acción local de la señalización de los factores de crecimiento desempeña un papel especialmente importante en el desarrollo de los tejidos. Además, el ácido retinoico , la forma activa de la vitamina A , funciona de manera paracrina para regular la expresión génica durante el desarrollo embrionario en animales superiores. [49] En los insectos, la alatostatina controla el crecimiento a través de la acción paracrina sobre los cuerpos alados. [ cita requerida ]
En los organismos maduros, la señalización paracrina está involucrada en las respuestas a los alérgenos , la reparación de tejidos, la formación de tejido cicatricial y la coagulación sanguínea . [ cita requerida ] La histamina es una paracrina que es liberada por las células inmunes en el árbol bronquial. La histamina hace que las células musculares lisas de los bronquios se contraigan, estrechando las vías respiratorias. [ 50 ]
Véase también
- Vía dependiente de AMPc
- Diafonía (biología)
- Señalización lipídica
- Hormona local : ya sea una hormona paracrina o una hormona que actúa tanto de manera paracrina como endocrina.
- Vía de señalización MAPK
- Netpath : un recurso seleccionado de vías de transducción de señales en humanos
- Regulador paracrino
Referencias
- ^ "Factores paracrinos" . Consultado el 27 de julio de 2018 .
- ^ Gospodarowicz, D.; Ferrara, N.; Schweigerer, L.; Neufeld, G. (1987). "Caracterización estructural y funciones biológicas del factor de crecimiento de fibroblastos". Endocrine Reviews . 8 (2): 95–114. doi :10.1210/edrv-8-2-95. PMID 2440668.
- ^ Rifkin, Daniel B.; Moscatelli, David (1989). "Desarrollos recientes en la biología celular del factor de crecimiento básico de fibroblastos". The Journal of Cell Biology . 109 (1): 1–6. doi :10.1083/jcb.109.1.1. JSTOR 1613457. PMC 2115467. PMID 2545723 .
- ^ ab Lappi, Douglas A. (1995). "Focalización tumoral a través de receptores de factores de crecimiento de fibroblastos". Seminarios en biología del cáncer . 6 (5): 279–88. doi :10.1006/scbi.1995.0036. PMID 8562905.
- ^ ab Xu, J.; Xu, J; Colvin, JS; McEwen, DG; MacArthur, CA; Coulier, F; Gao, G; Goldfarb, M (1996). "Especificidad del receptor de la familia del factor de crecimiento de fibroblastos". Journal of Biological Chemistry . 271 (25): 15292–7. doi : 10.1074/jbc.271.25.15292 . PMID 8663044.
- ^ Logan, M. (2003). "Dedo de la mano o del pie: la base molecular de la identidad de las extremidades". Desarrollo . 130 (26): 6401–10. doi : 10.1242/dev.00956 . PMID 14660539.
- ^ ab Fantl, Wendy J; Johnson, Daniel E; Williams, Lewis T (1993). "Señalización por tirosina quinasas receptoras". Revisión anual de bioquímica . 62 : 453–81. doi :10.1146/annurev.bi.62.070193.002321. PMID 7688944.
- ^ Yarden, Yosef; Ullrich, Axel (1988). "Receptores de factores de crecimiento tirosina quinasas". Revisión anual de bioquímica . 57 : 443–78. doi :10.1146/annurev.bi.57.070188.002303. PMID 3052279.
- ^ Katz, Michael E; McCormick, Frank (1997). "Transducción de señales a partir de múltiples efectores Ras". Current Opinion in Genetics & Development . 7 (1): 75–9. doi :10.1016/S0959-437X(97)80112-8. PMID 9024640.
- ^ Zsebo, Krisztina M.; Williams, David A.; Geissler, Edwin N.; Broudy, Virginia C.; Martin, Francis H.; Atkins, Harry L.; Hsu, Rou-Yin; Birkett, Neal C.; Okino, Kenneth H.; Murdock, Douglas C.; Jacobsen, Frederick W.; Langley, Keith E.; Smith, Kent A.; Takeish, Takashi; Cattanach, Bruce M.; Galli, Stephen J.; Suggs, Sidney V. (1990). "El factor de células madre está codificado en el locus SI del ratón y es el ligando para el receptor de tirosina quinasa c-kit". Cell . 63 (1): 213–24. doi :10.1016/0092-8674(90)90302-U. Número de modelo: PMID 1698556. Número de modelo: S2CID 39924379.
- ^ Rönnstrand, L. (2004). "Transducción de señales a través del receptor del factor de células madre/c-Kit". Ciencias de la vida celular y molecular . 61 (19–20): 2535–48. doi :10.1007/s00018-004-4189-6. PMID 15526160. S2CID 2602233.
- ^ Melillo, Rosa Marina; Castellone, María Domenica; Guarino, Valentina; De Falcó, Valentina; Cirafici, Anna María; Salvatore, Giuliana; Caiazzo, Fiorina; Basolo, Fulvio; Giannini, Ricardo; Kruhoffer, Mogens; Orntoft, Torben; Fusco, Alfredo; Santoro, Máximo (2005). "La cascada de señalización lineal RET/PTC-RAS-BRAF media el fenotipo móvil y mitogénico de las células cancerosas de tiroides". Revista de investigación clínica . 115 (4): 1068–81. doi :10.1172/JCI22758. PMC 1062891 . PMID 15761501. (Retractado, ver doi :10.1172/JCI87345, PMID 27035814, Retraction Watch )
- ^ Kolch, Walter (2000). "Relaciones significativas: la regulación de la vía Ras/Raf/MEK/ERK mediante interacciones proteicas". The Biochemical Journal . 351 (2): 289–305. doi :10.1042/0264-6021:3510289. PMC 1221363 . PMID 11023813.
- ^ ab Aaronson, David S.; Horvath, Curt M. (2002). "Una hoja de ruta para quienes no conocen JAK-STAT". Science . 296 (5573): 1653–5. Bibcode :2002Sci...296.1653A. doi :10.1126/science.1071545. PMID 12040185. S2CID 20857536.
- ^ Rawlings, Jason S.; Rosler, Kristin M.; Harrison, Douglas A. (2004). "La vía de señalización JAK/STAT". Journal of Cell Science . 117 (8): 1281–3. doi : 10.1242/jcs.00963 . PMID 15020666.
- ^ O'Shea, John J; Gadina, Massimo; Schreiber, Robert D (2002). "Señalización de citocinas en 2002: nuevas sorpresas en la vía Jak/Stat". Cell . 109 (2): S121–31. doi : 10.1016/S0092-8674(02)00701-8 . PMID 11983158.
- ^ Shiang, Rita; Thompson, Leslie M.; Zhu, Ya-Zhen; Iglesia, Deanna M.; Fielder, Thomas J.; Bocian, Maureen; Winokur, Sara T.; Wasmuth, John J. (1994). "Las mutaciones en el dominio transmembrana de FGFR3 provocan la forma genética más común de enanismo, la acondroplasia". Celúla . 78 (2): 335–42. doi :10.1016/0092-8674(94)90302-6. PMID 7913883. S2CID 20325070.
- ^ Kalluri, Raghu; Weinberg, Robert A. (2009). "Los fundamentos de la transición epitelial-mesenquimal". Revista de investigación clínica . 119 (6): 1420–8. doi :10.1172/JCI39104. PMC 2689101 . PMID 19487818.
- ^ Silver, Debra L.; Montell, Denise J. (2001). "La señalización paracrina a través de la vía JAK/STAT activa el comportamiento invasivo de las células epiteliales ováricas en Drosophila". Cell . 107 (7): 831–41. doi : 10.1016/S0092-8674(01)00607-9 . PMID 11779460.
- ^ Ingham, PW; McMahon, AP (2001). "Señalización de Hedgehog en el desarrollo animal: paradigmas y principios". Genes & Development . 15 (23): 3059–87. doi : 10.1101/gad.938601 . PMID 11731473.
- ^ Bitgood, Mark J.; McMahon, Andrew P. (1995). "Los genes Hedgehog y Bmp se coexpresan en muchos sitios diversos de interacción célula-célula en el embrión de ratón". Biología del desarrollo . 172 (1): 126–38. doi : 10.1006/dbio.1995.0010 . PMID 7589793.
- ^ ab Jacob, L.; Lum, L. (2007). "Vía de señalización de Hedgehog". Science's STKE . 2007 (407): cm6. doi :10.1126/stke.4072007cm6. PMID 17925577. S2CID 35653781.
- ^ Johnson, Ronald L; Scott, Matthew P (1998). "Nuevos actores y enigmas en la vía de señalización Hedgehog". Current Opinion in Genetics & Development . 8 (4): 450–6. doi : 10.1016/S0959-437X(98)80117-2 . PMID 9729722.
- ^ Nybakken, K; Perrimon, N (2002). "Transducción de señales de Hedgehog: hallazgos recientes". Current Opinion in Genetics & Development . 12 (5): 503–11. doi :10.1016/S0959-437X(02)00333-7. PMID 12200154.
- ^ Lubik AA, Nouri M, Truong S, Ghaffari M, Adomat HH, Corey E, Cox ME, Li N, Guns ES, Yenki P, Pham S, Buttyan R (2016). "La señalización paracrina de Sonic Hedgehog contribuye significativamente a la esteroidogénesis adquirida en el microambiente del tumor de próstata". Revista internacional del cáncer . 140 (2): 358–369. doi : 10.1002/ijc.30450 . PMID 27672740. S2CID 2354209.
- ^ Collins, RT; Cohen, SM (2005). "Un análisis genético en Drosophila para identificar nuevos componentes de la vía de señalización Hedgehog". Genética . 170 (1): 173–84. doi :10.1534/genetics.104.039420. PMC 1449730 . PMID 15744048.
- ^ Evangelista, M.; Tian, H.; De Sauvage, FJ (2006). "La vía de señalización Hedgehog en el cáncer". Investigación clínica del cáncer . 12 (20): 5924–8. doi : 10.1158/1078-0432.CCR-06-1736 . PMID: 17062662.
- ^ Taipale, Jussi; Beachy, Philip A. (2001). "Las vías de señalización Hedgehog y Wnt en el cáncer". Nature . 411 (6835): 349–54. Bibcode :2001Natur.411..349T. doi :10.1038/35077219. PMID 11357142. S2CID 4414768.
- ^ Cadigan, KM; Nusse, R. (1997). "Señalización Wnt: un tema común en el desarrollo animal". Genes & Development . 11 (24): 3286–305. doi : 10.1101/gad.11.24.3286 . PMID 9407023.
- ^ abc Dale, Trevor C. (1998). "Transducción de señales por la familia de ligandos Wnt". The Biochemical Journal . 329 (Pt 2): 209–23. doi :10.1042/bj3290209. PMC 1219034 . PMID 9425102.
- ^ abc Chen, Xi; Yang, Jun; Evans, Paul M; Liu, Chunming (2008). "Señalización de Wnt: lo bueno y lo malo". Acta Biochimica et Biophysica Sinica . 40 (7): 577–94. doi :10.1111/j.1745-7270.2008.00440.x. PMC 2532600 . PMID 18604449.
- ^ abc Komiya, Yuko; Habas, Raymond (2008). "Vías de transducción de señales Wnt". Organogénesis . 4 (2): 68–75. doi : 10.4161/org.4.2.5851. PMC 2634250 . PMID 19279717.
- ^ Logan, Catriona Y.; Nusse, Roel (2004). "La vía de señalización de Wnt en el desarrollo y la enfermedad". Revisión anual de biología celular y del desarrollo . 20 : 781–810. CiteSeerX 10.1.1.322.311 . doi :10.1146/annurev.cellbio.20.010403.113126. PMID 15473860.
- ^ Lustig, B; Behrens, J (2003). "La vía de señalización de Wnt y su papel en el desarrollo de tumores". Revista de investigación del cáncer y oncología clínica . 129 (4): 199–221. doi :10.1007/s00432-003-0431-0. PMID 12707770. S2CID 28959851.
- ^ Neth, Peter; Ries, Christian; Karow, Marisa; Egea, Virginia; Ilmer, Matthias; Jochum, Marianne (2007). "La vía de transducción de señales Wnt en células madre y células cancerosas: influencia en la invasión celular". Stem Cell Reviews . 3 (1): 18–29. doi :10.1007/s12015-007-0001-y. PMID 17873378. S2CID 25793825.
- ^ abc Bandyopadhyay, Amitabha; Tsuji, Kunikazu; Cox, Karen; Harfe, Brian D.; Rosen, Vicki; Tabin, Clifford J. (2006). "Análisis genético de los roles de BMP2, BMP4 y BMP7 en la formación de patrones de extremidades y la esqueletogénesis". PLOS Genetics . 2 (12): e216. doi : 10.1371/journal.pgen.0020216 . PMC 1713256 . PMID 17194222.
- ^ Attisano, Liliana; Wrana, Jeffrey L. (2002). "Transducción de señales por la superfamilia TGF-β". Science . 296 (5573): 1646–7. Bibcode :2002Sci...296.1646A. doi :10.1126/science.1071809. PMID 12040180. S2CID 84138159.
- ^ abc Wrana, Jeffrey L.; Ozdamar, Barish; Le Roy, Christine; Benchabane, Hassina (2008). "Receptores de señalización de la familia TGF-β". En Derynck, Rik; Miyazono, Kohei (eds.). La familia TGF-β . CSHL Press. págs. 151–77. ISBN 978-0-87969-752-5.
- ^ ten Dijke, Peter; Heldin, Carl-Henrik (2006). "La familia Smad". En ten Dijke, Peter; Heldin, Carl-Henrik (eds.). Transducción de señales Smad: Smads en proliferación, diferenciación y enfermedad . Proteínas y regulación celular. Vol. 5. Dordrecht: Springer. págs. 1–13. ISBN 978-1-4020-4709-1.
- ^ Moustakas, Aristidis (1 de septiembre de 2002). "Red de señalización Smad". Revista de Ciencias Celulares . 115 (17): 3355–6. doi : 10.1242/jcs.115.17.3355 . PMID 12154066.
- ^ Wu, Jia-Wei; Hu, Min; Chai, Jijie; Seoane, Joan; Casa, Morgan; Li, Carey; Rigotti, Daniel J.; Kyin, vio; Muir, Tom W.; Fairman, Robert; Massagué, Joan; Shi, Yigong (2001). "Estructura cristalina de un Smad2 fosforilado". Célula molecular . 8 (6): 1277–89. doi : 10.1016/S1097-2765(01)00421-X . PMID 11779503.
- ^ Pavletich, Nikola P.; Hata, Yigong; Lo, Akiko; Massagué, Roger S.; Pavletich, Joan (1997). "Una base estructural para la inactivación mutacional del supresor tumoral Smad4". Nature . 388 (6637): 87–93. Bibcode :1997Natur.388R..87S. doi : 10.1038/40431 . PMID 9214508.
- ^ Itoh, Fumiko; Asao, Hironobu; Sugamura, Kazuo; Heldin, Carl-Henrik; Ten Dijke, Peter; Itoh, Susumu (2001). "Promoción de la señalización de la proteína morfogenética ósea a través de la regulación negativa de Smads inhibidores". The EMBO Journal . 20 (15): 4132–42. doi :10.1093/emboj/20.15.4132. PMC 149146 . PMID 11483516.
- ^ Schmierer, Bernhard; Hill, Caroline S. (2007). "Transducción de señales TGFβ–SMAD: especificidad molecular y flexibilidad funcional". Nature Reviews Molecular Cell Biology . 8 (12): 970–82. doi :10.1038/nrm2297. PMID 18000526. S2CID 131895.
- ^ Moustakas, Aristidis; Heldin, Carl-Henrik (2005). "Señales de TGF-β no Smad". Journal of Cell Science . 118 (16): 3573–84. doi : 10.1242/jcs.02554 . PMID 16105881.
- ^ Ohkawara, Bisei; Iemura, Shun-Ichiro; Ten Dijke, Peter; Ueno, Naoto (2002). "El rango de acción de BMP se define por su núcleo de aminoácidos básicos N-terminal". Current Biology . 12 (3): 205–9. doi : 10.1016/S0960-9822(01)00684-4 . PMID 11839272.
- ^ Munir, Sadia; Xu, Guoxiong; Wu, Yaojiong; Yang, Burton; Lala, Peeyush K.; Peng, Chun (2004). "Nodal y ALK7 inhiben la proliferación e inducen apoptosis en células del trofoblasto humano". Journal of Biological Chemistry . 279 (30): 31277–86. doi : 10.1074/jbc.M400641200 . PMID 15150278.
- ^ Duester, Gregg (septiembre de 2008). "Síntesis y señalización del ácido retinoico durante la organogénesis temprana". Cell . 134 (6): 921–31. doi :10.1016/j.cell.2008.09.002. PMC 2632951 . PMID 18805086.
- ^ Este artículo incorpora texto disponible bajo la licencia CC BY 4.0. Betts, J Gordon; Desaix, Peter; Johnson, Eddie; Johnson, Jody E; Korol, Oksana; Kruse, Dean; Poe, Brandon; Wise, James; Womble, Mark D; Young, Kelly A (24 de julio de 2023). Anatomía y fisiología . Houston: OpenStax CNX. 17.1 Descripción general del sistema endocrino. ISBN 978-1-947172-04-3.
Enlaces externos
- Señalización paracrina+ en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- "paracrino" en el Diccionario médico de Dorland