Madres contra el homólogo decapentapléjico 4
| SMAD4 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | SMAD4 , DPC4, JIP, MADH4, MYHRS, miembro de la familia SMAD 4 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | OMIM : 600993; MGI : 894293; HomoloGene : 31310; Tarjetas genéticas : SMAD4; OMA :SMAD4 - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
SMAD4 , también llamada miembro 4 de la familia SMAD , Madres contra el homólogo decapentapléjico 4 o DPC4 (Deleted in Pancreatic Cancer-4) es una proteína altamente conservada presente en todos los metazoos . Pertenece a la familia SMAD de proteínas de factores de transcripción , que actúan como mediadores de la transducción de señales de TGF-β. La familia de citocinas TGFβ regula procesos críticos durante el ciclo de vida de los metazoos, con papeles importantes durante el desarrollo del embrión, la homeostasis tisular, la regeneración y la regulación inmunológica. [5]
SMAD 4 pertenece al grupo de co-SMAD ( mediador común SMAD), la segunda clase de la familia SMAD. SMAD4 es el único co-SMAD conocido en la mayoría de los metazoos. También pertenece a la familia Darwin de proteínas que modulan a miembros de la superfamilia de proteínas TGFβ , una familia de proteínas que desempeñan un papel en la regulación de las respuestas celulares. SMAD4 de mamíferos es un homólogo de la proteína de Drosophila " Madres contra decapentapléjicos " llamada Medea. [6]
SMAD4 interactúa con R-Smads, como SMAD2 , SMAD3 , SMAD1 , SMAD5 y SMAD8 (también llamado SMAD9) para formar complejos heterotriméricos. Los correguladores transcripcionales, como WWTR1 (TAZ) interactúan con SMADs para promover su función. Una vez en el núcleo, el complejo de SMAD4 y dos R-SMADS se une al ADN y regula la expresión de diferentes genes dependiendo del contexto celular. [6] Las reacciones intracelulares que involucran a SMAD4 son desencadenadas por la unión, en la superficie de las células, de factores de crecimiento de la familia TGFβ . La secuencia de reacciones intracelulares que involucran SMADS se llama vía SMAD o vía del factor de crecimiento transformante beta (TGF-β) ya que la secuencia comienza con el reconocimiento de TGF-β por las células.
Gene
En los mamíferos, el gen SMAD4 está codificado por un gen ubicado en el cromosoma 18. En los humanos, el gen SMAD4 contiene 54 829 pares de bases y se ubica desde el par n° 51 030 212 hasta el par n° 51 085 041 en la región 21.1 del cromosoma 18. [7] [8]

Proteína
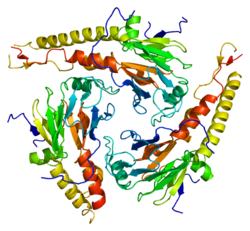
SMAD4 es un polipéptido de 552 aminoácidos con un peso molecular de 60,439 Da . SMAD4 tiene dos dominios funcionales conocidos como MH1 y MH2.
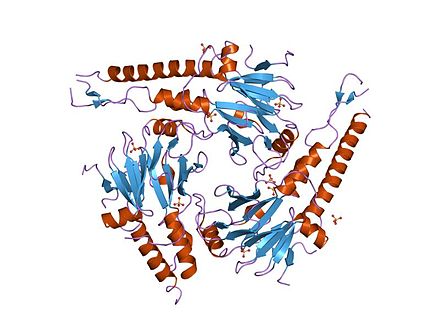
El complejo de dos SMAD3 (o de dos SMAD2) y un SMAD4 se une directamente al ADN a través de interacciones de sus dominios MH1. Estos complejos son reclutados a sitios en todo el genoma por factores de transcripción que definen el linaje celular (LDTF) que determinan la naturaleza dependiente del contexto de la acción de TGF-β. Los primeros conocimientos sobre la especificidad de unión al ADN de las proteínas Smad provinieron de los análisis de unión de oligonucleótidos, que identificaron el dúplex palindrómico 5'–GTCTAGAC–3' como una secuencia de unión de alta afinidad para los dominios MH1 de SMAD3 y SMAD4. [9] También se han identificado otros motivos en promotores y potenciadores. Estos sitios adicionales contienen el motivo CAGCC y las secuencias de consenso GGC(GC)|(CG), estas últimas también conocidas como sitios 5GC. [10] Los motivos 5GC están altamente representados como grupos de sitios, en regiones unidas a SMAD en todo el genoma. Estos grupos también pueden contener sitios CAG(AC)|(CC). El complejo SMAD3/SMAD4 también se une a los elementos promotores de genes sensibles a TPA, que tienen el motivo de secuencia TGAGTCAG. [11]
Estructuras
Complejos de dominio MH1 con motivos de ADN
La primera estructura de SMAD4 unida al ADN fue el complejo con el motivo palindrómico GTCTAGAC. [12] Recientemente, también se han determinado las estructuras del dominio MH1 de SMAD4 unidas a varios motivos 5GC. En todos los complejos, la interacción con el ADN implica una horquilla β conservada presente en el dominio MH1. La horquilla es parcialmente flexible en solución y su alto grado de flexibilidad conformacional permite el reconocimiento de las diferentes secuencias de 5 pb. Las interacciones eficientes con los sitios GC ocurren solo si un nucleótido G se encuentra en lo profundo del surco principal y establece enlaces de hidrógeno con el grupo guanidinio de Arg81. Esta interacción facilita un contacto superficial complementario entre la horquilla de unión al ADN de Smad y el surco principal del ADN. Otras interacciones directas involucran a Lys88 y Gln83. La estructura cristalina de rayos X de los dominios SMAD4 MH1 de Trichoplax adhaerens unidos al motivo GGCGC indica una alta conservación de esta interacción en los metazoos. [10]



Complejos de dominios MH2
El dominio MH2, correspondiente al extremo C , es responsable del reconocimiento del receptor y de la asociación con otros SMAD. Interactúa con el dominio MH2 de R-SMADS y forma heterodímeros y heterotrímeros. Algunas mutaciones tumorales detectadas en SMAD4 potencian las interacciones entre los dominios MH1 y MH2. [13]
Nomenclatura y origen del nombre
Los SMAD están altamente conservados entre especies, especialmente en el dominio MH1 de la terminal N y el dominio MH2 de la terminal C. Las proteínas SMAD son homólogas tanto de la proteína MAD de Drosophila como de la proteína SMA de C. elegans . El nombre es una combinación de las dos. Durante la investigación de Drosophila , se descubrió que una mutación en el gen MAD en la madre reprimió el gen decapentapléjico en el embrión. Se agregó la frase "Madres en contra", ya que las madres a menudo forman organizaciones que se oponen a varias cuestiones, por ejemplo, Madres contra la conducción en estado de ebriedad (MADD), lo que refleja "la mejora del efecto maternal de la dpp "; [14] y se basa en una tradición de nombres inusuales dentro de la comunidad de investigación. [15] SMAD4 también se conoce como DPC4, JIP o MADH4.
Función y mecanismo de acción
SMAD4 es una proteína definida como un efector esencial en la vía SMAD. SMAD4 actúa como mediador entre los factores de crecimiento extracelular de la familia TGFβ y los genes dentro del núcleo celular . La abreviatura co en co-SMAD significa mediador común . SMAD4 también se define como un transductor de señales.
En la vía del TGF-β, los dímeros de TGF-β son reconocidos por un receptor transmembrana, conocido como receptor tipo II. Una vez que el receptor tipo II se activa por la unión de TGF-β, fosforila un receptor tipo I. El receptor tipo I también es un receptor de superficie celular . Este receptor fosforila entonces SMADS regulados por receptores intracelulares (R-SMADS) como SMAD2 o SMAD3. Los R-SMADS fosforilados se unen entonces a SMAD4. La asociación R-SMADs-SMAD4 es un complejo heteromérico . Este complejo va a moverse desde el citoplasma hasta el núcleo: es la translocación. SMAD4 puede formar complejos heterotriméricos, heterohexaméricos o heterodiméricos con R-SMADS.
SMAD4 es un sustrato de la quinasa Erk / MAPK [16] y GSK3 . [17] La estimulación de la vía FGF ( factor de crecimiento de fibroblastos ) conduce a la fosforilación de Smad4 por Erk del sitio MAPK canónico ubicado en la treonina 277. Este evento de fosforilación tiene un doble efecto en la actividad de Smad4. Primero, permite que Smad4 alcance su pico de actividad transcripcional activando un dominio de activación de la transcripción regulado por un factor de crecimiento ubicado en la región de enlace de Smad4, SAD (dominio de activación de Smad). [18] En segundo lugar, MAPK prepara a Smad4 para las fosforilaciones mediadas por GSK3 que causan inhibición transcripcional y también generan un fosfodegrón utilizado como sitio de acoplamiento por la ligasa de ubiquitina E3 Beta-transducin Repeat Containing ( beta-TrCP ) que poliubiquitina Smad4 y lo dirige para su degradación en el proteasoma . [19] Se ha propuesto que las fosforilaciones de Smad4 GSK3 regulan la estabilidad de la proteína durante la progresión del cáncer de páncreas y de colon . [20]
En el núcleo, el complejo heteromérico se une a los promotores e interactúa con los activadores transcripcionales. Los complejos SMAD3 /SMAD4 pueden unirse directamente al SBE. Estas asociaciones son débiles y requieren factores de transcripción adicionales , como los miembros de la familia AP-1 , TFE3 y FoxG1, para regular la expresión génica . [21]
Muchos ligandos de TGFβ utilizan esta vía y posteriormente SMAD4 participa en muchas funciones celulares como la diferenciación , la apoptosis , la gastrulación , el desarrollo embrionario y el ciclo celular .
Importancia clínica
Para observar los efectos de un SMAD 4 disfuncional en el organismo estudiado, se pueden llevar a cabo experimentos genéticos como el knockout (KO), que consiste en modificar o inactivar un gen. Los experimentos se realizan a menudo en el ratón doméstico ( Mus musculus ).
Se ha demostrado que, en ratones KO de SMAD4 , las células de la granulosa , que secretan hormonas y factores de crecimiento durante el desarrollo del ovocito , sufren una luteinización prematura y expresan niveles más bajos de receptores de la hormona folículo estimulante (FSHR) y niveles más altos de receptores de la hormona luteinizante (LHR). Esto puede deberse en parte a la alteración de los efectos de la proteína morfogenética ósea 7 , ya que BMP-7 utiliza la vía de señalización SMAD4. [22] [23]
Las deleciones en los genes que codifican SMAD1 y SMAD5 también se han relacionado con tumores metastásicos de células de la granulosa en ratones. [24]
El SMAD4 se encuentra mutado en muchos tipos de cáncer. La mutación puede ser hereditaria o adquirida durante la vida de un individuo. Si es hereditaria, la mutación afecta tanto a las células somáticas como a las células de los órganos reproductores. Si la mutación SMAD 4 es adquirida, sólo existirá en ciertas células somáticas. De hecho, SMAD 4 no es sintetizado por todas las células. La proteína está presente en la piel, el páncreas, el colon, el útero y las células epiteliales. También es producida por fibroblastos . El SMAD 4 funcional participa en la regulación de la vía de transducción de señales TGF-β, que regula negativamente el crecimiento de las células epiteliales y la matriz extracelular (ECM). Cuando la estructura de SMAD 4 se altera, la expresión de los genes implicados en el crecimiento celular ya no está regulada y la proliferación celular puede continuar sin ninguna inhibición. El importante número de divisiones celulares conduce a la formación de tumores y luego al cáncer colorrectal multiploide y al carcinoma pancreático . Se encuentra inactivado en al menos el 50% de los cánceres pancreáticos. [25]
Se ha demostrado que las mutaciones somáticas encontradas en cánceres humanos del dominio MH1 de SMAD 4 inhiben la función de unión al ADN de este dominio.
El SMAD 4 también se encuentra mutado en el síndrome de poliposis juvenil (SPJ), una enfermedad autosómica dominante que se caracteriza por la presencia de pólipos hamartomatosos en el tracto gastrointestinal (GI). Estos pólipos suelen ser benignos, pero presentan un mayor riesgo de desarrollar cánceres gastrointestinales , en particular cáncer de colon . Se han identificado alrededor de 60 mutaciones que causan el SPJ y se han relacionado con la producción de un SMAD 4 más pequeño, con dominios faltantes que impiden que la proteína se una a R-SMADS y forme complejos heteroméricos . [8]
Las mutaciones en SMAD4 (principalmente sustituciones) pueden causar el síndrome de Myhre , un trastorno hereditario poco común caracterizado por discapacidades mentales, baja estatura, rasgos faciales inusuales y diversas anomalías óseas. [26] [27]
Referencias
- ^ abc GRCh38: Lanzamiento de Ensembl 89: ENSG00000141646 – Ensembl , mayo de 2017
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000024515 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia PubMed de ratón:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU . .
- ^ Massagué J (octubre de 2012). "Señalización de TGFβ en contexto". Nature Reviews. Molecular Cell Biology . 13 (10): 616–630. doi :10.1038/nrm3434. PMC 4027049 . PMID 22992590.
- ^ ab Massagué J (1998). "Transducción de señales de TGF-beta". Revista Anual de Bioquímica . 67 (1): 753–791. doi : 10.1146/annurev.biochem.67.1.753 . PMID 9759503.
- ^ "SMAD4 Miembro de la familia SMAD 4". Entrez Gene .
- ^ ab "SMAD 4". El sitio web de referencia de genética para el hogar .
- ^ Zawel L, Dai JL, Buckhaults P, Zhou S, Kinzler KW, Vogelstein B, et al. (Marzo de 1998). "Human Smad3 y Smad4 son activadores de transcripción específicos de secuencia". Célula molecular . 1 (4): 611–617. doi : 10.1016/s1097-2765(00)80061-1 . PMID 9660945.
- ^ ab Martin-Malpartida P, Batet M, Kaczmarska Z, Freier R, Gomes T, Aragón E, et al. (Diciembre de 2017). "Base estructural para el reconocimiento de todo el genoma de motivos GC de 5 pb mediante factores de transcripción SMAD". Comunicaciones de la naturaleza . 8 (1): 2070. Código Bib : 2017NatCo...8.2070M. doi :10.1038/s41467-017-02054-6. PMC 5727232 . PMID 29234012.
- ^ Zhang Y, Feng XH, Derynck R (agosto de 1998). "Smad3 y Smad4 cooperan con c-Jun/c-Fos para mediar la transcripción inducida por TGF-beta". Nature . 394 (6696): 909–913. Bibcode :1998Natur.394..909Z. doi :10.1038/29814. PMID 9732876. S2CID 4393852.
- ^ Baburajendran N, Jauch R, Tan CY, Narasimhan K, Kolatkar PR (octubre de 2011). "Base estructural para el reconocimiento cooperativo de ADN por dímeros Smad4 MH1". Nucleic Acids Research . 39 (18): 8213–8222. doi :10.1093/nar/gkr500. PMC 3185416 . PMID 21724602.
- ^ Hata A, Lo RS, Wotton D, Lagna G, Massagué J (julio de 1997). "Las mutaciones que aumentan la autoinhibición inactivan los supresores tumorales Smad2 y Smad4". Nature . 388 (6637): 82–87. Bibcode :1997Natur.388R..82H. doi : 10.1038/40424 . PMID 9214507. S2CID 4407819.
- ^ Sekelsky JJ, Newfeld SJ, Raftery LA, Chartoff EH, Gelbart WM (marzo de 1995). "Caracterización genética y clonación de madres contra dpp, un gen necesario para la función decapentapléjica en Drosophila melanogaster". Genética . 139 (3): 1347–1358. doi :10.1093/genetics/139.3.1347. PMC 1206461 . PMID 7768443.
- ^ White M (26 de septiembre de 2014). "Sonic Hedgehog, DICER y el problema de nombrar genes". Pacific Standard .
- ^ Roelen BA, Cohen OS, Raychowdhury MK, Chadee DN, Zhang Y, Kyriakis JM, et al. (octubre de 2003). "La fosforilación de la treonina 276 en Smad4 está implicada en la acumulación nuclear inducida por el factor de crecimiento transformante beta". Revista estadounidense de fisiología. Fisiología celular . 285 (4): C823–C830. doi :10.1152/ajpcell.00053.2003. PMID 12801888. S2CID 9326115.
- ^ Demagny H, Araki T, De Robertis EM (octubre de 2014). "El supresor tumoral Smad4/DPC4 está regulado por fosforilaciones que integran la señalización de FGF, Wnt y TGF-β". Cell Reports . 9 (2): 688–700. doi : 10.1016/j.celrep.2014.09.020 . PMID 25373906.
- ^ de Caestecker MP, Yahata T, Wang D, Parks WT, Huang S, Hill CS, et al. (enero de 2000). "El dominio de activación de Smad4 (SAD) es un dominio de activación transcripcional dependiente de p300 y rico en prolina". The Journal of Biological Chemistry . 275 (3): 2115–2122. doi : 10.1074/jbc.275.3.2115 . PMID 10636916.
- ^ Demagny H, De Robertis EM (marzo de 2016). "Smad4/DPC4: una barrera contra la progresión tumoral impulsada por la señalización RTK/Ras/Erk y Wnt/GSK3". Oncología molecular y celular . 3 (2): e989133. doi :10.4161/23723556.2014.989133. PMC 4905428 . PMID 27308623.
- ^ Demagny H, De Robertis EM (enero de 2016). "Las mutaciones puntuales en el supresor tumoral Smad4/DPC4 mejoran su fosforilación por GSK3 e inactivan reversiblemente la señalización de TGF-β". Oncología molecular y celular . 3 (1): e1025181. doi :10.1080/23723556.2015.1025181. PMC 4845174 . PMID 27308538.
- ^ Inman GJ (febrero de 2005). "Vinculación de Smads y activación transcripcional". The Biochemical Journal . 386 (Pt 1): e1–e3. doi :10.1042/bj20042133. PMC 1134782 . PMID 15702493.
- ^ Shi J, Yoshino O, Osuga Y, Nishii O, Yano T, Taketani Y (marzo de 2010). "La proteína morfogenética ósea 7 (BMP-7) aumenta la expresión del receptor de la hormona folículo estimulante (FSH) en células de la granulosa humana". Fertilidad y esterilidad . 93 (4): 1273–1279. doi : 10.1016/j.fertnstert.2008.11.014 . PMID 19108831.
- ^ Pangas SA, Li X, Robertson EJ, Matzuk MM (junio de 2006). "Luteinización prematura y defectos en las células del cúmulo en ratones knock-out específicos para Smad4 en el ovario". Endocrinología molecular . 20 (6): 1406–1422. doi : 10.1210/me.2005-0462 . PMID 16513794.
- ^ Middlebrook BS, Eldin K, Li X, Shivasankaran S, Pangas SA (diciembre de 2009). "Los ratones knock-out condicionales de ovario Smad1-Smad5 desarrollan un perfil de enfermedad similar a la forma juvenil de los tumores de células de la granulosa humanas". Endocrinología . 150 (12): 5208–5217. doi : 10.1210/en.2009-0644 . PMC 2819741 . PMID 19819941.
- ^ Cotran RS, Kumar V, Fausto N, Robbins SL, Abbas AK (2005). Robbins y Cotran: bases patológicas de la enfermedad (7.ª ed.). St. Louis, Mo: Elsevier Saunders. ISBN 0-7216-0187-1.
- ^ "Síndrome de deficiencia mental y de crecimiento de Myhre". Organización Nacional de Trastornos Raros . Archivado desde el original el 2 de abril de 2015. Consultado el 26 de marzo de 2015 .
- ^ Caputo V, Bocchinfuso G, Castori M, Traversa A, Pizzuti A, Stella L, et al. (Julio de 2014). "Nueva mutación SMAD4 que causa el síndrome de Myhre". Revista Estadounidense de Genética Médica. Parte A. 164A (7): 1835–1840. doi :10.1002/ajmg.a.36544. PMID 24715504. S2CID 5294309.
Lectura adicional
- Miyazono K (2000). "Señalización de TGF-beta por proteínas Smad". Cytokine & Growth Factor Reviews . 11 (1–2): 15–22. doi :10.1016/S1359-6101(99)00025-8. PMID 10708949.
- Wrana JL, Attisano L (2000). "La vía Smad". Cytokine & Growth Factor Reviews . 11 (1–2): 5–13. doi :10.1016/S1359-6101(99)00024-6. PMID 10708948.
- Verschueren K, Huylebroeck D (2000). "Versatilidad notable de las proteínas Smad en el núcleo de células activadas por el factor de crecimiento transformante beta". Cytokine & Growth Factor Reviews . 10 (3–4): 187–199. doi :10.1016/S1359-6101(99)00012-X. PMID 10647776.
- Massagué J (1998). "Transducción de señales de TGF-beta". Revista Anual de Bioquímica . 67 : 753–791. doi : 10.1146/annurev.biochem.67.1.753 . PMID 9759503.
- Klein-Scory S, Zapatka M, Eilert-Micus C, Hoppe S, Schwarz E, Schmiegel W, et al. (noviembre de 2007). "La reexpresión inducible de alto nivel de Smad4 en la línea celular de cáncer cervical C4-II está asociada con un perfil de expresión génica que predice un papel preferencial de Smad4 en la composición de la matriz extracelular". BMC Cancer . 7 : 209. doi : 10.1186/1471-2407-7-209 . PMC 2186346 . PMID 17997817.
- Kalo E, Buganim Y, Shapira KE, Besserglick H, Goldfinger N, Weisz L, et al. (diciembre de 2007). "El p53 mutante atenúa la vía de señalización del factor de crecimiento transformante beta1 (TGF-beta1) dependiente de SMAD al reprimir la expresión del receptor de TGF-beta tipo II". Biología molecular y celular . 27 (23): 8228–8242. doi :10.1128/MCB.00374-07. PMC 2169171 . PMID 17875924.
- Aretz S, Stienen D, Uhlhaas S, Stolte M, Entius MM, Loff S, et al. (noviembre de 2007). "Alta proporción de grandes deleciones genómicas y una actualización del fenotipo genotípico en 80 familias no relacionadas con síndrome de poliposis juvenil". Journal of Medical Genetics . 44 (11): 702–709. doi :10.1136/jmg.2007.052506. PMC 2752176 . PMID 17873119.
- Ali S, Cohen C, Little JV, Sequeira JH, Mosunjac MB, Siddiqui MT (octubre de 2007). "La utilidad de SMAD4 como marcador inmunohistoquímico de diagnóstico para el adenocarcinoma pancreático y su expresión en otros tumores sólidos". Citopatología diagnóstica . 35 (10): 644–648. doi :10.1002/dc.20715. PMID 17854080. S2CID 36682992.
- Milet J, Dehais V, Bourgain C, Jouanolle AM, Mosser A, Perrin M, et al. (octubre de 2007). "Variantes comunes en los genes BMP2, BMP4 y HJV de la vía de regulación de la hepcidina modulan la penetrancia de la hemocromatosis HFE". American Journal of Human Genetics . 81 (4): 799–807. doi :10.1086/520001. PMC 2227929 . PMID 17847004.
- Salek C, Benesova L, Zavoral M, Nosek V, Kasperova L, Ryska M, et al. (julio de 2007). "Evaluación de la relevancia clínica del examen de las mutaciones de K-ras, p16 y p53 junto con las pérdidas alélicas en 9p y 18q en muestras de aspiración con aguja fina guiadas por ecoendoscopia de pacientes con pancreatitis crónica y cáncer de páncreas". Revista Mundial de Gastroenterología . 13 (27): 3714–3720. doi : 10.3748/wjg.v13.i27.3714 . PMC 4250643 . PMID 17659731.
- Sebestyén A, Hajdu M, Kis L, Barna G, Kopper L (septiembre de 2007). "Efecto apoptótico independiente de Smad4 y dependiente de PP2A del factor de crecimiento transformante beta 1 exógeno en células de linfoma". Experimental Cell Research . 313 (15): 3167–3174. doi :10.1016/j.yexcr.2007.05.028. PMID 17643425.
- Levy L, Howell M, Das D, Harkin S, Episkopou V, Hill CS (septiembre de 2007). "Arkadia activa la transcripción dependiente de Smad3/Smad4 al desencadenar la degradación de SnoN inducida por señal". Biología molecular y celular . 27 (17): 6068–6083. doi :10.1128/MCB.00664-07. PMC 1952153 . PMID 17591695.
- Grijelmo C, Rodrigue C, Svrcek M, Bruyneel E, Hendrix A, de Wever O, et al. (agosto de 2007). "Actividad proinvasiva de BMP-7 a través de vías de señalización independientes de SMAD4/src y dependientes de ERK/Rac/JNK en células de cáncer de colon". Señalización celular . 19 (8): 1722–1732. doi :10.1016/j.cellsig.2007.03.008. PMID 17478078.
- Sonegawa H, Nukui T, Li DW, Takaishi M, Sakaguchi M, Huh NH (julio de 2007). "Participación del deterioro de la vía mediada por S100C/A11 en la resistencia de las líneas celulares de cáncer escamoso humano a la supresión del crecimiento inducida por TGFbeta". Journal of Molecular Medicine . 85 (7): 753–762. doi :10.1007/s00109-007-0180-7. PMID 17476473. S2CID 15667203.
- Sheikh AA, Vimalachandran D, Thompson CC, Jenkins RE, Nedjadi T, Shekouh A, et al. (junio de 2007). "La expresión de S100A8 en monocitos asociados al cáncer de páncreas está asociada con el estado de Smad4 de las células de cáncer de páncreas". Proteomics . 7 (11): 1929–1940. doi :10.1002/pmic.200700072. PMID 17469085. S2CID 35648264.
- Popović Hadzija M, Korolija M, Jakić Razumović J, Pavković P, Hadzija M, Kapitanović S (abril de 2007). "Mutaciones K-ras y Dpc4 en pancreatitis crónica: serie de casos". Revista médica croata . 48 (2): 218–224. PMC 2080529 . PMID 17436386.
- Losi L, Bouzourene H, Benhattar J (mayo de 2007). "La pérdida de la expresión de Smad4 predice la metástasis hepática en el cáncer colorrectal humano". Oncology Reports . 17 (5): 1095–1099. doi : 10.3892/or.17.5.1095 . PMID 17390050.
- Karlsson G, Blank U, Moody JL, Ehinger M, Singbrant S, Deng CX, et al. (marzo de 2007). "Smad4 es fundamental para la autorrenovación de las células madre hematopoyéticas". The Journal of Experimental Medicine . 204 (3): 467–474. doi :10.1084/jem.20060465. PMC 2137898 . PMID 17353364.
- Takano S, Kanai F, Jazag A, Ijichi H, Yao J, Ogawa H, et al. (marzo de 2007). "Smad4 es esencial para la regulación negativa de la E-cadherina inducida por TGF-beta en la línea celular de cáncer de páncreas PANC-1". Journal of Biochemistry . 141 (3): 345–351. doi :10.1093/jb/mvm039. PMID 17301079.
Enlaces externos
- Entrada en GeneReviews/NCBI/NIH/UW sobre la telangiectasia hemorrágica hereditaria
- Entrada en GeneReviews/NCBI/NIH/UW sobre el síndrome de poliposis juvenil
- Base de datos de variantes del gen SMAD4