Ligasa de ADN 1
| LIG1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | LIG1 , ADN ligasa 1, LIGI, hLig1, IMD96 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | OMIM : 126391; MGI : 101789; HomoloGene : 197; Tarjetas genéticas : LIG1; OMA :LIG1 - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
La ADN ligasa 1, también ADN ligasa I , es una enzima que en los humanos está codificada por el gen LIG1 . La ADN ligasa 1 es la única ADN ligasa eucariota conocida implicada tanto en la replicación como en la reparación del ADN , lo que la convierte en la más estudiada de las ligasas .
Descubrimiento
Se sabía que la replicación del ADN se producía a través de una rotura de doble cadena , pero la enzima responsable de volver a unir las cadenas y su mecanismo de acción eran desconocidos hasta que los laboratorios Lehman, Gellert, Richardson y Hurwitz hicieron importantes contribuciones al descubrimiento de la ADN ligasa en 1967. [5]

Reclutamiento y regulación
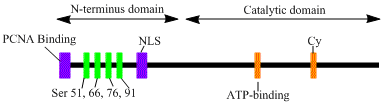
LIG1 codifica una enzima de 120 kDa, de 919 residuos de longitud, conocida como ADN ligasa 1. El polipéptido ADN ligasa 1 contiene una secuencia de dirección de fábrica de replicación N-terminal (RFTS), seguida de una secuencia de localización nuclear (NLS) y tres dominios funcionales. [6] Los tres dominios consisten en un dominio de unión al ADN N-terminal (DBD), una nucleotidiltransferasa catalítica (NTasa) y dominios de unión a oligonucleótidos / oligosacáridos (OB) C-terminales . Aunque el extremo N-terminal del péptido no tiene actividad catalítica, es necesario para la actividad dentro de las células. El extremo N-terminal de la proteína contiene una secuencia de dirección de fábrica de replicación que se utiliza para reclutarla a sitios de replicación de ADN conocidos como fábricas de replicación.
La activación y el reclutamiento de la ADN ligasa 1 parecen estar asociados con modificaciones postraduccionales. El dominio N-terminal se completa a través de la fosforilación de cuatro residuos de serina en este dominio, Ser51, Ser76 y Ser91 por la quinasa dependiente de ciclina (CDK) y Ser66 por la caseína quinasa II (CKII). Se ha demostrado que la fosforilación de estos residuos (Ser66 en particular) posiblemente regula la interacción entre el RFTS y el antígeno nuclear de la célula proliferante (PCNA) cuando la ligasa 1 se recluta a las fábricas de replicación durante la fase S. [6] [7] Rossi et al. propusieron que cuando Ser66 se desfosforila, el RFTS de la ligasa 1 interactúa con el PCNA, lo que fue confirmado in vitro por Tom et al. Ambos conjuntos de datos proporcionan evidencia plausible de que la región N-terminal de la ligasa I desempeña un papel regulador en la función in vivo de las enzimas en el núcleo. [7] [8] Además, se demostró mediante análisis mutacional que la identificación de un motivo de unión a ciclina (Cy) en el dominio catalítico del extremo C desempeña un papel en la fosforilación de las serinas 91 y 76. Juntas, las serinas del extremo N son sustratos de CDK y CKII, que parecen desempeñar un papel regulador importante en el reclutamiento de la ADN ligasa I a la fábrica de replicación durante la fase S del ciclo celular . [6] [9]
Función y mecanismo
LIG1 codifica la ADN ligasa 1, que funciona en la replicación del ADN y en el proceso de reparación por escisión de bases . [10]
La ADN ligasa 1 eucariota cataliza una reacción que es químicamente universal para todas las ligasas. La ADN ligasa 1 utiliza trifosfato de adenosina (ATP) para catalizar los eventos de ligadura energéticamente favorables tanto en la replicación como en la reparación del ADN . Durante la fase de síntesis (fase S) del ciclo celular eucariota , se produce la replicación del ADN. La ADN ligasa 1 es responsable de unir los fragmentos de Okazaki formados durante la síntesis de ADN discontinua en la hebra rezagada del ADN después de que la ADN polimerasa δ haya reemplazado los nucleótidos cebadores del ARN con nucleótidos de ADN. Si los fragmentos de Okazaki no se ligan correctamente, el ADN no ligado (que contiene una "mella") podría degradarse fácilmente hasta una rotura de doble hebra , un fenómeno que se sabe que causa mutaciones genéticas. Para ligar estos fragmentos, la ligasa progresa a través de tres pasos:
- La adición de un grupo monofosfato de adenosina (AMP) a la enzima, denominada adenililación,
- Transferencia de monofosfato de adenosina al ADN y
- Sellado de niquel, o formación de enlaces fosfodiéster. [8] [11]

Durante la adenililación , se produce un ataque nucleofílico al fosfato alfa del ATP de una lisina catalítica que da como resultado la producción de pirofosfato inorgánico (PPi) y un intermediario lisina-AMP unido covalentemente en el sitio activo de la ADN ligasa 1.
Durante el paso de transferencia de AMP, la ADN ligasa se asocia con el ADN, localiza una muesca y cataliza una reacción en el sitio de fosfato 5' de la muesca del ADN. Un oxígeno aniónico en el sitio de fosfato 5' de la muesca del ADN actúa como nucleófilo, atacando el fosfato alfa del AMP unido covalentemente, lo que hace que el AMP sea un intermediario unido covalentemente (intermediario ADN-AMP).
Para que se forme el enlace fosfodiéster, el intermediario ADN-AMP debe ser escindido. Para lograr esta tarea, se produce un ataque nucleofílico sobre el fosfato 5' desde el hidroxilo 3' aguas arriba, lo que da como resultado la formación del enlace fosfodiéster. Durante este ataque nucleofílico, el grupo AMP es expulsado del fosfato 5' como grupo saliente, lo que permite que la muesca se selle y se libere el AMP, completando así un ciclo de ligadura del ADN.
En condiciones subóptimas, la ligasa puede disociarse del ADN antes de que se complete la reacción completa. Se ha demostrado que los niveles de magnesio pueden ralentizar el proceso de sellado de la muesca, lo que hace que la ligasa se disocie del ADN, dejando un intermediario adenililado abortado incapaz de ser fijado sin la ayuda de una fosfodiesterasa . Se ha demostrado que la aprataxina (una fosfodiesterasa) actúa sobre intermediarios de ADN abortados a través de la hidrólisis del enlace AMP-fosfato, restaurando el ADN a su estado inicial antes de que la ligasa hubiera reaccionado. [12] [13]
Papel en la reparación de la base dañada
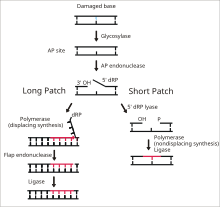
La ADN ligasa 1 funciona para ligar las roturas de ADN monocatenario en el paso final de la vía de reparación por escisión de bases (BER). [14] Las bases nitrogenadas del ADN suelen dañarse por peligros ambientales como especies reactivas de oxígeno , toxinas y radiación ionizante . BER es la principal vía de reparación responsable de escindir y reemplazar las bases dañadas. La ligasa I está involucrada en la vía LP-BER, mientras que la ligasa III está involucrada en la principal vía SN-BER (2). [15] LP-BER procede en 4 pasos catalíticos. Primero, una ADN glicosilasa escinde el enlace N-glucosídico , liberando la base dañada y creando un sitio AP, un sitio que carece de una base de purina o pirimidina . En el siguiente paso, una endonucleasa AP crea una muesca en el extremo 5' del sitio AP, generando un residuo de fosfato de desoxirribosa (dRP) colgante en lugar del sitio AP. La ADN polimerasa sintetiza varias bases nuevas en la dirección 5' a 3', generando un tramo colgante de ADN con el dRP en su extremo 5'. Es en este paso que SN-BER y LP-BER divergen en mecanismo: en SNBER, solo se agrega un único nucleótido y la ADN polimerasa actúa como una liasa para escindir el sitio AP. En LP-BER, se sintetizan varias bases, generando un colgajo colgante de ADN, que es cortado por una endonucleasa de colgajo . Esto deja atrás una cadena de ADN mellada que es detectada y ligada por la ADN ligasa. [14] [15] [16] La acción de la ligasa 1 es estimulada por otras enzimas LP-BER, particularmente la AP-endonucleasa y la ADN polimerasa. [16]
Importancia clínica
Las mutaciones en LIG1 que conducen a una deficiencia de la ADN ligasa 1 resultan en inmunodeficiencia y mayor sensibilidad a los agentes que dañan el ADN. [10]
Existen informes raros de pacientes que presentan deficiencia de ligasa 1 que resultó de alelos mutantes heredados. El primer caso se manifestó como retraso en el crecimiento y desarrollo y una inmunodeficiencia. Se creó un modelo de ratón basado en líneas celulares derivadas del paciente, lo que confirmó que la ligasa mutante confiere errores de replicación que conducen a inestabilidad genómica . Cabe destacar que los ratones mutantes también mostraron aumentos en la tumorigénesis . [8] Se informaron las características moleculares, celulares y clínicas de 5 pacientes de 3 familias con mutaciones bialélicas. Los pacientes exhibieron hipogammaglobulinemia, linfopenia, mayores proporciones de células γδT circulantes y glóbulos rojos muy grandes (macrocitosis). La gravedad clínica varió desde una deficiencia leve de anticuerpos hasta una inmunodeficiencia combinada que requirió trasplante de células madre hematopoyéticas. Se demostró que los defectos químicos y de radiación perjudican las vías de reparación del ADN. Los defectos en la ADN ligasa 1 pueden conducir a diferentes formas de deficiencia parcial autosómica recesiva de la ADN ligasa 1 que conducen a una inmunodeficiencia de gravedad variable. [17]
También se ha descubierto que la ligasa I está regulada positivamente en células tumorales en proliferación, a diferencia de las líneas celulares tumorales benignas y las células humanas normales. Además, se ha demostrado que inhibir la expresión de la ligasa I en estas células puede tener un efecto citotóxico, lo que sugiere que los inhibidores de la ligasa I pueden ser agentes quimioterapéuticos viables. [18]
Las deficiencias de aprataxina , una fosfodiesterasa responsable del reacondicionamiento del ADN (después de que la ADN ligasa I anula el intermediario del ADN adenililado), se han vinculado con la neurodegeneración . Esto sugiere que el ADN es incapaz de volver a ingresar a la vía de reparación sin una maquinaria de respaldo adicional para corregir los errores de la ligasa. [13]
Ahora que se conoce bien la estructura del ADN y se han identificado y caracterizado muchos de los componentes necesarios para su manipulación, reparación y uso, los investigadores están empezando a estudiar el desarrollo de maquinaria nanoscópica que se incorporaría a un organismo vivo que tendría la capacidad de tratar enfermedades, combatir el cáncer y liberar medicamentos en función de un estímulo biológico proporcionado por el organismo a la maquinaria nanoscópica. Lo más probable es que se debiera incorporar la ligasa del ADN a una máquina de este tipo. [19]
Referencias
- ^ abc GRCh38: Lanzamiento de Ensembl 89: ENSG00000105486 – Ensembl , mayo de 2017
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000056394 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia PubMed de ratón:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU . .
- ^ Kresge N, Simoni RD, Hill RL (enero de 2007). "Información sobre la unión del ADN: el trabajo de I. Robert Lehman sobre la ligasa del ADN". Journal of Biological Chemistry . 282 (2): e1–e3. doi : 10.1016/S0021-9258(20)73504-0 .
- ^ abc Ferrari G, Rossi R, Arosio D, Vindigni A, Biamonti G, Montecucco A (septiembre de 2003). "Fosforilación dependiente del ciclo celular de la ADN ligasa I humana en los sitios de cinasa dependientes de ciclina". The Journal of Biological Chemistry . 278 (39): 37761–7. doi : 10.1074/jbc.M304462200 . PMID 12851383.
- ^ ab Rossi R, Villa A, Negri C, Scovassi I, Ciarrocchi G, Biamonti G, Montecucco A (octubre de 1999). "La secuencia de destino de la fábrica de replicación/sitio de unión a PCNA es necesaria en G(1) para controlar el estado de fosforilación de la ADN ligasa I". The EMBO Journal . 18 (20): 5745–54. doi :10.1093/emboj/18.20.5745. PMC 1171641 . PMID 10523317.
- ^ abc Ellenberger T, Tomkinson AE (2008). "Ligasas de ADN eucariotas: perspectivas estructurales y funcionales". Revisión anual de bioquímica . 77 : 313–38. doi :10.1146/annurev.biochem.77.061306.123941. PMC 2933818 . PMID 18518823.
- ^ Prigent C, Lasko DD, Kodama K, Woodgett JR, Lindahl T (agosto de 1992). "Activación de la ligasa I de ADN de mamíferos a través de la fosforilación por la caseína quinasa II". The EMBO Journal . 11 (8): 2925–33. doi :10.1002/j.1460-2075.1992.tb05362.x. PMC 556774 . PMID 1639065.
- ^ ab "Gen Entrez: LIG1 ligasa I, ADN, dependiente de ATP".
- ^ Sriskanda V, Shuman S (enero de 1998). "Ligasa de ADN del virus Chlorella: reconocimiento de muescas y análisis mutacional". Nucleic Acids Research . 26 (2): 525–31. doi :10.1093/nar/26.2.525 (inactivo el 1 de noviembre de 2024). PMC 147278 . PMID 9421510.
{{cite journal}}: CS1 maint: DOI inactivo a partir de noviembre de 2024 ( enlace ) - ^ Taylor MR, Conrad JA, Wahl D, O'Brien PJ (julio de 2011). "El mecanismo cinético de la ligasa I de ADN humana revela cambios dependientes del magnesio en el paso limitante de la velocidad que comprometen la eficiencia de la ligación". The Journal of Biological Chemistry . 286 (26): 23054–62. doi : 10.1074/jbc.M111.248831 . PMC 3123073 . PMID 21561855.
- ^ ab Rass U, Ahel I, West SC (marzo de 2007). "Acciones de la aprataxina en múltiples vías de reparación del ADN". The Journal of Biological Chemistry . 282 (13): 9469–74. doi : 10.1074/jbc.M611489200 . PMID 17276982.
- ^ ab Sattler U, Frit P, Salles B, Calsou P (abril de 2003). "Síntesis de reparación de ADN de parche largo durante la reparación por escisión de bases en células de mamíferos". EMBO Reports . 4 (4): 363–7. doi :10.1038/sj.embor.embor796. PMC 1319152 . PMID 12671676.
- ^ ab Hegde ML, Hazra TK, Mitra S (enero de 2008). "Primeros pasos en la vía de reparación de la interrupción de cadena sencilla/escisión de bases de ADN en células de mamíferos". Cell Research . 18 (1): 27–47. doi :10.1038/cr.2008.8. PMC 2692221 . PMID 18166975.
- ^ ab Balakrishnan L, Brandt PD, Lindsey-Boltz LA, Sancar A, Bambara RA (mayo de 2009). "La reparación por escisión de la base de parche largo se lleva a cabo mediante la estimulación coordinada del complejo de reparación de ADN multienzimático". The Journal of Biological Chemistry . 284 (22): 15158–72. doi : 10.1074/jbc.M109.000505 . PMC 2685697 . PMID 19329425.
- ^ Maffucci P, Chavez J, Jurkiw TJ, O'Brien PJ, Abbott JK, Reynolds PR, et al. (diciembre de 2018). "Las mutaciones bialélicas en la ADN ligasa 1 son la base de un espectro de deficiencias inmunitarias". The Journal of Clinical Investigation . 128 (12): 5489–5504. doi :10.1172/JCI99629. PMC 6264644 . PMID 30395541.
- ^ Sun D, Urrabaz R, Nguyen M, Marty J, Stringer S, Cruz E, et al. (diciembre de 2001). "Expresión elevada de la ADN ligasa I en cánceres humanos". Clinical Cancer Research . 7 (12): 4143–8. PMID 11751514.
- ^ Macdonald J (noviembre de 2008). "ADN inteligente: programación de la molécula de la vida para el trabajo y el ocio [vista previa]". scientificamerican . Consultado el 22 de febrero de 2013 .
Lectura adicional
- Leonhardt H, Cardoso MC (1996). "Orientación y asociación de proteínas con dominios funcionales en el núcleo: la solución insoluble". Organización estructural y funcional de la matriz nuclear . Revista internacional de citología. Vol. 162B. págs. 303–35. doi :10.1016/S0074-7696(08)62620-0. ISBN . 978-0-12-364566-1. Número de identificación personal 8557490.
- Tomkinson AE, Mackey ZB (febrero de 1998). "Estructura y función de las ligasas de ADN de mamíferos". Mutation Research . 407 (1): 1–9. doi :10.1016/s0921-8777(97)00050-5. PMID 9539976.
- Perrigot M, Pierrot-Deseilligny E, Bussel B, Held JP (abril de 1976). "[Parálisis después de radiculografía con Dimer X]". La Nouvelle Presse Médicale . 5 (17): 1120–2. PMID 934827.
- Webster AD, Barnes DE, Arlett CF, Lehmann AR, Lindahl T (junio de 1992). "Retraso del crecimiento e inmunodeficiencia en un paciente con mutaciones en el gen de la ADN ligasa I". Lancet . 339 (8808): 1508–9. doi :10.1016/0140-6736(92)91266-B. PMID 1351188. S2CID 11874717.
- Barnes DE, Tomkinson AE, Lehmann AR, Webster AD, Lindahl T (mayo de 1992). "Mutaciones en el gen de la ADN ligasa I de un individuo con inmunodeficiencias e hipersensibilidad celular a agentes que dañan el ADN". Cell . 69 (3): 495–503. doi :10.1016/0092-8674(92)90450-Q. PMID 1581963. S2CID 11736507.
- Barnes DE, Kodama K, Tynan K, Trask BJ, Christensen M, De Jong PJ, et al. (enero de 1992). "Asignación del gen que codifica la ADN ligasa I al cromosoma humano 19q13.2-13.3". Genomics . 12 (1): 164–6. doi :10.1016/0888-7543(92)90422-O. PMID 1733856.
- Petrini JH, Huwiler KG, Weaver DT (septiembre de 1991). "Un gen de la ADN ligasa I de tipo salvaje se expresa en células del síndrome de Bloom". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 88 (17): 7615–9. Bibcode :1991PNAS...88.7615P. doi : 10.1073/pnas.88.17.7615 . PMC 52352 . PMID 1881902.
- Lasko DD, Tomkinson AE, Lindahl T (julio de 1990). "Ligasas de ADN de mamíferos. Biosíntesis y localización intracelular de la ligasa I de ADN". The Journal of Biological Chemistry . 265 (21): 12618–22. doi : 10.1016/S0021-9258(19)38388-7 . PMID 2197279.
- Barnes DE, Johnston LH, Kodama K, Tomkinson AE, Lasko DD, Lindahl T (septiembre de 1990). "ADN ligasa I humana: clonación y expresión funcional en Saccharomyces cerevisiae". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 87 (17): 6679–83. Bibcode :1990PNAS...87.6679B. doi : 10.1073/pnas.87.17.6679 . PMC 54600 . PMID 2204063.
- Montecucco A, Savini E, Weighardt F, Rossi R, Ciarrocchi G, Villa A, Biamonti G (noviembre de 1995). "El dominio N-terminal de la ADN ligasa I humana contiene la señal de localización nuclear y dirige la enzima a los sitios de replicación del ADN". The EMBO Journal . 14 (21): 5379–86. doi :10.1002/j.1460-2075.1995.tb00222.x. PMC 394647 . PMID 7489727.
- Maruyama K, Sugano S (enero de 1994). "Oligo-capping: un método simple para reemplazar la estructura de capuchón de los ARNm eucariotas con oligorribonucleótidos". Gene . 138 (1–2): 171–4. doi :10.1016/0378-1119(94)90802-8. PMID 8125298.
- Trask B, Fertitta A, Christensen M, Youngblom J, Bergmann A, Copeland A, et al. (enero de 1993). "Mapeo de hibridación in situ con fluorescencia del cromosoma humano 19: ubicación de la banda citogenética de 540 cósmidos y 70 genes o marcadores de ADN". Genomics . 15 (1): 133–45. doi :10.1006/geno.1993.1021. PMID 8432525.
- Petrini JH, Walsh ME, DiMare C, Chen XN, Korenberg JR, Weaver DT (septiembre de 1995). "Aislamiento y caracterización del homólogo humano MRE11". Genomics . 29 (1): 80–6. doi :10.1006/geno.1995.1217. PMID 8530104.
- Bentley D, Selfridge J, Millar JK, Samuel K, Hole N, Ansell JD, Melton DW (agosto de 1996). "La ADN ligasa I es necesaria para la eritropoyesis hepática fetal, pero no es esencial para la viabilidad de las células de los mamíferos". Nature Genetics . 13 (4): 489–91. doi :10.1038/ng0896-489. PMID 8696349. S2CID 20264173.
- Suzuki Y, Yoshitomo-Nakagawa K, Maruyama K, Suyama A, Sugano S (octubre de 1997). "Construcción y caracterización de una biblioteca de ADNc enriquecida en longitud completa y enriquecida en el extremo 5'". Gene . 200 (1–2): 149–56. doi :10.1016/S0378-1119(97)00411-3. PMID 9373149.
- Rossi R, Villa A, Negri C, Scovassi I, Ciarrocchi G, Biamonti G, Montecucco A (octubre de 1999). "La secuencia de orientación de la fábrica de replicación/sitio de unión a PCNA es necesaria en G(1) para controlar el estado de fosforilación de la ADN ligasa I". The EMBO Journal . 18 (20): 5745–54. doi :10.1093/emboj/18.20.5745. PMC 1171641 . PMID 10523317.
- Matsumoto Y, Kim K, Hurwitz J, Gary R, Levin DS, Tomkinson AE, Park MS (noviembre de 1999). "Reconstitución de la reparación dependiente de antígeno nuclear de células proliferantes de sitios apurínicos/apirimidínicos con proteínas humanas purificadas". The Journal of Biological Chemistry . 274 (47): 33703–8. doi : 10.1074/jbc.274.47.33703 . PMID 10559261.
- Vispé S, Satoh MS (septiembre de 2000). "Formación de rotura de ADN de doble cadena mediada por parches de reparación de ADN en células humanas". The Journal of Biological Chemistry . 275 (35): 27386–92. doi : 10.1074/jbc.M003126200 . PMID 10827190.







