Metilación de histonas
La metilación de histonas es un proceso por el cual los grupos metilo se transfieren a los aminoácidos de las proteínas histonas que forman los nucleosomas , alrededor de los cuales se enrolla la doble hélice del ADN para formar los cromosomas . La metilación de las histonas puede aumentar o disminuir la transcripción de genes, dependiendo de qué aminoácidos de las histonas estén metilados y de cuántos grupos metilo estén unidos. Los eventos de metilación que debilitan las atracciones químicas entre las colas de las histonas y el ADN aumentan la transcripción porque permiten que el ADN se desenrolle de los nucleosomas para que las proteínas de los factores de transcripción y la ARN polimerasa puedan acceder al ADN. Este proceso es fundamental para la regulación de la expresión génica que permite que diferentes células expresen diferentes genes.
Función
La metilación de histonas, como mecanismo para modificar la estructura de la cromatina , está asociada con la estimulación de vías neuronales que se sabe que son importantes para la formación de memorias a largo plazo y el aprendizaje. [1] La metilación de histonas es crucial para casi todas las fases del desarrollo embrionario animal . [2]
Los modelos animales han demostrado que la metilación y otros mecanismos de regulación epigenética están asociados con condiciones de envejecimiento, enfermedades neurodegenerativas y discapacidad intelectual [1] ( síndrome de Rubinstein-Taybi , discapacidad intelectual ligada al cromosoma X ). [3] La desregulación de H3K4, H3K27 y H4K20 está asociada con cánceres . [4] Esta modificación altera las propiedades del nucleosoma y afecta sus interacciones con otras proteínas, particularmente en lo que respecta a los procesos de transcripción genética.
- La metilación de histonas puede estar asociada con la represión o activación transcripcional . Por ejemplo, la trimetilación de la histona H3 en la lisina 4 ( H3K4me3 ) es una marca activa para la transcripción y se regula positivamente en el hipocampo una hora después del condicionamiento del miedo contextual en ratas. Sin embargo, la dimetilación de la histona H3 en la lisina 9 ( H3K9me2 ), una señal para el silenciamiento transcripcional, aumenta después de la exposición al condicionamiento del miedo o a un entorno nuevo solo. [5]
- La metilación de algunos residuos de lisina (K) y arginina (R) de las histonas da como resultado la activación transcripcional. Algunos ejemplos incluyen la metilación de la lisina 4 de la histona 3 ( H3K4me1 ) y de los residuos de arginina (R) en H3 y H4 .
- La adición de grupos metilo a las histonas por las metiltransferasas de histonas puede activar o reprimir aún más la transcripción, dependiendo del aminoácido que se esté metilando y de la presencia de otros grupos metilo o acetilo en las proximidades. [6]
- En la mayoría de los casos, en el caso de las metilaciones de lisina de histonas, las metiltransferasas de histonas (que metilan) se caracterizan como "escritoras", mientras que las desmetilasas se caracterizan como "borradores". [2] [4]
Mecanismo
La unidad fundamental de la cromatina , llamada nucleosoma , contiene ADN enrollado alrededor de un octámero proteico . Este octámero consta de dos copias de cada una de las cuatro proteínas histonas: H2A , H2B , H3 y H4 . Cada una de estas proteínas tiene una extensión de cola, y estas colas son los objetivos de la modificación del nucleosoma por metilación. La activación o inactivación del ADN depende en gran medida del residuo de cola específico metilado y su grado de metilación. Las histonas pueden metilarse solo en residuos de lisina (K) y arginina (R), pero la metilación se observa más comúnmente en residuos de lisina de las colas de histonas H3 y H4. [7] El extremo de la cola más alejado del núcleo del nucleosoma es el N-terminal (los residuos se numeran comenzando en este extremo). Los sitios comunes de metilación asociados con la activación genética incluyen H3K4, H3K48 y H3K79. Los sitios comunes para la inactivación de genes incluyen H3K9 y H3K27. [8] Los estudios de estos sitios han encontrado que la metilación de las colas de histonas en diferentes residuos sirven como marcadores para el reclutamiento de varias proteínas o complejos proteicos que sirven para regular la activación o inactivación de la cromatina.
Tanto los residuos de lisina como los de arginina contienen grupos amino, que les confieren características básicas e hidrofóbicas. La lisina puede mono-, di- o trimetilarse con un grupo metilo que reemplaza cada hidrógeno de su grupo NH3+. Con un grupo NH2 y NH2+ libre, la arginina puede mono- o dimetilarse. Esta dimetilación puede ocurrir asimétricamente en el grupo NH2 o simétricamente con una metilación en cada grupo. [9] Cada adición de un grupo metilo en cada residuo requiere un conjunto específico de enzimas proteicas con varios sustratos y cofactores. Generalmente, la metilación de un residuo de arginina requiere un complejo que incluye la proteína arginina metiltransferasa (PRMT), mientras que la lisina requiere una histona metiltransferasa (HMT) específica, que generalmente contiene un dominio SET conservado evolutivamente. [10]
Diferentes grados de metilación de residuos pueden conferir diferentes funciones, como se ejemplifica en la metilación del residuo H4K20 comúnmente estudiado. El H4K20 monometilado ( H4K20me 1) está involucrado en la compactación de la cromatina y, por lo tanto, en la represión transcripcional. Sin embargo, el H4K20me2 es vital en la reparación del ADN dañado. Cuando está dimetilado, el residuo proporciona una plataforma para la unión de la proteína 53BP1 involucrada en la reparación de roturas de ADN bicatenario mediante unión de extremos no homólogos. Se observa que el H4K20me3 se concentra en la heterocromatina y se observan reducciones en esta trimetilación en la progresión del cáncer. Por lo tanto, el H4K20me3 cumple una función adicional en la represión de la cromatina. [10] La reparación de roturas de doble cadena del ADN en la cromatina también ocurre por recombinación homóloga y también involucra la metilación de histonas ( H3K9me3 ) para facilitar el acceso de las enzimas reparadoras a los sitios de daño. [11]
Histona metiltransferasa

El genoma está fuertemente condensado en cromatina, que debe aflojarse para que se produzca la transcripción . Para detener la transcripción de un gen, el ADN debe enrollarse más fuertemente. Esto se puede hacer modificando las histonas en ciertos sitios mediante metilación. Las metiltransferasas de histonas son enzimas que transfieren grupos metilo de la S-adenosil metionina (SAM) a los residuos de lisina o arginina de las histonas H3 y H4. También hay casos en los que los dominios globulares centrales de las histonas están metilados.
Las metiltransferasas de histonas son específicas de la lisina o la arginina. Las transferasas específicas de la lisina se dividen en dos grupos: si tienen un dominio SET o si tienen un dominio no SET. Estos dominios especifican exactamente cómo la enzima cataliza la transferencia del metilo desde SAM a la proteína de transferencia y luego al residuo de histona. [12] Las metiltransferasas pueden agregar de 1 a 3 metilos a los residuos objetivo.
Estos metilos que se añaden a las histonas actúan para regular la transcripción bloqueando o favoreciendo el acceso del ADN a los factores de transcripción. De esta manera, la integridad del genoma y la herencia epigenética de los genes están bajo el control de las acciones de las metiltransferasas de las histonas. La metilación de las histonas es clave para distinguir la integridad del genoma y los genes que expresan las células, dándoles así su identidad.
Las histonas metiladas pueden reprimir o activar la transcripción. [12] Por ejemplo, mientras que H3K4me2, H3K4me3 y H3K79me3 generalmente se asocian con la actividad transcripcional, mientras que H3K9me2 , H3K9me3 , H3K27me2, H3K27me3 y H4K20me3 se asocian con la represión transcripcional. [13]
Epigenética
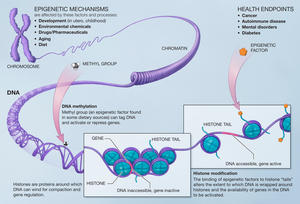
Las modificaciones que se realizan en las histonas tienen un efecto sobre los genes que se expresan en una célula, como ocurre cuando las metiltransferasas de las histonas añaden metilos a los residuos de histonas. [14] La metilación de las histonas desempeña un papel importante en el ensamblaje del mecanismo de la heterocromatina y en el mantenimiento de los límites genéticos entre los genes que se transcriben y los que no. Estos cambios se transmiten a la progenie y pueden verse afectados por el entorno al que están sujetas las células. Las alteraciones epigenéticas son reversibles, lo que significa que pueden ser objetivos de terapia.
Las actividades de las histonas metiltransferasas se ven contrarrestadas por la actividad de las histonas desmetilasas, lo que permite activar o desactivar la transcripción revirtiendo las modificaciones preexistentes. Es necesario que las actividades tanto de las histonas metiltransferasas como de las histonas desmetilasas estén reguladas estrictamente. La regulación incorrecta de cualquiera de ellas puede conducir a la expresión de genes que conducen a una mayor susceptibilidad a las enfermedades. Muchos cánceres surgen de los efectos epigenéticos inadecuados de la metilación mal regulada. [15] Sin embargo, debido a que estos procesos a veces son reversibles, existe interés en utilizar sus actividades en conjunto con terapias contra el cáncer. [15]
En la inactivación del cromosoma X
En los organismos femeninos, un espermatozoide que contiene un cromosoma X fertiliza el óvulo, lo que le da al embrión dos copias del cromosoma X. Las hembras, sin embargo, inicialmente no requieren ambas copias del cromosoma X, ya que solo duplicaría la cantidad de productos proteicos transcritos, como lo demuestra la hipótesis de la compensación de la dosis. El cromosoma X paterno se inactiva rápidamente durante las primeras divisiones. [16] Este cromosoma X inactivo (Xi) se empaqueta en una forma increíblemente compacta de cromatina llamada heterocromatina . [17] Este empaquetamiento ocurre debido a la metilación de los diferentes residuos de lisina que ayudan a formar diferentes histonas. En los humanos, la inactivación de X es un proceso aleatorio, que está mediado por el ARN no codificante XIST. [18]
Aunque la metilación de los residuos de lisina se produce en muchas histonas diferentes, la más característica de Xi se produce en la novena lisina de la tercera histona (H3K9). Si bien una única metilación de esta región permite que los genes unidos permanezcan transcripcionalmente activos, [19] en la heterocromatina este residuo de lisina suele metilarse dos o tres veces, H3K9me2 o H3K9me3 respectivamente, para garantizar que el ADN unido esté inactivo. Investigaciones más recientes han demostrado que H3K27me3 y H4K20me1 también son comunes en embriones tempranos. Otras marcas de metilación asociadas con áreas transcripcionalmente activas del ADN, H3K4me2 y H3K4me3, faltan en el cromosoma Xi junto con muchas marcas de acetilación. Aunque se sabía que ciertas marcas de metilación de la histona Xi se mantenían relativamente constantes entre especies, recientemente se ha descubierto que diferentes organismos e incluso diferentes células dentro de un solo organismo pueden tener diferentes marcas para su inactivación de X. [20] A través de la metilación de histonas se produce la impronta genética , de modo que el mismo homólogo X permanece inactivado a través de las replicaciones cromosómicas y las divisiones celulares.
Mutaciones
Debido a que la metilación de histonas regula gran parte de los genes que se transcriben, incluso cambios leves en los patrones de metilación pueden tener efectos nefastos en el organismo. Las mutaciones que se producen para aumentar o disminuir la metilación tienen grandes cambios en la regulación genética, mientras que las mutaciones en enzimas como la metiltransferasa y la desmetiltransferasa pueden alterar por completo qué proteínas se transcriben en una célula determinada. La metilación excesiva de un cromosoma puede provocar que ciertos genes necesarios para el funcionamiento normal de la célula se inactiven. En una determinada cepa de levadura, Saccharomyces cerevisiae , una mutación que hace que tres residuos de lisina en la tercera histona, H3K4, H3K36 y H3K79, se metilen provoca un retraso en el ciclo celular mitótico, ya que muchos genes necesarios para esta progresión se inactivan. Esta mutación extrema conduce a la muerte del organismo. Se ha descubierto que la eliminación de genes que eventualmente permitirán la producción de histona metiltransferasa permite que este organismo viva ya que sus residuos de lisina no están metilados. [21]
En los últimos años, los investigadores se han dado cuenta de que muchos tipos de cáncer se deben en gran medida a factores epigenéticos. El cáncer puede ser causado de diversas maneras debido a la metilación diferencial de las histonas. Desde el descubrimiento de los oncogenes , así como de los genes supresores de tumores , se sabe que un factor importante que causa y reprime el cáncer se encuentra dentro de nuestro propio genoma. Si las áreas alrededor de los oncogenes se desmetilan, estos genes causantes de cáncer tienen el potencial de transcribirse a un ritmo alarmante. Lo opuesto a esto es la metilación de los genes supresores de tumores. En los casos en que las áreas alrededor de estos genes estaban altamente metiladas, el gen supresor de tumores no estaba activo y, por lo tanto, era más probable que se produjera el cáncer. Estos cambios en el patrón de metilación a menudo se deben a mutaciones en la metiltransferasa y la desmetiltransferasa. [22] Otros tipos de mutaciones en proteínas como la isocitrato deshidrogenasa 1 (IDH1) y la isocitrato deshidrogenasa 2 (IDH2) pueden causar la inactivación de la histona desmetiltransferasa, lo que a su vez puede conducir a una variedad de cánceres, gliomas y leucemias, dependiendo de en qué células se produzca la mutación. [23]
El metabolismo de un carbono modifica la metilación de las histonas
En el metabolismo de un carbono, los aminoácidos glicina y serina se convierten a través de los ciclos de folato y metionina en precursores de nucleótidos y SAM. Múltiples nutrientes alimentan el metabolismo de un carbono, entre ellos la glucosa , la serina, la glicina y la treonina . Los altos niveles del donante de metilo SAM influyen en la metilación de las histonas, lo que puede explicar cómo los altos niveles de SAM previenen la transformación maligna. [24]
Véase también
- Código de histonas
- Acetilación y desacetilación de histonas
- Histona metiltransferasa
- Metilación
- Metillisina
- Impronta genética
- Metilación del ADN
Referencias
- ^ ab Kramer, Jamie M. (febrero de 2013). "Regulación epigenética de la memoria: implicaciones en los trastornos cognitivos humanos". Conceptos biomoleculares . 4 (1): 1–12. doi :10.1515/bmc-2012-0026. hdl : 2066/118379 . PMID 25436561. S2CID 6195513.
- ^ ab Jambhekar A, Dhall A, Shi Y (2019). "Funciones y regulación de la metilación de histonas en el desarrollo animal". Nature Reviews Molecular Cell Biology . 20 (10): 625–641. doi :10.1038/s41580-019-0151-1. PMC 6774358 . PMID 31267065.
- ^ Lilja, Tobias; Nina Heldring; Ola Hermanson (febrero de 2013). "Como una histona rodante: regulación epigenética de las células madre neuronales y el desarrollo cerebral por factores que controlan la acetilación y metilación de las histonas". Biochimica et Biophysica Acta (BBA) - Temas generales . 1830 (2): 2354–2360. doi : 10.1016/j.bbagen.2012.08.011 . PMID 22986149.
- ^ ab Hyun K, Jeon J, Park K, Kim J (2017). "Escritura, borrado y lectura de metilaciones de lisina de histonas". Medicina experimental y molecular . 49 (4): e324. doi :10.1038/emm.2017.11. PMC 6130214. PMID 28450737 .
- ^ Gupta, Swati; Se Y. Kim; Sonja Artis; David L. Molfese; Armin Schumacher; J. David Sweatt; Richard E. Paylor; Farah D. Lubin (10 de marzo de 2010). "La metilación de histonas regula la formación de la memoria". The Journal of Neuroscience . 30 (10): 3589–3599. doi :10.1523/JNEUROSCI.3732-09.2010. PMC 2859898 . PMID 20219993.
- ^ Greer, Eric L.; Shi, Yang (2012). "Metilación de histonas: una marca dinámica en la salud, la enfermedad y la herencia". Nature Reviews Genetics . 13 (5): 343–57. doi :10.1038/nrg3173. PMC 4073795 . PMID 22473383.
- ^ Jia, Songtao; Wang, Yu (2009). "Los títulos marcan la diferencia" (PDF) . Epigenética . 4 (5): 1–4. Archivado desde el original (PDF) el 1 de marzo de 2011 . Consultado el 3 de abril de 2013 .
- ^ Gilbert, SF (2010). Biología del desarrollo. (9.ª ed.). Sinauer Associates, Inc. 35-37.
- ^ Blanc, Roméo S; Richard, Stéphane (5 de enero de 2017). "Metilación de la arginina: la mayoría de edad". Molecular Cell . 65 (1): 8–24. doi : 10.1016/j.molcel.2016.11.003 . PMID 28061334.
- ^ ab Reinberg, Danny; Zhang, Yi (2001). "Regulación de la transcripción por metilación de histonas: interacción entre diferentes modificaciones covalentes de las colas de las histonas centrales". Genes & Development . 15 (18): 2343–2360. doi : 10.1101/gad.927301 . PMID 11562345.
- ^ Wei S, Li C, Yin Z, Wen J, Meng H, Xue L, Wang J (2018). "Metilación de histonas en la reparación del ADN y la práctica clínica: nuevos hallazgos durante los últimos 5 años". J Cancer . 9 (12): 2072–2081. doi :10.7150/jca.23427. PMC 6010677 . PMID 29937925.
- ^ ab Rice, Judd C.; Briggs, Scott D.; Ueberheide, Beatrix; Barber, Cynthia M.; Shabanowitz, Jeffrey; Hunt, Donald F.; Shinkai, Yoichi; Allis, C. David (2003). "Las histonas metiltransferasas dirigen diferentes grados de metilación para definir dominios de cromatina distintos". Molecular Cell . 12 (6): 1591–1598. doi : 10.1016/S1097-2765(03)00479-9 . PMID 14690610.
- ^ Kooistra, Susanne Marije; Helin, Kristian (4 de abril de 2012). "Mecanismos moleculares y funciones potenciales de las desmetilasas de histonas". Nature Reviews Molecular Cell Biology . 13 (5): 297–311. doi :10.1038/nrm3327. ISSN 1471-0072. PMID 22473470. S2CID 26181430.
- ^ Cheung, Peter; Lau, Priscilla (2005). "Regulación epigenética por metilación de histonas y variantes de histonas". Endocrinología molecular . 19 (3): 563–73. doi : 10.1210/me.2004-0496 . PMID 15677708.
- ^ ab Albert, Mareike; Helin, Kristian (2010). "Histonas metiltransferasas en cáncer". Seminarios en biología celular y del desarrollo . 21 (2): 209–220. doi :10.1016/j.semcdb.2009.10.007. PMID 19892027.
- ^ Akagi, Nobuo; Sasaki, Motomichi (1975). "Inactivación preferencial del cromosoma X de origen paterno en las membranas extraembrionarias del ratón". Nature . 256 (5519): 640–42. Bibcode :1975Natur.256..640T. doi :10.1038/256640a0. PMID 1152998. S2CID 4190616.
- ^ Elgin, Sarah CR; Grewal, Shiv IS (2003). "Heterocromatina: el silencio es oro". Current Biology . 13 (23): R895–898. doi : 10.1016/j.cub.2003.11.006 . PMID 14654010. S2CID 9728548.
- ^ Ahn, J.; Lee, J. (2008). "Cromosoma X: inactivación del cromosoma X". Nature Education . 1 (1): 24.
- ^ Barski, A.; Cuddapah, S.; Cui, K.; Roh, T.; Schones, D.; Wang, Z.; Wei, G.; Chepelev, I.; Zhao, K. (2007). "Perfiles de alta resolución de metilaciones de histonas en el genoma humano". Celúla . 129 (4): 823–37. doi : 10.1016/j.cell.2007.05.009 . PMID 17512414. S2CID 6326093.
- ^ Chow, Jennifer; Heard, Edith (2009). "Inactivación del cromosoma X y las complejidades del silenciamiento de un cromosoma sexual". Current Opinion in Cell Biology . 21 (3): 359–66. doi :10.1016/j.ceb.2009.04.012. PMID 19477626.
- ^ Jin, Y.; Rodríguez, AM; Stanton, JD; Kitazono, AA; Wyrick, JJ (2007). "La mutación simultánea de residuos de lisina metilada en la histona H3 provoca un mayor silenciamiento génico, defectos del ciclo celular y letalidad celular en Saccharomyces Cerevisiae". Biología molecular y celular . 27 (19): 6832–841. doi :10.1128/mcb.00745-07. PMC 2099221 . PMID 17664279.
- ^ Esteller, M (2006). "La epigenética proporciona una nueva generación de oncogenes y genes supresores de tumores". British Journal of Cancer . 94 (2): 179–83. doi :10.1038/sj.bjc.6602918. PMC 2361113 . PMID 16404435.
- ^ Lu, Chao; Ward, Patrick S.; Kapoor, Gurpreet S.; Rohle, Dan; Turcan, Sevin; Abdel-Wahab, Omar; Edwards, Christopher R.; Khanin, Raya; Figueroa, Maria E.; Melnick, Ari; Wellen, Kathryn E.; O'Rourke, Donald M.; Berger, Shelley L.; Chan, Timothy A.; Levine, Ross L.; Mellinghoff, Ingo K.; Thompson, Craig B. (2012). "La mutación de DH altera la desmetilación de histonas y da como resultado un bloqueo de la diferenciación celular". Nature . 483 (7390): 474–78. Bibcode :2012Natur.483..474L. doi :10.1038/nature10860. PMC 3478770 . Número de modelo: PMID22343901.
- ^ Wang YP, Lei QY (2018). "Recodificación metabólica de la epigenética en el cáncer". Cancer Commun (Londres) . 38 (1): 1–8. doi : 10.1186/s40880-018-0302-3 . PMC 5993135. PMID 29784032 .
Lectura adicional
- Orouji, Elias y Utikal, Jochen. (2018). Abordaje epigenético del melanoma maligno: metilación de la lisina de la histona. Clinical Epigenetics 2018 10:145 https://clinicalepigeneticsjournal.biomedcentral.com/articles/10.1186/s13148-018-0583-z
- Gozani, O., y Shi, Y. (2014). Metilación de histonas en la señalización de la cromatina. En: Fundamentals of Chromatin (pp. 213–256). Springer New York. doi :10.1007/978-1-4614-8624-4_5
- Shi, Yang; Whetstine, Johnathan R. (12 de enero de 2007). "Regulación dinámica de la metilación de la lisina de la histona por desmetilasas". Molecular Cell . 25 (1): 1–14. doi : 10.1016/j.molcel.2006.12.010 . PMID 17218267.