Helicasa

Las helicasas son una clase de enzimas que se cree que son vitales para todos los organismos . Su función principal es descomprimir el material genético de un organismo . Las helicasas son proteínas motoras que se mueven direccionalmente a lo largo de una cadena principal de ácido nucleico fosfodiéster , separando dos cadenas de ácido nucleico hibridadas (de ahí el nombre de helic- + -asa ), utilizando energía de la hidrólisis de ATP . Hay muchas helicasas, que representan la gran variedad de procesos en los que debe catalizarse la separación de cadenas. Aproximadamente el 1% de los genes eucariotas codifican helicasas. [1]
El genoma humano codifica 95 helicasas no redundantes: 64 helicasas de ARN y 31 helicasas de ADN. [2] Muchos procesos celulares, como la replicación del ADN , la transcripción , la traducción , la recombinación , la reparación del ADN y la biogénesis de los ribosomas, implican la separación de las cadenas de ácidos nucleicos que requieren el uso de helicasas. Algunas helicasas especializadas también participan en la detección de ácidos nucleicos virales durante la infección y cumplen una función inmunológica. Una helicasa es una enzima que desempeña un papel crucial en los procesos de replicación y reparación del ADN. Su función principal es desenrollar la molécula de ADN bicatenario rompiendo los enlaces de hidrógeno entre los pares de bases complementarios, lo que permite que las cadenas de ADN se separen. Esto crea una horquilla de replicación, que sirve como plantilla para sintetizar nuevas cadenas de ADN. La helicasa es un componente esencial de los mecanismos celulares que garantiza la replicación precisa del ADN y el mantenimiento de la información genética. La helicasa del ADN cataliza la regresión. RecG y la enzima PriA trabajan juntas para rebobinar el ADN dúplex, creando una unión Holliday. RecG libera las proteínas unidas y la helicasa PriA facilita la recarga del ADN para reanudar la replicación del ADN. RecG reemplaza a la proteína de unión de cadena sencilla (SSB), que regula los sitios de carga de la horquilla de la helicasa durante la regresión de la horquilla. La proteína SSB interactúa con las helicasas de ADN PriA y RecG para recuperar las horquillas de replicación de ADN estancadas. Estas enzimas deben unirse a la helicasa SSB para cargarse en las horquillas estancadas. El deslizamiento térmico y la unión del dúplex de ADN posiblemente estén respaldados por el dominio de cuña de la asociación de RecG con el enlazador SSB. En una reacción de regresión facilitada por RecG y ATP, se crean uniones Holliday para su posterior procesamiento.
Función
Las helicasas se utilizan a menudo para separar hebras de una doble hélice de ADN o una molécula de ARN autoanclada utilizando la energía de la hidrólisis de ATP , un proceso caracterizado por la ruptura de enlaces de hidrógeno entre bases de nucleótidos ancladas . También funcionan para eliminar proteínas asociadas a ácidos nucleicos y catalizar la recombinación de ADN homólogo . [3] Los procesos metabólicos del ARN, como la traducción, la transcripción, la biogénesis de ribosomas , el empalme de ARN , el transporte de ARN, la edición de ARN y la degradación de ARN, son todos facilitados por las helicasas. [3] Las helicasas se mueven de forma incremental a lo largo de una hebra de ácido nucleico del dúplex con una direccionalidad y procesividad específicas para cada enzima en particular.
Las helicasas adoptan diferentes estructuras y estados de oligomerización . Mientras que las helicasas similares a DnaB desenrollan el ADN como hexámeros en forma de anillo , se ha demostrado que otras enzimas son activas como monómeros o dímeros . Los estudios han demostrado que las helicasas pueden actuar pasivamente, esperando que se produzca el desenrollado no catalizado y luego translocándose entre cadenas desplazadas, [4] o pueden desempeñar un papel activo en la catálisis de la separación de cadenas utilizando la energía generada en la hidrólisis de ATP. [5] En el último caso, la helicasa actúa de manera comparable a un motor activo, desenrollándose y translocándose a lo largo de su sustrato como resultado directo de su actividad ATPasa. [6] Las helicasas pueden procesar mucho más rápido in vivo que in vitro debido a la presencia de proteínas accesorias que ayudan en la desestabilización de la unión en horquilla. [6]

Barrera de activación en la actividad de la helicasa
La acción de la helicasa enzimática, como el desenrollado de los ácidos nucleicos, se logra mediante la reducción de la barrera de activación ( ) de cada acción específica. [7] [5] [8] [9] La barrera de activación es el resultado de varios factores y se puede definir por
dónde
- = número de pares de bases desenrollados (bps),
- = energía libre de formación de pares de bases,
- = reducción de la energía libre debido a la helicasa, y
- = reducción de la energía libre debido a las fuerzas de apertura.
Los factores que contribuyen a la altura de la barrera de activación incluyen: la secuencia de ácido nucleico específica de la molécula involucrada, el número de pares de bases involucrados, la tensión presente en la horquilla de replicación y las fuerzas de desestabilización. [7] [5] [8] [9]
Helicasas activas y pasivas
El tamaño de la barrera de activación a superar por la helicasa contribuye a su clasificación como una helicasa activa o pasiva. En las helicasas pasivas, existe una barrera de activación significativa (definida como , donde es la constante de Boltzmann y es la temperatura del sistema). Debido a esta barrera de activación significativa, su progresión de desenrollado se ve afectada en gran medida por la secuencia de ácidos nucleicos dentro de la molécula a desenrollar y la presencia de fuerzas de desestabilización que actúan sobre la horquilla de replicación. [7] [5] [8] [9] Ciertas combinaciones de ácidos nucleicos disminuirán las tasas de desenrollado (es decir, guanina y citosina ), mientras que varias fuerzas desestabilizadoras pueden aumentar la tasa de desenrollado. [5] [8] [9] En sistemas pasivos, la tasa de desenrollado ( ) es menor que la tasa de translocación ( ) (translocación a lo largo del ácido nucleico monocatenario, ssNA), debido a su dependencia del desenrollado transitorio de los pares de bases en la horquilla de replicación para determinar su tasa de desenrollado. [7] [5] [8] [9]
En las helicasas activas, , donde el sistema carece de una barrera significativa, ya que la helicasa puede desestabilizar los ácidos nucleicos, desenrollando la doble hélice a una velocidad constante, independientemente de la secuencia de ácidos nucleicos. En las helicasas activas, está más cerca de , debido a la capacidad de la helicasa activa de desestabilizar directamente la horquilla de replicación para promover el desenrollado. [7] [5] [8] [9]
Las helicasas activas muestran un comportamiento similar cuando actúan sobre ácidos nucleicos de doble cadena, dsNA o ssNA, en lo que respecta a las tasas de desenrollado y las tasas de translocación, donde en ambos sistemas y son aproximadamente iguales.
Estas dos categorías de helicasas también pueden modelarse como mecanismos. En tales modelos, las helicasas pasivas se conceptualizan como trinquetes brownianos, impulsados por fluctuaciones térmicas y gradientes anisotrópicos subsiguientes a través de la red de ADN. Las helicasas activas, en cambio, se conceptualizan como motores paso a paso, también conocidos como motores de carrera de potencia, que utilizan un mecanismo de "gusano de pulgada" conformacional o un mecanismo de "caminar" mano sobre mano para progresar. [10] Dependiendo del organismo, dicho progreso de travesía de hélice puede ocurrir a velocidades de rotación en el rango de 5000 [11] a 10 000 [12] RPM.
Historia de las helicasas de ADN
Las helicasas de ADN se descubrieron en E. coli en 1976. Esta helicasa se describió como una "enzima desenrolladora de ADN" que "desnaturaliza los dúplex de ADN en una reacción dependiente de ATP, sin degradarse de manera detectable". [13] La primera helicasa de ADN eucariota descubierta fue en 1978 en la planta de lirio. [14] Desde entonces, se descubrieron y aislaron helicasas de ADN en otras bacterias, virus, levaduras, moscas y eucariotas superiores. [15] Hasta la fecha, se han aislado al menos 14 helicasas diferentes de organismos unicelulares, 6 helicasas de bacteriófagos, 12 de virus, 15 de levaduras, 8 de plantas, 11 de timo de ternera y aproximadamente 25 helicasas de células humanas. [16] A continuación se presenta una historia del descubrimiento de helicasas:
- 1976 – Descubrimiento y aislamiento de la helicasa de ADN basada en E. coli [13]
- 1978 – Descubrimiento de las primeras helicasas de ADN eucariotas, aisladas de la planta del lirio [14]
- 1982 – La "proteína T4 gen 41" es la primera helicasa de ADN de bacteriófago de la que se informa [15]
- 1985 – Se aíslan las primeras helicasas de ADN de mamíferos a partir del timo de ternera [17]
- 1986 – Se informa que el antígeno tumoral grande SV40 es una helicasa viral (la primera proteína viral informada que se determinó que sirve como una helicasa de ADN) [18]
- 1986 – Se determina que la ATPasa III, una proteína de levadura, es una helicasa de ADN [19]
- 1988 – Descubrimiento de siete dominios de aminoácidos conservados que se determinó que eran motivos de helicasa
- 1989 – Designación de la superfamilia I y la superfamilia II de las helicasas de ADN [20]
- 1989 – Identificación de la familia de helicasas de caja DEAD [21]
- 1990 – Aislamiento de una helicasa de ADN humana [22]
- 1992 – Aislamiento de la primera helicasa de ADN mitocondrial descrita (de cerebro bovino) [23]
- 1996 – Informe del descubrimiento de la primera helicasa de ADN de cloroplasto purificada del guisante [24]
- 2002 – Aislamiento y caracterización de la primera helicasa de ADN del parásito de la malaria bioquímicamente activa – Plasmodium cynomolgi . [25]
Características estructurales
La función común de las helicasas explica el hecho de que muestren un cierto grado de homología en la secuencia de aminoácidos ; todas poseen motivos de secuencia ubicados en el interior de su estructura primaria , involucrados en la unión de ATP , la hidrólisis de ATP y la translocación a lo largo del sustrato de ácido nucleico . La porción variable de la secuencia de aminoácidos está relacionada con las características específicas de cada helicasa.
La presencia de estos motivos de helicasa permite atribuir una supuesta actividad de helicasa a una proteína determinada, pero no necesariamente confirma que se trata de una helicasa activa. Sin embargo, los motivos conservados sí respaldan una homología evolutiva entre enzimas. Basándose en estos motivos de helicasa, se han distinguido varias superfamilias de helicasas.
Superfamilias
Las helicasas se clasifican en 6 grupos (superfamilias) según sus motivos de secuencia compartidos. [26] Las helicasas que no forman una estructura de anillo se encuentran en las superfamilias 1 y 2, y las helicasas que forman anillos forman parte de las superfamilias 3 a 6. [27] Las helicasas también se clasifican como α o β dependiendo de si funcionan con ADN de cadena simple o doble; las helicasas α funcionan con ADN de cadena simple y las helicasas β funcionan con ADN de cadena doble . También se clasifican por polaridad de translocación. Si la translocación ocurre 3'-5' la helicasa es de tipo A; si la translocación ocurre 5'-3' es de tipo B. [26]
- Superfamilia 1 (SF1) : Esta superfamilia se puede subdividir en helicasas SF1A y SF1B. [26] En este grupo, las helicasas pueden tener polaridad de translocación 3'-5' (subfamilia SF1A) o 5'-3' (subfamilia SF1B). [26] [28] Las helicasas SF1A más conocidas son Rep y UvrD en bacterias gramnegativas y la helicasa PcrA de bacterias grampositivas . [26] Las helicasas más conocidas del grupo SF1B son las helicasas RecD y Dda. [26] Tienen un núcleo de plegamiento similar a RecA. [27]
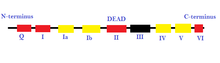
- Superfamilia 2 (SF2) : Este es el grupo más grande de helicasas que están involucradas en diversos procesos celulares. [26] [2] Se caracterizan por la presencia de nueve motivos conservados: Q, I, Ia, Ib y II a VI. [2] Este grupo está compuesto principalmente por helicasas de ARN de caja DEAD. [27] Algunas otras helicasas incluidas en SF2 son la familia similar a RecQ y las enzimas similares a Snf2. [26] La mayoría de las helicasas SF2 son de tipo A con algunas excepciones como la familia XPD. [26] Tienen un núcleo de plegamiento similar a RecA. [27]
- Superfamilia 3 (SF3) : La superfamilia 3 está formada por helicasas AAA+ codificadas principalmente por virus de ADN pequeños y algunos virus de ADN nucleocitoplasmático grandes. [29] [30] Tienen una direccionalidad de translocación 3'-5', lo que significa que todas son helicasas de tipo A. [26] La helicasa SF3 más conocida es la helicasa E1 del virus del papiloma. [26]
- Superfamilia 4 (SF4) : Todas las helicasas de la familia SF4 tienen una polaridad de tipo B (5'-3'). Tienen un pliegue RecA. [26] La helicasa SF4 más estudiada es la gp4 del bacteriófago T7. [26]
- Superfamilia 5 (SF5) : las proteínas Rho conforman el grupo SF5. Tienen un pliegue RecA. [26]
- Superfamilia 6 (SF6) : Contienen el núcleo AAA+ que no está incluido en la clasificación SF3. [26] Algunas proteínas del grupo SF6 son: mantenimiento de mini cromosomas MCM , RuvB, RuvA y RuvC. [26]
Todas las helicasas son miembros de una familia que contiene un bucle P o un motivo Walker .
Trastornos y enfermedades de la helicasa
Mutaciones de la helicasa ATRX
El gen ATRX codifica la helicasa dependiente de ATP, ATRX (también conocida como XH2 y XNP) de la familia del subgrupo SNF2, que se cree que es responsable de funciones como la remodelación de la cromatina, la regulación genética y la metilación del ADN. [31] [32] [33] [34] Estas funciones ayudan a prevenir la apoptosis, lo que resulta en la regulación del tamaño cortical, así como una contribución a la supervivencia de las estructuras hipocampales y corticales, afectando la memoria y el aprendizaje. [31] Esta helicasa se encuentra en el cromosoma X (Xq13.1-q21.1), en la heterocromatina pericentromérica y se une a la proteína heterocromatina 1. [ 31] [33] Los estudios han demostrado que ATRX desempeña un papel en la metilación del ADNr y es esencial para el desarrollo embrionario. [35] Se han encontrado mutaciones en toda la proteína ATRX , y más del 90% de ellas se encuentran en los dominios de dedo de zinc y helicasa. [36] Las mutaciones de ATRX pueden provocar retraso mental asociado a talasemia alfa ligada al cromosoma X ( síndrome ATR-X ). [31]
Se ha descubierto que varios tipos de mutaciones encontradas en ATRX están asociadas con ATR-X, incluidas las mutaciones más comunes de una sola base sin sentido, así como las mutaciones sin sentido, de cambio de marco y de deleción. [34] Las características de ATR-X incluyen: microcefalia, anomalías esqueléticas y faciales, retraso mental, anomalías genitales, convulsiones, uso y capacidad limitados del lenguaje y alfa-talasemia. [31] [35] [32] El fenotipo observado en ATR-X sugiere que la mutación del gen ATRX causa la regulación negativa de la expresión genética, como los genes de alfa-globina. [32] Todavía se desconoce qué causa la expresión de las diversas características de ATR-X en diferentes pacientes. [35]
Mutaciones puntuales de la helicasa XPD
XPD (factor D del xeroderma pigmentoso, también conocido como proteína ERCC2) es una helicasa dependiente de ATP de la superfamilia II, de 5'-3', que contiene dominios de grupos de hierro-azufre. [26] [37] Se ha demostrado que las mutaciones puntuales heredadas en la helicasa XPD están asociadas con trastornos de envejecimiento acelerado como el síndrome de Cockayne (SC) y la tricotiodistrofia (TTD). [38] El síndrome de Cockayne y la tricotiodistrofia son trastornos del desarrollo que implican sensibilidad a la luz ultravioleta y envejecimiento prematuro, y el síndrome de Cockayne presenta retraso mental grave desde el momento del nacimiento. [38] La mutación de la helicasa XPD también se ha relacionado con el xeroderma pigmentoso (XP), un trastorno caracterizado por la sensibilidad a la luz ultravioleta y que resulta en un aumento de varios miles de veces en el desarrollo de cáncer de piel. [38]
XPD es un componente esencial del complejo TFIIH , un factor de transcripción y reparación en la célula. [38] [39] [40] [41] [42] Como parte de este complejo, facilita la reparación por escisión de nucleótidos desenrollando el ADN. [38] TFIIH ayuda a reparar el ADN dañado, como el daño solar. [38] [39] [40] [41] [42] Una mutación en la helicasa XPD que ayuda a formar este complejo y contribuye a su función causa la sensibilidad a la luz solar observada en las tres enfermedades, así como el mayor riesgo de cáncer observado en XP y el envejecimiento prematuro observado en la tricotiodistrofia y el síndrome de Cockayne. [38]
Las mutaciones de la helicasa XPD que conducen a la tricotiodistrofia se encuentran en toda la proteína en varias ubicaciones involucradas en las interacciones proteína-proteína. [38] Esta mutación da como resultado una proteína inestable debido a su incapacidad para formar interacciones estabilizadoras con otras proteínas en los puntos de mutación. [38] Esto, a su vez, desestabiliza todo el complejo TFIIH, lo que conduce a defectos en los mecanismos de transcripción y reparación de la célula. [38]
Se ha sugerido que las mutaciones de la helicasa XPD que conducen al síndrome de Cockayne podrían ser el resultado de mutaciones dentro de XPD, que causan rigidez de la proteína y la posterior incapacidad de cambiar de funciones de reparación a funciones de transcripción debido a un "bloqueo" en el modo de reparación. [38] Esto podría hacer que la helicasa corte segmentos de ADN destinados a la transcripción. [38] Aunque la evidencia actual apunta a un defecto en la helicasa XPD que resulta en una pérdida de flexibilidad en la proteína en casos de síndrome de Cockayne, aún no está claro cómo esta estructura de proteína conduce a los síntomas descritos en el síndrome de Cockayne. [38]
En el xeroderma pigmentosa, la mutación de la helicasa XPD existe en el sitio de unión del ATP o del ADN. [38] Esto da como resultado una helicasa estructuralmente funcional capaz de facilitar la transcripción, sin embargo inhibe su función en el desenrollado del ADN y la reparación del ADN. [38] La falta de la capacidad de una célula para reparar mutaciones, como las causadas por el daño solar, es la causa de la alta tasa de cáncer en pacientes con xeroderma pigmentosa.
Mutaciones de la familia RecQ

Las helicasas RecQ (3'-5') pertenecen al grupo de helicasas de la Superfamilia II, que ayudan a mantener la estabilidad del genoma y suprimen la recombinación inapropiada. [43] [44] Las deficiencias y/o mutaciones en las helicasas de la familia RecQ muestran una recombinación genética aberrante y/o replicación de ADN, lo que conduce a la inestabilidad cromosómica y una disminución general de la capacidad de proliferación. [43] Se ha demostrado que las mutaciones en las helicasas de la familia RecQ BLM, RECQL4 y WRN, que desempeñan un papel en la regulación de la recombinación homóloga, dan lugar a las enfermedades autosómicas recesivas síndrome de Bloom (BS), síndrome de Rothmund-Thomson (RTS) y síndrome de Werner (WS), respectivamente. [44] [45]
El síndrome de Bloom se caracteriza por una predisposición al cáncer de aparición temprana, con una edad media de aparición de 24 años. [44] [46] Las células de los pacientes con síndrome de Bloom muestran una alta frecuencia de intercambio recíproco entre cromátidas hermanas (SCE) y daño cromosómico excesivo. [47] Hay evidencia que sugiere que BLM desempeña un papel en el rescate de la replicación de ADN interrumpida en las horquillas de replicación. [47]
El síndrome de Werner es un trastorno de envejecimiento prematuro, con síntomas que incluyen la aparición temprana de aterosclerosis y osteoporosis y otras enfermedades relacionadas con la edad, una alta incidencia de sarcoma y muerte que a menudo ocurre por infarto de miocardio o cáncer en la cuarta a sexta década de la vida. [44] [48] Las células de los pacientes con síndrome de Werner exhiben una vida reproductiva reducida con roturas y translocaciones cromosómicas, así como grandes deleciones de componentes cromosómicos, lo que causa inestabilidad genómica. [48]
El síndrome de Rothmund-Thomson, también conocido como poiquilodermia congénita , se caracteriza por envejecimiento prematuro, anomalías cutáneas y esqueléticas, erupción cutánea, poiquilodermia , cataratas juveniles y predisposición a cánceres como osteosarcomas. [44] [49] En las células de los pacientes con síndrome de Rothmund-Thomson se encuentran reordenamientos cromosómicos que causan inestabilidad genómica. RecQ es una familia de enzimas helicasas de ADN que se encuentran en varios organismos, incluidas bacterias, arqueas y eucariotas (como los humanos). Estas enzimas desempeñan papeles importantes en el metabolismo del ADN durante la replicación, recombinación y reparación del ADN. Hay cinco proteínas helicasas RecQ conocidas en humanos: RecQ1, BLM, WRN, RecQ4 y RecQ5. Las mutaciones en algunos de estos genes están asociadas con trastornos genéticos. Por ejemplo, las mutaciones en el gen BLM causan el síndrome de Bloom, que se caracteriza por un mayor riesgo de cáncer y otros problemas de salud. [50] Las mutaciones en el gen WRN conducen al síndrome de Werner, una afección caracterizada por el envejecimiento prematuro y un mayor riesgo de enfermedades relacionadas con la edad. Las helicasas RecQ son cruciales para mantener la estabilidad e integridad genómicas. Ayudan a prevenir la acumulación de anomalías genéticas que pueden conducir a enfermedades como el cáncer. La integridad del genoma depende de la familia de helicasas de ADN RecQ, que incluye procesos de reparación, recombinación, replicación y transcripción del ADN. La inestabilidad del genoma y el envejecimiento prematuro son afecciones que surgen de mutaciones en las helicasas RecQ humanas. [51] La helicasa RecQ Sgs1 falta en las células de levadura, lo que las convierte en modelos útiles para comprender las anomalías celulares humanas y la función de la helicasa RecQ. [52] El miembro de la familia de helicasas RecQ, RECQ1, está conectado a una pequeña cantidad de trastornos genéticos de cáncer poco comunes en individuos. Participa en la transcripción, el ciclo celular y la reparación del ADN. Según una investigación reciente, las mutaciones sin sentido en el gen RECQ1 pueden desempeñar un papel en el desarrollo del cáncer de mama familiar. Las helicasas de ADN son frecuentemente atraídas a regiones de ADN dañado y son esenciales para la replicación, recombinación, reparación y transcripción del ADN celular. La manipulación química de sus procesos moleculares puede cambiar la velocidad a la que se dividen las células cancerosas, así como la eficiencia de las transacciones y la homeostasis celular. El atrapamiento inducido por moléculas pequeñas de las helicasas de ADN, un tipo de proteína metabólica del ADN, puede tener consecuencias nocivas para las células cancerosas que proliferan rápidamente, lo que podría ser eficaz en el tratamiento del cáncer.
Durante la meiosis, las roturas de doble cadena de ADN y otros daños en el ADN de una cromátida se reparan mediante recombinación homóloga utilizando la cromátida hermana o una cromátida no hermana homóloga como plantilla. Esta reparación puede dar como resultado un recombinante cruzado (CO) o, más frecuentemente, un recombinante no cruzado (NCO). En la levadura Schizosaccharomyces pombe, la helicasa de ADN FmI1 de la familia FANCM dirige la formación de la recombinación NCO durante la meiosis. [53] La helicasa de tipo RecQ Rqh1 también dirige la recombinación meiótica NCO. [54] Estas helicasas, a través de su capacidad para desenrollar intermediarios del bucle D , promueven la recombinación NCO mediante el proceso de recocido de cadena dependiente de la síntesis .
En la planta Arabidopsis thaliana , la helicasa FANCM promueve la NCO y antagoniza la formación de recombinantes de CO. [55] Otra helicasa, RECQ4A/B, también reduce de forma independiente los CO. Se sugirió que los CO están restringidos debido a los costos a largo plazo de la recombinación de CO, es decir, la ruptura de combinaciones genéticas favorables de alelos construidas por la selección natural pasada . [55]
Helicasas de ARN

Las helicasas de ARN son esenciales para la mayoría de los procesos del metabolismo del ARN, como la biogénesis de los ribosomas , el empalme del pre-ARNm y la iniciación de la traducción . También desempeñan un papel importante en la detección de ARN virales. [56] Las helicasas de ARN están involucradas en la mediación de la respuesta inmune antiviral porque pueden identificar ARN extraños en vertebrados. Aproximadamente el 80% de todos los virus son virus de ARN y contienen sus propias helicasas de ARN. [57] Las helicasas de ARN defectuosas se han relacionado con cánceres, enfermedades infecciosas y trastornos neurodegenerativos. [56] Algunos trastornos neurológicos asociados con helicasas de ARN defectuosas son: esclerosis lateral amiotrófica , atrofia muscular espinal , ataxia espinocerebelosa tipo 2 , enfermedad de Alzheimer y síndrome de contractura congénita letal . [57]
Las helicasas de ARN y las helicasas de ADN se pueden encontrar juntas en todas las superfamilias de helicasas excepto en SF6. [58] [59] Todas las helicasas de ARN eucariotas que se han identificado hasta la fecha no forman anillos y son parte de SF1 y SF2. Por otro lado, se han encontrado helicasas de ARN formadoras de anillos en bacterias y virus. [56] Sin embargo, no todas las helicasas de ARN exhiben actividad helicasa según lo definido por la función enzimática, es decir, proteínas de la familia Swi/Snf. Aunque estas proteínas llevan los motivos helicasa típicos, hidrolizan ATP de una manera dependiente del ácido nucleico y están construidas alrededor de un núcleo de helicasa, en general, no se observa actividad de desenrollado. [60]
Las helicasas de ARN que exhiben actividad de desenrollado se han caracterizado por al menos dos mecanismos diferentes: desenrollado de dúplex canónico y separación local de la cadena. El desenrollado de dúplex canónico es la separación direccional por pasos de una cadena de dúplex, como se describió anteriormente, para el desenrollado de ADN. Sin embargo, la separación local de la cadena ocurre mediante un proceso en el que la enzima helicasa se carga en cualquier lugar a lo largo del dúplex. Esto suele ser ayudado por una región monocatenaria del ARN, y la carga de la enzima está acompañada por la unión de ATP. [61] Una vez que la helicasa y el ATP están unidos, ocurre la separación local de la cadena, que requiere la unión de ATP pero no el proceso real de hidrólisis de ATP. [62] Presentado con menos pares de bases, el dúplex se disocia sin más ayuda de la enzima. Este modo de desenrollado es utilizado por las helicasas de caja DEAD/DEAH . [63]
Actualmente hay disponible en línea una base de datos de helicasas de ARN [64] que contiene una lista completa de helicasas de ARN con información como secuencia, estructura y funciones bioquímicas y celulares. [56]
Herramientas de diagnóstico para la medición de helicasas
Medición y seguimiento de la actividad de la helicasa
Se utilizan varios métodos para medir la actividad de la helicasa in vitro . Estos métodos van desde ensayos cualitativos (ensayos que generalmente implican resultados que no involucran valores o mediciones) hasta cuantitativos (ensayos con resultados numéricos que pueden utilizarse en análisis estadísticos y numéricos). En 1982-1983, se desarrolló el primer ensayo bioquímico directo para medir la actividad de la helicasa. [15] [65] Este método se denominó "ensayo de desplazamiento de cadena".
- El ensayo de desplazamiento de cadena implica el radiomarcado de los dúplex de ADN. Después del tratamiento con helicasa, el ADN monocatenario se detecta visualmente como separado del ADN bicatenario mediante electroforesis PAGE no desnaturalizante . Después de la detección del ADN monocatenario, se cuantifica la cantidad de etiqueta radiactiva que se encuentra en el ADN monocatenario para dar un valor numérico de la cantidad de ADN bicatenario desenrollado.El ensayo de desplazamiento de cadena es aceptable para el análisis cualitativo, su incapacidad para mostrar resultados para más de un punto de tiempo, su consumo de tiempo y su dependencia de compuestos radiactivos para el etiquetado justificaron la necesidad de desarrollar diagnósticos que puedan monitorear la actividad de la helicasa en tiempo real.
Posteriormente se desarrollaron otros métodos que incorporaban algunos, si no todos, los siguientes: mecánica de alto rendimiento, el uso de marcaje de nucleótidos no radiactivos, tiempo de reacción más rápido/menor consumo de tiempo, monitoreo en tiempo real de la actividad de la helicasa (usando medición cinética en lugar de análisis de punto final/punto único). Estas metodologías incluyen: "un método de flujo de extinción rápido, ensayos basados en fluorescencia, ensayos de filtración, un ensayo de proximidad de centelleo , un ensayo de transferencia de energía de resonancia de fluorescencia con resolución temporal , un ensayo basado en tecnología de placa flash, ensayos de extinción de fluorescencia homogéneos con resolución temporal y ensayos de helicasa basados en electroquimioluminiscencia". [16] Con el uso de ecuaciones matemáticas especializadas, algunos de estos ensayos se pueden utilizar para determinar cuántos nucleótidos apareados de bases puede romper una helicasa por hidrólisis de 1 molécula de ATP. [66]
También se encuentran disponibles comercialmente kits de diagnóstico. Uno de estos kits es el ensayo de diagnóstico "Trupoint" de PerkinElmer , Inc. Este ensayo es un ensayo de extinción de fluorescencia con resolución temporal que utiliza la tecnología "SignalClimb" de PerkinElmer, que se basa en dos etiquetas que se unen en estrecha proximidad entre sí, pero en cadenas de ADN opuestas. Una etiqueta es un quelato de lantánido fluorescente, que sirve como etiqueta que se monitorea a través de un lector de placa de 96/384 pocillos adecuado. La otra etiqueta es una molécula extintora orgánica. La base de este ensayo es la "extinción" o represión de la señal del quelato de lantánido por la molécula extintora orgánica cuando los dos están en estrecha proximidad, como lo estarían cuando el dúplex de ADN está en su estado nativo. Tras la actividad de la helicasa en el dúplex, las etiquetas extintora y lantánida se separan a medida que se desenrolla el ADN. Esta pérdida de proximidad anula la capacidad de los inhibidores de reprimir la señal de los lantánidos, lo que provoca un aumento detectable de la fluorescencia que es representativo de la cantidad de ADN desenrollado y puede utilizarse como una medida cuantificable de la actividad de la helicasa. La ejecución y el uso de técnicas de obtención de imágenes de fluorescencia de moléculas individuales, centrándose en métodos que incluyen el atrapamiento óptico junto con la obtención de imágenes epifluorescentes, y también la inmovilización de la superficie junto con la visualización de fluorescencia por reflexión interna total. Combinadas con celdas de flujo de microcanales y control microfluídico, permiten obtener imágenes y realizar un seguimiento de moléculas de ADN y proteínas marcadas con fluorescencia individuales, lo que permite medir el desenrollado y la translocación del ADN con una resolución de una sola molécula. [67]
Determinación de la polaridad de la helicasa
La polaridad de la helicasa, que también se denomina "direccionalidad", se define como la dirección (caracterizada como 5'→3' o 3'→5') del movimiento de la helicasa en la cadena sencilla de ADN/ARN a lo largo de la cual se está moviendo. Esta determinación de la polaridad es vital para, por ejemplo, determinar si la helicasa probada se une a la cadena principal de ADN o a la cadena rezagada de ADN. Para caracterizar esta característica de la helicasa, se utiliza un ADN parcialmente dúplex como sustrato que tiene una región central de ADN monocatenario con diferentes longitudes de regiones dúplex de ADN (una región corta que corre 5'→3' y una región más larga que corre 3'→5') en ambos lados de esta región. [68] Una vez que la helicasa se agrega a esa región central monocatenaria, la polaridad se determina mediante la caracterización en el ADN monocatenario recién formado.
Véase también
- Proteína de unión al ADN de la helicasa de cromodominio : CHD1 , CHD1L , CHD2 , CHD3 , CHD4 , CHD5 , CHD6, CHD7 , CHD8 , CHD9
- Caja muerta / caja muerta/muerta helicasa : DDX3X , DDX5 , DDX6 , DDX10 , DDX11 , DDX12, DDX58 , DHX8 , DHX9 , DHX37, DHX40, DHX58
- ASCC3 , BLM , BRIP1 , DNA2 , FBXO18, FBXO30, HELB, HELLS , HELQ, HELZ, HFM1 , HLTF , IFIH1 , NAV2 , PIF1 , RECQL , RTEL1, SHPRH , SMARCA4 , SMARCAL1 , WRN , WRNIP1
- Base de datos de helicasas de ARN
Referencias
- ^ Wu Y (2012). "Desenrollado y rebobinado: ¿caras dobles de la helicasa?". Journal of Nucleic Acids . 2012 : 140601. doi : 10.1155/2012/140601 . PMC 3409536. PMID 22888405 .
- ^ abc Umate P, Tuteja N, Tuteja R (enero de 2011). "Análisis exhaustivo de todo el genoma de las helicasas humanas". Biología comunicativa e integradora . 4 (1): 118–137. doi :10.4161/cib.13844. PMC 3073292 . PMID 21509200.
- ^ ab Patel SS, Donmez I (julio de 2006). "Mecanismos de las helicasas". The Journal of Biological Chemistry . 281 (27): 18265–18268. doi : 10.1074/jbc.R600008200 . PMID 16670085.
- ^ Lionnet T, Spiering MM, Benkovic SJ, Bensimon D, Croquette V (diciembre de 2007). "La observación en tiempo real de la helicasa gp41 del bacteriófago T4 revela un mecanismo de desenrollado". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (50): 19790–19795. Bibcode :2007PNAS..10419790L. doi : 10.1073/pnas.0709793104 . PMC 2148377 . PMID 18077411.
- ^ abcdefg Johnson DS, Bai L, Smith BY, Patel SS, Wang MD (junio de 2007). "Estudios de moléculas individuales revelan la dinámica del desenrollado del ADN por la helicasa T7 en forma de anillo". Cell . 129 (7): 1299–1309. doi :10.1016/j.cell.2007.04.038. PMC 2699903 . PMID 17604719.
- ^ ab "Investigadores resuelven el misterio de cómo se separan las cadenas de ADN". 2007-07-03 . Consultado el 2007-07-05 .
- ^ abcde Betterton MD, Jülicher F (31 de agosto de 2005). "Fe de erratas: Apertura de cadenas dobles de ácidos nucleicos por helicasas: apertura activa frente a apertura pasiva [Phys. Rev. E 71, 011904 (2005)]". Physical Review E . 72 (2): 029906. Bibcode :2005PhRvE..72b9906B. doi :10.1103/PhysRevE.72.029906.
- ^ abcdef Manosas M, Xi XG, Bensimon D, Croquette V (septiembre de 2010). "Mecanismos activos y pasivos de las helicasas". Nucleic Acids Research . 38 (16): 5518–5526. doi :10.1093/nar/gkq273. PMC 2938219 . PMID 20423906.
- ^ abcdef Jarillo J, Ibarra B, Cao-García FJ (2021). "Replicación de ADN: análisis de datos y modelos de manipulación de moléculas individuales in vitro". Revista de biotecnología estructural y computacional . 19 : 3765–3778. doi :10.1016/j.csbj.2021.06.032. PMC 8267548 . PMID 34285777.
- ^ Wu, CG y Spies, M.: Descripción general: ¿Qué son las helicasas? En: Spies, M. (Ed.): [1]. Springer Science+Business Media, NY, 2013
- ^ "Bioquímica de Kevin Ahern (BB 451/551) en la Universidad Estatal de Oregón". oregonstate.edu . Archivado desde el original el 26 de enero de 2021 . Consultado el 3 de enero de 2024 .
- ^ Biblioteca de animación 3D; Replicación: [2] (Avanzada)
- ^ ab Abdel-Monem M, Dürwald H, Hoffmann-Berling H (junio de 1976). "Desenrollado enzimático del ADN. 2. Separación de cadenas por una enzima de desenrollado del ADN dependiente de ATP". Revista Europea de Bioquímica . 65 (2): 441–449. doi : 10.1111/j.1432-1033.1976.tb10359.x . PMID 133023.
- ^ ab Hotta Y, Stern H (mayo de 1978). "Proteína desenrolladora de ADN de células meióticas de Lilium". Bioquímica . 17 (10): 1872–1880. doi :10.1021/bi00603a011. PMID 207302.
- ^ abc Venkatesan M, Silver LL, Nossal NG (octubre de 1982). "La proteína del gen 41 del bacteriófago T4, necesaria para la síntesis de cebadores de ARN, también es una helicasa de ADN". The Journal of Biological Chemistry . 257 (20): 12426–12434. doi : 10.1016/S0021-9258(18)33731-1 . PMID 6288720.
- ^ ab Tuteja N, Tuteja R (mayo de 2004). "Helicasas de ADN procariotas y eucariotas. Proteínas motoras moleculares esenciales para la maquinaria celular". Revista Europea de Bioquímica . 271 (10): 1835–1848. doi :10.1111/j.1432-1033.2004.04093.x. PMC 7164108 . PMID 15128294.
- ^ Hübscher U, Stalder HP (agosto de 1985). "ADN helicasa de mamíferos". Investigación de ácidos nucleicos . 13 (15): 5471–5483. doi : 10.1093/nar/13.15.5471. PMC 321884 . PMID 3162158.
- ^ Stahl H, Dröge P, Knippers R (agosto de 1986). "Actividad de helicasa de ADN del antígeno tumoral grande SV40". The EMBO Journal . 5 (8): 1939–1944. doi :10.1002/j.1460-2075.1986.tb04447.x. PMC 1167061 . PMID 3019672.
- ^ Sugino A, Ryu BH, Sugino T, Naumovski L, Friedberg EC (septiembre de 1986). "Una nueva ATPasa dependiente de ADN que estimula la ADN polimerasa I de levadura y tiene actividad de desenrollado del ADN". The Journal of Biological Chemistry . 261 (25): 11744–11750. doi : 10.1016/S0021-9258(18)67306-5 . PMID 3017945.
- ^ Gorbalenya AE, Koonin EV, Donchenko AP, Blinov VM (junio de 1989). "Dos superfamilias relacionadas de supuestas helicasas implicadas en la replicación, recombinación, reparación y expresión de genomas de ADN y ARN". Nucleic Acids Research . 17 (12): 4713–4730. doi :10.1093/nar/17.12.4713. PMC 318027 . PMID 2546125.
- ^ Linder, P., Lasko, PF, Ashburner, M., Leroy, P., Nielson, PJ, Nishi, K., Schneir, J., Slonimski, PP (1989) Nacimiento de la caja muerta. Nature (Londres) 337, 121-122.
- ^ Tuteja N, Tuteja R, Rahman K, Kang LY, Falaschi A (diciembre de 1990). "Una helicasa de ADN de células humanas". Nucleic Acids Research . 18 (23): 6785–6792. doi :10.1093/nar/18.23.6785. PMC 332732 . PMID 1702201.
- ^ Hehman GL, Hauswirth WW (septiembre de 1992). "Helicasa de ADN de mitocondrias de mamíferos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 89 (18): 8562–8566. Bibcode :1992PNAS...89.8562H. doi : 10.1073/pnas.89.18.8562 . PMC 49960 . PMID 1326759.
- ^ Tuteja N, Phan TN, Tewari KK (mayo de 1996). "Purificación y caracterización de una helicasa de ADN del cloroplasto de guisante que se transloca en la dirección 3' a 5'". Revista Europea de Bioquímica . 238 (1): 54–63. doi : 10.1111/j.1432-1033.1996.0054q.x . PMID 8665952.
- ^ Tuteja R, Malhotra P, Song P, Tuteja N, Chauhan VS (2002). "Aislamiento y caracterización de un homólogo de eIF-4A de Plasmodium cynomolgi". Parasitología molecular y bioquímica . 124 (1–2): 79–83. doi :10.1016/S0166-6851(02)00205-0. PMID 12387853.
- ^ abcdefghijklmnopq Singleton MR, Dillingham MS, Wigley DB (2007). "Estructura y mecanismo de las helicasas y translocasas de ácidos nucleicos". Revisión anual de bioquímica . 76 : 23–50. doi :10.1146/annurev.biochem.76.052305.115300. PMID 17506634.
- ^ abcd Fairman-Williams ME, Guenther UP, Jankowsky E (junio de 2010). "Helicasas SF1 y SF2: asuntos de familia". Current Opinion in Structural Biology . 20 (3): 313–324. doi :10.1016/j.sbi.2010.03.011. PMC 2916977 . PMID 20456941.
- ^ Stelter M, Acajjaoui S, McSweeney S, Timmins J (2013). "Información estructural y mecanicista sobre el desenrollado del ADN por Deinococcus radiodurans UvrD". PLOS ONE . 8 (10): e77364. Bibcode :2013PLoSO...877364S. doi : 10.1371/journal.pone.0077364 . PMC 3797037 . PMID 24143224.
- ^ Iyer LM, Aravind L, Koonin EV (diciembre de 2001). "Origen común de cuatro familias diversas de grandes virus de ADN eucariotas". Revista de Virología . 75 (23): 11720–11734. doi :10.1128/JVI.75.23.11720-11734.2001. PMC 114758 . PMID 11689653.
- ^ Iyer LM, Leipe DD, Koonin EV, Aravind L (2004). "Historia evolutiva y clasificación de orden superior de las ATPasas AAA+". Journal of Structural Biology . 146 (1–2): 11–31. doi :10.1016/j.jsb.2003.10.010. PMID 15037234.
- ^ abcde Ropers HH, Hamel BC (enero de 2005). "Retardo mental ligado al cromosoma X". Nature Reviews. Genetics . 6 (1): 46–57. doi :10.1038/nrg1501. PMID 15630421. S2CID 427210.
- ^ abc Gibbons RJ, Picketts DJ, Villard L, Higgs DR (marzo de 1995). "Las mutaciones en un supuesto regulador transcripcional global causan retraso mental ligado al cromosoma X con talasemia alfa (síndrome ATR-X)". Cell . 80 (6): 837–845. doi : 10.1016/0092-8674(95)90287-2 . PMID 7697714.
- ^ ab Nextprot Online Protein Database. "ATRX-Regulador transcripcional ATRX.", consultado el 12 de noviembre de 2012.
- ^ ab Picketts DJ, Higgs DR, Bachoo S, Blake DJ, Quarrell OW, Gibbons RJ (diciembre de 1996). "ATRX codifica un nuevo miembro de la familia de proteínas SNF2: las mutaciones apuntan a un mecanismo común subyacente al síndrome ATR-X". Human Molecular Genetics . 5 (12): 1899–1907. doi : 10.1093/hmg/5.12.1899 . PMID 8968741.
- ^ abc Gibbons R (mayo de 2006). "Talasemia alfa-retardo mental, ligada al cromosoma X". Orphanet Journal of Rare Diseases . 1 : 15. doi : 10.1186/1750-1172-1-15 . PMC 1464382 . PMID 16722615.
- ^ Stevenson RE (1993). Adam MP, Everman DB, Mirzaa GM, Pagon RA, Wallace SE, Bean LJ, Gripp KW, Amemiya A (eds.). "Síndrome de discapacidad intelectual ligado al cromosoma X y alfa-talasemia". GeneReviews . Seattle (WA): Universidad de Washington, Seattle. PMID 20301622.
- ^ Rudolf J, Rouillon C, Schwarz-Linek U, White MF (enero de 2010). "La helicasa XPD desenrolla las estructuras de burbujas y no se detiene por las lesiones del ADN eliminadas por la vía de reparación por escisión de nucleótidos". Nucleic Acids Research . 38 (3): 931–941. doi :10.1093/nar/gkp1058. PMC 2817471 . PMID 19933257.
- ^ abcdefghijklmno Fan L, Fuss JO, Cheng QJ, Arvai AS, Hammel M, Roberts VA, et al. (mayo de 2008). "Estructuras y actividades de la helicasa XPD: perspectivas sobre los fenotipos del cáncer y el envejecimiento a partir de mutaciones XPD". Cell . 133 (5): 789–800. doi :10.1016/j.cell.2008.04.030. PMC 3055247 . PMID 18510924.
- ^ ab Lainé JP, Mocquet V, Egly JM (2006). "Actividades enzimáticas de TFIIH en la transcripción y reparación por escisión de nucleótidos". Reparación de ADN, Parte A. Métodos en enzimología. Vol. 408. págs. 246–263. doi :10.1016/S0076-6879(06)08015-3. ISBN 9780121828134. Número de identificación personal 16793373.
- ^ ab Tirode F, Busso D, Coin F, Egly JM (enero de 1999). "Reconstitución del factor de transcripción TFIIH: asignación de funciones para las tres subunidades enzimáticas, XPB, XPD y cdk7". Molecular Cell . 3 (1): 87–95. doi : 10.1016/S1097-2765(00)80177-X . PMID 10024882.
- ^ ab Sung P, Bailly V, Weber C, Thompson LH, Prakash L, Prakash S (octubre de 1993). "El gen del grupo D del xeroderma pigmentoso humano codifica una helicasa de ADN". Nature . 365 (6449): 852–855. Bibcode :1993Natur.365..852S. doi :10.1038/365852a0. PMID 8413672. S2CID 4334960.
- ^ ab Schaeffer L, Roy R, Humbert S, Moncollin V, Vermeulen W, Hoeijmakers JH, et al. (Abril de 1993). "Helicasa reparadora de ADN: un componente del factor de transcripción básico BTF2 (TFIIH)". Ciencia . 260 (5104): 58–63. Código Bib : 1993 Ciencia... 260... 58S. doi : 10.1126/ciencia.8465201. PMID 8465201.
- ^ ab Hanada K, Hickson ID (septiembre de 2007). "Genética molecular de los trastornos de la helicasa RecQ". Ciencias de la vida celular y molecular . 64 (17): 2306–2322. doi :10.1007/s00018-007-7121-z. PMC 11136437 . PMID 17571213. S2CID 29287970.
- ^ abcde Opresko PL, Cheng WH, Bohr VA (abril de 2004). "Unión de la bioquímica de la helicasa RecQ y la enfermedad humana". The Journal of Biological Chemistry . 279 (18): 18099–18102. doi : 10.1074/jbc.R300034200 . PMID 15023996.
- ^ Ouyang KJ, Woo LL, Ellis NA (2008). "Recombinación homóloga y mantenimiento de la integridad del genoma: cáncer y envejecimiento a través del prisma de las helicasas RecQ humanas". Mecanismos de envejecimiento y desarrollo . 129 (7–8): 425–440. doi :10.1016/j.mad.2008.03.003. PMID 18430459. S2CID 6804631.
- ^ Ellis NA, Groden J, Ye TZ, Straughen J, Lennon DJ, Ciocci S, et al. (noviembre de 1995). "El producto del gen del síndrome de Bloom es homólogo a las helicasas RecQ". Celúla . 83 (4): 655–666. doi : 10.1016/0092-8674(95)90105-1 . PMID 7585968.
- ^ ab Selak N, Bachrati CZ, Shevelev I, Dietschy T, van Loon B, Jacob A, et al. (septiembre de 2008). "La helicasa del síndrome de Bloom (BLM) interactúa física y funcionalmente con p12, la subunidad más pequeña de la ADN polimerasa delta humana". Nucleic Acids Research . 36 (16): 5166–5179. doi :10.1093/nar/gkn498. PMC 2532730 . PMID 18682526.
- ^ ab Gray MD, Shen JC, Kamath-Loeb AS, Blank A, Sopher BL, Martin GM, et al. (septiembre de 1997). "La proteína del síndrome de Werner es una helicasa de ADN". Nature Genetics . 17 (1): 100–103. doi :10.1038/ng0997-100. PMID 9288107. S2CID 20587915.
- ^ Kitao S, Shimamoto A, Goto M, Miller RW, Smithson WA, Lindor NM, Furuichi Y (mayo de 1999). "Las mutaciones en RECQL4 causan un subconjunto de casos de síndrome de Rothmund-Thomson". Nature Genetics . 22 (1): 82–84. doi :10.1038/8788. PMID 10319867. S2CID 195211275.
- ^ Gupta, Sonia Vidushi; Schmidt, Kristina Hildegard (18 de febrero de 2020). "Mantenimiento de la integridad del genoma de la levadura mediante helicasas de ADN de la familia RecQ". Genes . 11 (2): 205. doi : 10.3390/genes11020205 . ISSN 2073-4425. PMC 7074392 . PMID 32085395.
- ^ Debnath S., Sharma S. Helicasa RECQ1 en estabilidad genómica y cáncer. Genes. 2020:11. doi: 10.3390/genes11060622], Debnath S., Sharma S. Helicasa RECQ1 en estabilidad genómica y cáncer. Genes. 2020:11. doi: 10.3390/genes11060622].
{{cite journal}}: Requiere citar revista|journal=( ayuda ) ; Falta o está vacío|title=( ayuda )Mantenimiento CS1: nombres múltiples: lista de autores ( enlace ) Mantenimiento CS1: nombres numéricos: lista de autores ( enlace ) - ^ Hendrickson, WA; Ward, KB (27 de octubre de 1975). "Modelos atómicos para las cadenas principales polipeptídicas de miohemeritrina y hemeritrina". Comunicaciones de investigación bioquímica y biofísica . 66 (4): 1349–1356. doi :10.1016/0006-291x(75)90508-2. ISSN 1090-2104. PMID 5.
- ^ Lorenz A, Osman F, Sun W, Nandi S, Steinacher R, Whitby MC (junio de 2012). "El ortólogo de la levadura de fisión FANCM dirige la recombinación sin entrecruzamiento durante la meiosis". Science . 336 (6088): 1585–1588. Bibcode :2012Sci...336.1585L. doi :10.1126/science.1220111. PMC 3399777 . PMID 22723423.
- ^ Lorenz A, Mehats A, Osman F, Whitby MC (diciembre de 2014). "Los parálogos y mediadores de Rad51/Dmc1 se oponen a las helicasas de ADN para limitar la formación de ADN híbrido y promover los entrecruzamientos durante la recombinación meiótica". Nucleic Acids Research . 42 (22): 13723–13735. doi :10.1093/nar/gku1219. PMC 4267644 . PMID 25414342.
- ^ ab Séguéla-Arnaud M, Crismani W, Larchevêque C, Mazel J, Froger N, Choinard S, et al. (abril de 2015). "Múltiples mecanismos limitan los entrecruzamientos meióticos: TOP3α y dos homólogos de BLM antagonizan los entrecruzamientos en paralelo a FANCM". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (15): 4713–4718. Bibcode :2015PNAS..112.4713S. doi : 10.1073/pnas.1423107112 . PMC 4403193 . PMID 25825745.
- ^ abcd Jankowsky A, Guenther UP, Jankowsky E (enero de 2011). "La base de datos de helicasas de ARN". Nucleic Acids Research . 39 (número de la base de datos): D338–D341. doi :10.1093/nar/gkq1002. PMC 3013637 . PMID 21112871.
- ^ Jankowsky E, Fairman-Williams ME (2010). "Introducción a las helicasas de ARN: superfamilias, familias y temas principales". En Jankowsky E (ed.). Helicasas de ARN (RSC Biomolecular Sciences) . Cambridge, Inglaterra: Royal Society of Chemistry. p. 5. ISBN 978-1-84755-914-2.
- ^ Ranji A, Boris-Lawrie K (2010). "Helicasas de ARN: funciones emergentes en la replicación viral y la respuesta innata del huésped". RNA Biology . 7 (6): 775–787. doi :10.4161/rna.7.6.14249. PMC 3073335 . PMID 21173576.
- ^ Jankowsky E (enero de 2011). "Las helicasas de ARN en acción: unión y reorganización". Tendencias en ciencias bioquímicas . 36 (1): 19–29. doi :10.1016/j.tibs.2010.07.008. PMC 3017212 . PMID 20813532.
- ^ Yang Q, Del Campo M, Lambowitz AM, Jankowsky E (octubre de 2007). "Las proteínas DEAD-box desenrollan dúplex mediante separación local de cadenas". Molecular Cell . 28 (2): 253–263. doi : 10.1016/j.molcel.2007.08.016 . PMID 17964264.
- ^ Liu F, Putnam A, Jankowsky E (diciembre de 2008). "La hidrólisis de ATP es necesaria para el reciclaje de proteínas DEAD-box pero no para el desenrollado del dúplex". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (51): 20209–20214. Bibcode :2008PNAS..10520209L. doi : 10.1073/pnas.0811115106 . PMC 2629341 . PMID 19088201.
- ^ Jarmoskaite I, Russell R (2011). "Proteínas DEAD-box como helicasas de ARN y chaperonas". Wiley Interdisciplinary Reviews. ARN . 2 (1): 135–152. doi :10.1002/wrna.50. PMC 3032546 . PMID 21297876.
- ^ "Índice de /". www.rnahelicase.org . Archivado desde el original el 18 de diciembre de 2014 . Consultado el 7 de diciembre de 2012 .
- ^ Matson SW, Tabor S, Richardson CC (noviembre de 1983). "La proteína del gen 4 del bacteriófago T7. Caracterización de la actividad de la helicasa". The Journal of Biological Chemistry . 258 (22): 14017–14024. doi : 10.1016/S0021-9258(17)44018-X . PMID 6315716.
- ^ Sarlós K, Gyimesi M, Kovács M (junio de 2012). "La helicasa RecQ se transloca a lo largo del ADN monocatenario con una procesividad moderada y un acoplamiento mecanoquímico ajustado". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (25): 9804–9809. Bibcode :2012PNAS..109.9804S. doi : 10.1073/pnas.1114468109 . PMC 3382518 . PMID 22665805.
- ^ Pavankumar TL, Exell JC, Kowalczykowski SC (1 de enero de 2016). "Imágenes fluorescentes directas de translocación y desenrollado por helicasas de ADN individuales". Enzimología de moléculas individuales: métodos de alto rendimiento y basados en fluorescencia . Métodos en enzimología. Vol. 581. págs. 1–32. doi :10.1016/bs.mie.2016.09.010. ISBN . 9780128092675. PMC 5854184 . PMID 27793277.
- ^ Borowiec JA (1996). "Helicasas de ADN". En DePamphilis ML (ed.). Replicación de ADN en células eucariotas . Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. págs. 545–574. ISBN 978-0-87969-459-3.OCLC 246537432 .
Enlaces externos
- Helicasas de ADN+ en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Helicasas de ARN+ en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.















