Virus de ARN de cadena negativa
Se ha sugerido que este artículo se divida en artículos titulados Virus de ARN de cadena negativa y Negarnaviricota . ( discutir ) ( octubre de 2024 ) |
| Negarnaviricota | |
|---|---|
 | |
| Un montaje de micrografías electrónicas de transmisión de algunos virus del filo Negarnaviricota . No a escala. Especies de izquierda a derecha, de arriba a abajo: Ébolavirus Zaire , ortohantavirus Sin Nombre , ortopneumovirus humano , henipavirus Hendra , un rabdovirus no identificado , morbilivirus del sarampión . | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Negarnaviricota |
| Subtaxones | |
Ver texto | |
| Sinónimos [1] [2] | |
| |
Los virus de ARN de cadena negativa ( virus −ssRNA ) son un grupo de virus relacionados que tienen genomas monocatenarios de sentido negativo hechos de ácido ribonucleico (ARN). Tienen genomas que actúan como cadenas complementarias a partir de las cuales se sintetiza el ARN mensajero (ARNm) por la enzima viral ARN polimerasa dependiente de ARN (RdRp). Durante la replicación del genoma viral, RdRp sintetiza un antígeno de sentido positivo que utiliza como plantilla para crear ARN genómico de sentido negativo. Los virus de ARN de cadena negativa también comparten otras características: la mayoría contiene una envoltura viral que rodea la cápside, que encierra el genoma viral, los genomas de los virus −ssRNA suelen ser lineales y es común que su genoma esté segmentado.
Los virus de ARN de cadena negativa constituyen el filo Negarnaviricota , en el reino Orthornavirae y reino Riboviria . Descienden de un ancestro común que era un virus de ARN bicatenario (dsRNA) , y se consideran un clado hermano de los reovirus , que son virus dsRNA. Dentro del filo, hay dos ramas principales que forman dos subfilos: Haploviricotina , cuyos miembros son en su mayoría no segmentados y que codifican un RdRp que sintetiza capuchones en el ARNm, y Polyploviricotina , cuyos miembros están segmentados y que codifican un RdRp que arrebata capuchones de los ARNm del huésped. Se reconocen un total de seis clases en el filo.
Los virus de ARN de cadena negativa están estrechamente asociados con los artrópodos y pueden dividirse informalmente entre aquellos que dependen de los artrópodos para su transmisión y aquellos que descienden de los virus de los artrópodos pero que ahora pueden replicarse en vertebrados sin la ayuda de los artrópodos. Los virus de ARN de cadena negativa más destacados transmitidos por artrópodos incluyen el virus de la fiebre del Valle del Rift y el virus del marchitamiento moteado del tomate . Los virus de ARN de cadena negativa más destacados de vertebrados incluyen el virus del Ébola , los hantavirus , los virus de la gripe , el virus de la fiebre de Lassa y el virus de la rabia .
Etimología
Negarnaviricota toma la primera parte de su nombre del latín nega , que significa negativo, la parte media rna se refiere al ARN, y la parte final, viricota , es el sufijo utilizado para los filos de virus. El subfilo Haploviricotina toma la primera parte de su nombre, Haplo , del griego antiguo ἁπλός, que significa simple, y 'viricotina es el sufijo utilizado para los subfilos de virus. El subfilo Polyploviricotina sigue el mismo patrón, Polyplo se toma del griego antiguo πολύπλοκος, que significa complejo. [1]
Características
Genoma

Todos los virus de Negarnaviricota son virus de ARN monocatenario (−ssRNA) de sentido negativo. Tienen genomas hechos de ARN, que son monocatenarios en lugar de bicatenarios. Sus genomas son de sentido negativo, lo que significa que el ARN mensajero (ARNm) puede sintetizarse directamente a partir del genoma por la enzima viral ARN polimerasa dependiente de ARN (RdRp), también llamada ARN replicasa, que está codificada por todos los virus −ssRNA. Excluyendo los virus del género Tenuivirus y algunos de la familia Chuviridae , todos los virus −ssRNA tienen genomas lineales en lugar de circulares, y los genomas pueden estar segmentados o no segmentados. [1] [3] [4] Todos los genomas −ssRNA contienen repeticiones terminales invertidas , que son secuencias de nucleótidos palindrómicas en cada extremo del genoma. [5]
Replicación y transcripción
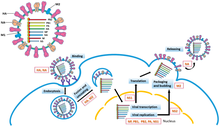
La replicación de los genomas de −ssRNA se ejecuta mediante RdRp, que inicia la replicación uniéndose a una secuencia líder en el extremo 3' (que suele pronunciarse como "tres extremos principales") del genoma. A continuación, RdRp utiliza el genoma de sentido negativo como plantilla para sintetizar un antígeno de sentido positivo. Al replicar el antígeno, RdRp se une primero a la secuencia final en el extremo 3' del antígeno. A continuación, RdRp ignora todas las señales de transcripción en el antígeno y sintetiza una copia del genoma mientras utiliza el antígeno como plantilla. [6] La replicación se ejecuta mientras el genoma está dentro de la nucleocápside, y RdRp revela la cápside y se transloca a lo largo del genoma durante la replicación. A medida que RdRp sintetiza nuevas secuencias de nucleótidos, las proteínas de la cápside se ensamblan y encapsidan el ARN viral recién replicado. [2]
La transcripción del ARNm del genoma sigue el mismo patrón direccional que la producción del antígeno. En la secuencia líder, RdRp sintetiza un ARN líder trifosfato en el extremo 5' (que suele pronunciarse "five prime end") y, en el caso del subfilo Haploviricotina , cubre el extremo 5' o, en el caso del subfilo Polyploviricotina , toma una cubierta de un ARNm hospedador y la adhiere al ARNm viral para que los ribosomas de la célula hospedadora puedan traducirlo . [7] [8] [9]
Después de tapar el ARNm, RdRp inicia la transcripción en una señal de inicio del gen y luego termina la transcripción al alcanzar una señal de fin del gen. [10] Al final de la transcripción, RdRp sintetiza una cola poliadenilada (cola poli (A)) que consiste en cientos de adeninas en el extremo 3 del ARNm, lo que puede hacerse tartamudeando en una secuencia de uracilos . [11] [12] Después de que se construye la cola poli (A), RdRp libera el ARNm. En genomas que codifican más de una porción transcribible, RdRp puede continuar escaneando hasta la siguiente secuencia de inicio para continuar con la transcripción. [10] [7] [13]
Algunos virus −ssRNA son ambisense , lo que significa que tanto la cadena genómica negativa como el antígeno positivo codifican por separado proteínas diferentes. Para transcribir los virus ambisense, se realizan dos rondas de transcripción: primero, el ARNm se produce directamente a partir del genoma; segundo, el ARNm se crea a partir del antígeno. Todos los virus ambisense contienen una estructura de bucle de horquilla para detener la transcripción después de que se haya transcrito el ARNm de la proteína. [14]
Morfología

Los virus de ARN de cadena negativa contienen un complejo de ribonucleoproteína compuesto por el genoma y una RdRp unida a cada segmento del genoma rodeado por una cápside. [15] La cápside está compuesta por proteínas cuya estructura plegada contiene cinco hélices alfa en el lóbulo N-terminal (motivo 5-H) y tres hélices alfa en el lóbulo C-terminal (motivo 3-H). Dentro de la cápside, el genoma está intercalado entre estos dos motivos. [2] Excluyendo la familia Aspiviridae , los virus de ARN monocatenario contienen una envoltura viral externa , un tipo de membrana lipídica que rodea la cápside. La forma de la partícula viral, llamada virión, de los virus de ARN monocatenario varía y puede ser filamentosa, pleomórfica, esférica o tubular. [16]
Evolución
La segmentación del genoma es un rasgo destacado entre muchos virus −ssRNA, y los virus −ssRNA varían desde tener genomas con un segmento, típico de los miembros del orden Mononegavirales , hasta genomas con diez segmentos, como es el caso de Tilapia tilapinevirus . [5] [17] No hay una tendencia clara a lo largo del tiempo que determine el número de segmentos, y la segmentación del genoma entre los virus −ssRNA parece ser un rasgo flexible ya que ha evolucionado de forma independiente en múltiples ocasiones. La mayoría de los miembros del subfilo Haploviricotina no están segmentados, mientras que la segmentación es universal en Polyploviricotina . [2] [5]
Filogenética

El análisis filogenético basado en RdRp muestra que los virus −ssRNA descienden de un ancestro común y que probablemente sean un clado hermano de los reovirus , que son virus dsRNA. Dentro del filo, hay dos ramas claras, asignadas a dos subfilos, en función de si RdRp sintetiza una tapa en el ARNm viral o toma una tapa del ARNm del huésped y la adhiere al ARNm viral. [1] [3]
Dentro del filo, los virus −ssRNA que infectan a los artrópodos parecen ser los basales y los ancestros de todos los demás virus −ssRNA. Los artrópodos con frecuencia viven juntos en grupos grandes, lo que permite que los virus se transmitan fácilmente. Con el tiempo, esto ha llevado a que los virus −ssRNA de los artrópodos adquieran un alto nivel de diversidad. Si bien los artrópodos albergan grandes cantidades de virus, existe desacuerdo sobre el grado en que se produce la transmisión entre especies de virus −ssRNA de los artrópodos entre artrópodos. [4] [5]
Los virus ARN monocatenarios de plantas y vertebrados tienden a estar genéticamente relacionados con los virus infectados por artrópodos. Además, la mayoría de los virus ARN monocatenarios fuera de los artrópodos se encuentran en especies que interactúan con ellos. Por lo tanto, los artrópodos sirven como hospedadores clave y vectores de transmisión de los virus ARN monocatenarios. En términos de transmisión, los virus ARN monocatenarios no artrópodos se pueden distinguir entre aquellos que dependen de los artrópodos para su transmisión y aquellos que pueden circular entre vertebrados sin la ayuda de los artrópodos. Es probable que este último grupo se haya originado a partir del primero, adaptándose a la transmisión solo entre vertebrados. [5]
Clasificación
Negarnaviricota pertenece al reino Orthornavirae , que abarca todos los virus de ARN que codifican RdRp, y al reino Riboviria , que incluye Orthornavirae así como todos los virus que codifican la transcriptasa inversa en el reino Pararnavirae . Negarnaviricota contiene dos subfilos, que contienen seis clases combinadas, cinco de las cuales son monotípicas hasta taxones inferiores: [2] [9] [18]

- Subfilo: Haploviricotina , que contiene virus −ssRNA que codifican un RdRp que sintetiza una estructura de capuchón en el ARNm viral y que generalmente tienen genomas no segmentados.
- Clase: Chunquiviricetes
- Orden: Muvirales
- Familia: Qinviridae
- Género: Yingvirus
- Familia: Qinviridae
- Orden: Muvirales
- Clase: Milneviricetes
- Orden: Serpentovirales
- Familia: Aspviridae
- Género: Ophiovirus
- Familia: Aspviridae
- Orden: Serpentovirales
- Clase: Monjiviricetes
- Clase: Yunchangviricetes
- Orden: Goujianvirales
- Familia: Yueviridae
- Género: Yuyuevirus
- Familia: Yueviridae
- Orden: Goujianvirales
- Clase: Chunquiviricetes
- Subfilo: Polyploviricotina , que contiene virus −ssRNA que codifican un RdRp que toma una tapa del ARNm del huésped para usarla como tapa en el ARNm viral y que tienen genomas segmentados.
- Clase: Ellioviricetes
- Orden: Bunyavirales
- Clase: Insthoviricetes
- Orden: Articulavirales
- Clase: Ellioviricetes
Los virus de ARN de cadena negativa se clasifican como Grupo V en el sistema de clasificación de Baltimore , que agrupa a los virus en función de su forma de producción de ARNm y que suele utilizarse junto con la taxonomía estándar de virus, que se basa en la historia evolutiva. Por lo tanto, el Grupo V y Negarnaviricota son sinónimos. [1]
Enfermedad
Los virus de ARN de cadena negativa causaron muchas enfermedades ampliamente conocidas. Muchas de ellas son transmitidas por artrópodos, incluido el virus de la fiebre del Valle del Rift y el virus del marchitamiento moteado del tomate . [19] [20] Entre los vertebrados, los murciélagos y los roedores son vectores comunes de muchos virus, incluido el virus del Ébola y el virus de la rabia , transmitidos por murciélagos y otros vertebrados, [21] [22] y el virus de la fiebre de Lassa y los hantavirus , transmitidos por roedores. [23] [24] Los virus de la influenza son comunes entre las aves y los mamíferos. [25] Los virus de ARN monocatenario específicos de los humanos incluyen el virus del sarampión y el virus de las paperas . [26] [27]
Historia
A lo largo de la historia se han conocido muchas enfermedades causadas por virus −ssRNA, incluidas la infección por hantavirus, el sarampión y la rabia. [28] [29] [30] En la historia moderna, algunas como el ébola y la gripe han causado brotes de enfermedades mortales. [31] [32] El virus de la estomatitis vesicular , aislado por primera vez en 1925 y uno de los primeros virus animales en ser estudiados porque podía estudiarse bien en cultivos celulares , fue identificado como un virus −ssRNA, lo que era único en ese momento porque otros virus de ARN que se habían descubierto eran de sentido positivo. [33] [34] A principios del siglo XXI, la enfermedad bovina peste bovina , causada por el virus de la peste bovina −ssRNA, se convirtió en la segunda enfermedad en ser erradicada, después de la viruela , causada por un virus de ADN. [35]
En el siglo XXI, la metagenómica viral se ha vuelto común para identificar virus en el medio ambiente. Para los virus −ssRNA, esto permitió identificar una gran cantidad de virus de invertebrados, y especialmente de artrópodos, lo que ayudó a proporcionar información sobre la historia evolutiva de los virus −ssRNA. Con base en el análisis filogenético de RdRp que muestra que los virus −ssRNA descienden de un ancestro común, Negarnaviricota y sus dos subfilos se establecieron en 2018, y se colocaron en el reino Riboviria , entonces recién establecido . [1] [36]
Galería
- Rabia ( Rhabdoviridae )
Notas
- ^ El virus de la hepatitis D se suele denominar virus, pero se puede describir más específicamente como una cadena de ARN monocatenario patógena de tipo virusoide . Se excluye de Negarnaviricota porque, aunque es ARN monocatenario, no codifica RdRp, que es el rasgo unificador de los virus en Orthornavirae .
Referencias
- ^ abcdef Wolf Y, Krupovic M, Zhang YZ, Maes P, Dolji V, Koonin EV (21 de agosto de 2017). "Megataxonomía de virus de ARN de sentido negativo" (docx) . Comité Internacional de Taxonomía de Virus (ICTV) . Consultado el 6 de agosto de 2020 .
- ^ abcde Luo M, Terrel JR, Mcmanus SA (30 de julio de 2020). "Estructura de la nucleocápside del virus de ARN de cadena negativa". Viruses . 12 (8): 835. doi : 10.3390/v12080835 . PMC 7472042 . PMID 32751700.
- ^ ab Wolf YI, Kazlauskas D, Iranzo J, Lucia-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Kooning EV (27 de noviembre de 2018). "Orígenes y evolución del viroma de ARN global". mBio . 9 (6): e02329-18. doi :10.1128/mBio.02329-18. PMC 6282212 . PMID 30482837.
- ^ ab Käfer S, Paraskevopoulou S, Zirkel F, Wieseke N, Donath A, Petersen M, Jones TC, Liu S, Zhou X, Middendorf M, Junglen S, Misof B, Drosten C (12 de diciembre de 2019). "Reevaluación de la diversidad de virus de ARN de cadena negativa en insectos". PLOS Patog . 15 (12): e1008224. doi : 10.1371/journal.ppat.1008224 . PMC 6932829 . PMID 31830128.
- ^ abcde Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (29 de enero de 2015). "La diversidad genómica sin precedentes de los virus de ARN en artrópodos revela la ascendencia de los virus de ARN de sentido negativo". eLife . 4 (4): e05378. doi : 10.7554/eLife.05378 . PMC 4384744 . PMID 25633976.
- ^ "Replicación del virus ARN de cadena negativa". ViralZone . Instituto Suizo de Bioinformática . Consultado el 6 de agosto de 2020 .
- ^ ab "Transcripción del virus ARN de cadena negativa". ViralZone . Instituto Suizo de Bioinformática . Consultado el 6 de agosto de 2020 .
- ^ "Cap snatching". ViralZone . Instituto Suizo de Bioinformática . Consultado el 6 de agosto de 2020 .
- ^ ab Kuhn JH, Wolf YI, Krupovic M, Zhang YZ, Maes P, Dolja VV, Koonin EV (febrero de 2019). "Clasificar los virus: la ganancia vale la pena". Nature . 566 (7744): 318–320. Bibcode :2019Natur.566..318K. doi :10.1038/d41586-019-00599-8. PMC 11165669 . PMID 30787460. S2CID 67769904.
- ^ ab Fearns, Rachel; Plemper, Richard K (15 de abril de 2017). "Polimerasas de paramixovirus y neumovirus". Virus Research . 234 : 87–102. doi :10.1016/j.virusres.2017.01.008. ISSN 0168-1702. PMC 5476513 . PMID 28104450.
- ^ Harmon, Shawn B.; Megaw, A. George; Wertz, Gail W. (1 de enero de 2001). "Secuencias de ARN implicadas en la terminación transcripcional del virus respiratorio sincitial". Journal of Virology . 75 (1): 36–44. doi :10.1128/JVI.75.1.36-44.2001. PMC 113895 . PMID 11119571.
- ^ Jacques, JP; Kolakofsky, D. (1 de mayo de 1991). "Transcripción pseudo-templada en organismos procariotas y eucariotas". Genes & Development . 5 (5): 707–713. doi : 10.1101/gad.5.5.707 . ISSN 0890-9369. PMID 2026325.
- ^ "Tartamudeo de la polimerasa del virus ARN de cadena negativa". ViralZone . Instituto Suizo de Bioinformática . Consultado el 6 de agosto de 2020 .
- ^ "Transcripción ambisense en virus de ARN de cadena negativa". ViralZone . Instituto Suizo de Bioinformática . Consultado el 6 de agosto de 2020 .
- ^ Zhou H, Sun Y, Guo Y, Lou Z (septiembre de 2013). "Perspectiva estructural sobre la formación del complejo ribonucleoproteico en virus de ARN monocatenario de sentido negativo". Trends Microbiol . 21 (9): 475–484. doi :10.1016/j.tim.2013.07.006. PMID 23953596.
- ^ Fermin, G. (2018). Virus: biología molecular, interacciones con el huésped y aplicaciones a la biotecnología. Elsevier. pp. 19–27, 43. doi :10.1016/B978-0-12-811257-1.00002-4. ISBN 9780128112571.S2CID89706800 .
- ^ Bacharach E, Mishra N, Briese T, Zody MC, Kembou Tsofack JE, Zamostiano R, Berkowitz A, Ng J, Nitido A, Corvelo A, Toussaint NC, Abel Nielsen SC, Hornig M, Del Pozo J, Bloom T, Ferguson H, Eldar A, Lipkin WI (5 de abril de 2016). "Caracterización de un nuevo virus similar a ortomixo que provoca muertes masivas de tilapia". mBio . 7 (2): e00431-16. doi :10.1128/mBio.00431-16. PMC 4959514 . PMID 27048802.
- ^ "Taxonomía de virus: publicación de 2019". talk.ictvonline.org . Comité Internacional de Taxonomía de Virus . Consultado el 6 de agosto de 2020 .
- ^ Hartman A (junio de 2017). "Fiebre del Valle del Rift". Clin Lab Med . 37 (2): 285–301. doi :10.1016/j.cll.2017.01.004. PMC 5458783 . PMID 28457351.
- ^ Scholthof KB, Adkins S, Czosnek H, Palukaitis P, Jacquot E, Hohn T, Hohn B, Saunders K, Candresse T, Ahlquist P, Hemenway C, Foster GD (diciembre de 2011). "Los 10 principales virus de plantas en patología molecular de plantas". Mol Plant Pathol . 12 (9): 938–954. doi :10.1111/j.1364-3703.2011.00752.x. PMC 6640423 . PMID 22017770.
- ^ Muñoz-Fontela C, McElroy AK (2017). "Enfermedad del virus del Ébola en humanos: fisiopatología e inmunidad". Marburg- and Ebolaviruses . Temas actuales en microbiología e inmunología. Vol. 411. págs. 141–169. doi :10.1007/82_2017_11. ISBN 978-3-319-68946-3. PMC 7122202 . PMID 28653186.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Fisher CR, Streicker DG, Schnell MJ (abril de 2018). "La propagación y evolución del virus de la rabia: conquistando nuevas fronteras". Nat Rev Microbiol . 16 (4): 241–255. doi :10.1038/nrmicro.2018.11. PMC 6899062 . PMID 29479072.
- ^ Yun NE, Walker DH (9 de octubre de 2012). "Patogénesis de la fiebre de Lassa". Viruses . 4 (10): 2031–2048. doi : 10.3390/v4102031 . PMC 3497040 . PMID 23202452.
- ^ Avsic-Zupanc T, Saksida A, Korva M (abril de 2019). "Infecciones por hantavirus". Clin Microbiol Infect . 21S : e6–e16. doi : 10.1111/1469-0691.12291 . PMID 24750436 . Consultado el 6 de agosto de 2020 .
- ^ Borkenhagen LK, Salman MD, Ma MJ, Gray GC (noviembre de 2019). "Infecciones por el virus de la influenza animal en humanos: un comentario". Int J Infect Dis . 88 : 113–119. doi : 10.1016/j.ijid.2019.08.002 . PMID 31401200 . Consultado el 6 de agosto de 2020 .
- ^ "Transmisión del sarampión". cdc.gov . Centros para el Control y la Prevención de Enfermedades (CDC). 5 de febrero de 2018 . Consultado el 6 de agosto de 2020 .
- ^ Rubin S, Eckhaus M, Rennick LJ, Bamford CG, Duprex WP (enero de 2015). "Biología molecular, patogénesis y patología del virus de las paperas". J Pathol . 235 (2): 242–252. doi :10.1002/path.4445. PMC 4268314 . PMID 25229387.
- ^ Jiang H, Zheng X, Wang L, Du H, Wang P, Bai X (2017). "Infección por hantavirus: un desafío zoonótico global". Virol Sin . 32 (1): 32–43. doi :10.1007/s12250-016-3899-x. PMC 6598904 . PMID 28120221.
- ^ "Historia del sarampión". cdc.gov . Centros para el Control y la Prevención de Enfermedades (CDC). 5 de febrero de 2018 . Consultado el 6 de agosto de 2020 .
- ^ Velasco-Villa A, Mauldin MR, Shi M, Escobar LE, Gallardo-Romero NF, Damon I, Olson VA, Streicker DG, Emerson G (octubre de 2017). "La historia de la rabia en el hemisferio occidental". Antiviral Res . 146 : 221–232. doi :10.1016/j.antiviral.2017.03.013. PMC 5620125 . PMID 28365457.
- ^ Zawilinska B, Kosz-Vnenchak M (2014). "Introducción general a la biología y la enfermedad del virus del Ébola" (PDF) . Folia Med Cracov . 54 (3): 57–65. PMID 25694096 . Consultado el 6 de agosto de 2020 .
- ^ Krammer F, Smith G, Fouchier R, Peiris M, Kedzierska K, Doherty PC, Palese P, Shaw ML, Treanor J, Webster RG, García-Sastre A (28 de junio de 2018). "Influenza". Imprimaciones Nat Rev Dis . 4 (1): 3. doi :10.1038/s41572-018-0002-y. PMC 7097467 . PMID 29955068.
- ^ "Virus de la estomatitis vesicular" (PDF) . Swine Health Information Center . Centro de Seguridad Alimentaria y Salud Pública, Facultad de Medicina Veterinaria, Universidad Estatal de Iowa. Noviembre de 2015 . Consultado el 6 de agosto de 2020 .
- ^ Kolakofsky D (abril de 2015). "Una breve historia sesgada de los virus ARN". ARN . 21 (4): 667–669. doi :10.1261/rna.049916.115. PMC 4371325 . PMID 25780183 . Consultado el 6 de agosto de 2020 .
- ^ Greenwood B (12 de mayo de 2014). "La contribución de la vacunación a la salud mundial: pasado, presente y futuro". Philos Trans R Soc Lond B Biol Sci . 369 (1645): 20130433. doi :10.1098/rstb.2013.0433. PMC 4024226 . PMID 24821919.
- ^ "Historia de la taxonomía ICTV: Negarnaviricota". Comité Internacional de Taxonomía de Virus (ICTV) . Consultado el 6 de agosto de 2020 .









_EM_PHIL_2175_lores.jpg/440px-Respiratory_Syncytial_Virus_(RSV)_EM_PHIL_2175_lores.jpg)


_EM_18_lores.jpg/440px-Vesicular_stomatitis_virus_(VSV)_EM_18_lores.jpg)
