Virus de la hepatitis C
| Hepacivirus C | |
|---|---|
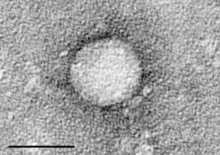 | |
| Micrografía electrónica de Hepacivirus C purificado a partir de cultivo celular. Barra de escala = 50 nanómetros | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Kitrinoviricota |
| Clase: | Flasuviricetes |
| Orden: | Virus amarillos |
| Familia: | Flaviviridae |
| Género: | Hepacivirus |
| Especies: | Hepacivirus C |
| Sinónimos [1] [2] | |
Virus de la hepatitis C | |
El virus de la hepatitis C ( VHC ) [3] es un virus ARN monocatenario de polaridad positiva, pequeño (de 55 a 65 nm de tamaño), con envoltura , de la familia Flaviviridae . El virus de la hepatitis C es la causa de la hepatitis C y de algunos cánceres como el cáncer de hígado ( carcinoma hepatocelular , abreviado como HCC) y los linfomas en humanos. [4] [5]
Taxonomía
El virus de la hepatitis C pertenece al género Hepacivirus , un miembro de la familia Flaviviridae . Antes de 2011, se consideraba que era el único miembro de este género. Sin embargo, se ha descubierto un miembro de este género en perros : el hepacivirus canino . [6] También existe al menos un virus de este género que infecta a los caballos. [7] Se han descrito varios virus adicionales del género en murciélagos y roedores. [8] [9]
Estructura

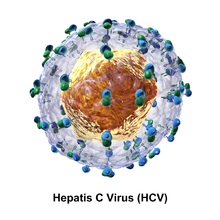
La partícula del virus de la hepatitis C está formada por una membrana lipídica que tiene un diámetro de entre 55 y 65 nm. [10] [11] Dos glicoproteínas de la membrana viral , E1 y E2 , están incrustadas en la membrana lipídica. [12] Participan en la unión y la entrada del virus en la célula. [10] Dentro de la membrana hay un núcleo icosaédrico que tiene un diámetro de entre 33 y 40 nm. [11] Dentro del núcleo se encuentra el material de ARN del virus. [10]
Glicoproteínas E1 y E2
E1 y E2 están unidos covalentemente cuando están incrustados en la envoltura del VHC y se estabilizan mediante enlaces disulfuro . E2 es globular y parece sobresalir 6 nm de la membrana de la envoltura según las imágenes del microscopio electrónico. [11]
Estas glicoproteínas desempeñan un papel importante en las interacciones que la hepatitis C tiene con el sistema inmunológico. Una región hipervariable , la región hipervariable 1 (HVR1), se puede encontrar en la glicoproteína E2. [10] La HVR1 es flexible y bastante accesible a las moléculas circundantes. [13] La HVR1 ayuda a E2 a proteger al virus del sistema inmunológico. Impide que el CD81 se adhiera a su respectivo receptor en el virus. [13] Además, E2 puede proteger a E1 del sistema inmunológico. [13] Aunque la HVR1 es bastante variable en la secuencia de aminoácidos, esta región tiene características químicas, físicas y conformacionales similares en muchas glicoproteínas E2. [14]
Genoma

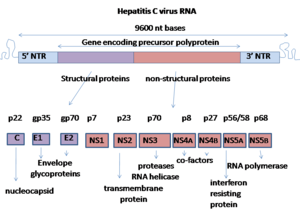
El virus de la hepatitis C tiene un genoma de ARN monocatenario de sentido positivo . El genoma consta de un único marco de lectura abierto de 9600 bases de nucleótidos de longitud. [15] Este único marco de lectura abierto se traduce para producir un único producto proteico, que luego se procesa para producir proteínas activas más pequeñas. Por eso, en las bases de datos disponibles públicamente, como el Instituto Europeo de Bioinformática , el proteoma viral solo consta de 2 proteínas. [ cita requerida ]
En los extremos 5′ y 3′ del ARN se encuentran las regiones no traducidas (UTR), que no se traducen en proteínas pero son importantes para la traducción y replicación del ARN viral. La UTR 5′ tiene un sitio de unión al ribosoma [16] o sitio interno de entrada al ribosoma (IRES) que inicia la traducción de una proteína muy larga que contiene alrededor de 3000 aminoácidos. El dominio central del IRES del VHC contiene una unión Holliday helicoidal de cuatro vías que está integrada dentro de un pseudonudo previsto . [17] La conformación de este dominio central restringe la orientación del marco de lectura abierto para el posicionamiento en la subunidad ribosómica 40S . La preproteína grande es escindida posteriormente por proteasas celulares y virales en las 10 proteínas más pequeñas que permiten la replicación viral dentro de la célula huésped, o se ensamblan en las partículas virales maduras. [18] Las proteínas estructurales producidas por el virus de la hepatitis C incluyen la proteína central, E1 y E2; Las proteínas no estructurales incluyen NS2 , NS3 , NS4A , NS4B , NS5A y NS5B . [ cita requerida ]
Biología molecular
Esta sección necesita citas adicionales para su verificación . ( Mayo de 2016 ) |
Las proteínas de este virus se organizan a lo largo del genoma en el siguiente orden: terminal N-núcleo-envoltura (E1)–E2–p7-proteína no estructural 2 (NS2)–NS3–NS4A–NS4B–NS5A–NS5B–terminal C. La generación de proteínas no estructurales maduras (NS2 a NS5B) depende de la actividad de las proteinasas virales. [19] La unión NS2/NS3 es escindida por una proteinasa autocatalítica dependiente de metales codificada dentro de NS2 y el extremo N de NS3. Las escisiones restantes aguas abajo de este sitio son catalizadas por una serina proteasa también contenida dentro de la región N-terminal de NS3.
- La proteína central tiene 191 aminoácidos y se puede dividir en tres dominios en función de su hidrofobicidad: el dominio 1 (residuos 1-117) contiene principalmente residuos básicos con dos regiones hidrofóbicas cortas ; el dominio 2 (residuos 118-174) es menos básico y más hidrofóbico y su extremo C está al final de p21; el dominio 3 (residuos 175-191) es altamente hidrofóbico y actúa como una secuencia señal para la proteína de envoltura E1.
- Ambas proteínas de la envoltura (E1 y E2) están altamente glucosiladas y son importantes para la entrada en la célula. La E1 actúa como subunidad fusogénica y la E2 actúa como proteína de unión al receptor. La E1 tiene entre 4 y 5 glicanos unidos a N y la E2 tiene 11 sitios de glucosilación en N.
- La proteína NS1 (p7) es indispensable para la replicación del genoma viral, pero desempeña un papel fundamental en la morfogénesis del virus. Esta proteína es una proteína de 63 aminoácidos que atraviesa la membrana y se encuentra en el retículo endoplasmático . La escisión de p7 está mediada por las peptidasas de señal del retículo endoplasmático . Dos dominios transmembrana de p7 están conectados por un bucle citoplasmático y están orientados hacia el lumen del retículo endoplasmático.
- La proteína NS2 es una proteína transmembrana de 21 a 23 kilodalton (kDa) con actividad de proteasa.
- La NS3 es una proteína de 67 kDa cuyo extremo N-terminal tiene actividad de serina proteasa y cuyo extremo C-terminal tiene actividad de NTPasa / helicasa . Se encuentra dentro del retículo endoplasmático y forma un complejo heterodimérico con la NS4A, una proteína de membrana de 54 aminoácidos que actúa como cofactor de la proteasa.
- NS4A: una proteína de membrana de 54 aminoácidos que actúa como cofactor de la proteinasa.
- La NS4B es una proteína de membrana integral hidrofóbica pequeña (27 kDa) con cuatro dominios transmembrana. Se encuentra dentro del retículo endoplasmático y desempeña un papel importante en el reclutamiento de otras proteínas virales. Induce cambios morfológicos en el retículo endoplasmático formando una estructura denominada red membranosa.
- La NS5A es una fosfoproteína hidrófila que desempeña un papel importante en la replicación viral, la modulación de las vías de señalización celular y la respuesta al interferón . Se sabe que se une a las proteínas VAP humanas ancladas al retículo endoplásmico . [20]
- La proteína NS5B (65 kDa) es la ARN polimerasa dependiente de ARN viral . NS5B tiene la función clave de replicar el ARN viral del VHC utilizando la cadena de ARN de sentido positivo viral como su plantilla y cataliza la polimerización de los trifosfatos de ribonucleósido (rNTP) durante la replicación del ARN . [21] [22] [23] Se han determinado varias estructuras cristalinas de la polimerasa NS5B en varias formas cristalinas basándose en la misma secuencia de consenso BK (HCV-BK, genotipo 1). [24] La estructura se puede representar mediante una forma de mano derecha con dedos, palma y pulgar. El sitio activo rodeado , exclusivo de NS5B, está contenido dentro de la estructura de palma de la proteína. Estudios recientes sobre la estructura de la cepa J4 (HC-J4) de genotipo 1b de la proteína NS5B indican la presencia de un sitio activo donde se produce un posible control de la unión de nucleótidos y el inicio de la síntesis de ARN de novo. La técnica de novo añade los iniciadores necesarios para iniciar la replicación del ARN. [25] La investigación actual intenta unir estructuras a este sitio activo para alterar su funcionalidad y así evitar una mayor replicación del ARN viral. [26]
También se ha descrito una undécima proteína. [27] [28] Esta proteína está codificada por un desplazamiento del marco de lectura +1 en el gen de la cápside. Parece ser antigénica, pero se desconoce su función.
Replicación

La replicación del VHC implica varios pasos. El virus se replica principalmente en los hepatocitos del hígado , donde se estima que cada célula infectada produce diariamente aproximadamente cincuenta viriones (partículas virales) con un total calculado de un billón de viriones generados. El virus también puede replicarse en células mononucleares de sangre periférica , lo que potencialmente explica los altos niveles de trastornos inmunológicos encontrados en pacientes con VHC infectados crónicamente. En el hígado, las partículas de VHC son llevadas a los sinusoides hepáticos por el flujo sanguíneo. Estos sinusoides son vecinos de las células de los hepatocitos. [10] El VHC puede atravesar el endotelio de los sinusoides y llegar a la superficie basolateral de las células de los hepatocitos. [10]
El VHC tiene una amplia variedad de genotipos y muta rápidamente debido a una alta tasa de error por parte de la ARN polimerasa dependiente del ARN del virus . La tasa de mutación produce tantas variantes del virus que se considera una cuasiespecie en lugar de una especie de virus convencional. [29] La entrada a las células huésped se produce a través de interacciones complejas entre los viriones, especialmente a través de sus glicoproteínas, y las moléculas de la superficie celular CD81 , receptor de LDL , SR-BI , DC-SIGN , Claudina-1 y Ocludina . [30] [31]
La envoltura del VHC es similar a las lipoproteínas de muy baja densidad (VLDL) y las lipoproteínas de baja densidad (LDL). [10] Debido a esta similitud, se cree que el virus puede asociarse con apolipoproteínas . Podría rodearse de lipoproteínas, cubriendo parcialmente E1 y E2. Investigaciones recientes indican que estas apolipoproteínas interactúan con el receptor depurador B1 (SR-B1). El SR-B1 puede eliminar lípidos de las lipoproteínas alrededor del virus para permitir un mejor contacto con HVR1. La claudina 1, que es una proteína de unión estrecha , y el CD81 se unen para crear un complejo, preparándolos para procesos posteriores de infección por VHC. A medida que se activa el sistema inmunológico, los macrófagos aumentan la cantidad de TNF-α alrededor de los hepatocitos que se están infectando. Esto desencadena la migración de ocludina, que es otro complejo de unión estrecha, a la membrana basolateral. La partícula del VHC está lista para ingresar a la célula. [10]
Estas interacciones conducen a la endocitosis de la partícula viral, proceso que se ve facilitado por las proteínas clatrina. Una vez dentro de un endosoma temprano, el endosoma y la envoltura viral se fusionan y el ARN puede ingresar al citoplasma. [10]
El VHC se hace cargo de partes de la maquinaria intracelular para replicarse. [32] El genoma del VHC se traduce para producir una sola proteína de alrededor de 3011 aminoácidos. Luego, la poliproteína es procesada proteolíticamente por proteasas virales y celulares para producir tres proteínas estructurales (asociadas al virión) y siete proteínas no estructurales (NS). Alternativamente, puede ocurrir un cambio de marco de lectura en la región Core para producir una proteína de marco de lectura alternativo (ARFP). [33] El VHC codifica dos proteasas, la cisteína autoproteasa NS2 y la serina proteasa NS3-4A. Las proteínas NS luego reclutan el genoma viral en un complejo de replicación de ARN, que está asociado con membranas citoplasmáticas reorganizadas. La replicación de ARN se lleva a cabo a través de la ARN polimerasa dependiente de ARN viral NS5B, que produce un intermediario de ARN de cadena negativa. El ARN de cadena negativa luego sirve como plantilla para la producción de nuevos genomas virales de cadena positiva. Los genomas nacientes pueden luego ser traducidos, replicados o empaquetados dentro de nuevas partículas virales. [ cita requerida ]
El virus se replica en las membranas lipídicas intracelulares. [34] El retículo endoplasmático en particular se deforma en estructuras de membrana de forma única denominadas "redes membranosas". Estas estructuras pueden ser inducidas por la expresión única de la proteína viral NS4B. [35] La proteína central se asocia con gotitas de lípidos y utiliza microtúbulos y dineínas para alterar su ubicación a una distribución perinuclear . [36] La liberación del hepatocito puede involucrar la vía secretora de VLDL. [37] Otra hipótesis establece que la partícula viral puede ser secretada desde el retículo endoplasmático a través de la vía del complejo de clasificación endosómica requerida para el transporte (ESCRT). [10] Esta vía se utiliza normalmente para hacer brotar vesículas fuera de la célula. La única limitación a esta hipótesis es que la vía se utiliza normalmente para la gemación celular , y no se sabe cómo el VHC se apoderaría de la vía ESCRT para su uso con el retículo endoplasmático. [10]
Genotipos
Según las diferencias genéticas entre los aislados del VHC, la especie del virus de la hepatitis C se clasifica en seis genotipos (1-6) con varios subtipos dentro de cada genotipo (representados por letras minúsculas). [38] [39] Los subtipos se dividen a su vez en cuasiespecies según su diversidad genética. Los genotipos difieren en un 30-35% de los sitios de nucleótidos en todo el genoma. [40] La diferencia en la composición genómica de los subtipos de un genotipo suele ser del 20-25%. Los subtipos 1a y 1b se encuentran en todo el mundo y causan el 60% de todos los casos. [ cita requerida ]
Importancia clínica
Algunas partes de este artículo (las relacionadas con los medicamentos antivirales de acción directa) deben actualizarse . Ayúdenos ( Enero de 2024 ) |
El genotipo es clínicamente importante para determinar la respuesta potencial a la terapia basada en interferón y la duración requerida de dicha terapia. Los genotipos 1 y 4 responden menos al tratamiento basado en interferón que los otros genotipos (2, 3, 5 y 6). [41] La duración de la terapia estándar basada en interferón para los genotipos 1 y 4 es de 48 semanas, mientras que el tratamiento para los genotipos 2 y 3 se completa en 24 semanas. Se producen respuestas virológicas sostenidas en el 70% de los casos de genotipo 1, ~90% de los genotipos 2 y 3, ~65% del genotipo 4 y ~80% del genotipo 6. [42] Además, las personas de ascendencia africana tienen muchas menos probabilidades de responder al tratamiento cuando se infectan con los genotipos 1 o 4. [43] Se propone que la proporción sustancial de esta falta de respuesta al tratamiento se debe a un polimorfismo de un solo nucleótido (SNP) en el cromosoma 19 del genoma humano que predice el éxito del tratamiento. [44] Los genotipos 1 y 4 del VHC se han distribuido de forma endémica en áreas superpuestas de África occidental y central, infectando durante siglos a poblaciones humanas portadoras del polimorfismo genético en cuestión. Esto ha llevado a los científicos a sugerir que la persistencia prolongada de los genotipos 1 y 4 del VHC en personas de origen africano es una adaptación evolutiva del VHC a lo largo de muchos siglos a las respuestas inmunogenéticas de estas poblaciones. [45]
La infección con un genotipo no confiere inmunidad contra los demás, y es posible la infección simultánea con dos cepas. En la mayoría de estos casos, una de las cepas supera a la otra en poco tiempo. Este hallazgo puede ser útil en el tratamiento, al reemplazar cepas que no responden a la medicación por otras más fáciles de tratar. [46]
Recombinación
Cuando dos virus infectan la misma célula, puede ocurrir una recombinación genética . [47] Aunque es poco frecuente, se ha observado recombinación del VHC entre diferentes genotipos, entre subtipos del mismo genotipo e incluso entre cepas del mismo subtipo. [47]
Epidemiología
El virus de la hepatitis C es un virus que se transmite predominantemente por la sangre , con un riesgo muy bajo de transmisión sexual o vertical . [48] Debido a este modo de propagación, los grupos clave en riesgo son los usuarios de drogas intravenosas (UDI), los receptores de productos sanguíneos y, a veces, los pacientes en hemodiálisis . El entorno común para la transmisión del VHC también es la transmisión intrahospitalaria ( nosocomial ), cuando las prácticas de higiene y esterilización no se siguen correctamente en la clínica. [49] Se han propuesto varias prácticas culturales o rituales como un posible modo histórico de propagación del VHC, incluida la circuncisión, la mutilación genital, la escarificación ritual, el tatuaje tradicional y la acupuntura. [48] También se ha argumentado que, dados los períodos extremadamente prolongados de persistencia del VHC en humanos, incluso tasas muy bajas e indetectables de transmisión mecánica a través de insectos que pican pueden ser suficientes para mantener la infección endémica en los trópicos, donde las personas reciben una gran cantidad de picaduras de insectos. [50]
Evolución
La identificación del origen de este virus ha sido difícil, pero los genotipos 1 y 4 parecen compartir un origen común. [51] Un análisis bayesiano sugiere que los genotipos principales divergieron hace unos 300 a 400 años a partir del virus ancestro común . [52] Los genotipos menores divergieron hace unos 200 años a partir de sus genotipos principales. Todos los genotipos existentes parecen haber evolucionado a partir del genotipo 1 subtipo 1b. [ cita requerida ]
Un estudio de cepas del genotipo 6 sugiere una fecha de evolución anterior: aproximadamente entre 1100 y 1350 años antes del presente . [53] La tasa estimada de mutación fue de 1,8 × 10 −4 . Un estudio experimental estimó la tasa de mutación en 2,5–2,9 × 10 −3 sustituciones de bases por sitio por año. [54] Este genotipo puede ser el ancestro de los otros genotipos. [53]
Un estudio de aislamientos europeos, estadounidenses y japoneses sugirió que la fecha de origen del genotipo 1b fue aproximadamente en el año 1925. [55] Las fechas estimadas de origen de los tipos 2a y 3a fueron 1917 y 1943 respectivamente. El tiempo de divergencia de los tipos 1a y 1b se estimó en 200-300 años. [ cita requerida ]
Un estudio de los genotipos 1a y 1b estimó que las fechas de origen fueron 1914-1930 para el tipo 1a y 1911-1944 para el tipo 1b. [56] Tanto el tipo 1a como el 1b experimentaron expansiones masivas en su tamaño poblacional efectivo entre 1940 y 1960. La expansión del subtipo 1b del VHC precedió a la del subtipo 1a por al menos 16 años. Ambos tipos parecen haberse propagado desde el mundo desarrollado al mundo en desarrollo.
Las cepas de genotipo 2 de África se pueden dividir en cuatro clados que se correlacionan con su país de origen: (1) Camerún y República Centroafricana (2) Benin, Ghana y Burkina Faso (3) Gambia, Guinea, Guinea-Bissau y Senegal (4) Madagascar. [57] También hay evidencia sólida de la diseminación del genotipo 2 del VHC desde África occidental hasta el Caribe a través del comercio transatlántico de esclavos . [58]
Se cree que el genotipo 3 tiene su origen en el sudeste asiático. [59]
Los datos de estos distintos países sugieren que el virus puede haber evolucionado en el sudeste asiático y que los comerciantes de Europa occidental lo propagaron hasta África occidental. [60] Más tarde, cuando se levantó el aislamiento autoimpuesto de ese país, se introdujo en Japón . Una vez introducido en un país, su propagación se ha visto influida por muchos factores locales, como las transfusiones de sangre, los programas de vacunación, el uso intravenoso de drogas y los regímenes de tratamiento. Dada la reducción de la tasa de propagación una vez que se implementó la detección del VHC en los productos sanguíneos en la década de 1990, parecería que anteriormente la transfusión de sangre era un método importante de propagación. Se requieren más estudios para determinar las fechas de evolución de los distintos genotipos y el momento de su propagación por todo el mundo. [ cita requerida ]
Vacunación
A diferencia de la hepatitis A y B, actualmente no existe una vacuna para prevenir la infección por hepatitis C. [61]
Investigación actual
La exactitud de este artículo puede verse comprometida debido a información desactualizada . La razón esgrimida es que el VHC ahora es curable como resultado de la investigación médica: https://web.archive.org/web/20180919174730/http://www.who.int/hepatitis/news-events/direct-acting-antiviral-cure-hepatitis-c/en/. ( Noviembre de 2019 ) |
El estudio del VHC se ha visto obstaculizado por el estrecho rango de hospedadores del VHC. [62] El uso de replicones ha tenido éxito, pero estos recién se descubrieron recientemente. [63] El VHC, como la mayoría de los virus de ARN, existe como una cuasiespecie viral , lo que hace que sea muy difícil aislar una sola cepa o tipo de receptor para su estudio. [64] [65]
La investigación actual se centra en los inhibidores de moléculas pequeñas de la proteasa viral , la ARN polimerasa y otros genes no estructurales. Dos agentes, boceprevir de Merck [66] y telaprevir de Vertex Pharmaceuticals , ambos inhibidores de la proteasa NS3, fueron aprobados para su uso el 13 de mayo de 2011 y el 23 de mayo de 2011, respectivamente.
Se ha informado de una posible asociación entre niveles bajos de vitamina D y una respuesta deficiente al tratamiento. [67] [68] [69] [70] Los trabajos in vitro han demostrado que la vitamina D puede reducir la replicación viral. [71] Si bien este trabajo parece prometedor [72] [73], los resultados de los ensayos clínicos están pendientes. [74] [75] Sin embargo, se ha propuesto que la suplementación con vitamina D es importante además del tratamiento estándar, con el fin de mejorar la respuesta al tratamiento. [76]
Se ha demostrado que la naringenina , un flavonoide que se encuentra en la toronja y otras frutas y hierbas, bloquea el ensamblaje de partículas virales infecciosas intracelulares sin afectar los niveles intracelulares del ARN o la proteína viral. [76]
Otros agentes que se encuentran bajo investigación incluyen inhibidores de análogos de nucleósidos y nucleótidos e inhibidores no nucleósidos de la ARN polimerasa dependiente de ARN, inhibidores de NSP5A y compuestos dirigidos al huésped como inhibidores de ciclofilina y silibinina . [77]
El 6 de diciembre de 2013, la FDA aprobó el uso de sofosbuvir contra la infección crónica por hepatitis C. Se ha informado que es el primer fármaco que ha demostrado seguridad y eficacia para tratar ciertos tipos de infección por VHC sin la necesidad de la administración conjunta de interferón. [78] El 22 de noviembre, la FDA aprobó el uso de simeprevir en combinación con peginterferón-alfa y ribavirina . [79] El simeprevir ha sido aprobado en Japón para el tratamiento de la infección crónica por hepatitis C, genotipo 1. [80]
También hay investigaciones experimentales actuales sobre terapias no relacionadas con fármacos. La oximatrina , por ejemplo, es un extracto de raíz que se encuentra en el continente asiático y que, según se ha informado, tiene actividad antiviral contra el VHC en cultivos celulares y estudios con animales. Ensayos humanos pequeños y prometedores han mostrado resultados beneficiosos y ningún efecto secundario grave, pero fueron demasiado pequeños para generalizar conclusiones. [76]
El 5 de octubre de 2020, se anunció que Harvey J. Alter , Michael Houghton y Charles M. Rice habían recibido el Premio Nobel de Fisiología o Medicina 2020 por el descubrimiento del VHC. [81]
Véase también
- Enfermedad transmitida por la sangre
- Virus del cáncer
- Descubrimiento y desarrollo de inhibidores de NS5A
- IRES del VHC
- Virus de la hepatitis C, tallo-bucle VII
- Elemento 3′X del virus de la hepatitis C
- Elemento de replicación que actúa en cis (CRE) del virus de la hepatitis C (VHC)
- Infección de transmisión sexual
Referencias
- ^ "Hepacivirus C". Centro Nacional de Información Biotecnológica (NCBI) . Consultado el 3 de enero de 2022 .
- ^ Smith, Donald B.; et al. (23 de junio de 2016). "Crear 13 nuevas especies en el género Hepacivirus y renombrar 1 especie (familia Flaviviridae)" (PDF) . Comité Internacional de Taxonomía de Virus (ICTV) . Consultado el 13 de marzo de 2019 .
- ^ "Género: Hepacivirus". Comité Internacional de Taxonomía de Virus . Julio de 2018. Archivado desde el original el 19 de febrero de 2020.
- ^ Ferri, Clodoveo (2015). "Síndrome del VHC: una constelación de trastornos autoinmunes específicos de órganos y no órganos, linfoma no Hodgkin de células B y cáncer". Revista Mundial de Hepatología . 7 (3): 327–43. doi : 10.4254/wjh.v7.i3.327 . ISSN 1948-5182. PMC 4381161 . PMID 25848462.
- ^ Rusyn I, Lemon SM (2014). "Mecanismos del cáncer de hígado inducido por el VHC: ¿qué aprendimos de los estudios in vitro y en animales?". Cancer Lett . 345 (2): 210–5. doi :10.1016/j.canlet.2013.06.028. PMC 3844040 . PMID 23871966.
- ^ Kapoor A, et al. (2011). "Caracterización de un homólogo canino del virus de la hepatitis C". Proc Natl Acad Sci USA . 108 (28): 11608–13. Bibcode :2011PNAS..10811608K. doi : 10.1073/pnas.1101794108 . PMC 3136326 . PMID 21610165.
- ^ Burbelo PD, Dubovi EJ, Simmonds P, et al. (junio de 2012). "Descubrimiento de hepacivirus genéticamente diversos en un nuevo huésped gracias a la serología". J. Virol . 86 (11): 6171–8. doi :10.1128/JVI.00250-12. PMC 3372197 . PMID 22491452.
- ^ Quan PL, Firth C, Conte JM, et al. (mayo de 2013). "Los murciélagos son un importante reservorio natural de hepacivirus y pegivirus". Proc. Natl. Sci. USA . 110 (20): 8194–9. Bibcode :2013PNAS..110.8194Q. doi : 10.1073/pnas.1303037110 . PMC 3657805 . PMID 23610427.
- ^ Kapoor A, Simmonds P, Scheel TK, et al. (2013). "Identificación de homólogos de roedores del virus de la hepatitis C y pegivirus". mBio . 4 (2): e00216–13. doi :10.1128/mBio.00216-13. PMC 3622934 . PMID 23572554.
- ^ abcdefghijk Dubuisson, Jean; Cosset, François-Loïc (2014). "Virología y biología celular del ciclo de vida del virus de la hepatitis C: una actualización". Journal of Hepatology . 61 (1): S3–S13. doi : 10.1016/j.jhep.2014.06.031 . hdl : 20.500.12210/28720 . PMID 25443344.
- ^ abc Kaito, Masahiko; Ishida, Satoshi; Tanaka, Hideaki; Horiike, Shinichiro; Fujita, Naoki; Adachi, Yukihiko; Kohara, Michinori; Konishi, Masayoshi; Watanabe, Shozo (junio de 2006). "Morfología de las partículas del virus de la hepatitis C y la hepatitis B detectadas mediante microscopía electrónica inmuno-oro". Morfología molecular médica . 39 (2): 63–71. doi : 10.1007/s00795-006-0317-8 . ISSN 1860-1480. PMID 16821143. S2CID 24668769.
- ^ Op De Beeck A, Dubuisson J (2003). "Topología de las glicoproteínas de la envoltura del virus de la hepatitis C". Rev. Med. Virol . 13 (4): 233–41. doi :10.1002/rmv.391. PMID 12820185. S2CID 22280227.
- ^ abc Castelli, Matteo; Clementi, Nicola; Pfaff, Jennifer; Sautto, Giuseppe A.; Diotti, Roberta A.; Burioni, Roberto ; Doranz, Benjamín J.; Dal Peraro, Matteo; Clementi, Massimo (16 de marzo de 2017). "Un modelo estructural de heterodímero VHC E1E2 biológicamente validado". Informes científicos . 7 (1): 214. Código bibliográfico : 2017NatSR...7..214C. doi :10.1038/s41598-017-00320-7. ISSN 2045-2322. PMC 5428263 . PMID 28303031.
- ^ Basu, Arnab; Beyene, Aster; Meyer, Keith; Ray, Ranjit (mayo de 2004). "La región hipervariable 1 de la glicoproteína E2 del virus de la hepatitis C se une a los glicosaminoglicanos, pero esta unión no conduce a la infección en un sistema pseudotípico". Journal of Virology . 78 (9): 4478–4486. doi :10.1128/JVI.78.9.4478-4486.2004. ISSN 0022-538X. PMC 387685 . PMID 15078928.
- ^ Kato N (2000). "Genoma del virus de la hepatitis C humana (VHC): organización génica, diversidad de secuencias y variación". Microb. Comp. Genom . 5 (3): 129–51. doi :10.1089/mcg.2000.5.129. PMID 11252351.
- ^ Jubin R (2001). "IRES de la hepatitis C: traducir la traducción en un objetivo terapéutico". Curr. Opin. Mol. Ther . 3 (3): 278–87. PMID 11497352.
- ^ Berry KE, Waghray S, Mortimer SA, Bai Y, Doudna JA (octubre de 2011). "La estructura cristalina del dominio central IRES del VHC revela la estrategia para el posicionamiento del codón de inicio". Structure . 19 (10): 1456–66. doi :10.1016/j.str.2011.08.002. PMC 3209822 . PMID 22000514.
- ^ Dubuisson J (2007). "Proteínas del virus de la hepatitis C". World J. Gastroenterol . 13 (17): 2406–15. doi : 10.3748/wjg.v13.i17.2406 . PMC 4146758 . PMID 17552023.
- ^ De Francesco R (1999). "Virología molecular del virus de la hepatitis C". J Hepatol . 31 (Supl 1): 47–53. doi :10.1016/S0168-8278(99)80374-2. PMID 10622560.
- ^ Gupta G, Qin H, Song J (2012). "El dominio 3 intrínsecamente no estructurado de la proteína NS5A del virus de la hepatitis C forma un "complejo difuso" con el dominio VAPB-MSP que porta mutaciones que causan ELA". PLOS ONE . 7 (6): e39261. Bibcode :2012PLoSO...739261G. doi : 10.1371/journal.pone.0039261 . PMC 3374797 . PMID 22720086.
- ^ Jin, Z; Leveque, V; Ma, H; Johnson, KA; Klumpp, K (2012). "Ensamblaje, purificación y análisis cinético de estado preestacionario del complejo de elongación de la ARN polimerasa dependiente de ARN activa". Journal of Biological Chemistry . 287 (13): 10674–83. doi : 10.1074/jbc.M111.325530 . PMC 3323022 . PMID 22303022.
- ^ Moradpour D, Penin F, Rice CM (junio de 2007). "Replicación del virus de la hepatitis C". Nat. Rev. Microbiol . 5 (6): 453–63. doi :10.1038/nrmicro1645. PMID 17487147. S2CID 13176201.
- ^ Rigat K, Wang Y, Hudyma TW, et al. (noviembre de 2010). "Cambios inducidos por ligando en la estructura de la polimerasa NS5B del virus de la hepatitis C". Antiviral Res . 88 (2): 197–206. doi :10.1016/j.antiviral.2010.08.014. PMID 20813137.
- ^ Biswal BK, Cherney MM, Wang M, et al. (mayo de 2005). "Las estructuras cristalinas del genotipo 2a de la ARN polimerasa dependiente de ARN del virus de la hepatitis C revelan dos conformaciones y sugieren mecanismos de inhibición por inhibidores no nucleósidos". J. Biol. Chem . 280 (18): 18202–10. doi : 10.1074/jbc.M413410200 . PMID 15746101.
- ^ O'Farrell D, Trowbridge R, Rowlands D, Jäger J (febrero de 2003). "Complejos de sustrato de la ARN polimerasa del virus de la hepatitis C (HC-J4): evidencia estructural de la importación de nucleótidos y la iniciación de novo". J. Mol. Biol . 326 (4): 1025–35. doi :10.1016/s0022-2836(02)01439-0. PMID 12589751.
- ^ Biswal BK, Wang M, Cherney MM, et al. (agosto de 2006). "Los inhibidores no nucleósidos que se unen a la polimerasa NS5B del virus de la hepatitis C revelan un nuevo mecanismo de inhibición". J. Mol. Biol . 361 (1): 33–45. doi :10.1016/j.jmb.2006.05.074. PMID 16828488.
- ^ Walewski JL, Keller TR, Stump DD, Branch AD (2001). "Evidencia de un nuevo antígeno del virus de la hepatitis C codificado en un marco de lectura superpuesto". ARN . 7 (5): 710–721. doi :10.1017/S1355838201010111 (inactivo 2024-11-02). PMC 1370123 . PMID 11350035.
{{cite journal}}: CS1 maint: DOI inactive as of November 2024 (link) - ^ Baghbani-arani F, Roohvand F, Aghasadeghi MR, Eidi A, Amini S, Motevalli F, Sadat SM, Memarnejadian A, Khalili G, et al. (2012). "Expresión y caracterización de la proteína ARFP/F del virus de la hepatitis C derivada de Escherichia coli". Mol Biol (Mosk) . 46 (2): 251–9. doi :10.1134/S0026893312020033. PMID 22670521. S2CID 7379944.
- ^ Bartenschlager R, Lohmann V (julio de 2000). "Replicación del virus de la hepatitis C". J. Gen. Virol . 81 (Pt 7): 1631–48. CiteSeerX 10.1.1.319.8775 . doi :10.1099/0022-1317-81-7-1631 (inactivo el 1 de noviembre de 2024). PMID 10859368. Archivado desde el original el 2009-12-03 . Consultado el 2010-07-16 .
{{cite journal}}: CS1 maint: DOI inactive as of November 2024 (link) - ^ Zeisel, M.; Barth, H.; Schuster, C.; Baumert, T. (2009). "Entrada del virus de la hepatitis C: mecanismos moleculares y objetivos para la terapia antiviral". Frontiers in Bioscience . 14 (8): 3274–3285. Bibcode :2009CNSNS..14.3274H. doi :10.1016/j.cnsns.2008.11.006. PMC 3235086 . PMID 19273272.
- ^ Kohaar, I.; Ploss, A.; Korol, E.; Mu, K.; Schoggins, J.; O'Brien, T.; Rice, C.; Prokunina-Olsson, L. (2010). "Diversidad de empalme del gen OCLN humano y su importancia biológica para la entrada del virus de la hepatitis C". Journal of Virology . 84 (14): 6987–6994. doi :10.1128/JVI.00196-10. PMC 2898237 . PMID 20463075.
- ^ Lindenbach B, Rice C (2005). "Descifrando la replicación del virus de la hepatitis C desde el genoma hasta la función". Nature . 436 (7053): 933–8. Bibcode :2005Natur.436..933L. doi : 10.1038/nature04077 . PMID 16107832.
- ^ Branch, AD; Stump, DD; Gutierrez, JA; Eng, F.; Walewski, JL (2005). "El marco de lectura alternativo (ARF) del virus de la hepatitis C y su familia de productos novedosos: la proteína de marco de lectura alternativo/proteína F, la proteína de doble desplazamiento del marco de lectura y otros". Seminarios sobre enfermedades hepáticas . 25 (1): 105–117. doi :10.1055/s-2005-864786. PMID 15732002. S2CID 260316882.
- ^ Dubuisson J, Penin F, Moradpour D (2002). "Interacción de las proteínas del virus de la hepatitis C con las membranas y los lípidos de las células huésped". Trends Cell Biol . 12 (11): 517–523. doi :10.1016/S0962-8924(02)02383-8. PMID 12446113.
- ^ Egger D, Wölk B, Gosert R, Bianchi L, Blum HE, Moradpour D, Bienz K (2002). "La expresión de proteínas del virus de la hepatitis C induce alteraciones de membrana distintivas, incluido un complejo de replicación viral candidato". J Virol . 76 (12): 5974–84. doi :10.1128/JVI.76.12.5974-5984.2002. PMC 136238 . PMID 12021330.
- ^ Boulant S, Douglas MW, Moody L, Budkowska A, Targett-Adams P, McLauchlan J (2008). "La proteína central del virus de la hepatitis C induce la redistribución de las gotas de lípidos de una manera dependiente de los microtúbulos y la dineína". Traffic . 9 (8): 1268–82. doi : 10.1111/j.1600-0854.2008.00767.x . PMID 18489704. S2CID 20609887.
- ^ Syed GH, Amako Y, Siddiqui A (2010). "El virus de la hepatitis C secuestra el metabolismo lipídico del huésped". Trends Endocrinol Metab . 21 (1): 33–40. doi :10.1016/j.tem.2009.07.005. PMC 2818172 . PMID 19854061.
- ^ Simmonds P, Holmes EC, Cha TA, et al. (noviembre de 1993). "Clasificación del virus de la hepatitis C en seis genotipos principales y una serie de subtipos mediante análisis filogenético de la región NS-5" (PDF) . J. Gen. Virol . 74 (Pt 11): 2391–9. CiteSeerX 10.1.1.325.7888 . doi :10.1099/0022-1317-74-11-2391. PMID 8245854. S2CID 17597460 . Consultado el 10 de julio de 2020 .
- ^ Nakano, Tatsunori; Lau, Gillian MG; Lau, Grace ML; Sugiyama, Masaya; Mizokami, Masashi (9 de octubre de 2011). "Análisis actualizado de los genotipos y subtipos del virus de la hepatitis C basado en la región codificante completa". Liver International . 32 (2): 339–45. doi :10.1111/j.1478-3231.2011.02684.x. PMID 22142261. S2CID 23271017.
- ^ Ohno O, Mizokami M, Wu RR, Saleh MG, Ohba K, Orito E, Mukaide M, Williams R, Lau JY, et al. (2007). "Nuevo sistema de genotipado del virus de la hepatitis C (VHC) que permite la identificación de los genotipos 1a, 1b, 2a, 2b, 3a, 3b, 4, 5a y 6a". J Clin Microbiol . 35 (1): 201–7. doi :10.1128/JCM.35.1.201-207.1997. PMC 229539 . PMID 8968908.
- ^ Simmonds P; Bukh J; Combate C; Delegado G; Enomoto N; Piedra fina S; Halfon P; Inchauspé G; Kuiken C; Maertens G; Mizokami M; Murphy, director general; Okamoto, H; Pawlotsky, JM; Peñín, F; Sablón, E; Shin-I, T; Stuyver, LJ; Thiel, HJ; Viazov, S; Weiner, AJ; Widell, A (2005). "Propuestas de consenso para un sistema unificado de nomenclatura de genotipos del virus de la hepatitis C". Hepatología . 42 (4): 962–73. doi : 10.1002/hep.20819 . PMID 16149085. S2CID 21393716.
- ^ Yu ML, Chuang WL (2009). "Tratamiento de la hepatitis C crónica en Asia: cuando Oriente se encuentra con Occidente". J Gastroenterol Hepatol . 24 (3): 336–345. doi : 10.1111/j.1440-1746.2009.05789.x . PMID 19335784. S2CID 27333980.
- ^ Muir, AJ; Bornstein, JD; Killenberg, PG; Atlantic Coast Hepatitis Treatment Group (2004). "Peginterferón alfa-2b y ribavirina para el tratamiento de la hepatitis C crónica en negros y blancos no hispanos". N Engl J Med . 350 (22): 2265–71. doi : 10.1056/NEJMoa032502 . PMID 15163776.Fe de erratas: doi :10.1056/nejm200409163511229
- ^ Ge, D; Fellay, J; Thompson, AJ; Simon, SJ; Shianna, KV; Urban, TJ; Heinzen, EL; et al. (2009). "La variación genética en IL28B predice la eliminación viral inducida por el tratamiento de la hepatitis C". Nature . 461 (7262): 399–401. Bibcode :2009Natur.461..399G. doi :10.1038/nature08309. PMID 19684573. S2CID 1707096.
- ^ Rose, R; Markov, PV; Lam, TT; Pybus, OG (2013). "La evolución viral explica las asociaciones entre el genotipo del virus de la hepatitis C, los resultados clínicos y la variación genética humana". Infect Genet Evol . 20 : 418–21. Bibcode :2013InfGE..20..418R. doi :10.1016/j.meegid.2013.09.029. hdl : 10722/221827 . PMID 24140473.
- ^ Laskus T, Wang LF, Radkowski M, Vargas H, Nowicki M, Wilkinson J, Rakela J (2001). "La exposición de receptores con ARN positivo para el virus de la hepatitis C (VHC) a donantes de sangre con ARN positivo para el VHC da como resultado un rápido predominio de una única cepa del donante y la exclusión y/o supresión de la cepa del receptor". Journal of Virology . 75 (5): 2059–66. doi :10.1128/JVI.75.5.2059-2066.2001. PMC 114790 . PMID 11160710.
- ^ ab González-Candelas F, López-Labrador FX, Bracho MA (octubre de 2011). "Recombinación en el virus de la hepatitis C". Virus . 3 (10): 2006–24. doi : 10.3390/v3102006 . PMC 3205392 . PMID 22069526.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ^ ab Shepard, CW; Finelli, L; Alter, MJ (septiembre de 2005). "Epidemiología global de la infección por el virus de la hepatitis C". Lancet Infect Dis . 5 (9): 558–67. doi :10.1016/S1473-3099(05)70216-4. PMID 16122679.
- ^ Alter, MJ (noviembre de 2011). "Vías de transmisión del VHC: lo que se siembra se cosecha". Semin Liver Dis . 31 (4): 340–6. doi :10.1055/s-0031-1297923. PMID 22189974. S2CID 260310609.
- ^ Pybus, OG; Markov, PV; Wu, A; Tatem, AJ (julio de 2007). "Investigación de la transmisión endémica del virus de la hepatitis C". Int J Parasitol . 37 (8–9): 839–49. doi :10.1016/j.ijpara.2007.04.009. PMID 17521655.
- ^ Salemi M, Vandamme AM (2002). "Estudio de los patrones evolutivos del virus de la hepatitis C mediante el análisis de secuencias del genoma completo". J Mol Evol . 54 (1): 62–70. Bibcode :2002JMolE..54...62S. doi : 10.1007/s00239-001-0018-9 . PMID 11734899. S2CID 35899454.
- ^ Sarwar MT, et al. (2011). "La proteína NS4A como marcador de la historia del VHC sugiere que diferentes genotipos del VHC evolucionaron originalmente a partir del genotipo 1b". Virol. J. 8 : 317. doi : 10.1186/1743-422X-8-317 . PMC 3145594. PMID 21696641 .
- ^ ab Pybus OG, Barnes E, Taggart R, Lemey P, Markov PV, Rasachak B, Syhavong B, Phetsouvanah R, Sheridan I, et al. (2009). "Historia genética del virus de la hepatitis C en el este de Asia". J Virol . 83 (2): 1071–82. doi :10.1128/JVI.01501-08. PMC 2612398 . PMID 18971279.
- ^ Kato N, Ueda Y, Sejima H, Gu W, Satoh S, Dansako H, Ikeda M, Shimotohno K (2019) Estudio de múltiples variaciones genéticas causadas por la replicación persistente del virus de la hepatitis C en cultivos celulares a largo plazo. Arch Virol
- ^ Simmonds P, Smith DB (1997). "Investigación del patrón de diversidad del virus de la hepatitis C en relación con los tiempos de transmisión". Journal of Viral Hepatitis . 4 (Supl 1): 69–74. doi :10.1111/j.1365-2893.1997.tb00163.x. PMID 9097281. S2CID 41594303.
- ^ Magiorkinis G, Magiorkinis E, Paraskevis D, et al. (Diciembre de 2009). "La propagación global del virus de la hepatitis C 1a y 1b: un análisis filodinámico y filogeográfico". PLOS Med . 6 (12): e1000198. doi : 10.1371/journal.pmed.1000198 . PMC 2795363 . PMID 20041120.
- ^ Markov PV, Pepin J, Frost E, Deslandes S, Labbé AC, Pybus OG (septiembre de 2009). "Filogeografía y epidemiología molecular del genotipo 2 del virus de la hepatitis C en África". J. General Virol . 90 (parte 9): 2086–96. doi : 10.1099/vir.0.011569-0 . PMID 19474244.
- ^ Markov, PV; van de Laar, TJ; Tomás, XV; Aronson, SJ; Weegink, CJ; van den Berk, GE; Prins, M.; et al. (2012). "La historia colonial y la transmisión contemporánea dan forma a la diversidad genética del genotipo 2 del virus de la hepatitis C en Ámsterdam". J Virol . 86 (14): 7677–7687. doi :10.1128/JVI.06910-11. PMC 3416291 . PMID 22573865.
- ^ Simmonds P (noviembre de 2004). "Diversidad genética y evolución del virus de la hepatitis C: 15 años después". J. Gen. Virol . 85 (Pt 11): 3173–88. doi : 10.1099/vir.0.80401-0 . PMID: 15483230.
- ^ Simmonds P (2001). "Reconstrucción de los orígenes de los virus de la hepatitis humana". Philos Trans R Soc Lond B Biol Sci . 356 (1411): 1013–26. doi :10.1098/rstb.2001.0890. PMC 1088496. PMID 11516379 .
- ^ Yu CI, Chiang BL (2010). "Una nueva perspectiva sobre el desarrollo de la vacuna contra la hepatitis C". J. Biomed. Biotechnol . 2010 : 1–12. doi : 10.1155/2010/548280 . PMC 2896694. PMID 20625493 .
- ^ Rauch, A.; Gaudieri, S.; Thio, C.; Bochud, PY (2009). "Determinantes genéticos del huésped de la eliminación espontánea de la hepatitis C". Farmacogenómica . 10 (11): 1819–1837. doi :10.2217/pgs.09.121. PMID 19891557.
- ^ Meier V, Ramadori G (abril de 2009). "Virología del virus de la hepatitis C y nuevos objetivos de tratamiento". Expert Rev Anti Infect Ther . 7 (3): 329–50. doi :10.1586/eri.09.12. PMID 19344246. S2CID 38411966.
- ^ Manns MP, Foster GR, Rockstroh JK, Zeuzem S, Zoulim F, Houghton M (diciembre de 2007). "El camino a seguir en el tratamiento del VHC: encontrar el camino correcto". Nat Rev Drug Discov . 6 (12): 991–1000. doi : 10.1038/nrd2411 . PMID 18049473. S2CID 52874660.
- ^ Ahmed, Ali Mahmoud; Doheim, Mohamed Fahmy; Mattar, Omar Mohamed; Sherif, Nourin Ali; Truong, Duy Hieu; Pham TL, Hoa; Hirayama, Kenji; Huy, Nguyen Tien (mayo de 2018). "Beclabuvir en combinación con asunaprevir y daclatasvir para la infección por el virus de la hepatitis C de genotipo 1: una revisión sistemática y un metanálisis". J Med Virol . 90 (5): 907–918. doi :10.1002/jmv.24947. PMID 28892235. S2CID 3829214.
- ^ "La FDA aprueba Victrelis para la hepatitis C" (nota de prensa) . FDA. 13 de mayo de 2011.
- ^ Gutierrez JA, Parikh N, Branch AD (2011). "Funciones clásicas y emergentes de la vitamina D en la infección por el virus de la hepatitis C". Semin Liver Dis . 31 (4): 387–398. doi :10.1055/s-0031-1297927. PMC 4107414 . PMID 22189978.
- ^ Lange CM, Bojunga J, Ramos-Lopez E, von Wagner M, Hassler A, Vermehren J, Herrmann E, Badenhoop K, Zeuzem S, et al. (2011). "La deficiencia de vitamina D y un polimorfismo del promotor CYP27B1-1260 están asociados con la hepatitis C crónica y una respuesta deficiente a la terapia basada en interferón alfa". J Hepatol . 54 (5): 887–893. doi :10.1016/j.jhep.2010.08.036. PMID 21145801 . Consultado el 10 de julio de 2020 .
- ^ Baur K, Mertens JC, Schmitt J, et al. (2012). "El haplotipo bAt (CCA) del gen del receptor de vitamina D afecta la respuesta a la terapia basada en interferón pegilado/ribavirina en pacientes con hepatitis C crónica". Antivir. Ther . 17 (3): 541–7. doi : 10.3851/IMP2018 . PMID 22300961. S2CID 32175340.
- ^ Bitetto D, Fattovich G, Fabris C, Ceriani E, Falleti E, Fornasiere E, Pasino M, Ieluzzi D, Cussigh A, et al. (2011). "Papel complementario de la deficiencia de vitamina D y el polimorfismo C/T de la interleucina-28B rs12979860 en la predicción de la respuesta antiviral en la hepatitis C crónica". Hepatología . 53 (4): 1118–26. doi : 10.1002/hep.24201 . PMID 21480318. S2CID 5329252.
- ^ Gal-Tanamy M, Bachmetov L, Ravid A, Koren R, Erman A, Tur-Kaspa R, Zemel R (2011). "Vitamina D: un agente antiviral innato que suprime el virus de la hepatitis C en los hepatocitos humanos". Hepatología . 54 (5): 1570–9. doi : 10.1002/hep.24575 . PMID 21793032. S2CID 10090454.
- ^ Abu-Mouch S, Fireman Z, Jarchovsky J, Zeina AR, Assy N (2011). "La suplementación con vitamina D mejora la respuesta virológica sostenida en pacientes sin tratamiento previo con hepatitis C crónica (genotipo 1)". World J Gastroenterol . 17 (47): 5184–90. doi : 10.3748/wjg.v17.i47.5184 . PMC 3243885 . PMID 22215943.
- ^ Bitetto D, Fabris C, Fornasiere E, Pipan C, Fumolo E, Cussigh A, Bignulin S, Cmet S, Fontanini E, et al. (2011). "La suplementación con vitamina D mejora la respuesta al tratamiento antiviral para la hepatitis C recurrente". Transpl. Int . 24 (1): 43–50. doi :10.1111/j.1432-2277.2010.01141.x. PMID 20649944. S2CID 22124427.
- ^ Cholongitas E, Theocharidou E, Goulis J, Tsochatzis E, Akriviadis E, Burroughs K (marzo de 2012). "Artículo de revisión: los efectos extraesqueléticos de la vitamina D en la infección crónica por hepatitis C". Aliment. Pharmacol. Ther . 35 (6): 634–46. doi : 10.1111/j.1365-2036.2012.05000.x . PMID 22316435. S2CID 25534747.
- ^ Cacopardo B, Camma C, Petta S, Pinzone MR, Cappellani A, Zanghi A, Nicolosi A, Nunnari G (2012). "Papel diagnóstico y terapéutico de la vitamina D en la infección crónica por el virus de la hepatitis C". Frente Biosci . 1 (4): 1276–1286. doi :10.2741/e458. PMID 22201953.
- ^ abc Halegoua-De Marzio, Dina; Fenkel, Jonathan (27 de enero de 2014). "Medicamentos alternativos en la infección por hepatitis C". Revista Mundial de Hepatología . 6 (1): 9–16. doi : 10.4254/wjh.v6.i1.9 . PMC 3953807 . PMID 24653790.
- ^ Sarrazin C, Hézode C, Zeuzem S, Pawlotsky JM (2012). "Estrategias antivirales en la infección por el virus de la hepatitis C". J. Hepatol . 56 (Supl 1): S88–100. doi :10.1016/S0168-8278(12)60010-5. PMID 22300469.
- ^ "Comunicado de prensa, FDA, 6 de diciembre de 2013". Administración de Alimentos y Medicamentos .
- ^ "La FDA aprueba un nuevo tratamiento para el virus de la hepatitis C". Administración de Alimentos y Medicamentos . 22 de noviembre de 2013.
- ^ "Medivir: Simeprevir ha sido aprobado en Japón para el tratamiento de la infección crónica por hepatitis C de genotipo 1". The Wall Street Journal. 27 de septiembre de 2013.
- ^ "El Premio Nobel de Fisiología o Medicina 2020". NobelPrize.org .
Enlaces externos
- Artículos académicos sobre los seis genotipos del VHC Archivado el 11 de octubre de 2020 en Wayback Machine Clodovero Ferri
- Bases de datos de inmunología y secuencias del VHC en el Laboratorio Nacional de Los Álamos
- Base de datos y recursos de análisis de virus y patógenos (ViPR): Flaviviridae



