Sulfato de heparán

El heparán sulfato ( HS ) es un polisacárido lineal que se encuentra en todos los tejidos animales. [1] Se presenta como un proteoglicano (HSPG, es decir, heparán sulfato proteoglicano) en el que dos o tres cadenas de HS están unidas en estrecha proximidad a las proteínas de la superficie celular o de la matriz extracelular . [2] [3] En esta forma, el HS se une a una variedad de ligandos proteicos , incluido Wnt , [4] [5] y regula una amplia gama de actividades biológicas, incluidos los procesos de desarrollo, la angiogénesis , la coagulación sanguínea , la abolición de la actividad de desprendimiento por GrB ( Granzima B ), [6] y la metástasis tumoral . También se ha demostrado que el HS sirve como receptor celular para varios virus, incluido el virus respiratorio sincitial . [7] Un estudio sugiere que el heparán sulfato celular tiene un papel en la infección por SARS-CoV-2, particularmente cuando el virus se une con ACE2. [8]
Proteoglicanos
Los principales HSPG de la membrana celular son los sindecanos transmembrana y los glipicanos anclados al glicosilfosfatidilinositol (GPI) . [9] [10] Otras formas menores de HSPG de membrana incluyen betaglicano [11] y la isoforma V-3 de CD44 presente en los queratinocitos y monocitos activados . [12]
En la matriz extracelular, especialmente las membranas basales y las fractonas , [13] las proteínas centrales multidominio perlecano , [14] agrina [15] y colágeno XVIII [16] son las principales especies portadoras de HS.
Estructura y diferencias con la heparina
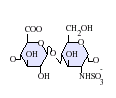
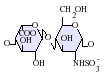
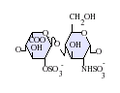
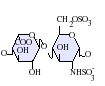
El heparán sulfato es un miembro de la familia de carbohidratos de los glicosaminoglicanos (GAG) y su estructura está muy relacionada con la de la heparina . Ambos están formados por una unidad de disacárido repetida con sulfatación variable . Las principales unidades de disacárido presentes en el heparán sulfato y la heparina se muestran a continuación.
La unidad de disacárido más común dentro del sulfato de heparán está compuesta por un ácido glucurónico (GlcA) unido a N -acetilglucosamina (GlcNAc), que normalmente constituye alrededor del 50% de las unidades de disacárido totales. Compárese esto con la heparina, donde IdoA(2S)-GlcNS(6S) constituye el 85% de las heparinas de pulmón de res y alrededor del 75% de las de mucosa intestinal porcina. Surgen problemas al definir GAG híbridos que contienen estructuras tanto "similares a la heparina" como "similares a HS". Se ha sugerido que un GAG debería calificar como heparina solo si su contenido de grupos N-sulfato excede en gran medida el de grupos N-acetilo y la concentración de grupos O-sulfato excede la de N-sulfato. De lo contrario, debería clasificarse como HS. [17]
A continuación no se muestran los raros disacáridos que contienen una glucosamina 3-O-sulfatada (GlcNS(3S,6S)) o un grupo amina libre (GlcNH 3 + ). En condiciones fisiológicas, los grupos sulfato de éster y amida se desprotonan y atraen contraiones cargados positivamente para formar una sal. [18] Se cree que el HS existe en esta forma en la superficie celular.
- GlcA-GlcNAc
- GlcA-GlcNS
- IdoA-GlcNS
- IdoA(2S)-GlcNS
- IdoA-GlcNS(6S)
- IdoA(2S)-GlcNS(6S)
Abreviaturas
- GAG = glicosaminoglicano
- GlcA = β- D - ácido glucurónico
- IdoA = ácido α- L - idurónico
- IdoA(2S) = ácido 2- O -sulfo-α- L -idurónico
- GlcNAc = 2-desoxi-2-acetamido-α- D -glucopiranosil
- GlcNS = 2-desoxi-2-sulfamido-α- D -glucopiranosil
- GlcNS(6S) = 2-desoxi-2-sulfamido-α- D -glucopiranosil-6- O -sulfato
Biosíntesis
Muchos tipos de células diferentes producen cadenas de HS con muchas estructuras primarias diferentes. Por lo tanto, existe una gran variabilidad en la forma en que se sintetizan las cadenas de HS, lo que produce una diversidad estructural englobada en el término "heparanoma", que define la gama completa de estructuras primarias producidas por una célula, tejido u organismo en particular. [19] Sin embargo, esencial para la formación de HS independientemente de la secuencia primaria es una gama de enzimas biosintéticas. Estas enzimas consisten en múltiples glicosiltransferasas , sulfotransferasas y una epimerasa . Estas mismas enzimas también sintetizan heparina .
En la década de 1980, Jeffrey Esko fue el primero en aislar y caracterizar mutantes de células animales alterados en el ensamblaje del sulfato de heparán. [20] Muchas de estas enzimas ya han sido purificadas, clonadas molecularmente y sus patrones de expresión estudiados. A partir de este trabajo y de los primeros trabajos sobre las etapas fundamentales de la biosíntesis de HS/heparina utilizando un sistema libre de células de mastocitoma de ratón, se sabe mucho sobre el orden de las reacciones enzimáticas y su especificidad. [21]
Iniciación de la cadena

La síntesis de HS se inicia con la transferencia de xilosa desde la UDP -xilosa por la xilosiltransferasa (XT) a residuos de serina específicos dentro del núcleo de la proteína. La unión de dos residuos de galactosa (Gal) por las galactosiltransferasas I y II (GalTI y GalTII) y ácido glucurónico (GlcA) por la glucuronosiltransferasa I (GlcATI) completa la formación de un cebador tetrasacárido O -ligado a una serina de la proteína central: [22]
βGlcUA-(1→3)-βGal-(1→3)-βGal-(1→4)-βXyl- O -Ser.
Las vías de biosíntesis de HS/heparina o de sulfato de condroitina (CS) y de dermatán sulfato (DS) divergen después de la formación de esta estructura de enlace tetrasacárido común. La siguiente enzima en actuar, GlcNAcT-I o GalNAcT-I, dirige la síntesis, ya sea hacia HS/heparina o hacia CS/DS, respectivamente. [23]
Se cree que la unión de la xilosa a la proteína central ocurre en el retículo endoplásmico (RE) y que el ensamblaje posterior de la región de enlace y el resto de la cadena se produce en el aparato de Golgi . [22] [23]
Alargamiento de la cadena
Después de la unión del primer residuo de N -acetilglucosamina (GlcNAc), la elongación del tetrasacárido enlazador continúa mediante la adición gradual de residuos de GlcA y GlcNAc. Estos se transfieren desde sus respectivos nucleótidos de UDP-azúcar. Esto lo llevan a cabo las proteínas de la familia EXT con actividades de glicosiltransferasa. Los genes de la familia EXT son supresores de tumores. [22] [24]
Las mutaciones en los loci del gen EXT1-3 en humanos provocan una incapacidad de las células para producir HS y el desarrollo de la enfermedad denominada exostosis hereditaria múltiple (EMH). La EHM se caracteriza por tumores con cubierta de cartílago, conocidos como osteocondromas o exostosis, que se desarrollan principalmente en los huesos largos de los individuos afectados desde la primera infancia hasta la pubertad. [25]
Modificación de cadena
A medida que una cadena HS se polimeriza, sufre una serie de reacciones de modificación llevadas a cabo por cuatro clases de sulfotransferasas y una epimerasa. La disponibilidad del donante de sulfato PAPS es crucial para la actividad de las sulfotransferasas. [26] [27]
N-desacetilación/N-sulfatación
La primera modificación del polímero es la N-desacetilación/N-sulfatación de los residuos de GlcNAc en GlcNS. Este es un prerrequisito para todas las reacciones de modificación posteriores, y lo llevan a cabo uno o más miembros de una familia de cuatro enzimas GlcNAc N-desacetilasa/N-sulfotransferasa (NDST). En estudios tempranos, se demostró que las enzimas modificadoras podían reconocer y actuar sobre cualquier residuo N-acetilado en el polímero en formación. [28] Por lo tanto, la modificación de los residuos de GlcNAc debería ocurrir aleatoriamente a lo largo de la cadena. Sin embargo, en HS, los residuos N-sulfatados se agrupan principalmente y se separan por regiones de N-acetilación donde la GlcNAc permanece sin modificar.
Existen cuatro isoformas de NDST (NDST1–4). Las actividades de N-desacetilasa y N-sulfotransferasa están presentes en todas las isoformas de NDST, pero difieren significativamente en sus actividades enzimáticas. [29]
Generación de GlcNH2
Debido a que la N-desacetilasa y la N-sulfotransferasa son llevadas a cabo por la misma enzima, la N-sulfatación normalmente está estrechamente acoplada a la N-acetilación. Se han encontrado residuos de GlcNH2 resultantes de un aparente desacoplamiento de las dos actividades en la heparina y algunas especies de HS. [30]
Epimerización y 2-O-sulfatación
La epimerización es catalizada por una enzima, la epimerasa C5 de GlcA o heparosano-N-sulfato-glucuronato 5-epimerasa ( EC 5.1.3.17). Esta enzima epimeriza la GlcA a ácido idurónico (IdoA). El reconocimiento del sustrato requiere que el residuo de GlcN unido al lado no reductor de un objetivo potencial de GlcA esté N-sulfatado. La uronosil-2-O-sulfotransferasa (2OST) sulfata los residuos de IdoA resultantes.
6-O-sulfatación
Se han identificado tres glucosaminil 6-O-transferasas (6OST) que dan lugar a la formación de GlcNS(6S) adyacente a IdoA sulfatado o no sulfatado. La GlcNAc(6S) también se encuentra en cadenas HS maduras.
3-O-sulfatación
Actualmente se sabe que existen siete glucosaminil 3- O -sulfotransferasas (3OST, HS3ST) en mamíferos (ocho en el pez cebra). [31] [32] Las enzimas 3OST crean varios disacáridos 3- O -sulfatados posibles, incluidos GlcA-GlcNS(3S±6S) (modificado por HS3ST1 y HS3ST5), IdoA(2S)-GlcNH 2 (3S±6S) (modificado por HS3ST3A1 , HS3ST3B1 , HS3ST5 y HS3ST6) y GlcA/IdoA(2S)-GlcNS(3S) (modificado por HS3ST2 y HS3ST4). [33] [34] [35] [36] Al igual que todas las demás sulfotransferasas de HS, las 3OST utilizan 3'-fosfoadenosina-5'-fosfosulfato (PAPS) como donante de sulfato. A pesar de ser la familia más grande de enzimas de modificación de HS, las 3OST producen la modificación de HS más rara, la 3- O -sulfatación de residuos específicos de glucosamina en la fracción C3-OH. [37]
Las 3OST se dividen en dos subcategorías funcionales, las que generan un sitio de unión de la antitrombina III ( HS3ST1 y HS3ST5) y las que generan un sitio de unión de la glicoproteína D del virus del herpes simple 1 (HSV-1 gD) ( HS3ST2 , HS3ST3A1 , HS3ST3B1 , HS3ST4, HS3ST5 y HS3ST6). [33] [34] [35] [36] [38] [39] [40] [41] [42] [43] [44] Como las 3OST son la familia más grande de enzimas de modificación de HS y sus acciones son limitantes de la velocidad, específicas del sustrato y producen modificaciones raras, se ha planteado la hipótesis de que la HS modificada por 3OST desempeña un papel regulador importante en los procesos biológicos. [36] [39] Se ha demostrado que la 3- O -sulfatación puede mejorar la unión de Wnt al glipicano y puede desempeñar un papel en la regulación de Wnt en el cáncer. [5] [10]
Unión de ligando
El heparán sulfato se une a una gran cantidad de proteínas extracelulares. A menudo, se las denomina colectivamente “interactoma de heparina” o “proteínas de unión a heparina”, porque se aíslan mediante cromatografía de afinidad en el polisacárido relacionado heparina, aunque el término “interactoma de heparán sulfato” es más correcto. Las funciones de las proteínas de unión al heparán sulfato varían desde componentes de la matriz extracelular hasta enzimas y factores de coagulación, y la mayoría de los factores de crecimiento, citocinas, quimiocinas y morfógenos [45]. El laboratorio de Mitchell Ho en el NCI aisló el anticuerpo monoclonal humano HS20 con alta afinidad por el heparán sulfato mediante presentación en fagos [46] . El anticuerpo se une al heparán sulfato, no al condroitín sulfato [5] . La unión del HS20 al heparán sulfato requiere sulfatación tanto en la posición C2 como en la posición C6. HS20 bloquea la unión de Wnt al sulfato de heparán [5] y también inhibe la entrada infecciosa del poliomavirus JC patógeno. [47]
Interferón-γ
La región de unión del receptor de la superficie celular del interferón-γ se superpone con la región de unión del HS, cerca del extremo C de la proteína. La unión del HS bloquea el sitio de unión del receptor y, como resultado, los complejos proteína-HS quedan inactivos. [48]
No lo sé
El glipicano-3 (GPC3) interactúa con Wnt y Frizzled para formar un complejo y desencadena la señalización descendente. [4] [10] Se ha establecido experimentalmente que Wnt reconoce un motivo de sulfato de heparán en GPC3, que contiene IdoA2S y GlcNS6S, y que la 3-O-sulfatación en GlcNS6S3S mejora la unión de Wnt al glipicano. [5]
También se están estudiando las propiedades de unión al HS de varias otras proteínas:
- Antitrombina III
- Factor de crecimiento de fibroblastos
- Factor de crecimiento de hepatocitos
- Interleucina-8
- Factor de crecimiento endotelial vascular
- Wnt/Sin alas
- Endostatina
Análogo de sulfato de heparán
Se cree que los análogos de heparán sulfato muestran propiedades idénticas a las de heparán sulfato, con la excepción de que son estables en un entorno proteolítico como una herida. [49] [50] Debido a que el heparán sulfato se descompone en heridas crónicas por la heparanasa, los análogos solo se unen a sitios donde el heparán sulfato natural está ausente y, por lo tanto, son resistentes a la degradación enzimática. [51] Además, la función de los análogos de heparán sulfato es la misma que la del heparán sulfato, protegiendo una variedad de ligandos proteicos como factores de crecimiento y citocinas. Al mantenerlos en su lugar, el tejido puede usar los diferentes ligandos proteicos para la proliferación.
Condiciones asociadas
La exostosis múltiple hereditaria (también conocida como exostosis hereditaria múltiple u osteocondromas múltiples) es una enfermedad hereditaria con mutaciones en los genes EXT1 y EXT2 que afectan la biosíntesis de heparán sulfato. [52] [53]
Referencias
- ^ Medeiros GF, Mendes A, Castro RA, Baú EC, Nader HB, Dietrich CP (julio de 2000). "Distribución de glicosaminoglicanos sulfatados en el reino animal: presencia generalizada de compuestos similares a la heparina en invertebrados". Biochimica et Biophysica Acta (BBA) - Temas generales . 1475 (3): 287–94. doi :10.1016/S0304-4165(00)00079-9. PMID 10913828.
- ^ Gallagher JT, Lyon M (2000). "Estructura molecular del sulfato de heparán e interacciones con factores de crecimiento y morfógenos". En Iozzo MV (ed.). Proteoglicanos: estructura, biología e interacciones moleculares . Nueva York, Nueva York: Marcel Dekker Inc. págs. 27–59.
- ^ Iozzo RV (1998). "Proteoglicanos de matriz: del diseño molecular a la función celular". Revisión anual de bioquímica . 67 : 609–52. doi : 10.1146/annurev.biochem.67.1.609 . PMID 9759499. S2CID 14638091.
- ^ ab Gao W, Kim H, Feng M, Phung Y, Xavier CP, Rubin JS, Ho M (agosto de 2014). "Inactivación de la señalización de Wnt por un anticuerpo humano que reconoce las cadenas de sulfato de heparán de glipicano-3 para la terapia del cáncer de hígado". Hepatología . 60 (2): 576–87. doi :10.1002/hep.26996. PMC 4083010 . PMID 24492943.
- ^ abcde Gao W, Xu Y, Liu J, Ho M (mayo de 2016). "Mapeo de epítopos por un anticuerpo bloqueador de Wnt: evidencia del dominio de unión a Wnt en heparán sulfato". Scientific Reports . 6 : 26245. Bibcode :2016NatSR...626245G. doi :10.1038/srep26245. PMC 4869111 . PMID 27185050.
- ^ Buzza MS, Zamurs L, Sun J, Bird CH, Smith AI, Trapani JA, et al. (junio de 2005). "Remodelación de la matriz extracelular por la granzima B humana a través de la escisión de vitronectina, fibronectina y laminina". The Journal of Biological Chemistry . 280 (25): 23549–58. doi : 10.1074/jbc.M412001200 . PMID 15843372.
- ^ Hallak LK, Spillmann D, Collins PL, Peeples ME (noviembre de 2000). "Requerimientos de sulfatación de glicosaminoglicanos para la infección por el virus respiratorio sincitial". Journal of Virology . 74 (22): 10508–13. doi :10.1128/JVI.74.22.10508-10513.2000. PMC 110925 . PMID 11044095.
- ^ Clausen TM, Sandoval DR, Spliid CB, Pihl J, Perrett HR, Painter CD, et al. (14 de septiembre de 2020). "La infección por SARS-CoV-2 depende del heparán sulfato celular y de la ECA2". The Journal of Cell . 183 (4): 1043–1057.e15. doi :10.1016/j.cell.2020.09.033. PMC 7489987 . PMID 32970989.
- ^ Ho M, Kim H (febrero de 2011). "Glypican-3: un nuevo objetivo para la inmunoterapia contra el cáncer". Revista Europea del Cáncer . 47 (3): 333–8. doi :10.1016/j.ejca.2010.10.024. PMC 3031711 . PMID 21112773.
- ^ abc Li N, Gao W, Zhang YF, Ho M (noviembre de 2018). "Glipícanos como dianas terapéuticas contra el cáncer". Tendencias en el cáncer . 4 (11): 741–754. doi :10.1016/j.trecan.2018.09.004. PMC 6209326 . PMID 30352677.
- ^ Andres JL, DeFalcis D, Noda M, Massagué J (marzo de 1992). "Unión de dos familias de factores de crecimiento a dominios separados del proteoglicano betaglicano". The Journal of Biological Chemistry . 267 (9): 5927–30. doi : 10.1016/S0021-9258(18)42643-9 . PMID 1556106.
- ^ Jackson DG, Bell JI, Dickinson R, Timans J, Shields J, Whittle N (febrero de 1995). "Las formas de proteoglicanos del receptor de localización de linfocitos CD44 son variantes empalmadas alternativamente que contienen el exón v3". The Journal of Cell Biology . 128 (4): 673–85. doi :10.1083/jcb.128.4.673. PMC 2199896 . PMID 7532175.
- ^ Mercier, Frederic (2016). "Fractonas: nicho de la matriz extracelular que controla el destino de las células madre y la actividad de los factores de crecimiento en el cerebro en condiciones de salud y enfermedad". Ciencias de la vida celular y molecular . 73 (24): 4661–4674. doi :10.1007/s00018-016-2314-y. PMC 11108427 . PMID 27475964. S2CID 28119663.
- ^ Arikawa-Hirasawa E, Wilcox WR, Le AH, Silverman N, Govindraj P, Hassell JR, Yamada Y (abril de 2001). "La displasia disegmentaria, tipo Silverman-Handmaker, es causada por mutaciones funcionales nulas del gen perlecan". Nature Genetics . 27 (4): 431–4. doi :10.1038/86941. PMID 11279527. S2CID 22934192.
- ^ Verbeek, Marcel M.; Otte-Höller, Irene; van den Born, Jacob; van den Heuvel, Lambert PWJ; David, Guido; Wesseling, Pieter; de Waal, Robert MW (1999). "La agrina es un proteoglicano de sulfato de heparán importante que se acumula en el cerebro de la enfermedad de Alzheimer". La Revista Estadounidense de Patología . 155 (6). Elsevier BV: 2115–2125. doi :10.1016/s0002-9440(10)65529-0. ISSN 0002-9440. PMC 1866925 . PMID 10595940.
- ^ Kawashima, Hiroto; Watanabe, Norifumi; Hirose, Mayumi; Sol, Xin; Atarashi, Kazuyuki; Kimura, Tetsuya; Shikata, Kenichi; Matsuda, Mitsuhiro; Ogawa, Daisuke; Heljasvaara, Ritva; Rehn, Marko; Pihlajaniemi, Taina; Miyasaka, Masayuki (2003). "El colágeno XVIII, un proteoglicano de heparán sulfato de la membrana basal, interactúa con la L-selectina y la proteína quimioatrayente de monocitos-1". Revista de Química Biológica . 278 (15). Elsevier BV: 13069–13076. doi : 10.1074/jbc.m212244200 . ISSN 0021-9258. PMID 12556525.
- ^ Gallagher JT, Walker A (septiembre de 1985). "Distinciones moleculares entre el heparán sulfato y la heparina. El análisis de los patrones de sulfatación indica que el heparán sulfato y la heparina son familias separadas de polisacáridos N-sulfatados". The Biochemical Journal . 230 (3): 665–74. doi :10.1042/bj2300665. PMC 1152670 . PMID 2933029.
- ^ LA, Fransson; I, Silverberg; I, Carlstedt (1985). "Estructura de la región de enlace de la proteína con el heparán sulfato. Demostración de la secuencia galactosil-galactosil-xilosa-2-fosfato". Revista de química biológica . 260 (27). J Biol Chem: 14722–14726. doi : 10.1016/S0021-9258(17)38632-5 . ISSN 0021-9258. PMID 2932448.
- ^ Turnbull J, Powell A, Guimond S (febrero de 2001). "Sulfato de heparán: decodificación de un regulador celular multifuncional dinámico". Tendencias en biología celular . 11 (2): 75–82. doi :10.1016/s0962-8924(00)01897-3. PMID 11166215.
- ^ Esko JD, Stewart TE, Taylor WH (mayo de 1985). "Animal cell mutants flawless in glycosaminoglycan biosynthesis" (Mutantes de células animales defectuosos en la biosíntesis de glicosaminoglicanos). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 82 (10): 3197–201. Bibcode :1985PNAS...82.3197E. doi : 10.1073/pnas.82.10.3197 . PMC 397742 . PMID 3858816.
- ^ Lindahl U, Kusche-Gullberg M, Kjellén L (septiembre de 1998). "Diversidad regulada de heparán sulfato". The Journal of Biological Chemistry . 273 (39): 24979–82. doi : 10.1074/jbc.273.39.24979 . PMID 9737951.
- ^ abc Kreuger, Johan; Kjellén, Lena (4 de octubre de 2012). "Biosíntesis de sulfato de heparán". Revista de histoquímica y citoquímica . 60 (12). Publicaciones SAGE: 898–907. doi :10.1369/0022155412464972. ISSN 0022-1554. PMC 3527889 . PMID 23042481.
- ^ ab Jones, Courtney L.; Liu, Jian; Xu, Ding (2010). "Estructura, biosíntesis y función de los glicosaminoglicanos". Comprehensive Natural Products II . Elsevier. págs. 407–427. doi :10.1016/b978-008045382-8.00132-5. ISBN 9780080453828.
- ^ Busse-Wicher, Marta; Wicher, Krzysztof B.; Kusche-Gullberg, Marion (2014). "La familia de las extostosinas: proteínas con múltiples funciones". Matrix Biology . 35 . Elsevier BV: 25–33. doi :10.1016/j.matbio.2013.10.001. hdl : 1956/10590 . ISSN 0945-053X.
- ^ Beltrami G, Ristori G, Scoccianti G, Tamburini A, Capanna R (2016). "Exostosis múltiples hereditarias: una revisión de la apariencia clínica y el patrón metabólico". Casos clínicos en metabolismo mineral y óseo . 13 (2): 110–118. doi :10.11138/ccmbm/2016.13.2.110. PMC 5119707. PMID 27920806 .
- ^ Silbert JE (noviembre de 1967). "Biosíntesis de heparina. 3. Formación de un glicosaminoglicano sulfatado con una preparación microsomal a partir de tumores de mastocitos". The Journal of Biological Chemistry . 242 (21): 5146–52. doi : 10.1016/S0021-9258(18)99487-1 . PMID 4228675.
- ^ Carlsson P, Presto J, Spillmann D, Lindahl U, Kjellén L (julio de 2008). "Biosíntesis de heparina/sulfato de heparán: formación procesiva de dominios N-sulfatados". The Journal of Biological Chemistry . 283 (29): 20008–14. doi : 10.1074/jbc.M801652200 . PMID 18487608.
- ^ Höök M, Lindahl U, Hallén A, Bäckström G (agosto de 1975). "Biosíntesis de heparina. Estudios sobre el proceso de sulfatación microsomal". The Journal of Biological Chemistry . 250 (15): 6065–71. doi : 10.1016/S0021-9258(19)41159-9 . PMID 807579.
- ^ Aikawa J, Grobe K, Tsujimoto M, Esko JD (febrero de 2001). "Múltiples isozimas de heparán sulfato/heparina GlcNAc N-desacetilasa/GlcN N-sulfotransferasa. Estructura y actividad del cuarto miembro, NDST4". The Journal of Biological Chemistry . 276 (8): 5876–82. doi : 10.1074/jbc.M009606200 . PMID 11087757.
- ^ Toida T, Yoshida H, Toyoda H, Koshiishi I, Imanari T, Hileman RE, et al. (marzo de 1997). "Diferencias estructurales y presencia de grupos amino no sustituidos en sulfatos de heparán de diferentes tejidos y especies". The Biochemical Journal . 322 (Parte 2) (Parte 2): 499–506. doi :10.1042/bj3220499. PMC 1218218 . PMID 9065769.
- ^ Cadwallader AB, Yost HJ (febrero de 2007). "Patrones de expresión combinatoria de sulfotransferasas de heparán sulfato en pez cebra: III. 2-O-sulfotransferasa y C5-epimerasas". Dinámica del desarrollo . 236 (2): 581–6. doi :10.1002/dvdy.21051. PMID 17195182. S2CID 38249813.
- ^ Xu D, Tiwari V, Xia G, Clement C, Shukla D, Liu J (enero de 2005). "Caracterización de la isoforma 6 de la heparán sulfato 3-O-sulfotransferasa y su función en la asistencia a la entrada del virus del herpes simple tipo 1". The Biochemical Journal . 385 (Pt 2): 451–9. doi :10.1042/BJ20040908. PMC 1134716 . PMID 15303968.
- ^ ab Shukla D, Liu J, Blaiklock P, Shworak NW, Bai X, Esko JD, et al. (octubre de 1999). "Una nueva función del heparán sulfato 3-O-sulfatado en la entrada 1 del virus del herpes simple". Cell . 99 (1): 13–22. doi : 10.1016/s0092-8674(00)80058-6 . PMID 10520990. S2CID 14139940.
- ^ ab Xia G, Chen J, Tiwari V, Ju W, Li JP, Malmstrom A, et al. (octubre de 2002). "La isoforma 5 de la heparán sulfato 3-O-sulfotransferasa genera tanto un sitio de unión a la antitrombina como un receptor de entrada para el virus del herpes simple, tipo 1". The Journal of Biological Chemistry . 277 (40): 37912–9. doi : 10.1074/jbc.m204209200 . PMID 12138164.
- ^ ab Xu D, Tiwari V, Xia G, Clement C, Shukla D, Liu J (enero de 2005). "Caracterización de la isoforma 6 de la heparán sulfato 3-O-sulfotransferasa y su función en la asistencia a la entrada del virus del herpes simple tipo 1". The Biochemical Journal . 385 (Pt 2): 451–9. doi :10.1042/bj20040908. PMC 1134716 . PMID 15303968.
- ^ abc Lawrence R, Yabe T, Hajmohammadi S, Rhodes J, McNeely M, Liu J, et al. (julio de 2007). "Las principales 3-O-sulfotransferasas neuronales de tipo gD y sus productos en los tejidos del sistema nervioso central y periférico". Matrix Biology . 26 (6): 442–55. doi :10.1016/j.matbio.2007.03.002. PMC 1993827 . PMID 17482450.
- ^ Shworak NW, HajMohammadi S, de Agostini AI, Rosenberg RD (2003). "Ratones deficientes en heparán sulfato 3-O-sulfotransferasa-1: hemostasia normal con fenotipos perinatales inesperados". Glycoconjugate Journal . 19 (4–5): 355–61. doi :10.1023/a:1025377206600. PMID 12975616. S2CID 21853086.
- ^ Liu J, Shworak NW, Fritze LM, Edelberg JM, Rosenberg RD (octubre de 1996). "Purificación de la heparán sulfato D-glucosaminil 3-O-sulfotransferasa". The Journal of Biological Chemistry . 271 (43): 27072–82. doi : 10.1074/jbc.271.43.27072 . PMID 8900198.
- ^ ab Shworak NW, Liu J, Fritze LM, Schwartz JJ, Zhang L, Logeart D, Rosenberg RD (octubre de 1997). "Clonación molecular y expresión de ADNc de ratón y humano que codifican heparán sulfato D-glucosaminil 3-O-sulfotransferasa". The Journal of Biological Chemistry . 272 (44): 28008–19. doi : 10.1074/jbc.272.44.28008 . PMID 9346953.
- ^ Shworak NW, Liu J, Petros LM, Zhang L, Kobayashi M, Copeland NG, et al. (febrero de 1999). "Múltiples isoformas de la heparán sulfato D-glucosaminil 3-O-sulfotransferasa. Aislamiento, caracterización y expresión de ADNc humanos e identificación de loci genómicos distintos". The Journal of Biological Chemistry . 274 (8): 5170–84. doi : 10.1074/jbc.274.8.5170 . PMID 9988767.
- ^ Chen J, Duncan MB, Carrick K, Pope RM, Liu J (noviembre de 2003). "Biosíntesis de heparán sulfato 3-O-sulfatado: especificidad de sustrato única de la isoforma 5 de la heparán sulfato 3-O-sulfotransferasa". Glycobiology . 13 (11): 785–94. doi : 10.1093/glycob/cwg101 . PMID 12907690.
- ^ Duncan MB, Chen J, Krise JP, Liu J (marzo de 2004). "La biosíntesis del anticoagulante heparán sulfato por la isoforma 5 de heparán sulfato 3-O-sulfotransferasa". Biochimica et Biophysica Acta (BBA) - Temas generales . 1671 (1–3): 34–43. doi :10.1016/j.bbagen.2003.12.010. PMID 15026143.
- ^ Chen J, Liu J (septiembre de 2005). "Caracterización de la estructura del heparán sulfato que se une a la antitrombina generado por la heparán sulfato 3-O-sulfotransferasa 5". Biochimica et Biophysica Acta (BBA) - Temas generales . 1725 (2): 190–200. doi :10.1016/j.bbagen.2005.06.012. PMID 16099108.
- ^ Girardin EP, Hajmohammadi S, Birmele B, Helisch A, Shworak NW, de Agostini AI (noviembre de 2005). "La síntesis de proteoglicanos de heparán sulfato con actividad anticoagulante por las células epiteliales glomerulares implica múltiples isoformas de 3-O-sulfotransferasa y un grupo de precursores limitantes". The Journal of Biological Chemistry . 280 (45): 38059–70. doi : 10.1074/jbc.m507997200 . PMID 16107334.
- ^ Ori A, Wilkinson MC, Fernig DG (mayo de 2008). "El heparanoma y la regulación de la función celular: estructuras, funciones y desafíos". Frontiers in Bioscience . 13 (13): 4309–38. doi : 10.2741/3007 . PMID 18508513.
- ^ Kim H, Ho M (noviembre de 2018). "Aislamiento de anticuerpos contra el sulfato de heparán en glipicanos mediante visualización en fagos". Protocolos actuales en ciencia de proteínas . 94 (1): e66. doi :10.1002/cpps.66. PMC 6205898. PMID 30091851 .
- ^ Geoghegan EM, Pastrana DV, Schowalter RM, Ray U, Gao W, Ho M, et al. (octubre de 2017). "Entrada infecciosa y neutralización de poliomavirus JC patógenos". Cell Reports . 21 (5): 1169–1179. doi :10.1016/j.celrep.2017.10.027. PMC 5687836 . PMID 29091757.
- ^ Sadir R, Forest E, Lortat-Jacob H (mayo de 1998). "La secuencia de unión del heparán sulfato del interferón-gamma aumentó la tasa de formación del complejo interferón-gamma-receptor de interferón-gamma". The Journal of Biological Chemistry . 273 (18): 10919–25. doi : 10.1074/jbc.273.18.10919 . PMID 9556569.
- ^ Tong M, Tuk B, Hekking IM, Vermeij M, Barritault D, van Neck JW (2009). "Neovascularización estimulada, resolución de la inflamación y maduración del colágeno en la curación de heridas cutáneas de ratas mediante un mimético de glicosaminoglicano de heparán sulfato, OTR4120". Reparación y regeneración de heridas . 17 (6): 840–52. doi :10.1111/j.1524-475X.2009.00548.x. PMID 19903305. S2CID 17262546.
- ^ Tong M, Tuk B, Hekking IM, Pleumeekers MM, Boldewijn MB, Hovius SE, van Neck JW (2011). "Un mimético de glicosaminoglicano de heparán sulfato mejora la cicatrización de las úlceras por presión en un modelo de rata de lesión cutánea por isquemia-reperfusión". Reparación y regeneración de heridas . 19 (4): 505–14. doi :10.1111/j.1524-475X.2011.00704.x. PMID 21649786. S2CID 7380997.
- ^ Tong, Miao; Tuk, Bastiaan; Hekking, Ineke M.; Vermeij, Marcel; Barritault, Denis; van Neck, Johan W. (2009). "Neovascularización estimulada, resolución de la inflamación y maduración del colágeno en la curación de heridas cutáneas de ratas mediante un mimético de glicosaminoglicano de heparán sulfato, OTR4120". Reparación y regeneración de heridas . 17 (6). Wiley: 840–852. doi :10.1111/j.1524-475x.2009.00548.x. ISSN 1067-1927. PMID 19903305. S2CID 17262546.
- ^ Zak, Beverly M; Crawford, Brett E; Esko, Jeffrey D (19 de diciembre de 2002). "Exostosis múltiples hereditarias y polimerización de heparán sulfato". Biochimica et Biophysica Acta (BBA) - Temas generales . Glicobiología del desarrollo. 1573 (3): 346–355. doi :10.1016/S0304-4165(02)00402-6. ISSN 0304-4165.
- ^ Meneghetti, Maria CZ; Hughes, Ashley J.; Rudd, Timothy R.; Nader, Helena B.; Powell, Andrew K.; Yates, Edwin A.; Lima, Marcelo A. (6 de septiembre de 2015). "Interacciones de heparán sulfato y heparina con proteínas". Journal of the Royal Society, Interface . 12 (110): 0589. doi :10.1098/rsif.2015.0589. ISSN 1742-5662. PMC 4614469 . PMID 26289657.