Proteasa del VIH-1
| Proteasa del VIH-1 (retropepsina) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
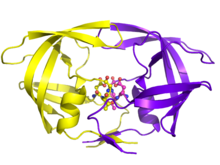
 Dímero de proteasa del VIH-1 en blanco y gris, con sustrato peptídico en negro y cadenas laterales de aspartato del sitio activo en rojo. ( PDB : 1KJF ) | |||||||||
| Identificadores | |||||||||
| N.º CE | 3.4.23.16 | ||||||||
| N.º CAS | 144114-21-6 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| Ontología genética | AmiGO / QuickGO | ||||||||
| |||||||||
La proteasa del VIH-1 o PR es una aspartil proteasa retroviral (retropepsina), una enzima involucrada en la hidrólisis del enlace peptídico en los retrovirus, que es esencial para el ciclo de vida del VIH , el retrovirus que causa el SIDA . [1] [2] La PR del VIH-1 escinde poliproteínas recién sintetizadas (a saber, Gag y Gag- Pol [3] ) en nueve sitios de escisión para crear los componentes proteicos maduros de un virión del VIH , la forma infecciosa de un virus fuera de la célula huésped. [4] Sin una PR eficaz del VIH-1, los viriones del VIH siguen sin ser infecciosos. [5] [6]
Estructura

La proteasa madura del VIH existe como un homodímero de 22 kDa , con cada subunidad formada por 99 aminoácidos. [1] Un único sitio activo se encuentra entre las subunidades idénticas y tiene la secuencia de tríada catalítica Asp - Thr - Gly (Asp25, Thr26 y Gly27) característica común a las proteasas aspárticas. [8] Como la PR del VIH-1 solo puede funcionar como un dímero, la proteasa madura contiene dos aminoácidos Asp25, uno de cada monómero, que actúan en conjunto como residuos catalíticos. [9] Además, la proteasa del VIH tiene dos "aletas" moleculares que se mueven una distancia de hasta 7 Å cuando la enzima se asocia con un sustrato. [10] Esto se puede visualizar con animaciones de las aletas abriéndose y cerrándose.
Biosíntesis

Precursor
La poliproteína Gag-Pol, que contiene proteínas de codificación prematura, incluyendo el PR del VIH-1. [9] El PR se encuentra entre la transcriptasa inversa (que está en el extremo C del PR) y la pol p6 (que está en el extremo N del PR) de la región transframe (TFR). [11]
Para que este precursor se convierta en una proteína funcional, cada monómero debe asociarse con otro monómero PR del VIH-1 para formar un sitio activo catalítico funcional, cada uno contribuyendo con el Asp25 de sus respectivas tríadas catalíticas. [9]
Mecanismo de síntesis
Cuando el ARN viral del VIH entra en la célula, va acompañado de una transcriptasa inversa , una integrasa y un PR del VIH-1 maduro. La transcriptasa inversa convierte el ARN viral en ADN, lo que facilita el papel de la integrasa en la incorporación de la información genética viral con el ADN de la célula huésped. [2] El ADN viral puede permanecer latente en el núcleo o ser transcrito en ARNm y traducido por la célula huésped en la poliproteína Gag-Pol, que luego sería escindida en proteínas funcionales individuales (incluido un PR del VIH-1 recién sintetizado) por el PR del VIH-1 maduro. [9]
El precursor PR del VIH-1 cataliza su propia producción al facilitar su escisión de la poliproteína Gag-Pol en un mecanismo conocido como autoprocesamiento. El autoprocesamiento del PR del VIH-1 se caracteriza por dos pasos secuenciales: (1) la escisión intramolecular del extremo N en el sitio de escisión de la proteasa p6 pol , que sirve para finalizar el procesamiento del PR y aumentar la actividad enzimática con el intermediario PR-transcriptasa inversa recién formado, y (2) la escisión intermolecular del extremo C en el sitio de escisión de la proteasa-transcriptasa inversa, que conduce al ensamblaje de dos subunidades de PR en dímeros maduros. [12] [13] La dimerización de las dos subunidades permite la formación de un sitio activo combinado completamente funcional, caracterizado por dos residuos catalíticos Asp25 (uno de cada monómero). [14]
Función
El PR del VIH-1 cumple una doble función. El PR precursor del VIH-1 es responsable de catalizar su propia producción en enzimas PR maduras a través del autoprocesamiento de PR. [15] La proteasa madura es capaz de hidrolizar enlaces peptídicos en las poliproteínas Gag-Pol en nueve sitios específicos, procesando las subunidades resultantes en proteínas maduras y completamente funcionales. Estas proteínas escindidas, que incluyen la transcriptasa inversa, la integrasa y la ARNasa H, están codificadas por los componentes de la región codificante necesarios para la replicación viral. [4]
Mecanismo
Como proteasa aspártica, la PR del VIH-1 dimerizada funciona a través del complejo del grupo aspartilo, para realizar la hidrólisis. De los dos residuos Asp25 en el sitio activo catalítico combinado de la PR del VIH-1, uno está desprotonado mientras que el otro está protonado, debido a las diferencias de pKa con el microambiente. [16]
En un mecanismo general de proteasa aspártica, una vez que el sustrato está unido correctamente al sitio activo de la enzima, el aminoácido catalítico Asp25 desprotonado sufre catálisis básica, convirtiendo a la molécula de agua entrante en un mejor nucleófilo al desprotonarla. El ion hidroxilo resultante ataca al carbono carbonilo del enlace peptídico, formando un intermedio con un oxianión transitorio, que es estabilizado por el Asp25 inicialmente protonado. El oxianión vuelve a formar un doble enlace, lo que lleva a la escisión del enlace peptídico entre los dos aminoácidos, mientras que el Asp25 inicialmente desprotonado sufre catálisis ácida para donar su protón al grupo amino, haciendo que el grupo amino sea un mejor grupo saliente para la escisión completa del enlace peptídico y volviendo a su estado desprotonado original. [2] [17]
Si bien la PR del VIH-1 comparte muchas de las mismas características que una proteasa aspártica no viral, algunas evidencias han demostrado que la PR del VIH-1 cataliza la hidrólisis de manera concertada; en otras palabras, la molécula de agua nucleófila y el Asp25 protonado atacan simultáneamente el enlace peptídico escindible durante la catálisis. [17] [18]

Como objetivo farmacológico
La proteasa del VIH, por su papel fundamental en la replicación del VIH, ha sido un objetivo primordial de la terapia farmacológica. Los inhibidores de la proteasa del VIH funcionan uniéndose específicamente al sitio activo imitando el intermediario tetraédrico de su sustrato y, en esencia, “quedándose pegados”, desactivando la enzima. Después del ensamblaje y la gemación, las partículas virales que carecen de proteasa activa no pueden madurar y convertirse en viriones infecciosos. Se han autorizado varios inhibidores de la proteasa para la terapia del VIH. [19]
Existen diez inhibidores de la PR del VIH-1 que están aprobados actualmente por la Administración de Alimentos y Medicamentos : indinavir , saquinavir , ritonavir , nelfinavir , lopinavir , amprenavir , fosamprenevir , atazanavir , tipranavir y darunavir . Muchos de los inhibidores tienen diferentes componentes moleculares y, por lo tanto, diferentes acciones mecanísticas, como el bloqueo del sitio activo. Sus funciones también se extienden a la influencia de las concentraciones circulantes de otros fármacos inhibidores (ritonavir) y se utilizan solo para ciertas circunstancias en las que el virus muestra tolerancia a otros inhibidores (tipranavir). [4] [20]
Evolución y resistencia
Debido a las altas tasas de mutación de los retrovirus, especialmente debido a las regiones sensibles a las mutaciones (en particular la región que contiene la secuencia de la tríada catalítica), y considerando que los cambios en unos pocos aminoácidos dentro de la proteasa del VIH pueden hacerla mucho menos visible para un inhibidor, el sitio activo de esta enzima puede cambiar rápidamente cuando está bajo la presión selectiva de fármacos inhibidores de la replicación. [21] [22]
En general, se asocian dos tipos de mutaciones con el aumento de la resistencia a los fármacos: las mutaciones "principales" y las mutaciones "secundarias". Las mutaciones principales implican una mutación en el sitio activo del PR del VIH-1, que impide que los inhibidores selectivos se unan a él. Las mutaciones secundarias se refieren a cambios moleculares en la periferia de la enzima debido a la exposición prolongada a sustancias químicas similares, que pueden afectar la especificidad del inhibidor para el PR del VIH-1. [3]
Una estrategia para minimizar el desarrollo de resistencia a los fármacos en el VIH es administrar una combinación de fármacos que inhiban varios aspectos clave del ciclo de replicación del VIH simultáneamente, en lugar de un fármaco a la vez. Otros objetivos de la terapia farmacológica incluyen la transcriptasa inversa , la adhesión del virus, la fusión de membranas, la integración del ADNc y el ensamblaje del virión. [23] [24]
Véase también
Enlaces externos
- Base de datos en línea MEROPS para peptidasas y sus inhibidores: A02.001
- Proteopedia HIV-1_proteasa : la estructura de la proteasa del VIH-1 en 3D interactivo.
- Proteopedia Flaps_Morph_for_HIV_Protease - Animación de la apertura y cierre de las aletas basada en estructuras cristalinas de rayos X.
- Proteasa del VIH-1+ en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
Referencias
- ^ ab Davies DR (1990). "La estructura y función de las proteinasas aspárticas". Revisión anual de biofísica y química biofísica . 19 (1): 189–215. doi :10.1146/annurev.bb.19.060190.001201. PMID 2194475.
- ^ abc Brik A, Wong CH (enero de 2003). "Proteasa del VIH-1: mecanismo y descubrimiento de fármacos". Química orgánica y biomolecular . 1 (1): 5–14. doi :10.1039/b208248a. PMID 12929379.
- ^ ab Huang X, Britto MD, Kear-Scott JL, Boone CD, Rocca JR, Simmerling C, Mckenna R, Bieri M, Gooley PR, Dunn BM, Fanucci GE (junio de 2014). "El papel de los polimorfismos de subtipos selectos en el muestreo y la dinámica conformacional de la proteasa del VIH-1". The Journal of Biological Chemistry . 289 (24): 17203–14. doi : 10.1074/jbc.M114.571836 . PMC 4059161 . PMID 24742668.
- ^ abc Lv Z, Chu Y, Wang Y (abril de 2015). "Inhibidores de la proteasa del VIH: una revisión de la selectividad molecular y la toxicidad". VIH/SIDA: investigación y cuidados paliativos . 7 : 95–104. doi : 10.2147/hiv.s79956 . PMC 4396582 . PMID 25897264.
- ^ Kräusslich HG, Ingraham RH, Skoog MT, Wimmer E, Pallai PV, Carter CA (febrero de 1989). "Actividad de la proteinasa biosintética purificada del virus de la inmunodeficiencia humana en sustratos naturales y péptidos sintéticos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 86 (3): 807–11. Bibcode :1989PNAS...86..807K. doi : 10.1073/pnas.86.3.807 . PMC 286566 . PMID 2644644.
- ^ Kohl NE, Emini EA, Schleif WA, Davis LJ, Heimbach JC, Dixon RA, Scolnick EM, Sigal IS (julio de 1988). "Se requiere la proteasa activa del virus de la inmunodeficiencia humana para la infectividad viral". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 85 (13): 4686–90. Bibcode :1988PNAS...85.4686K. doi : 10.1073/pnas.85.13.4686 . PMC 280500 . PMID 3290901.
- ^ Perryman AL, Lin JH, McCammon JA (abril de 2004). "Dinámica molecular de la proteasa del VIH-1 de un tipo salvaje y del mutante V82F/I84V: posibles contribuciones a la resistencia a los fármacos y un nuevo sitio diana potencial para los fármacos" (PDF) . Protein Science . 13 (4): 1108–23. doi :10.1110/ps.03468904. PMC 2280056 . PMID 15044738. Archivado desde el original (PDF) el 16 de diciembre de 2008.
- ^ Chatterjee A, Mridula P, Mishra RK, Mittal R, Hosur RV (marzo de 2005). "El plegamiento regula el autoprocesamiento del precursor de la proteasa del VIH-1". The Journal of Biological Chemistry . 280 (12): 11369–78. doi : 10.1074/jbc.M412603200 . PMID 15632156.
- ^ abcd Pettit SC, Everitt LE, Choudhury S, Dunn BM, Kaplan AH (agosto de 2004). "La escisión inicial del precursor GagPol del virus de la inmunodeficiencia humana tipo 1 por su proteasa activada se produce mediante un mecanismo intramolecular". Revista de Virología . 78 (16): 8477–85. doi :10.1128/JVI.78.16.8477-8485.2004. PMC 479095 . PMID 15280456.
- ^ Miller M, Schneider J, Sathyanarayana BK, Toth MV, Marshall GR, Clawson L, Selk L, Kent SB, Wlodawer A (diciembre de 1989). "Estructura del complejo de la proteasa sintética del VIH-1 con un inhibidor basado en sustrato a una resolución de 2,3 A". Science . 246 (4934): 1149–52. doi :10.1126/science.2686029. PMID 2686029.
- ^ Louis JM, Clore GM, Gronenborn AM (septiembre de 1999). "El autoprocesamiento de la proteasa del VIH-1 está estrechamente relacionado con el plegamiento de proteínas". Nature Structural Biology . 6 (9): 868–75. doi :10.1038/12327. PMID 10467100. S2CID 6375519.
- ^ Louis JM, Nashed NT, Parris KD, Kimmel AR, Jerina DM (agosto de 1994). "Cinética y mecanismo de autoprocesamiento de la proteasa del virus de inmunodeficiencia humana tipo 1 a partir de un análogo de la poliproteína Gag-Pol". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (17): 7970–4. Bibcode :1994PNAS...91.7970L. doi : 10.1073/pnas.91.17.7970 . PMC 44526 . PMID 8058744.
- ^ Wondrak EM, Nashed NT, Haber MT, Jerina DM, Louis JM (febrero de 1996). "Un precursor transitorio de la proteasa del VIH-1. Aislamiento, caracterización y cinética de la maduración". The Journal of Biological Chemistry . 271 (8): 4477–81. doi : 10.1074/jbc.271.8.4477 . PMID 8626801.
- ^ Zhang S, Kaplan AH, Tropsha A (noviembre de 2008). "Estudios de la estructura y la función de la proteasa del VIH-1 con el método de análisis de vecindad simplicial del empaquetamiento de proteínas". Proteins . 73 (3): 742–53. doi :10.1002/prot.22094. PMC 2765824 . PMID 18498108.
- ^ Huang L, Chen C (julio de 2013). "Comprensión del autoprocesamiento de la proteasa del VIH-1 para el desarrollo de nuevas terapias". Future Medicinal Chemistry . 5 (11): 1215–29. doi :10.4155/fmc.13.89. PMC 3826259 . PMID 23859204.
- ^ Smith R, Brereton IM, Chai RY, Kent SB (noviembre de 1996). "Estados de ionización de los residuos catalíticos en la proteasa del VIH-1". Nature Structural Biology . 3 (11): 946–50. doi :10.1038/nsb1196-946. PMID 8901873. S2CID 1076528.
- ^ ab Liu H, Müller-Plathe F, van Gunsteren WF (agosto de 1996). "Un estudio combinado de dinámica molecular cuántica/clásica del mecanismo catalítico de la proteasa del VIH". Journal of Molecular Biology . 261 (3): 454–69. doi :10.1006/jmbi.1996.0476. PMID 8780786.
- ^ Jaskólski M, Tomasselli AG, Sawyer TK, Staples DG, Heinrikson RL, Schneider J, Kent SB, Wlodawer A (febrero de 1991). "Estructura con una resolución de 2,5 A de la proteasa del virus de inmunodeficiencia humana tipo 1 sintetizada químicamente en complejo con un inhibidor basado en hidroxietileno". Bioquímica . 30 (6): 1600–9. doi :10.1021/bi00220a023. PMID 1993177.
- ^ Rang HP (2007). Farmacología de Rang y Dale (6.ª ed.). Filadelfia, Pensilvania, EE. UU.: Churchill Livingstone/Elsevier. ISBN 9780808923541.
- ^ Griffin L, Annaert P, Brouwer KL (septiembre de 2011). "Influencia de las proteínas de transporte de fármacos en la farmacocinética y las interacciones farmacológicas de los inhibidores de la proteasa del VIH". Journal of Pharmaceutical Sciences . 100 (9): 3636–54. doi :10.1002/jps.22655. PMC 3750718 . PMID 21698598.
- ^ Watkins T, Resch W, Irlbeck D, Swanstrom R (febrero de 2003). "Selección de resistencia de alto nivel a los inhibidores de la proteasa del virus de inmunodeficiencia humana tipo 1". Agentes antimicrobianos y quimioterapia . 47 (2): 759–69. doi :10.1128/AAC.47.2.759-769.2003. PMC 151730. PMID 12543689 .
- ^ Loeb DD, Swanstrom R, Everitt L, Manchester M, Stamper SE, Hutchison CA (agosto de 1989). "Mutagénesis completa de la proteasa del VIH-1". Nature . 340 (6232): 397–400. Bibcode :1989Natur.340..397L. doi :10.1038/340397a0. PMID 2666861. S2CID 4351388.
- ^ Moore JP, Stevenson M (octubre de 2000). "Nuevos objetivos para los inhibidores de la replicación del VIH-1". Nature Reviews. Molecular Cell Biology . 1 (1): 40–9. doi :10.1038/35036060. PMID 11413488. S2CID 10811618.
- ^ De Clercq E (diciembre de 2007). "El diseño de fármacos para el VIH y el VHC". Nature Reviews. Drug Discovery . 6 (12): 1001–18. doi :10.1038/nrd2424. PMID 18049474. S2CID 37859193.
