Transposón de ADN
Los transposones de ADN son secuencias de ADN, a veces denominadas "genes saltarines", que pueden moverse e integrarse en diferentes ubicaciones dentro del genoma . [1] Son elementos transponibles (ET) de clase II que se mueven a través de un intermediario de ADN , a diferencia de los ET de clase I, retrotransposones , que se mueven a través de un intermediario de ARN . [2] Los transposones de ADN pueden moverse en el ADN de un organismo a través de un intermediario de ADN monocatenario o bicatenario. [3] Se han encontrado transposones de ADN en organismos procariotas y eucariotas . Pueden constituir una parte significativa del genoma de un organismo, particularmente en eucariotas. En procariotas, los ET pueden facilitar la transferencia horizontal de resistencia a antibióticos u otros genes asociados con la virulencia . Después de replicarse y propagarse en un huésped, todas las copias de transposón se inactivan y se pierden a menos que el transposón pase a un genoma iniciando un nuevo ciclo de vida con transferencia horizontal. [4] Es importante señalar que los transposones de ADN no se insertan aleatoriamente en el genoma, sino que muestran preferencia por sitios específicos.
En cuanto al movimiento, los transposones de ADN pueden clasificarse como autónomos y no autónomos. [5] Los autónomos pueden moverse por sí solos, mientras que los no autónomos requieren la presencia del gen de otro elemento transponible, la transposasa , para moverse. Existen tres clasificaciones principales para el movimiento de los transposones de ADN: "cortar y pegar", [6] " círculo rodante " (Helitrones), [7] y "autosintetizadores" (Polintones). [8] Estos distintos mecanismos de movimiento les permiten moverse por el genoma de un organismo. Dado que los transposones de ADN no pueden sintetizar ADN, se replican utilizando la maquinaria de replicación del huésped. Estas tres clases principales se dividen a su vez en 23 superfamilias diferentes caracterizadas por su estructura, secuencia y mecanismo de acción. [9]
Los transposones de ADN son una causa de alteraciones en la expresión génica . Como ADN recién insertado en secuencias codificantes activas , pueden alterar las funciones normales de las proteínas y causar mutaciones. Los TE de clase II constituyen aproximadamente el 3% del genoma humano. Hoy en día, no hay transposones de ADN activos en el genoma humano. Por lo tanto, los elementos que se encuentran en el genoma humano se denominan fósiles.
Mecanismos de acción
Cortar y pegar

Tradicionalmente, los transposones de ADN se mueven en el genoma mediante un método de cortar y pegar. El sistema requiere una enzima transposasa que cataliza el movimiento del ADN desde su ubicación actual en el genoma y lo inserta en una nueva ubicación. La transposición requiere tres sitios de ADN en el transposón : dos en cada extremo del transposón llamados repeticiones invertidas terminales y uno en el sitio objetivo. La transposasa se unirá a las repeticiones invertidas terminales del transposón y mediará la sinapsis de los extremos del transposón. Luego, la enzima transposasa desconecta el elemento del ADN flanqueante del sitio donante original y media la reacción de unión que vincula el transposón al nuevo sitio de inserción. La adición del nuevo ADN en el sitio objetivo causa pequeños espacios a cada lado del segmento insertado. [10] Los sistemas hospedadores reparan estos espacios dando como resultado la duplicación de la secuencia objetivo (TSD) que es característica de la transposición. En muchas reacciones, el transposón se extirpa por completo del sitio donante en lo que se denomina una transposición de "cortar y pegar" [11] y se inserta en el ADN diana para formar una inserción simple. En ocasiones, también se copia y se mueve material genético que no se encontraba originalmente en el elemento transponible.
Helitrones
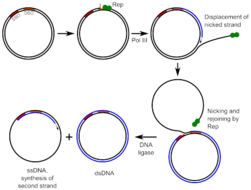
Los helitrones también son un grupo de TE eucariotas de clase II. Los helitrones no siguen el mecanismo clásico de "cortar y pegar". En cambio, se plantea la hipótesis de que se mueven por el genoma a través de un mecanismo similar al de un círculo rodante. Este proceso implica hacer un corte en una hebra circular mediante una enzima, que separa el ADN en dos hebras simples. La proteína de iniciación luego permanece unida al fosfato 5' en la hebra cortada, exponiendo el hidroxilo 3' de la hebra complementaria. Esto permite que una enzima polimerasa comience la replicación en la hebra no cortada. Finalmente, se replica toda la hebra, momento en el que el ADN recién sintetizado se disocia y se replica en paralelo con la hebra molde original. [12] Los helitrones codifican una proteína desconocida que se cree que tiene función de endonucleasa HUH, así como actividad de helicasa 5' a 3' . Esta enzima haría un corte de una sola hebra en el ADN, lo que explica la falta de duplicaciones del sitio objetivo encontradas en los helitrones. Los helitrones también fueron la primera clase de elementos transponibles que se descubrieron computacionalmente y marcaron un cambio de paradigma en la forma en que se estudiaban los genomas completos. [13]
Polintones

Los polintones también son un grupo de TE eucariotas de clase II. Como uno de los transposones de ADN más complejos conocidos en eucariotas, conforman los genomas de protistas , hongos y animales , como la entamoeba , la roya de la soja y el pollo , respectivamente. Contienen genes con homología con las proteínas virales y que a menudo se encuentran en genomas eucariotas , como la polimerasa y la integrasa retroviral . Sin embargo, no se conoce ninguna proteína funcionalmente similar a las proteínas de la cápside o la envoltura viral . Comparten sus muchas características estructurales con plásmidos lineales , bacteriófagos y adenovirus , que se replican utilizando polimerasas de ADN cebadas con proteínas. Se ha propuesto que los polintones pasan por una autosíntesis similar por su polimerasa. Los polintones, de 15 a 20 kb de longitud, codifican hasta 10 proteínas individuales. Para la replicación , utilizan una ADN polimerasa B activada por proteínas , una integrasa retroviral , una cisteína proteasa y una ATPasa . En primer lugar, durante la replicación del genoma del huésped, se escinde un elemento Polinton extracromosómico monocatenario del ADN del huésped utilizando la integrasa, formando una estructura similar a una raqueta. En segundo lugar, el Polinton experimenta una replicación utilizando la ADN polimerasa B, con la iniciación iniciada por una proteína terminal, que puede estar codificada en algunos plásmidos lineales. Una vez que se genera el Polinton bicatenario, la integrasa sirve para insertarlo en el genoma del huésped. Los Polintons exhiben una alta variabilidad entre diferentes especies y pueden estar estrechamente regulados, lo que resulta en una baja tasa de frecuencia en muchos genomas. [14]
Clasificación
A partir de la actualización más reciente en 2015, se reconocieron y anotaron 23 superfamilias de transposones de ADN en Repbase, una base de datos de elementos repetitivos de ADN mantenida por el Instituto de Investigación de Información Genética : [15]
Efectos de los transposones

Los transposones de ADN , como todos los transposones, tienen un gran impacto en la expresión genética. Una secuencia de ADN puede insertarse en un gen previamente funcional y crear una mutación . Esto puede suceder de tres maneras distintas: 1. alteración de la función, 2. reordenamiento cromosómico y 3. una fuente de material genético nuevo. [16] Dado que los transposones de ADN pueden llevarse partes de secuencias genómicas con ellos, puede ocurrir una reorganización de exones. La reorganización de exones es la creación de nuevos productos genéticos debido a la nueva ubicación de dos exones previamente no relacionados a través de la transposición. [17] Debido a su capacidad para alterar la expresión del ADN , los transposones se han convertido en un objetivo importante de la investigación en ingeniería genética .
_shown_in_her_laboratory_in_1947.jpg/440px-Barbara_McClintock_(1902-1992)_shown_in_her_laboratory_in_1947.jpg)
Ejemplos
Maíz
Barbara McClintock fue la primera en descubrir y describir los transposones de ADN en Zea mays [ 18] durante la década de 1940; este es un logro que le valdría el Premio Nobel en 1983. Describió el sistema Ac/Ds donde la unidad Ac (activador) era autónoma pero la unidad genómica Ds requería la presencia del activador para poder moverse. Este TE es uno de los más obvios visualmente, ya que fue capaz de hacer que el maíz cambiara de color de amarillo a marrón/manchado en granos individuales.
Moscas de la fruta
El transposón Mariner/Tc1 , presente en muchos animales pero estudiado en Drosophila , fue descrito por primera vez por Jacobson y Hartl. [19] El transposón Mariner es bien conocido por poder extirparse e insertarse horizontalmente en un nuevo organismo. [20] Se han encontrado miles de copias del TE dispersas en el genoma humano y en otros animales.
Los transposones Hobo en Drosophila han sido ampliamente estudiados debido a su capacidad de causar disgenesia gonadal . [21] La inserción y posterior expresión de secuencias similares a las de Hobo resultan en la pérdida de células germinales en las gónadas de las moscas en desarrollo.
Bacteria
Los transposones bacterianos son especialmente buenos para facilitar la transferencia horizontal de genes entre microbios. La transposición facilita la transferencia y acumulación de genes de resistencia a los antibióticos . En las bacterias, los elementos transponibles pueden saltar fácilmente entre el genoma cromosómico y los plásmidos . En un estudio de 1982 realizado por Devaud et al., se aisló y examinó una cepa de Acinetobacter resistente a múltiples fármacos . La evidencia apuntaba a la transferencia de un plásmido a la bacteria, donde los genes de resistencia se transponían al genoma cromosómico. [22]
Diversidad genética
Los transposones pueden tener un efecto en la promoción de la diversidad genética de muchos organismos. Los transposones de ADN pueden impulsar la evolución de los genomas al promover la reubicación de secciones de secuencias de ADN. Como resultado, esto puede alterar las regiones reguladoras de los genes y los fenotipos. [23] El descubrimiento de los transposones fue realizado por Barbara McClintock , quien notó que estos elementos podían realmente cambiar el color de las plantas de maíz que estaba estudiando, proporcionando evidencia rápida de un resultado del movimiento de transposones. [24] Otro ejemplo es el transposón de ADN Tol2 en el pez medaka que se dice que es el resultado de su variedad en los patrones de pigmentación. [25] Estos ejemplos muestran que los transposones pueden influir en gran medida en el proceso de evolución al inducir rápidamente cambios en el genoma.
Inactivación
Todos los transposones de ADN están inactivos en el genoma humano . [26] Los transposones inactivados o silenciados no producen un resultado fenotípico y no se mueven por el genoma. Algunos son inactivos porque tienen mutaciones que afectan su capacidad de moverse entre cromosomas, mientras que otros son capaces de moverse pero permanecen inactivos debido a las defensas epigenéticas, como la metilación del ADN y la remodelación de la cromatina . Por ejemplo, las modificaciones químicas del ADN pueden constreñir ciertas áreas del genoma de tal manera que las enzimas de transcripción no pueden alcanzarlas. El ARNi , específicamente el silenciamiento de ARNi y miARN , es un mecanismo natural que, además de regular la expresión génica eucariota, previene la transcripción de transposones de ADN. Otro modo de inactivación es la inhibición de la sobreproducción. Cuando la transposasa excede una concentración umbral, la actividad del transposón disminuye. [27] Dado que la transposasa puede formar monómeros inactivos o menos activos que disminuirán la actividad de transposición en general, también se producirá una disminución en la producción de transposasa cuando aumenten las copias grandes de esos elementos menos activos en el genoma del huésped.
Transferencia horizontal
La transferencia horizontal se refiere al movimiento de información de ADN entre células de diferentes organismos. La transferencia horizontal puede implicar el movimiento de TE de un organismo al genoma de otro. La inserción en sí permite que el TE se convierta en un gen activado en el nuevo huésped. Los transposones de ADN utilizan la transferencia horizontal para evitar la inactivación y la pérdida completa del transposón. Esta inactivación se denomina inactivación vertical, lo que significa que el transposón de ADN está inactivo y permanece como un fósil. Este tipo de transferencia no es la más común, pero se ha visto en el caso de la proteína de virulencia del trigo ToxA, que se transfirió entre los diferentes patógenos fúngicos Parastagonospora nodorum , Pyrenophora tritici-repentis y Bipolaris sorokiniana. [28] Otros ejemplos incluyen la transferencia entre crustáceos marinos , insectos de diferentes órdenes y organismos de diferentes filos , como los humanos y los nematodos . [29]
Evolución
Los genomas eucariotas difieren en el contenido de transposones. Recientemente, un estudio de las diferentes superfamilias de transposones revela que existen similitudes sorprendentes entre los grupos. Se ha planteado la hipótesis de que muchos de ellos están representados en dos o más supergrupos eucariotas. Esto significa que la divergencia de las superfamilias de transposones podría incluso ser anterior a la divergencia de los supergrupos eucariotas. [30]
Recombinación V(D)J
La recombinación V(D)J , aunque no es un TE de ADN, es notablemente similar a los transposones . La recombinación V(D)J es el proceso por el cual se crea la gran variación en los sitios de unión de los anticuerpos. En este mecanismo, el ADN se recombina para crear diversidad genética. [31] Debido a esto, se ha planteado la hipótesis de que estas proteínas, particularmente Rag1 y Rag2 [32], se derivan de elementos transponibles. [33]
Extinción en el genoma humano
Hay evidencia que sugiere que al menos 40 familias de transposones de ADN humano estuvieron activas durante la radiación de los mamíferos y el linaje temprano de los primates. Luego, hubo una pausa en la actividad transposicional durante la última parte de la radiación de los primates, con una detención completa del movimiento de transposones en un antepasado primate antropoide. No hay evidencia de ningún elemento transponible más joven que unos 37 millones de años. [34]
Referencias
- ^ "Transposón | genética". Enciclopedia Británica . Consultado el 28 de octubre de 2019 .
- ^ Wicker, Thomas; Sabot, François; Hua-Van, Aurélie; Bennetzen, Jeffrey L.; Capy, Pierre; Chalhoub, Boulos; Flavell, Andrew; Leroy, Philippe; Morgante, Michele (2007). "Un sistema de clasificación unificado para elementos transponibles eucariotas". Nature Reviews Genetics . 8 (12): 973–982. doi :10.1038/nrg2165. PMID 17984973. S2CID 32132898.
- ^ Feschotte, Cédric; Pritham, Ellen J. (diciembre de 2007). "Transposones de ADN y la evolución de los genomas eucariotas". Revista Anual de Genética . 41 (1): 331–368. doi :10.1146/annurev.genet.40.110405.090448. PMC 2167627 . PMID 18076328.
- ^ Muñoz-López, Martín; García-Pérez, José L. (abril de 2010). "Transposones de ADN: naturaleza y aplicaciones en genómica". Genómica actual . 11 (2): 115-128. doi :10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221 . PMID 20885819.
- ^ "Transposones | Aprende ciencia en Scitable" www.nature.com . Consultado el 28 de octubre de 2019 .
- ^ Craig, Nancy L. (13 de octubre de 1995). "Unidad en reacciones de transposición". Science . 270 (5234): 253–4. Bibcode :1995Sci...270..253C. doi :10.1126/science.270.5234.253. ISSN 0036-8075. PMID 7569973. S2CID 29930180.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (17 de julio de 2001). "Transposones de círculo rodante en eucariotas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (15): 8714–8719. Bibcode :2001PNAS...98.8714K. doi : 10.1073/pnas.151269298 . ISSN 0027-8424. PMC 37501 . PMID 11447285.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (21 de marzo de 2006). "Transposones de ADN autosintetizadores en eucariotas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (12): 4540–4545. Bibcode :2006PNAS..103.4540K. doi : 10.1073/pnas.0600833103 . ISSN 0027-8424. PMC 1450207 . PMID 16537396.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (21 de marzo de 2006). "Transposones de ADN autosintetizadores en eucariotas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (12): 4540–4545. Bibcode :2006PNAS..103.4540K. doi : 10.1073/pnas.0600833103 . ISSN 0027-8424. PMC 1450207 . PMID 16537396.
- ^ Berg y Howe, Douglas E. y Martha M. (1989). ADN móvil II . ASM Press. pág. 98. ISBN 9781555812096.
- ^ Madigan M, Martinko J, eds. (2006). Brock Biología de microorganismos (11.ª ed.). Prentice Hall. ISBN 0-13-144329-1.
- ^ "Replicación por círculo rodante - Wiki de la Facultad de Ciencias Biomédicas". teaching.ncl.ac.uk . Consultado el 6 de octubre de 2019 .
- ^ Thomas J, Pritham EJ (agosto de 2015). "Helitrones, los elementos transponibles en círculo rodante eucariotas". Microbiology Spectrum . 3 (4): 893–926. doi : 10.1128/microbiolspec.MDNA3-0049-2014 . ISBN 9781555819200. Número de identificación personal 26350323.
- ^ Kapitonov VV, Jurka J (marzo de 2006). "Transposones de ADN autosintetizadores en eucariotas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (12): 4540–5. Bibcode :2006PNAS..103.4540K. doi : 10.1073/pnas.0600833103 . PMC 1450207 . PMID 16537396.
- ^ Bao W, Kojima KK, Kohany O (2 de junio de 2015). "Actualización de Repbase, una base de datos de elementos repetitivos en genomas eucariotas". ADN móvil . 6 (1): 11. doi : 10.1186/s13100-015-0041-9 . PMC 4455052 . PMID 26045719.
- ^ Feschotte C, Pritham EJ (2007). "Transposones de ADN y la evolución de los genomas eucariotas". Revista Anual de Genética . 41 : 331–68. doi :10.1146/annurev.genet.40.110405.090448. PMC 2167627 . PMID 18076328.
- ^ Morgante M, Brunner S, Pea G, Fengler K, Zuccolo A, Rafalski A (septiembre de 2005). "La duplicación de genes y la redistribución de exones por transposones similares a helitrones generan diversidad intraespecie en el maíz". Nature Genetics . 37 (9): 997–1002. doi :10.1038/ng1615. PMID 16056225. S2CID 10401931.
- ^ McClintock, Barbara (1950). "El origen y comportamiento de los loci mutables en el maíz". Proc Natl Acad Sci USA . 36 (6): 344–55. Bibcode :1950PNAS...36..344M. doi : 10.1073/pnas.36.6.344 . PMC 1063197 . PMID 15430309.
- ^ Jacobson, JW; Medhora, MM; Hartl, DL (noviembre de 1986). "Estructura molecular de un elemento transponible somáticamente inestable en Drosophila". Proc Natl Acad Sci USA . 83 (22): 8684–8. Bibcode :1986PNAS...83.8684J. doi : 10.1073/pnas.83.22.8684 . PMC 386995 . PMID 3022302.
- ^ Lohe, AR; Moriyama, EN; Lidholm, DA; Hartl, DL (1995). "Transmisión horizontal, inactivación vertical y pérdida estocástica de elementos transponibles de tipo mariner". Mol. Biol. Evol . 12 (1): 62–72. doi : 10.1093/oxfordjournals.molbev.a040191 . PMID 7877497.
- ^ Deprá, M; Valente, VL; Margis, R; Loreto, EL (2009). "El transposón hobo y los elementos relacionados con los hobo se expresan como genes de desarrollo en Drosophila". Gene . 448 (1): 57–63. doi : 10.1016/j.gene.2009.08.012 . PMID 19720121.
- ^ Devaud, M; Kayser, FH; Bächi, B (agosto de 1982). "Resistencia a múltiples antibióticos mediada por transposones en cepas de Acinetobacter". Antimicrob Agents Chemother . 22 (2): 323–9. doi :10.1128/aac.22.2.323. PMC 183733 . PMID 6100428.
- ^ "Transposones | Aprende ciencia en Scitable" www.nature.com . Consultado el 1 de noviembre de 2019 .
- ^ "Barbara McClintock y el descubrimiento de los genes saltarines (transposones) | Aprenda ciencias en Scitable" www.nature.com . Consultado el 7 de octubre de 2019 .
- ^ Koga, Akihiko; Iida, Atsuo; Hori, Hiroshi; Shimada, Atsuko; Shima, Akihiro (julio de 2006). "El transposón de ADN de vertebrados como mutador natural: el elemento Tol2 del pez medaka contribuye a la variación genética sin dejar rastros reconocibles". Biología molecular y evolución . 23 (7): 1414–1419. doi : 10.1093/molbev/msl003 . ISSN 0737-4038. PMID 16672286.
- ^ "Transposones | Aprende ciencia en Scitable" www.nature.com . Consultado el 1 de noviembre de 2019 .
- ^ Muñoz-López, Martín; García-Pérez, José L. (abril de 2010). "Transposones de ADN: naturaleza y aplicaciones en genómica". Genómica actual . 11 (2): 115-128. doi :10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221 . PMID 20885819.
- ^ McDonald, Megan C.; Taranto, Adam P.; Hill, Erin; Schwessinger, Benjamin; Liu, Zhaohui; Simpfendorfer, Steven; Milgate, Andrew; Solomon, Peter S. (2019-10-29). "Transferencia horizontal mediada por transposón de la proteína de virulencia específica del huésped ToxA entre tres hongos patógenos del trigo". mBio . 10 (5). doi :10.1128/mBio.01515-19. ISSN 2150-7511. PMC 6737239 . PMID 31506307.
- ^ Muñoz-López, Martín; García-Pérez, José L. (abril de 2010). "Transposones de ADN: naturaleza y aplicaciones en genómica". Genómica actual . 11 (2): 115-128. doi :10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221 . PMID 20885819.
- ^ Feschotte C, Pritham EJ (2007). "Transposones de ADN y la evolución de los genomas eucariotas". Revista Anual de Genética . 41 : 331–68. doi :10.1146/annurev.genet.40.110405.090448. PMC 2167627 . PMID 18076328.
- ^ Jung D, Alt FW (enero de 2004). "Descifrando la recombinación V(D)J; perspectivas sobre la regulación génica". Cell . 116 (2): 299–311. doi : 10.1016/S0092-8674(04)00039-X . PMID 14744439. S2CID 16890458.
- ^ Bourque G, Burns KH, Gehring M, Gorbunova V, Seluanov A, Hammell M, et al. (noviembre de 2018). "Diez cosas que debería saber sobre los elementos transponibles". Genome Biology . 19 (1): 199. doi : 10.1186/s13059-018-1577-z . PMC 6240941 . PMID 30454069.
- ^ Muñoz-López M, García-Pérez JL (abril de 2010). "Transposones de ADN: naturaleza y aplicaciones en genómica". Current Genomics . 11 (2): 115–28. doi :10.2174/138920210790886871. PMC 2874221 . PMID 20885819.
- ^ Pace JK, Feschotte C (abril de 2007). "La historia evolutiva de los transposones de ADN humano: evidencia de una intensa actividad en el linaje de los primates". Genome Research . 17 (4): 422–32. doi :10.1101/gr.5826307. PMC 1832089 . PMID 17339369.
Enlaces externos
- Dfam, una base de datos de secuencias repetidas de ADN
- Repbase, una base de datos y un sistema de clasificación para secuencias repetidas de ADN
- Genes derivados de transposones de ADN, en la base de datos HGNC