Cruce de vacaciones

Una unión de Holliday es una estructura de ácido nucleico ramificada que contiene cuatro brazos bicatenarios unidos. Estos brazos pueden adoptar una de varias conformaciones dependiendo de las concentraciones de sal del tampón y la secuencia de nucleobases más cercana a la unión. La estructura recibe su nombre de Robin Holliday , el biólogo molecular que propuso su existencia en 1964.
En biología, las uniones de Holliday son un intermediario clave en muchos tipos de recombinación genética , así como en la reparación de roturas de doble cadena . Estas uniones suelen tener una secuencia simétrica y, por lo tanto, son móviles, lo que significa que los cuatro brazos individuales pueden deslizarse a través de la unión en un patrón específico que preserva en gran medida el apareamiento de bases . Además, las uniones de cuatro brazos similares a las uniones de Holliday aparecen en algunas moléculas de ARN funcionales .
Los científicos crearon artificialmente uniones de Holliday inmóviles, con secuencias asimétricas que bloquean las hebras en una posición específica, para estudiar su estructura como modelo para las uniones de Holliday naturales. Estas uniones también se utilizaron posteriormente como bloques estructurales básicos en la nanotecnología del ADN , donde múltiples uniones de Holliday se pueden combinar en geometrías diseñadas específicamente que proporcionan a las moléculas un alto grado de rigidez estructural .
Estructura
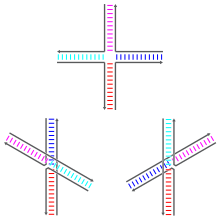
Las uniones de Holliday pueden existir en una variedad de isómeros conformacionales con diferentes patrones de apilamiento coaxial entre los cuatro brazos de doble hélice. El apilamiento coaxial es la tendencia de los extremos romos de los ácidos nucleicos a unirse entre sí, mediante interacciones entre las bases expuestas. Hay tres posibles conformadores: una forma no apilada (o abierta-X) y dos formas apiladas. La forma no apilada domina en ausencia de cationes divalentes como Mg 2+ , debido a la repulsión electrostática entre las cadenas principales cargadas negativamente. En presencia de al menos aproximadamente 0,1 mM Mg 2+ , la repulsión electrostática se contrarresta y predominan las estructuras apiladas. A partir de 2000, no se sabía con certeza si el blindaje electrostático era el resultado de la unión específica del sitio de los cationes a la unión, o la presencia de una colección difusa de los iones en solución. [1]
La forma no apilada es una conformación plana y extendida casi cuadrada. Por otro lado, los conformadores apilados tienen dos dominios continuos de doble hélice separados por un ángulo de aproximadamente 60° en una dirección hacia la derecha . Dos de las cuatro cadenas permanecen aproximadamente helicoidales, permaneciendo dentro de cada uno de los dos dominios de doble hélice, mientras que las otras dos se cruzan entre los dos dominios de manera antiparalela . [1]
Las dos posibles formas apiladas difieren en los pares de brazos que se apilan entre sí; cuál de los dos domina depende en gran medida de las secuencias de bases más cercanas a la unión. Algunas secuencias dan como resultado un equilibrio entre los dos confórmeros, mientras que otras prefieren fuertemente un único confórmero. En particular, las uniones que contienen la secuencia A-CC que une el punto de unión parecen preferir fuertemente el confórmero que permite que se forme un enlace de hidrógeno entre la segunda citosina y uno de los fosfatos en el punto de unión. Si bien la mayoría de los estudios se han centrado en las identidades de las cuatro bases más cercanas a la unión en cada brazo, es evidente que las bases más alejadas también pueden afectar las conformaciones de apilamiento observadas. [1]
En las uniones con secuencias simétricas, el punto de ramificación es móvil y puede migrar en un proceso de recorrido aleatorio . La velocidad de migración de la ramificación varía drásticamente con la concentración de iones, con tiempos de un solo paso que aumentan de 0,3 a 0,4 ms sin iones a 270−300 ms con 10 mM de Mg 2+ . El cambio en la velocidad se correlaciona con la formación de las estructuras apiladas frente a las no apiladas. [1]
Las uniones de Holliday con una mella , o ruptura en una de las hebras, en el punto de unión adoptan una orientación perpendicular y siempre prefieren el confórmero de apilamiento que coloca la mella en una hebra cruzada en lugar de una hebra helicoidal. [1]
Las uniones de Holliday del ARN asumen una conformación apilada antiparalela a altas concentraciones de magnesio, una conformación apilada perpendicular a concentraciones moderadas y rotan a una conformación apilada paralela a bajas concentraciones, mientras que incluso pequeñas concentraciones de iones de calcio favorecen al confórmero antiparalelo. [1]
Función biológica

La unión de Holliday es un intermediario clave en la recombinación homóloga , un proceso biológico que aumenta la diversidad genética al cambiar genes entre dos cromosomas , así como en eventos de recombinación específicos del sitio que involucran integrasas . Además, están involucradas en la reparación de roturas de doble cadena . [1] Además, pueden surgir estructuras cruciformes que involucran uniones de Holliday para aliviar la tensión helicoidal en secuencias simétricas en superenrollamientos de ADN . [2] Si bien las uniones de cuatro brazos también aparecen en moléculas de ARN funcionales , como el ARN espliceosómico U1 y la ribozima en horquilla del virus de la mancha anular del tabaco , estas generalmente contienen nucleótidos no apareados entre los dominios de doble hélice apareados y, por lo tanto, no adoptan estrictamente la estructura de Holliday. [1]
Las uniones de Holliday en la recombinación homóloga se dan entre secuencias idénticas o casi idénticas, lo que da lugar a una disposición simétrica de las secuencias alrededor de la unión central. Esto permite que se produzca un proceso de migración de ramificaciones en el que las hebras se mueven a través del punto de unión. [1] La escisión, o resolución, de la unión de Holliday puede producirse de dos formas. La escisión del conjunto original de hebras da lugar a dos moléculas que pueden mostrar conversión génica pero no entrecruzamiento cromosómico , mientras que la escisión del otro conjunto de dos hebras hace que las moléculas recombinantes resultantes muestren entrecruzamiento. Todos los productos, independientemente de la escisión, son heterodúplex en la región de migración de la unión de Holliday. [3]
Muchas proteínas son capaces de reconocer o distorsionar la estructura de la unión de Holliday. Una de estas clases contiene enzimas que resuelven la unión y la cortan, a veces de una manera específica para la secuencia. Estas proteínas distorsionan la estructura de la unión de varias maneras, a menudo tirando de la unión hacia una conformación no apilada, rompiendo los pares de bases centrales y/o cambiando los ángulos entre los cuatro brazos. Otras clases son las proteínas de migración de ramificación que aumentan la tasa de intercambio en órdenes de magnitud y las recombinasas específicas del sitio . [1] En los procariotas, las resolvasas de la unión de Holliday se dividen en dos familias, integrasas y nucleasas, que son estructuralmente similares aunque sus secuencias no se conservan. [3]
En los eucariotas, dos modelos primarios de cómo la recombinación homóloga repara las roturas de doble cadena en el ADN son la vía de reparación de rotura de doble cadena (DSBR) (a veces llamada el modelo de doble unión de Holliday ) y la vía de recocido de cadena dependiente de síntesis (SDSA). [4] En el caso de la rotura de doble cadena, el extremo 3' se degrada y el extremo 5' más largo invade la cromátida hermana contigua, formando una burbuja de replicación. A medida que esta burbuja se acerca al ADN roto, la cadena antisentido 5' más larga invade nuevamente la cadena sentido de esta porción de ADN, transcribiendo una segunda copia. Cuando termina la replicación, ambas colas se reconectan para formar dos uniones de Holliday, que luego son escindidas en una variedad de patrones por las proteínas. [5] Una animación de este proceso se puede ver aquí. [6]
Las roturas de doble cadena de ADN en bacterias son reparadas por la vía RecBCD de recombinación homóloga. Se cree que las roturas que ocurren en solo una de las dos cadenas de ADN, conocidas como brechas de cadena simple, se reparan mediante la vía RecF . Tanto la vía RecBCD como la RecF incluyen una serie de reacciones conocidas como migración de rama, en la que se intercambian cadenas simples de ADN entre dos moléculas entrecruzadas de ADN dúplex, y resolución , en la que esas dos moléculas entrecruzadas de ADN se separan y se restauran a su estado normal de doble cadena. [7] La recombinación homóloga ocurre en varios grupos de virus. En los virus de ADN como el herpesvirus , la recombinación ocurre a través de un mecanismo de ruptura y unión como en bacterias y eucariotas. [8] En las bacterias, la migración de rama es facilitada por el complejo RuvABC o proteína RecG, motores moleculares que utilizan la energía de la hidrólisis de ATP para mover la unión. La unión debe entonces resolverse en dos dúplex separados, restaurando ya sea la configuración parental o una configuración cruzada. La resolución puede ocurrir de manera horizontal o vertical durante la recombinación homóloga, dando lugar a productos de parche (si están en la misma orientación durante la reparación de la rotura de doble cadena) o productos de empalme (si están en diferentes orientaciones durante la reparación de la rotura de doble cadena). [9] [10] RuvA y RuvB son proteínas de migración de ramificaciones, mientras que RuvC es una enzima de resolución de uniones. [1]
Hay evidencia de recombinación en algunos virus ARN , específicamente virus ssRNA de sentido positivo como retrovirus , picornavirus y coronavirus . Existe controversia sobre si la recombinación homóloga ocurre en virus ssRNA de sentido negativo como la influenza . [11]
Resolución
En la levadura en ciernes Saccharomyces cerevisiae , las uniones de Holliday se pueden resolver mediante cuatro vías diferentes que explican esencialmente toda la resolución de las uniones de Holliday in vivo . [12] La vía que produce la mayoría de los entrecruzamientos en la levadura en ciernes S. cerevisiae , y posiblemente en los mamíferos, involucra a las proteínas EXO1 , el heterodímero MLH1 - MLH3 (llamado MutL gamma) y SGS1 (ortólogo de la helicasa del síndrome de Bloom ). [12] El heterodímero MLH1-MLH3 se une preferentemente a las uniones de Holliday. [13] Es una endonucleasa que realiza roturas de cadena simple en el ADN bicatenario superenrollado. [13] [14] El heterodímero MLH1-MLH3 promueve la formación de recombinantes de entrecruzamiento . [15] Si bien las otras tres vías, que involucran a las proteínas MUS81 -MMS4, SLX1 y YEN1, respectivamente, pueden promover la resolución de la unión de Holliday in vivo, la ausencia de las tres nucleasas tiene solo un impacto modesto en la formación de productos de cruce.
Los mutantes dobles eliminados tanto de MLH3 (vía principal) como de MMS4 (vía secundaria) mostraron una reducción drástica del entrecruzamiento en comparación con el tipo salvaje (de 6 a 17 veces); sin embargo, la viabilidad de las esporas fue razonablemente alta (62%) y la disyunción cromosómica pareció mayoritariamente funcional. [15]
Aunque MUS81 es un componente de una vía de cruce menor en la meiosis de levaduras en gemación, plantas y vertebrados, [16] en el protozoo Tetrahymena thermophila , MUS81 parece ser parte de una vía de cruce esencial, si no la predominante. [16] La vía MUS81 también parece ser la vía de cruce predominante en la levadura de fisión Schizosaccharomyces pombe . [16]
Las proteínas MSH4 y MSH5 forman una estructura hetero-oligomérica (heterodímero) en levaduras y humanos. [17] [18] [19] En la levadura Saccharomyces cerevisiae, MSH4 y MSH5 actúan específicamente para facilitar los entrecruzamientos entre cromosomas homólogos durante la meiosis. [17] El complejo MSH4/MSH5 se une y estabiliza las uniones dobles de Holliday y promueve su resolución en productos de entrecruzamiento. Un mutante hipomórfico MSH4 (parcialmente funcional) de S. cerevisiae mostró una reducción del 30% en todo el genoma en los números de entrecruzamientos y una gran cantidad de meiosis con cromosomas sin intercambio. [20] Sin embargo, este mutante dio lugar a patrones de viabilidad de esporas que sugieren que la segregación de cromosomas sin intercambio ocurrió de manera eficiente. Por lo tanto, en S. cerevisiae, la segregación adecuada aparentemente no depende completamente de entrecruzamientos entre pares homólogos.
Uso en nanotecnología del ADN

La nanotecnología del ADN consiste en el diseño y la fabricación de estructuras artificiales de ácidos nucleicos como materiales de ingeniería para la nanotecnología, en lugar de como portadores de información genética en células vivas. Este campo utiliza estructuras de ADN ramificadas como componentes fundamentales para crear estructuras más complejas y diseñadas racionalmente. Las uniones de Holliday son, por tanto, componentes de muchas de estas estructuras de ADN. Como los complejos de uniones de Holliday aislados son demasiado flexibles para ensamblarse en grandes conjuntos ordenados, se utilizan motivos estructurales con múltiples uniones de Holliday para crear " baldosas " rígidas que luego pueden ensamblarse en "conjuntos" más grandes. [22] [23]

El motivo más común es el complejo de doble cruce (DX), que contiene dos uniones de Holliday muy próximas entre sí, lo que da como resultado una estructura rígida que puede autoensamblarse en matrices más grandes. La estructura de la molécula DX obliga a las uniones de Holliday a adoptar una conformación con los dominios de doble hélice directamente uno al lado del otro, en contraste con su ángulo preferido de aproximadamente 60°. El complejo puede diseñarse para obligar a las uniones a adoptar una orientación paralela o antiparalela, pero en la práctica la variedad antiparalela se comporta mejor y la versión paralela rara vez se utiliza. [22] [23]
El motivo estructural DX es el elemento fundamental del método de origami de ADN , que se utiliza para crear estructuras bidimensionales y tridimensionales más grandes de forma arbitraria. En lugar de utilizar piezas DX individuales, una sola hebra larga de andamiaje se pliega en la forma deseada mediante una serie de hebras cortas. Cuando se ensamblan, la hebra de andamiaje es continua a través de los dominios de doble hélice, mientras que las hebras cortas participan en las uniones de Holliday como hebras cruzadas. [25]
Se han demostrado algunos tipos de mosaicos que conservan el ángulo nativo de 60° de la unión de Holliday. Una de estas matrices utiliza mosaicos que contienen cuatro uniones de Holliday en una disposición de paralelogramo. Esta estructura tenía el beneficio de permitir que el ángulo de unión se visualizara directamente a través de microscopía de fuerza atómica . Se han utilizado mosaicos de tres uniones de Holliday en forma triangular para hacer matrices tridimensionales periódicas para su uso en cristalografía de rayos X de biomoléculas. Estas estructuras reciben su nombre por su similitud con las unidades estructurales basadas en el principio de tensegridad , que utiliza miembros tanto en tensión como en compresión. [22] [23]
Historia
Robin Holliday propuso la estructura de unión que ahora lleva su nombre como parte de su modelo de recombinación homóloga en 1964, basándose en su investigación sobre los organismos Ustilago maydis y Saccharomyces cerevisiae . El modelo proporcionó un mecanismo molecular que explicaba tanto la conversión génica como el entrecruzamiento cromosómico . Holliday se dio cuenta de que la vía propuesta crearía segmentos de ADN heterodúplex con desajustes de bases entre diferentes versiones de un solo gen. Predijo que la célula tendría un mecanismo para la reparación de desajustes, que se descubrió más tarde. [3] Antes del modelo de Holliday, el modelo aceptado implicaba un mecanismo de elección de copia [26] donde la nueva hebra se sintetiza directamente a partir de partes de las diferentes hebras parentales. [27]
En el modelo original de Holliday para la recombinación homóloga, las roturas de una sola hebra se producen en el mismo punto en una hebra de cada ADN parental. Los extremos libres de cada hebra rota migran entonces a la otra hélice de ADN. Allí, las hebras invasoras se unen a los extremos libres que encuentran, lo que da lugar a la unión de Holliday. A medida que cada hebra cruzada se rehibrida con su hebra compañera original, desplaza a la hebra complementaria original que se encuentra delante de ella. Esto hace que la unión de Holliday migre, creando los segmentos heterodúplex. Dependiendo de qué hebra se utilizó como plantilla para reparar la otra, las cuatro células resultantes de la meiosis podrían acabar con tres copias de un alelo y sólo una del otro, en lugar de las dos normales de cada uno, una propiedad conocida como conversión génica. [3]
El modelo original de Holliday suponía que el ADN heterodúplex estaría presente en ambos cromosomas, pero los datos experimentales en levaduras refutaron esta idea. Un modelo actualizado por Matt Meselson y Charley Radding en 1975 introdujo la idea de la migración de ramas. [26] Otras observaciones en la década de 1980 llevaron a la propuesta de mecanismos alternativos para la recombinación, como el modelo de ruptura de doble cadena (por Jack Szostak , Frank Stahl y otros) y el modelo de recocido de cadena simple. Un tercero, el modelo de recocido de cadena dependiente de la síntesis, no involucraba uniones de Holliday. [3]
La primera evidencia experimental de la estructura de la unión de Holliday provino de estudios de microscopía electrónica a fines de la década de 1970, donde la estructura de cuatro brazos era claramente visible en imágenes de ADN de plásmidos y bacteriófagos . Más tarde, en la década de 1980, se identificaron las enzimas responsables de iniciar la formación y la unión a las uniones de Holliday, aunque a partir de 2004, la identificación de las resolvasas de la unión de Holliday de mamíferos seguía siendo esquiva (sin embargo, consulte la sección "Resolución de las uniones de Holliday", más arriba para obtener información más reciente). En 1983, Nadrian Seeman construyó por primera vez moléculas artificiales de la unión de Holliday a partir de oligonucleótidos sintéticos , lo que permitió un estudio más directo de sus propiedades físicas. Gran parte del análisis inicial de la estructura de la unión de Holliday se dedujo de la electroforesis en gel , la FRET y los estudios de huellas de radicales hidroxilo y nucleasas . En la década de 1990, se dispuso de métodos de cristalografía y RMN de ácidos nucleicos , así como de herramientas de modelado molecular computacional . [1] [3] [28]
Inicialmente, los genetistas asumieron que la unión adoptaría una conformación paralela en lugar de antiparalela , porque eso colocaría a los dúplex homólogos en una alineación más cercana entre sí. [1] El análisis químico en la década de 1980 mostró que la unión en realidad prefería la conformación antiparalela, un hallazgo que se consideró controvertido, y el propio Robin Holliday inicialmente dudó de los hallazgos. [1] [3] La estructura antiparalela más tarde fue ampliamente aceptada debido a los datos de cristalografía de rayos X en moléculas in vitro , aunque a partir de 2004 las implicaciones para la estructura in vivo seguían sin estar claras, especialmente la estructura de las uniones a menudo se altera por las proteínas unidas a ella. [3]
La base conceptual para la nanotecnología del ADN fue establecida por primera vez por Nadrian Seeman a principios de la década de 1980. [29] En ese momento se conocían varias estructuras de ADN ramificadas naturales, incluida la horquilla de replicación del ADN y la unión móvil de Holliday, pero la idea de Seeman fue que las uniones de ácidos nucleicos inmóviles se podían crear diseñando adecuadamente las secuencias de hebras para eliminar la simetría en la molécula ensamblada, y que estas uniones inmóviles en principio se podían combinar en redes cristalinas rígidas. El primer artículo teórico que proponía este esquema se publicó en 1982, y la primera demostración experimental de una unión de ADN inmóvil se publicó el año siguiente. [23] [30] Seeman desarrolló el motivo de doble cruce (DX) más rígido , adecuado para formar redes bidimensionales, demostrado en 1998 por él y Erik Winfree . [22] En 2006, Paul Rothemund demostró por primera vez la técnica del origami de ADN para crear de manera fácil y robusta estructuras de ADN plegadas de forma arbitraria. Este método permitió la creación de estructuras mucho más grandes que las que eran posibles anteriormente y que son menos exigentes técnicamente para diseñar y sintetizar. [31] La síntesis de una red tridimensional fue finalmente publicada por Seeman en 2009, casi treinta años después de que se había propuesto lograrla. [32]
Referencias
- ^ abcdefghijklmn Lilley, David MJ (2000). "Estructuras de uniones helicoidales en ácidos nucleicos". Quarterly Reviews of Biophysics . 33 (2): 109–159. doi :10.1017/S0033583500003590. PMID 11131562. S2CID 40501795.
- ^ Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Jr., Ignacio (2000). Ácidos nucleicos: estructuras, propiedades y funciones . Sausalito, California: University Science Books. pág. 468. ISBN 0935702490.
- ^ abcdefgh Liu Y, West S (2004). "Felices fiestas: 40 aniversario del cruce de Holliday". Nature Reviews Molecular Cell Biology . 5 (11): 937–44. doi :10.1038/nrm1502. PMID 15520813. S2CID 24520723.
- ^ Sung, P; Klein, H (octubre de 2006). "Mecanismo de recombinación homóloga: los mediadores y las helicasas asumen funciones reguladoras". Nature Reviews Molecular Cell Biology . 7 (10): 739–750. doi :10.1038/nrm2008. PMID 16926856. S2CID 30324005.
- ^ Hartel, Daniel L.; Jones, Elizabeth W. (2009). "Capítulo 6: Biología molecular de la replicación y recombinación del ADN". Genética: análisis de la genética y los genomas . Burlington: Jones & Bartlett. ISBN 9780763758684.
- ^ Helleday, T. (20 de noviembre de 2018). "Reparación de rotura de doble cadena mediante uniones de doble Holliday (modelo de Szostak)". Animación . MIT.
- ^ Rocha, EPC; Cornet, E; Michel, B (agosto de 2005). "Análisis comparativo y evolutivo de los sistemas de recombinación homóloga bacteriana". PLOS Genetics . 1 (2): e15. doi : 10.1371/journal.pgen.0010015 . PMC 1193525 . PMID 16132081.

- ^ Fleischmann Jr, WR (1996). "Capítulo 43". Microbiología médica (4.ª ed.). Rama médica de la Universidad de Texas en Galveston. ISBN 0-9631172-1-1.
- ^ West SC (2003). "Visiones moleculares de las proteínas de recombinación y su control". Nature Reviews Molecular Cell Biology . 4 (6): 435–45. doi :10.1038/nrm1127. PMID 12778123. S2CID 28474965.
- ^ Kowalczykowski SC (2000). "Inicio de la recombinación genética y replicación dependiente de la recombinación". Tendencias en ciencias bioquímicas . 25 (4): 156–65. doi :10.1016/S0968-0004(00)01569-3. PMID 10754547.
- ^ Boni, MF; de Jong, MD; van Doorn, HR; Holmes, EC; Martin, Darren P. (3 de mayo de 2010). Martin, Darren P. (ed.). "Directrices para la identificación de eventos de recombinación homóloga en el virus de la influenza A". PLOS ONE . 5 (5): e10434. Bibcode :2010PLoSO...510434B. doi : 10.1371/journal.pone.0010434 . PMC 2862710 . PMID 20454662.

- ^ ab Zakharyevich, K; Tang, S; Ma, Y; Hunter, N (abril de 2012). "La delineación de las vías de resolución de moléculas conjuntas en la meiosis identifica una resolvasa específica de cruce". Cell . 149 (2): 334–47. doi :10.1016/j.cell.2012.03.023. PMC 3377385 . PMID 22500800.
- ^ ab Ranjha, L; Anand, R; Cejka, P (2014). "El heterodímero Mlh1-Mlh3 de Saccharomyces cerevisiae es una endonucleasa que se une preferentemente a las uniones de Holliday". J. Biol. Chem . 289 (9): 5674–86. doi : 10.1074/jbc.M113.533810 . PMC 3937642. PMID 24443562 .
- ^ Rogacheva MV, Manhart CM, Chen C, Guarne A, Surtees J, Alani E (2014). "Mlh1-Mlh3, un factor de reparación de errores de emparejamiento y cruce meiótico de ADN, es una endonucleasa estimulada por Msh2-Msh3". J. Biol. Chem . 289 (9): 5664–73. doi : 10.1074/jbc.M113.534644 . PMC 3937641. PMID 24403070 .
- ^ ab Sonntag Brown M, Lim E, Chen C, Nishant KT, Alani E (2013). "El análisis genético de las mutaciones mlh3 revela interacciones entre los factores que promueven el entrecruzamiento durante la meiosis en la levadura de panadería". G3: Genes, Genomas, Genética . 3 (1): 9–22. doi :10.1534/g3.112.004622. PMC 3538346 . PMID 23316435.
- ^ abc Lukaszewicz A, Howard-Till RA, Loidl J (2013). "La nucleasa Mus81 y la helicasa Sgs1 son esenciales para la recombinación meiótica en un protista que carece de un complejo sinaptonémico". Nucleic Acids Res . 41 (20): 9296–309. doi :10.1093/nar/gkt703. PMC 3814389 . PMID 23935123.
- ^ ab Pochart P, Woltering D, Hollingsworth NM (1997). "Propiedades conservadas entre homólogos de MutS funcionalmente distintos en levadura". J. Biol. Chem . 272 (48): 30345–9. doi : 10.1074/jbc.272.48.30345 . PMID 9374523.
- ^ Winand NJ, Panzer JA, Kolodner RD (1998). "Clonación y caracterización de los homólogos humanos y de Caenorhabditis elegans del gen MSH5 de Saccharomyces cerevisiae". Genómica . 53 (1): 69–80. doi : 10.1006/geno.1998.5447 . PMID 9787078.
- ^ Bocker T, Barusevicius A, Snowden T, Rasio D, Guerrette S, Robbins D, Schmidt C, Burczak J, Croce CM, Copeland T, Kovatich AJ, Fishel R (1999). "hMSH5: un homólogo de MutS humano que forma un nuevo heterodímero con hMSH4 y se expresa durante la espermatogénesis". Cancer Res . 59 (4): 816–22. PMID 10029069.
- ^ Krishnaprasad GN, Anand MT, Lin G, Tekkedil MM, Steinmetz LM, Nishant KT (2015). "La variación en las frecuencias de cruce perturba la seguridad del cruce sin afectar la segregación cromosómica meiótica en Saccharomyces cerevisiae". Genética . 199 (2): 399–412. doi :10.1534/genetics.114.172320. PMC 4317650 . PMID 25467183.
- ^ Mao, Chengde (diciembre de 2004). "El surgimiento de la complejidad: lecciones del ADN". PLOS Biology . 2 (12): 2036–2038. doi : 10.1371/journal.pbio.0020431 . PMC 535573 . PMID 15597116.
- ^ abcd Seeman, Nadrian C. (junio de 2004). "Nanotecnología y la doble hélice". Scientific American . 290 (6): 64–75. Bibcode :2004SciAm.290f..64S. doi :10.1038/scientificamerican0604-64. PMID 15195395.
- ^ abcd Seeman, Nadrian C. (2010). "Nanomateriales basados en ADN". Revista Anual de Bioquímica . 79 : 65–87. doi :10.1146/annurev-biochem-060308-102244. PMC 3454582 . PMID 20222824.
- ^ Pan, Keyao; Kim, Do-Nyun; Zhang, Fei; Adendorff, Mateo R.; Yan, Hao; Báñese, Mark (3 de diciembre de 2014). "Predicción sin celosía de la estructura tridimensional de conjuntos de ADN programados". Comunicaciones de la naturaleza . 5 : 5578. Código Bib : 2014NatCo...5.5578P. doi : 10.1038/ncomms6578. PMC 4268701 . PMID 25470497.
- ^ Saccà, Barbara; Niemeyer, Christof M. (2012). "DNA Origami: The Art of Folding DNA" (PDF) . Angewandte Chemie International Edition . 51 (1): 58–66. doi :10.1002/anie.201105846. PMID 22162047. S2CID 8014597 . Consultado el 25 de febrero de 2015 .
- ^ ab Stahl FW (1 de octubre de 1994). "La unión de Holliday en su trigésimo aniversario" ( PDF ) . Genética . 138 (2): 241–246. doi :10.1093/genetics/138.2.241. PMC 1206142 . PMID 7828807.
- ^ Avances en genética. Academic Press. 1971. ISBN 9780080568027.
- ^ Hays FA, Watson J, Ho PS (2003). "¡Precaución! Cruce de ADN: estructuras cristalinas de las uniones de Holliday". J Biol Chem . 278 (50): 49663–49666. doi : 10.1074/jbc.R300033200 . PMID: 14563836.
- ^ Pelesko, John A. (2007). Autoensamblaje: la ciencia de las cosas que se ensamblan por sí mismas . Nueva York: Chapman & Hall/CRC. pp. 201, 242, 259. ISBN 978-1-58488-687-7.
- ^ Pinheiro, AV; Han, D.; Shih, WM; Yan, H. (diciembre de 2011). "Desafíos y oportunidades para la nanotecnología estructural del ADN". Nature Nanotechnology . 6 (12): 763–772. Bibcode :2011NatNa...6..763P. doi :10.1038/nnano.2011.187. PMC 3334823 . PMID 22056726.
- ^ Rothemund, Paul WK (2006). "Origami de ADN con andamiaje: desde multicruces generalizados hasta redes poligonales". En Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz (eds.). Nanotecnología: ciencia y computación . Natural Computing Series. Nueva York: Springer. pp. 3–21. doi :10.1007/3-540-30296-4_1. ISBN . 978-3-540-30295-7.
- ^ Service, Robert F. (3 de junio de 2011). "La nanotecnología del ADN crece". Science . 332 (6034): 1140–1143. Bibcode :2011Sci...332.1140S. doi :10.1126/science.332.6034.1140. PMID 21636754.
Enlaces externos
- Holliday+junctions en los encabezamientos de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Cambio conformacional de la unión de Holliday
- Análisis de las actividades de migración de ramificaciones de proteínas utilizando sustratos de ADN sintéticos (un protocolo)
