Picornavirus
| Picornaviridae | |
|---|---|
 | |
| Micrografía electrónica del virus de la poliomielitis | |
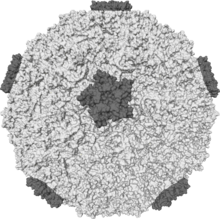 | |
| Isosuperficie de un rinovirus humano que muestra picos de proteína | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Pisuviricota |
| Clase: | Pisoniviricetes |
| Orden: | Picornaviral |
| Familia: | Picornaviridae |
| Géneros [1] | |
Ver texto | |
Los picornavirus son un grupo de virus ARN sin envoltura relacionados que infectan a vertebrados , incluidos peces , [2] mamíferos y aves . Son virus que representan una gran familia de virus ARN monocatenarios pequeños de sentido positivo con una cápside icosaédrica de 30 nm . Los virus de esta familia pueden causar una variedad de enfermedades, incluido el resfriado común , la poliomielitis , la meningitis , la hepatitis y la parálisis . [3] [4] [5] [6]
Los picornavirus constituyen la familia Picornaviridae , orden Picornavirales y reino Riboviria . Hay 158 especies en esta familia, asignadas a 68 géneros. Ejemplos notables son los géneros Enterovirus (incluidos Rhinovirus y Poliovirus ), Aphthovirus , Cardiovirus y Hepatovirus . [1] [7]
Etimología
El nombre "picornavirus" tiene una doble etimología . En primer lugar, el nombre deriva de picorna- , que es un acrónimo de "poliovirus, insensibilidad al éter, coxsackievirus, virus huérfano, rinovirus y ácido ribonucleico " . En segundo lugar , el nombre deriva de pico- , que designa una unidad de medida muy pequeña (equivalente a 10 −12 ), combinada con rna para describir este grupo de virus ARN muy pequeños . [8]
Historia
El primer virus animal descubierto (1897) fue el virus de la fiebre aftosa (FMDV). Es el miembro prototípico del género Aphthovirus en la familia Picornaviridae . [5] El ensayo de placa se desarrolló utilizando poliovirus ; el descubrimiento de la replicación viral en cultivo también fue con poliovirus en 1949. Esta fue la primera vez que se produjo un virus infeccioso en células cultivadas. [9] La síntesis de poliproteínas , los sitios de entrada internos de los ribosomas y el ARNm sin capuchón se descubrieron mediante el estudio de células infectadas por poliovirus, y un clon de poliovirus fue el primer clon de ADN infeccioso hecho de un virus ARN en animales. Junto con el rinovirus , el poliovirus fue el primer virus animal en tener su estructura determinada por cristalografía de rayos X. La ARN polimerasa dependiente de ARN se descubrió en Mengovirus , un género de picornavirus. [10]
Virología
Estructura

Los picornavirus no tienen envoltura y poseen una cápside icosaédrica . [4] La cápside es una disposición de 60 protómeros en una estructura icosaédrica muy compacta. Cada protómero consta de cuatro polipéptidos conocidos como VP (proteína viral) 1, 2, 3 y 4. Los polipéptidos VP2 y VP4 se originan a partir de un protómero conocido como VP0 que se escinde para dar los diferentes componentes de la cápside. Se dice que la cápside icosaédrica tiene un número de triangulación de 3, esto significa que en la estructura icosaédrica cada uno de los 60 triángulos que forman la cápside se divide en tres pequeños triángulos con una subunidad en la esquina. [ cita requerida ]
Muchos picornavirus tienen una hendidura profunda formada alrededor de cada uno de los 12 vértices de los icosaedros. La superficie exterior de la cápside está compuesta por regiones de VP1, VP2 y VP3. Alrededor de cada uno de los vértices hay un cañón revestido con los extremos C de VP1 y VP3. La superficie interior de la cápside está compuesta por VP4 y los extremos N de VP1. J. Esposito y Frederick A. Murphy demuestran la estructura de hendidura conocida como cañones, utilizando cristalografía de rayos X y microscopía crioelectrónica. [9]
Dependiendo del tipo y grado de deshidratación, la partícula viral tiene un diámetro de alrededor de 30 a 32 nm. [7] El genoma viral tiene una longitud de alrededor de 2500 nm, por lo que está empaquetado firmemente dentro de la cápside junto con sustancias como iones de sodio para equilibrar las cargas negativas en el ARN causadas por los grupos fosfato . [ cita requerida ]
Genoma

Los picornavirus se clasifican en el sistema de clasificación viral de Baltimore como virus del grupo IV, ya que contienen un genoma de ARN monocatenario de sentido positivo . Su genoma tiene una longitud de entre 6,7 y 10,1 kilobases . [7] Como la mayoría de los genomas de ARN de sentido positivo, el material genético por sí solo es infeccioso; aunque sustancialmente menos virulento que si estuviera contenido dentro de la partícula viral, el ARN puede tener una mayor infectividad cuando se transfecta en células. El genoma de ARN es inusual porque tiene una proteína en el extremo 5' que se usa como cebador para la transcripción por la ARN polimerasa . Este cebador se llama genoma VPg y tiene un tamaño de entre 2 y 3 kb. VPg contiene un residuo de tirosina en el extremo 3'. La tirosina como fuente de –OH para el extremo 5' del ARN está ligada covalentemente. [9] [11]
El genoma no está segmentado y es de sentido positivo (el mismo sentido que el ARNm de los mamíferos, que se lee de 5' a 3'). A diferencia del ARNm de los mamíferos , los picornavirus no tienen una tapa 5' , sino una proteína codificada por el virus conocida como VPg . Sin embargo, al igual que el ARNm de los mamíferos, el genoma tiene una cola de poli(A) en el extremo 3'. En ambos extremos del genoma del picornavirus se encuentra una región no traducida (UTR). La UTR 5' suele ser más larga, con una longitud de alrededor de 500 a 1200 nucleótidos (nt), en comparación con la UTR 3', que tiene alrededor de 30 a 650 nt. Se cree que la UTR 5' es importante en la traducción, y la 3' en la síntesis de cadena negativa; sin embargo, el extremo 5' también puede tener un papel que desempeñar en la virulencia del virus. El resto del genoma codifica proteínas estructurales en el extremo 5' y proteínas no estructurales en el extremo 3' en una única poliproteína. [ cita requerida ]
La poliproteína está organizada de la siguiente manera: L-1ABCD-2ABC-3ABCD, donde cada letra representa una proteína, pero existen variaciones en esta disposición. [ cita requerida ]
Las proteínas 1A, 1B, 1C y 1D son las proteínas de la cápside VP4, VP2, VP3 y VP1, respectivamente. Las proteasas codificadas por el virus realizan las escisiones, algunas de las cuales son intramoleculares. La poliproteína se corta primero para producir P1, P2 y P3. P1 se miristila en el extremo N antes de escindirse en VP0, VP3 y VP1, las proteínas que formarán las procápsides; VP0 se escindirá más tarde para producir VP2 y VP4. Otros productos de escisión incluyen 3B (VPg), 2C (una ATPasa) y 3D (la ARN polimerasa). [9] [12]
Replicación
Elementos del ARN

Los ARN genómicos de los picornavirus poseen múltiples elementos de ARN, y son necesarios para la síntesis de ARN de cadena negativa y positiva. El elemento de replicación que actúa en cis (CRE) es necesario para la replicación. La estructura de tallo-bucle que contiene el CRE es independiente de la posición, pero cambia con la ubicación entre los tipos de virus cuando se ha identificado. Además, los elementos del extremo 3' del ARN viral son importantes y eficientes para la replicación de ARN de los picornavirus. El extremo 3' del picornavirus contiene un tracto poli(A), que es necesario para la infectividad. Sin embargo, se plantea la hipótesis de que la síntesis de ARN ocurre en esta región. El NCR del extremo 3' del poliovirus no es necesario para la síntesis de cadena negativa, pero es un elemento importante para la síntesis de cadena positiva. Además, el NCR del extremo 5' que contiene elementos estructurales secundarios es necesario para la replicación de ARN y el inicio de la traducción del poliovirus. Los sitios de entrada internos al ribosoma son estructuras de ARN que permiten el inicio de la traducción independiente de la tapa y pueden iniciar la traducción en medio de un ARN mensajero. [13]
Ciclo vital

La partícula viral se une a los receptores de la superficie celular. Los receptores de la superficie celular se caracterizan para cada serotipo de picornavirus. Por ejemplo, el receptor del poliovirus es la glicoproteína CD155, que es un receptor especial para los seres humanos y algunas otras especies de primates. Por esta razón, el poliovirus no podía producirse en muchos laboratorios hasta que se desarrollaron ratones transgénicos con un receptor CD155 en sus superficies celulares en la década de 1990. Estos animales pueden ser infectados y utilizados para estudios de replicación y patogénesis. [9] La unión provoca un cambio conformacional en las proteínas de la cápside viral y se libera ácido mirístico . El ácido forma un poro en la membrana celular a través del cual se inyecta el ARN. [14]
Una vez dentro de la célula, el ARN se desenvuelve y el genoma de ARN de la cadena (+) se replica a través de un intermediario de ARN de doble cadena que se forma utilizando la ARN polimerasa dependiente del ARN viral. La traducción por los ribosomas de la célula huésped no se inicia por una tapa 5' G como es habitual, sino que se inicia por un sitio de entrada interno al ribosoma. El ciclo de vida viral es muy rápido, y todo el proceso de replicación se completa en promedio en 8 horas. Sin embargo, tan solo 30 minutos después de la infección inicial, la síntesis de proteínas celulares disminuye a casi cero: esencialmente, se detiene la síntesis macromolecular de proteínas celulares. Durante las siguientes 1-2 horas, se produce una pérdida de marginación de la cromatina y homogeneidad en el núcleo, antes de que las proteínas virales comiencen a sintetizarse y aparezca una vacuola en el citoplasma cerca del núcleo que comienza a extenderse gradualmente a medida que el tiempo después de la infección llega a aproximadamente 3 horas. Después de este tiempo, la membrana plasmática celular se vuelve permeable; Entre las 4 y las 6 horas, las partículas virales se reúnen y, a veces, se las puede ver en el citoplasma. Alrededor de las 8 horas, la célula está efectivamente muerta y se lisa para liberar las partículas virales. [ cita requerida ]
Los datos experimentales obtenidos a partir de experimentos de crecimiento en un solo paso han permitido observar la replicación de los picornavirus con gran detalle. Toda la replicación ocurre dentro del citoplasma de la célula huésped y la infección puede ocurrir incluso en células que no contienen núcleo (enucleadas) y en aquellas tratadas con actinomicina D (este antibiótico inhibiría la replicación viral si esta ocurriera en el núcleo). [ cita requerida ]
La traducción se lleva a cabo mediante el desplazamiento del marco de lectura del ribosoma -1, la iniciación viral y la omisión del ribosoma. El virus sale de la célula huésped mediante lisis y viroporinas. Los vertebrados son los huéspedes naturales. Las vías de transmisión son fecal-oral, contacto, ingestión y partículas transportadas por el aire. [4]
Proteína viral (VPg)
Los picornavirus tienen una proteína viral ( VPg ) unida covalentemente al extremo 5' de sus genomas en lugar de una tapa de 7-metilguanosina como los ARNm celulares. Las ARN polimerasas del virus utilizan VPg como cebador. VPg como cebador utiliza síntesis de ARN de cadena positiva y negativa. La replicación de los picornavirus se inicia mediante la uridilación de VPg. Se uridila en el grupo hidroxilo de un residuo de tirosina. [3] Un mecanismo de cebador VPg es utilizado por los picornavirus (entero- afto- y otros), grupos de virus adicionales (poty-, como-, calici- y otros) y supergrupos de virus de ARN similares a picornavirus (coronavirus, notavirus, etc.). El mecanismo ha sido mejor estudiado para los enterovirus (que incluyen muchos patógenos humanos, como el poliovirus y los virus coxsackie ), así como para el aftovirus, un patógeno animal que causa la fiebre aftosa. [ cita requerida ]
En este grupo, la síntesis de ARN dependiente de cebadores utiliza una pequeña proteína viral de 22 a 25 aminoácidos de longitud unida al VPg [15] para iniciar la actividad de la polimerasa, donde el cebador se une covalentemente al extremo 5' de la plantilla de ARN. [16] La uridilación ocurre en un residuo de tirosina en la tercera posición del VPg. Un CRE, que es una estructura de bucle de tallo de ARN, sirve como plantilla para la uridilación de VPg, lo que resulta en la síntesis de VPgpUpUOH. Las mutaciones dentro de la estructura CRE-ARN previenen la uridilación de VPg, y las mutaciones dentro de la secuencia de VPg pueden disminuir gravemente la actividad catalítica de RdRp. [17] Si bien el hidroxilo de tirosina de VPg puede iniciar la síntesis de ARN de cadena negativa de una manera independiente de CRE y VPgpUpUOH, la síntesis de VPgpUpUOH dependiente de CRE es absolutamente necesaria para la síntesis de ARN de cadena positiva. La uridilación de VPg dependiente de CRE reduce la K m¬ de UTP necesaria para la replicación del ARN viral y la síntesis de VPgpUpUOH dependiente de CRE, y es necesaria para una síntesis eficiente de ARN de cadena negativa, especialmente cuando las concentraciones de UTP son limitantes. [18] El cebador VPgpUpUOH se transfiere al extremo 3' de la plantilla de ARN para la elongación, que puede continuar mediante la adición de bases de nucleótidos por RdRp. Las estructuras cristalinas parciales para VPgs del virus de la fiebre aftosa [19] y el virus coxsackie B3 [20] sugieren que puede haber dos sitios en la polimerasa viral para los VPgs pequeños de los picornavirus. Las estructuras de la solución de RMN de VPg del poliovirus [21] y VPgpU [22] muestran que la uridilación estabiliza la estructura del VPg, que de otro modo es bastante flexible en solución. El segundo sitio puede usarse para la uridilación, después de lo cual el VPgpU puede iniciar la síntesis de ARN. Los iniciadores VPg de los calicivirus, cuyas estructuras apenas están comenzando a revelarse, [23] son mucho más grandes que los de los picornavirus. Los mecanismos de uridilación y iniciación pueden ser bastante diferentes en todos estos grupos. [ cita requerida ]
La uridilación de VPg puede incluir el uso de proteínas precursoras, lo que permite la determinación de un posible mecanismo para la ubicación del precursor diuridilado que contiene VPg en el extremo 3' del ARN de cadena positiva o negativa para la producción de ARN de longitud completa. Los determinantes de la eficiencia de la uridilación de VPg sugieren la formación y/o colapso o liberación del producto uridilado como el paso limitante de la velocidad in vitro dependiendo del donante de VPg empleado. [24] Las proteínas precursoras también tienen un efecto sobre la especificidad y estabilidad de VPg-CRE. [25] El bucle del tallo superior del ARN, al que se une VPg, tiene un impacto significativo tanto en la retención como en el reclutamiento de VPg y Pol. El bucle del tallo de CRE se desenrollará parcialmente, lo que permitirá que los componentes precursores se unan y recluten VPg y Pol4. El bucle de CRE tiene una secuencia de consenso definida a la que se unen los componentes de iniciación, pero no existe una secuencia de consenso para el tallo de soporte, lo que sugiere que solo es importante la estabilidad estructural del CRE. [26]
Ensamblaje y organización del complejo ribonucleoproteico VPg del picornavirus
- Dos moléculas 3CD (complejo VPg) se unen a CRE con los dominios 3C (dominio VPg) en contacto con el tallo superior y los dominios 3D (dominio VPg) en contacto con el tallo inferior.
- El dímero 3C abre el tallo del ARN formando una interacción más estable con las cadenas individuales que forman el tallo.
- La 3Dpol se recluta y se retiene en este complejo mediante una interacción física entre el subdominio posterior del pulgar de 3Dpol y una superficie de uno o ambos subdominios 3C de 3CD.
La VPg también puede desempeñar un papel importante en el reconocimiento específico del genoma viral por parte de las proteínas de movimiento (MP), que son proteínas no estructurales codificadas por muchos, si no todos, los virus de plantas para permitir su movimiento de una célula infectada a células vecinas. [27] La MP y la VPg interactúan para proporcionar especificidad para el transporte de ARN viral de una célula a otra. Para satisfacer los requisitos de energía, la MP también interactúa con P10, que es una ATPasa celular. [ cita requerida ]
Enfermedades
Los picornavirus causan una variedad de enfermedades. Los enterovirus de la familia de los picornavirus infectan el tracto entérico , lo que se refleja en su nombre. Los rinovirus infectan principalmente la nariz y la garganta . Los enterovirus se replican a 37 °C, mientras que los rinovirus crecen mejor a 33 °C, ya que esta es la temperatura más baja de la nariz. Los enterovirus son estables en condiciones ácidas, por lo que pueden sobrevivir a la exposición al ácido gástrico . Por el contrario, los rinovirus son lábiles al ácido (se inactivan o destruyen en condiciones de pH bajo ), por lo que las infecciones por rinovirus se limitan a la nariz y la garganta. [ cita requerida ]
Taxonomía
Se reconocen estos géneros: [1]
- Virus Aali
- Virus Ailuri
- Ampivirus
- Anativirus
- Aftovirus
- Virus aquama
- Hepatovirus avi
- Avisivirus
- Virus Boosepi
- virus de la bopiosis
- Cecilivirus
- Cardiovirus
- Cosavirus
- Crahelivirus
- Crohivirus
- Virus del danipi
- Dicipivirus
- Diresapivirus
- Enterovirus
- Erbovirus
- Felipivirus
- Fipivirus
- Gallivirus
- Virus Gruheli
- Grusopivirus
- virus harka
- Hemipivirus
- Hepatovirus
- Virus huni
- Virus Kobu
- Virus Kunsagi
- Limnipivirus
- Livupivirus
- Ludopivirus
- Malgachevirus
- Marsupivirus
- Megrivirus
- Virus Mischi
- Virus de mosa
- Mupivirus
- Mirropivirus
- Orivirus
- Oscivirus
- Parabovirus
- Parechovirus
- Pasivirus
- Paserivirus
- Virus pemapi
- Poecivirus
- Virus Potamipi
- Pygoscepivirus
- Rabovirus
- Virus Rafi
- Virus rajidapi
- Rohelivirus
- Virus rosa
- Virus Sakobu
- Salivirus
- Sapelovirus
- virus seneca
- Virus Shanba
- Virus sicininiano
- Virus simapi
- Teschovirus
- Virus de la antorcha
- Tottorivirus
- Tremovirus
- Tropivirus
Véase también
Referencias
- ^ abc «Taxonomía de virus: publicación de 2020». Comité Internacional de Taxonomía de Virus (ICTV). Marzo de 2021. Consultado el 20 de mayo de 2021 .
- ^ Altan E, Kubiski SV, Boros Á, Reuter G, Sadeghi M, Deng X, Creighton EK, Crim MJ, Delwart E (2019). "Un picornavirus altamente divergente que infecta el epitelio intestinal del pez cebra (Danio rerio) en instituciones de investigación de todo el mundo". Pez cebra . 16 (3): 291–299. doi : 10.1089/zeb.2018.1710 . PMID 30939077. S2CID 92999901.
- ^ ab Ryu WS (2016). "Capítulo 11 – Picornavirus". Virología molecular de virus patógenos humanos . Corea: Academic Press. págs. 153–64. doi :10.1016/b978-0-12-800838-6.00011-4. ISBN 978-0-12-800838-6.
- ^ abc "Viral Zone". ExPASy . Consultado el 15 de junio de 2015 .
- ^ ab Martínez-Salas E, Saiz M, Sobrino F (2008). "Virus de la fiebre aftosa". En Mettenleiter TC, Sobrino F (eds.). Virus animales: biología molecular. Norfolk, Reino Unido: Caister Academic Press. ISBN 978-1-904455-22-6.
- ^ Lau SK, Woo PC, Lai KK, Huang Y, Yip CC, Shek CT, Lee P, Lam CS, Chan KH, Yuen KY (septiembre de 2011). "Análisis completo del genoma de tres nuevos picornavirus de diversas especies de murciélagos". Revista de Virología . 85 (17): 8819–28. doi :10.1128/JVI.02364-10. PMC 3165794 . PMID 21697464.
- ^ abc «Picornaviridae - Picornaviridae - Picornavirales». Comité Internacional de Taxonomía de Virus (ICTV) . Archivado desde el original el 21 de septiembre de 2017. Consultado el 12 de junio de 2020 .
- ^ "Picornaviridae". Comité Internacional de Taxonomía de Virus (ICTV) . Octubre de 2017. Archivado desde el original el 21 de septiembre de 2017. Consultado el 5 de febrero de 2019 .
- ^ abcde Carter JB, Saunders VA (2007). "Picornavirus (y otros virus de ARN de cadena positiva)". Virología: principios y aplicaciones . Chichester, Inglaterra: John Wiley & Sons. págs. 160–65. ISBN 978-0-470-02386-0.
- ^ Knipe DM, Howley P (2013). Virología de Fields. Lippincott Williams & Wilkins. ISBN 978-1-4698-3066-7.
- ^ Zabel P, Moerman M, Lomonossoff G, Shanks M, Beyreuther K (julio de 1984). "Virus del mosaico del caupí VPg: la secuenciación de la proteína modificada radioquímicamente permite el mapeo del gen en el ARN B". The EMBO Journal . 3 (7): 1629–34. doi :10.1002/j.1460-2075.1984.tb02021.x. PMC 557569 . PMID 16453534.
- ^ Acheson NH (2011). Fundamentos de virología molecular (2.ª ed.). John Wiley & Sons, Inc. ISBN 978-0470900598.
- ^ Daijogo S, Semler BL (2011). "Intersecciones mecanicistas entre la traducción de picornavirus y la replicación de ARN". Avances en la investigación de virus . 80 : 1–24. doi :10.1016/B978-0-12-385987-7.00001-4. ISBN . 9780123859877. Número de identificación personal 21762819.
- ^ "Patología, Microbiología e Inmunología - Facultad de Medicina de Columbia | Universidad de Carolina del Sur".
- ^ Flanegan JB, Baltimore D (septiembre de 1977). "Poliovirus-specific primer-dependent RNA polymerase able to copy poly(A)" (Poliovirus-specific cebador-dependiente RNA polimerasa capaz de copiar poly(A)). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 74 (9): 3677–80. Bibcode :1977PNAS...74.3677F. doi : 10.1073/pnas.74.9.3677 . PMC 431685 . PMID 198796.
- ^ Ambros V, Baltimore D (agosto de 1978). "La proteína está unida al extremo 5' del ARN del poliovirus mediante un enlace fosfodiéster a la tirosina". The Journal of Biological Chemistry . 253 (15): 5263–66. doi : 10.1016/S0021-9258(17)30361-7 . PMID 209034.
- ^ Gu C, Zeng T, Li Y, Xu Z, Mo Z, Zheng C (octubre de 2009). "Análisis de la estructura y la función de complejos de ARN polimerasa dependientes de ARN mutantes con VPg". Bioquímica. Biokhimiia . 74 (10): 1132–41. doi :10.1134/S0006297909100095. PMID 19916926. S2CID 24968119.
- ^ Steil BP, Barton DJ (octubre de 2008). "La uridilación de VPg dependiente del elemento de replicación que actúa en cis del poliovirus reduce la Km del nucleósido trifosfato iniciador de la replicación del ARN viral". Journal of Virology . 82 (19): 9400–08. doi :10.1128/JVI.00427-08. PMC 2546976 . PMID 18653453.
- ^ Ferrer-Orta C, Arias A, Agudo R, Pérez-Luque R, Escarmís C, Domingo E, Verdaguer N (febrero de 2006). "La estructura de un complejo proteína cebador-polimerasa en el inicio de la replicación del genoma". La Revista EMBO . 25 (4): 880–88. doi :10.1038/sj.emboj.7600971. PMC 1383552 . PMID 16456546.
- ^ Gruez A, Selisko B, Roberts M, Bricogne G, Bussetta C, Jabafi I, et al. (octubre de 2008). "La estructura cristalina de la ARN polimerasa dependiente de ARN del virus Coxsackie B3 en complejo con su cebador proteico VPg confirma la existencia de un segundo sitio de unión de VPg en las polimerasas de Picornaviridae". Journal of Virology . 82 (19): 9577–90. doi :10.1128/JVI.00631-08. PMC 2546979 . PMID 18632861.
- ^ Schein CH, Oezguen N, Volk DE, Garimella R, Paul A, Braun W (julio de 2006). "Estructura de RMN del péptido viral ligado al genoma (VPg) del poliovirus". Péptidos . 27 (7): 1676–84. doi :10.1016/j.peptides.2006.01.018. PMC 1629084 . PMID 16540201.
- ^ Schein CH, Oezguen N, van der Heden van Noort GJ, Filippov DV, Paul A, Kumar E, Braun W (agosto de 2010). "Estructura de la solución de RMN del péptido uridilado del poliovirus vinculado al genoma (VPgpU)". Péptidos . 31 (8): 1441–48. doi :10.1016/j.peptides.2010.04.021. PMC 2905501 . PMID 20441784.
- ^ Leen EN, Kwok KY, Birtley JR, Simpson PJ, Subba-Reddy CV, Chaudhry Y, et al. (mayo de 2013). "Estructuras de los dominios del núcleo helicoidal compacto de las proteínas VPg del calicivirus felino y del norovirus murino" (PDF) . Revista de Virología . 87 (10): 5318–30. doi :10.1128/JVI.03151-12. PMC 3648151 . PMID 23487472.
- ^ Pathak HB, Oh HS, Goodfellow IG, Arnold JJ, Cameron CE (noviembre de 2008). "Replicación del genoma de picornavirus: funciones de las proteínas precursoras y pasos limitantes de la velocidad en la uridilación de VPg dependiente de oriI". The Journal of Biological Chemistry . 283 (45): 30677–88. doi : 10.1074/jbc.M806101200 . PMC 2576561 . PMID 18779320.
- ^ Shen M, Wang Q, Yang Y, Pathak HB, Arnold JJ, Castro C, Lemon SM, Cameron CE (noviembre de 2007). "Los mutantes con ganancia de función del rinovirus humano tipo 14 para la utilización de oriI definen residuos de 3C(D) y 3Dpol que contribuyen al ensamblaje y la estabilidad del complejo de uridilación de VPg del picornavirus". J. Virol . 81 (22): 12485–95. doi :10.1128/JVI.00972-07. PMC 2169002 . PMID 17855535.
- ^ Yang Y, Rijnbrand R, McKnight KL, Wimmer E, Paul A, Martin A, Lemon SM (agosto de 2002). "Requisitos de secuencia para la replicación del ARN viral y la uridilación de VPg dirigida por el elemento de replicación interno que actúa en cis (CRE) del rinovirus humano tipo 14". Journal of Virology . 76 (15): 7485–94. doi :10.1128/JVI.76.15.7485-7494.2002. PMC 136355 . PMID 12097561.
- ^ Roy Chowdhury S, Savithri HS (enero de 2011). Pfeffer S (ed.). "Interacción de la proteína de movimiento del virus del mosaico de Sesbania con VPg y P10: implicación en la especificidad del reconocimiento del genoma". PLOS ONE . 6 (1): e15609. Bibcode :2011PLoSO...615609R. doi : 10.1371/journal.pone.0015609 . PMC 3016346 . PMID 21246040.
Lectura adicional
- Kahn CM, Line S, eds. (2005). "Encefalomielitis enteroviral porcina". Manual veterinario Merck (novena edición). Merck . ISBN 978-0-911910-50-6.
- Thompson JR, Dasgupta I, Fuchs M, Iwanami T, Karasev AV, Petrzik K, Sanfaçon H, Tzanetakis I, van der Vlugt R, Wetzel T, Yoshikawa N, et al. (Consorcio Informe ICTV) (abril de 2017). "Perfil de taxonomía de virus ICTV: Secoviridae". La Revista de Virología General . 98 (4): 529–531. doi :10.1099/jgv.0.000779. PMC 5657025 . PMID 28452295.
- Thompson JR (2020). "Secovirus (Secoviridae)". Módulo de referencia en ciencias de la vida . págs. 692–702. doi :10.1016/B978-0-12-809633-8.21253-3. ISBN 978-0-12-809633-8.S2CID241194454 .
Enlaces externos
- Büchen-Osmond C, ed. (2006). "ICTVdB – The Universal Virus Database, version 4". Gestión de ICTVdB, Escuela de Salud Pública Mailman . Nueva York: Universidad de Columbia. Archivado desde el original el 10 de marzo de 2010.
- "Picornaviridae". Encabezamientos de materias médicas (MeSH) . Biblioteca Nacional de Medicina . Consultado el 3 de septiembre de 2007 .
- "Picornavirus". The Pirbright Institute . Consultado el 3 de septiembre de 2007 .
- Informe en línea de ICTV sobre Picornaviridae
- Listas maestras de especies del Comité Internacional de Taxonomía de Virus del ICTV Archivado el 5 de marzo de 2016 en Wayback Machine
- Comité Internacional de Taxonomía de Virus del ICTV (sitio oficial) Archivado el 3 de febrero de 2017 en Wayback Machine
- Picornavirus: descripción, replicación, enfermedades
- Picornavirus en el navegador de taxonomía del NCBI
- Clasificación de Picornaviridae por el Comité Internacional de Taxonomía de Virus
- Virus animales
- Zona viral: Picornaviridae
- Base de datos y recurso de análisis de virus y patógenos (ViPR): Picornaviridae
- Televisión ICTV
