Retrovirus
| Retrovirus | |
|---|---|
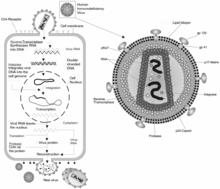 | |
| Esquema de infección celular, producción del virus y estructura del virus del retrovirus VIH | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Pararnavirus |
| Filo: | Arteverviricota |
| Clase: | Revtraviricetes |
| Orden: | Virus ortopédicos |
| Familia: | Retrovirus |
| Subfamilias y géneros [1] | |
| |
Un retrovirus es un tipo de virus que inserta una copia de ADN de su genoma de ARN en el ADN de una célula huésped que invade, modificando así el genoma de esa célula. [2] Después de invadir el citoplasma de una célula huésped , el virus utiliza su propia enzima transcriptasa inversa para producir ADN a partir de su genoma de ARN, al revés del patrón habitual, es decir, retro (hacia atrás). El nuevo ADN se incorpora entonces al genoma de la célula huésped mediante una enzima integrasa , momento en el que el ADN retroviral se denomina provirus . La célula huésped trata entonces el ADN viral como parte de su propio genoma, transcribiendo y traduciendo los genes virales junto con los genes de la propia célula, produciendo las proteínas necesarias para ensamblar nuevas copias del virus. Muchos retrovirus causan enfermedades graves en humanos, otros mamíferos y aves. [3]
Los retrovirus tienen muchas subfamilias en tres grupos básicos.
- Los oncoretrovirus (retrovirus que causan cáncer) incluyen el virus linfotrópico T humano (HTLV), que causa un tipo de leucemia en humanos, y los virus de leucemia murina (MLV) en ratones. [4]
- Los lentivirus (virus lentos) incluyen el VIH-1 y el VIH-2 , causantes del síndrome de inmunodeficiencia adquirida ( SIDA ) en humanos.
- Los espumavirus (virus espumosos) son benignos y no están relacionados con ninguna enfermedad en humanos o animales. [5]
Las enzimas especializadas de infiltración de ADN de los retrovirus los convierten en valiosas herramientas de investigación en biología molecular y se han utilizado con éxito en sistemas de administración de genes. [6]
La evidencia de los retrovirus endógenos (ADN de provirus heredado en genomas animales) sugiere que los retrovirus han estado infectando a los vertebrados durante al menos 450 millones de años. [7]
Estructura
Los viriones , virus en forma de partículas independientes de los retrovirus, consisten en partículas envueltas de unos 100 nm de diámetro. La envoltura lipídica externa está formada por glicoproteína. [8] Los viriones también contienen dos moléculas de ARN monocatenario idénticas de 7 a 10 kilobases de longitud. Las dos moléculas están presentes como un dímero, formado por el apareamiento de bases entre secuencias complementarias. Los sitios de interacción entre las dos moléculas de ARN se han identificado como un " asa de tallo besándose ". [3] Aunque los viriones de diferentes retrovirus no tienen la misma morfología o biología, todos los componentes del virión son muy similares. [9]
Los principales componentes del virión son:
- Envoltura : compuesta de lípidos (obtenidos de la membrana plasmática del huésped durante el proceso de gemación ) así como de glucoproteína codificada por el gen env . La envoltura retroviral cumple tres funciones distintas: protección del entorno extracelular a través de la bicapa lipídica , lo que permite al retrovirus entrar y salir de las células huésped a través del tráfico de membrana endosómica y la capacidad de entrar directamente en las células fusionándose con sus membranas.
- ARN : consiste en un ARN dímero . Tiene una tapa en el extremo 5' y una cola de poli(A) en el extremo 3'. El ARN genómico (gRNA) se produce como resultado de la actividad de la ARN polimerasa II (Pol II) del huésped y al agregar una tapa de metilo 5' y una cola de poli-A 3' se procesa como un ARNm del huésped. [10] El genoma del ARN también tiene regiones terminales no codificantes, que son importantes en la replicación, y regiones internas que codifican proteínas del virión para la expresión génica . El extremo 5' incluye cuatro regiones, que son R, U5, PBS y L. La región R es una secuencia corta repetida en cada extremo del genoma que se usa durante la transcripción inversa para asegurar una transferencia correcta de extremo a extremo en la cadena en crecimiento. U5, por otro lado, es una secuencia corta única entre R y PBS. PBS (sitio de unión del cebador) consta de 18 bases complementarias al extremo 3' del cebador de ARNt. La región L es una región líder no traducida que da la señal para el empaquetamiento del ARN del genoma. El extremo 3' incluye tres regiones, que son PPT (tracto de polipurina), U3 y R. El PPT es un cebador para la síntesis de ADN de cadena positiva durante la transcripción inversa . U3 es una secuencia entre PPT y R, que sirve como señal que el provirus puede usar en la transcripción . R es la secuencia terminal repetida en el extremo 3'.
- Proteínas : consisten en proteínas gag, proteasa (PR), proteínas pol y proteínas env.
- Las proteínas de antígeno específico de grupo (gag) son componentes principales de la cápside viral , de las cuales hay alrededor de 2000 a 4000 copias por virión. Gag posee dos dominios de unión a ácidos nucleicos, que incluyen la matriz (MA) y la nucleocápside (NC). Reconocer, unir y empaquetar específicamente el ARN genómico retroviral en viriones ensamblables es una de las funciones importantes de la proteína Gag. Las interacciones de Gag con los ARN celulares también regulan aspectos del ensamblaje. [11] La expresión de gag por sí sola da lugar al ensamblaje de partículas inmaduras similares a virus que brotan de la membrana plasmática. En todos los retrovirus, la proteína Gag es el precursor de la proteína estructural interna. [12]
- La proteasa (pro) se expresa de forma diferente en distintos virus. Su función es la de realizar cortes proteolíticos durante la maduración del virión para producir proteínas gag y pol maduras. Las proteínas Gag retrovirales son responsables de coordinar muchos aspectos del ensamblaje del virión.
- Las proteínas Pol son responsables de la síntesis del ADN viral y de su integración en el ADN del huésped después de la infección.
- Las proteínas Env desempeñan un papel en la asociación y la entrada de viriones en la célula huésped. [13] La posesión de una copia funcional de un gen env es lo que hace que los retrovirus se distingan de los retroelementos . [14] La capacidad del retrovirus de unirse a su célula huésped objetivo utilizando receptores específicos de la superficie celular está dada por el componente de superficie (SU) de la proteína Env, mientras que la capacidad del retrovirus de entrar en la célula a través de la fusión de membranas está impartida por el componente transmembrana anclado a la membrana (TM). Por lo tanto, es la proteína Env la que permite que el retrovirus sea infeccioso.
- Varias especies de proteínas están asociadas con el ARN en el virión del retrovirus. La proteína nucleocápside (NC) es la proteína más abundante, que recubre el ARN; mientras que otras proteínas, presentes en cantidades mucho menores y tienen actividades enzimáticas. Algunas actividades enzimáticas que están presentes en el virión del retrovirus incluyen la ADN polimerasa dependiente de ARN (transcriptasa inversa; RT), la ADN polimerasa dependiente de ADN, la Ribonucleasa H (RNasa H) Integrasa y la Proteasa. [15] Se ha demostrado que las ARNasas retrovirales H codificadas por todos los retrovirus, incluido el VIH, muestran tres modos diferentes de escisión: interna, dirigida al extremo 3' del ADN y dirigida al extremo 5' del ARN. Los tres modos de escisión constituyen papeles en la transcripción inversa. Por lo tanto, la actividad de la ARNasa H es esencial en varios aspectos de la transcripción inversa. El uso de la actividad de la ARNasa H durante la replicación retroviral muestra una estrategia única para copiar un genoma de ARN monocatenario en un ADN bicatenario, ya que el ADN de cadena negativa es complementario y realiza apareamiento de bases con el genoma del retrovirus en el primer ciclo de síntesis de ADN. [16] La actividad de la ribonucleasa ARNasa H también es necesaria en el ciclo de vida retroviral, ya que genera y elimina cebadores esenciales para la transcriptasa inversa (RT) para el inicio de la síntesis de ADN. Los retrovirus que carecen de la actividad de la ARNasa H no son infecciosos. [17]

Estructura genómica
El genoma retroviral está empaquetado en forma de partículas virales, que son dímeros de moléculas de ARN lineales de cadena sencilla y sentido positivo. [10]
Los retrovirus (y los ortervirus en general) siguen una disposición de 5'– gag – pro – pol – env –3' en el genoma del ARN. gag y pol codifican poliproteínas, cada una de las cuales gestiona la cápside y la replicación. La región pol codifica enzimas necesarias para la replicación viral, como la transcriptasa inversa, la proteasa y la integrasa. [19] Dependiendo del virus, los genes pueden superponerse o fusionarse en cadenas de poliproteínas más grandes. Algunos virus contienen genes adicionales. El género lentivirus, el género spumavirus, el género HTLV/virus de la leucemia bovina (BLV) y un género de virus de peces recientemente introducido son retrovirus clasificados como complejos. Estos virus tienen genes llamados genes accesorios, además de los genes gag, pro, pol y env. Los genes accesorios se encuentran entre pol y env, aguas abajo del env, incluida la región U3 de LTR, o en el env y las porciones superpuestas. Si bien los genes accesorios tienen funciones auxiliares, también coordinan y regulan la expresión génica viral. Además, algunos retrovirus pueden portar genes llamados oncogenes o genes onc de otra clase. Los retrovirus con estos genes (también llamados virus transformantes) son conocidos por su capacidad de causar rápidamente tumores en animales y transformar células en cultivo en un estado oncogénico. [20]
Las poliproteínas se dividen en proteínas más pequeñas, cada una con su propia función. Los nucleótidos que las codifican se conocen como subgenes . [18]
Multiplicación

Cuando los retrovirus han integrado su propio genoma en la línea germinal , su genoma se transmite a una generación siguiente. Estos retrovirus endógenos (ERV), en contraste con los exógenos , ahora constituyen el 5-8% del genoma humano. [21] La mayoría de las inserciones no tienen una función conocida y a menudo se las denomina " ADN basura ". Sin embargo, muchos retrovirus endógenos desempeñan papeles importantes en la biología del huésped, como el control de la transcripción genética, la fusión celular durante el desarrollo placentario en el curso de la germinación de un embrión y la resistencia a la infección retroviral exógena. Los retrovirus endógenos también han recibido especial atención en la investigación de patologías relacionadas con la inmunología , como las enfermedades autoinmunes como la esclerosis múltiple , aunque aún no se ha demostrado que los retrovirus endógenos desempeñen ningún papel causal en esta clase de enfermedad. [22]
Aunque clásicamente se pensaba que la transcripción ocurría solo del ADN al ARN, la transcriptasa inversa transcribe el ARN en ADN. El término "retro" en retrovirus se refiere a esta inversión (convertir ADN a ARN) de la dirección habitual de la transcripción. Todavía obedece al dogma central de la biología molecular , que establece que la información puede transferirse de ácido nucleico a ácido nucleico pero no puede transferirse de proteína a proteína o ácido nucleico. La actividad de la transcriptasa inversa fuera de los retrovirus se ha encontrado en casi todos los eucariotas , lo que permite la generación e inserción de nuevas copias de retrotransposones en el genoma del huésped. Estas inserciones son transcritas por enzimas del huésped en nuevas moléculas de ARN que ingresan al citosol. A continuación, algunas de estas moléculas de ARN se traducen en proteínas virales. Las proteínas codificadas por los genes gag y pol se traducen de ARNm de longitud de genoma en poliproteínas Gag y Gag-Pol. Por ejemplo, para el gen gag ; se traduce en moléculas de la proteína de la cápside, y para el gen pol ; La transcripción inversa permite la traducción de la proteína Gag a moléculas de la transcriptasa inversa. Los retrovirus necesitan muchas más proteínas Gag que proteínas Pol y han desarrollado sistemas avanzados para sintetizar la cantidad necesaria de cada una. Por ejemplo, después de la síntesis de Gag, casi el 95 por ciento de los ribosomas terminan la traducción, mientras que otros ribosomas continúan la traducción para sintetizar Gag-Pol. En el retículo endoplasmático rugoso comienza la glicosilación y el gen env se traduce a partir de ARNm empalmados en el retículo endoplasmático rugoso, en moléculas de la proteína de la envoltura. Cuando las moléculas de la proteína de la envoltura son transportadas al complejo de Golgi, una proteasa del huésped las divide en glicoproteína de superficie y glicoproteína transmembrana. Estos dos productos de glicoproteína permanecen en estrecha afiliación y son transportados a la membrana plasmática después de una glicosilación adicional. [3]
Es importante señalar que un retrovirus debe "traer" su propia transcriptasa inversa en su cápside , de lo contrario no puede utilizar las enzimas de la célula infectada para llevar a cabo la tarea, debido a la naturaleza inusual de producir ADN a partir de ARN. [ cita requerida ]
Los fármacos industriales diseñados como inhibidores de la proteasa y la transcriptasa inversa se fabrican de manera que se dirijan a sitios y secuencias específicos dentro de sus respectivas enzimas. Sin embargo, estos fármacos pueden volverse rápidamente ineficaces debido al hecho de que las secuencias genéticas que codifican la proteasa y la transcriptasa inversa mutan rápidamente. Estos cambios en las bases hacen que cambien codones y sitios específicos con las enzimas y, por lo tanto, evitan la focalización del fármaco al perder los sitios a los que el fármaco realmente se dirige. [ cita requerida ]
Como la transcripción inversa carece de la corrección habitual de la replicación del ADN, un retrovirus muta muy a menudo. Esto le permite desarrollar rápidamente resistencia a los fármacos antivirales e impide el desarrollo de vacunas e inhibidores eficaces para el retrovirus. [23]
Una dificultad a la que se enfrentan algunos retrovirus, como el retrovirus de Moloney, es que las células deben estar dividiendo activamente para que se produzca la transducción. Como resultado, células como las neuronas son muy resistentes a la infección y la transducción por retrovirus. Esto da lugar a la preocupación de que la mutagénesis insercional debida a la integración en el genoma del huésped pueda provocar cáncer o leucemia. Esto es diferente a lo que ocurre con los lentivirus , un género de Retroviridae , que son capaces de integrar su ARN en el genoma de células huésped que no se dividen. [ cita requerida ]
Recombinación
En cada partícula de retrovirus se encuentran empaquetados dos genomas de ARN , pero, después de una infección, cada virus genera solo un provirus . [24] Después de la infección, se produce la transcripción inversa y este proceso va acompañado de recombinación . La recombinación implica el cambio de la cadena molde entre las dos copias del genoma (recombinación por elección de copia) durante la transcripción inversa. En cada ciclo de replicación se producen de 5 a 14 eventos de recombinación por genoma. [25] La recombinación genética parece ser necesaria para mantener la integridad del genoma y como mecanismo de reparación para salvar genomas dañados. [24]
Transmisión
- De célula a célula [26]
- Fluidos
- Se transmite por el aire, como el retrovirus de la oveja Jaagsiekte .
Provirus
El ADN formado después de la transcripción inversa (el provirus) es más largo que el genoma del ARN porque cada uno de los terminales tiene las secuencias U3 - R - U5 llamadas repetición terminal larga (LTR). Así, el terminal 5' tiene la secuencia U3 adicional, mientras que el otro terminal tiene la secuencia U5. [15] Las LTR son capaces de enviar señales para que se lleven a cabo tareas vitales como el inicio de la producción de ARN o la gestión de la tasa de transcripción. De esta manera, las LTR pueden controlar la replicación, por lo tanto, todo el progreso del ciclo viral. [27] Aunque se encuentra en el núcleo, el ADNc retroviral no integrado es un sustrato muy débil para la transcripción. Por esta razón, un provirus integrado es necesario para la expresión permanente y efectiva de los genes retrovirales. [10]
Este ADN puede incorporarse al genoma del huésped como un provirus que puede transmitirse a las células de la progenie. El ADN del retrovirus se inserta al azar en el genoma del huésped. Debido a esto, puede insertarse en los oncogenes . De esta manera, algunos retrovirus pueden convertir células normales en células cancerosas. Algunos provirus permanecen latentes en la célula durante un largo período de tiempo antes de que se activen por el cambio en el entorno celular. [ cita requerida ]
Evolución temprana
Los estudios de retrovirus condujeron a la primera síntesis demostrada de ADN a partir de plantillas de ARN, un modo fundamental para transferir material genético que ocurre tanto en eucariotas como en procariotas . Se ha especulado que los procesos de transcripción de ARN a ADN utilizados por los retrovirus pueden haber causado que el ADN se usara como material genético. En este modelo, la hipótesis del mundo del ARN , los organismos celulares adoptaron el ADN más estable químicamente cuando los retrovirus evolucionaron para crear ADN a partir de las plantillas de ARN . [ cita requerida ]
Una estimación de la fecha de evolución de los retrovirus endógenos de tipo espumoso sitúa el tiempo del ancestro común más reciente en hace > 450 millones de años . [28]
Terapia génica
Se han desarrollado vectores gammaretrovirales y lentivirales para terapia génica que median la modificación genética estable de las células tratadas mediante la integración cromosómica de los genomas de los vectores transferidos. Esta tecnología es útil, no solo para fines de investigación, sino también para la terapia génica clínica destinada a la corrección a largo plazo de defectos genéticos, por ejemplo, en células madre y progenitoras. Se han diseñado partículas de vectores retrovirales con tropismo para varias células diana. Hasta ahora, los vectores gammaretrovirales y lentivirales se han utilizado en más de 300 ensayos clínicos, abordando opciones de tratamiento para varias enfermedades. [6] [29] Se pueden desarrollar mutaciones retrovirales para crear modelos de ratones transgénicos para estudiar varios cánceres y sus modelos metastásicos . [ cita requerida ]
Cáncer
Los retrovirus que causan el crecimiento de tumores incluyen el virus del sarcoma de Rous y el virus del tumor mamario del ratón . El cáncer puede ser desencadenado por protooncogenes que se incorporaron por error al ADN proviral o por la alteración de los protooncogenes celulares. El virus del sarcoma de Rous contiene el gen src que desencadena la formación de tumores. Más tarde se descubrió que un gen similar en las células está involucrado en la señalización celular, que probablemente fue extirpado con el ADN proviral. Los virus no transformantes pueden insertar aleatoriamente su ADN en protooncogenes, alterando la expresión de proteínas que regulan el ciclo celular. El promotor del ADN del provirus también puede causar una sobreexpresión de genes reguladores. Los retrovirus pueden causar enfermedades como el cáncer y la inmunodeficiencia. Si el ADN viral se integra en los cromosomas del huésped, puede provocar infecciones permanentes. Por lo tanto, es importante descubrir la respuesta del cuerpo a los retrovirus. Los retrovirus exógenos están especialmente asociados con enfermedades patógenas. Por ejemplo, los ratones tienen el virus del tumor mamario del ratón (MMTV), que es un retrovirus. Este virus pasa a los ratones recién nacidos a través de la leche mamaria. Cuando tienen 6 meses de edad, los ratones portadores del virus contraen cáncer mamario a causa del retrovirus. Además, el virus de la leucemia I (HTLV-1), que se encuentra en las células T humanas, se ha encontrado en humanos durante muchos años. Se estima que este retrovirus causa leucemia en las edades de 40 y 50 años. [30] Tiene una estructura replicable que puede inducir cáncer. Además de la secuencia genética habitual de los retrovirus, el HTLV-1 contiene una cuarta región, PX. Esta región codifica las proteínas reguladoras Tax, Rex, p12, p13 y p30. La proteína Tax inicia el proceso leucémico y organiza la transcripción de todos los genes virales en el ADN proviral HTLV integrado. [31]
Clasificación

Exógeno
Es posible que sea necesario limpiar esta sección para cumplir con los estándares de calidad de Wikipedia . El problema específico es: No es el taxón "Retroviridae" del que habla el artículo; sugiero que se traslade a la clasificación de Baltimore#Virus de transcripción inversa o Revtraviricetes . ( febrero de 2021 ) |
Los retrovirus exógenos son virus infecciosos que contienen ARN o ADN y que se transmiten de un organismo a otro. En el sistema de clasificación de Baltimore , que agrupa a los virus en función de su forma de síntesis de ARN mensajero , se clasifican en dos grupos: Grupo VI: virus de ARN monocatenario con un intermediario de ADN en su ciclo de vida, y Grupo VII: virus de ADN bicatenario con un intermediario de ARN en su ciclo de vida. [ cita requerida ]
Virus del grupo VI
Todos los miembros del Grupo VI utilizan la transcriptasa inversa codificada por el virus , una ADN polimerasa dependiente del ARN, para producir ADN a partir del genoma de ARN del virión inicial. Este ADN suele integrarse en el genoma del huésped, como en el caso de los retrovirus y pseudovirus , donde es replicado y transcrito por el huésped.
El grupo VI incluye:
- Ordenar Ortervirales
- Familia Belpaoviridae
- Familia Metaviridae
- Familia Pseudoviridae
- Familia Retroviridae – Retrovirus, p. ej., VIH
- Familia Caulimoviridae : una familia de virus del grupo VII (ver más abajo)
La familia Retroviridae se dividía anteriormente en tres subfamilias ( Oncovirinae , Lentivirinae y Spumavirinae ), pero ahora se divide en dos: Orthoretrovirinae y Spumaretrovirinae . El término oncovirus se utiliza ahora comúnmente para describir un virus que causa cáncer. Esta familia ahora incluye los siguientes géneros:
- Subfamilia Orthoretrovirinae :
- Género Alpharetrovirus ; incluye el virus de la leucosis aviar y el virus del sarcoma de Rous
- Género Betaretrovirus ; incluye el virus del tumor mamario del ratón
- Género Gammaretrovirus ; incluye el virus de la leucemia murina y el virus de la leucemia felina
- Género Deltaretrovirus ; incluye el virus de la leucemia bovina y el virus linfotrópico T humano causante de cáncer
- Género Epsilonretrovirus
- Género Lentivirus ; incluye el virus de inmunodeficiencia humana 1 y los virus de inmunodeficiencia simia y felina
- Subfamilia Spumaretrovirinae :
- Género Bovispumavirus
- Género Equispumavirus
- Género Felispumavirus
- Género Prosimiispumavirus
- Género Simiispumavirus
Cabe señalar que, según ICTV 2017, el género Spumavirus se ha dividido en cinco géneros, y su antigua especie tipo, el virus espumoso de los simios , ahora se ha actualizado al género Simiispumavirus con no menos de 14 especies, incluida la nueva especie tipo, el virus espumoso de los simios del chimpancé oriental . [32]
Virus del grupo VII
Ambas familias del grupo VII tienen genomas de ADN contenidos en las partículas del virus invasor. El genoma de ADN se transcribe en ARNm, para su uso como transcripción en la síntesis de proteínas, y ARN pregenómico, para su uso como plantilla durante la replicación del genoma. La transcriptasa inversa codificada por el virus utiliza el ARN pregenómico como plantilla para la creación de ADN genómico.
El grupo VII incluye:
- Familia Caulimoviridae — p. ej. Virus del mosaico de la coliflor
- Familia Hepadnaviridae — p. ej. el virus de la hepatitis B
La última familia está estrechamente relacionada con la recién propuesta
- Familia Nackednaviridae — por ejemplo, el nackednavirus de los cíclidos africanos (ACNDV), anteriormente llamado virus de la hepatitis B de los cíclidos africanos (ACHBV). [33]
mientras que las familias Belpaoviridae , Metaviridae , Pseudoviridae , Retroviridae y Caulimoviridae constituyen el orden Ortervirales . [34]
Endógeno
Los retrovirus endógenos no están incluidos formalmente en este sistema de clasificación y se clasifican en tres clases, según su relación con los géneros exógenos:
- La clase I es la más similar a los gammaretrovirus.
- La clase II son los más similares a los betaretrovirus y alfaretrovirus.
- La clase III es la más similar a los espumavirus.
Controversia
Los retrovirus han sido el foco de varias afirmaciones y afirmaciones recientes que han sido ampliamente desacreditadas por la comunidad científica. Un estudio inicial en 2009 pareció hacer nuevos hallazgos que podrían cambiar algunos de los conocimientos establecidos sobre este tema. Sin embargo, aunque investigaciones posteriores refutaron algunas de las afirmaciones realizadas sobre los retrovirus, hay varias figuras controvertidas que continúan haciendo afirmaciones que en general se considera que no tienen ninguna base válida o consenso que las respalde. [35] [36] [37]
Tratamiento
Los fármacos antirretrovirales son medicamentos para el tratamiento de la infección por retrovirus, principalmente el VIH . Diferentes clases de fármacos antirretrovirales actúan en diferentes etapas del ciclo de vida del VIH . La combinación de varios fármacos antirretrovirales (normalmente tres o cuatro) se conoce como terapia antirretroviral de gran actividad (TARGA). [38]
Tratamiento de los retrovirus veterinarios
Las infecciones por el virus de la leucemia felina y el virus de la inmunodeficiencia felina se tratan con productos biológicos , incluido el único inmunomodulador actualmente autorizado para la venta en los Estados Unidos, el modulador inmunológico de células T de linfocitos (LTCI). [39]
Referencias
- ^ "Taxonomía de virus: versión 2018b". Comité Internacional de Taxonomía de Virus (ICTV) . Marzo de 2019. Consultado el 16 de marzo de 2019 .
- ^ "retrovirus". Diccionario Oxford de inglés . Archivado desde el original el 26 de septiembre de 2018. Consultado el 25 de septiembre de 2018 .
- ^ abc Carter JB, Saunders VA (2007). Virología: principios y aplicaciones (1.ª ed.). Chichester, Inglaterra: John Wiley & Sons. pág. 191. ISBN 978-0-470-02386-0.OCLC 124160564 .
- ^ Coffin JM, Hughes SH, Varmus HE, eds. (1997). Retrovirus . Laboratorio Cold Spring Harbor. ISBN 978-0-87969-571-2 .
- ^ {Miller, AD (2006). Vectores retrovirales en terapia génica. Enciclopedia de ciencias de la vida. doi:10.1038/npg.els.0005741}
- ^ ab Kurth R, Bannert N, eds. (2010). Retrovirus: biología molecular, genómica y patogénesis. Horizon Scientific. ISBN 978-1-904455-55-4.
- ^ Zheng, Jialu; Wei, Yutong; Han, Guan-Zhu (1 de febrero de 2022). "La diversidad y evolución de los retrovirus: perspectivas a partir de los "fósiles" virales". Virologica Sinica . 37 (1): 11–18. doi :10.1016/j.virs.2022.01.019. ISSN 1995-820X. PMC 8922424 . PMID 35234634.
- ^ Coffin, John M.; Hughes, Stephen H.; Varmus, Harold E. (1997). El lugar de los retrovirus en la biología. Cold Spring Harbor Laboratory Press.
- ^ Coffin JM (1992). "Estructura y clasificación de los retrovirus". En Levy JA (ed.). The Retroviridae . Vol. 1 (1.ª ed.). Nueva York: Plenum. p. 20. ISBN 978-0-306-44074-8.
- ^ abc Painter, Mark M.; Collins, Kathleen L. (1 de enero de 2019), "VIH y retrovirus", en Schmidt, Thomas M. (ed.), Enciclopedia de microbiología (cuarta edición) , Academic Press, págs. 613–628, doi :10.1016/b978-0-12-801238-3.66202-5, ISBN 978-0-12-811737-8, S2CID 188750910 , consultado el 3 de mayo de 2020
- ^ Olson ED, Musier-Forsyth K (febrero de 2019). "Interacciones proteína Gag retroviral-ARN: implicaciones para el empaquetamiento de ARN genómico específico y el ensamblaje de viriones". Seminarios en biología celular y del desarrollo . SI: Células dendríticas humanas. 86 : 129–139. doi :10.1016/j.semcdb.2018.03.015. PMC 6167211. PMID 29580971 .
- ^ Coffin JM, Hughes SH, Varmus HE (1997). Proteínas viriones. Prensa de laboratorio Cold Spring Harbor. ISBN 978-0-87969-571-2.
- ^ Ataúd 1992, págs. 26-34
- ^ Kim FJ, Battini JL, Manel N, Sitbon M (enero de 2004). "Aparición de retrovirus vertebrados y captura de envoltura". Virology . 318 (1): 183–91. doi : 10.1016/j.virol.2003.09.026 . PMID 14972546.
- ^ ab Carter JB, Saunders VA (2007). Virología: principios y aplicaciones . Chichester, Inglaterra: John Wiley & Sons. ISBN 978-0-470-02386-0.OCLC 124160564 .
- ^ Champoux JJ, Schultz SJ (junio de 2009). "Actividad de la ARNasa H: estructura, especificidad y función en la transcripción inversa". The FEBS Journal . 134 (1–2): 86–103. doi :10.1016/j.virusres.2007.12.007. PMC 2464458 . PMID 18261820.
- ^ Moelling K, Broecker F, Kerrigan JE (2014). "RNasa H: especificidad, mecanismos de acción y diana antiviral". Retrovirus humanos . Métodos en biología molecular. Vol. 1087. págs. 71–84. doi :10.1007/978-1-62703-670-2_7. ISBN . 978-1-62703-669-6. Número de identificación personal 24158815.
- ^ ab Vargiu L, Rodríguez-Tomé P, Sperber GO, Cadeddu M, Grandi N, Blikstad V, et al. (Enero de 2016). "Clasificación y caracterización de retrovirus endógenos humanos; las formas en mosaico son comunes". Retrovirología . 13 : 7. doi : 10.1186/s12977-015-0232-y . PMC 4724089 . PMID 26800882.
- ^ Peters, PJ, Marston, BJ, Weidle, PJ y Brooks, JT (2013). Infección por el virus de la inmunodeficiencia humana. Hunter's Tropical Medicine and Emerging Infectious Disease, 217–247. doi:10.1016/b978-1-4160-4390-4.00027-8
- ^ Coffin JM, Hughes SH, Varmus HE (1997). "Organización genética". Retrovirus . Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-571-2.
- ^ Belshaw R, Pereira V, Katzourakis A, Talbot G, Paces J, Burt A, Tristem M (abril de 2004). "Reinfección a largo plazo del genoma humano por retrovirus endógenos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 101 (14): 4894–9. Bibcode :2004PNAS..101.4894B. doi : 10.1073/pnas.0307800101 . PMC 387345 . PMID 15044706.
- ^ Medstrand P, van de Lagemaat LN, Dunn CA, Landry JR, Svenback D, Mager DL (2005). "Impacto de los elementos transponibles en la evolución de la regulación génica de los mamíferos". Cytogenetic and Genome Research . 110 (1–4): 342–52. doi :10.1159/000084966. PMID 16093686. S2CID 25307890.
- ^ Svarovskaia ES, Cheslock SR, Zhang WH, Hu WS, Pathak VK (enero de 2003). "Tasas de mutación retroviral y fidelidad de la transcriptasa inversa". Frontiers in Bioscience . 8 (1–3): d117–34. doi : 10.2741/957 . PMID 12456349.
- ^ ab Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (noviembre de 2018). "La recombinación es necesaria para la replicación eficiente del VIH-1 y el mantenimiento de la integridad del genoma viral". Nucleic Acids Research . 46 (20): 10535–10545. doi :10.1093/nar/gky910. PMC 6237782 . PMID 30307534.
- ^ Cromer D, Grimm AJ, Schlub TE, Mak J, Davenport MP (enero de 2016). "Estimación de la tasa de recombinación y cambio de plantilla del VIH in vivo". SIDA . 30 (2): 185–92. doi : 10.1097/QAD.0000000000000936 . PMID 26691546. S2CID 20086739.
- ^ Jolly C (marzo de 2011). "Transmisión de retrovirus de célula a célula: inmunidad innata y factores de restricción inducidos por interferón". Virology . 411 (2): 251–9. doi :10.1016/j.virol.2010.12.031. PMC 3053447 . PMID 21247613.
- ^ MacLachlan, N. James; Dubovi, Edward J. (2011). Fenner's Veterinary Virology (Cuarta edición). Academic Press. pág. 250. ISBN 978-0-12-375159-1. Recuperado el 6 de mayo de 2020 .
- ^ Aiewsakun P, Katzourakis A (enero de 2017). "Origen marino de los retrovirus en la era paleozoica temprana". Nature Communications . 8 : 13954. Bibcode :2017NatCo...813954A. doi :10.1038/ncomms13954. PMC 5512871 . PMID 28071651.
- ^ Desport M, ed. (2010). Lentivirus y macrófagos: interacciones moleculares y celulares . Caister Academic. ISBN 978-1-904455-60-8.
- ^ Ross, SR (2018). Respuestas inmunitarias celulares a los retrovirus. En Retrovirus-Cell Interactions (págs. 401–420). Elsevier. https://doi.org/10.1016/B978-0-12-811185-7.00011-X
- ^ Burrell, CJ, Howard, CR y Murphy, FA (2017). Retrovirus. En Fenner and White's Medical Virology (pp. 317–344). Elsevier. https://doi.org/10.1016/b978-0-12-375156-0.00023-0
- ^ Navegador de taxonomías ICTV
- ^ Lauber C, Seitz S, Mattei S, Suh A, Beck J, Herstein J, et al. (septiembre de 2017). "Descifrando el origen y la evolución de los virus de la hepatitis B por medio de una familia de virus de peces sin envoltura". Cell Host & Microbe . 22 (3): 387–399.e6. doi :10.1016/j.chom.2017.07.019. PMC 5604429 . PMID 28867387. y PDF
- ^ Krupovic M, Blomberg J, Coffin JM, Dasgupta I, Fan H, Geering AD, et al. (junio de 2018). "Ortervirales: nuevo orden de virus que unifica cinco familias de virus de transcripción inversa". Journal of Virology . 92 (12). doi :10.1128/JVI.00515-18. PMC 5974489 . PMID 29618642.
- ^ Verificación de hechos de Judy Mikovits, la controvertida viróloga que ataca a Anthony Fauci en un video conspirativo viral, por Martin Enserink, Jon Cohen, 8 de mayo de 2020, consultado el 17 de junio de 2022, sitio web science.org.
- ^ Neil, Stuart JD; Campbell, Edward M. (2020). "Ciencia falsa: XMRV, COVID-19 y el legado tóxico de la Dra. Judy Mikovits". Investigación sobre el SIDA y retrovirus humanos . 36 (7): 545–549. doi :10.1089/aid.2020.0095. PMC 7398426 . PMID 32414291.
- ^ Los conspiradores del virus elevan a un nuevo campeón, por Davey Alba , 9 de mayo de 2020, nytimes.com
- ^ Rutherford GW, Sangani PR, Kennedy GE (2003). "Regímenes de mantenimiento con antirretrovirales de tres o cuatro fármacos versus dos fármacos para la infección por VIH". Base de datos Cochrane de revisiones sistemáticas (4): CD002037. doi :10.1002/14651858.CD002037. PMID 14583945.
- ^ Gingerich DA (2008). "Inmunomodulador de células T de linfocitos (LTCI): Revisión de la inmunofarmacología de un nuevo producto biológico" (PDF) . Revista Internacional de Investigación Aplicada en Medicina Veterinaria . 6 (2): 61–68. ISSN 1559-470X.
Lectura adicional
- Coffin JM, Hughes SH, Varmus HE (1997). Coffin JM, Hughes SH, Varmus HE (eds.). Retrovirus. Laboratorio Cold Spring Harbor. ISBN 978-0-87969-571-2. Número de identificación personal 21433340. NBK19376.
- Specter M (3 de diciembre de 2007). "Anales de la ciencia: la sorpresa de Darwin". The New Yorker .
- Dostálková, A; Vokatá, B; Kaufman, F; Ulbrich, P; Ruml, T; Rumlová, M (2021). "Efecto de los polianiones pequeños en el ensamblaje in vitro de miembros seleccionados de retrovirus alfa, beta y gamma". Viruses . 13 (1): 129. doi : 10.3390/v13010129 . PMC 7831069 . PMID 33477490.
Enlaces externos
- ViralZone Un recurso del Instituto Suizo de Bioinformática para todas las familias virales, que proporciona información molecular y epidemiológica general (siga los enlaces para "Virus retrotranscriptivos" )
- Animación de retrovirus (se requiere Flash)
- Revista científica Retrovirología
- Capítulo sobre el ciclo de vida de los retrovirus De Kimball's Biology (páginas del libro de texto de biología en línea)


