Flora de la piel
La flora cutánea , también llamada microbiota cutánea , se refiere a la microbiota ( comunidades de microorganismos ) que residen en la piel , normalmente la piel humana .
Muchas de ellas son bacterias de las que hay alrededor de 1.000 especies en la piel humana pertenecientes a diecinueve filos . [1] [2] La mayoría se encuentran en las capas superficiales de la epidermis y en las partes superiores de los folículos pilosos .
La flora cutánea no suele ser patógena y es comensal (no es dañina para su huésped) o mutualista (ofrece un beneficio). Los beneficios que pueden ofrecer las bacterias incluyen evitar que los organismos patógenos transitorios colonicen la superficie de la piel, ya sea compitiendo por nutrientes, secretando sustancias químicas contra ellos o estimulando el sistema inmunológico de la piel . [3] Sin embargo, los microbios residentes pueden causar enfermedades cutáneas y entrar en el sistema sanguíneo , creando enfermedades potencialmente mortales, en particular en personas inmunodeprimidas . [3]
Una importante flora cutánea no humana es Batrachochytrium dendrobatidis , un hongo zoospórico quítrido y no hifal que causa quitridiomicosis , una enfermedad infecciosa que se cree que es responsable de la disminución de las poblaciones de anfibios . [4]
Variedad de especies
Bacteria

La estimación del número de especies presentes en las bacterias de la piel ha cambiado radicalmente gracias al uso del ARN ribosómico 16S para identificar las especies bacterianas presentes en las muestras de piel directamente a partir de su material genético. Anteriormente, dicha identificación dependía de cultivos microbiológicos en los que muchas variedades de bacterias no crecían y, por lo tanto, permanecían ocultas a la ciencia. [1]
A partir de investigaciones basadas en cultivos, se pensaba que Staphylococcus epidermidis y Staphylococcus aureus eran las especies dominantes. Sin embargo, las investigaciones sobre el ARN ribosómico 16S han descubierto que, si bien son comunes, estas especies representan solo el 5 % de las bacterias de la piel. Sin embargo, la variedad de la piel proporciona un hábitat rico y diverso para las bacterias . La mayoría proviene de cuatro filos: Actinomycetota (51,8 %), Bacillota (24,4 %), Pseudomonadota (16,5 %) y Bacteroidota (6,3 %). [5]

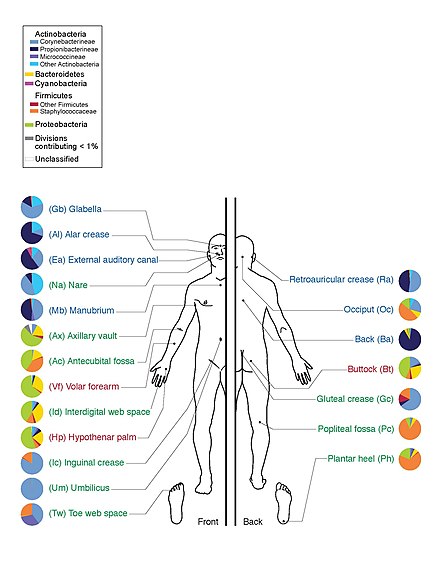
Hay tres áreas ecológicas principales: sebácea, húmeda y seca. Las especies de Propionibacteria y Staphylococci fueron las principales especies en las áreas sebáceas . En los lugares húmedos del cuerpo , dominan Corynebacteria junto con Staphylococci . En las áreas secas, hay una mezcla de especies, pero Betaproteobacteria y Flavobacteriales son dominantes. Ecológicamente, las áreas sebáceas tuvieron una mayor riqueza de especies que las húmedas y secas. Las áreas con menor similitud entre las personas en cuanto a especies fueron los espacios entre los dedos de las manos , los espacios entre los dedos de los pies , las axilas y el muñón del cordón umbilical . Las más similares fueron al lado de la fosa nasal , las fosas nasales (dentro de la fosa nasal) y en la espalda. [1]
Frecuencia de los microbios de la piel mejor estudiados [3] Organismo Observaciones Patogenicidad Estafilococo epidermidis Común Ocasionalmente patógeno Estafilococo áureo Infrecuente Generalmente patógeno Estafilococo warneri Infrecuente Ocasionalmente patógeno Estreptococo pyogenes Infrecuente Generalmente patógeno Estreptococo mitis Frecuente Ocasionalmente patógeno Cutibacterium acnes Frecuente Ocasionalmente patógeno Bacterias del género Corynebacterium . Frecuente Ocasionalmente patógeno Acinetobacter johnsonii Frecuente Ocasionalmente patógeno Pseudomonas aeruginosa Infrecuente Ocasionalmente patógeno
Hongo
Un estudio del área entre los dedos de los pies en 100 adultos jóvenes encontró 14 géneros diferentes de hongos. Estos incluyen levaduras como Candida albicans , Rhodotorula rubra , Torulopsis y Trichosporon cutaneum , dermatofitos (hongos que viven en la piel) como Microsporum gypseum y Trichophyton rubrum y hongos no dermatofitos (hongos oportunistas que pueden vivir en la piel) como Rhizopus stolonifer , Trichosporon cutaneum , Fusarium , Scopulariopsis brevicaulis , Curvularia , Alternaria alternata , Paecilomyces , Aspergillus flavus y especies de Penicillium . [6]
Un estudio del Instituto Nacional de Investigación del Genoma Humano en Bethesda, Maryland , investigó el ADN de los hongos de la piel humana en 14 lugares diferentes del cuerpo: el canal auditivo, el entrecejo, la nuca, detrás de la oreja, el talón, las uñas de los pies, entre los dedos de los pies, el antebrazo, la espalda, la ingle, las fosas nasales, el pecho, la palma y el pliegue del codo. El estudio mostró una gran diversidad de hongos en todo el cuerpo, siendo el talón el hábitat más rico, que alberga alrededor de 80 especies de hongos. En contraste, hay unas 60 especies en recortes de uñas de los pies y 40 entre los dedos de los pies. Otras áreas ricas son la palma, el antebrazo y la parte interior del codo, con entre 18 y 32 especies. La cabeza y el tronco albergaban entre 2 y 10 cada uno. [7]
Microbioma umbilical
El ombligo, o umbilical , es un área del cuerpo que rara vez está expuesta a la luz ultravioleta, jabones o secreciones corporales [8] (el ombligo no produce ninguna secreción o aceite) [9] y debido a que es una comunidad de bacterias casi inalterada [10] es una excelente parte del microbioma de la piel para estudiar. [11] El ombligo, o umbilical, es un microbioma húmedo del cuerpo [12] (con alta humedad y temperaturas), [13] que contiene una gran cantidad de bacterias, [14] especialmente bacterias que favorecen las condiciones húmedas como Corynebacterium [15] y Staphylococcus . [13]
El Proyecto de Biodiversidad del Ombligo comenzó en la Universidad Estatal de Carolina del Norte a principios de 2011 con dos grupos iniciales de 35 y 25 voluntarios. [10] A los voluntarios se les dieron hisopos de algodón estériles y se les pidió que los insertaran en sus ombligos, que giraran el hisopo de algodón tres veces y luego lo devolvieran a los investigadores en un frasco [16] que contenía 0,5 ml de tampón salino de fosfato al 10%. [10] Los investigadores de la Universidad Estatal de Carolina del Norte, dirigidos por Jiri Hulcr, [17] luego cultivaron las muestras en un cultivo hasta que las colonias bacterianas fueron lo suficientemente grandes como para ser fotografiadas y luego estas imágenes se publicaron en el sitio web del Proyecto de Biodiversidad del Ombligo (los voluntarios recibieron números de muestra para que pudieran ver sus propias muestras en línea). [16] Luego, estas muestras se analizaron utilizando bibliotecas de ADNr 16S para que se pudieran identificar las cepas que no crecieron bien en los cultivos. [10]
Los investigadores de la Universidad Estatal de Carolina del Norte descubrieron que, si bien era difícil predecir cada cepa de bacterias en el microbioma del ombligo, podían predecir qué cepas prevalecerían y qué cepas de bacterias serían bastante raras en el microbioma. [10] Se encontró que los microbiomas del ombligo solo contenían unos pocos tipos prevalentes de bacterias ( Staphylococcus , Corynebacterium , Actinobacteria, Clostridiales y Bacilli) y muchos tipos diferentes de bacterias raras. [10] Se descubrieron otros tipos de organismos raros dentro de los ombligos de los voluntarios, incluidos tres tipos de Archaea, dos de los cuales se encontraron en un voluntario que afirmó no haberse bañado ni duchado durante muchos años. [10]
Staphylococcus y Corynebacterium se encontraban entre los tipos de bacterias más comunes encontrados en los ombligos de los voluntarios de este proyecto y se ha descubierto que estos tipos de bacterias son los tipos de bacterias más comunes encontrados en la piel humana en estudios más amplios del microbioma de la piel [18] (del cual el Proyecto de Biodiversidad del Ombligo es parte). [10] (En estos estudios más amplios se ha descubierto que las mujeres generalmente tienen más Staphylococcus viviendo en sus microbiomas de la piel [18] (generalmente Staphylococcus epidermidis ) [16] y que los hombres tienen más Corynebacterium viviendo en sus microbiomas de la piel). [18]
Según el Proyecto de Biodiversidad del Ombligo [10] de la Universidad Estatal de Carolina del Norte, en el ombligo y las zonas circundantes se encuentran dos tipos de microorganismos. Las bacterias transitorias (bacterias que no se reproducen) [12] constituyen la mayoría de los organismos que se encuentran en el ombligo, y se estima que se encontraron 1400 cepas diferentes en el 95 % de los participantes del estudio. [19]
El Proyecto de Biodiversidad del Ombligo está en marcha y ya ha tomado muestras de más de 500 personas. [10] El proyecto fue diseñado con el objetivo de contrarrestar la idea errónea de que las bacterias siempre son dañinas para los humanos [20] y que los humanos están en guerra con las bacterias. [21] En realidad, la mayoría de las cepas de bacterias son inofensivas [13], si no beneficiosas, para el cuerpo humano. [22] Otro de los objetivos del proyecto es fomentar el interés público en la microbiología. [17] Trabajando en conjunto con el Proyecto del Microbioma Humano, el Proyecto de Biodiversidad del Ombligo también estudia las conexiones entre los microbiomas humanos y los factores de edad, sexo, etnia, ubicación [17] y salud general. [23]
Relación con el anfitrión
La microflora de la piel puede ser comensal , mutualista o patógena . A menudo pueden ser las tres dependiendo de la fuerza del sistema inmunológico de la persona . [3] La investigación sobre el sistema inmunológico en el intestino y los pulmones ha demostrado que la microflora ayuda al desarrollo de la inmunidad: sin embargo, dicha investigación solo ha comenzado a determinar si este es el caso de la piel. [3] Pseudomonas aeruginosa es un ejemplo de una bacteria mutualista que puede convertirse en un patógeno y causar enfermedades: si ingresa al sistema circulatorio, puede provocar infecciones en los huesos, las articulaciones, el sistema gastrointestinal y el respiratorio. También puede causar dermatitis . Sin embargo, P. aeruginosa produce sustancias antimicrobianas como el ácido pseudomónico (que se explotan comercialmente como la mupirocina ). Esto funciona contra las infecciones estafilocócicas y estreptocócicas . P. aeruginosa también produce sustancias que inhiben el crecimiento de especies de hongos como Candida krusei , Candida albicans , Torulopsis glabrata , Saccharomyces cerevisiae y Aspergillus fumigatus . [24] También puede inhibir el crecimiento de Helicobacter pylori . [25] Sus acciones antimicrobianas son tan importantes que se ha observado que "la eliminación de P. aeruginosa de la piel, mediante el uso de antibióticos orales o tópicos, puede permitir inversamente la colonización e infección aberrantes por levaduras". [3]
Otro aspecto de las bacterias es la generación de olor corporal . El sudor no tiene olor, pero varias bacterias pueden consumirlo y crear subproductos que pueden ser considerados pútridos por los humanos (a diferencia de las moscas, por ejemplo, que pueden encontrarlos atractivos). Algunos ejemplos son:
- Las propionibacterias en las glándulas sebáceas de adolescentes y adultos pueden convertir sus aminoácidos en ácido propiónico . [26]
- El Staphylococcus epidermidis crea olor corporal al descomponer el sudor en ácido isovalérico (ácido 3-metilbutanoico). [27]
- Bacillus subtilis produce un fuerte olor en los pies. [28]
Defensas de la piel
Péptidos antimicrobianos
La piel crea péptidos antimicrobianos como las catelicidinas que controlan la proliferación de microbios de la piel. Las catelicidinas no solo reducen directamente el número de microbios, sino que también provocan la secreción de citocinas que inducen inflamación , angiogénesis y reepitelización . Afecciones como la dermatitis atópica se han relacionado con la supresión de la producción de catelicidina. [29] En la rosácea, el procesamiento anormal de la catelicidina causa inflamación. La psoriasis se ha relacionado con el ADN propio creado a partir de péptidos de catelicidina que causa autoinflamación . Un factor importante que controla la catelicidina es la vitamina D 3. [30 ]
Acidez
Las capas superficiales de la piel son naturalmente ácidas ( pH 4-4,5) debido al ácido láctico del sudor y producido por las bacterias de la piel. [31] A este pH, la flora mutualista como los estafilococos , micrococos , corinebacterias y propionibacterias crecen, pero no las bacterias transitorias como las bacterias gramnegativas como Escherichia y Pseudomonas o las grampositivas como Staphylococcus aureus . [31] Otro factor que afecta al crecimiento de bacterias patológicas es que las sustancias antimicrobianas secretadas por la piel se potencian en condiciones ácidas. [31] En condiciones alcalinas, las bacterias dejan de estar adheridas a la piel y se desprenden más fácilmente. Se ha observado que la piel también se hincha en condiciones alcalinas y se abre permitiendo el movimiento bacteriano a la superficie. [31]
Sistema inmunitario
Si se activa, el sistema inmunitario de la piel produce inmunidad celular contra microbios como los dermatofitos (hongos de la piel). [32] Una reacción es aumentar la renovación del estrato córneo y, de este modo, eliminar el hongo de la superficie de la piel. Los hongos de la piel como el Trichophyton rubrum han evolucionado para crear sustancias que limitan la respuesta inmunitaria a ellos. [32] El desprendimiento de la piel es un medio general para controlar la acumulación de flora en la superficie de la piel. [33]
Enfermedades de la piel
Los microorganismos desempeñan un papel en enfermedades cutáneas no infecciosas como la dermatitis atópica , [34] la rosácea , la psoriasis , [35] y el acné . [36] La piel dañada puede hacer que las bacterias no patógenas se vuelvan patógenas . [37] La diversidad de especies en la piel está relacionada con el desarrollo posterior de la dermatitis. [38]
Acné vulgar
El acné vulgar es una afección cutánea común que se caracteriza por una producción excesiva de sebo por parte de la unidad pilosebácea y por la inflamación de la piel. [39] Las zonas afectadas suelen estar colonizadas por Propionibacterium acnes , un miembro de la microbiota comensal incluso en aquellas personas sin acné. [40] Las poblaciones elevadas de P. acnes están relacionadas con el acné vulgar, aunque solo ciertas cepas están fuertemente asociadas con el acné, mientras que otras lo están con la piel sana. La población relativa de P. acnes es similar entre las personas con acné y las que no lo tienen. [39] [40]
El tratamiento actual incluye fármacos antibacterianos tópicos y sistémicos que dan como resultado una disminución de la colonización y/o actividad de P. acnes . [41] El posible tratamiento probiótico incluye el uso de Staphylococcus epidermidis para inhibir el crecimiento de P. acnes . S. epidermidis produce ácido succínico que ha demostrado inhibir el crecimiento de P. acnes . [42] También se ha demostrado que Lactobacillus plantarum actúa como un antiinflamatorio y mejora las propiedades antimicrobianas de la piel cuando se aplica tópicamente. También se ha demostrado que es eficaz para reducir el tamaño de las lesiones de acné. [43]
Dermatitis atópica
Las personas con dermatitis atópica han mostrado un aumento en las poblaciones de Staphylococcus aureus tanto en la piel lesionada como en la no lesionada. [40] Los brotes de dermatitis atópica están asociados con una baja diversidad bacteriana debido a la colonización por S. aureus y, después del tratamiento estándar , se ha observado que la diversidad bacteriana aumenta. [ cita requerida ]
Los tratamientos actuales incluyen combinaciones de antibióticos tópicos o sistémicos, corticosteroides y baños de lejía diluida. [44] Los posibles tratamientos probióticos incluyen el uso de la bacteria comensal de la piel, S. epidermidis , para inhibir el crecimiento de S. aureus . Durante los brotes de dermatitis atópica, se ha demostrado que los niveles de población de S. epidermidis aumentan como un intento de controlar las poblaciones de S. aureus . [40] [44]
La baja diversidad microbiana intestinal en los bebés se ha asociado con un mayor riesgo de dermatitis atópica. [45] Los bebés con eccema atópico tienen niveles bajos de Bacteroides y niveles altos de Bacillota . Los Bacteroides tienen propiedades antiinflamatorias que son esenciales contra la dermatitis. [45] (Ver microbiota intestinal )
Psoriasis vulgar
La psoriasis vulgar generalmente afecta las zonas más secas de la piel, como los codos y las rodillas . Las zonas secas de la piel tienden a tener una alta diversidad microbiana y menos poblaciones que las zonas sebáceas. [41] Un estudio que utilizó técnicas de muestreo con hisopos muestra que las zonas ricas en Bacillota (principalmente Streptococcus y Staphylococcus ) y Actinomycetota (principalmente Corynebacterium y Propionibacterium ) están asociadas con la psoriasis. [46] Mientras que otro estudio que utilizó biopsias asocia niveles elevados de Bacillota y Actinomycetota con piel sana. [47] Sin embargo, la mayoría de los estudios muestran que las personas afectadas por psoriasis tienen una menor diversidad microbiana en las zonas afectadas.
Los tratamientos para la psoriasis incluyen agentes tópicos, fototerapia y agentes sistémicos. [48] La investigación actual sobre el papel de la microbiota de la piel en la psoriasis es inconsistente, por lo tanto, no existen tratamientos probióticos potenciales.
Rosácea
La rosácea suele estar relacionada con las zonas sebáceas de la piel. El ácaro de la piel Demodex folliculorum produce lipasas que le permiten utilizar el sebo como fuente de alimento, por lo que tiene una gran afinidad por las zonas sebáceas de la piel. Aunque forma parte de la microbiota comensal de la piel, los pacientes afectados de rosácea muestran un aumento de D. folliculorum en comparación con los individuos sanos, lo que sugiere patogenicidad . [49]
Bacillus oleronius , un microbio asociado a Demodex , no se encuentra típicamente en la microbiota comensal de la piel, pero inicia vías inflamatorias cuyo mecanismo de inicio es similar al de los pacientes con rosácea. [40] También se han aislado poblaciones de S. epidermidis de pústulas de pacientes con rosácea. Sin embargo, es posible que Demodex las haya trasladado a áreas que favorecen el crecimiento, ya que se ha demostrado que Demodex transporta bacterias por la cara. [50]
Los tratamientos actuales incluyen antibióticos tópicos y orales y terapia con láser. [51] Como la investigación actual aún no ha demostrado un mecanismo claro de la influencia de Demodex en la rosácea, no existen tratamientos probióticos potenciales.
Clínico
Dispositivos infectados
Los microbios de la piel son una fuente potencial de dispositivos médicos infectados, como los catéteres . [52]
Higiene
La piel humana alberga numerosas especies bacterianas y fúngicas, algunas de las cuales se sabe que son dañinas, otras que son beneficiosas y la gran mayoría de ellas no han sido investigadas. El uso de jabones bactericidas y fungicidas conducirá inevitablemente a poblaciones bacterianas y fúngicas resistentes a los productos químicos empleados (véase resistencia a los medicamentos ).
Contagio
La flora cutánea no pasa fácilmente entre personas: 30 segundos de fricción moderada y contacto con las manos secas dan como resultado una transferencia de solo el 0,07 % de la flora natural de las manos desnudas, con un porcentaje mayor si se utiliza con guantes. [53]
Eliminación
El lavado antimicrobiano más eficaz (reducción del 60-80%) es con etanol , isopropanol y n-propanol . Los virus son los más afectados por concentraciones altas (95%) de etanol, mientras que las bacterias son más afectadas por n-propanol. [54]
Los jabones no medicinales no son muy eficaces, como lo ilustran los siguientes datos: los trabajadores de la salud se lavaron las manos una vez con jabón líquido no medicinal durante 30 segundos, mientras que los estudiantes y técnicos lo hicieron 20 veces. [55]
Flora cutánea en dos grupos hospitalarios en unidades formadoras de colonias por ml . Estado de la piel de las manos y del grupo sucio lavado Trabajadores de la salud saludables 3.47 3.15 Trabajadores de la salud dañados 3.33 3.29 Estudiantes/técnicos sanos 4.39 3.54 Estudiantes/técnicos dañados 4.58 4.43
Un uso importante del lavado de manos es prevenir la transmisión de la flora cutánea resistente a los antibióticos que causa infecciones adquiridas en el hospital, como el Staphylococcus aureus resistente a la meticilina . Si bien dicha flora se ha vuelto resistente a los antibióticos debido a los antibióticos, no hay evidencia de que los antisépticos o desinfectantes recomendados seleccionen organismos resistentes a los antibióticos cuando se usan en el lavado de manos. [56] Sin embargo, muchas cepas de organismos son resistentes a algunas de las sustancias utilizadas en jabones antibacterianos, como el triclosán . [56]
Un estudio sobre jabones en barra en clínicas dentales descubrió que todos tenían su propia flora y, en promedio, de dos a cinco géneros diferentes de microorganismos, y los más utilizados tenían más probabilidades de tener más variedades de especies. [57] Otro estudio sobre jabones en barra en baños públicos encontró incluso más flora. [58] Otro estudio descubrió que los jabones muy secos no están colonizados, mientras que todos los que descansan en charcos de agua sí lo están. [59] Sin embargo, un experimento con jabones inoculados con Pseudomonas aeruginosa y Escherichia coli demostró que lavarse con jabón en barra inoculado no transmitía estas bacterias a las manos de los participantes. [60]
Piel dañada
El lavado repetido de la piel puede dañar la capa externa protectora y provocar pérdida transepidérmica de agua. Esto se puede observar en asperezas caracterizadas por descamación y sequedad, picor, dermatitis provocada por microorganismos y alérgenos que penetran en la capa córnea y enrojecimiento. El uso de guantes puede causar más problemas ya que produce un ambiente húmedo que favorece el crecimiento de microbios y también contiene irritantes como el látex y el talco . [61]
Lavarse las manos puede dañar la piel porque la capa superior del estrato córneo de la piel consta de 15 a 20 capas de discos de queratina , corneocitos , cada uno de los cuales está rodeado por una fina película de lípidos de la piel que pueden eliminarse con alcoholes y detergentes . [62]
También se ha descubierto que la piel dañada definida por un agrietamiento extenso de la superficie de la piel, enrojecimiento generalizado o sangrado ocasional está colonizada con mayor frecuencia por Staphylococcus hominis y es más probable que estos sean resistentes a la meticilina . [61] Aunque no está relacionado con una mayor resistencia a los antibióticos, la piel dañada también tenía más probabilidades de ser colonizada por Staphylococcus aureus , bacterias gramnegativas , enterococos y Candida . [61]
Comparación con otras especies de flora
La flora de la piel es diferente de la del intestino, que es predominantemente Bacillota y Bacteroidota . [63] También hay un bajo nivel de variación entre las personas que no se encuentra en los estudios intestinales. [5] Sin embargo, tanto la flora intestinal como la de la piel carecen de la diversidad que se encuentra en la flora del suelo . [1]
Véase también
Referencias
- ^ abcd Grice EA, Kong HH, Conlan S (2009). "Diversidad topográfica y temporal del microbioma de la piel humana". Science . 324 (5931): 1190–92. Bibcode :2009Sci...324.1190G. doi :10.1126/science.1171700. PMC 2805064 . PMID 19478181.
- ^ "Tu cuerpo es un país de las maravillas... de bacterias". www.science.org . 28 de mayo de 2009 . Consultado el 2 de enero de 2023 .
- ^ abcdef Cogen AL, Nizet V, Gallo RL (2008). "Microbiota cutánea: ¿fuente de enfermedad o defensa?". Br J Dermatol . 158 (3): 442–55. doi :10.1111/j.1365-2133.2008.08437.x. PMC 2746716 . PMID 18275522.
- ^ Voyles, Jamie; Young, Sam; Berger, Lee; Campbell, Craig; Voyles, Wyatt F.; Dinudom, Anuwat; Cook, David; Webb, Rebecca; Alford, Ross A.; Skerratt, Lee F.; Speare, Rick (23 de octubre de 2009). "Patogénesis de la quitridiomicosis, una causa de la catastrófica disminución de los anfibios". Science . 326 (5952): 582–585. Bibcode :2009Sci...326..582V. doi :10.1126/science.1176765. ISSN 0036-8075. PMID 19900897. S2CID 52850132.
- ^ ab Grice EA, Kong HH, Renaud G, Young AC, Bouffard GG, Blakesley RW, Wolfsberg TG, Turner ML, Segre JA (2008). "Un perfil de diversidad de la microbiota de la piel humana". Genome Res . 18 (7): 1043–50. doi :10.1101/gr.075549.107. PMC 2493393 . PMID 18502944.
- ^ Oyeka CA, Ugwu LO (2002). "Flora fúngica de las membranas de los dedos de los pies humanos". Micosis . 45 (11–12): 488–91. doi :10.1046/j.1439-0507.2002.00796.x. PMID 12472726. S2CID 8789635.
- ↑ Helen Briggs (22 de mayo de 2013). «Los pies albergan más de 100 hongos». BBC News . Consultado el 2 de enero de 2023 .
- ^ Ecological Society of America (4 de agosto de 2011). "Bellybutton microbiomes: Ecological research on the human biome" (Comunicado de prensa) . ScienceDaily . Consultado el 20 de abril de 2013 .
- ^ Nierenberg, Cari (14 de abril de 2011). «Nuevo significado de la expresión «mirarse el ombligo»: los científicos estudian las bacterias del ombligo» . Consultado el 29 de septiembre de 2013 .
- ^ abcdefghij Hulcr, Jirir; Andrew M. Latimer; Jessica B. Henley; Nina R. Rountree; Noah Fierer; Andrea Lucky; Margaret D. Lowman; Robert R. Dunn (7 de noviembre de 2012). "Hay una jungla ahí dentro: las bacterias de los ombligos son muy diversas, pero predecibles". PLOS ONE . 7 (11): e47712. Bibcode :2012PLoSO...747712H. doi : 10.1371/journal.pone.0047712 . PMC 3492386 . PMID 23144827.
- ^ "La vida salvaje de tu cuerpo" . Consultado el 1 de septiembre de 2013 .
- ^ ab Kong, Hiedi (17 de junio de 2011). "Microbioma de la piel: perspectivas genómicas sobre la diversidad y el papel de los microbios de la piel". Trends Mol. Med . 17 (6): 320–8. doi :10.1016/j.molmed.2011.01.013. PMC 3115422. PMID 21376666 .
- ^ abc Grice, Elizabeth; Julia Segre (9 de abril de 2011). "El microbioma de la piel". Nat Rev Microbiol . 9 (4): 244–53. doi :10.1038/nrmicro2537. PMC 3535073 . PMID 21407241.
- ^ Kaplan, Karen (1 de junio de 2009). «Estudio demuestra que estás cubierto de bacterias: vive con ello». The Star . Archivado desde el original el 11 de noviembre de 2013. Consultado el 29 de septiembre de 2013 .
- ^ Grice, Elizabeth; Heidi H. Kong; Sean Conlan; Clayton B. Deming; Joie Davis; Alice C. Young; Gerard G. Bouffard; Robert W. Blakesley; Patrick R. Murray; Eric D. Green; Maria L. Turner; Julia A. Segre (29 de mayo de 2009). "Diversidad topográfica y temporal del microbioma de la piel humana". Science . 324 (5931): 1190–2. Bibcode :2009Sci...324.1190G. doi :10.1126/science.1171700. PMC 2805064 . PMID 19478181.
- ^ abc Parker-Pope, Tara (14 de abril de 2011). "What's in Your Belly Button" (¿Qué hay en tu ombligo?) . Consultado el 29 de septiembre de 2013 .
- ^ abc Nierenberg, Cari (14 de abril de 2011). "Nuevo significado de 'mirarse el ombligo': los científicos estudian las bacterias del ombligo". NBC News . Consultado el 29 de septiembre de 2013 .
- ^ abc Callewaert, Chris; Frederiek-Maarten Kerckhof; Michael S. Granitsiotis; Mireille Van Gele; Tom Van de Wiele; Nico Boon (12 de agosto de 2013). "Caracterización de grupos de estafilococos y corynebacterium en la región axilar humana". MÁS UNO . 8 (8): e70538. Código Bib : 2013PLoSO...870538C. doi : 10.1371/journal.pone.0070538 . PMC 3741381 . PMID 23950955.
- ^ Saunders, Chris (12 de julio de 2011). "Mirarse el ombligo en NC State conduce a un descubrimiento importante". Red & White for Life :: Asociación de exalumnos de la NC State University . Consultado el 20 de abril de 2013 .
- ^ Aldhous, Peter. "El bioma del ombligo es más que un trozo de pelusa". Archivado desde el original el 2013-10-02 . Consultado el 2013-09-29 .
- ^ "Microbios humanos" . Consultado el 29 de septiembre de 2013 .
- ^ Ahmad, Salar; Shailly Anand; Rup Lal (septiembre de 2012). "Los comensales de la piel regulan la inmunidad cutánea". Indian J. Microbiol . 52 (3): 517–8. doi :10.1007/s12088-012-0301-z. PMC 3460106 . PMID 23997352.
- ^ Grice, Isabel; Julia Segre (6 de junio de 2012). "El microbioma humano: nuestro segundo genoma". Annu Rev Genom Hum Genet . 13 (1): 151–70. doi :10.1146/annurev-genom-090711-163814. PMC 3518434 . PMID 22703178.
- ^ Kerr JR (1994). "Supresión del crecimiento fúngico exhibida por Pseudomonas aeruginosa". J Clin Microbiol . 32 (2): 525–7. doi :10.1128/JCM.32.2.525-527.1994. PMC 263067 . PMID 8150966.
- ^ Krausse R, Piening K, Ullmann U (2005). "Efectos inhibidores de varios microorganismos en el crecimiento de Helicobacter pylori". Lett Appl Microbiol . 40 (1): 81–6. doi : 10.1111/j.1472-765X.2004.01632.x . PMID 15613007. S2CID 2253604.
- ^ Himmi, EH; Bories, A.; Boussaid, A.; Hassani, L. (1 de abril de 2000). "Fermentación de glicerol y glucosa con ácido propiónico por Propionibacterium acidipropionici y Propionibacterium freudenreichii ssp.shermanii". Microbiología Aplicada y Biotecnología . 53 (4): 435–440. doi :10.1007/s002530051638. ISSN 1432-0614. PMID 10803900.
- ^ Ara K, Hama M, Akiba S, et al. (2006). "Olor de pies debido al metabolismo microbiano y su control". Can. J. Microbiol . 52 (4): 357–64. doi :10.1139/w05-130. PMID 16699586. S2CID 36221022.
- ^ Ara K, Hama M, Akiba S, Koike K, Okisaka K, Hagura T, Kamiya T, Tomita F (2006). "Olor de pies debido al metabolismo microbiano y su control". Can J Microbiol . 52 (4): 357–64. doi :10.1139/w05-130. PMID 16699586.[ enlace muerto permanente ]
- ^ Patra, Vijaykumar; Mayer, Gerlinde; Gruber-Wackernagel, Alexandra; Horn, Michael; Lembo, Serena; Wolf, Peter (2018). "Perfil único de expresión de péptidos antimicrobianos en lesiones polimórficas de erupción lumínica en comparación con piel sana, dermatitis atópica y psoriasis". Fotodermatología, fotoinmunología y fotomedicina . 34 (2): 137–144. doi :10.1111/phpp.12355. PMC 5888155 . PMID 29044786.
- ^ Schauber J, Gallo RL (2008). "Péptidos antimicrobianos y el sistema de defensa inmunitaria de la piel". J Allergy Clin Immunol . 122 (2): 261–6. doi :10.1016/j.jaci.2008.03.027. PMC 2639779 . PMID 18439663.
- ^ abcd Lambers H, Piessens S, Bloem A, Pronk H, Finkel P (2006). "El pH natural de la superficie de la piel está en promedio por debajo de 5, lo que es beneficioso para su flora residente". Revista Internacional de Ciencias Cosméticas . 28 (5): 359–70. doi :10.1111/j.1467-2494.2006.00344.x. PMID 18489300. S2CID 25191984.
- ^ ab Dahl MV (1993). "Supresión de la inmunidad y la inflamación por productos producidos por dermatofitos". J Am Acad Dermatol . 28 (5 Pt 1): S19–S23. doi :10.1016/s0190-9622(09)80303-4. PMID 8496406.
- ^ Percival, Steven L; Emanuel, Charlotte; Cutting, Keith F; Williams, David W (febrero de 2012). "Microbiología de la piel y el papel de las biopelículas en la infección". Revista Internacional de Heridas . 9 (1): 14–32. doi :10.1111/j.1742-481X.2011.00836.x. ISSN 1742-4801. PMC 7950481 . PMID 21973162.
- ^ Baker BS (2006). "El papel de los microorganismos en la dermatitis atópica". Clin Exp Immunol . 144 (1): 1–9. doi :10.1111/j.1365-2249.2005.02980.x. PMC 1809642 . PMID 16542358.
- ^ Paulino LC, Tseng CH, Strober BE, Blaser MJ (2006). "Análisis molecular de la microbiota fúngica en muestras de piel humana sana y lesiones psoriásicas". J Clin Microbiol . 44 (8): 2933–41. doi :10.1128/JCM.00785-06. PMC 1594634 . PMID 16891514.
- ^ Holland KT, Cunliffe WJ, Roberts CD (1977). "Acné vulgar: una investigación sobre el número de difteroides anaeróbicos y miembros de Micrococcaceae en piel normal y con acné". Br J Dermatol . 96 (6): 623–6. doi :10.1111/j.1365-2133.1977.tb05206.x. PMID 141301. S2CID 37507292.
- ^ Roth RR, James WD (1988). "Ecología microbiana de la piel". Annu Rev Microbiol . 42 (1): 441–64. doi :10.1146/annurev.mi.42.100188.002301. PMID 3144238.
- ^ Williams, Michael R.; Gallo, Richard L. (2017). "Evidencia de que la disbiosis del microbioma de la piel humana promueve la dermatitis atópica". Revista de dermatología investigativa . 137 (12): 2460–2461. doi :10.1016/j.jid.2017.09.010. PMC 5814121 . PMID 29169458.
- ^ ab Fitz-Gibbon, S; Shuta, T; Bor-Han, C; Nguyen, L; Du, C; Minghsun, L; Elashoff, D; Erfe, MC; Loncaric, A; Kim, J; Modlin, RL; Miller, JF; Sodergren, E; Craft, N; Weinstock, GM; Li, H (2013). "Poblaciones de cepas de Propionibacterium acnes en el microbioma de la piel humana asociadas con el acné". J Invest Dermatol . 133 (9): 2152–2160. doi :10.1038/jid.2013.21. PMC 3745799 . PMID 23337890.
- ^ abcde Grice, EA (2014). "El microbioma de la piel: potencial para nuevos enfoques diagnósticos y terapéuticos para las enfermedades cutáneas". Semin Cutan Med Surg . 33 (2): 98–103. doi :10.12788/j.sder.0087. PMC 4425451 . PMID 25085669. Archivado desde el original el 11 de abril de 2015.
- ^ ab Hannigan, GD; Grice, EA (2013). "Ecología microbiana de la piel en la era de la metagenómica y la microbiología molecular". Cold Spring Harb Perspect Med . 3 (12): a015362. doi :10.1101/cshperspect.a015362. PMC 3839604. PMID 24296350 .
- ^ Muya, S; Wang, Y; Yu, J; Kuo, S; Coda, A; Jiang, Y; Gallo, RL; Huang, CM (2013). "Fermentación de Propionibacterium acnes, una bacteria comensal en el microbioma de la piel humana, como probióticos cutáneos contra Staphylococcus aureus resistente a la meticilina". PLOS ONE . 8 (2): e55380. Bibcode :2013PLoSO...855380S. doi : 10.1371/journal.pone.0055380 . PMC 3566139 . PMID 23405142.
- ^ Muizzuddin, N; Maher, W; Sullivan, M; Schnittger, S; Mammone, T (2012). "Efecto fisiológico de los probióticos en la piel". J Cosmet Sci . 63 (6): 385–95. PMID 23286870.
- ^ ab Kong, HH; Oh, J; Deming, C; Conlan, S; Grice, EA; Beatson, MA; Nomicos, E; Polley, EC; Komarow, HD; NISC Comparative Sequence Program; Murray, PR; Turner, ML; Segre, JA (2012). "Cambios temporales en el microbioma de la piel asociados con brotes de enfermedades y tratamiento en niños con dermatitis atópica". Genome Res . 22 (5): 850–9. doi :10.1101/gr.131029.111. PMC 3337431 . PMID 22310478.
- ^ ab Abrahamsson, TR; Jakobsson, ÉL; Andersson, AF; Björkstén, B; Engstrand, L; Jenmalm, MC (2012). "Baja diversidad de la microbiota intestinal en bebés con eccema atópico". J Alergia Clin Immunol . 129 (2): 434–40, 440.e1–2. doi : 10.1016/j.jaci.2011.10.025 . PMID 22153774.
- ^ Alekseyenko, AV; Perez-Perez, GI; De Souza, A; Strober, B; Gao, Z; Bihan, M; Li, K; Methé, BA; Blaser, MJ (2013). "Diferenciación comunitaria de la microbiota cutánea en la psoriasis". Microbioma . 1 (1): 31. doi : 10.1186/2049-2618-1-31 . PMC 4177411 . PMID 24451201.
- ^ Fahlén, A; Engstrand, L; Baker, BS; Powles, A; Fry, L (2012). "Comparación de la microbiota bacteriana en biopsias de piel de piel normal y psoriásica". Arch Dermatol Res . 304 (1): 15–22. doi :10.1007/s00403-011-1189-x. PMID 22065152. S2CID 9169314.
- ^ Menter, A; Griffiths, CE (2007). "Manejo actual y futuro de la psoriasis". Lancet . 370 (9583): 272–84. doi :10.1016/S0140-6736(07)61129-5. PMID 17658398. S2CID 7907468.
- ^ Casas, C; Paul, C; Lahfa, M; Livideanu, B; Lejeune, O; Alvarez-Georges, S; Saint-Martory, C; Degouy, A; Mengeaud, V; Ginisty, H; Durbise, E; Schmitt, AM; Redoulès, D (2012). "Cuantificación de Demodex folliculorum por PCR en rosácea y su relación con la activación inmunitaria innata de la piel". Exp Dermatol . 21 (12): 906–10. doi :10.1111/exd.12030. PMID 23171449. S2CID 19722615.
- ^ Jarmuda, S; O'Reilly, N; Zaba, R; Jakubowicz, O; Szkaradkiewicz, A; Kavanagh, K (2012). "Potencial papel de los ácaros y bacterias Demodex en la inducción de la rosácea". J Med Microbiol . 61 (Pt 11): 1504–10. doi : 10.1099/jmm.0.048090-0 . PMID 22933353.
- ^ Cohen, AF; Tiemstra, JD (2002). "Diagnóstico y tratamiento de la rosácea". J Am Board Fam Pract . 15 (3): 214–7. PMID 12038728.
- ^ Martín-Rabadán P, Gijón P, Alcalá L, Rodríguez-Créixems M, Alvarado N, Bouza E (2008). "Propionibacterium acnes es un colonizador común de catéteres intravasculares". J Infectar . 56 (4): 257–60. doi :10.1016/j.jinf.2008.01.012. PMID 18336916.
- ^ Lingaas E, Fagernes M (2009). "Desarrollo de un método para medir la transferencia bacteriana de las manos". J Hosp Infect . 72 (1): 43–9. doi :10.1016/j.jhin.2009.01.022. PMID 19282052.
- ^ Kampf G, Kramer A (2004). "Antecedentes epidemiológicos de la higiene de manos y evaluación de los agentes más importantes para frotar y frotar". Clin Microbiol Rev . 17 (4): 863–93. doi :10.1128/CMR.17.4.863-893.2004. PMC 523567 . PMID 15489352.
- ^ Borges LF, Silva BL, Gontijo Filho PP (2007). "Lavado de manos: cambios en la flora cutánea". Am J Infect Control . 35 (6): 417–20. doi :10.1016/j.ajic.2006.07.012. PMID 17660014.
- ^ ab Weber DJ, Rutala WA (2006). "Uso de germicidas en el hogar y en el ámbito sanitario: ¿existe una relación entre el uso de germicidas y la resistencia a los antibióticos?". Infect Control Hosp Epidemiol . 27 (10): 1107–19. doi :10.1086/507964. PMID 17006819. S2CID 20734025.
- ^ Hegde PP, Andrade AT, Bhat K (2006). "Contaminación microbiana de las pastillas de jabón "en uso" en las clínicas dentales". Indian J Dent Res . 17 (2): 70–3. doi : 10.4103/0970-9290.29888 . PMID 17051871.
- ^ Kabara JJ, Brady MB (1984). "Contaminación de jabones en barra en condiciones de "uso"". J Environ Pathol Toxicol Oncol . 5 (4–5): 1–14. PMID 6394740.
- ^ Afolabi BA, Oduyebo OO, Ogunsola FT (2007). "Flora bacteriana de jabones de uso común en tres hospitales de Nigeria". East Afr Med J. 84 ( 10): 489–95. doi : 10.4314/eamj.v84i10.9567 . PMID: 18232270.
- ^ Heinze JE, Yackovich F (1988). "Es poco probable que el lavado con jabón en barra contaminado transfiera bacterias". Epidemiol Infect . 101 (1): 135–42. doi :10.1017/s0950268800029290. PMC 2249330 . PMID 3402545.
- ^ abc Larson EL, Hughes CA, Pyrek JD, Sparks SM, Cagatay EU, Bartkus JM (1998). "Cambios en la flora bacteriana asociados con daño cutáneo en las manos del personal sanitario". Am J Infect Control . 26 (5): 513–21. doi : 10.1016/s0196-6553(98)70025-2 . PMID 9795681.
- ^ Kownatzki E (2003). "Higiene de manos y salud de la piel". J Hosp Infect . 55 (4): 239–45. doi :10.1016/j.jhin.2003.08.018. PMID 14629966.
- ^ Eckburg PB, Bik EM, Bernstein CN, Purdom E, Dethlefsen L, Sargent M, Gill SR, Nelson KE, Relman DA (2005). "Diversidad de la flora microbiana intestinal humana". Science . 308 (5728): 1635–8. Bibcode :2005Sci...308.1635E. doi :10.1126/science.1110591. PMC 1395357 . PMID 15831718.
Enlaces externos
- Celulitis Infección de la piel
- Proyecto del microbioma humano
- Libro de texto en línea de bacteriología de Todar
- Higiene de la piel: ¿cuándo lo limpio es demasiado limpio?



