Fosforilación oxidativa
La fosforilación oxidativa (UK / ɒ k ˈ s ɪ d . ə . t ɪ v / , US / ˈ ɑː k . s ɪ ˌ d eɪ . t ɪ v / [1] ) o fosforilación ligada al transporte de electrones u oxidación terminal es la vía metabólica en la que las células utilizan enzimas para oxidar nutrientes , liberando así energía química para producir trifosfato de adenosina (ATP). En eucariotas , esto tiene lugar dentro de las mitocondrias . Casi todos los organismos aeróbicos llevan a cabo la fosforilación oxidativa. Esta vía es tan omnipresente porque libera más energía que los procesos de fermentación alternativos como la glucólisis anaeróbica .
La energía almacenada en los enlaces químicos de la glucosa es liberada por la célula en el ciclo del ácido cítrico , produciendo dióxido de carbono y los energéticos donadores de electrones NADH y FADH . La fosforilación oxidativa utiliza estas moléculas y O 2 para producir ATP , que se utiliza en toda la célula siempre que se necesita energía. Durante la fosforilación oxidativa, los electrones se transfieren desde los donadores de electrones a una serie de aceptores de electrones en una serie de reacciones redox que terminan en oxígeno, cuya reacción libera la mitad de la energía total. [2]
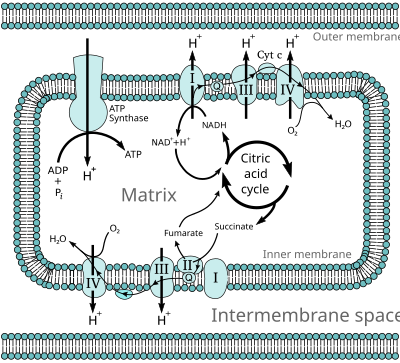
En los eucariotas , estas reacciones redox son catalizadas por una serie de complejos proteicos dentro de la membrana interna de las mitocondrias de la célula, mientras que, en los procariotas , estas proteínas se encuentran en la membrana externa de la célula. Estos conjuntos de proteínas enlazadas se denominan cadena de transporte de electrones . En los eucariotas, intervienen cinco complejos proteicos principales, mientras que en los procariotas están presentes muchas enzimas diferentes, que utilizan una variedad de donantes y aceptores de electrones.
La energía transferida por los electrones que fluyen a través de esta cadena de transporte de electrones se utiliza para transportar protones a través de la membrana mitocondrial interna , en un proceso llamado transporte de electrones . Esto genera energía potencial en forma de gradiente de pH y el potencial eléctrico resultante a través de esta membrana. Esta reserva de energía se aprovecha cuando los protones fluyen de regreso a través de la membrana y hacia abajo por el gradiente de energía potencial, a través de una enzima grande llamada ATP sintasa en un proceso llamado quimiosmosis . La ATP sintasa usa la energía para transformar el difosfato de adenosina (ADP) en trifosfato de adenosina, en una reacción de fosforilación . La reacción es impulsada por el flujo de protones, que fuerza la rotación de una parte de la enzima. La ATP sintasa es un motor mecánico rotatorio.
Aunque la fosforilación oxidativa es una parte vital del metabolismo, produce especies reactivas de oxígeno como el superóxido y el peróxido de hidrógeno , que conducen a la propagación de radicales libres , dañando las células y contribuyendo a la enfermedad y, posiblemente, al envejecimiento y la senescencia . Las enzimas que llevan a cabo esta vía metabólica también son el objetivo de muchos medicamentos y venenos que inhiben sus actividades.
Quimiosmosis
La fosforilación oxidativa funciona mediante el uso de reacciones químicas que liberan energía para impulsar reacciones que requieren energía. Se dice que los dos conjuntos de reacciones están acoplados . Esto significa que uno no puede ocurrir sin el otro. La cadena de reacciones redox que impulsa el flujo de electrones a través de la cadena de transporte de electrones, desde los donantes de electrones como NADH hasta los aceptores de electrones como el oxígeno y el hidrógeno (protones), es un proceso exergónico : libera energía, mientras que la síntesis de ATP es un proceso endergónico , que requiere un aporte de energía. Tanto la cadena de transporte de electrones como la ATP sintasa están incrustadas en una membrana, y la energía se transfiere de la cadena de transporte de electrones a la ATP sintasa mediante movimientos de protones a través de esta membrana, en un proceso llamado quimiosmosis . [3] Una corriente de protones es impulsada desde el lado N negativo de la membrana al lado P positivo a través de las enzimas de bombeo de protones de la cadena de transporte de electrones. El movimiento de protones crea un gradiente electroquímico a través de la membrana, llamado fuerza protón-motriz . Tiene dos componentes: una diferencia en la concentración de protones (un gradiente de H + , Δ pH ) y una diferencia en el potencial eléctrico , con el lado N con carga negativa. [4]
La ATP sintasa libera esta energía almacenada completando el circuito y permitiendo que los protones fluyan por el gradiente electroquímico, de regreso al lado N de la membrana. [5] El gradiente electroquímico impulsa la rotación de parte de la estructura de la enzima y acopla este movimiento a la síntesis de ATP.
Los dos componentes de la fuerza protón-motriz son termodinámicamente equivalentes: en las mitocondrias, la mayor parte de la energía es proporcionada por el potencial; en las bacterias alcalófilas , la energía eléctrica incluso tiene que compensar una diferencia inversa de pH. A la inversa, los cloroplastos funcionan principalmente en ΔpH. Sin embargo, también requieren un pequeño potencial de membrana para la cinética de la síntesis de ATP. En el caso de la fusobacteria Propionigenium modestum, impulsa la contrarrotación de las subunidades a y c del motor F O de la ATP sintasa. [4]
La cantidad de energía liberada por la fosforilación oxidativa es alta, comparada con la cantidad producida por la fermentación anaeróbica . La glucólisis produce solo 2 moléculas de ATP, pero entre 30 y 36 ATP se producen por la fosforilación oxidativa de las 10 moléculas de NADH y 2 de succinato que se forman al convertir una molécula de glucosa en dióxido de carbono y agua [6] , mientras que cada ciclo de beta oxidación de un ácido graso produce alrededor de 14 ATP. Estos rendimientos de ATP son valores máximos teóricos; en la práctica, algunos protones se filtran a través de la membrana, lo que reduce el rendimiento de ATP. [7]
Moléculas de transferencia de electrones y protones
La cadena de transporte de electrones transporta tanto protones como electrones, pasando electrones de donantes a aceptores y transportando protones a través de una membrana. Estos procesos utilizan moléculas de transferencia tanto solubles como unidas a proteínas. En las mitocondrias, los electrones se transfieren dentro del espacio intermembrana por la proteína de transferencia de electrones soluble en agua citocromo c . [8] Esta transporta solo electrones, y estos se transfieren por la reducción y oxidación de un átomo de hierro que la proteína mantiene dentro de un grupo hemo en su estructura. El citocromo c también se encuentra en algunas bacterias, donde se encuentra dentro del espacio periplásmico . [9]
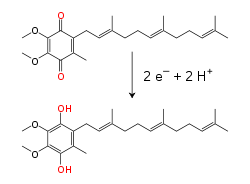
Dentro de la membrana mitocondrial interna, la coenzima Q10 (Q), transportadora de electrones soluble en lípidos, transporta electrones y protones mediante un ciclo redox . [10] Esta pequeña molécula de benzoquinona es muy hidrófoba , por lo que se difunde libremente dentro de la membrana. Cuando Q acepta dos electrones y dos protones, se reduce a la forma ubiquinol (QH 2 ); cuando QH 2 libera dos electrones y dos protones, se oxida de nuevo a la forma ubiquinona (Q). Como resultado, si dos enzimas se organizan de modo que Q se reduzca en un lado de la membrana y QH 2 se oxide en el otro, la ubiquinona acoplará estas reacciones y transportará protones a través de la membrana. [11] Algunas cadenas de transporte de electrones bacterianas utilizan diferentes quinonas, como la menaquinona , además de la ubiquinona. [12]
Dentro de las proteínas, los electrones se transfieren entre cofactores de flavina , [5] [13] cúmulos de hierro-azufre y citocromos. Hay varios tipos de cúmulos de hierro-azufre. El tipo más simple que se encuentra en la cadena de transferencia de electrones consiste en dos átomos de hierro unidos por dos átomos de azufre inorgánico ; estos se denominan cúmulos [2Fe–2S]. El segundo tipo, llamado [4Fe–4S], contiene un cubo de cuatro átomos de hierro y cuatro átomos de azufre. Cada átomo de hierro en estos cúmulos está coordinado por un aminoácido adicional , generalmente por el átomo de azufre de la cisteína . Los cofactores de iones metálicos experimentan reacciones redox sin unirse ni liberar protones, por lo que en la cadena de transporte de electrones sirven únicamente para transportar electrones a través de las proteínas. Los electrones se mueven distancias bastante largas a través de las proteínas saltando a lo largo de las cadenas de estos cofactores. [14] Esto ocurre por efecto túnel cuántico , que es rápido en distancias de menos de 1,4 × 10 −9 m. [15]
Cadenas de transporte de electrones eucariotas
Muchos procesos bioquímicos catabólicos , como la glucólisis , el ciclo del ácido cítrico y la beta oxidación , producen la coenzima reducida NADH . Esta coenzima contiene electrones que tienen un alto potencial de transferencia ; en otras palabras, liberarán una gran cantidad de energía al oxidarse. Sin embargo, la célula no libera esta energía de una sola vez, ya que sería una reacción incontrolable. En cambio, los electrones se eliminan del NADH y pasan al oxígeno a través de una serie de enzimas que liberan cada una una pequeña cantidad de energía. Este conjunto de enzimas, que consta de los complejos I a IV, se llama cadena de transporte de electrones y se encuentra en la membrana interna de la mitocondria . El succinato también se oxida mediante la cadena de transporte de electrones, pero alimenta la vía en un punto diferente.
En los eucariotas , las enzimas de este sistema de transporte de electrones utilizan la energía liberada del O2 por el NADH para bombear protones a través de la membrana interna de la mitocondria. Esto hace que los protones se acumulen en el espacio intermembrana y genera un gradiente electroquímico a través de la membrana. La energía almacenada en este potencial es luego utilizada por la ATP sintasa para producir ATP. La fosforilación oxidativa en la mitocondria eucariota es el ejemplo mejor entendido de este proceso. La mitocondria está presente en casi todos los eucariotas, con la excepción de los protozoos anaeróbicos como Trichomonas vaginalis que, en cambio, reducen los protones a hidrógeno en una mitocondria remanente llamada hidrogenosoma . [16]
| Enzima respiratoria | Par redox | Potencial de punto medio (Voltios) |
|---|---|---|
| NADH deshidrogenasa | NAD + / NADH | -0,32 [17] |
| Succinato deshidrogenasa | FMN o FAD / FMNH 2 o FADH 2 | -0,20 [17] |
| Complejo citocromo bc 1 | Coenzima Q10 buey / Coenzima Q10 roja | +0,06 [17] |
| Complejo citocromo bc 1 | Citocromo b ox / Citocromo b rojo | +0,12 [17] |
| Complejo IV | Citocromo c ox / Citocromo c rojo | +0,22 [17] |
| Complejo IV | Citocromo a buey / Citocromo a rojo | +0,29 [17] |
| Complejo IV | O2 / HO− | +0,82 [17] |
| Condiciones: pH = 7 [17] | ||
NADH-coenzima Q oxidorreductasa (complejo I)
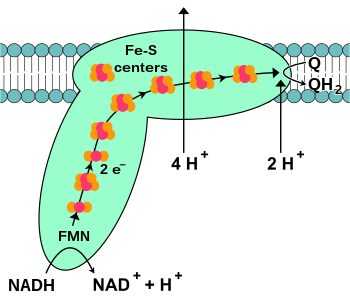
La NADH-coenzima Q oxidorreductasa , también conocida como NADH deshidrogenasa o complejo I , es la primera proteína en la cadena de transporte de electrones. [18] El complejo I es una enzima gigante con el complejo I de mamíferos que tiene 46 subunidades y una masa molecular de aproximadamente 1.000 kilodaltons (kDa). [19] La estructura se conoce en detalle solo de una bacteria; [20] [21] en la mayoría de los organismos el complejo se asemeja a una bota con una gran "bola" que sobresale de la membrana hacia la mitocondria. [22] [23] Los genes que codifican las proteínas individuales están contenidos tanto en el núcleo celular como en el genoma mitocondrial , como es el caso de muchas enzimas presentes en la mitocondria.
La reacción que cataliza esta enzima es la oxidación de dos electrones del NADH por la coenzima Q10 o ubiquinona (representada como Q en la ecuación siguiente), una quinona liposoluble que se encuentra en la membrana mitocondrial:
| ( 1 ) |
El inicio de la reacción, y de hecho de toda la cadena electrónica, es la unión de una molécula de NADH al complejo I y la donación de dos electrones. Los electrones entran al complejo I a través de un grupo prostético unido al complejo, el mononucleótido de flavina (FMN). La adición de electrones al FMN lo convierte en su forma reducida, FMNH 2 . Los electrones luego se transfieren a través de una serie de grupos de hierro-azufre: el segundo tipo de grupo prostético presente en el complejo. [20] Hay grupos de hierro-azufre tanto [2Fe–2S] como [4Fe–4S] en el complejo I.
A medida que los electrones pasan a través de este complejo, se bombean cuatro protones desde la matriz hacia el espacio intermembrana. No está claro exactamente cómo ocurre esto, pero parece implicar cambios conformacionales en el complejo I que hacen que la proteína se una a los protones en el lado N de la membrana y los libere en el lado P de la membrana. [24] Finalmente, los electrones se transfieren desde la cadena de grupos de hierro-azufre a una molécula de ubiquinona en la membrana. [18] La reducción de la ubiquinona también contribuye a la generación de un gradiente de protones, ya que se toman dos protones de la matriz a medida que se reduce a ubiquinol (QH 2 ).
Succinato-Q oxidorreductasa (complejo II)

La succinato-Q oxidorreductasa , también conocida como complejo II o succinato deshidrogenasa , es un segundo punto de entrada a la cadena de transporte de electrones. [25] Es inusual porque es la única enzima que forma parte tanto del ciclo del ácido cítrico como de la cadena de transporte de electrones. El complejo II consta de cuatro subunidades proteicas y contiene un cofactor de dinucleótido de flavina y adenina (FAD) unido, grupos de hierro-azufre y un grupo hemo que no participa en la transferencia de electrones a la coenzima Q, pero se cree que es importante para disminuir la producción de especies reactivas de oxígeno. [26] [27] Oxida el succinato a fumarato y reduce la ubiquinona. Como esta reacción libera menos energía que la oxidación de NADH, el complejo II no transporta protones a través de la membrana y no contribuye al gradiente de protones.
| ( 2 ) |
En algunos eucariotas, como el gusano parásito Ascaris suum , una enzima similar al complejo II, la fumarato reductasa (menaquinol: fumarato oxidorreductasa o QFR), opera a la inversa para oxidar el ubiquinol y reducir el fumarato. Esto permite que el gusano sobreviva en el ambiente anaeróbico del intestino grueso , llevando a cabo la fosforilación oxidativa anaeróbica con el fumarato como aceptor de electrones. [28] Otra función no convencional del complejo II se observa en el parásito de la malaria Plasmodium falciparum . Aquí, la acción inversa del complejo II como oxidasa es importante para regenerar el ubiquinol, que el parásito utiliza en una forma inusual de biosíntesis de pirimidina . [29]
Oxidorreductasa de flavoproteína-Q de transferencia de electrones
La flavoproteína-ubiquinona oxidorreductasa de transferencia de electrones (ETF-Q oxidorreductasa), también conocida como flavoproteína deshidrogenasa de transferencia de electrones , es un tercer punto de entrada a la cadena de transporte de electrones. Es una enzima que acepta electrones de la flavoproteína de transferencia de electrones en la matriz mitocondrial y utiliza estos electrones para reducir la ubiquinona. [30] Esta enzima contiene una flavina y un grupo [4Fe–4S], pero, a diferencia de los otros complejos respiratorios, se adhiere a la superficie de la membrana y no atraviesa la bicapa lipídica. [31]
| ( 3 ) |
En los mamíferos, esta vía metabólica es importante en la beta oxidación de los ácidos grasos y el catabolismo de los aminoácidos y la colina , ya que acepta electrones de múltiples acetil-CoA deshidrogenasas. [32] [33] En las plantas, la oxidorreductasa ETF-Q también es importante en las respuestas metabólicas que permiten la supervivencia en períodos prolongados de oscuridad. [34]
Q-citocromo c oxidorreductasa (complejo III)

La Q-citocromo c oxidorreductasa también se conoce como citocromo c reductasa , complejo citocromo bc 1 o simplemente complejo III . [35] [36] En los mamíferos, esta enzima es un dímero , y cada complejo de subunidades contiene 11 subunidades proteicas, un grupo hierro-azufre [2Fe-2S] y tres citocromos : un citocromo c 1 y dos citocromos b . [37] Un citocromo es un tipo de proteína de transferencia de electrones que contiene al menos un grupo hemo . Los átomos de hierro dentro de los grupos hemo del complejo III alternan entre un estado ferroso reducido (+2) y férrico oxidado (+3) a medida que los electrones se transfieren a través de la proteína.
La reacción catalizada por el complejo III es la oxidación de una molécula de ubiquinol y la reducción de dos moléculas de citocromo c , una proteína hemo asociada de forma vaga a la mitocondria. A diferencia de la coenzima Q, que lleva dos electrones, el citocromo c lleva sólo un electrón.
| ( 4 ) |
Como sólo uno de los electrones puede ser transferido desde el donante QH 2 a un aceptor de citocromo c a la vez, el mecanismo de reacción del complejo III es más elaborado que los de los otros complejos respiratorios, y ocurre en dos pasos llamados ciclo Q. [38] En el primer paso, la enzima se une a tres sustratos, primero, QH 2 , que luego se oxida, pasando un electrón al segundo sustrato, el citocromo c. Los dos protones liberados de QH 2 pasan al espacio intermembrana. El tercer sustrato es Q, que acepta el segundo electrón del QH 2 y se reduce a Q .− , que es el radical libre ubisemiquinona . Los primeros dos sustratos se liberan, pero este intermediario ubisemiquinona permanece unido. En el segundo paso, una segunda molécula de QH 2 se une y nuevamente pasa su primer electrón a un aceptor de citocromo c. El segundo electrón pasa a la ubisemiquinona unida, reduciéndola a QH 2 al ganar dos protones de la matriz mitocondrial. Luego, este QH 2 se libera de la enzima. [39]
A medida que la coenzima Q se reduce a ubiquinol en el lado interno de la membrana y se oxida a ubiquinona en el otro, se produce una transferencia neta de protones a través de la membrana, lo que aumenta el gradiente de protones. [5] El mecanismo de dos pasos, bastante complejo, por el que esto ocurre es importante, ya que aumenta la eficiencia de la transferencia de protones. Si, en lugar del ciclo Q, se utilizara una molécula de QH 2 para reducir directamente dos moléculas de citocromo c, la eficiencia se reduciría a la mitad, y solo se transferiría un protón por cada citocromo c reducido. [5]
Citocromo c oxidasa (complejo IV)

La citocromo c oxidasa , también conocida como complejo IV , es el complejo proteico final en la cadena de transporte de electrones. [40] La enzima de los mamíferos tiene una estructura extremadamente complicada y contiene 13 subunidades, dos grupos hemo, así como múltiples cofactores de iones metálicos: en total, tres átomos de cobre , uno de magnesio y uno de zinc . [41]
Esta enzima media la reacción final en la cadena de transporte de electrones y transfiere electrones al oxígeno y al hidrógeno (protones), mientras bombea protones a través de la membrana. [42] El oxígeno, el último aceptor de electrones , se reduce a agua en este paso. Tanto el bombeo directo de protones como el consumo de protones de la matriz en la reducción del oxígeno contribuyen al gradiente de protones. La reacción catalizada es la oxidación del citocromo c y la reducción del oxígeno:
| ( 5 ) |
Reductasas y oxidasas alternativas
Muchos organismos eucariotas tienen cadenas de transporte de electrones que difieren de las enzimas de mamíferos descritas anteriormente, que han sido muy estudiadas. Por ejemplo, las plantas tienen oxidasas de NADH alternativas, que oxidan el NADH en el citosol en lugar de en la matriz mitocondrial, y pasan estos electrones al depósito de ubiquinona. [43] Estas enzimas no transportan protones y, por lo tanto, reducen la ubiquinona sin alterar el gradiente electroquímico a través de la membrana interna. [44]
Otro ejemplo de una cadena de transporte de electrones divergente es la oxidasa alternativa , que se encuentra en las plantas , así como en algunos hongos , protistas y posiblemente algunos animales. [45] [46] Esta enzima transfiere electrones directamente del ubiquinol al oxígeno. [47]
Las vías de transporte de electrones producidas por estas oxidasas alternativas de NADH y ubiquinona tienen rendimientos de ATP más bajos que la vía completa. Las ventajas producidas por una vía acortada no están del todo claras. Sin embargo, la oxidasa alternativa se produce en respuesta a estreses como el frío, las especies reactivas de oxígeno y la infección por patógenos, así como otros factores que inhiben la cadena de transporte de electrones completa. [48] [49] Por lo tanto, las vías alternativas podrían mejorar la resistencia de un organismo a las lesiones, al reducir el estrés oxidativo . [50]
Organización de complejos
El modelo original de cómo se organizan los complejos de la cadena respiratoria era que se difundían libre e independientemente en la membrana mitocondrial. [51] Sin embargo, datos recientes sugieren que los complejos podrían formar estructuras de orden superior llamadas supercomplejos o " respirasomas ". [52] En este modelo, los diversos complejos existen como conjuntos organizados de enzimas interactuantes. [53] Estas asociaciones podrían permitir la canalización de sustratos entre los diversos complejos enzimáticos, aumentando la velocidad y la eficiencia de la transferencia de electrones. [54] Dentro de estos supercomplejos mamíferos, algunos componentes estarían presentes en mayores cantidades que otros, con algunos datos que sugieren una relación entre los complejos I/II/III/IV y la ATP sintasa de aproximadamente 1:1:3:7:4. [55] Sin embargo, el debate sobre esta hipótesis del supercomplejo no está completamente resuelto, ya que algunos datos no parecen encajar con este modelo. [19] [56]
Cadenas de transporte de electrones procariotas
En contraste con la similitud general en la estructura y función de las cadenas de transporte de electrones en eucariotas, las bacterias y las arqueas poseen una gran variedad de enzimas de transferencia de electrones. Estas utilizan un conjunto igualmente amplio de sustancias químicas como sustratos. [57] Al igual que en eucariotas, el transporte de electrones en procariotas utiliza la energía liberada por la oxidación de un sustrato para bombear iones a través de una membrana y generar un gradiente electroquímico. En las bacterias, la fosforilación oxidativa en Escherichia coli se conoce con mayor detalle, mientras que los sistemas de las arqueas son actualmente poco conocidos. [58]
La principal diferencia entre la fosforilación oxidativa eucariota y procariota es que las bacterias y las arqueas utilizan muchas sustancias diferentes para donar o aceptar electrones. Esto permite que los procariotas crezcan en una amplia variedad de condiciones ambientales. [59] En E. coli , por ejemplo, la fosforilación oxidativa puede ser impulsada por una gran cantidad de pares de agentes reductores y agentes oxidantes, que se enumeran a continuación. El potencial de punto medio de una sustancia química mide cuánta energía se libera cuando se oxida o se reduce, y los agentes reductores tienen potenciales negativos y los agentes oxidantes, potenciales positivos.
| Enzima respiratoria | Par redox | Potencial de punto medio (Voltios) |
|---|---|---|
| Formato deshidrogenasa | Bicarbonato / Formato | -0,43 |
| Hidrogenasa | Protón / Hidrógeno | -0,42 |
| NADH deshidrogenasa | NAD + / NADH | -0,32 |
| Glicerol-3-fosfato deshidrogenasa | DHAP / Gly-3-P | -0,19 |
| Piruvato oxidasa | Acetato + dióxido de carbono / piruvato | ? |
| Lactato deshidrogenasa | Piruvato / Lactato | -0,19 |
| D- aminoácido deshidrogenasa | 2-oxoácido + amoniaco / D -aminoácido | ? |
| Glucosa deshidrogenasa | Gluconato / glucosa | -0,14 |
| Succinato deshidrogenasa | Fumarato / Succinato | +0,03 |
| Ubiquinol oxidasa | Oxígeno / Agua | +0,82 |
| Nitrato reductasa | Nitrato / Nitrito | +0,42 |
| Nitrito reductasa | Nitrito / Amoniaco | +0,36 |
| Dimetilsulfóxido reductasa | DMSO / DMS | +0,16 |
| Reductasa de N -óxido de trimetilamina | TMAO / TMA | +0,13 |
| Fumarato reductasa | Fumarato / Succinato | +0,03 |
Como se muestra arriba, E. coli puede crecer con agentes reductores como formiato, hidrógeno o lactato como donantes de electrones, y nitrato, DMSO u oxígeno como aceptores. [59] Cuanto mayor sea la diferencia en el potencial de punto medio entre un agente oxidante y reductor, más energía se libera cuando reaccionan. De estos compuestos, el par succinato/fumarato es inusual, ya que su potencial de punto medio es cercano a cero. Por lo tanto, el succinato se puede oxidar a fumarato si está disponible un agente oxidante fuerte como el oxígeno, o el fumarato se puede reducir a succinato usando un agente reductor fuerte como el formiato. Estas reacciones alternativas son catalizadas por la succinato deshidrogenasa y la fumarato reductasa , respectivamente. [61]
Algunos procariotas utilizan pares redox que tienen solo una pequeña diferencia en el potencial de punto medio. Por ejemplo, las bacterias nitrificantes como Nitrobacter oxidan el nitrito a nitrato, donando los electrones al oxígeno. La pequeña cantidad de energía liberada en esta reacción es suficiente para bombear protones y generar ATP, pero no suficiente para producir NADH o NADPH directamente para su uso en el anabolismo . [62] Este problema se resuelve utilizando una nitrito oxidorreductasa para producir suficiente fuerza protomomotriz para ejecutar parte de la cadena de transporte de electrones en sentido inverso, lo que hace que el complejo I genere NADH. [63] [64]
Los procariotas controlan el uso de estos donantes y aceptores de electrones al variar las enzimas que se producen, en respuesta a las condiciones ambientales. [65] Esta flexibilidad es posible porque diferentes oxidasas y reductasas utilizan el mismo grupo de ubiquinona. Esto permite que muchas combinaciones de enzimas funcionen juntas, unidas por el intermediario común ubiquinol. [60] Por lo tanto, estas cadenas respiratorias tienen un diseño modular , con conjuntos de sistemas enzimáticos fácilmente intercambiables.
Además de esta diversidad metabólica, los procariotas también poseen una gama de isoenzimas , diferentes enzimas que catalizan la misma reacción. Por ejemplo, en E. coli , hay dos tipos diferentes de ubiquinol oxidasa que utilizan el oxígeno como aceptor de electrones. En condiciones altamente aeróbicas, la célula utiliza una oxidasa con baja afinidad por el oxígeno que puede transportar dos protones por electrón. Sin embargo, si los niveles de oxígeno caen, cambian a una oxidasa que transfiere solo un protón por electrón, pero tiene una alta afinidad por el oxígeno. [66]
ATP sintasa (complejo V)
La ATP sintasa, también llamada complejo V , es la enzima final en la vía de la fosforilación oxidativa. Esta enzima se encuentra en todas las formas de vida y funciona de la misma manera tanto en procariotas como en eucariotas. [67] La enzima utiliza la energía almacenada en un gradiente de protones a través de una membrana para impulsar la síntesis de ATP a partir de ADP y fosfato (P i ). Las estimaciones de la cantidad de protones necesarios para sintetizar un ATP han oscilado entre tres y cuatro, [68] [69] y algunos sugieren que las células pueden variar esta proporción para adaptarse a diferentes condiciones. [70]
| ( 6 ) |
Esta reacción de fosforilación es un equilibrio que se puede alterar modificando la fuerza protón-motriz. En ausencia de una fuerza protón-motriz, la reacción de la ATP sintasa se desarrollará de derecha a izquierda, hidrolizando el ATP y bombeando protones fuera de la matriz a través de la membrana. Sin embargo, cuando la fuerza protón-motriz es alta, la reacción se ve obligada a desarrollarse en la dirección opuesta; procede de izquierda a derecha, permitiendo que los protones fluyan a favor de su gradiente de concentración y convirtiendo el ADP en ATP. [67] De hecho, en las H+-ATPasas de tipo vacuolar estrechamente relacionadas , la reacción de hidrólisis se utiliza para acidificar los compartimentos celulares, bombeando protones e hidrolizando el ATP. [71]
La ATP sintasa es un complejo proteico masivo con forma de hongo. El complejo enzimático de los mamíferos contiene 16 subunidades y tiene una masa de aproximadamente 600 kilodaltons . [72] La porción incrustada dentro de la membrana se llama F O y contiene un anillo de subunidades c y el canal de protones. El tallo y la cabeza esférica se llama F 1 y es el sitio de la síntesis de ATP. El complejo esférica al final de la porción F 1 contiene seis proteínas de dos tipos diferentes (tres subunidades α y tres subunidades β), mientras que el "tallo" consta de una proteína: la subunidad γ, con la punta del tallo extendiéndose hacia la bola de subunidades α y β. [73] Tanto las subunidades α como las β unen nucleótidos, pero solo las subunidades β catalizan la reacción de síntesis de ATP. A lo largo del costado de la porción F 1 y de regreso a la membrana se encuentra una subunidad larga con forma de varilla que ancla las subunidades α y β a la base de la enzima.
A medida que los protones cruzan la membrana a través del canal en la base de la ATP sintasa, el motor impulsado por protones F O gira. [74] La rotación puede ser causada por cambios en la ionización de los aminoácidos en el anillo de subunidades c que causan interacciones electrostáticas que impulsan el anillo de subunidades c más allá del canal de protones. [75] Este anillo giratorio a su vez impulsa la rotación del eje central (el tallo de la subunidad γ) dentro de las subunidades α y β. Las subunidades α y β no pueden girar por sí mismas mediante el brazo lateral, que actúa como un estator . Este movimiento de la punta de la subunidad γ dentro de la bola de subunidades α y β proporciona la energía para que los sitios activos en las subunidades β experimenten un ciclo de movimientos que produce y luego libera ATP. [76]

Esta reacción de síntesis de ATP se denomina mecanismo de cambio de enlace e implica que el sitio activo de una subunidad β pase por tres estados. [77] En el estado "abierto", el ADP y el fosfato entran en el sitio activo (que se muestra en marrón en el diagrama). A continuación, la proteína se cierra alrededor de las moléculas y las une de forma laxa: el estado "suelto" (que se muestra en rojo). A continuación, la enzima cambia de forma de nuevo y fuerza a estas moléculas a unirse, con el sitio activo en el estado "apretado" resultante (que se muestra en rosa) uniendo la molécula de ATP recién producida con una afinidad muy alta . Finalmente, el sitio activo vuelve al estado abierto, liberando ATP y uniendo más ADP y fosfato, listo para el siguiente ciclo.
En algunas bacterias y arqueas, la síntesis de ATP está impulsada por el movimiento de iones de sodio a través de la membrana celular, en lugar del movimiento de protones. [78] [79] Las arqueas como Methanococcus también contienen la A 1 A o sintasa, una forma de la enzima que contiene proteínas adicionales con poca similitud en secuencia con otras subunidades de ATP sintasa bacterianas y eucariotas. Es posible que, en algunas especies, la forma A 1 A o de la enzima sea una ATP sintasa especializada impulsada por sodio, [80] pero esto podría no ser cierto en todos los casos. [79]
Fosforilación oxidativa – energética
El transporte de electrones desde el par redox NAD + /NADH al par redox final 1/2 O2 / H2O se puede resumir como
1/2 O 2 + NADH + H + → H 2 O + NAD +
La diferencia de potencial entre estos dos pares redox es de 1,14 voltios, lo que equivale a -52 kcal/mol o -2600 kJ por 6 moles de O 2 .
Cuando un NADH se oxida a través de la cadena de transferencia de electrones, se producen tres ATP, lo que equivale a 7,3 kcal/mol x 3 = 21,9 kcal/mol.
La conservación de la energía se puede calcular mediante la siguiente fórmula
Eficiencia = (21,9 x 100%) / 52 = 42%
Así que podemos concluir que cuando el NADH se oxida, alrededor del 42% de la energía se conserva en forma de tres ATP y la energía restante (58%) se pierde en forma de calor (a menos que se haya subestimado la energía química del ATP en condiciones fisiológicas).
Especies reactivas de oxígeno
El oxígeno molecular es un buen aceptor terminal de electrones porque es un agente oxidante fuerte. La reducción del oxígeno implica intermediarios potencialmente dañinos. [81] Aunque la transferencia de cuatro electrones y cuatro protones reduce el oxígeno a agua, que es inofensiva, la transferencia de uno o dos electrones produce aniones superóxido o peróxido , que son peligrosamente reactivos.
| ( 7 ) |
Estas especies reactivas de oxígeno y sus productos de reacción, como el radical hidroxilo , son muy perjudiciales para las células, ya que oxidan las proteínas y provocan mutaciones en el ADN . Este daño celular puede contribuir a la aparición de enfermedades y se propone como una de las causas del envejecimiento . [82] [83]
El complejo citocromo c oxidasa es muy eficiente en la reducción del oxígeno a agua y libera muy pocos intermediarios parcialmente reducidos; sin embargo, la cadena de transporte de electrones produce pequeñas cantidades de anión superóxido y peróxido. [84] Particularmente importante es la reducción de la coenzima Q en el complejo III, ya que se forma un radical libre ubisemiquinona altamente reactivo como intermediario en el ciclo Q. Esta especie inestable puede provocar una "fuga" de electrones cuando los electrones se transfieren directamente al oxígeno, formando superóxido. [85] Como la producción de especies reactivas de oxígeno por estos complejos de bombeo de protones es mayor a potenciales de membrana altos, se ha propuesto que las mitocondrias regulan su actividad para mantener el potencial de membrana dentro de un rango estrecho que equilibra la producción de ATP frente a la generación de oxidantes. [86] Por ejemplo, los oxidantes pueden activar proteínas desacopladoras que reducen el potencial de membrana. [87]
Para contrarrestar estas especies reactivas de oxígeno, las células contienen numerosos sistemas antioxidantes , incluidas vitaminas antioxidantes como la vitamina C y la vitamina E , y enzimas antioxidantes como la superóxido dismutasa , la catalasa y las peroxidasas , [81] que desintoxican las especies reactivas, limitando el daño a la célula.
Fosforilación oxidativa en condiciones hipóxicas/anóxicas
Como el oxígeno es fundamental para la fosforilación oxidativa, una escasez en el nivel de O2 puede alterar las tasas de producción de ATP. En condiciones anóxicas, la ATP-sintasa cometerá una "traición celular" y funcionará en sentido inverso, forzando a los protones de la matriz a regresar al espacio de la membrana interna, consumiendo ATP en el proceso. [88] La fuerza motriz de protones y la producción de ATP se pueden mantener mediante la acidosis intracelular. [89] Los protones citosólicos que se han acumulado con la hidrólisis de ATP y la acidosis láctica pueden difundirse libremente a través de la membrana externa mitocondrial y acidificar el espacio intermembrana, contribuyendo así directamente a la fuerza motriz de protones y la producción de ATP.
Inhibidores
Existen varios fármacos y toxinas bien conocidos que inhiben la fosforilación oxidativa. Aunque cualquiera de estas toxinas inhibe sólo una enzima en la cadena de transporte de electrones, la inhibición de cualquier paso de este proceso detendrá el resto del proceso. Por ejemplo, si la oligomicina inhibe la ATP sintasa, los protones no pueden volver a pasar a la mitocondria. [90] Como resultado, las bombas de protones no pueden funcionar, ya que el gradiente se vuelve demasiado fuerte para que lo superen. El NADH ya no se oxida y el ciclo del ácido cítrico deja de funcionar porque la concentración de NAD + cae por debajo de la concentración que estas enzimas pueden utilizar.
Muchos inhibidores específicos de la cadena de transporte de electrones han contribuido al conocimiento actual de la respiración mitocondrial. La síntesis de ATP también depende de la cadena de transporte de electrones, por lo que todos los inhibidores específicos de la cadena también inhiben la formación de ATP. El veneno para peces rotenona , el fármaco barbitúrico amital y el antibiótico piericidina A inhiben el NADH y la coenzima Q. [91]
El monóxido de carbono, el cianuro, el sulfuro de hidrógeno y la azida inhiben eficazmente la citocromo oxidasa. El monóxido de carbono reacciona con la forma reducida del citocromo, mientras que el cianuro y la azida reaccionan con la forma oxidada. Un antibiótico, la antimicina A , y el anti-Lewisita británico , un antídoto utilizado contra las armas químicas, son los dos inhibidores importantes del sitio entre el citocromo B y C1. [91]
| Compuestos | Usar | Sitio de acción | Efecto sobre la fosforilación oxidativa |
|---|---|---|---|
| Cianuro Monóxido de carbono Azida Sulfuro de hidrógeno | Venenos | Complejo IV | Inhibe la cadena de transporte de electrones uniéndose más fuertemente que el oxígeno al centro Fe – Cu en la citocromo c oxidasa, previniendo la reducción del oxígeno. [92] |
| Oligomicina | Antibiótico | Complejo V | Inhibe la ATP sintasa al bloquear el flujo de protones a través de la subunidad F o . [90] |
| CCCP 2,4-Dinitrofenol | Venenos para adelgazar [N 1] | Membrana interna | Ionóforos que alteran el gradiente de protones al transportarlos a través de una membrana. Este ionóforo desacopla el bombeo de protones de la síntesis de ATP porque transporta protones a través de la membrana mitocondrial interna. [93] |
| Rotenona | Pesticida | Complejo I | Previene la transferencia de electrones del complejo I a la ubiquinona al bloquear el sitio de unión de la ubiquinona. [94] |
| Malonato y oxaloacetato | Venenos | Complejo II | Inhibidores competitivos de la succinato deshidrogenasa (complejo II). [95] |
| Antimicina A | Piscicida | Complejo III | Se une al sitio Qi de la citocromo c reductasa , inhibiendo así la oxidación del ubiquinol . |
No todos los inhibidores de la fosforilación oxidativa son toxinas. En el tejido adiposo pardo , los canales de protones regulados, llamados proteínas desacopladoras, pueden desacoplar la respiración de la síntesis de ATP. [96] Esta respiración rápida produce calor y es particularmente importante como forma de mantener la temperatura corporal de los animales que hibernan , aunque estas proteínas también pueden tener una función más general en las respuestas de las células al estrés. [97]
Historia
El campo de la fosforilación oxidativa comenzó con el informe en 1906 de Arthur Harden sobre un papel vital del fosfato en la fermentación celular , pero inicialmente solo se sabía que los fosfatos de azúcar estaban involucrados. [98] Sin embargo, a principios de la década de 1940, el vínculo entre la oxidación de azúcares y la generación de ATP fue firmemente establecido por Herman Kalckar , [99] confirmando el papel central del ATP en la transferencia de energía que había sido propuesto por Fritz Albert Lipmann en 1941. [100] Más tarde, en 1949, Morris Friedkin y Albert L. Lehninger demostraron que la coenzima NADH vinculaba vías metabólicas como el ciclo del ácido cítrico y la síntesis de ATP. [101] El término fosforilación oxidativa fue acuñado por Volodymyr Belitser en 1939. [102] [103]
Durante otros veinte años, el mecanismo por el cual se genera el ATP siguió siendo un misterio, con científicos buscando un esquivo "intermediario de alta energía" que vinculara las reacciones de oxidación y fosforilación. [104] Este rompecabezas fue resuelto por Peter D. Mitchell con la publicación de la teoría quimiosmótica en 1961. [105] Al principio, esta propuesta fue muy controvertida, pero poco a poco fue aceptada y Mitchell recibió el premio Nobel en 1978. [106] [107] La investigación posterior se concentró en purificar y caracterizar las enzimas involucradas, con importantes contribuciones de David E. Green sobre los complejos de la cadena de transporte de electrones, así como de Efraim Racker sobre la ATP sintasa. [108] Un paso crítico hacia la solución del mecanismo de la ATP sintasa fue proporcionado por Paul D. Boyer , con su desarrollo en 1973 del mecanismo de "cambio de enlace", seguido por su propuesta radical de catálisis rotacional en 1982. [77] [109] Trabajos más recientes han incluido estudios estructurales sobre las enzimas involucradas en la fosforilación oxidativa por John E. Walker , con Walker y Boyer recibiendo un Premio Nobel en 1997. [110]
Véase también
Notas
- ^ El DNP se utilizó ampliamente como medicamento contra la obesidad en la década de 1930, pero finalmente se dejó de utilizar debido a sus peligrosos efectos secundarios. Sin embargo, el uso ilícito del fármaco con este fin continúa en la actualidad. Consulte 2,4-Dinitrofenol#Ayuda para la dieta para obtener más información.
Referencias
- ^ "oxidativo Significado en el Diccionario Cambridge Inglés". dictionary.cambridge.org . Archivado desde el original el 24 de enero de 2018 . Consultado el 28 de abril de 2018 .
- ^ Voet, D.; Voet, JG (2004). "Bioquímica", 3ª ed., pág. 804, Wiley. ISBN 0-471-19350-X.
- ^ Mitchell P, Moyle J (enero de 1967). "Hipótesis quimiosmótica de la fosforilación oxidativa". Nature . 213 (5072): 137–139. Bibcode :1967Natur.213..137M. doi :10.1038/213137a0. PMID 4291593. S2CID 4149605.
- ^ ab Dimroth P, Kaim G, Matthey U (enero de 2000). "Función crucial del potencial de membrana para la síntesis de ATP por las sintasas de ATP F(1)F(o)". The Journal of Experimental Biology . 203 (Pt 1): 51–59. doi :10.1242/jeb.203.1.51. PMID 10600673. Archivado desde el original el 30 de septiembre de 2007.
- ^ abcd Schultz BE, Chan SI (2001). "Estructuras y estrategias de bombeo de protones de las enzimas respiratorias mitocondriales". Revisión anual de biofísica y estructura biomolecular . 30 : 23–65. doi :10.1146/annurev.biophys.30.1.23. PMID 11340051.
- ^ Rich PR (diciembre de 2003). "La maquinaria molecular de la cadena respiratoria de Keilin". Biochemical Society Transactions . 31 (Pt 6): 1095–1105. doi :10.1042/bst0311095. PMID 14641005.
- ^ Porter RK, Brand MD (septiembre de 1995). "La conductancia protónica mitocondrial y la relación H+/O son independientes de la tasa de transporte de electrones en hepatocitos aislados". The Biochemical Journal . 310 (Pt 2) (Pt 2): 379–382. doi :10.1042/bj3100379. PMC 1135905 . PMID 7654171.
- ^ Mathews FS (1985). "La estructura, función y evolución de los citocromos". Progreso en biofísica y biología molecular . 45 (1): 1–56. doi : 10.1016/0079-6107(85)90004-5 . PMID: 3881803.
- ^ Wood PM (diciembre de 1983). "¿Por qué existen los citocromos de tipo C?". FEBS Letters . 164 (2): 223–226. Bibcode :1983FEBSL.164..223W. doi : 10.1016/0014-5793(83)80289-0 . PMID 6317447. S2CID 7685958.
- ^ Crane FL (diciembre de 2001). "Funciones bioquímicas de la coenzima Q10". Revista del Colegio Americano de Nutrición . 20 (6): 591–598. doi :10.1080/07315724.2001.10719063. PMID 11771674. S2CID 28013583.
- ^ Mitchell P (diciembre de 1979). "El concepto de cadena respiratoria de Keilin y sus consecuencias quimiosmóticas". Science . 206 (4423): 1148–1159. Bibcode :1979Sci...206.1148M. doi :10.1126/science.388618. PMID 388618.
- ^ Søballe B, Poole RK (agosto de 1999). "Ubiquinonas microbianas: múltiples funciones en la respiración, la regulación genética y la gestión del estrés oxidativo" (PDF) . Microbiología . 145 (Pt 8) (8): 1817–1830. doi : 10.1099/13500872-145-8-1817 . PMID 10463148. Archivado (PDF) desde el original el 29 de mayo de 2008.
- ^ Johnson DC, Dean DR, Smith AD, Johnson MK (2005). "Estructura, función y formación de cúmulos biológicos de hierro y azufre". Revisión anual de bioquímica . 74 : 247–281. doi :10.1146/annurev.biochem.74.082803.133518. PMID 15952888.
- ^ Page CC, Moser CC, Chen X, Dutton PL (noviembre de 1999). "Principios de ingeniería natural del efecto túnel de electrones en la oxidación-reducción biológica". Nature . 402 (6757): 47–52. Bibcode :1999Natur.402...47P. doi :10.1038/46972. PMID 10573417. S2CID 4431405.
- ^ Leys D, Scrutton NS (diciembre de 2004). "Circuitos eléctricos en biología: principios emergentes de la estructura de proteínas". Current Opinion in Structural Biology . 14 (6): 642–647. doi :10.1016/j.sbi.2004.10.002. PMID 15582386.
- ^ Boxma B, de Graaf RM, van der Staay GW, van Alen TA, Ricard G, Gabaldón T, et al. (marzo de 2005). "Una mitocondria anaeróbica que produce hidrógeno". Nature . 434 (7029): 74–79. Bibcode :2005Natur.434...74B. doi :10.1038/nature03343. PMID 15744302. S2CID 4401178.
- ^ Compendio de QUÍMICA Médica abcdefgh . Por Anders Overgaard Pedersen y Henning Nielsen. Universidad de Aarhus. 2008
- ^ ab Hirst J (junio de 2005). "Transducción de energía por el complejo respiratorio I: una evaluación del conocimiento actual". Biochemical Society Transactions . 33 (Pt 3): 525–529. doi :10.1042/BST0330525. PMID 15916556.
- ^ ab Lenaz G, Fato R, Genova ML, Bergamini C, Bianchi C, Biondi A (2006). "Complejo mitocondrial I: aspectos estructurales y funcionales". Biochimica et Biophysica Acta (BBA) - Bioenergética . 1757 (9–10): 1406–1420. doi : 10.1016/j.bbabio.2006.05.007 . PMID 16828051.
- ^ ab Sazanov LA, Hinchliffe P (marzo de 2006). "Estructura del dominio hidrófilo del complejo respiratorio I de Thermus thermophilus". Science . 311 (5766): 1430–1436. Bibcode :2006Sci...311.1430S. doi : 10.1126/science.1123809 . PMID 16469879. S2CID 1892332.
- ^ Efremov RG, Baradaran R, Sazanov LA (mayo de 2010). "La arquitectura del complejo respiratorio I". Nature . 465 (7297): 441–445. Bibcode :2010Natur.465..441E. doi :10.1038/nature09066. PMID 20505720. S2CID 4372778.
- ^ Baranova EA, Holt PJ, Sazanov LA (febrero de 2007). "Estructura de proyección del dominio de membrana del complejo respiratorio I de Escherichia coli con una resolución de 8 A". Journal of Molecular Biology . 366 (1): 140–154. doi :10.1016/j.jmb.2006.11.026. PMID 17157874.
- ^ Friedrich T, Böttcher B (enero de 2004). "La estructura macroscópica del complejo respiratorio I: un sistema Lego". Biochimica et Biophysica Acta (BBA) - Bioenergética . 1608 (1): 1–9. doi : 10.1016/j.bbabio.2003.10.002 . PMID 14741580.
- ^ Hirst J (diciembre de 2009). "Hacia el mecanismo molecular del complejo respiratorio I". The Biochemical Journal . 425 (2): 327–339. doi :10.1042/BJ20091382. PMID 20025615.
- ^ Cecchini G (2003). "Función y estructura del complejo II de la cadena respiratoria". Revista Anual de Bioquímica . 72 : 77–109. doi :10.1146/annurev.biochem.72.121801.161700. PMID 14527321.
- ^ Yankovskaya V, Horsefield R, Törnroth S, Luna-Chavez C, Miyoshi H, Léger C, et al. (enero de 2003). "Arquitectura de la succinato deshidrogenasa y generación de especies reactivas de oxígeno". Science . 299 (5607): 700–704. Bibcode :2003Sci...299..700Y. doi :10.1126/science.1079605. PMID 12560550. S2CID 29222766.
- ^ Horsefield R, Iwata S, Byrne B (abril de 2004). "Complejo II desde una perspectiva estructural". Current Protein & Peptide Science . 5 (2): 107–118. doi :10.2174/1389203043486847. PMID 15078221.
- ^ Kita K, Hirawake H, Miyadera H, Amino H, Takeo S (enero de 2002). "Papel del complejo II en la respiración anaeróbica de las mitocondrias del parásito de Ascaris suum y Plasmodium falciparum". Biochimica et Biophysica Acta (BBA) - Bioenergética . 1553 (1–2): 123–139. doi : 10.1016/S0005-2728(01)00237-7 . PMID 11803022.
- ^ Painter HJ, Morrisey JM, Mather MW, Vaidya AB (marzo de 2007). "Función específica del transporte de electrones mitocondriales en el estadio sanguíneo de Plasmodium falciparum". Nature . 446 (7131): 88–91. Bibcode :2007Natur.446...88P. doi :10.1038/nature05572. PMID 17330044. S2CID 4421676.
- ^ Ramsay RR, Steenkamp DJ, Husain M (febrero de 1987). "Reacciones de la flavoproteína de transferencia de electrones y la flavoproteína de transferencia de electrones: ubiquinona oxidorreductasa". The Biochemical Journal . 241 (3): 883–892. doi :10.1042/bj2410883. PMC 1147643 . PMID 3593226.
- ^ Zhang J, Frerman FE, Kim JJ (octubre de 2006). "Estructura de la flavoproteína-ubiquinona oxidorreductasa de transferencia de electrones y transferencia de electrones al grupo de ubiquinona mitocondrial". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (44): 16212–16217. Bibcode :2006PNAS..10316212Z. doi : 10.1073/pnas.0604567103 . PMC 1637562 . PMID 17050691.
- ^ Ikeda Y, Dabrowski C, Tanaka K (enero de 1983). "Separación y propiedades de cinco acil-CoA deshidrogenasas distintas de mitocondrias de hígado de rata. Identificación de una nueva acil-CoA deshidrogenasa de cadena ramificada de 2-metilos". The Journal of Biological Chemistry . 258 (2): 1066–1076. doi : 10.1016/S0021-9258(18)33160-0 . PMID 6401712. Archivado desde el original el 29 de septiembre de 2007.
- ^ Ruzicka FJ, Beinert H (diciembre de 1977). "Una nueva flavoproteína de hierro y azufre de la cadena respiratoria. Un componente de la vía de la beta oxidación de ácidos grasos" (PDF) . The Journal of Biological Chemistry . 252 (23): 8440–8445. doi : 10.1016/S0021-9258(19)75238-7 . PMID 925004. Archivado (PDF) desde el original el 27 de septiembre de 2007.
- ^ Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ (septiembre de 2005). "El papel crítico de la flavoproteína de transferencia de electrones: ubiquinona oxidorreductasa de Arabidopsis durante la inanición inducida por la oscuridad". The Plant Cell . 17 (9): 2587–2600. doi :10.1105/tpc.105.035162. PMC 1197437 . PMID 16055629.
- ^ Berry EA, Guergova-Kuras M, Huang LS, Crofts AR (2000). "Estructura y función de los complejos del citocromo bc" (PDF) . Annual Review of Biochemistry . 69 : 1005–1075. CiteSeerX 10.1.1.319.5709 . doi :10.1146/annurev.biochem.69.1.1005. PMID 10966481. Archivado (PDF) desde el original el 28 de diciembre de 2015.
- ^ Crofts AR (2004). "El complejo citocromo bc1: función en el contexto de la estructura". Revista anual de fisiología . 66 : 689–733. doi :10.1146/annurev.physiol.66.032102.150251. PMID 14977419.
- ^ Iwata S, Lee JW, Okada K, Lee JK, Iwata M, Rasmussen B, et al. (julio de 1998). "Estructura completa del complejo de 11 subunidades del citocromo bc1 mitocondrial bovino". Science . 281 (5373): 64–71. Bibcode :1998Sci...281...64I. doi :10.1126/science.281.5373.64. PMID 9651245.
- ^ Trumpower BL (julio de 1990). "El ciclo Q protonmotriz. Transducción de energía mediante el acoplamiento de la translocación de protones a la transferencia de electrones por el complejo citocromo bc1" (PDF) . The Journal of Biological Chemistry . 265 (20): 11409–11412. doi : 10.1016/S0021-9258(19)38410-8 . PMID 2164001. Archivado (PDF) desde el original el 27 de septiembre de 2007.
- ^ Hunte C, Palsdottir H, Trumpower BL (junio de 2003). "Vías y mecanismos protonmotrices en el complejo citocromo bc1". FEBS Letters . 545 (1): 39–46. Bibcode :2003FEBSL.545...39H. doi : 10.1016/S0014-5793(03)00391-0 . PMID 12788490. S2CID 13942619.
- ^ Calhoun MW, Thomas JW, Gennis RB (agosto de 1994). "La superfamilia de bombas de protones impulsadas por rédox de la citocromo oxidasa". Tendencias en ciencias bioquímicas . 19 (8): 325–330. doi :10.1016/0968-0004(94)90071-X. PMID 7940677.
- ^ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, et al. (mayo de 1996). "Toda la estructura de la citocromo c oxidasa de 13 subunidades oxidada a 2,8 A". Science . 272 (5265): 1136–1144. Bibcode :1996Sci...272.1136T. doi :10.1126/science.272.5265.1136. PMID 8638158. S2CID 20860573.
- ^ Yoshikawa S, Muramoto K, Shinzawa-Itoh K, Aoyama H, Tsukihara T, Shimokata K, et al. (2006). "Mecanismo de bombeo de protones de la citocromo c oxidasa del corazón bovino". Biochimica et Biophysica Acta (BBA) - Bioenergética . 1757 (9–10): 1110–1116. doi : 10.1016/j.bbabio.2006.06.004 . PMID 16904626.
- ^ Rasmusson AG, Soole KL, Elthon TE (2004). "NAD(P)H deshidrogenasas alternativas de mitocondrias de plantas". Revisión anual de biología vegetal . 55 : 23–39. doi :10.1146/annurev.arplant.55.031903.141720. PMID 15725055.
- ^ Menz RI, Day DA (septiembre de 1996). "Purificación y caracterización de una NADH deshidrogenasa de 43 kDa insensible a la rotenona de mitocondrias de plantas". The Journal of Biological Chemistry . 271 (38): 23117–23120. doi : 10.1074/jbc.271.38.23117 . PMID 8798503. S2CID 893754.
- ^ McDonald A, Vanlerberghe G (junio de 2004). "Transporte electrónico mitocondrial ramificado en Animalia: presencia de oxidasa alternativa en varios filos animales". IUBMB Life . 56 (6): 333–341. doi : 10.1080/1521-6540400000876 . PMID 15370881.
- ^ Sluse FE, Jarmuszkiewicz W (junio de 1998). "Oxidasa alternativa en la red respiratoria mitocondrial ramificada: una descripción general de la estructura, función, regulación y papel". Revista Brasileña de Investigaciones Médicas y Biológicas = Revista Brasileira de Pesquisas Medicas e Biologicas . 31 (6): 733–747. doi : 10.1590/S0100-879X1998000600003 . PMID 9698817.
- ^ Moore AL, Siedow JN (agosto de 1991). "La regulación y la naturaleza de la oxidasa alternativa resistente al cianuro de las mitocondrias de las plantas". Biochimica et Biophysica Acta (BBA) - Bioenergetics . 1059 (2): 121–140. doi :10.1016/S0005-2728(05)80197-5. PMID 1883834.
- ^ Vanlerberghe GC, McIntosh L (junio de 1997). "OXIDASA ALTERNATIVA: del gen a la función". Revisión anual de fisiología vegetal y biología molecular vegetal . 48 : 703–734. doi :10.1146/annurev.arplant.48.1.703. PMID 15012279.
- ^ Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A (diciembre de 1997). "Los niveles de transcripción de genes de oxidasa alternativos dispuestos en tándem en el arroz aumentan con la baja temperatura". Gen. 203 (2): 121-129. doi :10.1016/S0378-1119(97)00502-7. PMID 9426242.
- ^ Maxwell DP, Wang Y, McIntosh L (julio de 1999). "La oxidasa alternativa reduce la producción de oxígeno reactivo mitocondrial en células vegetales". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 96 (14): 8271–8276. Bibcode :1999PNAS...96.8271M. doi : 10.1073/pnas.96.14.8271 . PMC 22224 . PMID 10393984.
- ^ Lenaz G (diciembre de 2001). "Una evaluación crítica del conjunto de coenzima Q mitocondrial". FEBS Letters . 509 (2): 151–155. Bibcode :2001FEBSL.509..151L. doi : 10.1016/S0014-5793(01)03172-6 . PMID 11741580. S2CID 46138989.
- ^ Heinemeyer J, Braun HP, Boekema EJ, Kouril R (abril de 2007). "Un modelo estructural del supercomplejo citocromo C reductasa/oxidasa de las mitocondrias de levadura". The Journal of Biological Chemistry . 282 (16): 12240–12248. doi : 10.1074/jbc.M610545200 . PMID 17322303. S2CID 18123642.
- ^ Schägger H, Pfeiffer K (abril de 2000). "Supercomplejos en las cadenas respiratorias de las mitocondrias de levaduras y mamíferos". The EMBO Journal . 19 (8): 1777–1783. doi :10.1093/emboj/19.8.1777. PMC 302020 . PMID 10775262.
- ^ Schägger H (septiembre de 2002). "Supercomplejos de la cadena respiratoria de mitocondrias y bacterias". Biochimica et Biophysica Acta (BBA) - Bioenergetics . 1555 (1–3): 154–159. doi : 10.1016/S0005-2728(02)00271-2 . PMID 12206908.
- ^ Schägger H, Pfeiffer K (octubre de 2001). "La proporción de complejos de fosforilación oxidativa IV en mitocondrias de corazón bovino y la composición de supercomplejos de la cadena respiratoria". The Journal of Biological Chemistry . 276 (41): 37861–37867. doi : 10.1074/jbc.M106474200 . PMID 11483615. Archivado desde el original el 29 de septiembre de 2007.
- ^ Gupte S, Wu ES, Hoechli L, Hoechli M, Jacobson K, Sowers AE, et al. (mayo de 1984). "Relación entre la difusión lateral, la frecuencia de colisión y la transferencia de electrones de los componentes de oxidación-reducción de la membrana interna mitocondrial". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 81 (9): 2606–2610. Bibcode :1984PNAS...81.2606G. doi : 10.1073/pnas.81.9.2606 . PMC 345118 . PMID 6326133.
- ^ Nealson KH (enero de 1999). "Microbiología post-vikinga: nuevos enfoques, nuevos datos, nuevos conocimientos". Orígenes de la vida y evolución de la biosfera . 29 (1): 73–93. Bibcode :1999OLEB...29...73N. doi :10.1023/A:1006515817767. PMID 11536899. S2CID 12289639.
- ^ Schäfer G, Engelhard M, Müller V (septiembre de 1999). "Bioenergética de las arqueas". Microbiology and Molecular Biology Reviews . 63 (3): 570–620. doi :10.1128/MMBR.63.3.570-620.1999. PMC 103747 . PMID 10477309.
- ^ ab Ingledew WJ, Poole RK (septiembre de 1984). "Las cadenas respiratorias de Escherichia coli". Microbiological Reviews . 48 (3): 222–271. doi :10.1128/mmbr.48.3.222-271.1984. PMC 373010 . PMID 6387427.
- ^ ab Unden G, Bongaerts J (julio de 1997). "Vías respiratorias alternativas de Escherichia coli: regulación energética y transcripcional en respuesta a aceptores de electrones". Biochimica et Biophysica Acta (BBA) - Bioenergetics . 1320 (3): 217–234. doi : 10.1016/S0005-2728(97)00034-0 . PMID 9230919.
- ^ Cecchini G, Schröder I, Gunsalus RP, Maklashina E (enero de 2002). "Succinato deshidrogenasa y fumarato reductasa de Escherichia coli". Biochimica et Biophysica Acta (BBA) - Bioenergetics . 1553 (1–2): 140–157. doi : 10.1016/S0005-2728(01)00238-9 . PMID 11803023.
- ^ Freitag A, Bock E (1990). "Conservación de energía en Nitrobacter". FEMS Microbiology Letters . 66 (1–3): 157–62. doi : 10.1111/j.1574-6968.1990.tb03989.x .
- ^ Starkenburg SR, Chain PS, Sayavedra-Soto LA, Hauser L, Land ML, Larimer FW, et al. (marzo de 2006). "Secuencia del genoma de la bacteria quimiolitoautotrófica oxidante de nitrito Nitrobacter winogradskyi Nb-255". Applied and Environmental Microbiology . 72 (3): 2050–2063. Bibcode :2006ApEnM..72.2050S. doi :10.1128/AEM.72.3.2050-2063.2006. PMC 1393235 . PMID 16517654.
- ^ Yamanaka T, Fukumori Y (diciembre de 1988). "El sistema de oxidación de nitrito de Nitrobacter winogradskyi". FEMS Microbiology Reviews . 54 (4): 259–270. doi : 10.1111/j.1574-6968.1988.tb02746.x . PMID 2856189.
- ^ Iuchi S, Lin EC (julio de 1993). "Adaptación de Escherichia coli a ambientes redox mediante expresión génica". Microbiología molecular . 9 (1): 9–15. doi :10.1111/j.1365-2958.1993.tb01664.x. PMID 8412675. S2CID 39165641.
- ^ Calhoun MW, Oden KL, Gennis RB, de Mattos MJ, Neijssel OM (mayo de 1993). "Eficiencia energética de Escherichia coli: efectos de las mutaciones en los componentes de la cadena respiratoria aeróbica" (PDF) . Journal of Bacteriology . 175 (10): 3020–3025. doi :10.1128/jb.175.10.3020-3025.1993. PMC 204621. PMID 8491720. Archivado (PDF) desde el original el 27 de septiembre de 2007.
- ^ ab Boyer PD (1997). "La ATP sintasa: una espléndida máquina molecular". Revista anual de bioquímica . 66 : 717–749. doi :10.1146/annurev.biochem.66.1.717. PMID 9242922.
- ^ Van Walraven HS, Strotmann H, Schwarz O, Rumberg B (febrero de 1996). "La relación de acoplamiento H+/ATP de la ATP sintasa de cloroplastos modulados por tiol y dos cepas de cianobacterias es cuatro". FEBS Letters . 379 (3): 309–313. Bibcode :1996FEBSL.379..309V. doi :10.1016/0014-5793(95)01536-1. PMID 8603713. S2CID 35989618.
- ^ Yoshida M, Muneyuki E, Hisabori T (septiembre de 2001). "ATP sintasa: un maravilloso motor rotatorio de la célula". Nature Reviews. Molecular Cell Biology . 2 (9): 669–677. doi :10.1038/35089509. PMID 11533724. S2CID 3926411.
- ^ Schemidt RA, Qu J, Williams JR, Brusilow WS (junio de 1998). "Efectos de la fuente de carbono en la expresión de genes F0 y en la estequiometría de la subunidad c en la ATPasa F1F0 de Escherichia coli". Journal of Bacteriology . 180 (12): 3205–3208. doi :10.1128/jb.180.12.3205-3208.1998. PMC 107823 . PMID 9620972.
- ^ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H (enero de 2000). "La biología celular de la generación de fuerza protónica por V-ATPases". The Journal of Experimental Biology . 203 (Pt 1): 89–95. doi :10.1242/jeb.203.1.89. PMID 10600677. Archivado desde el original el 30 de septiembre de 2007.
- ^ Rubinstein JL, Walker JE, Henderson R (diciembre de 2003). "Estructura de la ATP sintasa mitocondrial mediante criomicroscopía electrónica". The EMBO Journal . 22 (23): 6182–6192. doi :10.1093/emboj/cdg608. PMC 291849 . PMID 14633978.
- ^ Leslie AG, Walker JE (abril de 2000). "Modelo estructural de la F1-ATPasa y las implicaciones para la catálisis rotatoria". Philosophical Transactions of the Royal Society of London. Serie B, Ciencias Biológicas . 355 (1396): 465–471. doi :10.1098/rstb.2000.0588. PMC 1692760 . PMID 10836500.
- ^ Noji H, Yoshida M (enero de 2001). "La máquina rotatoria en la célula, la ATP sintasa". The Journal of Biological Chemistry . 276 (3): 1665–1668. doi : 10.1074/jbc.R000021200 . PMID 11080505. S2CID 30953216.
- ^ Capaldi RA, Aggeler R (marzo de 2002). "Mecanismo de la ATP sintasa de tipo F(1)F(0), un motor rotatorio biológico". Tendencias en ciencias bioquímicas . 27 (3): 154–160. doi :10.1016/S0968-0004(01)02051-5. PMID 11893513.
- ^ Dimroth P, von Ballmoos C, Meier T (marzo de 2006). "Ciclos catalíticos y mecánicos en las sintasas de F-ATP. Cuarto artículo de la serie de revisión de ciclos". EMBO Reports . 7 (3): 276–282. doi :10.1038/sj.embor.7400646. PMC 1456893 . PMID 16607397.
- ^ ab Gresser MJ, Myers JA, Boyer PD (octubre de 1982). "Cooperatividad del sitio catalítico de la adenosina trifosfatasa mitocondrial F1 de corazón de res. Correlaciones de la velocidad inicial, el intermedio ligado y las mediciones de intercambio de oxígeno con un modelo de tres sitios alternados". The Journal of Biological Chemistry . 257 (20): 12030–12038. doi : 10.1016/S0021-9258(18)33672-X . PMID 6214554. Archivado desde el original el 29 de septiembre de 2007.
- ^ Dimroth P (1994). "Energética bacteriana acoplada al ion sodio". Antonie van Leeuwenhoek . 65 (4): 381–395. doi :10.1007/BF00872221. PMID 7832594. S2CID 23763996.
- ^ ab Becher B, Müller V (mayo de 1994). "Delta mu Na+ impulsa la síntesis de ATP a través de una F1F0-ATP sintasa translocadora de delta mu Na(+) en vesículas de membrana de la arqueona Methanosarcina mazei Gö1". Journal of Bacteriology . 176 (9): 2543–2550. doi :10.1128/jb.176.9.2543-2550.1994. PMC 205391 . PMID 8169202.
- ^ Müller V (febrero de 2004). "Una variabilidad excepcional en el motor de las ATPases A1A0 de arqueas: de rotores multiméricos a monoméricos que comprenden de 6 a 13 sitios de unión de iones". Journal of Bioenergetics and Biomembranes . 36 (1): 115–125. doi :10.1023/B:JOBB.0000019603.68282.04. PMID 15168615. S2CID 24887884.
- ^ ab Davies KJ (1995). "Estrés oxidativo: la paradoja de la vida aeróbica". Simposio de la Sociedad Bioquímica . 61 : 1–31. doi :10.1042/bss0610001. PMID 8660387.
- ^ Rattan SI (diciembre de 2006). «Teorías del envejecimiento biológico: genes, proteínas y radicales libres» (PDF) . Free Radical Research . 40 (12): 1230–1238. CiteSeerX 10.1.1.476.9259 . doi :10.1080/10715760600911303. PMID 17090411. S2CID 11125090. Archivado desde el original (PDF) el 2014-06-14 . Consultado el 2017-10-27 .
- ^ Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J (2007). "Radicales libres y antioxidantes en funciones fisiológicas normales y enfermedades humanas". Revista Internacional de Bioquímica y Biología Celular . 39 (1): 44–84. doi :10.1016/j.biocel.2006.07.001. PMID 16978905.
- ^ Raha S, Robinson BH (octubre de 2000). "Mitocondrias, radicales libres de oxígeno, enfermedad y envejecimiento". Tendencias en ciencias bioquímicas . 25 (10): 502–508. doi :10.1016/S0968-0004(00)01674-1. PMID 11050436.
- ^ Finkel T, Holbrook NJ (noviembre de 2000). "Oxidantes, estrés oxidativo y biología del envejecimiento". Nature . 408 (6809): 239–247. Bibcode :2000Natur.408..239F. doi :10.1038/35041687. PMID 11089981. S2CID 2502238.
- ^ Kadenbach B, Ramzan R, Wen L, Vogt S (marzo de 2010). "Nueva extensión de la teoría de Mitchell para la fosforilación oxidativa en mitocondrias de organismos vivos". Biochimica et Biophysica Acta (BBA) - Temas generales . 1800 (3): 205–212. doi :10.1016/j.bbagen.2009.04.019. PMID 19409964.
- ^ Echtay KS, Roussel D, St-Pierre J, Jekabsons MB, Cadenas S, Stuart JA, et al. (enero de 2002). "El superóxido activa las proteínas desacopladoras mitocondriales". Nature . 415 (6867): 96–99. Bibcode :2002Natur.415...96E. doi :10.1038/415096a. PMID 11780125. S2CID 4349744.
- ^ St-Pierre J, Brand MD, Boutilier RG (julio de 2000). "Las mitocondrias como consumidoras de ATP: traición celular en la anoxia". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (15): 8670–8674. Bibcode :2000PNAS...97.8670S. doi : 10.1073/pnas.140093597 . PMC 27006 . PMID 10890886.
- ^ Devaux JB, Hedges CP, Birch N, Herbert N, Renshaw GM, Hickey AJ (enero de 2019). "La acidosis mantiene la función de las mitocondrias cerebrales en peces de triple aleta tolerantes a la hipoxia: ¿una estrategia para sobrevivir a la exposición hipóxica aguda?". Frontiers in Physiology . 9 : 1941. doi : 10.3389/fphys.2018.01941 . PMC 6346031 . PMID 30713504.
- ^ ab Joshi S, Huang YG (agosto de 1991). "Complejo de ATP sintasa de mitocondrias de corazón bovino: la proteína que confiere sensibilidad a la oligomicina es esencial para la ATPasa sensible a la diciclohexil carbodiimida". Biochimica et Biophysica Acta (BBA) - Biomembranas . 1067 (2): 255–258. doi :10.1016/0005-2736(91)90051-9. PMID 1831660.
- ^ ab Satyanarayana U (2002). Bioquímica (2ª ed.). Calcuta, India: libros y afines. ISBN 8187134801.OCLC 71209231 .
- ^ Tsubaki M (enero de 1993). "Estudio infrarrojo por transformada de Fourier de la unión del cianuro al sitio binuclear Fea3-CuB de la citocromo c oxidasa del corazón bovino: implicación del cambio conformacional vinculado a redox en el sitio binuclear". Bioquímica . 32 (1): 164–173. doi :10.1021/bi00052a022. PMID 8380331.
- ^ Heytler PG (1979). "Desacopladores de la fosforilación oxidativa". En Sidney Fleischer, Lester Packer (eds.). Biomembranas Parte F: Bioenergética: Fosforilación oxidativa . Métodos en enzimología. Vol. 55. págs. 462–472. doi :10.1016/0076-6879(79)55060-5. ISBN 978-0-12-181955-2. Número PMID 156853.
- ^ Lambert AJ, Brand MD (septiembre de 2004). "Los inhibidores del sitio de unión de la quinona permiten una rápida producción de superóxido a partir de la NADH:ubiquinona oxidorreductasa mitocondrial (complejo I)". The Journal of Biological Chemistry . 279 (38): 39414–39420. doi : 10.1074/jbc.M406576200 . PMID 15262965. S2CID 26620903.
- ^ Dervartanian DV, Veeger C (noviembre de 1964). "ESTUDIOS SOBRE LA SUCCINATO DESHIDROGENASA. I. PROPIEDADES ESPECTALES DE LA ENZIMA PURIFICADA Y FORMACIÓN DE COMPLEJOS INHIBIDORES ENZIMA-COMPETITIVOS". Biochimica et Biophysica Acta (BBA) - Sección especializada en temas enzimológicos . 92 (2): 233–247. doi :10.1016/0926-6569(64)90182-8. PMID 14249115.
- ^ Ricquier D, Bouillaud F (enero de 2000). "Los homólogos de las proteínas desacopladoras: UCP1, UCP2, UCP3, StUCP y AtUCP". The Biochemical Journal . 345 Pt 2 (Pt 2): 161–179. doi :10.1042/0264-6021:3450161. PMC 1220743 . PMID 10620491.
- ^ Borecký J, Vercesi AE (2005). "Proteína mitocondrial desacoplada de las plantas y oxidasa alternativa: metabolismo energético y estrés". Bioscience Reports . 25 (3–4): 271–286. doi :10.1007/s10540-005-2889-2. PMID 16283557. S2CID 18598358.
- ^ Harden A, Young WJ (1906). "La fermentación alcohólica del jugo de levadura". Actas de la Royal Society . B (77): 405–20. doi : 10.1098/rspb.1906.0029 .
- ^ Kalckar HM (noviembre de 1974). "Orígenes del concepto de fosforilación oxidativa". Bioquímica molecular y celular . 5 (1–2): 55–63. doi :10.1007/BF01874172. PMID 4279328. S2CID 26999163.
- ^ Lipmann F (1941). "Generación metabólica y utilización de la energía de los enlaces de fosfato". Adv Enzymol . 1 : 99–162. doi :10.4159/harvard.9780674366701.c141. ISBN 9780674366701.
- ^ Friedkin M, Lehninger AL (abril de 1949). "Esterificación de fosfato inorgánico acoplada al transporte de electrones entre el nucleótido dihidrodifosfopiridina y el oxígeno". The Journal of Biological Chemistry . 178 (2): 611–644. doi : 10.1016/S0021-9258(18)56879-4 . PMID 18116985. Archivado desde el original el 16 de diciembre de 2008.
- ^ Kalckar HM (1991). "50 años de investigación biológica: desde la fosforilación oxidativa hasta la regulación del transporte que requiere energía". Revisión anual de bioquímica . 60 : 1–37. doi : 10.1146/annurev.bi.60.070191.000245 . PMID 1883194.
- ^ Belitser VA, Tsibakova ET (1939). "Acerca del mecanismo de fosforilación acoplado a la respiración". Biokhimiya . 4 : 516–534.
- ^ Slater EC (noviembre de 1953). "Mecanismo de fosforilación en la cadena respiratoria". Nature . 172 (4387): 975–978. Bibcode :1953Natur.172..975S. doi :10.1038/172975a0. PMID 13111237. S2CID 4153659.
- ^ Mitchell P (julio de 1961). "Acoplamiento de la fosforilación a la transferencia de electrones e hidrógeno mediante un mecanismo de tipo quimiosmótico". Nature . 191 (4784): 144–148. Bibcode :1961Natur.191..144M. doi :10.1038/191144a0. PMID 13771349. S2CID 1784050.
- ^ Saier Jr MH. Peter Mitchell y la fuerza vital . OCLC 55202414.
- ^ Mitchell P (1978). "El concepto de cadena respiratoria de David Keilin y sus consecuencias quimiosmóticas" (PDF) . Conferencia Nobel . Fundación Nobel. Archivado (PDF) desde el original el 27 de septiembre de 2007. Consultado el 21 de julio de 2007 .
- ^ Pullman ME, Penefsky HS, Datta A, Racker E (noviembre de 1960). "Resolución parcial de las enzimas que catalizan la fosforilación oxidativa. I. Purificación y propiedades de la adenosina trifosfatasa soluble estimulada por dinitrofenol". The Journal of Biological Chemistry . 235 (11): 3322–3329. doi : 10.1016/S0021-9258(20)81361-1 . PMID 13738472. Archivado desde el original el 29 de septiembre de 2007.
- ^ Boyer PD, Cross RL, Momsen W (octubre de 1973). "Un nuevo concepto para el acoplamiento de energía en la fosforilación oxidativa basado en una explicación molecular de las reacciones de intercambio de oxígeno". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 70 (10): 2837–2839. Bibcode :1973PNAS...70.2837B. doi : 10.1073/pnas.70.10.2837 . PMC 427120 . PMID 4517936.
- ^ "El Premio Nobel de Química 1997". Fundación Nobel. Archivado desde el original el 25 de marzo de 2017. Consultado el 21 de julio de 2007 .
Lectura adicional
Introductorio
- Nelson DL, Cox MM (2004). Principios de bioquímica de Lehninger (4.ª ed.). WH Freeman. ISBN 0-7167-4339-6.
- Schneider ED, Sagan D (2006). Into the Cool: flujo de energía, termodinámica y vida (1.ª ed.). University of Chicago Press. ISBN 0-226-73937-6.
- Lane N (2006). Poder, sexo, suicidio: mitocondrias y el sentido de la vida (1.ª ed.). Oxford University Press, EE. UU. ISBN 0-19-920564-7.
Avanzado
- Nicholls DG, Ferguson SJ (2002). Bioenergética 3 (1.ª ed.). Academic Press. ISBN 0-12-518121-3.
- Haynie D (2001). Termodinámica biológica (1.ª ed.). Cambridge University Press. ISBN 0-521-79549-4.
- Rajan SS (2003). Introducción a la bioenergética (1.ª ed.). Anmol. ISBN 81-261-1364-2.
- Wikstrom M, ed. (2005). Aspectos biofísicos y estructurales de la bioenergética (1.ª ed.). Royal Society of Chemistry. ISBN 0-85404-346-2.
Recursos generales
- Diagramas animados que ilustran la fosforilación oxidativa Wiley and Co Conceptos en bioquímica
- Conferencias de biofísica en línea Archivado el 2 de mayo de 2009 en Wayback Machine Antony Crofts, Universidad de Illinois en Urbana–Champaign
- ATP sintasa Graham Johnson
Recursos estructurales
- Molécula del mes del PDB :
- ATP sintasa Archivado el 24 de julio de 2020 en Wayback Machine
- Citocromo c Archivado el 24 de julio de 2020 en Wayback Machine
- Citocromo c oxidasa Archivado el 24 de julio de 2020 en Wayback Machine
- Modelos moleculares interactivos en la Universidade Fernando Pessoa :
- NADH deshidrogenasa
- succinato deshidrogenasa
- Coenzima Q - citocromo c reductasa
- citocromo c oxidasa






![{\displaystyle {\ce {O2->[{\ce {e^{-}}}]{\underset {Superóxido}{O2^{\underline {\bullet }}}}->[{\ce {e ^{-}}}]{\underset {Peróxido}{O2^{2-}}}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a3d9bf9d3a61736aa6207fa53b8ce0165b9eebb6)