Desmetilasa
Las desmetilasas son enzimas que eliminan los grupos metilo (CH3 ) de los ácidos nucleicos , las proteínas (en particular, las histonas ) y otras moléculas. Las desmetilasas son proteínas epigenéticas importantes , ya que son responsables de la regulación transcripcional del genoma al controlar la metilación del ADN y las histonas y, por extensión, el estado de la cromatina en loci genéticos específicos .
Desmetilación de la lisina de la histona

La metilación de histonas se consideró inicialmente un proceso efectivamente irreversible ya que la vida media de la metilación de histonas era aproximadamente igual a la vida media de la histona. [1] La histona lisina desmetilasa LSD1 (más tarde clasificada como KDM1A) se identificó por primera vez en 2004 como un homólogo de la aminooxidasa nuclear. [2] Existen dos clases principales de histona lisina desmetilasas, definidas por sus mecanismos: las aminooxidasas dependientes de flavina adenina dinucleótido (FAD) y las hidroxilasas dependientes de α-cetoglutarato .
Las histonas lisinas desmetilasas poseen una variedad de dominios que son responsables del reconocimiento de histonas, la unión al ADN, la unión al sustrato de aminoácidos metilados y la actividad catalítica. Estos incluyen:
- Dominios de aminooxidasa dependientes de FAD que contienen el sitio catalítico activo de KDM1
- Dominios Jumonji-C que contienen el sitio catalítico activo de KDM2 a KDM8 [3] [4]
- Dominios Jumonji-N responsables de la estabilidad de la conformación del dominio Jumonji-C
- Dominios SWIRM (SWI3P, RSC8P y Moira) propuestos como un sitio de anclaje para sustratos de histonas y responsables de la estabilidad de la cromatina
- Dominios de dedo de zinc PHD, CXXC y C5HC2 responsables del reconocimiento y unión de histonas
Las desmetilasas de lisina de histonas se clasifican según sus dominios y especificidades de sustrato únicas. Los sustratos de lisina se identifican según su posición en la secuencia de aminoácidos de la histona correspondiente y el estado de metilación (por ejemplo, H3K9me3 se refiere a la lisina 9 de la histona 3 trimetilada).


- KDM1
- Los homólogos de KDM1 incluyen KDM1A y KDM1B . KDM1A desmetila H3K4me1/2 y H3K9me1/2, y KDM1B desmetila H3K4me1/2. La actividad de KDM1 es fundamental para la embriogénesis y la diferenciación específica de tejidos , así como para el crecimiento de los ovocitos. [1] La eliminación del gen de KDM1A puede tener efectos en el crecimiento y la diferenciación de las células madre embrionarias y es universalmente letal en ratones knockout . [5] [6] Se observa que la expresión del gen KDM1A está regulada positivamente en algunos cánceres, [7] [8] y, por lo tanto, la inhibición de KDM1A se ha considerado un posible tratamiento epigenético para el cáncer. [9] [10] [11] :KDM1B, sin embargo, está principalmente involucrado en el desarrollo de ovocitos . La eliminación de este gen conduce a la letalidad por efecto materno en ratones. [12] Los ortólogos de KDM1 en D. melanogaster y C. elegans parecen funcionar de manera similar a KDM1B en lugar de KDM1A. [13] [14]
- KDM2
- Los homólogos de KDM2 incluyen KDM2A y KDM2B . KDM2A y KDM2B desmetilan H3K4me3 y H3K36me2/3. KDM2A tiene funciones en la promoción o inhibición de la función tumoral, y KDM2B tiene funciones en la oncogénesis . [1] KDM2A y KDM2B poseen dominios de dedos de zinc CXXC responsables de la unión a islas CpG no metiladas, y se cree que pueden unirse a muchos elementos reguladores de genes en ausencia de factores de transcripción específicos de secuencia. [15] :Se ha observado KDM2B sobreexpresado en linfoma y adenocarcinoma humano , y se ha observado KDM2B subexpresado en cáncer de próstata humano y glioblastoma. Se ha demostrado además que KDM2B previene la senescencia en algunas células a través de la expresión ectópica . [16]
- KDM3
- Los homólogos de KDM3 incluyen KDM3A , KDM3B y KDM3C . KDM3A, KDM3B y KDM3C desmetilan H3K9me1/2. KDM3A tiene funciones en la espermatogénesis y las funciones metabólicas, sin embargo, la actividad de KDM3B y KDM3C no se conoce específicamente. [1] :Los estudios de supresión de KDM3A en ratones dieron como resultado infertilidad masculina y obesidad de inicio en la edad adulta. Estudios adicionales han indicado que KDM3A puede desempeñar un papel en la regulación de los genes dependientes del receptor de andrógenos, así como de los genes involucrados en la pluripotencia , lo que indica un papel potencial para KDM3A en la tumorigénesis. [17]
- KDM4
- Los homólogos de KDM4 incluyen KDM4A , KDM4B , KDM4C , KDM4D , KDM4E y KDM4F. KDM4A, KDM4B y KDM4C desmetilan H3K9me2/3, H3K9me3 y H3K36me2/3, y KDM4D, KDM4E y KDM4F desmetilan H3K9me2/3. KDM4A, KDM4B, KDM4C y KDM4D tienen funciones en la tumorigénesis , sin embargo, la actividad de KDM4E y KDM4F no se conoce específicamente. [1] Se ha observado una regulación positiva de KDM4B en el meduloblastoma, y se ha documentado una amplificación de KDM4C en el carcinoma escamoso esofágico, el meduloblastoma y el cáncer de mama. [18] [19] [20] [21] Otros datos de expresión genética también han sugerido que KDM4A, KDM4B y KDM4C están sobreexpresados en el cáncer de próstata. [22]
- KDM5
- Los homólogos de KDM5 incluyen KDM5A , KDM5B , KDM5C y KDM5D . KDM5A, KDM5B, KDM5C y KDM5D desmetilan H3K4me2/3. [1] La familia KDM5 parece regular funciones clave del desarrollo, incluyendo la diferenciación celular, la función mitocondrial y la progresión del ciclo celular . [23] [24] [25] [26] [27] [28] KDM5B y KDM5C también han demostrado interactuar con proteínas PcG, que están involucradas en la represión transcripcional. También se han observado mutaciones de KDM5C en el cromosoma X en pacientes con discapacidad intelectual ligada al cromosoma X. [29] El agotamiento de los homólogos de KDM5C en D. rerio ha mostrado defectos en el patrón cerebral y muerte de células neuronales. [30]
- KDM6
- La familia KDM6 incluye KDM6A , KDM6B y KDM6C . KDM6A y KDM6B desmetilan H3K27me2/3, y KDM4C desmetila H3K27me3. KDM6A y KDM6B poseen características supresoras de tumores. La supresión de KDM6A en fibroblastos conduce a un aumento inmediato de la población de fibroblastos. KDM6B expresado en fibroblastos induce oncogenes de la vía Ras/Raf/MEK/ERK. [31] Las mutaciones puntuales de KDM6A se han identificado como una causa del síndrome de Kabuki , un trastorno congénito que resulta en discapacidad intelectual. [32] [33] La eliminación de KDM6A en D. rerio da como resultado una expresión disminuida de los genes HOX, que desempeñan un papel en la regulación de los patrones corporales durante el desarrollo. [34] En estudios con mamíferos, se ha demostrado que KDM6A también regula los genes HOX. [35] [36] La mutación de KDM5B altera el desarrollo de las gónadas en C.elegans . [35] Otros estudios han demostrado que la expresión de KDM6B aumenta en los macrófagos activados y se expresa dinámicamente durante la diferenciación de las células madre . [37] [38]
Desmetilación de ésteres

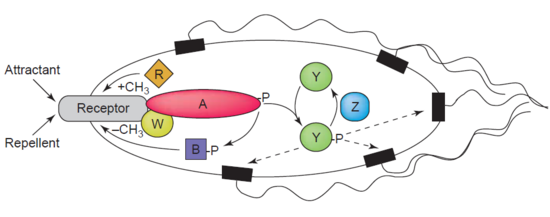
Otro ejemplo de una desmetilasa es la proteína-glutamato metilesterasa , también conocida como proteína CheB (EC 3.1.1.61), que desmetila las MCP ( proteínas de hemotaxis que aceptan metil ) a través de la hidrólisis de enlaces de éster carboxílico. La asociación de un receptor de quimiotaxis con un agonista conduce a la fosforilación de CheB. La fosforilación de la proteína CheB mejora su actividad catalítica de desmetilación de MCP, lo que resulta en la adaptación de la célula a los estímulos ambientales. [39] Las MCP responden a atrayentes y repelentes extracelulares en bacterias como E. coli en la regulación de la quimiotaxis . CheB se denomina más específicamente una metilesterasa , ya que elimina los grupos metilo de los residuos de metilglutamato ubicados en las MCP a través de la hidrólisis, produciendo glutamato acompañado de la liberación de metanol . [40]
CheB es de particular interés para los investigadores ya que puede ser un objetivo terapéutico para mitigar la propagación de infecciones bacterianas. [41]
Véase también
Referencias
- ^ abcdef Pedersen MT, Helin K (noviembre de 2010). "Histonas desmetilasas en el desarrollo y la enfermedad". Tendencias en biología celular . 20 (11): 662–71. doi :10.1016/j.tcb.2010.08.011. PMID 20863703.
- ^ Shi Y, Lan F, Matson C, Mulligan P, Whetstine JR, Cole PA, Casero RA, Shi Y (diciembre de 2004). "Desmetilación de histonas mediada por el homólogo de la aminooxidasa nuclear LSD1". Cell . 119 (7): 941–53. doi : 10.1016/j.cell.2004.12.012 . PMID 15620353.
- ^ Mosammaparast N, Shi Y (2010). "Reversión de la metilación de histonas: mecanismos bioquímicos y moleculares de las desmetilasas de histonas". Revisión anual de bioquímica . 79 : 155–79. doi :10.1146/annurev.biochem.78.070907.103946. PMID 20373914.
- ^ Aprelikova O, Chen K, El Touny LH, Brignatz-Guittard C, Han J, Qiu T, Yang HH, Lee MP, Zhu M, Green JE (abril de 2016). "El modificador epigenético JMJD6 se amplifica en tumores mamarios y coopera con c-Myc para mejorar la transformación celular, la progresión tumoral y la metástasis". Clin Epigenetics . 8 (38): 38. doi : 10.1186/s13148-016-0205-6 . PMC 4831179 . PMID 27081402.
- ^ Wang J, Hevi S, Kurash JK, Lei H, Gay F, Bajko J, Su H, Sun W, Chang H, Xu G, Gaudet F, Li E, Chen T (enero de 2009). "La lisina desmetilasa LSD1 (KDM1) es necesaria para el mantenimiento de la metilación global del ADN". Nature Genetics . 41 (1): 125–9. doi :10.1038/ng.268. PMID 19098913. S2CID 2010695.
- ^ Wang J, Scully K, Zhu X, Cai L, Zhang J, Prefontaine GG, Krones A, Ohgi KA, Zhu P, Garcia-Bassets I, Liu F, Taylor H, Lozach J, Jayes FL, Korach KS, Glass CK, Fu XD, Rosenfeld MG (abril de 2007). "Los complejos LSD1 opuestos funcionan en los programas de activación y represión de genes del desarrollo". Nature . 446 (7138): 882–7. Bibcode :2007Natur.446..882W. doi :10.1038/nature05671. PMID 17392792. S2CID 4387240.
- ^ Kahl P, Gullotti L, Heukamp LC, Wolf S, Friedrichs N, Vorreuther R, Solleder G, Bastian PJ, Ellinger J, Metzger E, Schüle R, Buettner R (diciembre de 2006). "Los coactivadores del receptor de andrógenos, la histona desmetilasa 1 específica de lisina y la proteína 2 de dominio LIM cuatro y medio, predicen el riesgo de recurrencia del cáncer de próstata". Cancer Research . 66 (23): 11341–7. doi :10.1158/0008-5472.CAN-06-1570. PMID 17145880.
- ^ Lim S, Janzer A, Becker A, Zimmer A, Schüle R, Buettner R, Kirfel J (marzo de 2010). "La desmetilasa 1 específica de lisina (LSD1) se expresa en gran medida en cánceres de mama ER-negativos y es un biomarcador que predice una biología agresiva". Carcinogénesis . 31 (3): 512–20. doi :10.1093/carcin/bgp324. PMID 20042638.
- ^ Metzger E, Wissmann M, Yin N, Müller JM, Schneider R, Peters AH, Günther T, Buettner R, Schüle R (septiembre de 2005). "LSD1 desmetila las marcas de histonas represivas para promover la transcripción dependiente del receptor de andrógenos". Naturaleza . 437 (7057): 436–9. Código Bib :2005Natur.437..436M. doi : 10.1038/naturaleza04020. PMID 16079795. S2CID 4308627.
- ^ Schulte JH, Lim S, Schramm A, Friedrichs N, Koster J, Versteeg R, Ora I, Pajtler K, Klein-Hitpass L, Kuhfittig-Kulle S, Metzger E, Schüle R, Eggert A, Buettner R, Kirfel J (marzo de 2009). "La desmetilasa 1 específica de lisina se expresa fuertemente en el neuroblastoma poco diferenciado: implicaciones para la terapia". Cancer Research . 69 (5): 2065–71. doi : 10.1158/0008-5472.CAN-08-1735 . PMID 19223552.
- ^ Wang Y, Zhang H, Chen Y, Sun Y, Yang F, Yu W, Liang J, Sun L, Yang X, Shi L, Li R, Li Y, Zhang Y, Li Q, Yi X, Shang Y (agosto de 2009). "LSD1 es una subunidad del complejo NuRD y se dirige a los programas de metástasis en el cáncer de mama". Cell . 138 (4): 660–72. doi : 10.1016/j.cell.2009.05.050 . PMID 19703393.
- ^ Ciccone DN, Su H, Hevi S, Gay F, Lei H, Bajko J, Xu G, Li E, Chen T (septiembre de 2009). "KDM1B es una desmetilasa de la histona H3K4 necesaria para establecer las improntas genómicas maternas". Nature . 461 (7262): 415–8. Bibcode :2009Natur.461..415C. doi :10.1038/nature08315. PMID 19727073. S2CID 1143128.
- ^ Rudolph T, Yonezawa M, Lein S, Heidrich K, Kubicek S, Schäfer C, Phalke S, Walther M, Schmidt A, Jenuwein T, Reuter G (abril de 2007). "La formación de heterocromatina en Drosophila se inicia mediante la eliminación activa de la metilación de H3K4 por el homólogo de LSD1 SU(VAR)3-3". Molecular Cell . 26 (1): 103–15. doi : 10.1016/j.molcel.2007.02.025 . PMID 17434130.
- ^ Di Stefano L, Ji JY, Moon NS, Herr A, Dyson N (mayo de 2007). "La mutación de Lsd1 de Drosophila altera la metilación de H3-K4, lo que produce defectos específicos de tejido durante el desarrollo". Current Biology . 17 (9): 808–12. doi :10.1016/j.cub.2007.03.068. PMC 1909692 . PMID 17462898.
- ^ Blackledge NP, Zhou JC, Tolstorukov MY, Farcas AM, Park PJ, Klose RJ (abril de 2010). "Las islas CpG reclutan una histona H3 lisina 36 desmetilasa". Molecular Cell . 38 (2): 179–90. doi :10.1016/j.molcel.2010.04.009. PMC 3098377 . PMID 20417597.
- ^ He J, Kallin EM, Tsukada Y, Zhang Y (noviembre de 2008). "La desmetilasa H3K36 Jhdm1b/Kdm2b regula la proliferación celular y la senescencia a través de p15(Ink4b)". Nature Structural & Molecular Biology . 15 (11): 1169–75. doi :10.1038/nsmb.1499. PMC 2612995 . PMID 18836456.
- ^ Loh YH, Zhang W, Chen X, George J, Ng HH (octubre de 2007). "Las desmetilasas de la histona H3 Lys 9 Jmjd1a y Jmjd2c regulan la autorrenovación en células madre embrionarias". Genes & Development . 21 (20): 2545–57. doi :10.1101/gad.1588207. PMC 2000320 . PMID 17938240.
- ^ Ehrbrecht A, Müller U, Wolter M, Hoischen A, Koch A, Radlwimmer B, Actor B, Mincheva A, Pietsch T, Lichter P, Reifenberger G, Weber RG (marzo de 2006). "Análisis genómico exhaustivo de meduloblastomas desmoplásicos: identificación de nuevos genes amplificados y evaluación separada de los diferentes componentes histológicos". The Journal of Pathology . 208 (4): 554–63. doi :10.1002/path.1925. PMID 16400626. S2CID 1463027.
- ^ Liu G, Bollig-Fischer A, Kreike B, van de Vijver MJ, Abrams J, Ethier SP, Yang ZQ (diciembre de 2009). "Amplificación genómica y propiedades oncogénicas del gen de la histona desmetilasa GASC1 en el cáncer de mama". Oncogene . 28 (50): 4491–500. doi :10.1038/onc.2009.297. PMC 2795798 . PMID 19784073.
- ^ Northcott PA, Nakahara Y, Wu X, Feuk L, Ellison DW, Croul S, Mack S, Kongkham PN, Peacock J, Dubuc A, Ra YS, Zilberberg K, McLeod J, Scherer SW, Sunil Rao J, Eberhart CG, Grajkowska W, Gillespie Y, Lach B, Grundy R, Pollack IF, Hamilton RL, Van Meter T, Carlotti CG, Boop F, Bigner D, Gilbertson RJ, Rutka JT, Taylor MD (abril de 2009). "Múltiples eventos genéticos recurrentes convergen en el control de la metilación de la lisina de la histona en el meduloblastoma". Nature Genetics . 41 (4): 465–72. doi :10.1038/ng.336. PMC 4454371 . PMID 19270706.
- ^ Yang ZQ, Imoto I, Fukuda Y, Pimkhaokham A, Shimada Y, Imamura M, Sugano S, Nakamura Y, Inazawa J (septiembre de 2000). "Identificación de un gen nuevo, GASC1, dentro de un amplicón en 9p23-24 detectado con frecuencia en líneas celulares de cáncer de esófago". Investigación del cáncer . 60 (17): 4735–9. PMID 10987278.
- ^ Cloos PA, Christensen J, Agger K, Maiolica A, Rappsilber J , Antal T, Hansen KH, Helin K (julio de 2006). "El supuesto oncogén GASC1 desmetila la lisina 9 trimetilada y dimetilada en la histona H3". Nature . 442 (7100): 307–11. Bibcode :2006Natur.442..307C. doi :10.1038/nature04837. PMID 16732293. S2CID 2874903.
- ^ Lee N, Erdjument-Bromage H, Tempst P, Jones RS, Zhang Y (marzo de 2009). "La H3K4 demethylase lid se asocia con la histona deacetilasa Rpd3 y la inhibe". Biología molecular y celular . 29 (6): 1401–10. doi :10.1128/MCB.01643-08. PMC 2648242 . PMID 19114561.
- ^ Benevolenskaya EV, Murray HL, Branton P, Young RA, Kaelin WG (junio de 2005). "La unión de pRB a la proteína PHD RBP2 promueve la diferenciación celular". Molecular Cell . 18 (6): 623–35. doi : 10.1016/j.molcel.2005.05.012 . PMID 15949438.
- ^ Lopez-Bigas N, Kisiel TA, Dewaal DC, Holmes KB, Volkert TL, Gupta S, Love J, Murray HL, Young RA, Benevolenskaya EV (agosto de 2008). "El análisis de todo el genoma de la histona desmetilasa H3K4 RBP2 revela un programa transcripcional que controla la diferenciación". Molecular Cell . 31 (4): 520–30. doi :10.1016/j.molcel.2008.08.004. PMC 3003864 . PMID 18722178.
- ^ Pasini D, Hansen KH, Christensen J, Agger K, Cloos PA, Helin K (mayo de 2008). "Regulación coordinada de la represión transcripcional por la desmetilasa RBP2 H3K4 y el complejo represivo Polycomb 2". Genes & Development . 22 (10): 1345–55. doi :10.1101/gad.470008. PMC 2377189 . PMID 18483221.
- ^ van Oevelen C, Wang J, Asp P, Yan Q, Kaelin WG, Kluger Y, Dynlacht BD (noviembre de 2008). "Un papel del Sin3 de los mamíferos en el silenciamiento genético permanente". Célula molecular . 32 (3): 359–70. doi :10.1016/j.molcel.2008.10.015. PMC 3100182 . PMID 18995834.
- ^ Zeng J, Ge Z, Wang L, Li Q, Wang N, Björkholm M, Jia J, Xu D (marzo de 2010). "La histona desmetilasa RBP2 se sobreexpresa en el cáncer gástrico y su inhibición desencadena la senescencia de las células cancerosas". Gastroenterología . 138 (3): 981–92. doi :10.1053/j.gastro.2009.10.004. PMID 19850045.
- ^ Jensen LR, Amende M, Gurok U, Moser B, Gimmel V, Tzschach A, Janecke AR, Tariverdian G, Chelly J, Fryns JP, Van Esch H, Kleefstra T, Hamel B, Moraine C, Gecz J, Turner G, Reinhardt R, Kalscheuer VM, Ropers HH, Lenzner S (febrero de 2005). "Las mutaciones en el gen JARID1C, que está implicado en la regulación transcripcional y la remodelación de la cromatina, causan retraso mental ligado al cromosoma X". American Journal of Human Genetics . 76 (2): 227–36. doi :10.1086/427563. PMC 1196368 . PMID 15586325.
- ^ Iwase S, Lan F, Bayliss P, de la Torre-Ubieta L, Huarte M, Qi HH, Whetstine JR, Bonni A, Roberts TM, Shi Y (marzo de 2007). "El gen de retraso mental ligado al cromosoma X SMCX/JARID1C define una familia de desmetilasas de la histona H3 lisina 4". Cell . 128 (6): 1077–88. doi : 10.1016/j.cell.2007.02.017 . PMID 17320160.
- ^ Agger K, Cloos PA, Rudkjaer L, Williams K, Andersen G, Christensen J, Helin K (mayo de 2009). "La desmetilasa H3K27me3 JMJD3 contribuye a la activación del locus INK4A-ARF en respuesta a la senescencia inducida por oncogenes y estrés". Genes & Development . 23 (10): 1171–6. doi :10.1101/gad.510809. PMC 2685535 . PMID 19451217.
- ^ Lederer D, Grisart B, Digilio MC, Benoit V, Crespin M, Ghariani SC, Maystadt I, Dallapiccola B, Verellen-Dumoulin C (enero de 2012). "Eliminación de KDM6A, una histona desmetilasa que interactúa con MLL2, en tres pacientes con síndrome de Kabuki". American Journal of Human Genetics . 90 (1): 119–24. doi :10.1016/j.ajhg.2011.11.021. PMC 3257878 . PMID 22197486.
- ^ Miyake N, Mizuno S, Okamoto N, Ohashi H, Shiina M, Ogata K, Tsurusaki Y, Nakashima M, Saitsu H, Niikawa N, Matsumoto N (enero de 2013). "Las mutaciones puntuales KDM6A causan el síndrome de Kabuki". Mutación humana . 34 (1): 108–10. doi : 10.1002/humu.22229 . PMID 23076834. S2CID 1745473.
- ^ Lan F, Bayliss PE, Rinn JL, Whetstine JR, Wang JK, Chen S, Iwase S, Alpatov R, Issaeva I, Canaani E, Roberts TM, Chang HY, Shi Y (octubre de 2007). "Una histona H3 lisina 27 desmetilasa regula el desarrollo posterior de los animales". Naturaleza . 449 (7163): 689–94. Código Bib :2007Natur.449..689L. doi : 10.1038/naturaleza06192. PMID 17851529. S2CID 612144.
- ^ ab Agger K, Cloos PA, Christensen J, Pasini D, Rose S, Rappsilber J, Issaeva I, Canaani E, Salcini AE, Helin K (octubre de 2007). "UTX y JMJD3 son histonas H3K27 desmetilasas implicadas en la regulación y el desarrollo del gen HOX". Naturaleza . 449 (7163): 731–4. Código Bib :2007Natur.449..731A. doi : 10.1038/naturaleza06145. PMID 17713478. S2CID 4413812.
- ^ Wang JK, Tsai MC, Poulin G, Adler AS, Chen S, Liu H, Shi Y, Chang HY (febrero de 2010). "La histona desmetilasa UTX permite el control del destino celular dependiente de RB". Genes & Development . 24 (4): 327–32. doi :10.1101/gad.1882610. PMC 2816731 . PMID 20123895.
- ^ De Santa F, Totaro MG, Prosperini E, Notarbartolo S, Testa G, Natoli G (septiembre de 2007). "La histona H3 lisina-27 desmetilasa Jmjd3 vincula la inflamación con la inhibición del silenciamiento génico mediado por polycomb". Cell . 130 (6): 1083–94. doi : 10.1016/j.cell.2007.08.019 . PMID 17825402.
- ^ Burgold T, Spreafico F, De Santa F, Totaro MG, Prosperini E, Natoli G, Testa G (2008). "La desmetilasa Jmjd3 específica de la lisina 27 de la histona H3 es necesaria para el compromiso neuronal". PLOS ONE . 3 (8): e3034. Bibcode :2008PLoSO...3.3034B. doi : 10.1371/journal.pone.0003034 . PMC 2515638 . PMID 18716661.
- ^ ab Vladimirov N, Løvdok L, Lebiedz D, Sourjik V (diciembre de 2008). "Dependencia de la quimiotaxis bacteriana en la forma del gradiente y la tasa de adaptación". PLOS Computational Biology . 4 (12): e1000242. Bibcode :2008PLSCB...4E0242V. doi : 10.1371/journal.pcbi.1000242 . PMC 2588534 . PMID 19096502.
- ^ Park SY, Borbat PP, Gonzalez-Bonet G, Bhatnagar J, Pollard AM, Freed JH, Bilwes AM, Crane BR (mayo de 2006). "Reconstrucción del ensamblaje de la quimiotaxis receptor-quinasa". Nature Structural & Molecular Biology . 13 (5): 400–7. doi :10.1038/nsmb1085. PMID 16622408. S2CID 859928.
- ^ West AH, Martinez-Hackert E, Stock AM (julio de 1995). "Estructura cristalina del dominio catalítico de la metilesterasa del receptor de quimiotaxis, CheB". Journal of Molecular Biology . 250 (2): 276–90. doi :10.1006/jmbi.1995.0376. PMID 7608974.
