Mutación por desplazamiento del marco de lectura

Una mutación por desplazamiento del marco de lectura (también llamada error de encuadre o cambio del marco de lectura ) es una mutación genética causada por indels ( inserciones o deleciones ) de un número de nucleótidos en una secuencia de ADN que no es divisible por tres. Debido a la naturaleza triplete de la expresión génica por codones , la inserción o deleción puede cambiar el marco de lectura (la agrupación de los codones), lo que resulta en una traducción completamente diferente de la original. Cuanto antes en la secuencia se produzca la deleción o inserción, más alterada será la proteína. [1] Una mutación por desplazamiento del marco de lectura no es lo mismo que un polimorfismo de un solo nucleótido en el que se reemplaza un nucleótido, en lugar de insertarlo o eliminarlo. Una mutación por desplazamiento del marco de lectura en general hará que la lectura de los codones después de la mutación codifique diferentes aminoácidos. La mutación por desplazamiento del marco de lectura también alterará el primer codón de terminación ("UAA", "UGA" o "UAG") encontrado en la secuencia. El polipéptido que se está creando podría ser anormalmente corto o anormalmente largo, y lo más probable es que no sea funcional. [2]
Las mutaciones por desplazamiento del marco de lectura son evidentes en enfermedades genéticas graves como la enfermedad de Tay-Sachs ; aumentan la susceptibilidad a ciertos cánceres y clases de hipercolesterolemia familiar ; en 1997, [3] una mutación por desplazamiento del marco de lectura se relacionó con la resistencia a la infección por el retrovirus VIH. Las mutaciones por desplazamiento del marco de lectura se han propuesto como una fuente de novedad biológica, como en el caso de la supuesta creación de la nilonasa ; sin embargo, esta interpretación es controvertida. Un estudio de Negoro et al. (2006) [4] encontró que era poco probable que una mutación por desplazamiento del marco de lectura fuera la causa y que, más bien, una sustitución de dos aminoácidos en el sitio activo de una esterasa ancestral dio lugar a la nilonasa.
Fondo
La información contenida en el ADN determina la función de las proteínas en las células de todos los organismos. La transcripción y la traducción permiten que esta información se comunique para la fabricación de proteínas. Sin embargo, un error en la lectura de esta comunicación puede provocar que la función de las proteínas sea incorrecta y, con el tiempo, causar enfermedades, incluso aunque la célula incorpore una variedad de medidas correctivas. La información genética se transmite por el ADN para la síntesis de proteínas dentro de las células. Una interpretación errónea puede provocar un funcionamiento defectuoso y enfermedades, a pesar de los mecanismos de corrección celular.
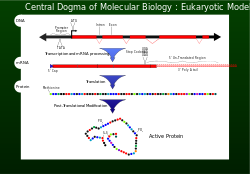
Dogma central
En 1956, Francis Crick describió el flujo de información genética desde el ADN hasta una disposición específica de aminoácidos para la fabricación de una proteína como el dogma central. [1] Para que una célula funcione correctamente, es necesario que las proteínas se produzcan de forma precisa para las actividades estructurales y catalíticas . Una proteína producida de forma incorrecta puede tener efectos perjudiciales en la viabilidad celular y, en la mayoría de los casos, hacer que el organismo superior se vuelva insalubre debido a funciones celulares anormales. Para garantizar que el genoma transmita correctamente la información, se incorporan en la replicación del ADN mecanismos de corrección , como las exonucleasas y los sistemas de reparación de errores de emparejamiento . [1]
Transcripción y traducción

Después de la replicación del ADN, la lectura de una sección seleccionada de información genética se logra mediante la transcripción . [1] Los nucleótidos que contienen la información genética ahora están en una plantilla de mensajero de una sola cadena llamada ARNm . El ARNm se incorpora con una subunidad del ribosoma e interactúa con un ARNr . La información genética transportada en los codones del ARNm ahora es leída (decodificada) por anticodones del ARNt. A medida que se lee cada codón (triplete), los aminoácidos se unen hasta que se alcanza un codón de terminación (UAG, UGA o UAA). En este punto, el polipéptido (proteína) se ha sintetizado y se libera. [1] Por cada 1000 aminoácidos incorporados a la proteína, no más de uno es incorrecto. Esta fidelidad del reconocimiento de codones, que mantiene la importancia del marco de lectura adecuado, se logra mediante el apareamiento adecuado de bases en el sitio del ribosoma A, la actividad de hidrólisis de GTP de EF-Tu , una forma de estabilidad cinética, y un mecanismo de corrección a medida que se libera EF-Tu. [1]
El desplazamiento del marco de lectura también puede ocurrir durante la traducción en profase , produciendo diferentes proteínas a partir de marcos de lectura abiertos superpuestos, como las proteínas retrovirales gag-pol-env . Esto es bastante común en los virus y también ocurre en bacterias y levaduras (Farabaugh, 1996). Se cree que la transcriptasa inversa , a diferencia de la ARN polimerasa II , es una causa más importante de la aparición de mutaciones por desplazamiento del marco de lectura. En experimentos, solo el 3-13% de todas las mutaciones por desplazamiento del marco de lectura se produjeron debido a la ARN polimerasa II. En procariotas, la tasa de error que induce mutaciones por desplazamiento del marco de lectura solo está en algún lugar en el rango de .0001 y .00001. [5]
Existen varios procesos biológicos que ayudan a prevenir las mutaciones por desplazamiento del marco de lectura. Las mutaciones inversas se producen cuando la secuencia mutada vuelve a la secuencia original de tipo salvaje . Otra posibilidad para la corrección de la mutación es el uso de una mutación supresora . Esto compensa el efecto de la mutación original creando una mutación secundaria, desplazando la secuencia para permitir que se lean los aminoácidos correctos. El ARN guía también se puede utilizar para insertar o eliminar uridina en el ARNm después de la transcripción, lo que permite el marco de lectura correcto. [1]
Importancia del triplete de codones

Un codón es un conjunto de tres nucleótidos , un triplete que codifica un determinado aminoácido . El primer codón establece el marco de lectura, por el cual comienza un nuevo codón. La secuencia de aminoácidos de la estructura de una proteína está definida por tripletes contiguos. [6] Los codones son clave para la traducción de la información genética para la síntesis de proteínas. El marco de lectura se establece cuando comienza la traducción del ARNm y se mantiene a medida que lee un triplete al siguiente. La lectura del código genético está sujeta a tres reglas que controlan los codones en el ARNm. Primero, los codones se leen en una dirección de 5' a 3'. Segundo, los codones no se superponen y el mensaje no tiene espacios. La última regla, como se indicó anteriormente, es que el mensaje se traduce en un marco de lectura fijo. [1]

Mecanismo
Las mutaciones por desplazamiento del marco de lectura pueden ocurrir de forma aleatoria o ser causadas por un estímulo externo. La detección de mutaciones por desplazamiento del marco de lectura puede ocurrir mediante varios métodos diferentes. Los desplazamientos del marco de lectura son solo un tipo de mutación que puede dar lugar a proteínas incompletas o incorrectas, pero representan un porcentaje significativo de errores en el ADN. En un gen inalterado, los codones (tripletes de nucleótidos) se interpretan secuencialmente, y cada codón codifica un aminoácido específico. Esto se conoce como el marco de lectura estándar. Sin embargo, en los casos de mutaciones por desplazamiento del marco de lectura, se inserta un nucleótido adicional (o más) en la secuencia de ADN, lo que altera el marco de lectura típico y provoca un cambio en la secuencia.
Esta inserción provoca un cambio en el marco de lectura debido a la naturaleza triplete del código genético. Por ejemplo, la adición de una "A" adicional conduce a un cambio de secuencia, lo que desencadena la lectura de un conjunto completamente diferente de codones. Esta desviación en la información genética hace que el ribosoma, que lee el ARNm para la síntesis de proteínas, interprete incorrectamente los datos genéticos. En consecuencia, se genera una serie completamente diferente de aminoácidos, lo que da como resultado la generación de una secuencia de proteína alterada. En la mayoría de los casos, el nuevo marco de lectura da como resultado un encuentro temprano con un codón de terminación, lo que conduce a la formación de una proteína acortada y generalmente inactiva. Esta forma de mutación se denomina codón de terminación temprano o mutación sin sentido.
Genético o ambiental
Se trata de una mutación genética a nivel de bases de nucleótidos. Se investiga continuamente por qué y cómo se producen las mutaciones por desplazamiento del marco de lectura. Se realizó un estudio ambiental, específicamente la producción de mutaciones por desplazamiento del marco de lectura inducidas por UV por ADN polimerasas deficientes en actividad exonucleasa 3′ → 5′. La secuencia normal 5′ GTC GTT TTA CAA 3′ se cambió a GTC GTT T TTA CAA (MIDT) de GTC GTT C TTA CAA (MIDC) para estudiar los desplazamientos del marco de lectura. Las enzimas mutantes de la ADN polimerasa de E. coli pol I Kf y T7 desprovistas de actividad exonucleasa 3′ → 5′ producen revertientes inducidos por UV con mayor frecuencia que sus contrapartes competentes en exonucleasa . Los datos indican que la pérdida de la actividad de corrección aumenta la frecuencia de los desplazamientos del marco de lectura inducidos por UV. [7]
Detección
Fluorescencia
Los efectos de las bases vecinas y la estructura secundaria para detectar la frecuencia de mutaciones por desplazamiento del marco de lectura se han investigado en profundidad utilizando fluorescencia . El ADN marcado con fluorescencia, por medio de análogos de bases, permite estudiar los cambios locales de una secuencia de ADN. [8] Los estudios sobre los efectos de la longitud de la cadena del cebador revelan que se observó una mezcla de equilibrio de cuatro conformaciones de hibridación cuando las bases de la plantilla se extendían formando un bulto, es decir, una estructura flanqueada a ambos lados por ADN dúplex. Por el contrario, se observó una estructura de doble bucle con una conformación de ADN no apilada inusual en su borde descendente cuando las bases extruidas se posicionaron en la unión cebador-plantilla, lo que demuestra que las desalineaciones pueden modificarse por la estructura secundaria del ADN vecino. [9]
Secuenciación
.jpg/440px-Frameshift_deletion_(13062713935).jpg)
La secuenciación de Sanger y la pirosecuenciación son dos métodos que se han utilizado para detectar mutaciones por desplazamiento del marco de lectura; sin embargo, es probable que los datos generados no sean de la más alta calidad. Aun así, se han identificado 1,96 millones de indeles mediante la secuenciación de Sanger que no se superponen con otras bases de datos. Cuando se observa una mutación por desplazamiento del marco de lectura, se compara con la Base de datos de mutaciones del genoma humano (HGMD) para determinar si la mutación tiene un efecto perjudicial. Esto se hace observando cuatro características. En primer lugar, la relación entre el ADN afectado y el conservado, en segundo lugar, la ubicación de la mutación en relación con la transcripción, en tercer lugar, la relación entre los aminoácidos conservados y afectados y, por último, la distancia del indel al final del exón . [10]
La secuenciación paralela masiva es un método más nuevo que se puede utilizar para detectar mutaciones. Con este método, se pueden secuenciar hasta 17 gigabases a la vez, a diferencia de los rangos limitados de la secuenciación Sanger de solo alrededor de 1 kilobase. Hay varias tecnologías disponibles para realizar esta prueba y se está estudiando su uso en aplicaciones clínicas. [11] Al realizar pruebas para diferentes carcinomas, los métodos actuales solo permiten observar un gen a la vez. La secuenciación paralela masiva puede probar una variedad de mutaciones causantes de cáncer a la vez, a diferencia de varias pruebas específicas. [12] Un experimento para determinar la precisión de este método de secuenciación más nuevo probó 21 genes y no tuvo falsos positivos para mutaciones por cambio de marco. [13]
Diagnóstico
Una patente estadounidense (5.958.684) de 1999 de Leeuwen detalla los métodos y reactivos para el diagnóstico de enfermedades causadas por o asociadas con un gen que tiene una mutación somática que da lugar a una mutación por desplazamiento del marco de lectura. Los métodos incluyen proporcionar una muestra de tejido o fluido y realizar un análisis genético para detectar la mutación por desplazamiento del marco de lectura o una proteína de este tipo de mutación. La secuencia de nucleótidos del gen sospechoso se proporciona a partir de secuencias genéticas publicadas o de la clonación y secuenciación del gen sospechoso. A continuación, se predice la secuencia de aminoácidos codificada por el gen. [14] Secuenciación NA: la secuenciación de Sanger o la secuenciación de próxima generación (NGS) se pueden utilizar para secuenciar directamente el ADN e identificar inserciones o deleciones. Reacción en cadena de la polimerasa (PCR): la PCR se puede utilizar para amplificar la región específica que contiene la mutación para su posterior análisis. Amplificación de sonda dependiente de ligación multiplex (MLPA): la MLPA es una técnica utilizada para detectar variaciones en el número de copias y pequeñas inserciones o deleciones. Hibridación genómica comparativa (CGH): la CGH se utiliza para detectar desequilibrios cromosómicos, que pueden incluir grandes inserciones o deleciones.
Frecuencia
A pesar de las reglas que rigen el código genético y los diversos mecanismos presentes en una célula para asegurar la correcta transferencia de información genética durante el proceso de replicación del ADN así como durante la traducción, se producen mutaciones; la mutación por desplazamiento del marco de lectura no es el único tipo. Existen al menos otros dos tipos de mutaciones puntuales reconocidas, específicamente la mutación sin sentido y la mutación sin sentido . [1] Una mutación por desplazamiento del marco de lectura puede cambiar drásticamente la capacidad de codificación (información genética) del mensaje. [1] Las pequeñas inserciones o deleciones (aquellas de menos de 20 pares de bases) constituyen el 24% de las mutaciones que se manifiestan en las enfermedades genéticas actualmente reconocidas. [10]
Se ha descubierto que las mutaciones por desplazamiento del marco de lectura son más comunes en las regiones repetidas del ADN. Una razón para esto es el deslizamiento de la enzima polimerasa en las regiones repetidas, lo que permite que las mutaciones entren en la secuencia . [15] Se pueden realizar experimentos para determinar la frecuencia de la mutación por desplazamiento del marco de lectura añadiendo o eliminando un número preestablecido de nucleótidos. Se han realizado experimentos añadiendo cuatro pares de bases, llamados experimentos +4, pero un equipo de la Universidad Emory observó la diferencia en la frecuencia de la mutación tanto añadiendo como eliminando un par de bases. Se demostró que no había diferencia en la frecuencia entre la adición y la eliminación de un par de bases. Sin embargo, hay una diferencia en el resultado de la proteína. [15]
La enfermedad de Huntington es uno de los nueve trastornos de reiteración de codones causados por mutaciones de expansión de poliglutamina, que incluyen la ataxia espinocerebelosa (SCA) 1, 2, 6, 7 y 3, la atrofia muscular espinobulbar y la dentatorubal-pallidoluysianatrofia. Puede haber un vínculo entre las enfermedades causadas por mutaciones de expansión de poliglutamina y polialanina, ya que el producto del gen SCA3 original que codifica CAG/poliglutaminas se convierte en GCA/polialaninas. Se ha propuesto que el deslizamiento ribosomal durante la traducción de la proteína SCA3 es el mecanismo que resulta en el cambio del marco que codifica poliglutamina al que codifica polialanina. Una eliminación de dinucleótidos o una inserción de un solo nucleótido dentro del tracto de poliglutamina del exón 1 de huntingtina cambiaría el marco codificante de poliglutamina CAG en +1 (+1 cambio de marco) al marco codificante de polialanina GCA e introduciría un nuevo epítopo en el extremo C del exón 1 de Htt (APAAAPAATRPGCG). [16]
Enfermedades
Varias enfermedades tienen mutaciones por desplazamiento del marco de lectura como al menos parte de la causa. Conocer las mutaciones prevalentes también puede ayudar en el diagnóstico de la enfermedad. Actualmente, existen intentos de utilizar las mutaciones por desplazamiento del marco de lectura de forma beneficiosa en el tratamiento de enfermedades, modificando el marco de lectura de los aminoácidos.


Cáncer
Se sabe que las mutaciones por desplazamiento del marco de lectura son un factor en el cáncer colorrectal , así como en otros cánceres con inestabilidad de microsatélites . Como se dijo anteriormente, es más probable que las mutaciones por desplazamiento del marco de lectura se produzcan en una región de secuencia repetida. Cuando la reparación de desajustes del ADN no corrige la adición o eliminación de bases, es más probable que estas mutaciones sean patógenas. Esto puede deberse en parte a que no se le dice al tumor que deje de crecer. Los experimentos en levaduras y bacterias ayudan a mostrar características de los microsatélites que pueden contribuir a una reparación defectuosa de los desajustes del ADN. Estas incluyen la longitud del microsatélite , la composición del material genético y la pureza de las repeticiones. Según los resultados experimentales, los microsatélites más largos tienen una mayor tasa de mutaciones por desplazamiento del marco de lectura. El ADN flanqueante también puede contribuir a las mutaciones por desplazamiento del marco de lectura. [17] En el cáncer de próstata, una mutación por desplazamiento del marco de lectura cambia el marco de lectura abierto (ORF) y evita que se produzca la apoptosis . Esto conduce a un crecimiento descontrolado del tumor . Si bien existen factores ambientales que contribuyen a la progresión del cáncer de próstata , también hay un componente genético. Durante las pruebas de las regiones codificantes para identificar mutaciones, se descubrieron 116 variantes genéticas, incluidas 61 mutaciones por desplazamiento del marco de lectura. [18] Hay más de 500 mutaciones en el cromosoma 17 que parecen desempeñar un papel en el desarrollo del cáncer de mama y de ovario en el gen BRCA1, muchas de las cuales son por desplazamiento del marco de lectura. [19]
Enfermedad de Crohn
La enfermedad de Crohn está asociada con el gen NOD2. La mutación es una inserción de una citosina en la posición 3020. Esto conduce a un codón de terminación prematuro, acortando la proteína que se supone que debe ser transcrita. Cuando la proteína puede formarse normalmente, responde a los liposacáridos bacterianos, donde la mutación 3020insC impide que la proteína responda. [20]
Fibrosis quística
La fibrosis quística (FQ) es una enfermedad basada en mutaciones en el gen regulador de la conductancia transmembrana de la FQ (CFTR). Se han identificado más de 1500 mutaciones, pero no todas causan la enfermedad. [21] La mayoría de los casos de fibrosis quística son resultado de la mutación ∆F508, que elimina el aminoácido completo. Dos mutaciones de cambio de marco son de interés para el diagnóstico de la FQ, CF1213delT y CF1154-insTC. Ambas de estas mutaciones ocurren comúnmente en tándem con al menos otra mutación. Ambas conducen a una pequeña disminución en la función de los pulmones y ocurren en aproximadamente el 1% de los pacientes evaluados. Estas mutaciones se identificaron mediante secuenciación de Sanger. [22]
VIH
El CCR5 es uno de los cofactores de entrada celular asociados con el VIH, que se relaciona con mayor frecuencia con cepas que no inducen sincitios y es más evidente en pacientes con VIH que en pacientes con SIDA. Se ha identificado una deleción de 32 pares de bases en el CCR5 como una mutación que niega la probabilidad de una infección por VIH. Esta región en el marco de lectura abierto ORF contiene una mutación de cambio de marco que conduce a un codón de terminación prematuro. Esto conduce a la pérdida de la función del correceptor del VIH in vitro. El CCR5-1 se considera el tipo salvaje y el CCR5-2 se considera el alelo mutante. Aquellos con una mutación heterocigótica para el CCR5 fueron menos susceptibles al desarrollo del VIH. En un estudio, a pesar de la alta exposición al virus del VIH, no hubo ningún homocigótico para la mutación del CCR5 que diera positivo para el VIH. [3]
Enfermedad de Tay-Sachs
La enfermedad de Tay-Sachs es una enfermedad mortal que afecta al sistema nervioso central. Se encuentra con mayor frecuencia en bebés y niños pequeños. La progresión de la enfermedad comienza en el útero , pero los síntomas no aparecen hasta aproximadamente los 6 meses de edad. No existe cura para la enfermedad. [23] Se sabe que las mutaciones en el gen de la β-hexosaminidasa A (Hex A) afectan la aparición de la enfermedad de Tay-Sachs, con 78 mutaciones descritas de diferentes tipos, 67 de las cuales se sabe que causan enfermedad. La mayoría de las mutaciones observadas (65/78) son sustituciones de una sola base o SNP, 11 deleciones, 1 grande y 10 pequeñas, y 2 inserciones. 8 de las mutaciones observadas son cambio de marco, 6 deleciones y 2 inserciones. Una inserción de 4 pares de bases en el exón 11 se observa en el 80% de los casos de enfermedad de Tay-Sachs en la población judía asquenazí . Las mutaciones por desplazamiento del marco de lectura dan lugar a un codón de terminación temprano, que se sabe que desempeña un papel en la enfermedad en los bebés. La enfermedad de aparición tardía parece estar causada por cuatro mutaciones diferentes, una de las cuales es una deleción de tres pares de bases. [24]
Síndrome de Smith-Magenis
El síndrome de Smith-Magenis (SMS) es un síndrome complejo que implica discapacidades intelectuales, trastornos del sueño, problemas de conducta y una variedad de anomalías craneofaciales, esqueléticas y viscerales. La mayoría de los casos de SMS albergan una deleción común de ~3,5 Mb que abarca el gen inducido por ácido retinoico-1 ( RAI1 ). Otros casos ilustran una variabilidad en el fenotipo SMS que no se había mostrado previamente para la mutación RAI1, incluida la pérdida de audición, conductas autoabusivas y retrasos globales leves. La secuenciación de RAI1 reveló una mutación de un tracto C heptamérico (CCC) en el exón 3 que resultó en mutaciones de cambio de marco. De las siete mutaciones de cambio de marco informadas que ocurren en tractos C poli en RAI1, cuatro casos (~57%) ocurren en este tracto C heptamérico. Los resultados indican que este tracto C heptamérico es un punto de inserción/deleción de recombinación preferencial (SNindels) y, por lo tanto, un objetivo primario para el análisis en pacientes sospechosos de mutaciones en RAI1. [25]
Miocardiopatía hipertrófica
La miocardiopatía hipertrófica es la causa más común de muerte súbita en personas jóvenes, incluidos los atletas entrenados, y está causada por mutaciones en genes que codifican proteínas del sarcómero cardíaco. Las mutaciones en el gen de la troponina C ( TNNC1 ) son una causa genética rara de miocardiopatía hipertrófica. Un estudio reciente ha indicado que una mutación por cambio de marco de lectura (c.363dupG o p.Gln122AlafsX30) en la troponina C fue la causa de la miocardiopatía hipertrófica (y muerte súbita cardíaca) en un varón de 19 años. [26]
Curas
Es poco frecuente encontrar una cura para las enfermedades causadas por mutaciones de cambio de marco de lectura. Se están realizando investigaciones al respecto. Un ejemplo es la inmunodeficiencia primaria (IDP), una enfermedad hereditaria que puede provocar un aumento de las infecciones. Hay 120 genes y 150 mutaciones que desempeñan un papel en las inmunodeficiencias primarias. El tratamiento estándar actual es la terapia génica , pero se trata de un tratamiento muy arriesgado y, a menudo, puede provocar otras enfermedades, como la leucemia. Los procedimientos de terapia génica incluyen la modificación de la proteína de fusión de la nucleasa de franja de zinc, escindiendo ambos extremos de la mutación, lo que a su vez la elimina de la secuencia. La omisión de exón mediada por oligonucleótidos antisentido es otra posibilidad para la distrofia muscular de Duchenne . Este proceso permite pasar por encima de la mutación para que el resto de la secuencia permanezca en marco de lectura y la función de la proteína se mantenga intacta. Sin embargo, esto no cura la enfermedad, solo trata los síntomas y solo es práctico en proteínas estructurales u otros genes repetitivos. Una tercera forma de reparación es el mosaicismo revertiente , que se produce de forma natural mediante la creación de una mutación inversa o una mutación en un segundo sitio que corrige el marco de lectura. Esta reversión puede ocurrir por recombinación intragénica , conversión génica mitótica , deslizamiento del ADN en el segundo sitio o reversión específica del sitio. Esto es posible en varias enfermedades, como la inmunodeficiencia combinada grave ligada al cromosoma X (SCID), el síndrome de Wiskott-Aldrich y el síndrome de Bloom . No existen medicamentos ni otros métodos farmacogenómicos que ayuden con las IDP. [27]
En 2003, Bork presentó una patente europea (EP1369126A1) que registra un método utilizado para la prevención de cánceres y el tratamiento curativo de cánceres y precánceres, como tumores esporádicos con deficiencia en la reparación de desajustes del ADN (MMR) y tumores asociados al HNPCC. La idea es utilizar inmunoterapia con mezclas combinatorias de péptidos derivados de mutaciones por desplazamiento del marco de lectura específicas del tumor para provocar una respuesta de células T citotóxicas dirigida específicamente contra las células tumorales. [28]
Véase también
- Cambio de marco traslacional
- Mutación
- Transcripción (genética)
- Traducción (biología)
- codón
- proteína
- marco de lectura
- mutación puntual
- Enfermedad de Crohn
- Enfermedad de Tay-Sachs
Referencias
- ^ abcdefghij Losick, Richard; Watson, James D.; Baker, Tania A.; Bell, Stephen; Gann, Alexander; Levine, Michael W. (2008). Biología molecular del gen (6.ª ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1.
- ^ "El ADN cambia constantemente a través del proceso de mutación". Nature . Consultado el 17 de mayo de 2019 .
- ^ ab Zimmerman PA, Buckler-White A, Alkhatib G, Spalding T, Kubofcik J, Combadiere C, Weissman D, Cohen O, Rubbert A, Lam G, Vaccarezza M, Kennedy PE, Kumaraswami V, Giorgi JV, Detels R, Hunter J, Chopek M, Berger EA, Fauci AS, Nutman TB, Murphy PM (enero de 1997). "Resistencia hereditaria al VIH-1 conferida por una mutación inactivante en el receptor 5 de quimiocina CC: estudios en poblaciones con fenotipos clínicos contrastantes, antecedentes raciales definidos y riesgo cuantificado". Medicina molecular . 3 (1): 23–36. PMC 2230106 . PMID 9132277.
- ^ Negoro S, Ohki T, Shibata N, Mizuno N, Wakitani Y, Tsurukame J, Matsumoto K, Kawamoto I, Takeo M, Higuchi Y (noviembre de 2005). "Análisis cristalográfico de rayos X de 6-aminohexanoato-dímero hidrolasa: base molecular para el nacimiento de una enzima que degrada el oligómero de nailon". J Biol Chem . 280 (47): 39644–52. doi : 10.1074/jbc.m505946200 . PMID 16162506.
- ^ Zhang, J (agosto de 2004). "La ARN polimerasa II del huésped hace contribuciones mínimas a las mutaciones de cambio de marco retroviral". The Journal of General Virology . 85 (Pt 8): 2389–95. doi : 10.1099/vir.0.80081-0 . PMID 15269381.
- ^ Cox, Michael; Nelson, David R.; Lehninger, Albert L (2008). Principios de bioquímica de Lehninger . San Francisco: WH Freeman. ISBN 978-0-7167-7108-1.
- ^ Sagher, Daphna; Turkington, Edith; Acharya, Sonia; Strauss, Bernard (julio de 1994). "Producción de mutaciones por desplazamiento del marco de lectura inducidas por UV in vitro por ADN polimerasas deficientes en actividad exonucleasa 3' → 5'". Journal of Molecular Biology . 240 (3): 226–242. doi :10.1006/jmbi.1994.1437. PMID 8028006.
- ^ Johnson, Neil P.; Walter A. Baase; Peter H. von Hippel (marzo de 2004). "Dicroísmo circular de baja energía del dinucleótido de 2-aminopurina como sonda de conformación local de ADN y ARN". Proc Natl Acad Sci USA . 101 (10): 3426–31. Bibcode :2004PNAS..101.3426J. doi : 10.1073/pnas.0400591101 . PMC 373478 . PMID 14993592.
- ^ Baase, Walter A.; Davis Jose; Benjamin C. Ponedel; Peter H. von Hippel; Neil P. Johnson (2009). "Modelos de ADN de deleciones por desplazamiento del marco de lectura de trinucleótidos: la formación de bucles y protuberancias en la unión cebador-molde". Nucleic Acids Research . 37 (5): 1682–9. doi :10.1093/nar/gkn1042. PMC 2655659 . PMID 19155277.
- ^ ab Hu, J; Ng, PC (9 de febrero de 2012). "Predicción de los efectos de las indeles con cambio de marco de lectura". Genome Biology . 13 (2): R9. doi : 10.1186/gb-2012-13-2-r9 . PMC 3334572 . PMID 22322200.
- ^ Tucker, Tracy; Marra, Marco; Friedman, Jan M. (2009). "Secuenciación masivamente paralela: el próximo gran avance en la medicina genética". The American Journal of Human Genetics . 85 (2): 142–154. doi :10.1016/j.ajhg.2009.06.022. PMC 2725244 . PMID 19679224.
- ^ Walsh, T.; Casadei, S.; Lee, MK; Pennil, CC; Nord, AS; Thornton, AM; Roeb, W.; Agnew, KJ; Stray, SM; Wickramanayake, A.; Norquist, B.; Pennington, KP; Garcia, RL; King, M.-C.; Swisher, EM (2011). "De la portada: mutaciones en 12 genes para carcinoma hereditario de ovario, trompa de Falopio y peritoneal identificadas mediante secuenciación masiva paralela". Proc Natl Acad Sci USA . 108 (44): 18032–7. Bibcode :2011PNAS..10818032W. doi : 10.1073/pnas.1115052108 . PMC 3207658 . PMID 22006311.
- ^ Walsh, T.; Lee, MK; Casadei, S.; Thornton, AM; Stray, SM; Pennil, C.; Nord, AS; Mandell, JB; Swisher, EM; King, M.-C. (2010). "Detección de mutaciones heredadas para cáncer de mama y ovario mediante captura genómica y secuenciación masiva paralela". Proc Natl Acad Sci USA . 107 (28): 12629–33. Bibcode :2010PNAS..10712629W. doi : 10.1073/pnas.1007983107 . PMC 2906584 . PMID 20616022.
- ^ Patente de EE. UU. 5.958.684 (28 de septiembre de 1999) "Diagnóstico de enfermedades neurodegenerativas" de Leeuwen et al.
- ^ ab Harfe, BD; Jinks-Robertson, S (julio de 1999). "Eliminación de intermediarios de cambio de marco de lectura por proteínas de reparación de desajustes en Saccharomyces cerevisiae". Biología molecular y celular . 19 (7): 4766–73. doi :10.1128/MCB.19.7.4766. PMC 84275 . PMID 10373526.
- ^ Davies, JE; Rubinsztein, DC (2006). "Productos de cambio de marco de polialanina y poliserina en la enfermedad de Huntington". Revista de genética médica . 43 (11): 893–896. doi :10.1136/jmg.2006.044222. PMC 2563184 . PMID 16801344.
- ^ Schmoldt, A; Benthe, HF; Haberland, G (1 de septiembre de 1975). "Metabolismo de la digitoxina por microsomas de hígado de rata". Farmacología bioquímica . 24 (17): 1639–41. doi :10.1016/0006-2952(75)90094-5. hdl : 10033/333424 . PMID 10.
- ^ Xu, XiaoLin; Zhu, KaiChang; Liu, Feng; Wang, Yue; Shen, Jianguo; Jin, Jizhong; Wang, Zhong; Chen, Lin; Li, Jia Dong; Xu, Min (mayo de 2013). "Identificación de mutaciones somáticas en cáncer de próstata humano mediante RNA-Seq". Gen. 519 (2): 343–7. doi :10.1016/j.gene.2013.01.046. PMID 23434521.
- ^ "Genómica del cáncer". Instituto Nacional del Cáncer del Instituto Nacional de Salud. Archivado desde el original el 18 de marzo de 2013. Consultado el 24 de marzo de 2013 .
- ^ Ogura Y, Bonen DK, Inohara N, Nicolae DL, Chen FF, Ramos R, Britton H, Moran T, Karaliuskas R, Duerr RH, Achkar JP, Brant SR, Bayless TM, Kirschner BS, Hanauer SB, Nuñez G, Cho JH (31 de mayo de 2001). "Una mutación de cambio de marco en NOD2 asociada con la susceptibilidad a la enfermedad de Crohn" (PDF) . Naturaleza . 411 (6837): 603–6. Código Bib :2001Natur.411..603O. doi :10.1038/35079114. hdl : 2027.42/62856 . PMID 11385577. S2CID 205017657.
- ^ Farrell PM, Rosenstein BJ, White TB, Accurso FJ, Castellani C, Cutting GR, Durie PR, Legrys VA, Massie J, Parad RB, Rock MJ, Campbell PW (2008). "Directrices para el diagnóstico de la fibrosis quística en recién nacidos hasta adultos mayores: Informe de consenso de la Cystic Fibrosis Foundation". The Journal of Pediatrics . 153 (2): S4–S14. doi :10.1016/j.jpeds.2008.05.005. PMC 2810958 . PMID 18639722.
- ^ Iannuzzi, MC; Stern, RC; Collins, FS; Hon, CT; Hidaka, N; Strong, T; Becker, L; Drumm, ML; White, MB; Gerrard, B (febrero de 1991). "Dos mutaciones de cambio de marco en el gen de la fibrosis quística". American Journal of Human Genetics . 48 (2): 227–31. PMC 1683026 . PMID 1990834.
- ^ "Aprender sobre la enfermedad de Tay-Sachs". Instituto Nacional de Investigación del Genoma Humano . Consultado el 24 de marzo de 2013 .
- ^ Myerowitz, R (1997). "Mutaciones causantes de la enfermedad de Tay-Sachs y polimorfismos neutrales en el gen Hex A". Human Mutation . 9 (3): 195–208. doi : 10.1002/(SICI)1098-1004(1997)9:3<195::AID-HUMU1>3.0.CO;2-7 . PMID 9090523.
- ^ Truong, Hoa T; Dudding, Tracy; Blanchard, Christopher L.; Elsea, Sarah H (2010). "Punto crítico de mutación por cambio de marco de lectura identificado en el síndrome de Smith-Magenis: informe de caso y revisión de la literatura". BMC Medical Genetics . 11 (1): 142. doi : 10.1186/1471-2350-11-142 . PMC 2964533 . PMID 20932317.
- ^ Chung WK, Kitner C, Maron BJ (junio de 2011). "Nueva mutación por cambio de marco de lectura en la troponina C (TNNC1) asociada con miocardiopatía hipertrófica y muerte súbita". Cardiol Young . 21 (3): 345–8. doi :10.1017/S1047951110001927. PMID 21262074. S2CID 46682245.
- ^ Hu, Hailiang; Gatti, Richard A (2008). "Nuevos enfoques para el tratamiento de las inmunodeficiencias primarias: reparación de mutaciones con productos químicos". Current Opinion in Allergy and Clinical Immunology . 8 (6): 540–6. doi :10.1097/ACI.0b013e328314b63b. PMC 2686128 . PMID 18978469.
- ^ Patente europea [1] (10 de diciembre de 2003) "Uso de péptidos derivados de mutaciones por desplazamiento del marco de lectura de la región microsatélite codificante para el tratamiento del cáncer" por Bork et al.
Lectura adicional
- Farabaugh PJ (1996). "Desplazamiento del marco de lectura de la traducción programada". Annu. Rev. Genet . 30 (1): 507–28. doi :10.1146/annurev.genet.30.1.507. PMC 239420 . PMID 8982463.
- Lewis, Ricki (2005). Genética humana: conceptos y aplicaciones (6.ª ed.). Boston, MA: McGraw Hill. Págs. 227-228. ISBN. 978-0-07-111156-0.
- "Enzimas nilonasas". 20 de abril de 2004. Consultado el 2 de junio de 2009 .
Enlaces externos
- Cambio de marco de lectura + mutación en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Base de datos dbSNP del NCBI: "un repositorio central tanto para sustituciones de nucleótidos de una sola base como para polimorfismos cortos de deleción e inserción"
- Wise2: alinea una proteína con una secuencia de ADN, lo que permite cambios de marco e intrones.
- FastY: compara una secuencia de ADN con una base de datos de secuencias de proteínas, lo que permite espacios y cambios de marco
- Path Archivado el 19 de julio de 2011 en Wayback Machine : herramienta que compara dos proteínas con desplazamiento del marco de lectura ( principio de retrotraducción )
- HGMD - Base de datos de mutaciones del genoma humano
